活的非可培养(viable but non-culturable,VBNC)状态是非芽孢菌为了应对包括极端温度、营养缺乏、高渗透压等因素在内的自然环境胁迫而自发形成的一种生存机制[1]。研究表明,VBNC现象普遍存在于微生物领域,至今已经有100多种微生物被证实可以进入VBNC状态[2]。当致病微生物进入VBNC状态时,它们会逐渐失去其可培养性,但仍保留细胞的某些代谢活性[3],在适宜的条件下能够恢复正常生长与致病性[4]。
副溶血弧菌(Vibrio parahaemolyticus)是一种主要存在于海洋环境中的革兰氏阴性菌,最早在1950年日本泉州集体食物中毒事件中分离得到[5]。作为贝类中危害最严重的致病微生物之一,它导致的水产品食品安全问题严重危害人类健康。当处于低温寡营养环境中[6],或遇到加热、冷藏、高渗、发酵、杀菌等常见的食品加工处理方法[7-10],副溶血弧菌都会应激转化为VBNC状态导致漏检,造成食品中极大的安全隐患[11]。常见的微生物检测技术中,平板计数法会因为VBNC状态微生物不能培养而造成假阴性检测结果;而常规PCR检测技术也因为不能够区分细菌的生命状态,造成假阳性结果。因此VBNC细菌检测技术的研究与优化对副溶血弧菌的防控有着重要意义。
对于细菌状态的精准区分与检测始终是VBNC领域的主要研究方向之一,人们将基于不同原理的方法结合起来,实现了更加快速有效的检测效果。目前普遍利用活菌计数方法与可培养菌计数方法相结合,当活菌数显著高于可培养菌数时,二者结果相减即为VBNC菌的数量。一般利用平板涂布计数法就能得到可培养菌数的准确结果,因此VBNC细菌检测的难点主要聚焦于活菌计数方法。近年来不断有报道证明,叠氮溴化丙锭联合实时荧光定量PCR法(propidium monoazide quantitative PCR,PMA-qPCR)在检测活菌方面具有广阔的应用前景[12-13]。TSENG等[14]利用PMA-qPCR方法检测医院空气环境中的耐药性和敏感性鲍曼不动杆菌。GOLPAYEGANI等[15]的报道表明对游泳池中可能存在的铜绿假单胞菌的检测过程中,与常规PCR、qPCR、平板法等相比,PMA-qPCR检测效率更高且更准确。叠氮溴化丙锭(propidium monoazide,PMA)能够穿过破损的细胞质膜进入死细胞内部与DNA形成共价氮碳键[16],细胞外残留的游离PMA光解后被除去。随后进行qPCR扩增时,死细胞的DNA扩增得到有效抑制[17-18]。但PMA对各种细菌的功效或毒性并不一样[19-20],因此关于PMA处理细菌的方法条件,需要根据菌种的不同进行探究与优化,才能保证其检测的准确性和实用性。
本研究以副溶血弧菌为研究对象,在已有研究基础上,进一步优化副溶血弧菌活菌检测的PMA-qPCR方法,提高其准确性与灵敏度,以建立与平板计数法相结合的VBNC状态副溶血弧菌定量检测方法。
1 材料与方法
1.1 菌种、试剂与仪器
1.1.1 菌种
副溶血弧菌ATCC 17802(Vp17802)、VIB461、VIB304,溶藻胶弧菌VIB283,地中海弧菌VIB296,拟态弧菌VIB298,双氮养弧菌VIB290,塔氏弧菌VIB309,哈维氏弧菌VIB410,均为中国海洋大学食品安全实验室保存。
1.1.2 试剂
PMA,美国Biotium公司;SYBR Green Realtime qPCR Master Mix,ToYoBo东洋纺(上海)生物科技有限公司;乙二胺四乙酸二钠(EDTA-2Na)、NaCl,国药集团化学试剂有限公司;LB液体培养基、LB固体培养基,青岛海博生物技术有限公司;Tris、Triton X-100,北京索莱宝科技有限公司。
1.1.3 仪器
1-14K低温高速离心机,德国Sigma公司;Synergy LX全波长酶标仪,美国BioTek公司;ZWY-2102C恒温振荡培养箱,上海智城分析仪器制造有限公司;MLS-3781L-PC高压蒸汽灭菌器,日本松下健康医疗器械株式会社;StepOnePlus Real-Time PCR仪,美国Applied Biosystems公司。
1.2 实验方法
1.2.1 菌液的制备
取-80 ℃下体积分数30%甘油保存的Vp17802菌种活化增殖,37 ℃培养至对数生长期,离心得到菌体沉淀用等体积生理盐水洗涤并复溶。测定菌悬液OD600 nm,根据本实验室已测定的OD值与菌液浓度的线性关系,将浓度调整至约5.0×107 CFU/mL。菌悬液分为2组,一组作为对照组,另一组沸水浴3 min,获得热灭活的死菌悬液为实验组。
平板计数法:将制备好的菌悬液在含30 g/L NaCl的LB固体平板上涂布计数,确定活菌悬液的准确浓度以及死菌悬液是否完全灭活。
1.2.2 细菌基因组的提取
配制DNA提取液:按照20 mmol/L Tris、2 mmol/L EDTA和体积分数0.6% Triton X-100的配比,分别取1.211 4 g Tris、0.336 2 g EDTA-2Na和3 mL Triton X-100,溶于400 mL超纯水,调节pH=8.0后再用超纯水定容至500 mL,灭菌后待用。
取500 μL已测定浓度的菌悬液于1.5 mL离心管中,10 000 r/min、4 ℃低温离心10 min,弃去上清液后,菌体沉淀用ddH2O洗涤1次,再加入等体积DNA提取液轻轻混匀,100 ℃金属浴加热10 min,取出立即冰上低温放置10 min,再4 ℃离心10 min[21],此时溶液中为提取出的DNA,吸取上清液可直接用于qPCR扩增。
1.2.3 引物的合成与验证
参考BEJ等[22]的报道,根据GenBank中已发表的副溶血弧菌特异性tlh基因序列(基因序列号M36437.1)设计引物,委托北京华大基因有限公司合成,引物序列为:上游引物5′-AAAGCGGATTATGCAGAAGCACTG-3′,下游引物5′-GCTACTTTCTAGCATTTTCTCTGC-3′,扩增得到的片段大小约为450 bp。分别提取Vp17802、VIB416、VIB304这3株副溶血弧菌和其余6株弧菌的DNA作为模板进行qPCR,验证上述引物的特异性。
1.2.4 qPCR检测体系的建立
qPCR反应体系共20 μL:DNA模板2 μL,SYBR Green Realtime qPCR Master Mix 10 μL,上下游引物各0.4 μL(终浓度0.2 μmol/L),ddH2O 7.2 μL。
优化后的qPCR反应程序设置:95 ℃预变性1 min,95 ℃变性15 s、60 ℃退火15 s、72 ℃延伸45 s,设置40个循环,同时收集荧光信号。熔解曲线分析:65~95 ℃,每5 s升高0.5 ℃。
1.2.5 PMA的作用条件优化
1.2.5.1 PMA工作液的配制
1 mg PMA完全溶解于200 μL ddH2O,配制成质量浓度为5 g/L的PMA储存液,-20 ℃避光保存。使用前用ddH2O稀释1/20作为工作液。
1.2.5.2 最小PMA作用浓度
取制备好的菌悬液500 μL,分别加入一定体积的PMA工作液使最终质量浓度为0~21 mg/L,混匀后于黑暗处静置孵育10 min,然后平铺于冰上,与650 W卤素灯保持15 cm间距进行10 min强光照射,再根据上述细菌基因组提取方法提取DNA进行qPCR。活菌与死菌做相同处理作为对照,每组设置3个平行。
1.2.5.3 最佳孵育时间
取制备好的菌悬液500 μL,加入PMA混匀后于黑暗处分别静置孵育0~15 min,然后平铺于冰上,后续步骤与1.2.5.2相同。活菌与死菌做相同处理作为对照,每组设置3个平行。
1.2.5.4 最佳光照时间
取制备好的菌悬液500 μL,加入PMA混匀后于黑暗处静置孵育10 min,然后平铺于冰上,与650 W卤素灯保持15 cm间距,分别进行0~20 min的强光照射,根据上述细菌基因组提取方法提取DNA进行qPCR。活菌与死菌做相同处理作为对照,每组设置3个平行。
1.2.6 PMA-qPCR标准曲线的建立
将培养至对数生长期的Vp17802菌液离心得到菌体沉淀,用等体积的生理盐水复溶,调整菌悬液OD600 nm,使菌液浓度保持在109 CFU/mL,取样于含3%NaCl的LB平板涂布计数。用生理盐水将菌悬液依次稀释,得到101、102、103、104、105、106、107、108、109 CFU/mL共9个浓度梯度,取9组菌液各500 μL,按1.2.5优化后的PMA处理条件进行处理,提取DNA模板进行qPCR定量检测,设置3组平行。分析平板涂布和qPCR检测结果的相关性,以扩增循环数(cycle threshold,Ct)和活菌浓度绘制标准曲线。
1.2.7 PMA-qPCR方法检测混合活死副溶血弧菌
培养至对数生长期的Vp17802菌液离心得到菌体沉淀,用等体积的0.85%生理盐水复溶,调整菌悬液OD600 nm,使菌液浓度保持在109 CFU/mL。菌悬液分为2组,一组进行10倍稀释,作为活菌悬液;另一组稀释100倍后沸水浴3 min,获得热灭活的死菌悬液。分别将生理盐水、101、102、103、104、105、106、107、108 CFU/mL活菌与106 CFU/mL死菌等体积混合,得到500 μL混合活死菌液,参照1.2.2提取DNA模板溶液,进行qPCR定量检测。同时对菌液进行平板涂布计数,与PMA-qPCR测定结果进行对比分析,将死菌悬液作为阴性对照,设置3组平行。
2 结果与分析
2.1 菌悬液测定浓度
涂布平板静置于37 ℃恒温过夜,选取平板上单菌落数在30~300内的为有效计数。经计算,活菌悬液浓度为4.70×107 CFU/mL;死菌悬液平板上均无菌落长出,表明沸水浴3 min使细菌完全热灭活,可以用于后续实验。
2.2 qPCR引物特异性验证
将实验室保存的多种弧菌平板活化后,液体增殖至对数生长期,根据本实验室前期测定的OD600 nm与菌液浓度的线性关系,将浓度均调整至108数量级。提取不同弧菌DNA作为模板进行qPCR,对设计合成的tlh基因引物的特异性进行验证。通过qPCR扩增曲线(图1-a)和熔解曲线(图1-b)可以看出,实验所用3株副溶血弧菌均在第15个循环前后开始检测到荧光信号并在88 ℃左右出现单一熔解峰;而其余弧菌在20个循环之后检测到一部分荧光信号,观察熔解曲线发现均为引物二聚体导致。因此该qPCR引物对副溶血弧菌具有高特异性。
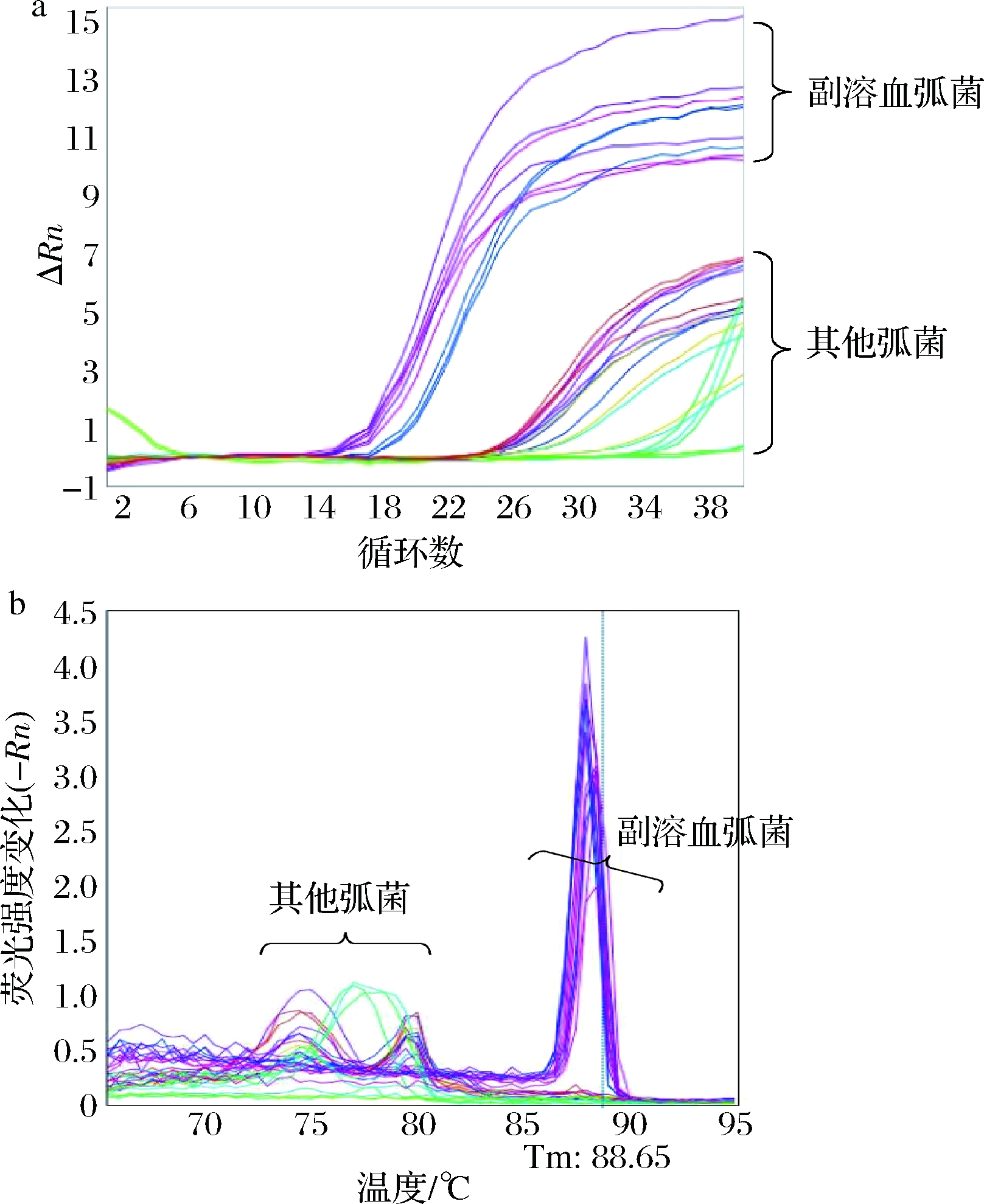
a-扩增曲线;b-熔解曲线
图1 不同弧菌的qPCR扩增曲线与熔解曲线
Fig.1 qPCR amplification plot and melt curve of different Vibrios strains
2.3 PMA处理条件优化结果
2.3.1 PMA最佳添加浓度
添加PMA作预处理时,由图2可知,PMA在质量浓度0~21 mg/L范围内,随着PMA添加量逐渐增加,活菌对照组qPCR扩增的Ct值始终在17左右,无显著性变化,表明ρ(PMA)<21 mg/L时不会影响活菌DNA的扩增。而死菌组qPCR扩增的Ct值逐渐升高,当ρ(PMA)增加至15 mg/L时,与活菌组Ct值结果差异达到最大,表明此时可以达到最大程度上抑制死菌DNA的扩增。当ρ(PMA)为18和21 mg/L时,活菌组与死菌组得到的Ct值结果差值与15 mg/L时无显著差异。因此,选择15 mg/L作为PMA添加的最佳质量浓度。
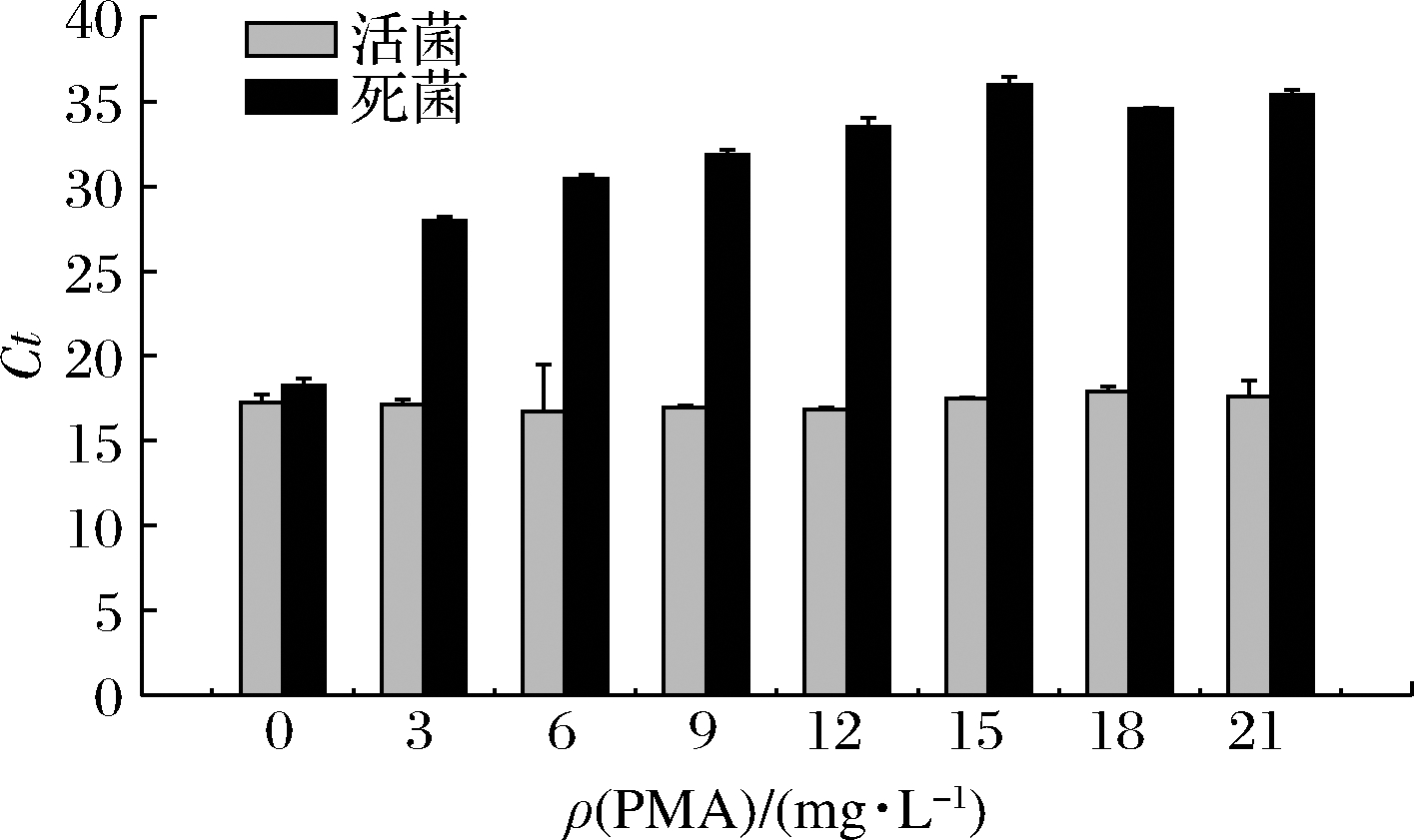
图2 PMA质量浓度对qPCR扩增结果的影响
Fig.2 Effects of PMA concentrations on the amplification results of qPCR
2.3.2 最佳黑暗孵育时间
添加终浓质量度为15 mg/L的PMA后,实验结果(图3)表明,在孵育0~15 min时间范围内,活菌对照组qPCR扩增的Ct值始终无显著性变化,而死菌组DNA的扩增始受到抑制,抑制效果随暗孵育时间而变化。当黑暗环境孵育时间为10 min时,活菌组与死菌组得到的Ct值结果差异达到最大,DNA扩增抑制效果最好。孵育时间多于或少于10 min的抑制效果都相对较弱,说明死菌DNA未被完全抑制扩增。
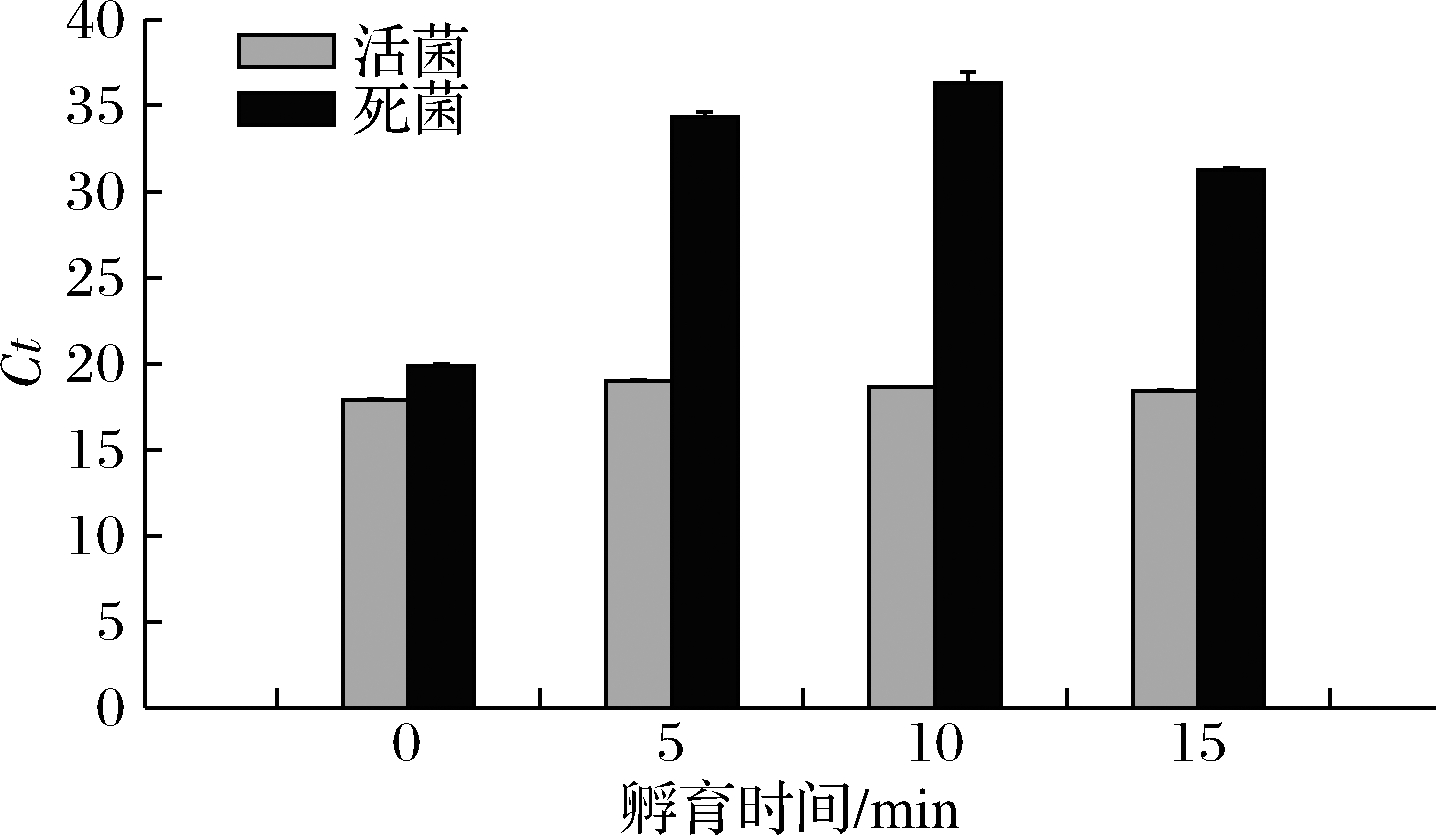
图3 孵育时间对qPCR扩增结果的影响
Fig.3 Effects of incubation time on the amplification results of qPCR
2.3.3 最佳强光照射时间
根据2.3.2得到的最佳暗处理时间,添加15 mg/L的PMA后置于黑暗环境孵育10 min,再平铺于冰上,距离光源15 cm进行照射。实验结果(图4)表明,光照时间在0~20 min范围内,活菌对照组qPCR扩增的Ct值始终无显著性变化,而死菌组DNA的扩增始终受到抑制,抑制效果随光照时间而变化,表现出先增强再减弱的趋势。当强光照射时间为15 min时,活菌组与死菌组得到的Ct值结果差异达到最大,DNA扩增抑制效果最好,光照时间多于或少于15 min,Ct值结果差异性均较小,抑制效果相对较弱说明死菌DNA未被完全抑制。
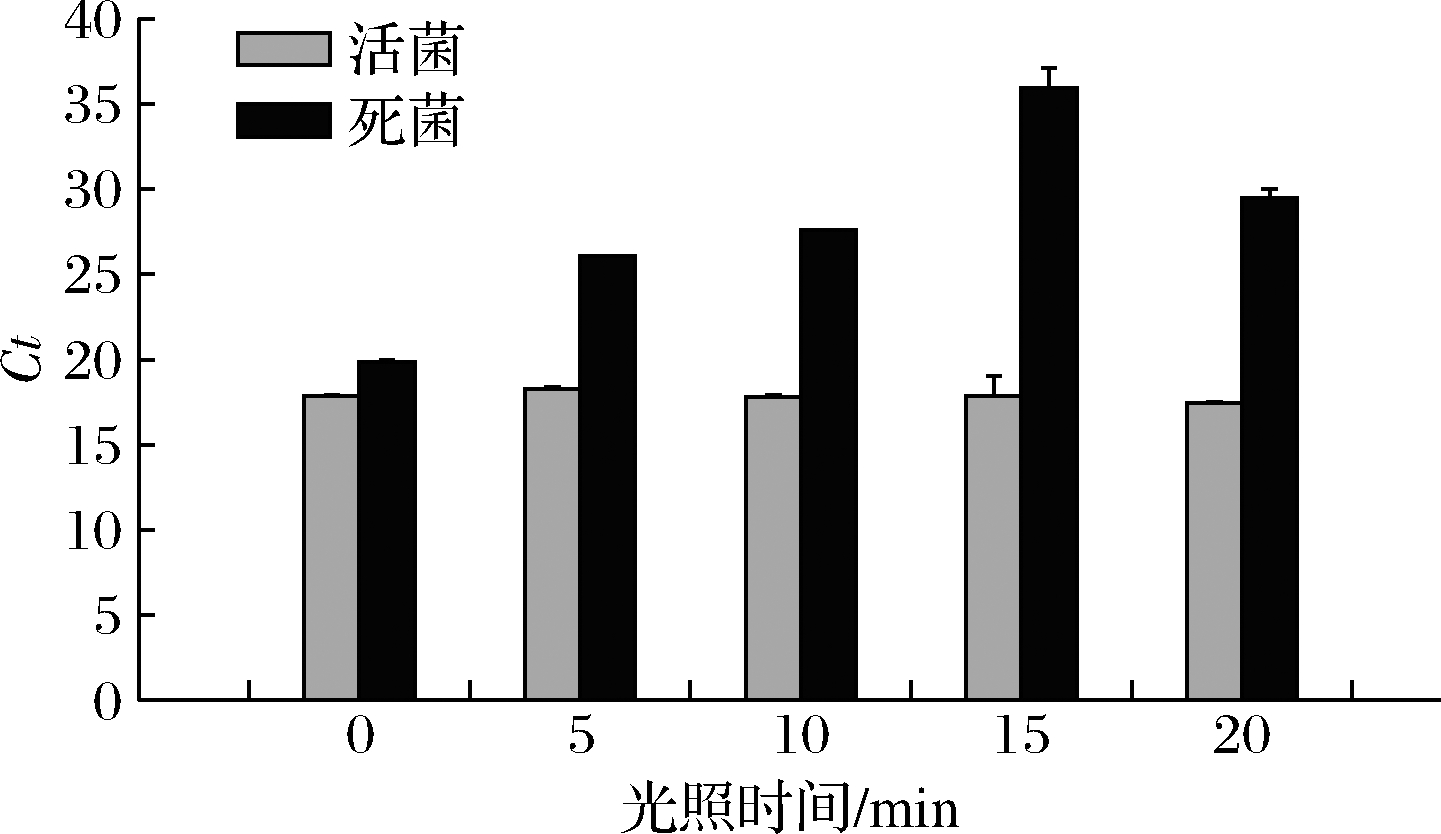
图4 光照时间对qPCR扩增结果的影响
Fig.4 Effects of lighting time on the amplification results of qPCR
2.4 PMA-qPCR法测定活菌数的标准曲线的建立
平板计数方式与2.1相同,最高活菌浓度为2.90×109 CFU/mL。根据上述实验获得的PMA-qPCR最佳处理条件,对9组不同浓度菌液进行PMA-qPCR定量检测,最终检测到qPCR扩增产物的荧光信号时所对应的扩增循环数即是所得到的Ct值。利用IBM SPSS软件对Ct值和平板计数法测得活菌数进行分析统计,结果如表1所示。在2.90×10~2.90×108 CFU/mL,活菌浓度与Ct值的相关系数为0.991,二者高度相关;显著性水平P=0.000<0.01,差异极显著。而当活菌浓度为2.90×109 CFU/mL时,其对应Ct值偏差较大,相关性较差,因此排除该组数据,不参与标准曲线的构建。
表1 实际活菌浓度与qPCR扩增Ct值的相关性分析
Table 1 Correlation analysis between actual viable bacteria concentration and Ct values of qPCR amplification
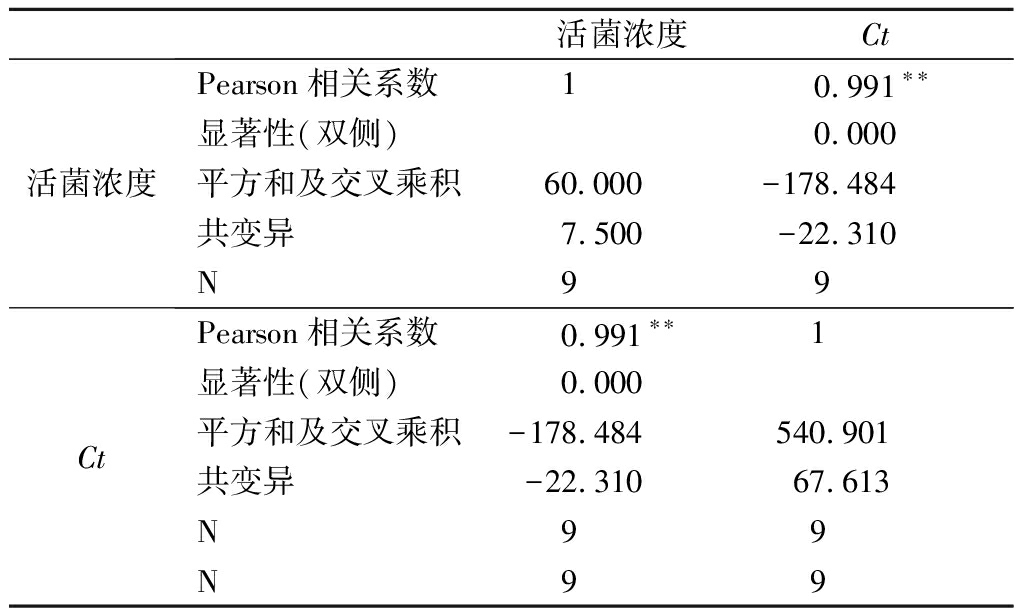
活菌浓度Ct活菌浓度Pearson相关系数 10.991∗∗显著性(双侧)0.000平方和及交叉乘积60.000-178.484共变异 7.500-22.310N 99CtPearson相关系数0.991∗∗ 1显著性(双侧)0.000平方和及交叉乘积-178.484540.901共变异-22.310 67.613N9 9N9 9
注:**,相关性在0.01水平(双侧)上显著
因此得到了在2.90×10~2.90×108 CFU/mL浓度范围内PMA-qPCR定量检测活菌数的标准曲线,其关系式为y=-3.193 4x+41.249,R2=0.992 4,经验证灵敏度与重复性较好,在后续实验中,Ct>36则认为是无效计数。
2.5 PMA-qPCR方法检测混合活死副溶血弧菌
根据平板计数,稀释后活菌悬液最高浓度为7.00×108 CFU/mL,以此作为理论活菌浓度。根据上述实验获得的PMA-qPCR最佳处理条件,对9组不同浓度混合活死菌液进行PMA-qPCR定量检测,再利用标准曲线换算得到实际测定的活菌浓度,利用Excel软件进行分析,结果见表2。通过对比理论与实测活菌浓度,得到各浓度组回收率在92.97%~99.57%,相对标准偏差为1.95%。以上结果表明在不同比例死菌、活菌的混合体系中,利用PMA抑制死菌DNA扩增,结合qPCR反应体系的高特异性,保证了上述PMA-qPCR标准曲线不会受到其他状态细菌的干扰,能够于混合体系中准确测定副溶血弧菌活菌数量,且方法重复性较好,基于培养物水平适用于VBNC副溶血弧菌的进一步探究。
表2 PMA-qPCR测定活菌数与理论值的对比分析
Table 2 Comparative analysis between viable bacteria count determined of PMA-qPCR and theoretical count
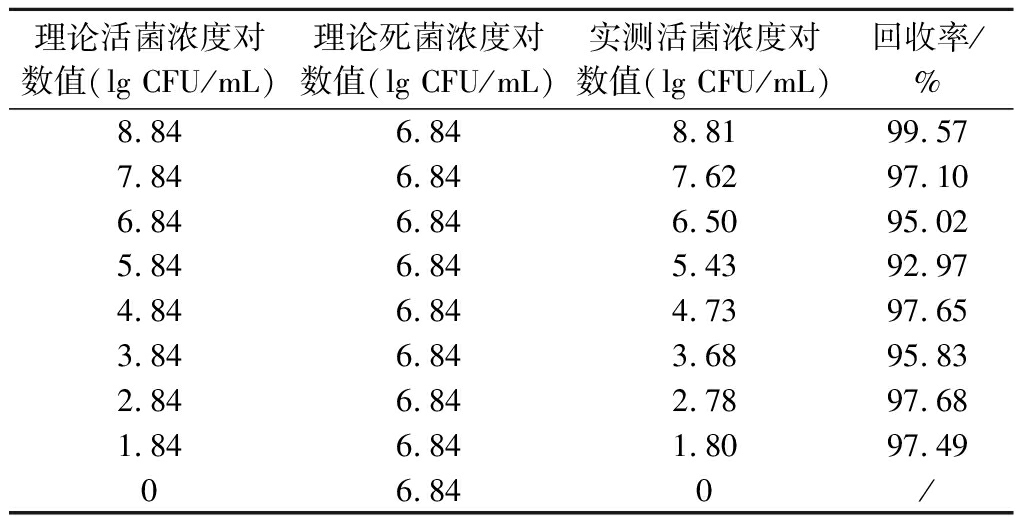
理论活菌浓度对数值(lg CFU/mL)理论死菌浓度对数值(lg CFU/mL)实测活菌浓度对数值(lg CFU/mL)回收率/%8.846.848.8199.577.846.847.6297.106.846.846.5095.025.846.845.4392.974.846.844.7397.653.846.843.6895.832.846.842.7897.681.846.841.8097.4906.840/
注:“/”表示无法计算
3 结论
PMA-qPCR具有高度特异性,不仅可以避免平板检测的漏检问题,也能有效排除普通PCR等核酸技术中受到死细菌信号的局限性,已成为近几年来区分细菌状态、定量活菌数量的重要技术之一[23-24],但PMA-qPCR对于副溶血弧菌活菌计数方面的应用报道较少[25]。利用改良基因组提取方法和副溶血弧菌特异性qPCR扩增反应体系,对PMA处理副溶血弧菌细胞的最佳条件进行探究,当添加质量浓度为15~21 mg/L时,能够基本抑制死菌的DNA扩增,且对活菌无影响。本研究实现了定量检测副溶血弧菌活菌的PMA-qPCR方法优化,检测限降低至101数量级且Ct值保持在更加合理有效的范围内。PMA-qPCR标准曲线扩增效率较高,在2.90×101~2.90×108 CFU/mL浓度范围内成立且合理,结合平板计数法建立了更加精准的VBNC副溶血弧菌定量检测方法。通过验证,不同状态菌体的存在不会干扰检测,但食品基质是否会干扰检测的灵敏度仍需进一步探究。目前该方法已成功应用于本课题组基于纯培养物对VBNC状态副溶血弧菌的机制研究,提供了可靠的技术手段。未来将通过人工模拟食品体系中存在VBNC副溶血弧菌,对该方法进行实际应用探究。
[1] PIENAAR J A,SINGH A,BARNARD T G.The viable but non-culturable state in pathogenic Escherichia coli:A general review[J].African Journal of Laboratory Medicine,2016,5(1):1-9.
[2] DONG K,PAN H X,YANG D,et al.Induction,detection,formation,and resuscitation of viable but non-culturable state microorganisms[J].Comprehensive Reviews in Food Science and Food Safety,2020,19(1):149-183.
[3] XU H S,ROBERTS N,SINGLETON F L,et al.Survival and viability of nonculturable Escherichia coli and Vibrio cholerae in the estuarine and marine environment[J].Microbial Ecology,1982,8(4):313-323.
[4] RAZAFIMAHEFA R M,LUDWIG-BEGALL L F,LE GUYADER F S,et al.Optimisation of a PMAxxTM-RT-qPCR assay and the preceding extraction method to selectively detect infectious murine norovirus particles in mussels[J].Food and Environmental Virology,2021,13(1):93-106.
[5] DANIELS N A,RAY B,EASTON A,et al.Emergence of a new Vibrio parahaemolyticus serotype in raw oysters:A prevention quandary[J].JAMA,2000,284(12):1 541-1 545.
[6] YOON J H,LEE S Y.Characteristics of viable-but-nonculturable Vibrio parahaemolyticus induced by nutrient-deficiency at cold temperature[J].Critical Reviews in Food Science and Nutrition,2020,60(8):1 302-1 320.
[7] 张蕊蕊,聂新颖,廖红梅,等.热辅助超声波处理中VBNC态鼠伤寒沙门氏菌产生及其机制[J].中国食品学报,2020,20(5):12-18.
ZHANG R R,NIE X Y,LIAO H M,et al.Occurrence of viable but non-culturable state in Salmonella enterica serovar typhimurium during thermosonication and its mechanism[J].Journal of Chinese Institute of Food Science and Technology,2020,20(5):12-18.
[8] WASFI R,ABDELLATIF G R,ELSHISHTAWY H M,et al.First-time characterization of viable but non-culturable Proteus mirabilis:induction and resuscitation[J].Journal of Cellular and Molecular Medicine,2020,24(5):2 791-2 801.
[9] 田聪. 两种食源性致病菌VBNC状态诱导与分子检测技术研究[D].广州:华南理工大学,2013.
TIAN C.Induction and molecular detecting techniques research on VBNC of two foodborne pathogenic bacteria[D].Guangzhou:South China University of Technology,2013.
[10] 黄新新, 杨娟,郑江,等.PMA-qPCR法检测冷冻基质中非可培养状态(VBNC)副溶血性弧菌[J].微生物学通报,2018,45(4):934-940.
HUANG X X,YANG J,ZHENG J,et al.Propidium monoazide based real-time PCR to detect Vibrio parahaemolyticus in a viable but nonculturable state[J].Microbiology China,2018,45(4):934-940.
[11] 谷立慧, 钟青萍,方祥,等.“活的非可培养态”食源致病细菌的研究进展[J].食品与发酵工业,2016,42(2):270-274.
GU L H,ZHONG Q P,FANG X,et al.Research progress on the viable but non-culturable state of food-borne pathogenic bacteria[J].Food and Fermentation Industries,2016,42(2):270-274.
[12] NOCKER A,CHEUNG C Y,CAMPER A K.Comparison of propidium monoazide with ethidium monoazide for differentiation of live vs.dead bacteria by selective removal of DNA from dead cells[J].Journal of Microbiological Methods,2006,67(2):310-320.
[13] 肖莉莉, 张昭寰,娄阳,等.叠氮溴化丙锭(PMA)在食源性致病菌检测中的应用[J].中国食品学报,2016,16(6):187-194.
XIAO L L,ZHANG Z H,LOU Y,et al.Application of propidium monoazide(PMA) for detection of foodborne pathogens[J].Journal of Chinese Institute of Food Science and Technology,2016,16(6):187-194.
[14] TSENG C C,HSIAO P K,CHANG K C,et al.Detection of viable antibiotic-resistant/sensitive Acinetobacter baumannii in indoor air by propidium monoazide quantitative polymerase chain reaction[J].Indoor Air,2015,25(5):475-487.
[15] GOLPAYEGANI A,DOURAGHI M,REZAEI F,et al.Propidium monoazide-quantitative polymerase chain reaction(PMA-qPCR) assay for rapid detection of viable and viable but non-culturable(VBNC) Pseudomonas aeruginosa in swimming pools[J].Journal of Environmental Health Science and Engineering,2019,17(1):407-416.
[16] KRALIK P,NOCKER A,PAVLIK I.Mycobacterium avium subsp. paratuberculosis viability determination using F57 quantitative PCR in combination with propidium monoazide treatment[J].International Journal of Food Microbiology,2010,141(S1):S80-S86.
[17] VAN FRANKENHUYZEN J K,TREVORS J T,LEE H,et al.Molecular pathogen detection in biosolids with a focus on quantitative PCR using propidium monoazide for viable cell enumeration[J].Journal of Microbiological Methods,2011,87(3):263-272.
[18] 陈卓君, 魏铭,林果,等.PMA-qPCR技术在发酵食品活菌计数中的应用[J].食品与发酵工业,2019,45(12):242-248.
CHEN Z J,WEI M,LIN G,et al.Application of PMA-qPCR in enumerating living microbes in fermented foods[J].Food and Fermentation Industries,2019,45(12):242-248.
[19] NOCKER A,SOSSA-FERNANDEZ P,BURR M D,et al.Use of propidium monoazide for live/dead distinction in microbial ecology[J].Applied and Environmental Microbiology,2007,73(16):5 111-5 117.
[20] YANEZ M A,NOCKER A,SORIA-SORIA E,et al.Quantification of viable Legionella pneumophila cells using propidium monoazide combined with quantitative PCR[J].Journal of Microbiological Methods,2011,85(2):124-130.
[21] ZHAO X H,WANG J,FORGHANI F,et al.Rapid detection of viable Escherichia coli O157 by coupling propidium monoazide with loop-mediated isothermal amplification[J].Journal of Microbiology and Biotechnology,2013,23(12):1 708-1 716.
[22] BEJ A K,PATTERSON D P,BRASHER C W,et al.Detection of total and hemolysin-producing Vibrio parahaemolyticus in shellfish using multiplex PCR amplification of tl,tdh and trh[J].Journal of Microbiological Methods,1999,36(3):215-225.
[23] BAE S,WUERTZ S.Rapid decay of host-specific fecal Bacteroidales cells in seawater as measured by quantitative PCR with propidium monoazide[J].Water Research,2009,43(19):4 850-4 859.
[24] NOCKER A,SOSSA K E,CAMPER A K.Molecular monitoring of disinfection efficacy using propidium monoazide in combination with quantitative PCR[J].Journal of Microbiological Methods,2007,70(2):252-260.
[25] LIU Y F,ZHONG Q P,WANG J,et al.Enumeration of Vibrio parahaemolyticus in VBNC state by PMA-combined real-time quantitative PCR coupled with confirmation of respiratory activity[J].Food Control,2018,91:85-91.