白酒作为我国的国粹,拥有众多的种类和品系,如清香型白酒、浓香型白酒、酱香型白酒和兼香型白酒等[1],其中酱香型白酒酿造工艺最为复杂[2],导致不同企业生产的产品风味及品质存在较大差异。在酱香型白酒生产过程中,高温大曲是其发酵重要的糖化酶和微生物来源[3],大曲质量在很大程度上决定着最终产品的品质。高温大曲在制作时,由于不同曲块在车间堆放发酵时所处的位置不同,各个曲块发酵时的微环境如温度、湿度、氧浓度等均存在一定差异,导致最终成品曲块的颜色有所不同。具体而言,如果发酵前期温度低,发酵后期干燥差,会生产出白色大曲;发酵前期温度适宜,发酵后期干燥充分,会生产出黄色大曲;发酵前期升温快,发酵后期干燥差,则会产生黑色曲块(较为少见)[4]。已有的研究表明不同颜色的高温大曲微生物群落结构及核心微生物类群等存在明显差异[4]。
茅台酒作为中国公认的高品质酱香型白酒,其风味浓郁醇厚、回味绵长,几乎代表了酱香型白酒制作的最高工艺。GAN等[5]研究发现,放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)是茅台高温大曲中的优势细菌门,同时不同颜色高温大曲菌群群落结构存在较大差异。以茅台酱香型高温大曲为标尺,在对中小型酱香型白酒企业高温大曲细菌类群进行测序的基础上,采用生物信息学和多元统计学手段对两者菌群群落结构和功能差异进行甄别,并以此为靶点在后续大曲制作过程中进行工艺改良对于中小型酱香型白酒生产企业产品品质的提升具有重要意义。
鉴于此,本研究将国际数据库中湖北省尧治河楚翁泉酒业有限公司(下简称尧治河)[6]和茅台高温大曲群落结构的细菌测序数据[5]进行了下载和归并分析,从菌群群落结构、功能以及细菌表型等多个维度全面探究了两者的共性和差异,通过本研究的开展以期为襄阳乃至湖北地区高温大曲制作工艺的改良提供理论依据和有益指导。
1 材料与方法
1.1 测序数据的获取
按照GAN等[5]的描述,本研究从CNGB国家基因库(https://db.cngb.org/cnsa/,编号:CNP0000-316)下载了茅台白色高温大曲(白曲)和黄色高温大曲(黄曲)测序数据各26份,同时整合了本研究团队前期采集自湖北省尧治河楚翁泉酒业有限公司的白曲和黄曲测序数据各10份(https://www.mg-rast.org/,编号∶mgp96594)[6],共计72份测序数据。
1.2 生物信息学分析
使用QIIME(V1.9.1)平台对测序数据进行细菌群落结构和多样性的分析,具体生物学分析过程包括:1)采用PyNAST软件对测序数据进行比对和校准[7];2)使用QIIME中的pick_closed_reference_otus.py命令结合Greengene数据库在97%的相似度对校准后的序列进行操作分类单元(operational taxonomic units,OTU)划分和注释[8-9];3)使用vsearch软件对属于嵌合体的OTU进行识别并去除[10];4)通过统计各OTU包含的序列数及对应的注释信息计算各样本中细菌在门和属水平的相对含量;5)基于各样品在属水平的相对含量分别基于Bray-Curtis距离和Binary Jaccard距离进行β多样性分析;6)通过计算样本相同测序深度下的α多样性指数(Chao1指数、Shannon指数、Observed species指数和Simpson指数)评估样品中菌群的丰度和多样性,具体操作为以测序量最低的样品为基准,各样品随机抽取相同的序列数,并计算上述4个α多样性指标,重复该过程1 000次,取各指标1 000次计算得到的平均值作为该样品最终的α多样性指数值;7)应用生物信息学开源软件PICRUSt对大曲菌群的功能进行预测[11],得到各样品直系同源基因簇(clusters of orthologous groups,COG)组成;8)使用BugBase工具(https://bugbase.cs.umn.edu/)对各酒曲样品细菌表型进行分类统计和可视化。
1.3 多元统计学分析
使用非参数的Mann-Whitney秩和检验对两组样品进行差异显著性检验,使用Benjaminiand Hochberg的方法校正多次检验的假阳性率[12]。使用置换多元方差分析(permutational multivariate analysis of variance,PERMANOVA)检验影响酒曲中菌群组成的关键因素。使用Spearman Rank 相关性分析确定茅台和尧治河酒曲中菌群彼此间的相关关系性。所有分析在R语言(V3.2.5)和STAMP(V2.1.3)软件中实现。
2 结果与分析
2.1 高温大曲中细菌菌群结构的整体组成
在高温大曲中共鉴定到23个细菌门和347个细菌属,其中优势细菌门(平均相对含量 >1%)主要包括厚壁菌门(82.32%)、放线菌门(7.72%)和变形菌门(5.80%)(图1-a);优势细菌属(平均相对含量 >1%)共有11个,主要包括芽胞杆菌属(Bacillus,23.12%)、嗜热放线菌属(Thermoactinomyces,19.55%)、葡萄球菌属(Staphylococcus,6.43%)、Kroppenstedtia(5.51%)、乳杆菌属(Lactobacillus,4.41%)和糖多孢菌属(Saccharopolyspora,4.26%)等。此外,各样品平均约24.12%的菌群无法鉴定到属水平。
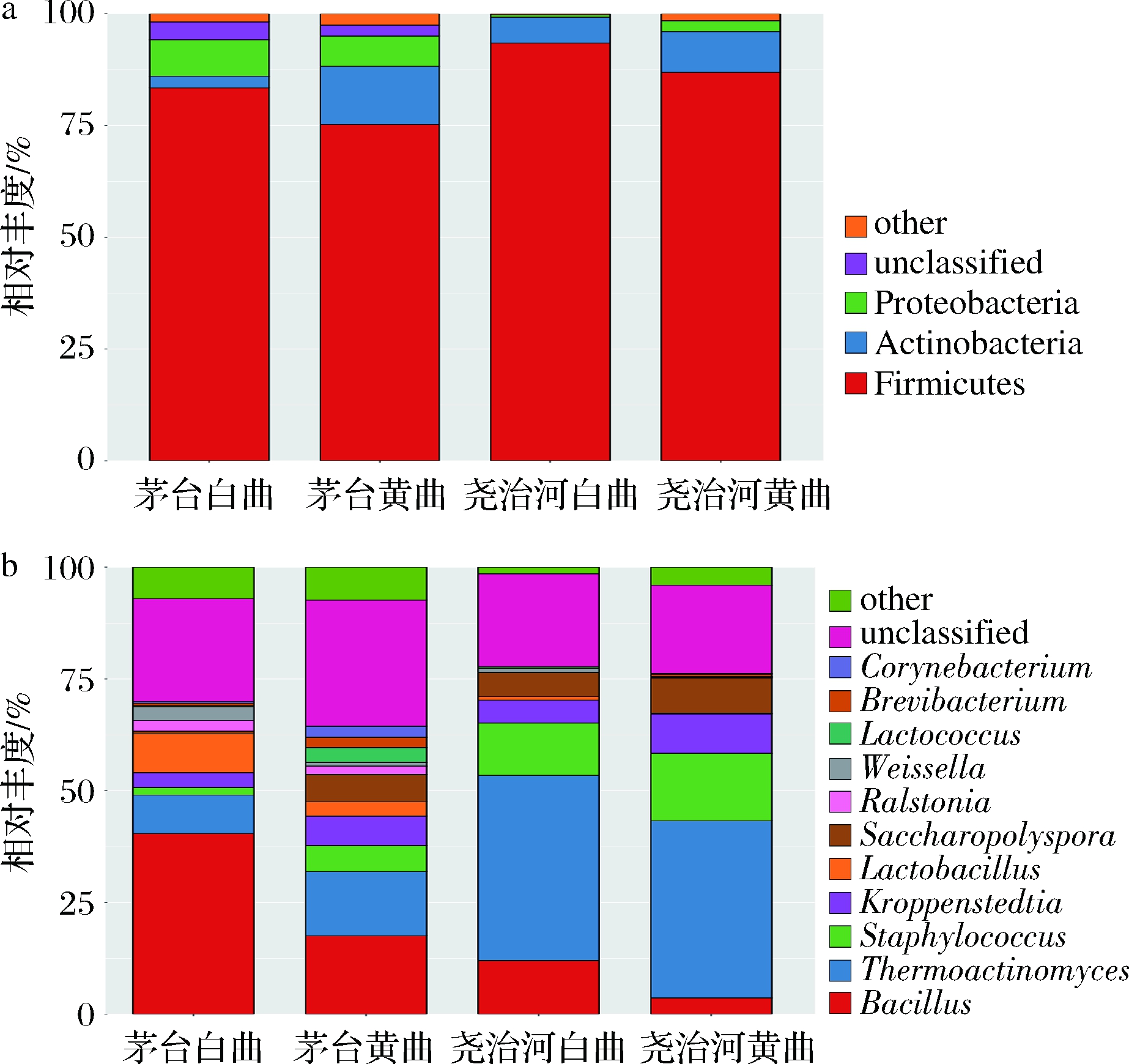
a-门水平;b-属水平
图1 不同地区和颜色高温大曲在门和属水平的菌群组成
Fig.1 Bacterial composition of high-temperature Daqu in different regions and colors at phylum and genus level
2.2 高温大曲核心菌群分析
本研究将在不同地区和不同颜色高温大曲中均出现的OTU定义为高温大曲的“核心OTU”,结果显示共有180个OTU满足该条件(图2)。这些OTU主要注释结果为嗜热放线菌属(45个)、芽胞杆菌属(34个)、Kroppenstedtia(12个)、乳杆菌属(9个)和葡萄球菌属(9个)等。“核心OTU”注释结果与酒曲中优势菌属的分析结果一致。值得注意的是,核心OTU中平均分别包含有尧治河大曲75.52%(22.33%~97.55%)的序列,而平均仅包含有茅台大曲中3.60%(0.38%~7.53%)的序列。同时,来自同一地区不同颜色的高温大曲亦拥有大量共有OTU,如在茅台和尧治河不同颜色高温大曲分别共有1 726和1 484个OTU。其中茅台黄曲和白曲共有OTU主要注释结果为芽孢杆菌属(8.86%)、嗜热放线菌属(4.52%)、乳杆菌属(4.40%)和Kroppenstedtia (2.14%)等。尧治河黄曲和白曲共有OTU主要注释结果为嗜热放线菌属(29.99%)、糖多孢菌属(10.85%)、芽孢杆菌属(4.18%)和Kroppenstedtia(4.11%)等。
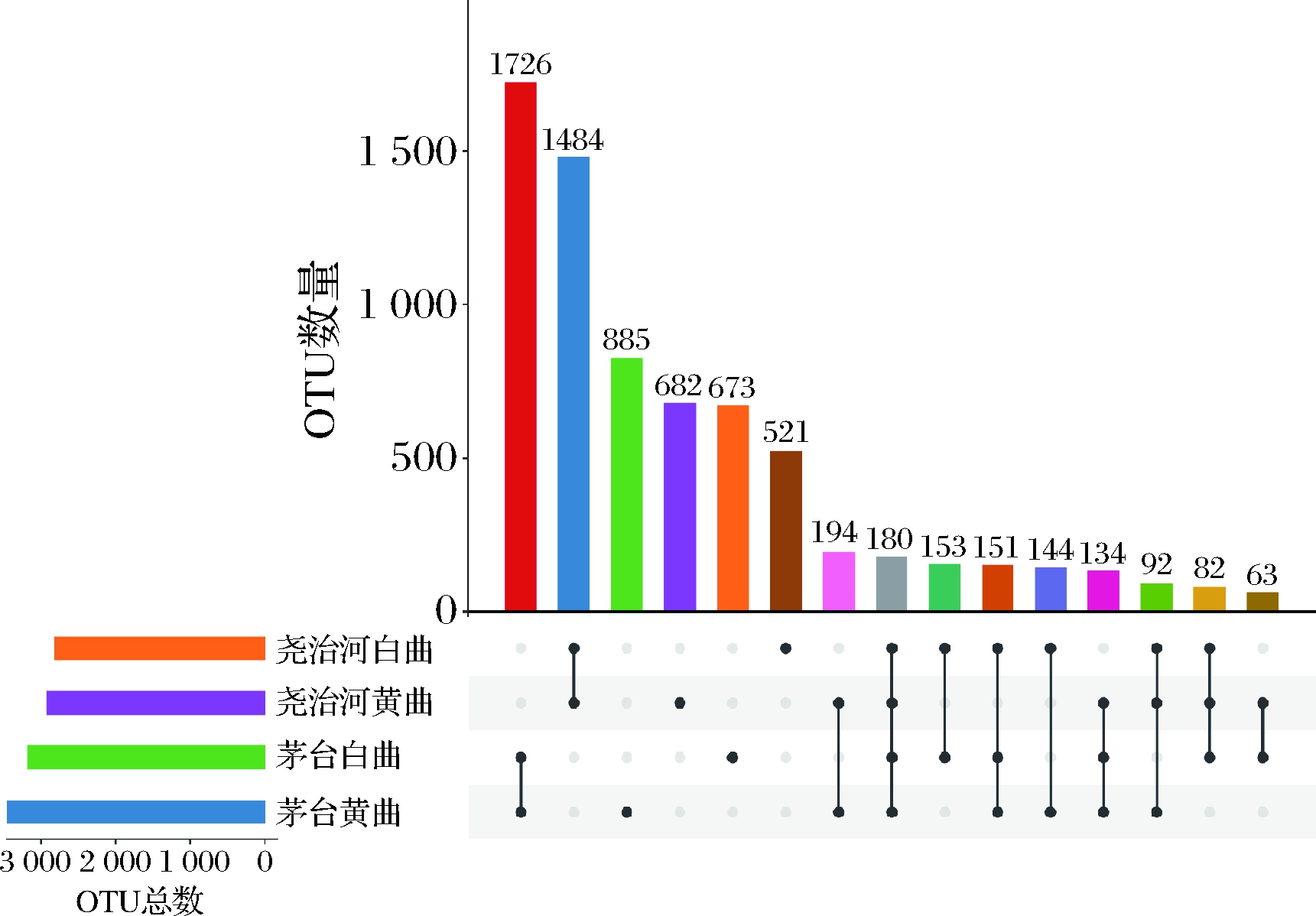
图2 不同地区和颜色高温大曲共有OTU分析
Fig.2 OTUs shared by high-temperature Daqu in different regions and colors
此外,茅台和尧治河不同颜色大曲共有OTU中分别有58.40%和34.30%无法鉴定到属水平。上述结果显示2个地区不同颜色高温大曲共有菌群存在较大差异,同时茅台大曲中存在较大比例的独有菌群,对于大曲中在属水平未能得到有效注释的菌群需要后续研究进一步挖掘和解析。
2.3 不同地区和颜色高温大曲菌群α多样性分析
α多样性指标可以反映不同样品中菌群的丰度和多样性差异。本研究分别使用Chao1指数、发现的OTU数及Shannon指数、Simpson指数表征高温大曲中菌群的丰度和多样性信息,结果如图3所示。各个指标的结果均显示尧治河高温大曲菌群的丰度和多样性显著低于茅台高温大曲(P<0.05)。同时,与黄曲相比,白曲中菌群的丰度和多样性会更高,但同一地区两种颜色大曲菌群α多样性差异不显著(P>0.05)。
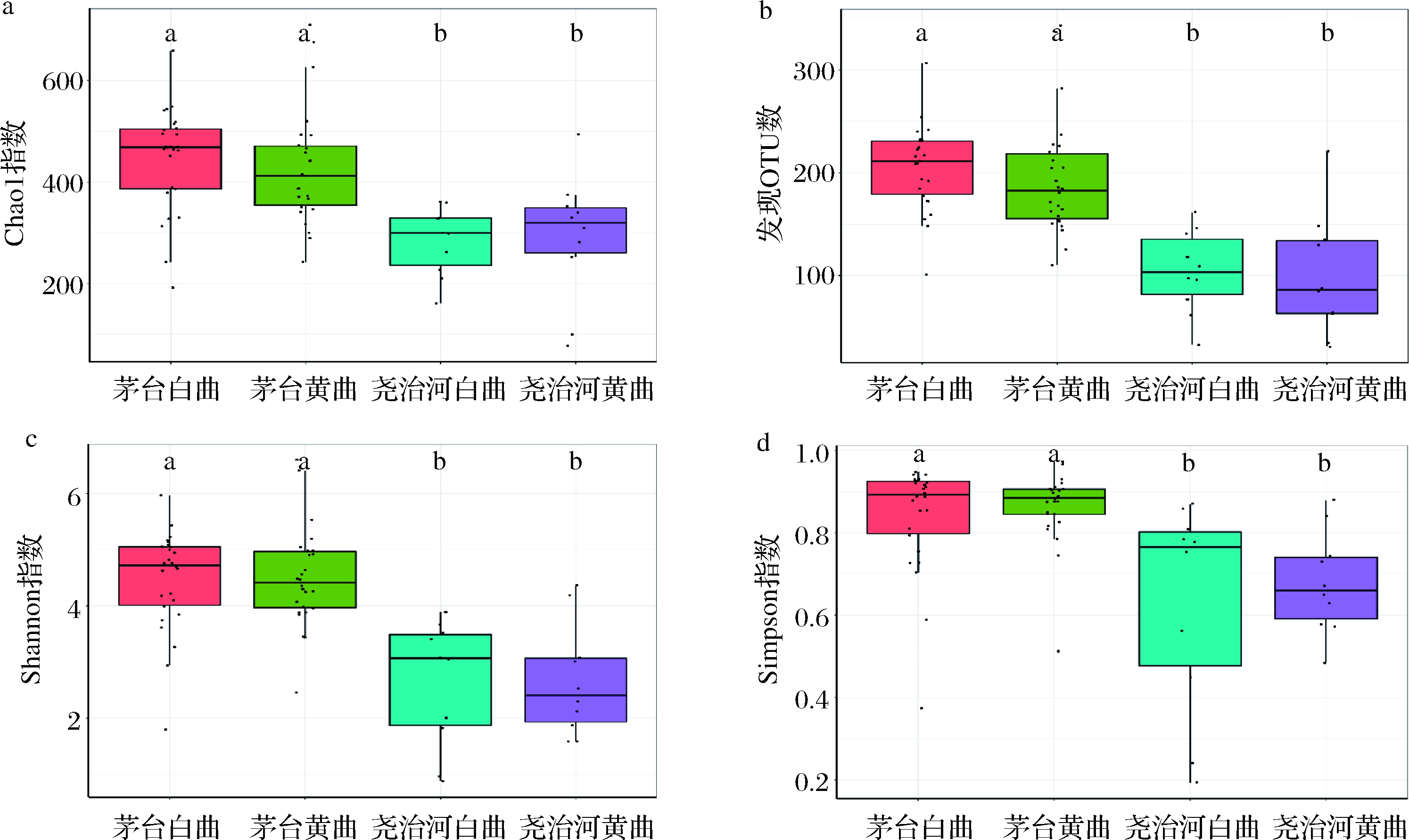
a-Chao1指数;b-发现的OTU数;c-Shannon指数;d-Simpson指数
图3 不同地区和颜色高温大曲菌群α多样性分析
Fig.3 α-Diversity analysis of high-temperature Daqu in different regions and colors
2.4 不同地区和颜色高温大曲菌群β多样性分析
为了直观的展现不同地区和不同颜色高温大曲菌群群落结构异同,本研究分别基于Binary Jaccard距离和Bray-Curtis距离进行了主坐标分析,结果如图4所示。基于Binary Jaccard距离的主坐标分析结果显示(图4-a),2个地区的样品间存在明显的分离趋势,而同一地区不同颜色的大曲样品间则存在明显的重叠。基于Bray-Curtis距离的主坐标分析结果显示(图4-b),茅台白曲与尧治河高温大曲间存在明显分离趋势,而茅台黄曲与尧治河高温大曲存在一定重叠现象。此外,基于上述两种距离的置换多元方差分析结果均表明,上述4组样品菌群群落结构差异具有显著性(P=0.001),并且地区对酒曲菌群的影响大于曲块颜色。
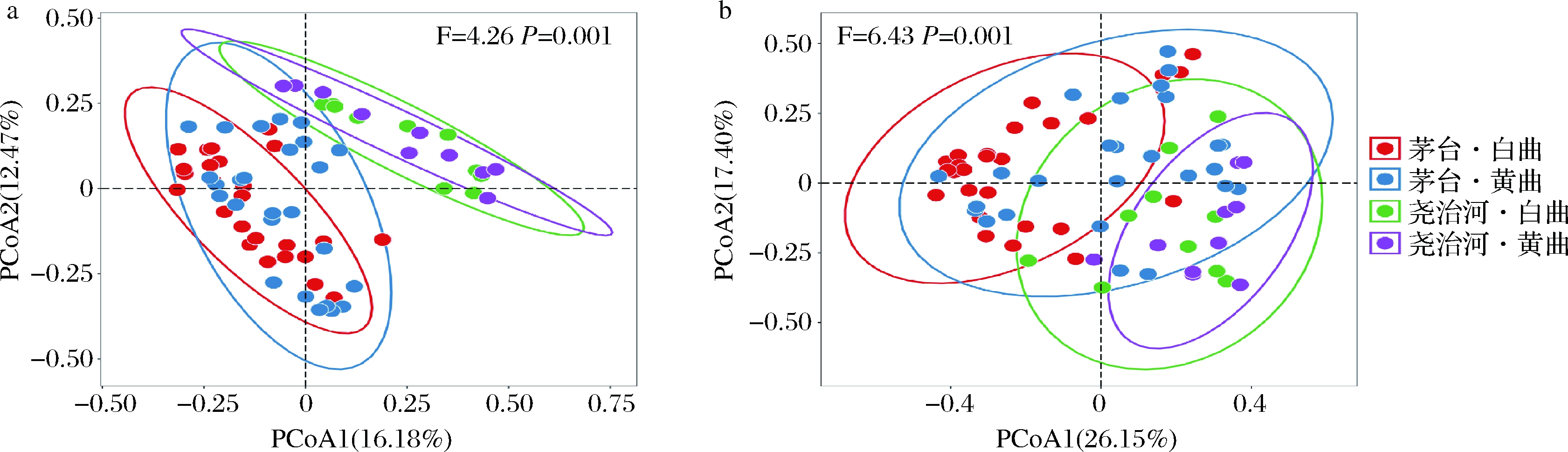
a-Binary Jaccard距离;b-Bray-Curtis距离
图4 基于Binary Jaccard距离和Bray-Curtis距离的主坐标分析
Fig.4 Principal coordinate analysis based on Binary Jaccard distance and Bray-Curtis distance
2.5 不同地区和颜色高温大曲菌群差异比较
为比较不同颜色高温大曲菌群群落结构的差异,分别使用Mann-Whitney秩和检验比较了同一地区不同颜色大曲菌群在属水平的差异,结果如表1所示。
由表1可知,2个地区白曲中芽胞杆菌属、乳杆菌属和魏斯氏菌属(Weissella)的相对含量均显著高于黄曲(P<0.05)。此外,在茅台白曲中,明串珠菌属(Leuconostoc)和溶杆菌属(Lysobacter)的相对含量显著高于黄曲(P<0.05),而糖多孢菌属相对含量显著低于黄曲(P<0.05)。为了分析不同地区高温大群菌落结构差异,本研究进一步对2个地区相同颜色的大曲菌群在属水平进行了Mann-Whitney秩和检验,结果如表2所示。
表1 相同地区不同颜色大曲间相对含量存在显著差异的菌属
Table 1 Bacterial genera with significant differences in relative abundance between Daqu of the same region and different colors
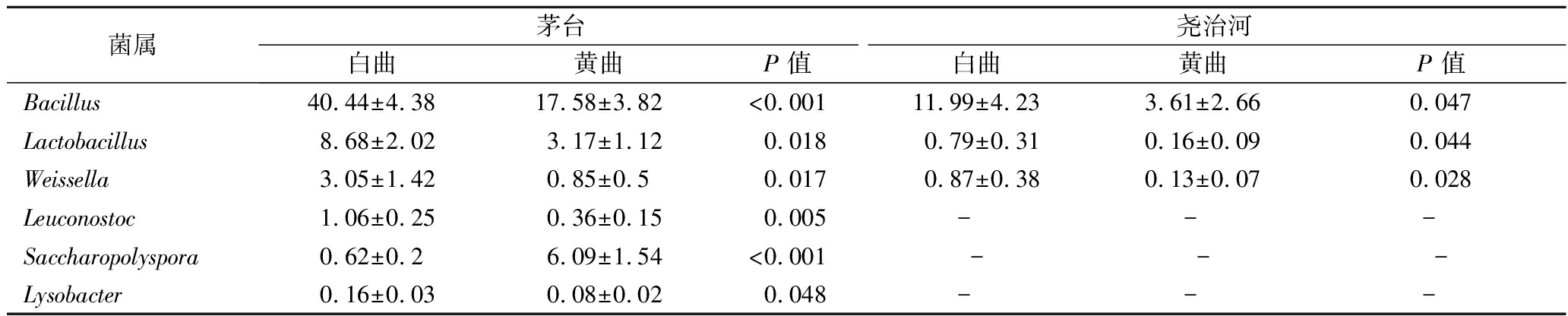
菌属茅台尧治河白曲黄曲P 值白曲黄曲P 值Bacillus40.44±4.3817.58±3.82<0.00111.99±4.233.61±2.660.047Lactobacillus8.68±2.023.17±1.120.0180.79±0.310.16±0.090.044Weissella3.05±1.420.85±0.50.0170.87±0.380.13±0.070.028Leuconostoc1.06±0.250.36±0.150.005- - - Saccharopolyspora0.62±0.26.09±1.54<0.001---Lysobacter0.16±0.030.08±0.020.048- - -
注∶“-”代表相应菌属在特定组之间不存在显著差异(下同)
表2 相同颜色不同地区大曲间相对含量存在显著差异的菌属
Table 2 Bacterial genera with significant differences in relative abundance between Daqu of same color and different regions
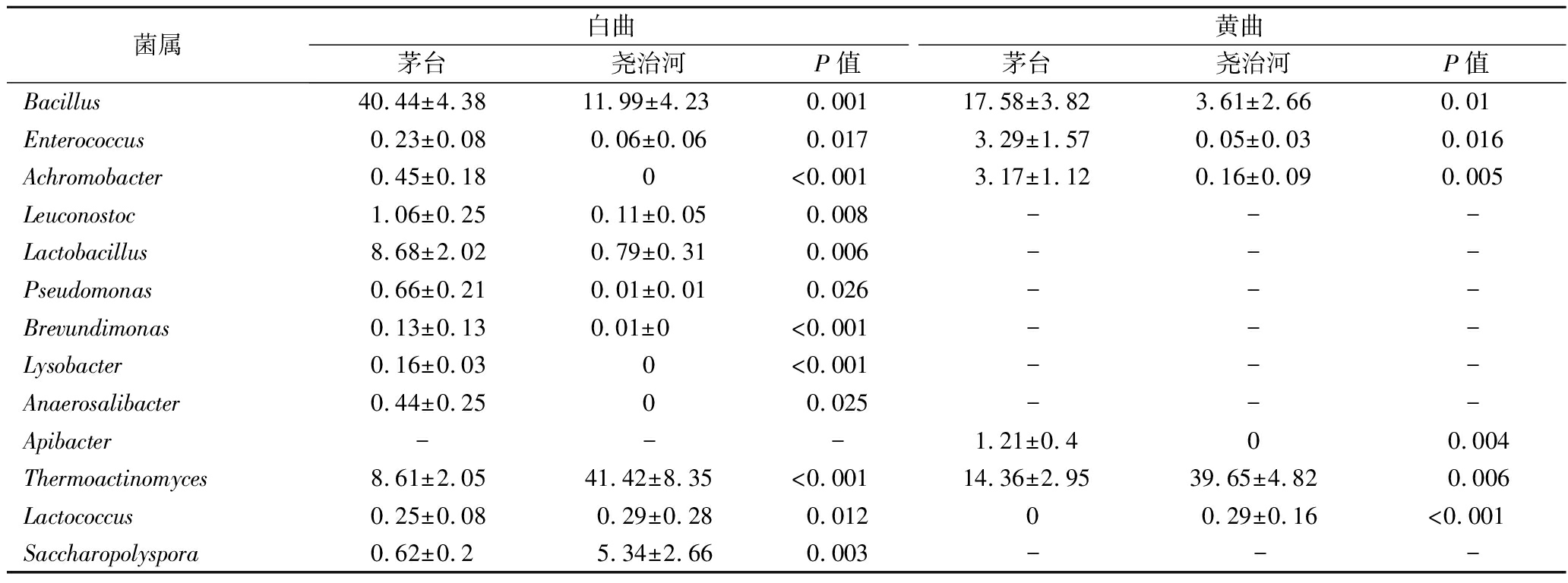
菌属白曲黄曲茅台尧治河P值茅台尧治河P值Bacillus40.44±4.3811.99±4.230.00117.58±3.823.61±2.660.01Enterococcus0.23±0.080.06±0.060.0173.29±1.570.05±0.030.016Achromobacter0.45±0.180<0.0013.17±1.120.16±0.090.005Leuconostoc1.06±0.250.11±0.050.008---Lactobacillus8.68±2.020.79±0.310.006---Pseudomonas0.66±0.210.01±0.010.026---Brevundimonas0.13±0.130.01±0 <0.001---Lysobacter0.16±0.030<0.001---Anaerosalibacter0.44±0.2500.025---Apibacter---1.21±0.400.004Thermoactinomyces8.61±2.0541.42±8.35<0.00114.36±2.9539.65±4.820.006Lactococcus0.25±0.080.29±0.280.01200.29±0.16<0.001Saccharopolyspora0.62±0.25.34±2.660.003---
由表2可知,尧治河大曲与茅台大曲相比,芽胞杆菌属、肠球菌属(Enterococcus)和无色杆菌属(Achromobacter)的相对含量显著较低(P<0.05)。此外,尧治河白曲中明串珠菌属、乳杆菌属、假单胞菌属(Pseudomonas)、短波单胞菌(Brevundimonas)、溶杆菌属和Anaerosalibacter的相对含量显著低于茅台白曲(P<0.05),尧治河黄曲中Apibacter相对含量显著低于茅台黄曲(P<0.05)。而嗜热放线菌属、乳球菌属(Lactococcus)在尧治河大曲中的相对含量显著较高(P<0.05),同时糖多孢菌属在尧治河白曲中的相对含量显著高于茅台白曲(P<0.05)。
2.6 高温大曲发酵菌群相关性分析
为了揭示高温大曲中发酵菌群彼此间的相关关系,本研究分别对茅台和尧治河高温大曲优势菌属进行了Spearman Rank相关性检验,彼此间具有显著相关关系(P<0.05)的菌属如图5所示。

a-茅台;b-尧治河
图5 茅台和尧治河高温大曲优势菌属相关性分析
Fig.5 Correlation analysis of dominant bacterial genera in Maotai and Yaozhihe high-temperature Daqu
与尧治河大曲菌群相比,茅台高温大曲中菌群彼此间相关关系更强。在茅台大曲中,芽胞杆菌属与魏斯氏菌属和明串珠菌属之间具有显著正相关性,而乳杆菌属与寡养单胞菌(Stenotrophomonas)、无色杆菌属彼此间呈显著负相关关系。值得注意的是,同属于变形菌门的劳尔氏菌属(Ralstonia)、寡养单胞菌、慢生根瘤菌(Bradyrhizobium)和无色杆菌属彼此间存在显著正相关关系。在尧治河大曲中,嗜热放线菌属与葡萄球菌属呈显著负相关关系,与Kroppenstedtia呈显著正相关关系。此外,芽胞杆菌属和魏斯氏菌属彼此间也存在显著正相关关系。
2.7 高温大曲发酵菌群功能差异分析
本研究使用PICRUSt软件对大曲中微生物的基因功能进行预测,并依据蛋白质COG进行功能注释。所有大曲样品共计注释到4 792 COGs,这些COGs分别属于23个功能大类,不同高温大曲间存在显著差异的功能大类如图6所示。由图6-a可知,茅台白曲与黄曲相比,COG功能大类M、D、O、S、T、U、L和J显著高于黄曲,COG功能大类K、E、I、Q、G显著低于黄曲。而尧治河白曲和黄曲菌群COG功能大类之间不存在显著差异。本研究进一步比较了相同颜色茅台大曲与尧治河大曲菌群功能差异,结果显示2个地区白曲功能差异较大(图6-b),而黄曲功能较为相似(图6-c)。具体而言,尧治河白曲与茅台白曲相比,COG功能大类I、R、V、K、E相对丰度显著较高,预示着其菌群对原料中的蛋白质和脂质潜在利用率更高,而O、M、U、T、S、Z显著较低;此外,尧治河黄曲中COG功能大类V和R相对丰度显著高于茅台黄曲。
2.8 高温大曲发酵菌群表型的差异分析
本研究使用BugBase工具对大曲中细菌表型进行预测,各组大曲表型分析结果如图7所示。尧治河白曲和黄曲菌群所有表型均不存在显著差异(P>0.05),而茅台白曲和黄曲菌群表型差异较大。同时,2个地区间的酒曲菌群表型也存在较大差异。具体而言,茅台白曲与黄曲相比,好氧菌、兼性厌氧菌、革兰氏阳性菌相对丰度显著较低,而革兰氏阴性菌相对丰度显著较高。尧治河高温大曲与茅台高温大曲相比,厌氧菌、革兰氏阳性菌、致病潜力相对丰度显著较高,而生物膜形成、移动原件含量、革兰氏阴性菌相对丰度显著较低。
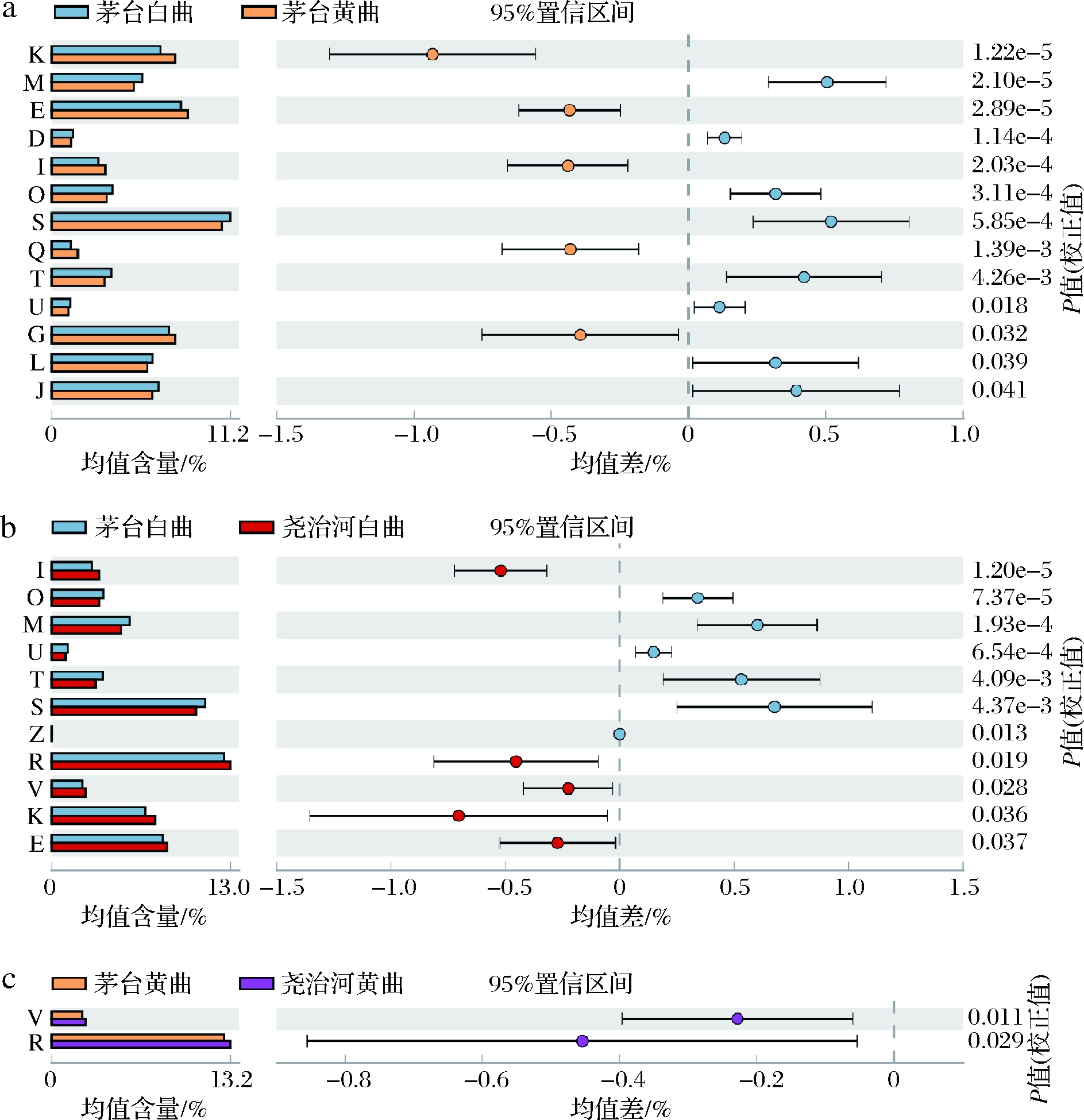
D-细胞周期控制,细胞分裂,染色体分割;E-氨基酸转运和代谢;G-碳水化合物运输和代谢;I-脂质运输和代谢;J-翻译、核糖体结构与生物合成;K-转录;L-复制、重组和修复;M-细胞壁/膜/包膜生物发生;O-翻译后修饰,蛋白质周转,伴侣;Q-次生代谢物的生物合成、运输和分解代谢;R-一般功能预测预测;S-功能未知;T-信号转导机制;U-细胞内运 输、分泌和囊泡运输;V-防御机制;Z-细胞骨架 a-茅台白曲和黄曲;b-茅台白曲和尧治河白曲;c-茅台黄曲和尧治河黄曲
图6 不同地区和不同颜色高温大曲菌群功能差异分析
Fig.6 Functional difference analysis of high-temperature Daqu flora in different regions and colors
3 讨论与结论
白酒的生产在我国呈现出明显的地域区分,如赤水河产区孕育了茅台、郎酒等酱香型白酒企业,而长江宜泸产区则孕育了五粮液、泸州老窖等浓香型白酒企业。由于白酒生产的区域性,国内的白酒科研团队亦常立足于某一区域的酿酒基质微生物开展研究,加之部分酿酒基质的保密性和稀缺性,最终导致很少有研究将不同地域、不同企业所蕴含的酿酒微生物进行比较分析。近10年来,以454焦磷酸测序、Illumina MiSeq和HiSeq测序为代表的第二代高通量测序技术在发酵食品微生物多样性解析中广泛应用[13-15],产生了海量的测序数据,其中部分数据上传至国际公共数据库。作为中国三大名酒之一,茅台酒在酱香型白酒中的地位不言而喻,多数酱香型白酒生产企业常以其作为标杆进行产品工艺的完善。本研究从国际公共数据库中下载了茅台大曲细菌多样性的测序数据,同时将其与湖北省襄阳市尧治河楚翁泉酒业酱香型白酒高温大曲的细菌群落结构、功能和表型进行了比较分析,一方面探究尧治河生产的高温大曲与茅台高温大曲之间的共性和差异,另一方面亦期望为襄阳乃至湖北地区酱香型白酒高温大曲制曲工艺的改良提供数据支撑和寻求方向。
本研究结果显示,2个地区的高温大曲细菌主要由芽胞杆菌属、嗜热放线菌属、葡萄球菌属、Kroppenstedtia等组成,优势菌群种类与之前的研究结果基本一致[16-17],这些菌属代表了高温大曲中3个主要的微生物组成部分:嗜热菌(嗜热放线菌属和Kroppenstedtia)、原料中的优势菌(芽胞杆菌属)和加工环境优势菌(葡萄球菌属)。嗜热放线菌属和Kroppenstedtia耐热能力强,在高温大曲中经常被检测到。嗜热放线菌属具有提供丰富风味物质和风味前体的潜在功能[18],例如,隶属于嗜热放线菌属的普通嗜热放线菌(Thermoactinomyces vulgaris)和糖化嗜热放线菌(Thermoactinomyces sacchari)不仅具有分泌淀粉酶、酯酶、纤维素酶、果胶酶和磷酸酶的能力,而且还能产生具有酱香味的风味物质,这对于白酒最终的风味至关重要[19-20]。然而,目前对于Kroppenstedtia的发酵特性鲜有报道,需要进一步的研究揭示。芽胞杆菌属是各类白酒发酵中的常见菌和优势菌,芽胞杆菌分泌的淀粉酶和蛋白酶能将淀粉和蛋白质转化为葡萄糖和氨基酸,促进白酒发酵过程中风味化合物前体如2-5-二甲基吡嗪和四甲基吡嗪的形成[21-23],因此其对于白酒发酵亦非常重要。之前基于酱油和韩国豆酱的研究发现,葡萄球菌属在发酵过程中可以提供各种酶和抗菌潜力[24-25],因此该菌属在白酒发酵中可能也具有一定的作用。值得注意的是,高温大曲核心菌群的分析结果显示,虽然在属水平2个地区的大曲优势菌群类似,但在更精细化的分类学水平上,如种水平或者株水平,两者之间存在较大差异;同时茅台大曲中存在较大比例的独有菌群,这应该是导致后续分析中2个地区大曲菌群功能存在较大差异的重要原因之一。
α多样性结果表明,尧治河高温大曲中微生物的丰度和多样性均显著低于茅台大曲。β多样性结果证实了2个地区大曲的群落结构亦存在明显的区别,且这种区别主要体现在白曲上。众所周知,黄色大曲形成的温度要高于白色大曲,因而为了寻找导致2个酒企高温大曲菌群群落结构差异的原因,本研究首先确认了温度(颜色)与大曲菌群之间的关系。结果发现,大曲中芽胞杆菌属、乳杆菌属、魏斯氏菌属、明串珠菌属和溶杆菌属的相对含量均随着温度的升高而下降,而糖多孢菌属相对含量随着温度升高而升高。通过对比尧治河和茅台酒曲中的差异菌群,如嗜热放线菌属和糖多孢菌属在尧治河酒曲中的相对含量显著较高,而芽胞杆菌属和乳杆菌属相对含量显著较低,我们不难推测导致尧治河酒曲与茅台酒曲菌群群落结构差异的一个重要因素是尧治河制曲时的发酵温度相较茅台酒曲更高。菌群相关性分析结果表明,茅台酒曲菌群间存在更为密切的相关关系,这一方面有利于酒曲中菌群群落结构的稳定[26],同时大量菌属彼此间存在显著正相关关系,对于微生物彼此相互协作产生丰富的代谢产物也具有积极作用,这可能是茅台酒风味幽雅细腻、持久稳定、回味悠长的关键因素之一。

a-好氧菌;b-厌氧菌;c-兼性厌氧菌;d-生物膜形成;e-移动元件;f-革兰氏阴性菌;g-革兰氏阳性菌;h-致病潜力;i-胁迫耐受
图7 不同地区和颜色高温大曲菌群表型差异分析
Fig.7 Phenotypic difference analysis of high-temperature Daqu in different regions and colors
高温大曲发酵菌群功能分析结果表明,黄曲中菌群的功能大类如E、I、Q、G相对丰度显著较高,这些功能对于菌群降解原料中的淀粉和蛋白质及产生次级代谢产物至关重要,这可能是黄色大曲用于酱香型白酒生产时产品品质最佳的原因[27],而白曲中相对含量显著较高的的功能如M、D、O、T、U、L和J更多的与菌群的生产繁殖有关,表明白曲中微生物彼此间的生长竞争更加激烈[28]。高温大曲发酵菌群表型分析结果显示,尧治河高温大曲中革兰氏阳性菌和致病潜力相对含量显著较高(P<0.05),这与尧治河大曲中嗜热放线菌属和葡萄球菌属的相对含量较高有关。同时,其菌群移动元件相对含量显著较低,这表明尧治河高温大曲中菌群对发酵环境的适应性要弱于茅台[29]。
综上所述,尧治河高温大曲制作温度相较茅台大曲更高,同时大曲中的重要发酵菌属芽胞杆菌属相对含量显著较低,因此,在后续的制曲过程中适当降低制曲温度,同时尝试具有优良发酵特性芽胞杆菌属的外源添加,可能对提升尧治河酱香型大曲及最终白酒的品质具有积极的意义。值得一提的是,由于本研究所选取的OTU划分方式、比对数据库和计算参数等与原来的研究可能不尽相同,因此可能导致本研究的部分结果与原有研究报道稍有不同。
[1] LIU X,GUO K L,ZHANG H X.Determination of microbial diversity in Daqu,a fermentation starter culture of Maotai liquor,using nested PCR-denaturing gradient gel electrophoresis[J].World Journal of Microbiology & Biotechnology,2012,28(6):2 375-2 381.
[2] WANG M Y,YANG J G,ZHAO Q S,et al.Research progress on flavor compounds and microorganisms of Maotai flavor Baijiu[J].Journal of Food Science,2019,84(1):6-18.
[3] WANG B W,WU Q,XU Y,et al.Synergistic effect of multiple saccharifying enzymes on alcoholic fermentation for Chinese Baijiu production[J].Applied and Environmental Microbiology,2020,86(8).DOI:10.1128/AEM.00 013-20.
[4] DENG L,MAO X,LIU D,et al.Comparative analysis of physicochemical properties and microbial composition in high-temperature Daqu with different colors[J].Frontiers in Microbiology,2020,11:588117.
[5] GAN S H,YANG F,SAHU S K,et al.Deciphering the composition and functional profile of the microbial communities in Chinese Moutai liquor starters[J].Frontiers in Microbiology,2019,10:1540.
[6] WANG Y R,CAI W C,WANG W P,et al.Analysis of microbial diversity and functional differences in different types of high-temperature Daqu[J].Food Science & Nutrition,2021,9(2):1 003-1 016.
[7] CAPORASO J G,BITTINGER K,BUSHMAN F D,et al.PyNAST:A flexible tool for aligning sequences to a template alignment[J].Bioinformatics,2010,26(2):266-267.
[8] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2 460-2 461.
[9] COLE J R,WANG Q,FISH J A,et al.Ribosomal Database Project:Data and tools for high throughput rRNA analysis[J].Nucleic Acids Research,2014,42(D1):D633-D642.
[10] ROGNES T,FLOURI T,NICHOLS B,et al.VSEARCH:a versatile open source tool for metagenomics[J].PeerJ,2016,4:e2584.
[11] LANGILLE M G I,ZANEVELD J,GREGORY CAPORASO J,et al.Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J].Nature Biotechnology,2013,31(9):814-821.
[12] BENJAMINI Y,HOCHBERG Y.Controlling the false discovery rate:A practical and powerful approach to multiple testing[J].Journal of the Royal Statistical Society:Series B(Methodological),1995,57(1):289-300.
[13] 李欣蔚, 丛敏,武俊瑞,等.基于16S rRNA基因V3-V4区高通量测序分析东北自然发酵酸菜中细菌群落结构[J].现代食品科技,2017,33(2):69-75;82.
LI X W,CONG M,WU J R,et al.Analysis of the bacterial community structure in naturally fermented cabbage of northeast China by high-throughput sequencing[J].Modern Food Science and Technology,2017,33(2):69-75;82.
[14] 倪慧, 葛东颖,杨甜甜,等.基于MiSeq高通量测序技术奶疙瘩细菌群落结构研究[J].中国乳品工业,2020,48(12):16-19.NI H,GE D Y,YANG T T,et al.Study on bacterial diversity of Kurut by MiSeq high-throughput sequencing technology[J].China Dairy Industry,2020,48(12):16-19.
[15] 魏本良, 刘长根,肖阳生,等.基于Illumina HiSeq技术分析浆水中细菌多样性及理化性质[J].食品科学,2019,40(6):62-68.
WEI B L,LIU C G,XIAO Y S,et al.Bacterial diversity analysis using Illumina HiSeq sequencing of Jiangshui,a Chinese fermented vegetable food,and its physicochemical properties[J].Food Science,2019,40(6):62-68.
[16] 沈毅, 陈波,王西,等.酱香型郎酒高温大曲、酒醅和窖泥中细菌群落结构分析[J].中国酿造,2020,39(2):89-93.
SHEN Y,CHEN B,WANG X,et al.Analysis of bacterial community structure in high temperature Daqu from sauce-flavor Langjiu,fermented grains and pit mud[J].China Brewing,2020,39(2):89-93.
[17] JIN Y,LI D Y,AI M,et al.Correlation between volatile profiles and microbial communities:A metabonomic approach to study Jiang-flavor liquor Daqu[J].Food Research International,2019,121:422-432.
[18] YOON J H,PARK Y H.Phylogenetic analysis of the genus Thermoactinomyces based on 16S rDNA sequences[J].International Journal of Systematic and Evolutionary Microbiology,2000,50 Pt 3:1 081-1 086.
[19] XIAO C,LU Z M,ZHANG X J,et al.Bio-heat is a key environmental driver shaping the microbial community of medium-temperature Daqu[J].Applied and Environmental Microbiology,2017,83(23):e01 550-e01 517.
[20] ZHENG X W,YAN Z,ROBERT NOUT M J,et al.Characterization of the microbial community in different types of Daqu samples as revealed by 16S rRNA and 26S rRNA gene clone libraries[J].World Journal of Microbiology and Biotechnology,2015,31(1):199-208.
[21] WANG H Y,GAO Y B,FAN Q W,et al.Characterization and comparison of microbial community of different typical Chinese liquor Daqus by PCR-DGGE[J].Letters in Applied Microbiology,2011,53(2):134-140.
[22] ZHENG X W,YAN Z,HAN B Z,et al.Complex microbiota of a Chinese “Fen” liquor fermentation starter(Fen-Daqu),revealed by culture-dependent and culture-independent methods[J].Food Microbiology,2012,31(2):293-300.
[23] HUANG Y H,YI Z L,JIN Y L,et al.New microbial resource:Microbial diversity,function and dynamics in Chinese liquor starter[J].Scientific Reports,2017,7:14577.
[24] NAM Y D,LEE S Y,LIM S I.Microbial community analysis of Korean soybean pastes by next-generation sequencing[J].International Journal of Food Microbiology,2012,155(1-2):36-42.
[25] YAN Y Z,QIAN Y L,JI F D,et al.Microbial composition during Chinese soy sauce koji-making based on culture dependent and independent methods[J].Food Microbiology,2013,34(1):189-195.
[26] 贺纪正, 李晶,郑袁明.土壤生态系统微生物多样性-稳定性关系的思考[J].生物多样性,2013,21(4):412-421.
HE J Z,LI J,ZHENG Y M.Thoughts on the microbial diversity-stability relationship in soil ecosystems[J].Biodiversity Science,2013,21(4):412-421.
[27] 覃建良, 莫于旺,覃旺远.酱香型丹泉酒制曲工艺初探[J].轻工科技,2015,31(12):26-27;44.
QIN J L,MO Y W,QIN W Y.Primary study on the technology of making Daqu with Maotai-flavor danquan wine[J].Light Industry Science and Technology,2015,31(12):26-27;44.
[28] JEONG S E,CHUN B H,KIM K H,et al.Genomic and metatranscriptomic analyses of Weissella koreensis reveal its metabolic and fermentative features during kimchi fermentation[J].Food Microbiology,2018,76:1-10.
[29] 钱玮, 陆震鸣,柴丽娟,等.泸型酒酒醅与窖泥中乳酸菌群落结构差异[J].应用与环境生物学报,2020,26(4):760-765.
QIAN W,LU Z M,CHAI L J,et al.Differences in the lactic acid bacterial community structure between Jiupei and pit mud during the fermentation of Luzhou-flavour baijiu[J].Chinese Journal of Applied and Environmental Biology,2020,26(4):760-765.