酱油是起源于我国的酿造调味品,已有数百年的产业发展历史,后传入韩国、日本,现已在国际市场上获得了欧美地区消费者的认可。岭南区日照充裕、气温较高,形成了酱油高盐稀态酿造工艺,这种工艺生产出的酱油味道鲜美、香味浓郁,在居民饮食中具有较高接受度。随着社会经济发展及人民生活水平提高,民众对于酱油这一必备调味品的品质及健康也有了更高、更多样化的需求。《中国居民膳食指南》推荐[1]:健康成人每人每天食盐摄入量不超过6 g;2~3岁幼儿摄入量不超过2 g;4~6岁幼儿不超过3 g;7~10岁儿童不超过4 g;65岁以上老人不超过5 g。因此,低盐是健康需求的新发展动向,高盐稀态酱油减盐发酵也已成为产业发展的新趋势,但现有技术存在基础研究依然薄弱。高盐稀态工艺包括制曲和发酵2个阶段,大豆/豆粕蒸煮后与小麦/麸皮混合,接入米曲霉进行固态制曲,完成后注入高浓度盐水进行液态发酵[2]。其中微生物扮演着极为重要的角色:制曲阶段,米曲霉产生的中性蛋白酶、酸性蛋白酶及纤维酶等,对曲料中的生物大分子物质进行初步水解;液态发酵阶段,酱醪中耐盐酵母菌与耐盐乳酸菌等多菌种产生更多酶系进一步对原料进行水解,生成更多的风味物质,包括氨基酸、肽、酮、醛、酸、酯、酚等;经过长周期的发酵,最终形成滋味和香气完美融合的岭南酱油[3-4]。这其中盐分对于发酵过程和产品风味至关重要:一是防腐抑菌作用,淘汰非耐盐性野生杂菌或有害微生物,这一作用主要通过降低发酵体系水活度,提高渗透压来实现[5],如沙门氏菌及大肠杆菌能够耐受的最低水分活度为0.95(相当于80~90 g/L的食盐含量)[6-7];此外,高浓度氯离子也能够有效抑制有害微生物的滋生[8],使得酱油能够安全的生产[9]。二是形成有效的有益微生物筛选机制[6];与酱油风味形成密切相关的嗜盐四链球菌(Tetragenococcus halophilus)能够耐受26%的高盐环境[10-11],鲁氏接合酵母(Zygosaccharomyces rouxii)及易变球拟酵母(Torulopsis versatilis)分别能够耐受180 g/L及250 g/L的食盐含量[12-13]。三是高浓度盐带来的咸味是酱油风味口感协调不可缺少因素;据报道,酱油的口感很大程度上取决于咸味及鲜味的平衡,咸味能够抑制苦味,提高鲜味[14]。酱油中咸味的缺乏在一定程度上破坏了酱油产品的总体风味及协调性,造成风味寡淡,影响感官品质。高盐稀态酱油酱醪通常酱醪中的盐分达到160~180 g/L,以维持菌群和风味的平衡,而低盐酱油酿造工艺也就会面临着高微生物污染及风味不协调的风险。CHUN等[15]报道了在韩式大酱酱醪的发酵中,90 g/L盐度大酱的pH值和有机酸变化远大于120、150和170 g/L盐度的大酱。
本课题对高盐稀态酱油不同盐浓度条件下发酵微生物菌落结构特征、变化规律及其对酱油理化指标和风味的影响进行研究分析,以期通过基础研究结果指引高盐稀态酱油减盐发酵技术的进一步升级开发。
1 材料与方法
1.1 材料与试剂
本研究所用曲料样品均取自佛山市海天调味食品股份有限公司。
表1 材料信息
Table 1 Material information

分组盐质量浓度/(g·L-1)发酵阶段/d编号L1205LI25LII55LIIIM1505MI25MII55MIIIH1705HI25HII55HIII
发酵工艺:将成曲用研钵碾碎,与盐水以体积比1.0∶2.0混合。设置3个初始盐度组,分别编号为L、M、H,其初始盐水质量浓度对应如下:第1组-13.7 g/100 mL、第2组-16.6 g/100 mL、第3组-20.4 g/100 mL。酱醪最终食盐含量约为120、150、170 g/L。将混合后的酱醪分装入2 L坛子中,不控温的情况下发酵55 d,期间每天搅拌1次。发酵第5天(I)、第25天(II)、第55天(III)移取各组酱醪样品,-20 ℃冰箱中保存待用。
土壤基因组提取试剂盒,美国MoBio公司;Qubit®dsDNA HS分析试剂盒,美国Thermo Fisher公司;SYBR® Premix Ex TaqTM II及配套产品,大连宝生生物工程公司;Pfu PCR MasterMix、核酸Maker和GoldView(GV),上海科晴生物技术有限公司;细菌基因组快速抽提试剂盒、PCR引物,上海生物工程有限公司。
1.2 仪器与设备
pH scan40笔式pH计,上海般特仪器制造公司;SynergyTM H1多功能酶标仪,德国BioTek公司;DK-8D三孔三温数显恒温水浴锅,常州申光仪器有限公司;5804R台式高速冷冻离心机,德国Eppendorf公司;SBA-40X三通道生物传感分析仪,济南延和生物科技有限公司。
1.3 实验方法
1.3.1 酱油感官评价的测定
酱醪过滤所得原油经巴氏杀菌后进行感官鉴评。设置鲜味、咸味、酸味、苦味、甜味、气味、色泽7个评价维度,组织20位有经验鉴评人员进行感官评分,满分为10分,其中高盐组(H)为对照,定为6分。评分标准为各指标比对照浓/好打6~10分,比对照淡/差打1~6分,每项指标的最终得分为20位鉴评人员所打分值的平均值。
1.3.2 微生物菌落总数的测定
将酱醪搅拌摇匀,并在超净台中取0.5~1 mL至15 mL离心管中,用无菌水将样品稀释至10-1、10-2、10-3、10-4浓度梯度;将10-2、10-3、10-4浓度梯度的稀释样品吸取100 μL分别涂布于PDA、PCA、MRS平板,每个梯度3个平行;将接种后的MRS平板培养基倒置于30 ℃无氧环境中培养,PDA和PCA培养基置于30 ℃环境中培养。
PCA培养基接种后培养24 h左右计数其菌落总数,PDA培养基接种后培养2~3 d对酵母进行计数,MRS培养基接种后培养2~3 d对乳酸菌进行计数。一般选取菌落数在30~300的稀释梯度进行计数,3个平板分别计为N1、N2、N3。菌落总数计算如公式(1)所示:
菌落总数=10×(N1+N2+N3)/(3×D)
(1)
式中:D,计数的稀释梯度。
1.3.3 理化指标测定
使用pH计直接测定酱醅样本的pH值;乳酸采用三通道生物传感分析仪测定;氨基酸态氮和总酸的测定参照GB 5009.40—2003 《酱卫生标准的分析方法》;还原糖的含量测定(以葡萄糖计)参考GB 5009.7—2016 《食品中还原糖的测定》;无盐固形物含量测定参考GB/T 18186—2000 《酿造酱油》。
1.3.4 高通量微生物基因组测序
测序流程:细菌测序区域为16S rRNA的V3~V4可变区,所用通用引物为341F(5′-CCTA-CGGGNGGCWGCAG-3′)和908R(5′-CCGTCAATTCMTTTGAGTTT-3′);真菌测序区域为18S rRNA的ITS1可变区,所用通用引物为ITS1F(5′CTTGGTCATTTAGAGGAAGTAA-3′)和ITS4R(5′TCCTCCGCTTATTGATATGC-3′)。PCR反应及扩增产物提取也按照文献的说明进行,提取获得的扩增子由北京百迈客生物科技有限公司应用Illumina HiSeq 2500平台进行测序。高通量测序得到的原始图像数据文件,经碱基识别(Base Calling)分析转化为原始测序序列(Sequenced Reads)进行信息分析。
1.3.5 信息分析流程
首先使用Trimmomatic v0.33软件[16],对测序得到的Raw Reads进行过滤;然后使用cutadapt 1.9.1软件[17]进行引物序列的识别与去除,获得Clean Reads。使用Usearch v10软件[18],对Clean Reads进行拼接,然后根据长度范围进行长度过滤。使用UCHIME v4.2软件[19],鉴定并去除嵌合体序列,得到最终有效数据(Effective Reads)。使用Usearch软件对Reads在97.0%的相似度水平下进行聚类,细菌以SILVA为参考数据库[20],真菌则以UNITE为参考数据库[21]使用朴素贝叶斯分类器结合比对的方法对特征序列进行分类学注释;利用QIIME软件[22]生成不同相对丰度、丰富度、多样性指数和相异性矩阵。使用PAST软件[23]及Excel进行非度量多维尺度分析(nonmetric multidimensional scale analysis,NMDS)数据绘图,使用SPSS 23进行显著性统计分析。
2 结果与分析
2.1 不同盐浓度酱醪发酵过程感官质量分析
不同发酵盐浓度酱油感官质量评价结果显示(图1),降低盐水浓度对提升酱油的鲜味有明显贡献,这可能与盐浓度降低后,曲霉酶系活性更佳,产生了更多鲜味氨基酸所致。但同时产生了较明显的酸味,酱油总体香气水平也明显下降,色泽明显较淡,影响酱油感官品质,L组更为突出。
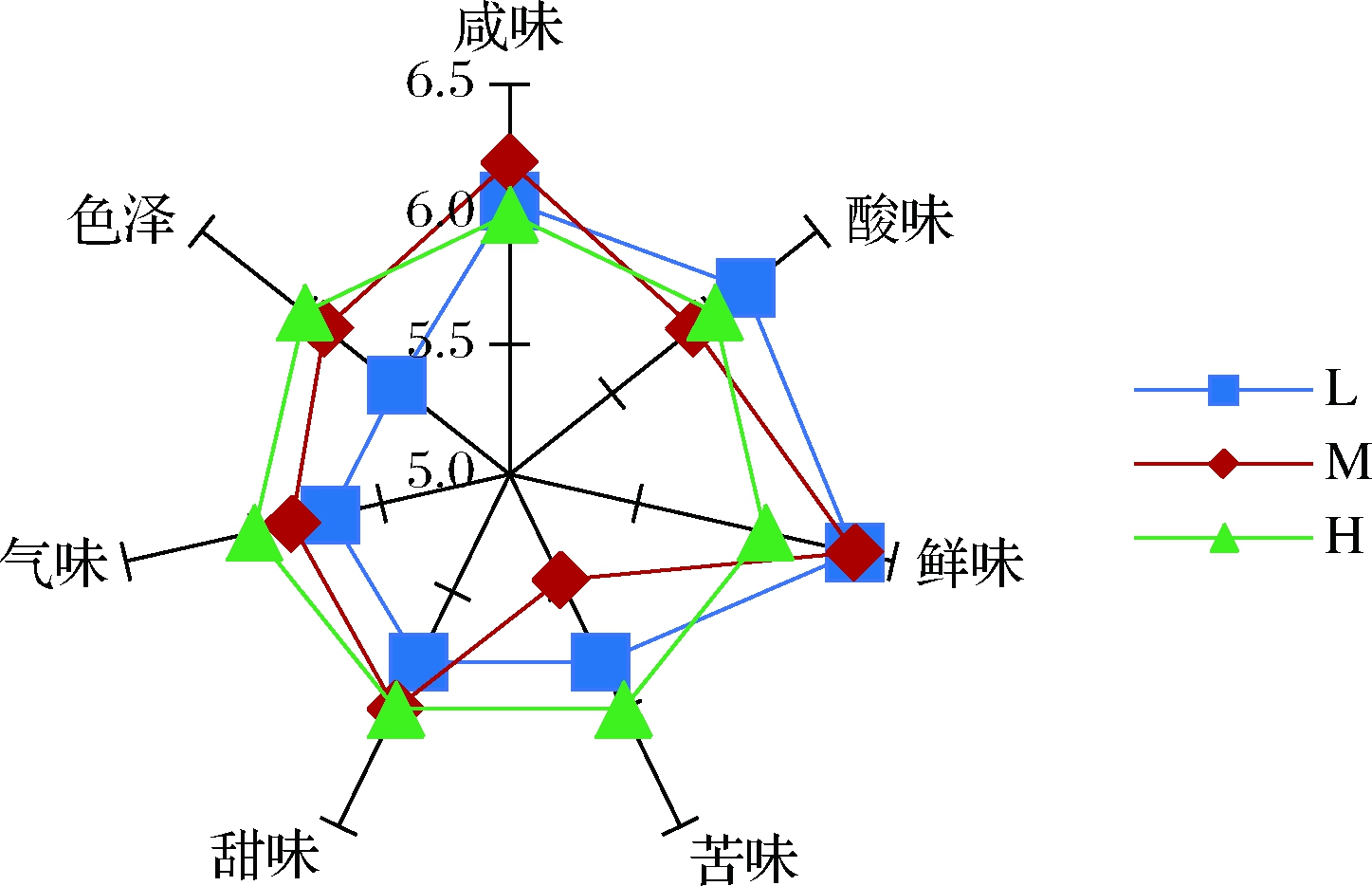
图1 不同盐浓度酱油的感官质量评价
Fig.1 Sensory evaluation of soy sauce with different salt concentration
2.2 不同盐浓度酱醪发酵过程微生物
不同盐浓度发酵过程酱醪中微生物含量如表2。不同盐分酱醪细菌含量在发酵初期并无显著性差异(P<0.05),在发酵过程中均呈现降低趋势。真菌及乳酸菌数量则呈现不同趋势,在发酵初期随着盐浓度的提高而降低,证明盐浓度的提高对真菌及乳酸菌形成有效抑制。其中真菌总数在发酵中期各组别均有显著的提升,但L组含量更高,数值达到6.46 lg CFU/mL,而M组和H组则分别为6.32和2.43 lg CFU/mL。在乳酸菌含量方面,L组在发酵初期与M组和H组的差异达到2个数量级;中期也呈现微量的提升但差异并不显著(P<0.05),发酵后期时又有所降低,其中L组乳酸菌含量对数值从中期至后期由6.37降至4.60,而M组和H组虽有所减少,但并无显著性差异,说明盐浓度对于微生物的影响贯穿于整个发酵周期内。
表2 不同盐浓度酱醪发酵过程中各类微生物菌落总数
Table 2 The total colonies forming unit in the fermentation process of sauce mash with different salt content
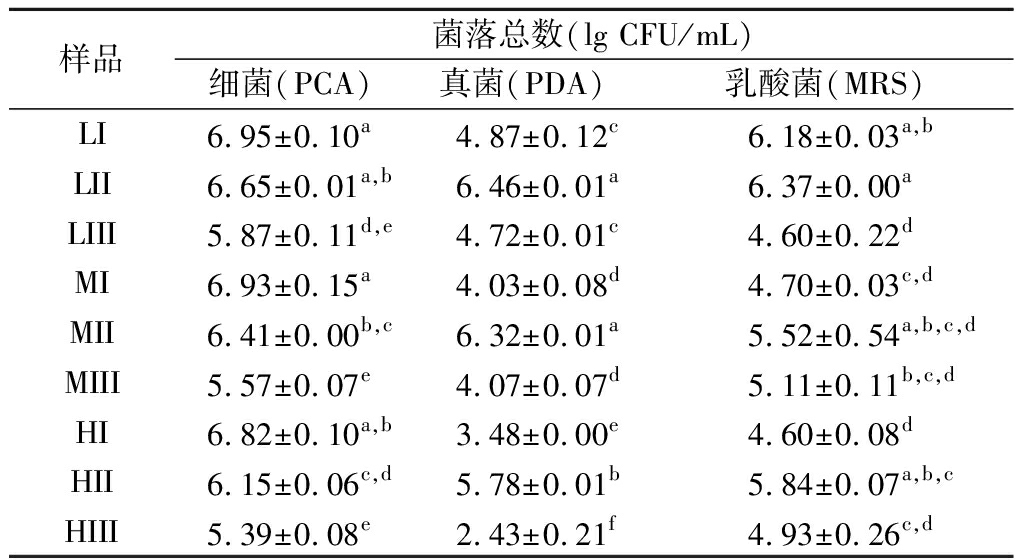
样品菌落总数(lg CFU/mL)细菌(PCA)真菌(PDA)乳酸菌(MRS)LI6.95±0.10a4.87±0.12c6.18±0.03a,bLII6.65±0.01a,b6.46±0.01a6.37±0.00aLIII5.87±0.11d,e4.72±0.01c4.60±0.22dMI6.93±0.15a4.03±0.08d4.70±0.03c,dMII6.41±0.00b,c6.32±0.01a5.52±0.54a,b,c,dMIII5.57±0.07e4.07±0.07d5.11±0.11b,c,dHI6.82±0.10a,b3.48±0.00e4.60±0.08dHII6.15±0.06c,d5.78±0.01b5.84±0.07a,b,cHIII5.39±0.08e2.43±0.21f4.93±0.26c,d
*注:L、M、H分别表示低盐组(120 g/L)、中盐组(150 g/L)和高盐组(170 g/L);I、II、III分别表示发酵第5天(I)、第25天(II)、第55天(III);Turkey HSD多重比较检验不同盐分组间差异,同一列不同小写字母标记的数值表示差异显著(P<0.05)(下同)
2.3 不同盐浓度酱醪发酵过程理化特性分析
总酸的高低直接影响酱油的滋味,对酱油的最终品质有重要影响,而乳酸菌等微生物代谢产生的有机酸是酱油中总酸的重要来源。由3种不同盐水浓度下生产酱油原油的总酸变化(表3)可知,发酵全程减盐条件(L、M)总酸含量相比H组明显提高,与文献报道相似[24],对比表2微生物变化情况,L组和H组总酸含量较高可能是由于盐分降低对产酸微生物(如乳酸菌)的抑制作用减弱所导致[25-26]。
氨基酸态氮的高低直接影响酱油的鲜味,是酱油国标中(GB 18186—2000)衡量酱油等级的重要参数,不同盐分酱醪发酵原油氨基酸态氮含量(表3)都达到了特级的标准(0.8 g/100 mL)。同一发酵时期,氨基酸态氮的含量与盐浓度成反比(55 d最明显),与文献研究结果一致[26]。在发酵进行的第55天,L组氨基态氮达到1.148 g/100 mL,比H组提高了49%。氨基酸是由微生物分泌的蛋白水解酶水解蛋白产生,高盐条件对微生物的生长及蛋白酶活力有一定抑制作用,而盐分降低后微生物含量更为丰富,对蛋白酶活的抑制作用更低,最终导致氨基酸态氮含量更高[25,27]。
表3 不同盐分酱醪发酵过程中基本理化指标含量
Table 3 Physiochemical characteristics of soy sauce mash with different salt content
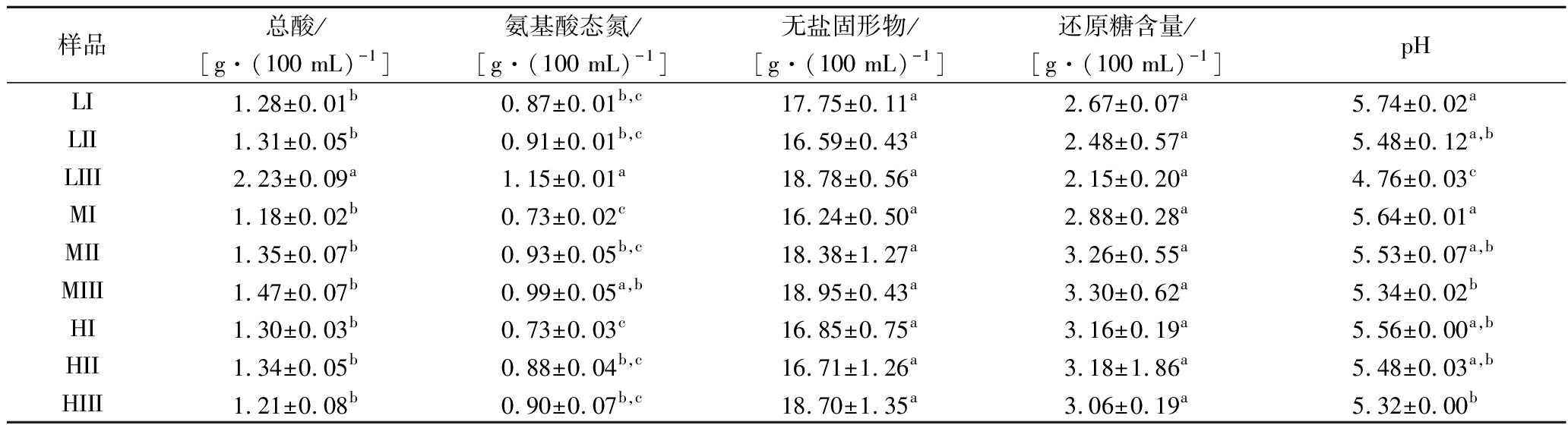
样品总酸/[g·(100 mL)-1]氨基酸态氮/[g·(100 mL)-1]无盐固形物/[g·(100 mL)-1]还原糖含量/[g·(100 mL)-1]pHLI1.28±0.01b0.87±0.01b,c17.75±0.11a2.67±0.07a5.74±0.02aLII1.31±0.05b0.91±0.01b,c16.59±0.43a2.48±0.57a5.48±0.12a,bLIII2.23±0.09a1.15±0.01a18.78±0.56a2.15±0.20a4.76±0.03cMI1.18±0.02b0.73±0.02c16.24±0.50a2.88±0.28a5.64±0.01aMII1.35±0.07b0.93±0.05b,c18.38±1.27a3.26±0.55a5.53±0.07a,bMIII1.47±0.07b0.99±0.05a,b18.95±0.43a3.30±0.62a5.34±0.02bHI1.30±0.03b0.73±0.03c16.85±0.75a3.16±0.19a5.56±0.00a,bHII1.34±0.05b0.88±0.04b,c16.71±1.26a3.18±1.86a5.48±0.03a,bHIII1.21±0.08b0.90±0.07b,c18.70±1.35a3.06±0.19a5.32±0.00b
可溶性无盐固形物是评价酱油好坏的一个重要指标,代表着发酵过程中原料物质的分解、转化产生各种小肽、氨基酸、还原糖类物质的水平。由表3可知,3组酱油无盐固形物的含量随时间的变化趋势相似,缓慢稳定上升,但并无显著性差异。还原糖是酱醪微生物代谢的物质基础,L组发酵全程还原糖含量明显较低,且下降迅速,25 d开始趋于平稳且较低的水平,表明微生物代谢更为旺盛,尤其是酵母及乳酸菌含量更高(表2),造成还原糖被相对充分利用,但总体上差异并不显著。
2.4 不同盐浓度酱醪发酵过程微生物组成及多样性分析
采用二代测序对120、150、170 g/L盐浓度发酵酱醪的不同阶段进行微生物多样性分析,进一步深入理解盐度降低后微生物的组成变化情况。
2.4.1 不同盐浓度酱醪中细菌群落分布分析
通过16S二代测序共获得2 158 201对Reads,每个样品至少产生79 374条Clean Reads,平均产生79 703条Clean Reads,各样品Q30都处于至少95%的水平,各样品Goods Coverage的中位数均在0.99以上,意味着数据采集具有足够的完整性及代表性。其中门及属水平群落结构分布随盐浓度和时间变化的结果分别如图2-a所示,其中丰度最高前3的细菌门分别是厚壁菌门(Firmicutes,79.58%~94.19%)、变形菌(Proteobacteria,1.96%~6.50%)及拟杆菌门(Bacteroidetes,1.88%~5.83%)。

a-门水平;b-属水平
图2 门水平和属水平下不同盐浓度酱醪发酵过程细菌菌群结构
Fig.2 Bacterial community structure during the fermentation process of soy sauce mash with different salt concentrations at the phylum level and genus level
魏斯氏菌属(Weissella)是酱醪样品中最主要的细菌,占据54.69%~78.00%(图2-b)。而葡萄球菌属(Staphylococcus)、明串珠菌属(Leuconostoc)和乳杆菌属(Lactobacillus)的相对丰度分别介于1.38%~9.22%、2.41%~5.75%及1.92%~10.81%。
在酱醪样品中前10个细菌属中,乳酸菌就占了5个,还包括了链球菌属(Streptococcus,0.79%~2.83%)及乳球菌属(Lactococcus)。
据文献报道,高盐稀态发酵酱油的优势细菌主要为魏斯氏菌属、芽孢杆菌属、乳杆菌属、葡萄球菌属等[28-30],与本研究结论一致。其中,魏斯氏菌属是酱醪样品中主要乳酸菌,丰度占比54%~78%,变化趋势较为稳定,在发酵全过程发挥重要作用。随着盐浓度降低,微生物菌群组成发生明显变化,其中L组乳杆菌含量在25 d(II)后有明显的增生,相对丰度超过10%;而葡萄球菌属(Staphylococcus)及乳球菌属(Lactococcus)均在L组中持续增长,在发酵L组55 d(III)其相对丰度明显高于盐分150 g/L(M)和170 g/L(H)的样品。以上结果表明,酱醪发酵过程中乳酸菌是主要的细菌,而盐分降低会导致乳杆菌、葡萄球菌、乳球菌等相对丰度升高,从而影响乳酸菌组成。
2.4.2 不同盐浓度酱醪中细菌α-生物多样性分析
从图3-a中可见,在57 130 reads的水平上,120 g/L(L)样品在发酵5 d(I)、25 d(II)后,其系统发育多样性均值是低于150 g/L(M)和170 g/L(H)盐分的样品,代表其菌群结构在系统发育树上有着较为亲密的关系。在observed species和Chao1指数分析中发现L组和M组酱醪在发酵初期(I)就有较高的微生物丰富度,并在发酵25 d(II)及55 d(III)后展示出比H组更高的细菌微生物物种丰富度及物种均匀度(香农指数)。说明高盐落黄发酵能够有效防止非耐盐细菌的生长[5],并在25 d后才达到较高的微生物丰度。

a-PD整树(PD whole tree);b-观察物种(Observed species);c-物种丰富度(Chao1);d-香浓指数(Shannon)
图3 基于57 130 reads水平不同盐浓度酱醪发酵过程的细菌α-多样性分析
Fig.3 Bacterial α-diversity analysis based on 57 130 reads during the fermentation process of soy sauce mash with different salt concentrations
2.4.3 不同盐浓度酱醪中细菌β-生物多样性分析
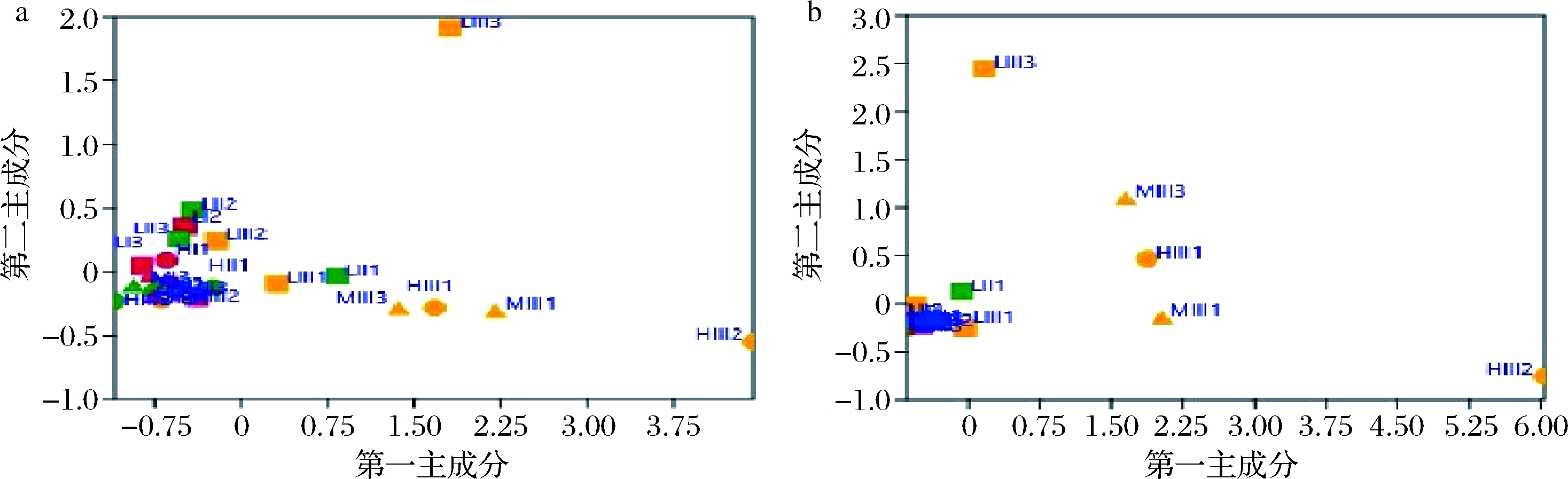
通过不同β-生物多样性距离矩阵与NMDS分析发现(图4),即不同盐份酱醪在初始阶段(I,发酵5 d,红色)虽然微生物物种及分布有所差异,但逐步在25 d发酵(II,绿色)后细菌菌群结构形成了较为集中的族群,并在发酵55 d(III,黄色)后形成了更为明显的族群。此外,β-生物多样性也展示了不同盐分酱醪在发酵过程中其细菌菌群结构随着发酵周期的延长会往同个方向演变,形成了明显的演变路线及规律,与之前的报道相符[31]。图4-a及图4-d中可知低盐组(L,黄色正方形)与中盐(M,黄色三角形)及高盐组(H,黄色圆形)有相较明显的区分,能够形成单一族群的。虽然随着发酵周期的延长,往同个方向演变,但低盐组在发酵后期(III)明显的脱离了中盐及高盐组的族群,微生物菌群上是有一定的差异。
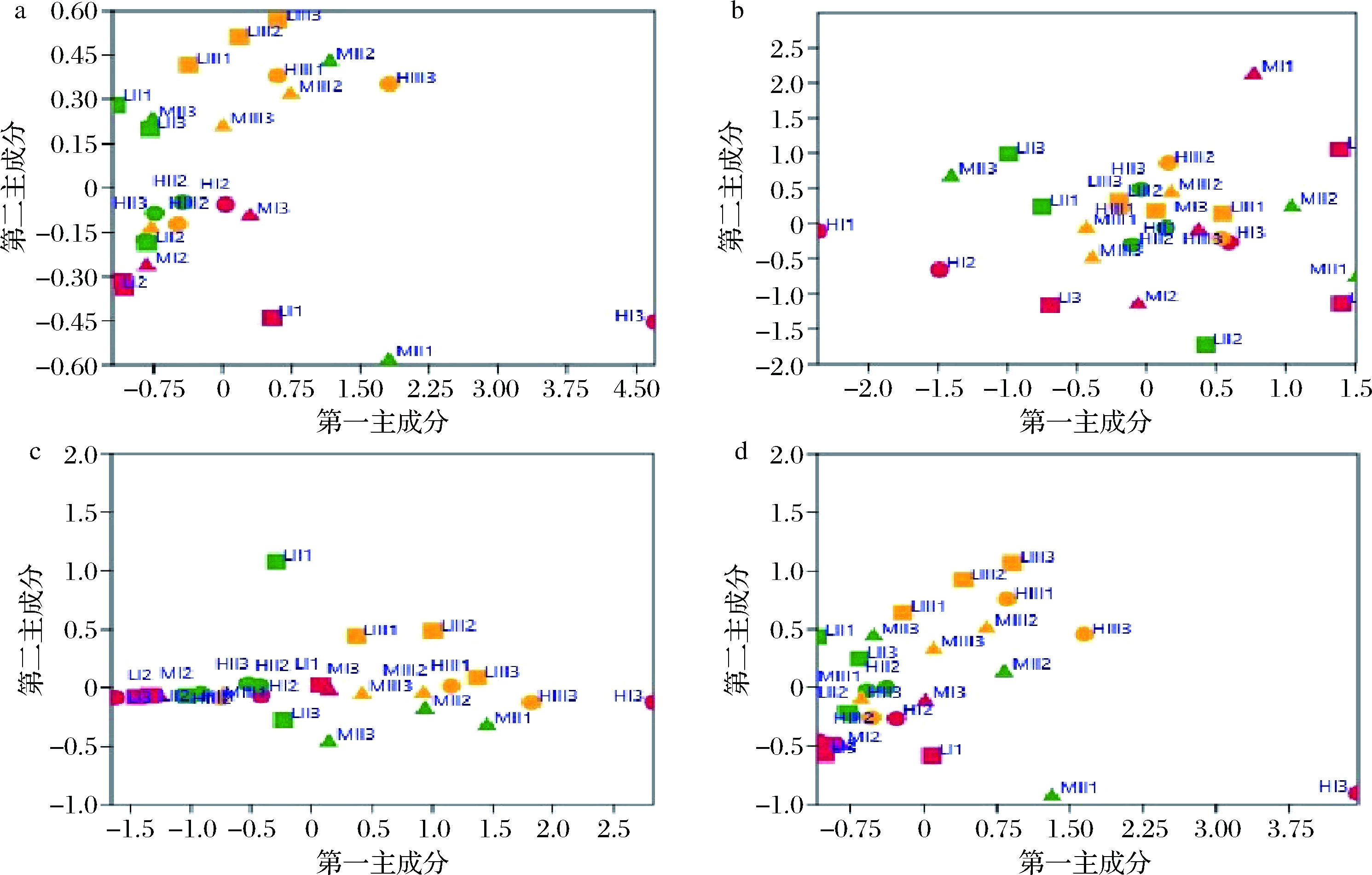
a-加权UniFrac;b-未加权UniFrac;c-Euclidean;d-Bray-Curtis
图4 不同盐浓度酱醪发酵过程的细菌β-生物多样性距离矩阵与NMDS分析
Fig.4 Bacterial β-diversity distance matrix and NMDS analysis during the fermentation of sauce mash with different salt concentrations
2.4.4 不同盐浓度酱醪中真菌群落分布分析
通过ITS全长二代测序后通过Barcode识别后共获得2 074 495对Reads。每个样品至少产生47 220条Clean Reads,平均产生76 618条Clean Reads,各样品Q30都处于至95%的水平,Goods Coverage的中位数均在0.99以上,意味着数据采集具有足够的完整性及代表性。其中门水平群落结构分布随盐浓度和时间变化的结果分别如图5-a所示,其中丰度最高前3的细菌门分别是子囊菌门(Ascomycota,94.27%~99.57%)、担子菌门(Basidiomycota,0.31%~3.99%)及被孢霉菌门(Mortierellomycota,0.05%~1.36%)。

a-门水平;b-属水平
图5 门水平和属水平下不同盐浓度酱醪发酵过程真菌菌群结构
Fig.5 Fungi community structure during the fermentation process of soy sauce mash with different salt concentrations at the phylum level and genus level
属水平群落结构如图5-b所示。酱油发酵酱醪中相对含量最为丰富的是曲霉菌属(Aspergillus,81.79%~97.01%)、假丝酵母菌属(Candida,0.43%~4.14%)、米勒氏酵母属(Millerozyma,0~8.60%)、镰刀菌属(Fusarium,0.10%~1.82%)、被孢霉属(Mortierella,0.05%~1.32%)、维希尼克氏酵母属(Vishniacozyma,0.21%)、粗糙孔菌属(Trechispora,0~1.62%)、链格孢属(Alternaria,0.04%~0.47%)、枝孢属(Cladosporium,0.06%~0.41%)。
酱醪中的曲霉是制曲期间人工引入的,是整个发酵过程的优势菌种,与文献报道相符[28]。除此之外酵母是主要的微生物。如图5-a,M组和H组的真菌菌群组成相似,与L组差异明显,其中假丝酵母属(Candida)和米勒氏酵母属(Millerozyma)含量在L组酱醪中显著提高。已有研究表明,在酱油酿造过程中,假丝酵母(Candida spp.)从开始的0.03%随着发酵周期的延长逐渐增加[30]。此外,有文献报道Millerozyma菌属在发酵中期出现并持续增长[29,31]。
2.4.5 不同盐浓度酱醪中真菌α-生物多样性分析
从图6中可见,L组样品在发酵5 d(I)、25 d(II)后,其系统发育多样性均值低于150 g/L(M)和170 g/L(H)盐分的样品,代表着菌群结构在系统发育树上有着较为亲密的关系。在observed species及Chao1分析中发现L组中在发酵初期(发酵5 d,I;发酵25 d,II)物种丰富度相比M组和H组酱醪较低。随着发酵周期的延长,各盐分浓度酱醪细菌组成逐步演变,物种丰富度和均匀度逐步增加(香农指数)。

a-PD整树(PD whole tree);b-观察物种(Observed species);c-物种丰富度(Chao1);d-香农指数(Shannon)
图6 基于62 450 reads水平不同盐浓度酱醪发酵过程的真菌α-多样性分析
Fig.6 Fungi α-diversity analysis based on 62 450 reads during the fermentation process of soy sauce mash with different salt concentrations
2.4.6 不同盐浓度酱醪中真菌β-生物多样性分析
在β-生物多样性方面,真菌保持着与细菌β-生物多样性相似的演变规律。图7表中通过Euclidean及Bray-Curtis差异矩阵算法得到了相同的趋势,即不同盐分酱醪在初始阶段(I,发酵5 d,红色)和25 d发酵(II,绿色)无法形成显著的差异,则在发酵55 d(III,黄色)后才形成了较明显的族群。
2.5 不同盐浓度酱醪发酵过程中微生物相对浓度与理化指标的关联
对总酸、pH值及细菌真菌丰度最高的3个菌科进行相关性分析。图8-a表明,发酵全程减盐条件(L、M)总酸含量相比高盐组(H)明显提高,L组总酸最高,pH值最低。L组酱醪中酵母菌目(Debaryo-mecetaceae)的含量在发酵初期就形成优势菌群并随着发酵时间的延长持续增长,在中期(发酵25 d,II)形成了显著的含量差异,并在发酵55 d(III)时,与M组和H组相对含量差异超过11%。而在科水平上,L组和M组在发酵前期(发酵第5天,I),细菌明串珠菌科(Leuconostocaceae)及乳杆菌科(Lactobacillaceae)的相对含量比H组更高,这可能是导致L组和M组总酸更高的原因。
a-Euclidean;b-Bray-Curtis
图7 不同盐浓度酱醪发酵过程的真菌β-生物多样性距离矩阵与NMDS分析
Fig.7 Fungi β-biodiversity distance matrix and NMDS analysis during the fermentation of sauce mash with different salt concentrations
结合L组展现较高的细菌α-多样性指数及较高的明串珠菌科(Leuconostocaceae)及乳杆菌科(Lactobacillaceae)相对含量可知,减盐酱醪由于较低的渗透压,无法对细菌,尤其是乳酸菌形成足够的压制,导致发酵前期总酸快速提升,pH值快速降低[24-25]。而这同时也为酵母菌快速形成了良好的生长环境[10-11],并在发酵中期(25 d)形成了明显的低真菌α-多样性的菌群结构(图6),对应为更高的假丝酵母属(Candida)和米勒氏酵母属(Millerozyma)含量(图5)。
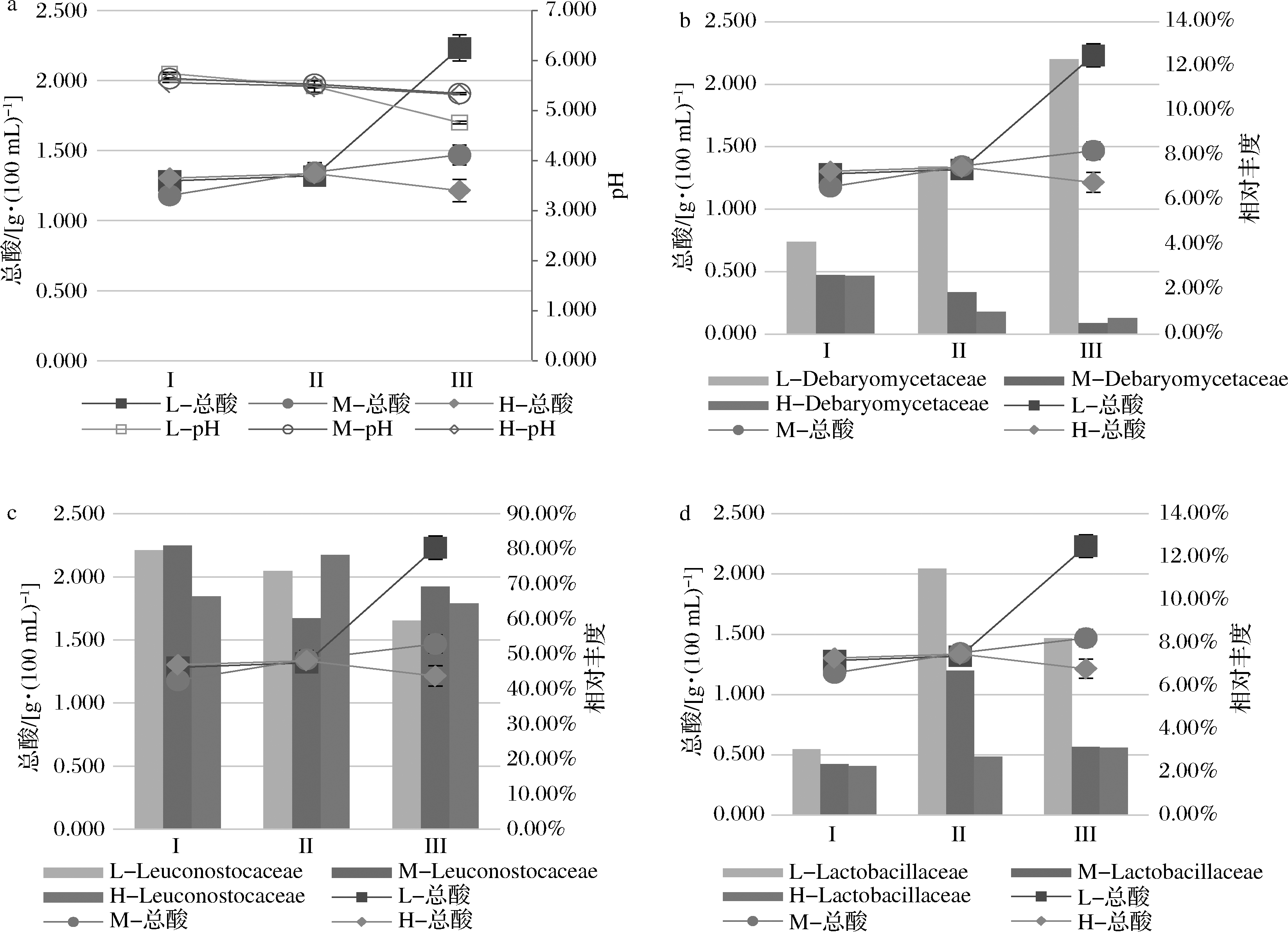
a-总酸与pH值;b-总酸与酵母菌目;c-总酸与明串珠菌科;d-总酸与乳杆菌科
图8 不同盐分及发酵周期中总酸、pH值及总酸与微生物的含量变化
Fig.8 Changes in total acid,pH and microbial relative abundance during the fermentation of sauce mash with different salt concentrations
3 结论
本研究基于微生物计数、理化指标及高通量基因序列分析发现低盐分酱醪体系中乳酸菌和真菌的总量更高,原油pH值更低,发酵后快速积累总酸、氨基酸态氮,消耗还原糖,同时乳杆菌(Lactobacillus)、葡萄球菌属(Staphylococcus)及乳球菌属(Lactococcus)含量在L组有明显的增生,拥有较高的α生物多样性。此外,假丝酵母属(Candida)和米勒氏酵母属(Millerozyma)含量在L组酱醪中明显较高,但真菌α多样性较M组和H组低。L组在发酵初期含有较高的乳酸菌含量,并快速形成了更加适合酵母菌目生长的酸性环境,导致酵母菌目在发酵中期形成了明显的优势菌群。该研究结果揭示了减盐发酵条件下酱油发酵微生物菌群结构演变规律,可为高盐稀态酱油减盐发酵过程中的微生物调控提供参考依据。
[1] 中国营养学会.中国居民膳食指南2016[M].北京:人民卫生出版社,2016:166-254.
Chinese Nutrition Society.Dietary Guidelines for Chinese Residents 2016[M].Beijing:People's Medical Publishing House,2016:166-254.
[2] 朱莉, 许长华.酱油关键风味物质及其功能与发酵工艺研究进展[J].食品与发酵工业,2018,44(6):287-292.
ZHU L,XU C H.Research advances on the key flavor substance ofsoysauce and its functions and fermentation technology[J].Food and Fermentation Industries,2018,44(6):287-292.
[3] 赵佳豪, 周韬,刘爽,等.中日两国酱油香味与滋味差异[J].食品与发酵工业,2020,46(17):232-236.
ZHAO J H,ZHOU T,LIU S,et al.Differences in the flavor and taste between Chinese and Japanese soy sauce[J].Food and Fermentation Industries,2020,46(17):232-236.
[4] 彭东, 蒋雪薇,陈幽,等.高盐稀态酱醪中耐盐生香酵母的筛选及生香特性研究[J].食品与发酵工业,2020,46(13):76-84.
PENG D,JIANG X W,CHEN Y,et al.Screening and aroma-producing characteristics of salt-tolerant aroma-producing yeasts from high-salt liquid-state Moromi[J].Food and Fermentation Industries,2020,46(13):76-84.
[5] 范智义, 邓维琴,李恒,等.传统发酵调味品低盐化的研究进展[J].中国调味品,2020,45(7):194-200.
FAN Z Y,DENG W Q,LI H,et al.Research progress on the desalination oftraditional fermented condiments[J].China Condiment,2020,45(7):194-200.
[6] CHRISTIAN J,SCOTT W J.Water relations of Salmonellae at 30 ℃[J].Australia Journal of Biological Science,1953,6(4):565-573.
[7] GIBSON A M,ROBERTS T A.The effect of pH,wateractivity,sodium nitrate and storage temperature on the growth of enteropathogenic Escherichia coli and Salmonellae in a laboratory medium[J].International Journal of Food Microbiology,1986,3(4):183-194.
[8] TAORMINA P J.Implications of salt and sodium reduction on microbial food safety[J].Critical Reviews in Food Science and Nutrition,2010,50(3):209-227.
[9] 包启安. 酱油科学与酿造技术[M].北京:中国轻工业出版社,2011:367-368.
BAO Q A.Soy Sauce Science and Brewing Technology[M].Beijing:China Light Industry Press,2011:367-368.
[10] GURTLER M,GANZLE M G,WOLF G,et al.Physiological diversity among strains of Tetragenococcus halophilus[J].Systematic and Applied Microbiology,1998,21(1):107-112.
[11] VAN DERSLUIS C,TRAMPER J,WIJFFELS R H.Enhancing and acceleratingflavour formation by salt-tolerant yeasts in Japanese soy-sauce processes[J].Trends in Food Science and Technology,2001,12(9):322-327.
[12] JANSEN M,VEURINK J H,EUVERINK G J W,et al.Growth of the salt-tolerant yeast Zygosaccharomyces rouxii in microtiter plates:Effects of NaCl,pH and temperature on growth and fusel alcohol production from branched-chain amino acid[J].FEMS Yeast Research,2003,3(3):313-318.
[13] SILVA-GRACA M,NEVES L,LUCAS C.Outlines for the definition of halotolerance/halophily in yeasts:Candida versatilis (halophila) CBS4019 as the archetype?[J].FEMS Yeast Research,2003,3(4):347-362.
[14] KURAMITSU R,SEGAWA D,NAKAMURA K,et al.Further studies on the preparation of low sodium chloride-containing soy sauce by using ornithyltaurine hydrochloride and its related compounds[J].Bioscience,Biotechnology and Biochemistry,1997,61(7):1 163-1 167.
[15] CHUN B H,KIM K H,JEONG S E,et al.The effect of salt concentrations on the fermentation of doenjang,a traditional Korean fermented soybean paste[J].Food Microbiology,2020,86:103329.
[16] BOLGER A M,LOHSE M,USADEL B.Trimmomatic:A flexible trimmer for Illumina sequence data[J].Bioinformatics,2014,30(15):2 114-2 120.
[17] MARTIN M.Cutadapt removes adapter sequences from high-throughput sequencing reads[J].EMBnet Journal,2011,17(1):10-12.
[18] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2011,26(19):2 460-2 461.
[19] EDGAR R C,HAAS B J,CLEMENTE J C,et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics,2011,27(16):2 194-2 200.
[20] QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNA gene database project:Improved data processing and web-based tools[J].Nucleic Acids Research,2013,41(D1):D590-D596.
[21] NILSSON R H,LARSSON K H,TAYLOR A F S,et al.The UNITE database for molecular identification of fungi:Handling dark taxa and parallel taxonomic classifications[J].Nucleic Acids Research,2019,47(D1):D259-D264.
[22] CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods,2010,7(5):335-336.
[23] HAMMER Ø S,HARPER D A T,RYAN P D.Past:Paleontological statistics software package for education and data analysis[J].Palaeontologia Electronica,2001,4(1):4-9.
[24] XIONG T,LI J B,LIANG F,et al.Effects of salt concentration on Chinese sauerkraut fermentation[J].LWT,2016,69:169-174.
[25] 杨希. 盐浓度对蚕豆酱发酵过程中原核微生物多样性及理化因子的影响[J/OL].食品与发酵工业,2021.https://doi.org/10.13995/j.cnki.11-1802/ts.027997.
YANG X.Effects of salt concentration on prokaryotic microbial diversity and physicochemical properties during broad bean paste fermentation[J/OL].Food and Fermentation Industries,2021.https://doi.org/10.13995/j.cnki.11-1802/ts.027997.
[26] 何炯灵, 张鉴平,姜佳丽,等.盐水浓度对酱油发酵过程中理化指标的影响[J].中国酿造,2016,290:34-37.
HE J L,ZHANG J P,JIANG J L,et al.Effects of brine concentration on physicochemical indexes during soy sauce fermentation[J].China Brewing,2016,35(4):34-37.
[27] JIA Y,NIU C T,LU Z M,et al.A bottom-up approach to develop a synthetic microbial community model:Application for efficient reduced-salt broad bean paste fermentation[J].Applied and Environmental Microbiology,2020,86(12):e00306-20.
[28] 刘婷婷, 蒋雪薇,周尚庭,等.高盐稀态发酵与低盐固态发酵酱油中次生菌群分析[J].食品与机械,2010,26(6):13-17.
LIU T T,JIANG X W,ZHOU S T,et al.Comparative analysis of the secondary microorganism constitutes in soy sauce fermentation between high-salt-level watery state and low-salt-level solid state technology[J].Food and Machinery,2010,26(6):13-17.
[29] WEI Q Z,WANG H B,CHEN Z X,et al.Profiling of dynamic changes in the microbial community during the soy sauce fermentation process[J].Applied Microbiology and Biotechnology,2013,97(20):9 111-9 119.
[30] SULAIMAN J,GAN H M,YIN W F,et al.Microbial succession and the functional potential during the fermentation of Chinese soy sauce brine[J].Frontiers in Microbiology,2014,5:556.
[31] HAN D M,CHUN B H,FENG T,et al.Dynamics of microbial communities and metabolites in ganjang,a traditional Korean fermented soy sauce,during fermentation[J].Food Microbiology,2020,92:103591.