戊二胺是杀菌剂、药物、织物柔软剂、可生物降解的塑料以及添加剂等的主要原料[1-2]。在工业生产中,戊二胺可作为高分子合成前体物质,通过聚合反应形成新的聚酰胺酯。目前,全球每年对聚酰胺产品需求量逐年上涨,但其主要合成方式依赖于以石油为原料的化工合成[3]。使用廉价且可再生原料(如葡萄糖、L-赖氨酸等)通过微生物法生产戊二胺已成为当前研究的热点[4-5]。先前已经设计了许多天然高L-赖氨酸/氨基酸生产菌株,特别是谷氨酸棒杆菌和大肠杆菌[6-7]。
氨基酸脱羧酶广泛应用于各种生物胺的合成,氨基酸脱羧酶主要分为2大类:磷酸吡哆醛(pyridoxal phosphate,PLP)依赖型和丙酮酸依赖型。PLP依赖型酶通常催化多种反应,其活性位点内的赖氨酸残基高度保守,从而促进其与辅因子PLP结合[8]。赖氨酸脱羧酶是催化L-赖氨酸脱羧生成戊二胺关键酶,其需要PLP作为辅因子。大肠杆菌赖氨酸脱羧酶ldcC通过破坏lysE并通过强合成启动子H30调节其表达,通过整合在谷氨酸棒杆菌PKC染色体上,从而稳定表达,经分批补料,产量约103.78 g/L戊二胺[9]。WANG等[10]应用定向进化,易错PCR诱变和DNA改组来改造H.alvei赖氨酸脱羧酶,在全细胞生物转化5 h后实现了63.9 g/L尸胺的生产,是原始菌株的1.48倍。KOU等[11]通过突变十聚体界面残基T88S,获得了一种具有良好热稳定性和pH稳定性的大肠杆菌赖氨酸脱羧酶变体。
脱羧酶应用广泛,但是由于反应的多样性,它们的工业应用仍然相对有限。因此,探索具有高效生物催化活性和生化特性(如热稳定性,独特的底物特异性)的脱羧酶已成为工业化合物生物合成的研究热点。目前,L-赖氨酸脱羧酶存在酶活力不高、稳定性较差、生物合成体系效率低的问题。本研究中,我们从不同的微生物中筛选并表征了4个L-赖氨酸脱羧酶,由于粘质沙雷氏菌来源的新型赖氨酸脱羧酶(SmcadA)具有相对较高的生物催化活性和良好的工业属性,对其活性位点附近氨基酸残基进行了定点突变,显著提高了其催化合成戊二胺的能力,为其工业化生产戊二胺提供了实验和理论基础。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
大肠杆菌Escherichia coli BL21(DE3)和质粒pMD18T、pET28a由本实验室保藏。
1.1.2 酶和试剂
BamH I、Hind III限制性核酸内切酶、DNA聚合酶等,TaKaRa公司;高保真酶、同源重组酶克隆试剂盒,南京诺维赞生物科技有限公司;琼脂糖凝胶DNA回收试剂盒、小量质粒提取试剂盒、细菌DNA基因组提取试剂盒,上海捷瑞生物工程有限公司;卡那霉素、氨苄青霉素,生工生物工程(上海)股份有限公司;L-Lys,麦克林(北京)股份有限公司;戊二胺标准品及其他分析纯试剂,Sigma公司。
1.1.3 培养基
LB液体培养基(g/L):酵母粉5.0,胰蛋白10.0,NaCl 10.0。
LB固体培养基(g/L):酵母粉5.0,胰蛋白栋10.0,NaCl 10.0,琼脂粉20。
1.2 实验方法
1.2.1 质粒提取和引物的设计
本文中涉及到的引物如表1所示。
表1 本研究使用到的引物
Table 1 Primers used in the study

引物名称引物序列(5′-3′)长度/bppET28a-SmcadA-FCCATCATCACCACAGCCAGGATCCATGAACGTTATC36pET28a-SmcadA-RCTTAAGCATTATGCGGCCGCAAGCTTTTATTT CGCCTTC39PpLDC-F Hind IIIGGCCGCAAGCTTTCACGCGCATTCCGTG28PpLDC-R BamH IGTCGCGGATCCATGTACAAGGACCTCAAGTTCC33GoLDC-F Hind IIIGCCGCAAGCTTTCAGCGCCCTGTTC25GoLDC-R BamH IGGTCGCGGATCCATGGCCGGACTGG25BlLDC-F EcoR ICTCGAATTCTCATGATTTCTCCTCTTCTATATATACAG38BlLDC-R BamH IGGTCGCGGATCCATGCAGAATGTCATATCGGC32Gly348Ala-FAGGCAAGTGCGCGATGAGCGGCG23Gly348Ala-RCGCCGCTCATCGCGCACTTGCCT23
注:下划线为限制性内切酶位点,粗体为突变位点
1.2.2 重组菌株的构建
分别以粘质沙雷氏菌Db11、氧化葡糖杆菌DSM 3504、恶臭假单胞菌KT2440、蜡状芽孢杆菌ATCC4342和地衣芽孢杆菌DSM13基因组DNA为模板,利用表1中列出的相关引物扩增赖氨酸脱羧酶编码基因。将扩增所得基因连接到事先用相应的限制酶消化的pET28a表达载体上,然后转化到E.coli BL21(DE3)感受态细胞,得到重组菌株分别命名为BL21/pET28a-SmcadA、BL21/pET28a-GoLDC、BL21/pET28a-PpLDC、BL21/pET28a-BcLDC和BL21/pET-28a-BlLDC。通过双酶切和测序验证转化是否成功。
1.2.3 SmcadA表达和纯化
挑选单菌落并接种于含有25 μg/mL氨苄青霉素的10 mL LB培养基中,在37 ℃、180 r/min下过夜培养。然后将1%的过夜培养物转移至50 mL含25 μg/mL氨苄青霉素的LB培养基中,在37 ℃、180 r/min下培养2 h,然后添加异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactopyranoside,IPTG)诱导,在28 ℃、180 r/min培养16 h。发酵培养后,在4 ℃下以8 000 r/min离心10 min,收集细胞,然后用0.01 mol/L PBS缓冲液(pH 7.4)洗涤后超声处理15 min,然后以10 000 r/min离心20 min收集上清酶液。使用配备GE Life Sciences公司的His-TrapTM HP色谱柱的AKTA Prime系统(瑞典GE Healthcare)纯化蛋白。将粗酶(5 mL)以0.5 mL/min的流速上样到色谱柱上,结合缓冲液为pH 7.4的0.02 mol/L Tris-HCl缓冲液和0.5 mol/L NaCl组成,然后在1 mL/min流速下以0~0.5 mol/L咪唑线性梯度洗脱。最后,使用pH 7.0的0.05 mol/L Tris-HCl缓冲液通过透析从纯化的蛋白中去除过量的咪唑[12]。纯化后的酶用于酶活性测定,并用十二烷基硫酸钠-聚丙烯酰氨凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)分析其纯度,剩余样品用于后续酶学性质表征实验。
1.2.4 催化合成戊二胺
将培养的发酵液离心,倒去上清液,收集菌体细胞,用50 mmol/L磷酸钾缓冲液(pH 7.4)洗涤2次,然后将收集到的细胞悬浮在50 mmol/L乙酸钠缓冲液中,并用1 mol/L HCl调节初始pH。反应混合物由500 mmol/L乙酸钠缓冲液、0.5 mol/L L-赖氨酸、1.5 mmol/L PLP和200 μL细胞(最终OD600 nm=10)组成,并添加优化的十六烷基三甲基溴化铵。将反应混合物在37~80 ℃下孵育1 h,然后将反应混合物在100 ℃加热5 min终止酶反应。最后,将样品以10 000 r/min离心20 min获得上清液,以备HPLC分析。
1.2.5 产物分析
对样品进行衍生化和分析,用乙氧基亚甲基丙二酸二乙酯(diethyl ethoxymethylidenemalonate,DEEMM)衍生L-赖氨酸和尸胺,衍生化体系包括:75 μL反应混合物样品、4.5 μL DEEMM、70.5 μL蒸馏水、150 μL 100%甲醇和450 μL 50 mmol/L硼酸盐缓冲液(pH 9.0)。将衍生化后混合物在涡旋混合仪中剧烈混合,然后在70 ℃加热2 h以去除多余的DEEMM和副产物。随后,将样品以10 000 r/min离心5 min后,用0.22 μm膜过滤,然后用HPLC系统(Platisil 5 μm ODS色谱柱,250 mm×4.6 mm)进行分析。柱温度设定在35 ℃,流动相由100%乙腈(A)和25 mmol/L乙酸钠水溶液(pH 4.8)(B)组成,流速1 mL/min,溶剂梯度如下:0~2 min,20%~25%A;2~20 min,25%~60%A;20~25 min,60%~20%A。
1.2.6 位点定向诱变
HotSpot Wizard 3(https://loschmidt.chemi.muni.cz/hotspotwizard)是以预测突变热点功能和进化数据为基础,而在蛋白质结构/功能上替换氨基酸是由在线工具PredictSNP(http://loschmidt.chemi.muni.cz/predictsnp)[10]和Mutpred2[11](http://mutpred2.mutdb.org)进行操作。将SmcadA与同源赖氨酸脱羧酶进行序列比对,在DNAMAN上验证突变残基。与主流的OE-PCR技术相比,用改良的组合重叠扩展PCR方法对重组蛋白进行定点突变是一种快速且准确对相关目的基因进行点突变的方法。简单地说,该方法涉及2个标准PCR反应:一级反应和从一级反应中提取的PCR扩增物作为二级反应模板。第一反应包含100 ng DNA模板,超保真DNA聚合酶2×Phanta Max Master mix 25 μL,引物1 μL,并加蒸馏水至50 μL。第一反应混合物,正向引物和反向引物见表1。
使用PCR程序如下:95 ℃初始变性5 min,95 ℃ 30 s,60 ℃退火30 s,72 ℃聚合1.5 min,72 ℃保持5 min。PCR产物(10 μL)在1%琼脂糖凝胶上用溴化乙锭染色。以pET28a-SmcadA-F和pET28a-SmcadA-R分别作为正向引物和反向引物进行2次PCR反应,每一个初级PCR产物(200 ng)作为模板。PCR程序的使用是相同的,除了退火步骤涉及10个周期,不同于初级阶段。2次PCR结果在1%琼脂糖凝胶上用溴化乙腈染色。将突变片段克隆到表达质粒中,转化到宿主细胞中,测序,核实测序结果证实构建成功。
1.2.7 定点突变及分子动力学分析
在引物中引入突变碱基,通过2轮PCR将突变位点引入到基因中,将第一轮PCR获得的2种PCR产物在第二轮PCR反应中用作模板,PCR反应条件如OSIRE等[12]所述。为了解定点突变对蛋白与底物相互作用的机制,在Gromacs中对SmcadA及其突变体与底物L-赖氨酸进行了分子动力学模拟[13]。通过添加氢离子和最小化共轭梯度,对蛋白分子(野生型和突变型)和赖氨酸底物结构单元进行了在线(https://www.swissparam.ch)分子对接。分析了主链均方根偏差(root mean squared deviation,RMSD)和蛋白质残基均方根波动(root mean squared fluctuation,RMSF)值,并在USCF Chimera软件和Graphpad Prism 8中评估了潜在的蛋白质-底物相互作用,并绘制图形说明。
2 结果与讨论
2.1 重组菌构建与表达
分别以氧化葡糖杆菌DSM3504(GoLDC)、地衣芽孢杆菌DSM13(BlLDC)、恶臭假单胞菌KT2440(Pp LDC)和粘质沙雷氏菌W2.3(SmcadA)的基因组为模板,利用表1中的引物,通过PCR扩增不同来源的赖氨酸脱羧酶编码基因,分别克隆到pET28a中,转化至大肠杆菌BL21。菌落PCR和基因测序验证重组菌构建成功。并在Expasy ProtParam网络服务器(https://web.expasy.org/protparam)进行蛋白质序列分析。
同源的赖氨酸脱羧酶蛋白序列,GoLDC,BlLDC,PpLDC和SmcadA蛋白的理论等电点(pI)为5.69、5.96、6.13和5.92,分子质量分别约为51.28 k、55.78 k、81.95 k和81.11 kDa。重组菌诱导表达16 h后,利用SDS-PAGE分析蛋白表达情况及其分子质量。图1结果表明,4种来源的赖氨酸脱羧酶均在大肠杆菌BL21中成功表达,BlLDC和GoLDC的分子质量为55和50 kDa,而PpLDC和SmcadA的分子质量约为75 kDa。
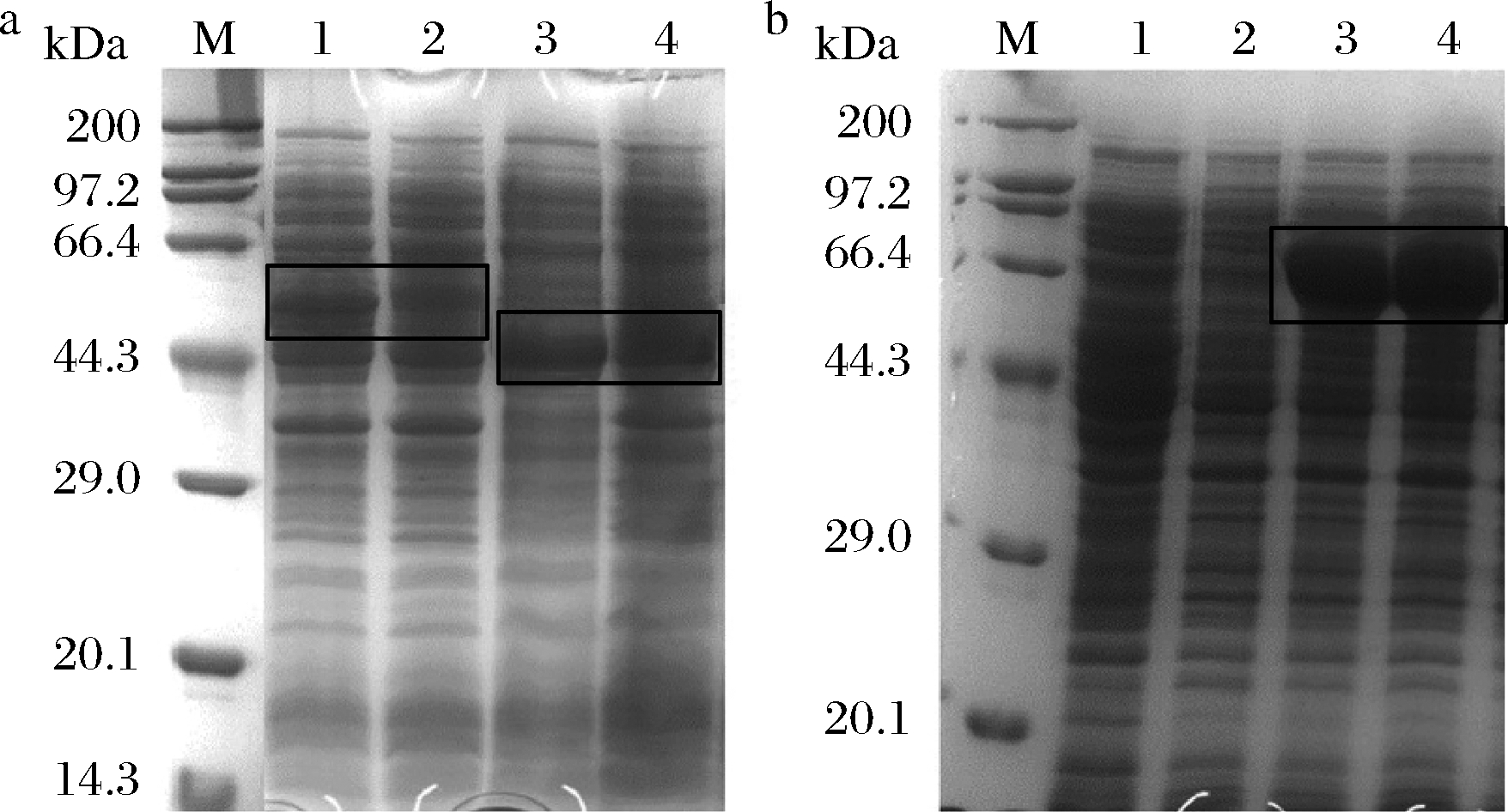
图1 SDS-PAGE表征蛋白表达
Fig.1 Protein expression analysis on SDS-PAGE a图:M-蛋白Marker;泳道1,2-E.coli BL21/pET28a-BlLDC细胞破碎上清液;泳道3,4-E.coli BL21/pET28a-GoLDC细胞破碎上清液;b图:M-蛋白Marker;泳道1-E.coli BL21细胞破碎上清液;泳道2-E.coli BL21/pET28a细胞破碎上清液;泳道3,4-分别是E.coli BL21/pET28a-PpLDC和E.coli BL21/pET28a-SmcadA细胞破碎上清液
2.2 赖氨酸脱羧酶酶学特性
赖氨酸脱羧酶酶活力受pH、温度、底物和辅因子浓度的影响[14]。因此,为了探究重组赖氨酸脱羧酶将L-赖氨酸转化为戊二胺的最佳条件,考察了其pH在4.0~10.0和温度在30~80 ℃下的相对酶活力。结果如图2所示,GoLDC和PpLDC在pH为9.0时均具有最高活性,SmcadA在pH为6.0时活性最高,而BlLDC在中性pH为7.0时具有最高活性。另外,GoLDC、PpLDC和SmcadA在5.0~9.0的pH范围内均具有较高活性。GoLDC和PpLDC的最适温度均为30 ℃,SmcadA的最适温度为40 ℃,而BlLDC的最适温度为60 ℃。
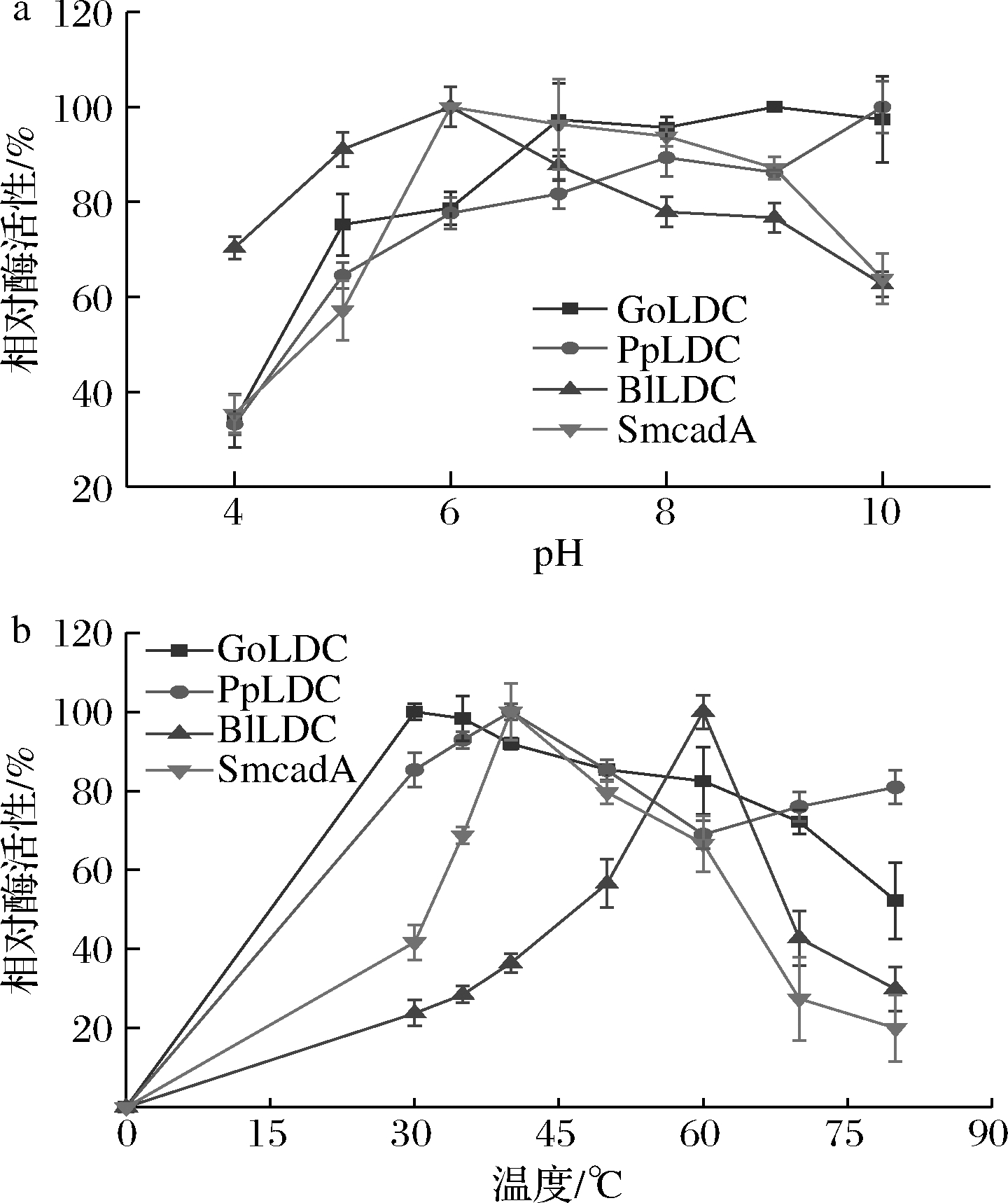
a-pH;b-温度
图2 pH和温度对赖氨酸脱羧酶活性的影响
Fig.2 Effects of pH and temperature on the activities of lysine decarboxylase 注:最适条件下比酶活力分别是PpLDC 201.79 U/mg、BlLDC 106.60 U/mg、GoLDC 143.76 U/mg和SmcadA 219.60 U/mg
2.3 全细胞催化合成戊二胺
反应体系包含:全细胞、500 mmol/L邻乙酸钠缓冲液、0.5 mol/L L-赖氨酸和辅因子PLP。然后将混合物在其各自的最佳条件下反应,每1 h回收细胞循环使用,结果如图3所示。
由图3可知,12 h后,含有来自粘质沙雷氏菌(SmcadA)LDC的重组菌株具有最高的赖氨酸转化率,转化率为98.1%,PpLDC为87.7%,GoLDC为71.0%,BlLDC为63.7%(图3-a)。图3-b结果还表明,利用固定后的SmcadA重组菌体进行转化,尸胺的产量为159.24 g/L,是未固定的SmcadA(cadA-Alg)的1.29倍(123.44 g/L)。由于SmcadA重组菌株具有很高的生物催化活性,因此对该菌株进行了进一步的工程设计和优化。
2.4 定点突变
前期研究发现,辅因子PLP通过与蛋白质残基和金属离子通过氢键和范德华力的直接/间接相互作用,改变了结合位点内残基的构象,并改变了辅助因子PLP结合方向,对SmcadA活性起关键调节作用[13],这些因此显著影响其生物催化活性。此外,KANJEE等[15]成功获得了LDC酶活力明显提高的单点突变体R206S。
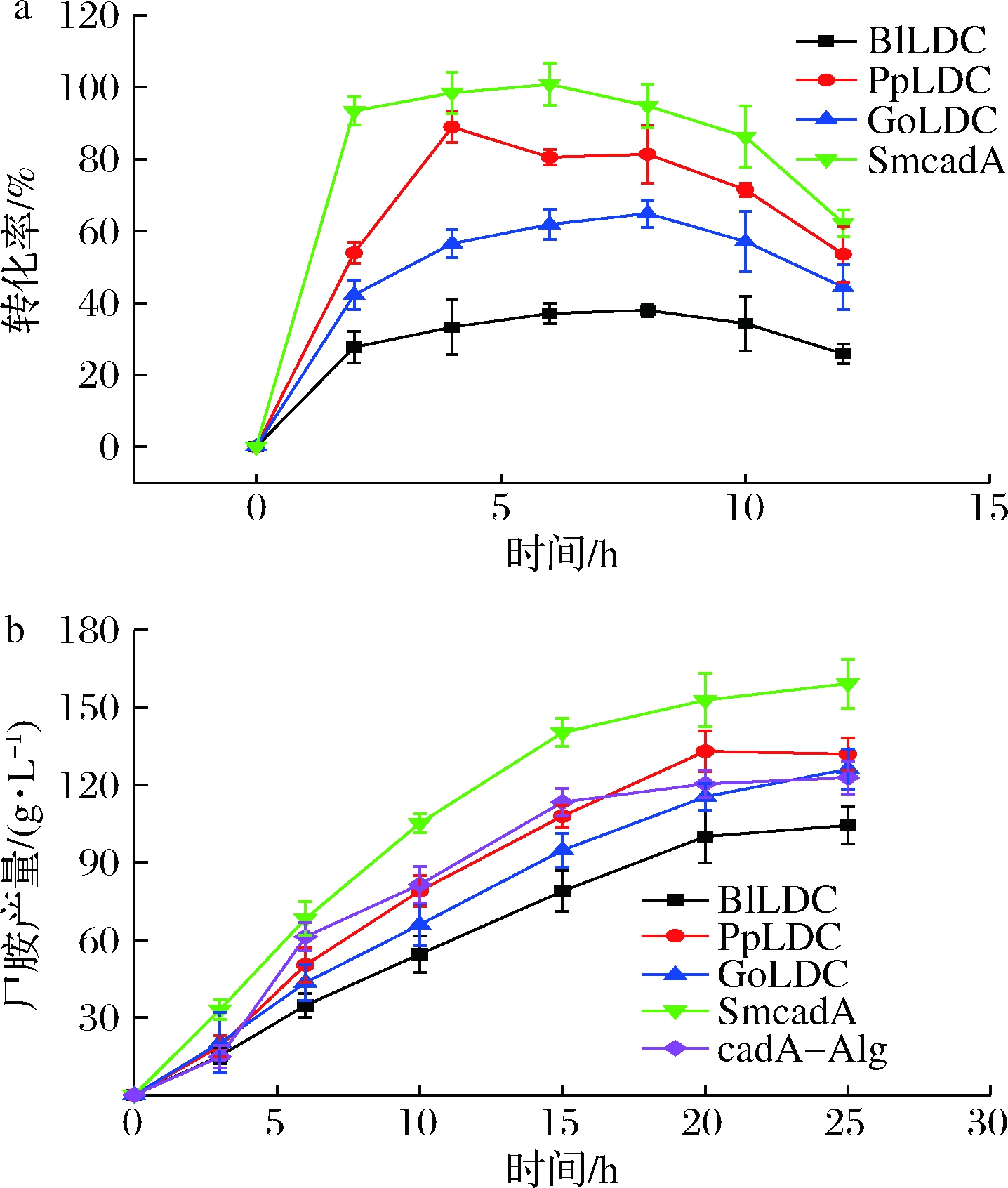
a-GoLDC、BlLDC、PpLDC和SmcadA尸胺的合成情况; b-尸胺在25 h内的生物合成情况
图3 尸胺的生物合成和优化
Fig.3 Cadaverine biosynthesis and optimization
利用在线工具HotSpot Wizard 3.0生成可行的突变热点。一些氨基酸残基被预测参与底物结合从而影响最终的产物合成。其中Gly348、Ala301、Pro530、Lys325和Val384预测为稳定性热点。对其进行定点突变,筛选尸胺合成效率提高和热稳定性提高的正向突变株。进一步进行蛋白质序列分析以确定同源相似性,并利用DAMAN分析软件来评估Gly348预测的可变区域的4个脱羧酶之间的进化差异。结果表明,GoLDC和SmcadA含有丙氨酸和甘氨酸残基,而BlLDC和PpLDC在相同位点具有赖氨酸残留物(图4)。
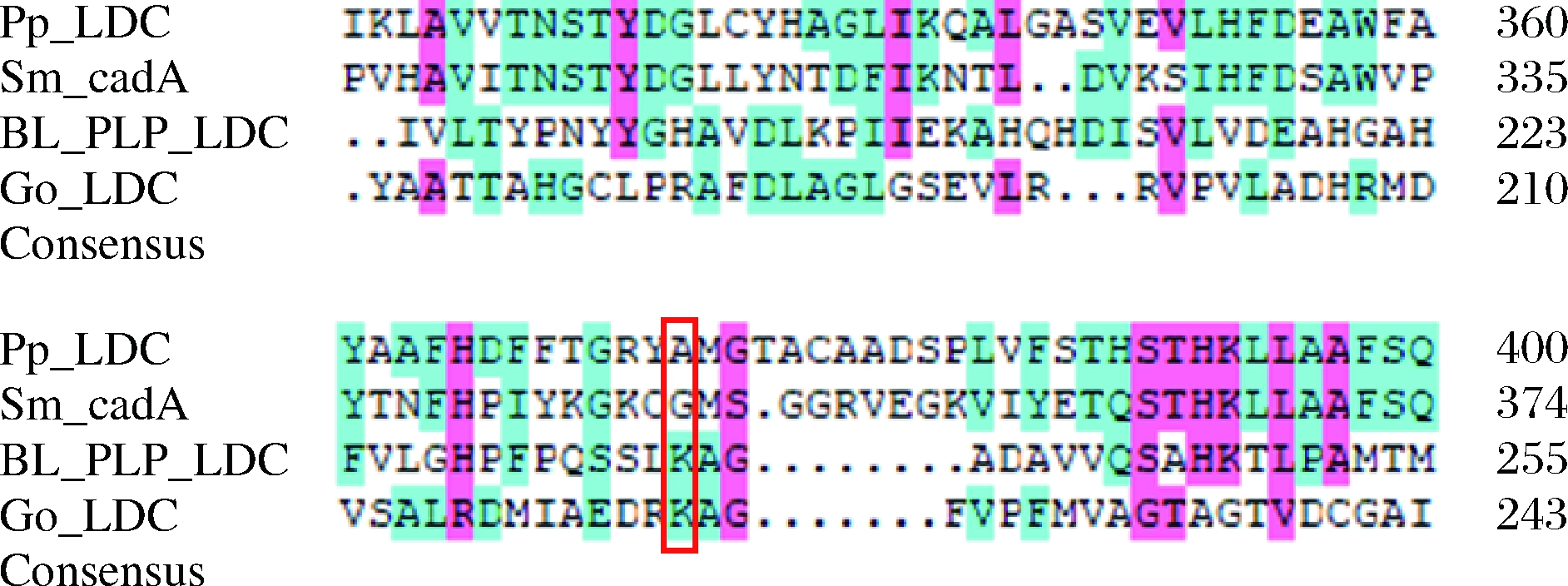
图4 蛋白质同源性分析
Fig.4 Protein homology analysis 注:Pseudomonas putida KT2440来源的赖氨酸脱羧酶简写为PpLDC;Serratia marcescens W2.3来源的赖氨酸脱羧酶简写为SmcadA,Bacillus licheniformis DSM 13来源的赖氨酸脱羧酶简写为BlLDC,Gluconobacter oxydans DSM 3504 来源的赖氨酸脱羧酶简写为GoLDC
2.4.1 突变体结构解析
本研究中,我们对活性位点内的关键残基Gly348进行了定点突变,提高了脱羧活性。在最佳条件下进行生物转化,突变菌株Gly348Ala在25 h内可转化320 g/L的L-赖氨酸生成218.16 g/L的戊二胺,转化率达到97.26%,生产效率达到8.73 g/(L·h)。这与HAN等对底物结合位点附近的负电荷残基进行点突变是一致的,他们发现底物结合位点对于L-赖氨酸转化生成尸胺具有重要影响。
为了解析突变株Gly348Ala较野生型SmcadA具有更好生物催化性能的原因,利用底物L-赖氨酸对这2种蛋白质模型进行了分子动力学模拟。模拟结果显示,突变株Gly348Ala中α氨基的赖氨酸与Arg558残基通过氢键相互作用,而原始菌株中,底物与周围金属离子直接形成氢键(图5)。此外,赖氨酸残基的羧基与Glu202形成氢键相互作用。除了一系列的交互Arg97和Glu74,先前的报道强调了Arg在蛋白质活性中的关键作用,这是因为强烈的相互作用力破坏活性位点残基的构象[16]。精氨酸胍基侧链具有特殊的水化特性,促进与活性位点内其他残基的非极性芳香族侧链和脂肪族侧链相互作用,在我们的模拟中,底物明显与Leu562、Leu564和Glu75形成范德华力相互作用[17]。

图5 Gly348Ala突变体的分子动力学模拟
Fig.5 Molecular dynamics simulations of Gly348Ala mutant 注:突变体Gly348Ala中蛋白质残基、底物L-赖氨酸和金属离子之间的相互 作用。青色棒代表氢键,紫色球代表金属离子,绿色球代表水分子(下同)。
另外,由于Arg残基的柔性性质,它容易与不同的底物相互作用。如先前报道的酶活性位点内,Arg558和Arg97通过形成盐桥从而促进酶与底物的相互作用[18]。据报道在某些PLP依赖性脱羧酶中,高度保守的Glu残基可通过氢键或盐桥与PLP吡啶环氮相互作用。结果表明,Glu202与底物L-赖氨酸的羧酸酯基团之间存在独特的氢键相互作用,并且这些相互作用可能会增强Gly348Ala突变菌株的生物催化活性。
在野生型SmcadA中,Lys134和Leu129与底物L-赖氨酸形成直接键(图5)。赖氨酸残基在脱羧酶活性方面的作用以前已有报道。赖氨酸负责形成希夫碱,促进脱羧酶催化反应中的转醛缩[19]。TONEY等[20]报道,Lys残基在质子转移中也起重要作用[21]。另一方面,亮氨酸残基被认为是恶臭假单胞菌3,4-二羟基苯基丙氨酸脱羧酶物特异性低的原因。
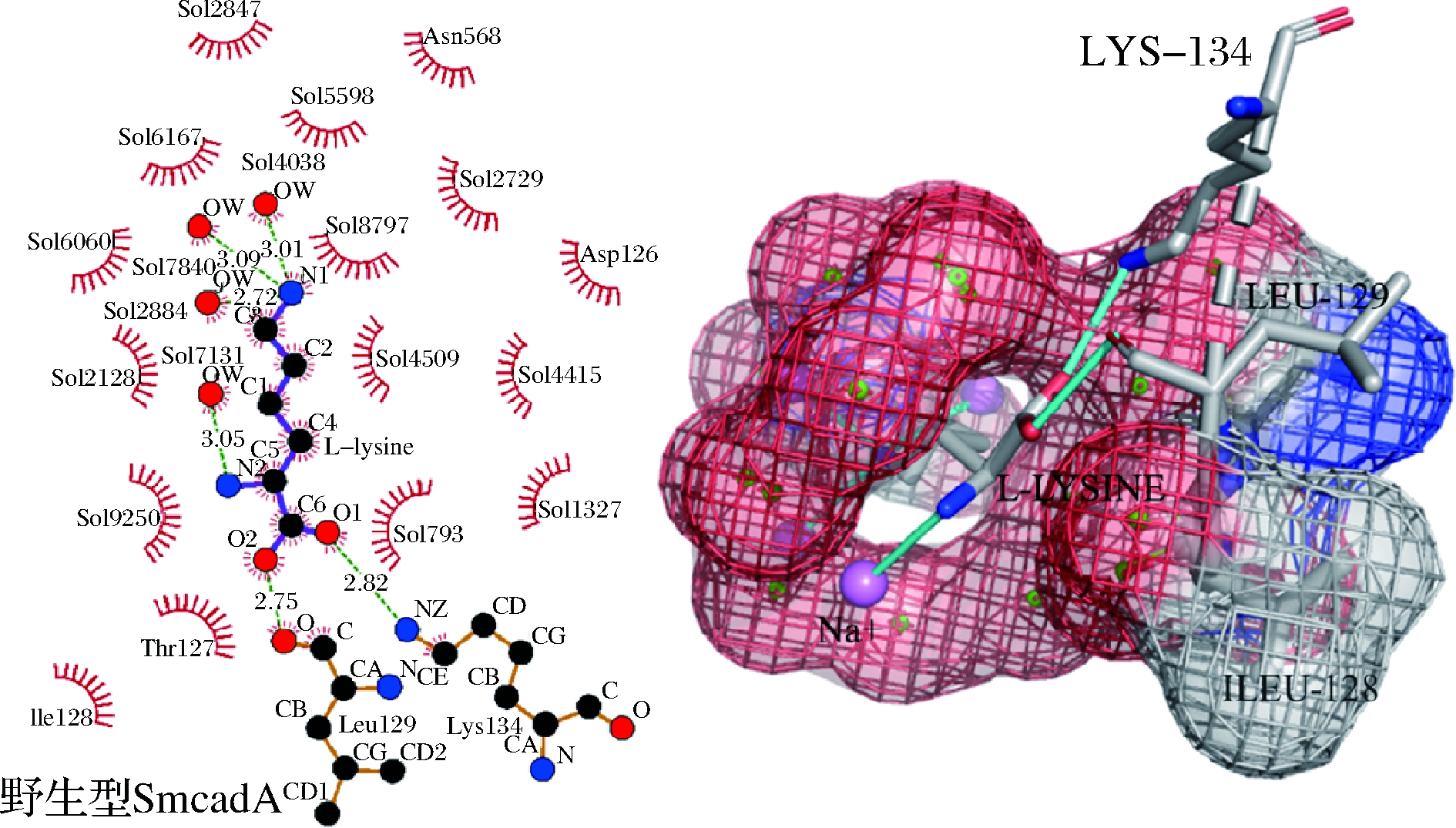
图6 野生型SmcadA的分子动力学模拟
Fig.6 Molecular dynamics simulations of SmcadA wildtype
活性位点上疏水残基原则上可以增强SmcadA的结构稳定性和底物结合亲和力。然而,菌株细胞的低活性可归因于酶的低活性,这可能是由于残基较大的侧链导致进入活性位点通道受限。如图7所示,RMSD和RMSF表明,与野生型SmcadA相比,Gly348Ala关键区域的柔韧性有所提高,这也是该突变体的催化活性提高的原因之一。
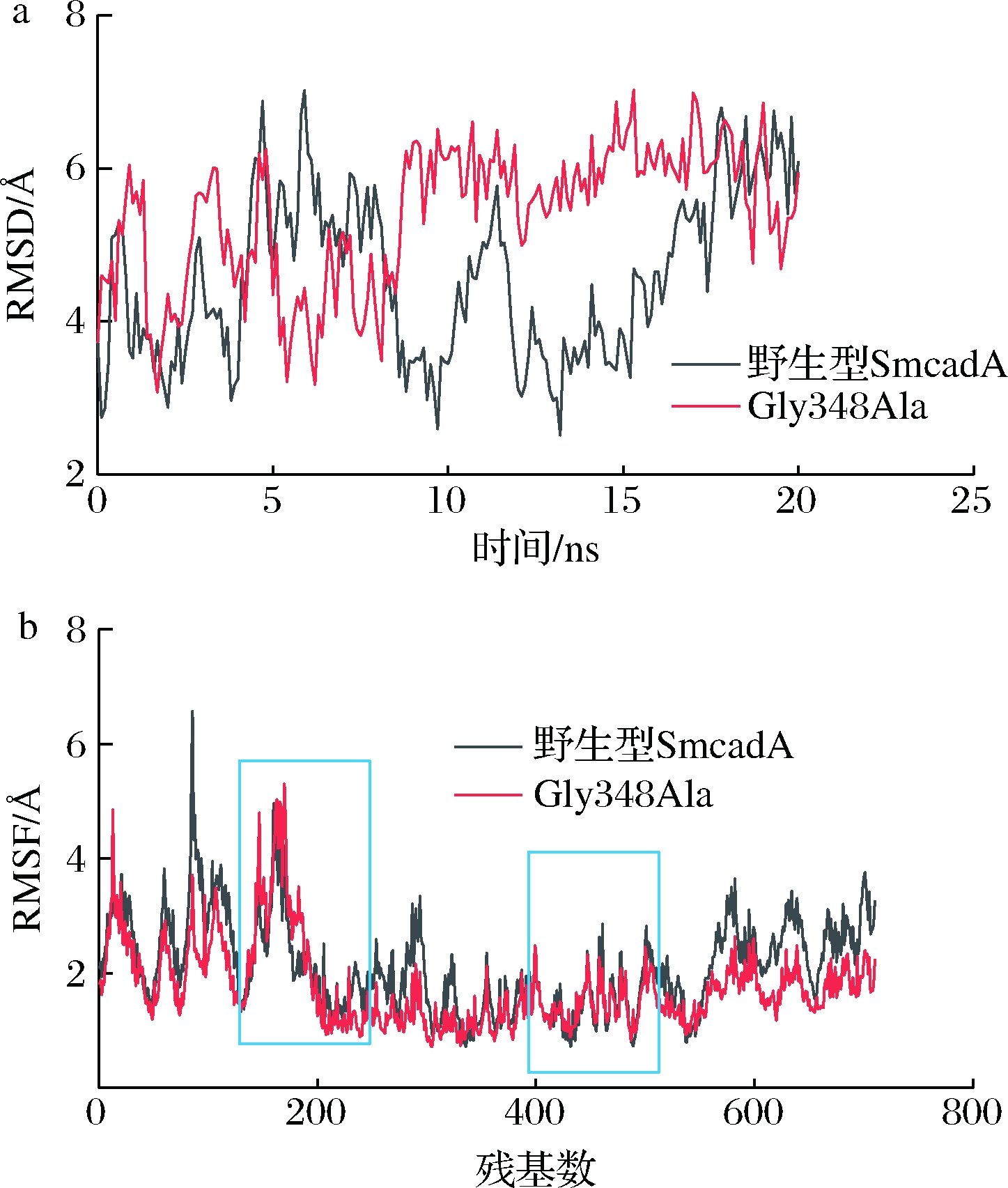
a-野生酶及突变酶的RMSD值;b-野生酶及突变酶的RMSF值
图7 蛋白质结构稳定性
Fig.7 Protein structure stability
3 结论与讨论
在本研究中,筛选和表征了4种不同微生物来源的赖氨酸脱羧酶,发现来源于粘质沙雷氏菌的SmcadA具有较好的戊二胺合成活性。为了进一步提高SmcadA脱羧活性,通过结构分析,确定对活性位点附近的Gly348位点进行定点突变,获得了活性明显提高的突变体Gly348Ala。为了进一步解析Gly348Ala突变脱羧活性提高的机理,将底物L-赖氨酸分别与SmcadA和突变体进行了分子动力学模拟,结果表明,突变蛋白中残基和底物之间的相互作用得到改善。Gly348Ala突变体与Arg558和Glu202形成了2个直接氢键,与周围的金属离子形成了另外5个氢键,而野生型SmcadA只有6个直接氢键,其中2个与Leu129和Lys134形成,其余与金属离子形成。突变菌株Gly348Ala在25 h内可转化320 g/L的L-赖氨酸生成218.16 g/L的戊二胺,转化率达到97.26%,生产效率达到8.73 g/(L·h),比野生型SmcadA提高了1.37倍。该研究结果为戊二胺的工业化生产应用提供了实验和理论基础,具有重要的参考价值。接下来,探究通过优化底物、辅因子需求以及调控氧气供应,进一步提高戊二胺合成效率,将是我们下一步工作的重点。
[1] MENG S Y,BENNETT G N.Nucleotide sequence of the Escherichia coli cad operon:A system for neutralization of low extracellular pH[J].Journal of Bacteriology,1992,174(8):2 659-2 669.
[2] TATENO T,OKADA Y,TSUCHIDATE T,et al.Direct production of cadaverine from soluble starch using Corynebacterium glutamicum coexpressing α-amylase and lysine decarboxylase[J].Applied Microbiology and Biotechnology,2009,82(1):115-121.
[3] LI X,LI L,CAO C,et al.Research progress of bio-based polyamides and their monomers[J].Engineering Plastics Application,2018,46(7):138-141.
[4] MA W C,CAO W J,ZHANG H,et al.Enhanced cadaverine production from L-lysine using recombinant Escherichia coli co-overexpressing CadA and CadB[J].Biotechnology Letters,2015,37(4):799-806.
[5] OH Y H,KANG K H,KWON M J,et al.Development of engineered Escherichia coli whole-cell biocatalysts for high-level conversion of L-lysine into cadaverine[J].Journal of Industrial Microbiology & Biotechnology,2015,42(11):1 481-1 491.
[6] KIND S,JEONG W K,SCHRÖDER H,et al.Systems-wide metabolic pathway engineering in Corynebacterium glutamicum for bio-based production of diaminopentane[J].Metabolic Engineering,2010,12(4):341-351.
[7] PARK S J,KIM E Y,NOH W,et al.Synthesis of nylon 4 from gamma-aminobutyrate(GABA) produced by recombinant Escherichia coli[J].Bioprocess and Biosystems Engineering,2013,36(7):885-892.
[8] LIANG J,HAN Q,TAN Y,et al.Current advances on structure-function relationships of pyridoxal 5′-phosphate-dependent enzymes[J].Frontiers in Molecular Biosciences,2019,6:4.
[9] KIM H T,BARITUGO K A,OH Y H,et al.Metabolic Engineering of Corynebacterium glutamicum for the high-levelproduction of cadaverine that can be used for the synthesis of biopolyamide 510[J].Acs Sustainable Chemistry & Engineering,2018,6(4):5 296-5 305.
[10] WANG C,ZHANG K,CHEN Z J,et al.Directed evolution and mutagenesis of lysine decarboxylase from Hafnia alvei AS1.1009 to improve its activity toward efficient cadaverine production[J].Biotechnology and Bioprocess Engineering,2015,20(3):439-446.
[11] KOU F Y,ZHAO J,LIU J,et al.Enhancement of the thermal and alkaline pH stability of Escherichia coli lysine decarboxylase for efficient cadaverine production[J].Biotechnology Letters,2018,40(4):719-727.
[12] OSIRE T,YANG T W,XU M J,et al.Lys-Arg mutation improved the thermostability of Bacillus cereus neutral protease through increased residue interactions[J].World Journal of Microbiology and Biotechnology,2019,35(11):173-184.
[13] OSIRE T,YANG T W,XU M J,et al.Integrated gene engineering synergistically improved substrate-product transport,cofactor generation and gene translation for cadaverine biosynthesis in E.coli[J].International Journal of Biological Macromolecules,2021,169:8-17.
[14] LEMKUL J A.From proteins to perturbed hamiltonians:A suite of tutorials for the GROMACS[J].Living J Comp Mol Sci,2019,1(1):1-53.
[15] KANJEE U,GUTSCHE I,ALEXOPOULOS E,et al.Linkage between the bacterial acid stress and stringent responses:The structure of the inducible lysine decarboxylase[J].The EMBO Journal,2011,30(5):931-944.
[16] LI L B,VOROBYOV I,ALLEN T W.The different interactions of lysine and arginine side chains with lipid membranes[J].The Journal of Physical Chemistry.B,2013,117(40):11 906-11 920.
[17] ARMSTRONG C T,MASON P E,ANDERSON J L R,et al.Arginine side chain interactions and the role of arginine as a gating charge carrier in voltage sensitive ion channels[J].Scientific Reports,2016,6:21759.
[18] MANTA B C,CASSIMJEE K E,HIMO F.Quantum chemical study of dual-substrate recognition in ω-transaminase[J].ACS Omega,2017,2(3):890-898.
[19] FUKUI T,SODA K.Molecular aspects of enzyme catalysis.Alanine racemase:Structure and function[J].Wiley-Blackwell,2007,8(1):86-190.
[20] TONEY M D,KIRSCH J F.Lysine 258 in aspartate aminotransferase:Enforcer of the circe effect for amino acid substrates and general-base catalyst for the 1,3-prototropic shift[J].Biochemistry,1993,32(6):1 471-1 479.
[21] KOYANAGI T,NAKAGAWA A,SAKURAMA H,et al.Eukaryotic-type aromatic amino acid decarboxylase from the root colonizer Pseudomonas putida is highly specific for 3,4-dihydroxyphenyl-L-alanine,an allelochemical in the rhizosphere[J].Microbiology,2012,158(12):2 965-2 974.