腐乳(sufu)是一种传统发酵食品,一般用于调味,以富含植物蛋白的豆腐为原材料,通过传统的固态发酵工艺制成,因此腐乳发酵过程中的微生物对腐乳的风味品质影响极大[1]。按照色泽与风味,腐乳可被分为青方腐乳、红方腐乳,白方腐乳、花色腐乳和酱腐乳等;同时按照发酵所用的微生物类型,又可以将腐乳分为毛霉型、根霉型和细菌型腐乳等[2]。
腐乳中含有丰富的植物蛋白质及被微生物降解产生的多肽和游离氨基酸,B族维生素,且不含胆固醇(或者胆固醇含量极低),另外还含有对人体有益的亚油酸,油酸,钙磷等无机盐矿物质,且经过微生物的发酵作用后,原有的抗营养成分被降解,呈豆腥味的分子被降解,变得易于消化[1]。据报道,腐乳还富含大豆异黄酮,大豆多肽,低聚肽,具有抗氧化、降血压、降胆固醇、改善阿尔兹海默病,还具有抗疲劳作用,这些都与腐乳制作过程中的微生物作用密不可分[1-3]。
花色腐乳与红方腐乳、白方腐乳的主要差异在于使用的辅料与工艺。研究表明,不同类型腐乳的菌群组成不同,白方腐乳和红方腐乳的菌群较为接近。红方腐乳中的主要微生物有乳球菌属(Lactococcus spp.),不动杆菌属(Acinetobacter spp.),四联菌属(Tetragenococcus spp.);白方腐乳的主要微生物有不动杆菌属,乳球菌属,假单胞菌属(Pseudomonas spp.),四联菌属,乳杆菌属(Lactobacillus spp.),金黄杆菌属(Chryseobacterium spp.)[4]。青方腐乳的主要微生物类群为乳杆菌属、Chishuiella spp.、四链球菌属。恩施地区的花色腐乳中的细菌主要为假单胞菌属、不动杆菌属、鞘氨醇杆菌属(Sphingobacterium spp.)、布丘氏菌(Buttiauxella)和草螺菌(Herbaspirillum)。张雅婷等[5]报道了腐乳中的主要真菌有霉菌,包括毛霉属(Mucor spp.),根霉属(Rhizopus spp.)等,以及酵母菌,包括裂殖酵母属(Schizosaccharomyces spp.),假丝酵母属(Candida spp.),毕赤酵母属(Pichia spp.)等。
风味是食品最重要的指标之一。尽管茨河花色腐乳风味较好,但该地区腐乳目前主要通过自然发酵法制作,无适用的微生物发酵剂,且发酵过程中腐乳菌群容易受到环境影响,造成产品质量不稳定,同时目前对该地区的花色腐乳中的微生物研究较少。第二代高通量测序技术具有成本低、通量高、可以快速产生大量数据的优点,能准确的分析发酵食品的微生物菌群组成。在食品检测中广泛应用的电子鼻与电子舌的分析结果不易受主观因素影响。因此本研究首先采集茨河地区花色腐乳样品,应用电子舌与电子鼻对腐乳的风味进行测定,继而通过MiSeq测序技术分析腐乳的真菌与细菌微生物多样性,并对腐乳的菌群与风味进行关联分析,以期为腐乳中有价值的微生物菌种的分离与筛选及腐乳的工业生产提供数据支撑。
1 材料与方法
1.1 材料与试剂
花色腐乳:采集自湖北省襄阳市谷城县茨河镇(111.85 °E, 32.04 °N)。首先将腐乳样品装入无菌瓶,然后装入低温样品箱,迅速带回微生物实验室,使用液氮冷冻后放入-70 ℃冰箱备用。腐乳样品要求无异味,色泽正常。腐乳样品编号为CH1~CH10,不同编号样品采集于不同商家或者农户。在使用MiSeq高通量测序技术对CH1腐乳细菌和真菌进行分析时,为便于区分同一样品分别编号为BCH1和FCH1,其他样品依次类推。
试剂:用于真菌ITS间区扩增的通用引物对ITS3F(5′-GCATCGATGAAGAACGCAGC-3′)和ITS4R(5′-TCCTCCGCTTATTGATATGC-3′)、用于细菌16S rRNA V3~V4区扩增的通用引物对338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′),由武汉天一辉远有限公司合成;dNTPs Mix、PrimeSTAR HS DNA Polymerase、5×PrimeSTAR Buffer,宝生物工程大连有限公司;E.Z.N.A.®土壤DNA试剂盒,美国Omega Bio-tek公司。电子鼻测定用阳离子溶液、阴离子溶液、内溶液和参比溶液,日本INSENT公司。
1.2 仪器与设备
Vetiri梯度基因扩增仪,美国ABI公司;WD-9413B型化学发光凝胶成像系统、DYY-6C型电泳仪,北京六一生物科技有限公司;ND-2000C型微量紫外分光光度计,美国Nano Drop公司;PEN3电子鼻,德国Airsense公司;SA-402B型电子舌,日本Insent公司。
1.3 实验方法
1.3.1 腐乳样品DNA提取,PCR扩增与高通量测序
腐乳样品的DNA提取使用E.Z.N.A.®土壤DNA试剂盒,参照说明书进行。将提取到的DNA使用琼脂糖凝胶电泳检测合格后,分别用于细菌和真菌的PCR扩增。对于真菌:参照邓长阳等[6]的方法,使用真菌带有barcode的通用引物ITS3F与ITS4R进行PCR扩增。PCR程序与体系均参照邓长阳等[6]的方法进行。PCR产物使用琼脂糖凝胶电泳与微量紫外分光度计检测。对于细菌:参照GUO等[7]的方法,使用细菌16S rRNA的V3~V4扩增的通用引物338F和806R。将真菌和细菌的PCR产物送上海美吉生物医药科技公司基于Illumina MiSeq第2代测序平台测序。
1.3.2 生物信息学分析
将MiSeq测序平台产生的原始序列使用QIIME 1.9.2平台进行生物信息学分析[8],步骤如下:使用FLASH将双末端序列基于重叠区域合并[9],去掉低质量序列,去掉接头;按照97%的相似度使用UCLUST[10]聚类产生分类操作单元(operational taxonomic unit,OTU),使用UCHIME[11]去掉嵌合体。对于真菌,基于UNITE数据库对OTU的代表性序列进行分类鉴定[12];对于细菌,基于Ribosomal Database Project (RDP) v11.5,Greengene database v13.5以及SILVA database v132数据库[13-15]进行联合鉴定。最后使用QIIME平台自带程序分别计算腐乳样品真菌和细菌的α与β多样性。将相对含量>1%的分类单元定义为优势分类单元。
1.3.3 腐乳滋味与气味测定
使用电子舌和电子鼻分别对腐乳滋味和气味进行测定与分析,具体参照崔梦君等[16]的方法进行。
1.3.4 统计分析与作图
使用R包vegan v2.5-7(https://cran.r-project.org/web/packages/vegan/index.html)进行去趋势对应分析(detrended correspondence analysis, DCA)和冗余分析(redundancy analysis, RDA);使用R自带的corr.test函数进行Pearson相关性分析;对数据的描述性统计使用R包pastecs(https://cran.microsoft.com/snapshot/2019-06-14/web/packages/pastecs/index.html)完成;文中所有图使用Origin 8.5软件或者R包ggplot2完成。
2 结果与分析
2.1 腐乳微生物多样性分析
通过高通量测序技术,分别以细菌16S rRNA基因的V3~V4区以及真菌的ITS区序列为靶点进行了MiSeq高通量测序与生物信息学分析。经过序列质控,共得到367 262条高质量16S reads,使用Uclust聚类分析,共得到4 652个OTU。接下来通过数据库Greengenes,RDP以及SILVA,将这些序列共注释为7个细菌门,11个纲,19个目,36个科,以及67个属(见表1)。
表1 茨河花色腐乳样品的序列与微生物多样性表
Table 1 Sequence abundance and microbial diversity of sufu samples from Cihe town

样品序列数分类操作单元门纲目科属发现物种指数Shannon指数BCH195 4542 3076101531502 101.925.94BCH1082 9362 2706101630502 207.566.12BCH2105 8662 6167111629512 291.246.20BCH387 4222 3846101630452 266.446.12BCH481 4742 1776101731502 130.725.83细菌BCH580 0162 231591326432 202.546.23BCH683 3322 1376101531572 071.545.80BCH783 8621 921591325441 855.745.30BCH879 9962 124691430482 099.245.92BCH989 1701 9057111529481 785.684.80平均值86 952.802 207.206.009.9015.0029.2048.602 101.265.83总数36 72624 652711193667——FCH163 491765516274454639.163.77FCH1078 033611410203747457.622.02FCH265 641813513243952678.644.26FCH342 140733511213747726.784.68FCH478 609996411264664794.544.95真菌FCH571 823705413213848563.822.99FCH679 318941512244462737.004.63FCH784 8541 006512244460789.564.81FCH879 893828413223956647.384.57FCH989 327921511224158714.224.79平均值73 312.90831.904.6012.2023.1040.9054.80674.874.15总数73 31291 9386173873111——
注:—表示无
2.2 腐乳菌群结构分析
对花色腐乳的优势细菌门进行了统计,结果见图1。由图1-a可知花色腐乳共有3个优势菌门,分别是变形菌门(Proteobacteria)[(55.38±9.29)%],厚壁菌门(Firmicutes)[(35.47±8.35)%],拟杆菌门(Bacteroidetes)[(7.25±2.68)%],其中变形菌门占一半以上。对相对含量>1%的细菌属进行统计分析见图1-b,发现花色腐乳中优势细菌属有6个:假单胞菌属[(51.67±9.68)%],乳杆菌属[(25.38±7.51)%],鞘氨醇杆菌属[(6.88±2.56)%],明串珠菌属[(Leuconostoc spp.)(5.10±2.22)%],肠球菌属[(Enterococcus spp.)(1.15±0.69)%],不动杆菌属[(1.03±0.47)%],其中假单胞菌占了50%以上,属于绝对优势属;另外对发酵食品比较重要的乳酸菌共占比31.63%。
尽管目前对花色腐乳中的微生物研究较少,然而已有不少研究对红方腐乳,白方腐乳和青方腐乳中的微生物多样性进行了解析。孙娜等[17]的研究表明,青方腐乳(臭豆腐)成品中的细菌以乳杆菌(23.44%)、Chishuiella(21.45%)、四链球菌属(16.28%)为主。红方腐乳成品中的优势菌属为乳球菌属、四联球菌属和不动杆菌属[4]。在红方腐乳和青方腐乳中均未出现占比超过50%的绝对优势属,且假单胞菌的相对含量也未超过1%[18]。通过可培养方法,对毛霉型腐乳的研究显示,优势菌属为乳球菌属、短杆菌属(Brevibacterium spp.)、稳杆菌属(Empedobacter spp.)、四联球菌属、鞘氨醇杆菌属,魏斯氏菌属(Weissella spp.)、盐厌氧菌属(Halanaerobium)和金葡杆菌属[19]。综合这些结果,不难看出,花色腐乳中细菌类群与其他类型的腐乳菌群差异较大,这也可能是导致其风味差异的重要原因。而同省的恩施州利川花色腐乳中的优势属与茨河花色腐乳较为接近,主要由假单胞菌属、不动杆菌属、鞘氨醇杆菌属等菌属构成[20]。另外也有研究表明,白方腐乳中也存在着假单胞菌,但是相对含量远低于花色腐乳[4]。
对真菌测序得到的原始序列进行质控与系列的双末端合并,共得到733 129个高质量序列,经过OTU聚类,并进行分类鉴定后,得到6个真菌门,17个真菌纲,38个真菌目,73个真菌科及111个真菌属。统计后发现,腐乳中真菌优势菌门有为子囊菌门(Ascomycota)[(39.52±16.02)%],担子菌门(Basidiomycota)[(35.69±18.88)%]和Mucoromycota[(24.66±19.75)%](图2-a);优势真菌属为红酵母属[(25.76±16.76)%],毛霉属[(24.64±19.74)%],青霉属[(12.14±8.25)%],间座壳属[(7.65±5.49)%],赤霉属[(5.17±2.69)%],Naganishia[(4.50±6.15)%],节担菌属(Wallemia spp.)[(2.70±1.50)%],棒孢菌属(Clavispora spp.)[(2.57±2.59)%],Guehomyces[(2.36±4.67)%],交链孢霉属(Alternaria spp.)[(2.23±1.53)%],分枝孢子霉属(Cladosporium spp.)[(1.88±1.13)%],曲霉属(Aspergillus spp.)[(1.54±1.12)%]和毕赤酵母属[(1.25±0.81)%],含5个酵母属与8个霉菌属。
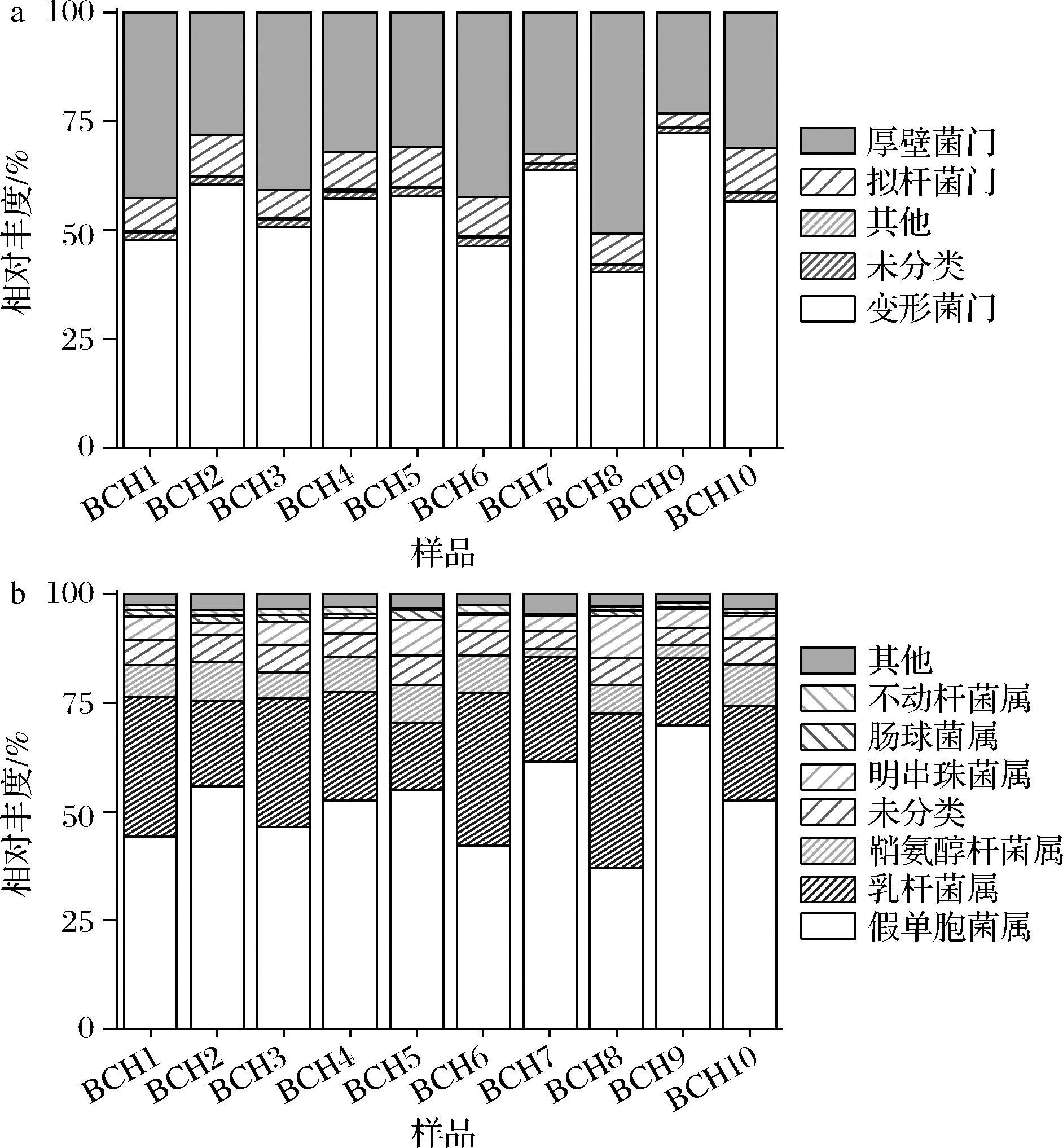
a-优势菌门;b-优势菌属
图1 茨河花色腐乳样品细菌优势菌门与优势菌属(n=10)
Fig.1 Dominant bacterial phyla and genera of huase sufu samples Cihe town (n=10)
对红方腐乳和另外一种毛霉型腐乳研究显示,其主要真菌组成为曲霉属(48.81%)和红曲霉属(Monascus spp., 37.18%),而重庆地区的腐乳中优势菌属为交链孢霉属、赤霉菌属,湖南白溪腐乳的优势真菌为地霉菌属(Geotrichum spp.)和被孢霉属(Mortierella spp.)[21-23],尽管不同的腐乳样品中优势菌群一般都是霉菌和酵母,但是在属水平上差异很大,而红酵母属是花色腐乳中含量最大且具有特色的真菌类群。红酵母属是担子菌门常见的酵母类群,可在土壤、海洋和湖水、果汁和牛奶中以及浴帘和牙刷上分离到,能分解蛋白质和脂肪产生氨基酸和小分子挥发性有机物[24],因此可能对腐乳风味具有作用。
Pearson相关系数取值范围为[-1,1],可用来衡量两个变量的关系,该值>0时,属于正相关,<0时,为负相关。生境中的微生物存在着复杂的生态关系,基于相对丰度,对微生物进行了相关性分析(图3),发现鞘氨醇杆菌与红酵母呈正相关(P<0.05),同时与青霉菌呈负相关(P<0.01);不动杆菌与Naganishia呈正相关(P<0.05),肠球菌与节担菌属呈正相关(P<0.01),而假单胞菌与毕赤酵母呈显著负相关(P<0.05)。这表明,细菌中占比最高的假单胞菌和毕赤酵母可能存在相互抑制,而真菌中相对含量最高的菌属红酵母属与鞘氨醇杆菌属有相互促进的代谢作用。
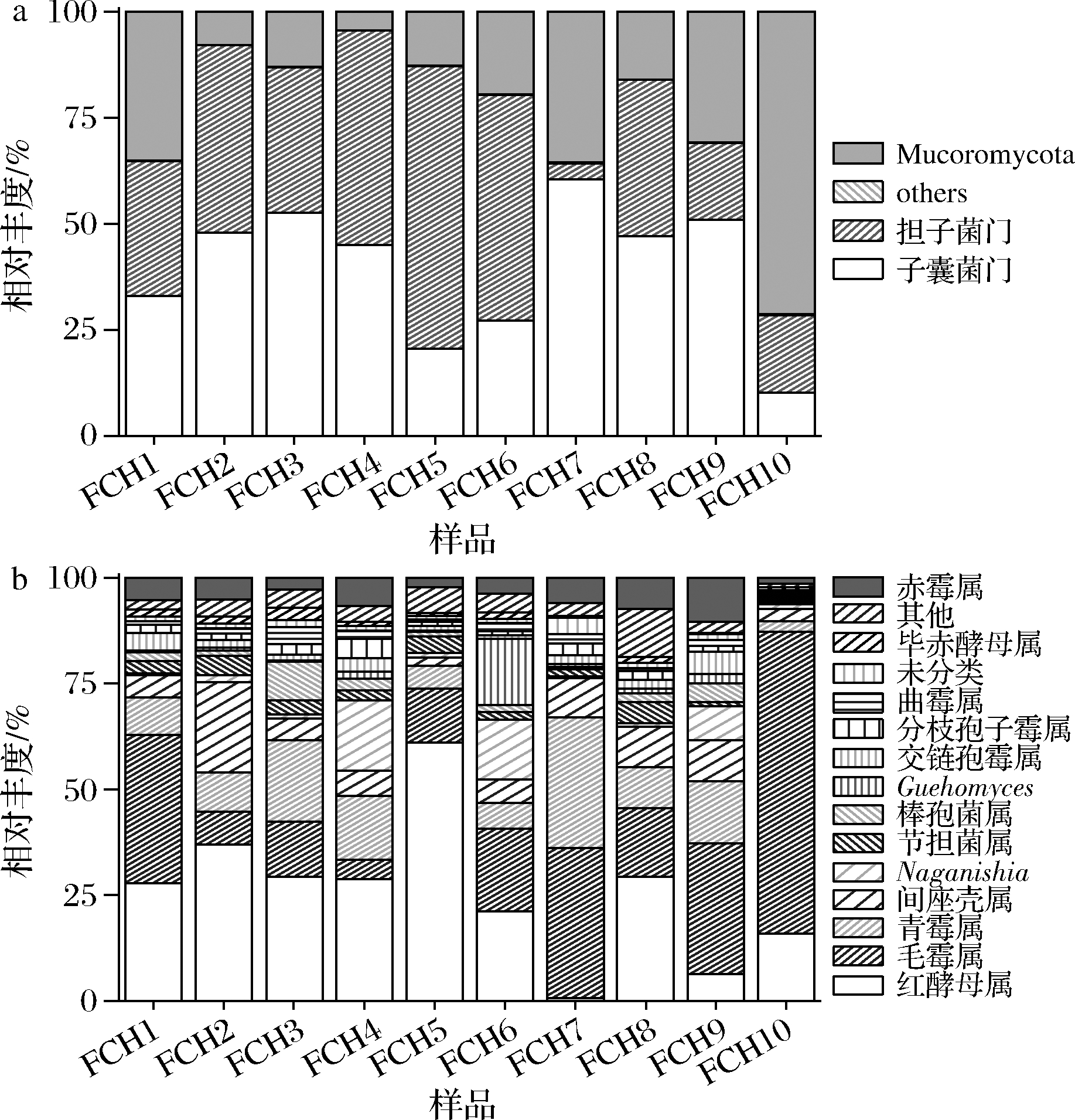
a-优势菌门;b-优势菌属
图2 茨河花色腐乳样品真菌优势菌门与优势菌属(n=10)
Fig.2 Dominant fungi phyla and genera of huase sufu samples Cihe town (n=10)
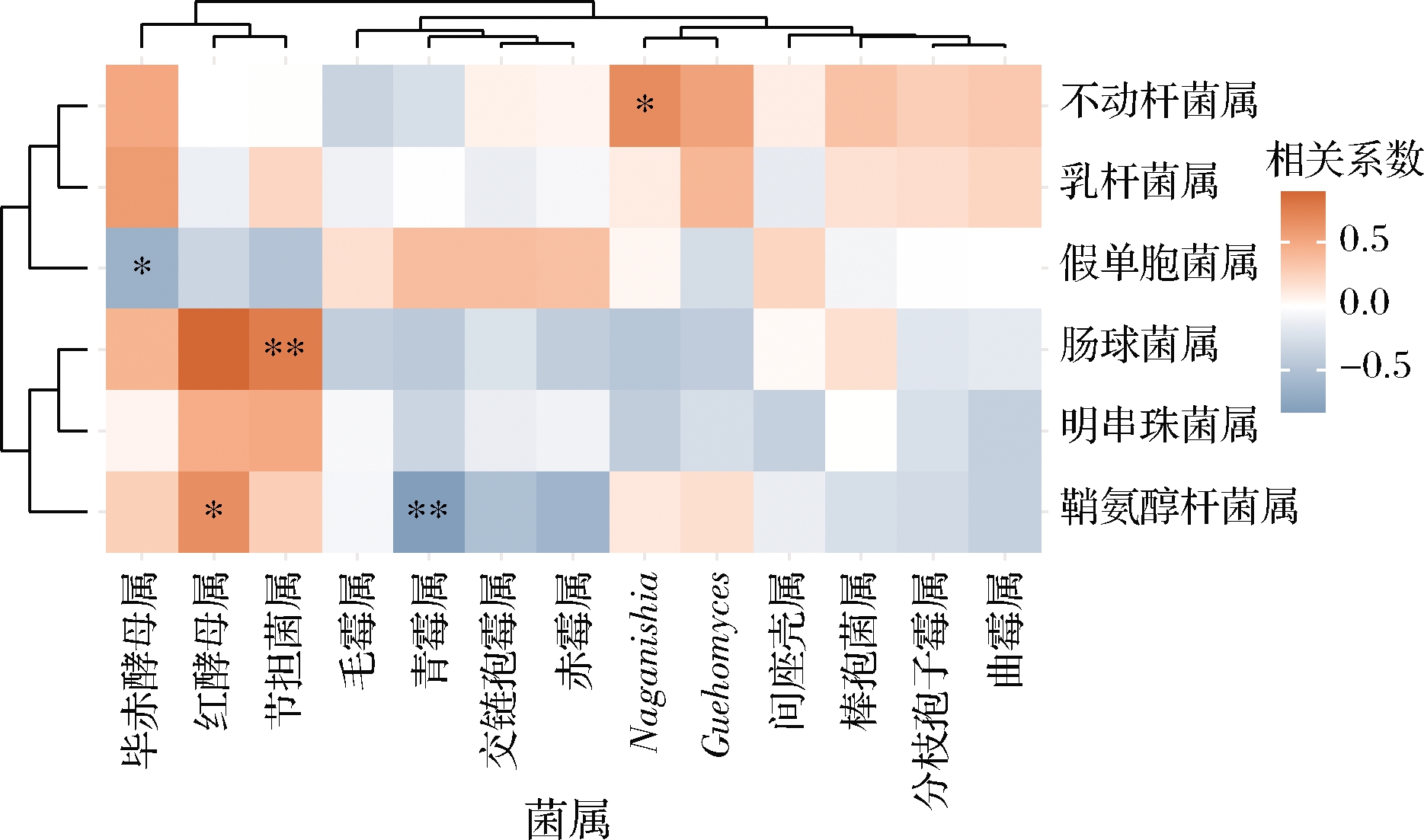
图3 茨河花色腐乳样品中真菌与细菌属相关性分析
Fig.3 Correlation analysis between fungi and bacteria in huase sufu samples from Cihe town 注:*,P<0.05;**, P<0.01;***, P<0.001(下同)
2.3 腐乳滋味与风味品质测定
滋味品质是食品的重要特征。使用电子舌对腐乳的滋味品质进行了测定与统计分析,结果见表2,10个腐乳样品的酸味与咸味的差异最大,极差值均>5,且苦味,涩味,鲜味,后味A(涩味的回味),后味B(苦味的回味)与丰厚度(鲜味的回味)的极差值也>1,表明这些腐乳样品在滋味上的差异可以通过感官分辨出来。
表2 基于电子舌的花色腐乳样品的各滋味 指标相对强度(n=10)
Table 2 Relative intensity of different taste indexes of sufu samples using electronic tongue (n=10)

滋味指标最小值最大值极差值中位数平均值酸味-7.030.217.24-0.64-2.58苦味-2.720.623.34-0.01-0.44涩味-4.400.064.46-0.12-1.28咸味-1.075.746.810.612.00鲜味-0.102.702.800.261.00后味A-1.580.011.59-0.03-0.28后味B-0.720.431.150.060.00丰厚度-0.831.762.59-0.120.27
同样的,通过电子鼻对腐乳的气味进行了测定与统计分析,电子鼻各传感器对腐乳的响应值见图4。结果表明,10个茨河花色腐乳的气味各指标存在差异,其中W1S,W5S与W1W传感器的响应值与极差值较大,极差值均超过了10,且W5S与W1W的响应值与极差值最大,极差值超过了70,表明采集的10个腐乳样品的差异主要体现在氮氧化物,有机硫化物与萜类化合物,甲烷和乙醇。
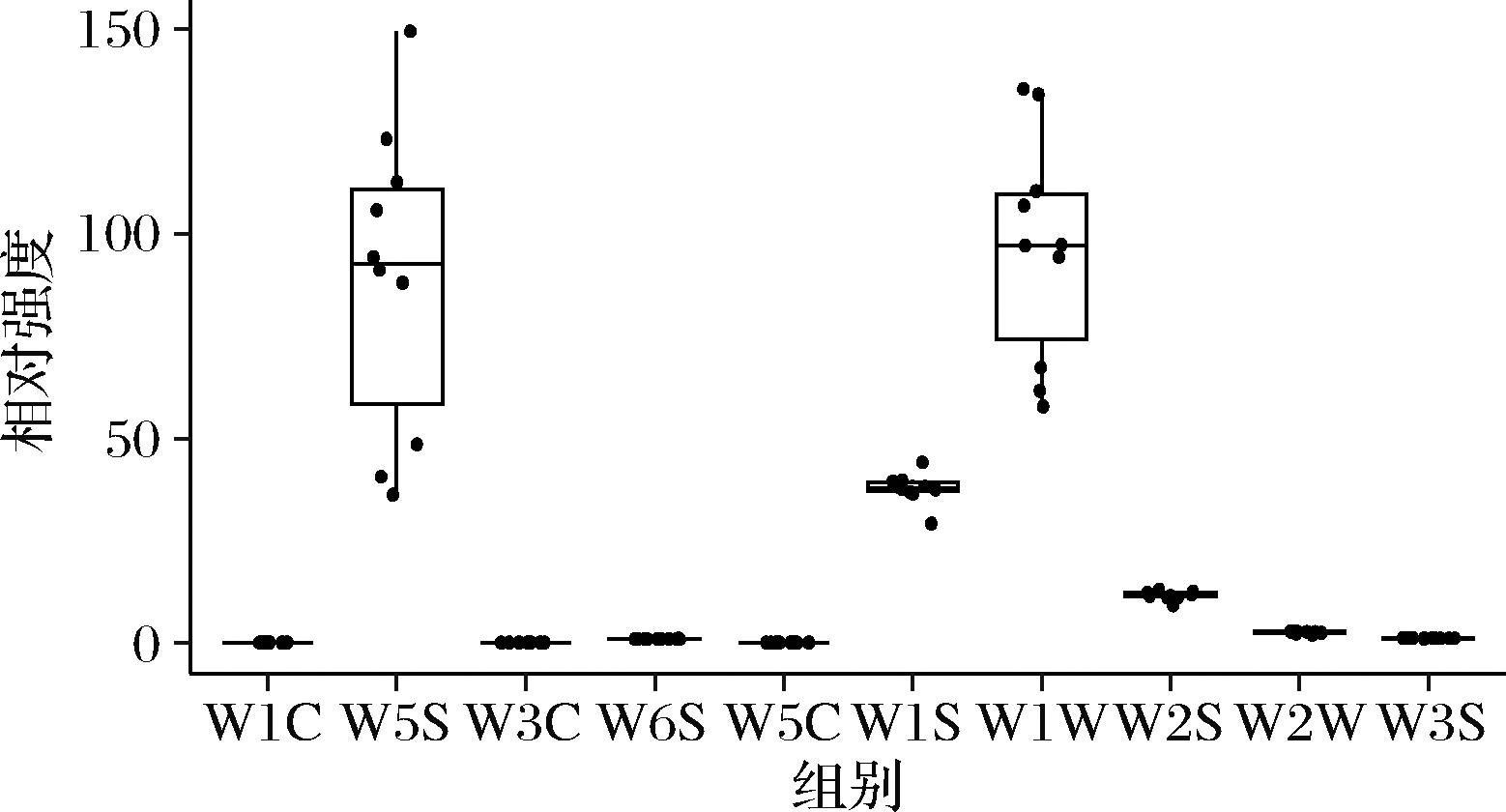
图4 基于电子鼻的茨河花色腐乳样品气味各传感器 强度箱型图(n=10) Table 4 Boxplot of relative intensity of taste indexes for sufu samples using electronic nose Cihe town (n=10) 注:W1C-对芳香味物质灵敏;W3C-芳香类物质灵敏;W5C-对烷烃; 芳香类物质灵敏;W1S-对甲烷灵敏;W2S-对乙醇灵敏;W3S-对烷烃灵敏; W5S-对氮氧化物灵敏;W6S-对氢气灵敏;W1W-对有机硫化物; 萜类物质灵敏;W2W-对有机硫化物灵敏
2.4 菌群与腐乳品质关联分析
对花色腐乳样品优势属相对含量与滋味、气味指标数据进行DCA,其最大梯度长都<1,因此判断腐乳样品数据更适合用线性模型分析。基于线性模型的RDA结果见图5。

图5 茨河花色腐乳样品滋味与气味品质与 菌群优势属的RDA
Fig.5 RDA analysis of taste and smell and dominant genera of sufu samples from Cihe town
图5显示,轴1能解释97.62%的茨河花色腐乳风味品质,而轴2能解释1.25%的腐乳风味品质,总体上来看,茨河花色腐乳样品的品质与肠球菌属、红酵母属、鞘氨醇单胞菌属、假单胞菌属和不动杆菌属关联度最强:与肠球菌属、红酵母属、鞘氨醇单胞菌属呈负相关,而与假单胞菌属、不动杆菌属呈正相关。同样的,也有研究显示,肠球菌与腐乳的大部分香味物质呈负相关关系[18]。然而,由于RDA并不能判断出来菌群具体与哪种滋味或者气味品质的关联,因此我们使用Pearson相关分析来进一步做了判定,结果见图6。总体上来说,与茨河花色腐乳滋味显著相关的微生物有青霉、毛霉、节担菌、鞘氨醇单胞菌、肠球菌和红酵母;与腐乳滋味相关的微生物有交链孢霉属、赤霉属、假单胞菌属、明串珠菌、乳杆菌、肠球菌、红酵母和Naganishia;同时与腐乳滋味与气味相关的微生物有肠球菌、红酵母和Naganishia。具体来说,红酵母和肠球菌与腐乳咸味呈显著负相关(P<0.05);仅青霉与腐乳的后味A与后味B呈负相关(P<0.05);对于丰厚度,有多种微生物(节担菌属、鞘氨醇单胞菌和肠球菌)均呈现出负相关关系(P<0.05),与之相反,毛霉呈极显著正相关(P<0.01),因此推测毛霉与青霉对腐乳的滋味品质具有促进作用,而节担菌等的代谢作用则可能产生不好的口感。总体来看,青霉与毛霉都对腐乳的滋味品质有促进作用,前者有助于去除涩味与苦味的回味,而毛霉能促进腐乳的丰厚度,但未发现与腐乳酸味,苦味,涩味,鲜味显著相关的细菌或真菌。

图6 茨河花色腐乳风味品质与菌群相关性分析
Fig.6 Correlation analysis between flavor quality and microbiol flora of huase sufu Cihe town
从气味上来说,与腐乳的芳香味呈正相关的菌有交链孢霉属和赤霉属(P<0.05);与腐乳有机硫化物传感器信号强度呈正相关的有肠球菌,呈负相关的有赤霉菌和交链孢霉菌(P<0.05);与对氮氧化物呈正相关的菌有肠球菌,呈负相关的有交链孢霉属和赤霉属(P<0.05);与腐乳氢气呈正相关的有明串珠菌和乳杆菌,呈负相关的为假单胞菌(P<0.05);与腐乳乙醇呈负相关的为赤霉菌和交链孢菌(P<0.05);与甲烷呈负相关的为赤霉菌和交链孢菌,呈正相关的为红酵母(P<0.05)。由于氮氧化物,有机硫化物,芳香味和乙醇等信号强度与腐乳的特征指标,因此交链孢霉菌、赤霉菌以及肠球菌对茨河花色腐乳具有重要的影响,可以后续从腐乳中筛选这些真菌,以明确这些菌的具体功能,以期在腐乳生产中应用。
3 结论
茨河花色腐乳中的优势真菌有红酵母属、毛霉属、青霉属、间座壳霉属、赤霉属、Naganishia、节担菌属、棒孢菌属、Guehomyces、交链孢霉属、分枝孢子霉属、曲霉属和毕赤酵母属,其中红酵母,毛霉和青霉相对含量均超过了10%;优势细菌属有假单胞菌属、乳杆菌属、鞘氨醇杆菌属、明串珠菌属、肠球菌属和不动杆菌属,其中假单胞菌属作为绝对优势属,其相对含量超过了50%以上。茨河花色腐乳滋味差异体现在咸味和酸味上,而气味差异主要在于氮氧化合物,有机硫化物或/和萜类化合物上。相关性分析表明,交链孢霉属和赤霉属与腐乳的芳香味呈显著正相关,而青霉和毛霉可能有助于改善腐乳的滋味。本研究能为腐乳的生产及发酵用菌的筛选提供数据支持。
[1] HAN B Z, ROMBOUTS F M, NOUT M J R.A Chinese fermented soybean food[J].International Journal of Food Microbiology, 2001, 65(1-2):1-10.
[2] 张兰威. 发酵食品工艺学[M].北京:中国轻工业出版社, 2011.
ZHANG L W.Fermented Food Technology[M].Beijing:China Light Industry Press, 2011.
[3] 张婷,张琪,罗洁,等.腐乳的营养价值及功能性研究进展[J].食品工业, 2019, 40(8):258-262.
ZHANG T, ZHANG Q, LUO J, et al.The functional research progress and nutrition of sufu[J].The Food Industry, 2019, 40(8):258-262.
[4] TAN G L, HU M, LI X Y, et al.2020.High-throughput sequencing and metabolomics reveal differences in bacterial diversity and metabolites between red and white sufu[J].Frontiers in Microbiology, 2020,758.
[5] 张雅婷,孙娜,于寒松,等.红腐乳发酵过程中菌群结构与风味相关性研究进展[J].食品与发酵工业,2020,46(1):287-294.
ZHANG Y T, SUN N, YU H S, et al.Advances in research on correlation between flora structure and product flavor during red sufu fermentation[J].Food and Fermentation Industries, 2020,46(1):287-294.
[6] 邓长阳,黎婷玉,刘文汇,等.襄阳地区中高温大曲曲皮和曲心真菌多样性解析[J].中国酿造,2021,40(6):65-69.
DENG C Y, LI T Y, LIU W H, et al.Fungal diversity in surface and center of medium-high temperature Daqu in xiangyang area [J].China Brewing, 2021,40(6):65-69.
[7] GUO Z, WANG Y R, XIANG F S, et al.Bacterial diversity in pickled cowpea (Vigna unguiculata [Linn.] Walp) as determined by Illumina MiSeq sequencing and culturedependent methods[J].Current Microbiology, 2021, 78(4):1 286-1 297.
[8] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods, 2010, 7(5):335-336.
[9] ![]() , SALZBERG S L.FLASH:Fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics, 2011, 27(21):2 957-2 963.
, SALZBERG S L.FLASH:Fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics, 2011, 27(21):2 957-2 963.
[10] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics, 2020, 26(19):2 460-2 461.
[11] EDGAR R C, HAAS B J, CLEMENTE J C, et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics, 2011, 27(16):2 194-2 200.
[12] ABARENKOV K, HENRIK NILSSON R, LARSSON K H, et al.The UNITE database for molecular identification of fungi-recent updates and future perspectives[J].New Phytologist, 2010, 186(2):281-285.
[13] COLE J R, CHAI B, FARRIS R J, et al.The ribosomal database project (RDP-II):Introducing my RDP space and quality controlled public data[J].Nucleic Acids Research, 2006, 35:169-172.
[14] DESANTIS T Z, HUGENHOLTZ P, LARSEN N, et al.Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J].Applied and Environmental Microbiology, 2006, 72(7):5 069-5 072.
[15] QUAST C, PRUESSE E, YILMAZ P, et al.The SILVA ribosomal RNA gene database project:Improved data processing and web-based tools[J].Nucleic Acids Research, 2012, 41:D590-D596.
[16] 崔梦君,张振东,万舒曼,等.农家豆瓣酱细菌多样性及其对品质影响的评价[J].食品与发酵工业, 2020, 46(5):68-73.
CUI M J, ZHANG Z D, WAN S M, et al.Characterization of bacterial microflora and its influence on product quality of soybean paste[J].Food and Fermentation Industries, 2020, 46(5):68-73.
[17] 孙娜,张雅婷,于寒松,等.发酵型青腐乳菌群结构与风味物质及其相关性分析[J].食品科学,2020,41(22):177-183.
SUN N, ZHANG Y T, YU H S, et al.Microflora structure and flavor components and correlation between them in fermented stinky tofu[J].Food Science, 2020,41(22):177-183.
[18] 陈卓,吴学凤,穆冬冬,等.红腐乳后酵期风味物质与细菌菌群分析[J].食品科学,2021,42(6):118-125.
CHEN Z, WU X F, MU D D, et al.Determination and correlation analysis of flavor components with bacterial community in post-fermented red sufu[J].Food Science, 2021,42(6):118-125.
[19] XIE C, ZENG H, WANG C, et al.Volatile flavor components, microbiota and their correlations in different sufu, a Chinese fermented soybean food[J].Journal of Applied Microbiology, 2018, 125(6):1 761-1 773.
[20] 李娜,崔梦君,马佳佳,等.自然发酵腐乳中细菌多样性评价[J].食品研究与开发,2019,40(16):165-171.
LI N, CUI M J, MA J J, et al.Evaluation of bacterial diversity in natural fermented sufu[J].Food Research and Development, 2019,40(16):165-171.
[21] XU D D, WANG P, ZHANG X, et al.High-throughput sequencing approach to characterize dynamic changes of the fungal and bacterial communities during the production of sufu, a traditional Chinese fermented soybean food[J].Food Microbiology, 2020, 86:103340.
[22] 石黎琳, 牟方婷,李安,等.基于高通量测序技术分析腐乳自然发酵过程微生物多样性[J].中国酿造, 2021,40(2):144-149.
SHI L L, MU F T, LI A, et al.Analysis of microbial diversity in sufu during natural fermentation using high-throughput sequencing technology[J].China Brewing, 2021,40(2):144-149.
[23] WAN H F, LIU T, SU C W, et al.Evaluation of bacterial and fungal communities during the fermentation of Baixi sufu, a traditional spicy fermented bean curd[J].Journal of the Science of Food and Agriculture, 2020, 100(4):1 448-1 457.
[24] BATT C A, TORTORELLO M L. Encyclopedia of Food Microbiology (Second Edition)[M]. Salt Lake City: Academic Press, 2014.