中国传统食醋是指以谷物为主要原料,经过多种微生物共同发酵,再经长时间陈酿而成的一种酸性液体调味料,在中国已经有3 000多年的历史[1]。按照食醋的醋酸发酵工艺,可将食醋分为固态发酵食醋和液态发酵食醋。山西老陈醋、镇江香醋、四川麸醋、赤水晒醋和天津独流醋等中国传统谷物醋都是典型的固态发酵食醋[2];固态发酵食醋的开放式发酵、多菌种协同、长时间陈酿、多代谢产物共存的特点赋予了食醋丰富的营养物质和风味物质,使食醋具有开胃、抗菌杀菌、抗氧化和减肥等功效[3]。
赤水晒醋是贵州省赤水市生产的一种极具地方特色的优质食醋产品,其工序繁多、选料考究、品质精良,在中国西南地区享有盛誉。传统的赤水晒醋用中草药制曲、经蒸煮、酒精发酵和醋酸发酵、晒醅、淋醋、陈酿、灭菌等几十道工序精制而成[4]。目前有众多的研究者对“中国四大名醋”的发酵机理、营养成分、风味形成和功能作用等各个方面进行了研究,但对赤水晒醋的研究较少。目前仅有卢叶等[5]研究了赤水晒醋的有机酸组成,发现除乙酸外,不挥发酸中琥珀酸和乳酸的含量最高;石庆叠等[6]从赤水晒醋中筛选了3株醋酸菌,并研究了其发酵特性,发现C6菌株能耐9%(体积分数)的乙醇、8 g/L的氯化钠和40 g/L的乙酸;唐杰等[7]研究了赤水晒醋的香气成分,从晒醋中共鉴定出39种主要香气成分;吴震等[8]研究了赤水晒醋生产过程的理化指标和功能性指标的变化,揭示了发酵规律;何银等[9]、杨新等[10-11]从赤水晒醋中筛选了腐败微生物并研究了其发酵特性,主要包括地衣芽孢杆菌、解淀粉芽孢杆菌、索诺拉沙漠芽孢杆菌、酸居芽孢杆菌、耐热芽孢杆菌、枯草芽孢杆菌等;李荣源等[12]研究了赤水晒醋各生产阶段终点的微生物群落结构组成,其在晒醅阶段检测到194种真菌和400种细菌;但目前尚没有针对赤水晒醋醋酸发酵过程原核微生物群落结构变化的相关研究。赤水晒醋醋酸发酵阶段具有开放式和多菌协同的特点,因此研究赤水晒醋醋酸发酵过程原核微生物群落结构变化对解析赤水晒醋的发酵机理及通过微生物调控技术改善赤水晒醋的品质具有重要意义。
1 材料与方法
1.1 材料与试剂
醋醅样品,贵州省赤水市某醋厂。
DNA抽提试剂盒,赛默飞世尔科技(中国)有限公司;琼脂糖,上海阿拉丁生化科技股份有限公司;蛋白酶K,上海生物工程有限公司;Premix ET Taq聚合酶,宝生物工程(大连)有限公司。
1.2 仪器与设备
DW-25L262医用低温保存箱,青岛海尔特种电器有限公司;5425R高速离心机,德国Eppendorf公司;NanoDrop 2000超微量分光光度计,美国Thermo Fisher Scientific公司;HiSeq测序仪,美国Illumina公司;ELx 800酶标仪,美国BioTek公司;QL-901旋涡混合器,海门其林贝尔仪器制造有限公司。
1.3 实验方法
1.3.1 取样方法
采用五点取样法从贵州省赤水市某醋厂同一生产批次的3个不同的醋酸发酵池(A、B、C)中取样,每隔3天取样1次,每次取3个平行样品,共采集18个样品,将样品置于-80 ℃冰箱中保存。
1.3.2 DNA提取
取50 g保存于冰箱中的醋醅样品置于研钵中,添加液氮研磨5 min,然后使用DNA抽提试剂盒提取醋醅内含物微生物DNA,使用NanoDrop 2000超微量分光光度计检测DNA浓度及质量。
1.3.3 PCR扩增及测序
通过扩增原核微生物16S rRNA V3~V4区研究醋醅的原核微生物群落结构,采用的上游引物为338F (5′-ACTCCTACGGGAGGCAGC-3′),下游引物为806R (5′-GGACTACNNGGGTATCTAAT-3′)。对指定测序范围合成带barcode的特异性引物进行PCR扩增及产物纯化,构建PE文库后进行Illumina HiSeq 2500平台测序,由上海美吉生物医药科技有限公司完成。
1.3.4 理化指标的检测
总酸、乙醇、还原糖和乳酸含量的检测方法参考文献[13]。
1.4 数据处理
利用QIIME 1.9对测序数据进行处理,使用Usearch去除嵌合体序列,并按照97%相似度对序列进行操作分类单元(operational taxonomic units,OTU)聚类划分,选取最丰富序列(most abundant)为OTU代表序列。细菌采用GreenGenes数据库(http://greengenes.secondgenome.com/)对OTU代表序列进行分类注释。然后根据注释结果,使用R 3.6、Graphpad Prism 7和Canoco 5等软件进行后续分析绘图。
2 结果与分析
2.1 α多样性分析
赤水晒醋醋酸发酵过程中醋醅样品原核微生物群落的测序情况见表1,18个醋醅样品检测到的有效序列数为41 713~47 029条,总序列数为794 635条。按97%的相似度对测序数据进行聚类划分,18个样品的OTU数为51~156个,共有213个OTU。且OTU数随着醋酸发酵的不断进行而逐渐下降。所有样品文库的覆盖率均>99.9%,表明样品中序列没有被检测出的概率极低。将测序数据置于GreenGenes数据库中进行比对,得到各个样品中的物种数,可见物种数与OTU数变化趋势一致。
根据97%相似度水平下的OTU信息,采用表征α多样性的ACE指数及Shannon指数对样品微生物物种丰富度和多样性进行评估[14],结果见表1。ACE指数用于评估样本中OTU数目的多少,指数越大,表明OTU数越多;Shannon指数用于评估样本中微生物的多样性,Shannon指数越大,群落的多样性越高。从表1可以看出,赤水晒醋醋酸发酵过程醋醅样品的原核微生物群落OTU数和多样性在第0~3天略微上升,在第3~15天逐渐下降。
基于97%相似度的OTU水平绘制的赤水晒醋醋酸发酵过程的原核微生物群落稀释曲线如图1所示,当检测到的序列数>30 000以后,曲线趋于平缓,说明测序深度较好,测序数据量合理。
2.2 原核微生物群落组成分析
由图2-a可知,赤水晒醋的醋酸发酵过程中醋醅的原核微生物群落在门水平上可分为变形菌门(Proteobacteria,41.7%)、厚壁菌门(Firmicutes,43.25%)、蓝细菌门(Cyanobacteria,14.29%)、拟杆菌门(Bacteroidetes,0.48%)和放线菌门(Actiobacteria,0.28%)。由图2-b可知,醋酸发酵第0天,变形菌门、厚壁菌门和蓝细菌门的总相对丰度>98%,随着醋酸发酵的进行,蓝细菌门的相对丰度由42.11%下降至1.45%;而厚壁菌门的相对丰度由15.53%上升至62.43%;变形菌门的相对丰度由第0天的40.86%上升至第9天的48.83%,然后再下降至第15天的38.19%;拟杆菌门和放线菌门的相对丰度在第12天即下降至0.1%以下。
表1 醋醅中原核微生物群落α多样性
Table 1 Alpha diversity of prokaryotic microbial community in Cupei
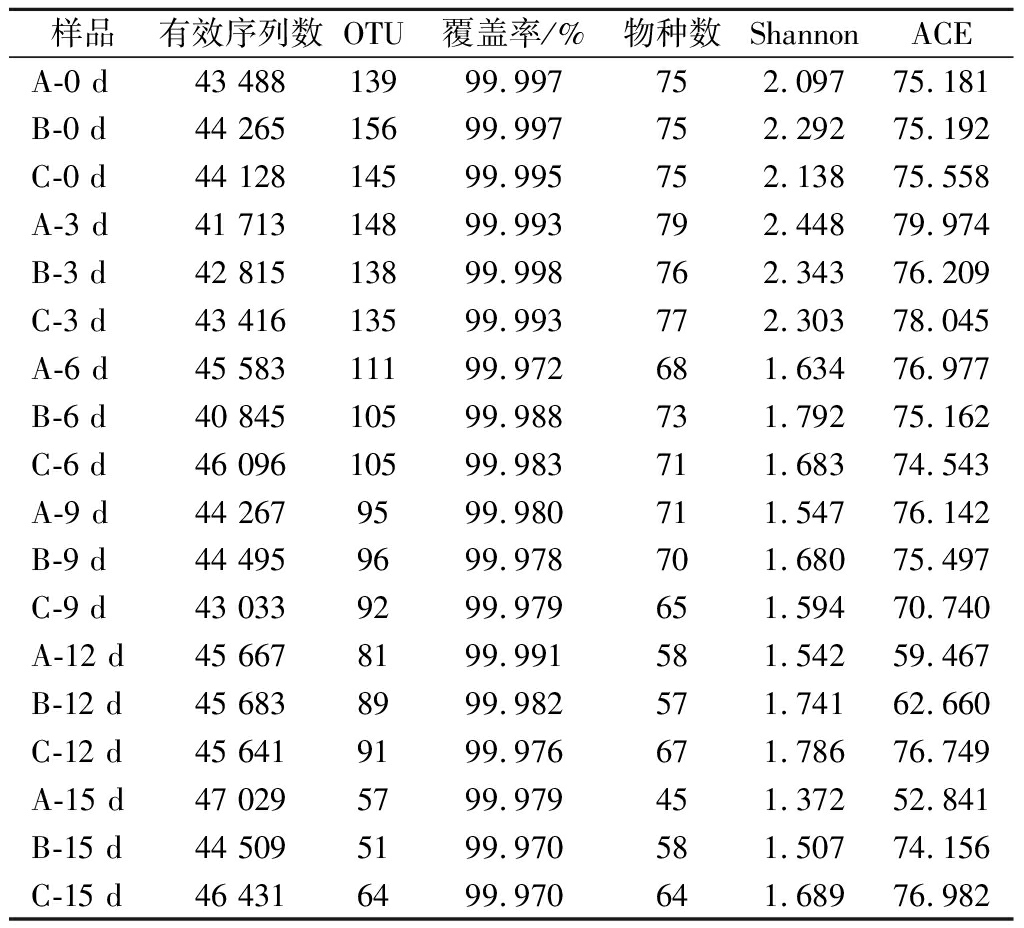
样品有效序列数OTU覆盖率/%物种数ShannonACEA-0 d43 48813999.997752.09775.181B-0 d44 26515699.997752.29275.192C-0 d44 12814599.995752.13875.558A-3 d41 71314899.993792.44879.974B-3 d42 81513899.998762.34376.209C-3 d43 41613599.993772.30378.045A-6 d45 58311199.972681.63476.977B-6 d40 84510599.988731.79275.162C-6 d46 09610599.983711.68374.543A-9 d44 2679599.980711.54776.142B-9 d44 4959699.978701.68075.497C-9 d43 0339299.979651.59470.740A-12 d45 6678199.991581.54259.467B-12 d45 6838999.982571.74162.660C-12 d45 6419199.976671.78676.749A-15 d47 0295799.979451.37252.841B-15 d44 5095199.970581.50774.156C-15 d46 4316499.970641.68976.982

图1 醋醅中原核微生物群落稀释曲线
Fig.1 Rarefaction curve of prokaryotic microbial community in Cupei
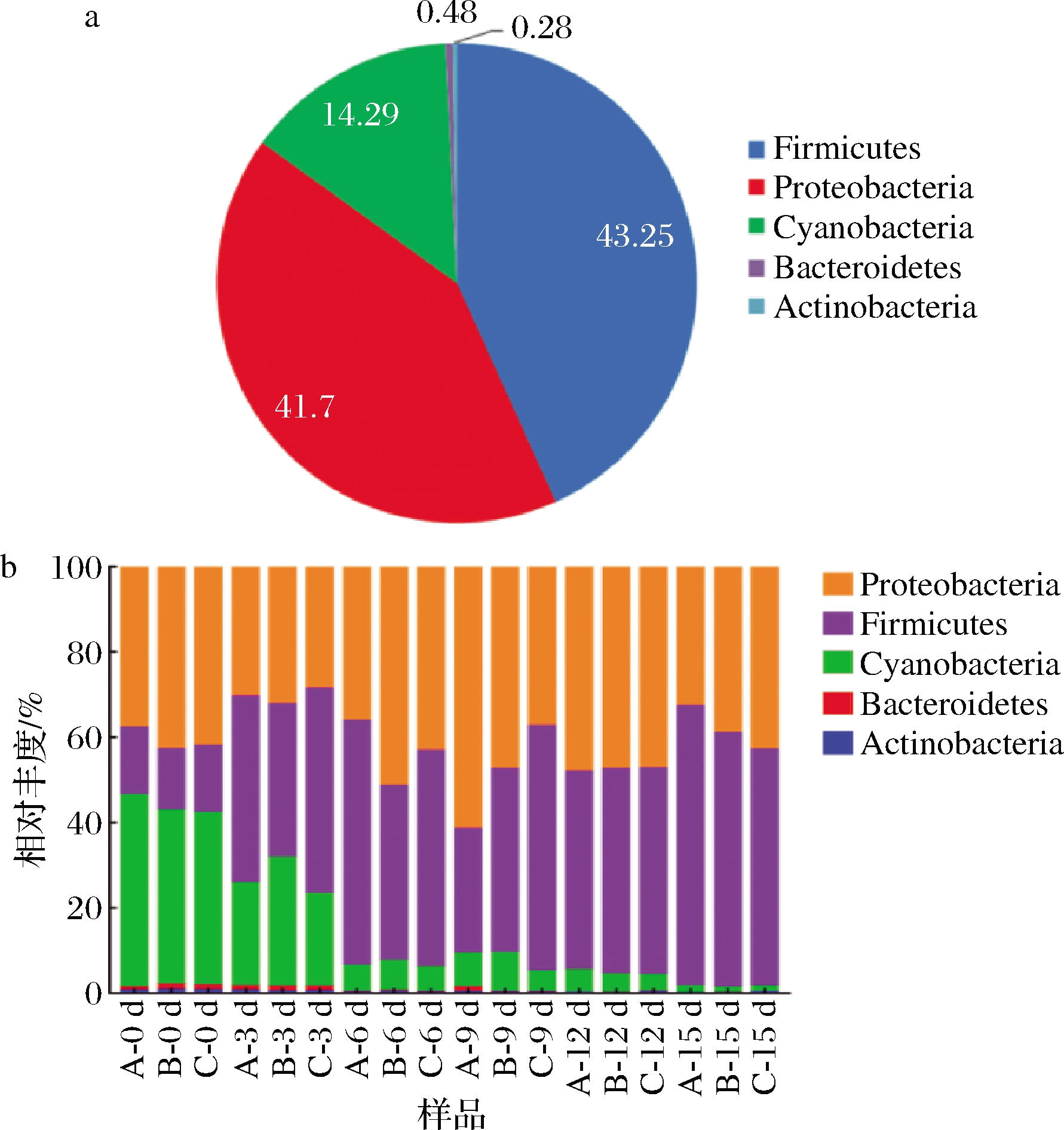
a-门水平的组成;b-相对丰度变化
图2 原核微生物群落在门水平的组成及相对丰度变化
Fig.2 The composition and relative abundance changes of the prokaryotic microbial community at the phylum level 注:图2-a中原核微生物群落在门水平所占的比例为所有 样品的平均值
如图3-a所示,18个醋醅样品中的原核微生物属主要为乳杆菌属(Lactobacillus,33.88%)、醋杆菌属(Acetobacter,28.19%)、蓝藻细菌属(Acaryochloris,14.21%)、醋酸乳杆菌属(Acetilactobacillus,8.97%)、未分类的红螺菌属(Rhodospirillaceae_unclassified,4.84%)、农杆菌属(Agrobacterium,1.98%)、鞘氨醇单胞菌属(Sphingomonas,1.69%)和葡糖醋杆菌属(Gluconacetobacter,1.23%)。由图3-b可知,随着醋酸发酵的进行,乳杆菌属的相对丰度由第0天的15.03%上升至第6天的39.30%,然后下降至第15天的23.05%;醋杆菌属在第0天时的相对丰度为0.32%,第3天时上升至8.85%,第6~12天相对丰度为43.77%,第15天又下降至32.28%;而醋酸乳杆菌属和葡糖醋杆菌属在第0~6天的相对丰度极低,在第9~15天相对丰度逐渐上升,到第15天时,醋酸乳杆菌属和葡糖醋杆菌属的相对丰度分别达36.78%和4.76%;蓝藻细菌属、未分类的红螺菌属、农杆菌属、鞘氨醇单胞菌属、未注释到的菌属(not_assigned)和黄单胞菌属(Xanthomonas)、甲基杆菌属(Methylobacterium)、寡养单胞菌属(Stenotrophomonas)、假单胞菌属(Pseudomonas)和泛菌属(Pantoea)等菌属的相对丰度随着醋酸发酵过程的进行而下降;李荣源等[12]研究发现醋酸发酵结束的醋醅中主要的细菌属为乳杆菌属和醋杆菌属,这可能是数据处理方式的区别导致醋酸乳杆菌属未被注释到。另外可以发现赤水晒醋醋酸发酵的细菌群落组成与镇江香醋[15]、山西老陈醋[16]、凉州熏醋[17]等固态发酵食醋的细菌群落较为相似,而与浙江玫瑰醋[18]和永春老醋[19]等液态发酵食醋的细菌群落有较大差异,这表明醋酸发酵工艺对微生物群落结构有较大影响。由于醋酸发酵过程主要是微生物利用乙醇和糖类物质生成乙酸和其他有机酸并产生大量生物热的过程,前期乳杆菌属利用原料中的糖类繁殖代谢,相对丰度升高,随后由于小分子糖被耗尽以及酸度的升高,乳杆菌属逐渐死亡,相对丰度下降;醋杆菌属前期利用乙醇大量生成乙酸,后期由于乙醇几乎被耗尽且酸度升高,因此相对丰度下降;蓝藻细菌属、未分类的红螺菌属、农杆菌属、鞘氨醇单胞菌属、未注释到的菌属和黄单胞菌属、甲基杆菌属、寡养单胞菌属、假单胞菌属和泛菌属等菌属主要来自原料、空气和生产工具[15],醋酸发酵体系的高乙醇、高乙酸和高温度使其凋亡;而醋酸乳杆菌属和葡糖醋杆菌属由于具有耐酸的特性[20-22],因此在醋酸发酵后期其相对丰度逐渐升高。

a-属水平的组成;b-相对丰度变化
图3 原核微生物群落在属水平的组成及相对丰度变化
Fig.3 The composition and relative abundance changes of the prokaryotic microbial community at the genus level 注:图3-a中原核微生物群落在属水平所占的比例为所有样品的平均值
2.3 β多样性分析
基于Bray-Curtis距离算法,以18个醋醅样品中的OTU组成为原始数据进行主坐标分析(principal coordinate analysis,PCoA)(图4-a)和聚类分析(cluster analysis,CA)(图4-b)。由图4-a可以看出第一主坐标轴(PCoA1)和第二主坐标轴(PCoA2)对赤水晒醋醋酸发酵过程醋醅中的原核微生物群落的解释度分别为75.9%和15.2%,不同醋醅样品基本按照发酵天数聚在一起,并随着发酵时间在PCoA1上逐渐移动。由图4-b可以将赤水晒醋的醋酸发酵过程分为3个阶段:发酵前期(第0~3天)、发酵中期(第6~12天)和发酵末期(第15天);另外平行样品之间的距离小于不同时间样品之间的距离,可以说明平行样品之间的一致性较好。
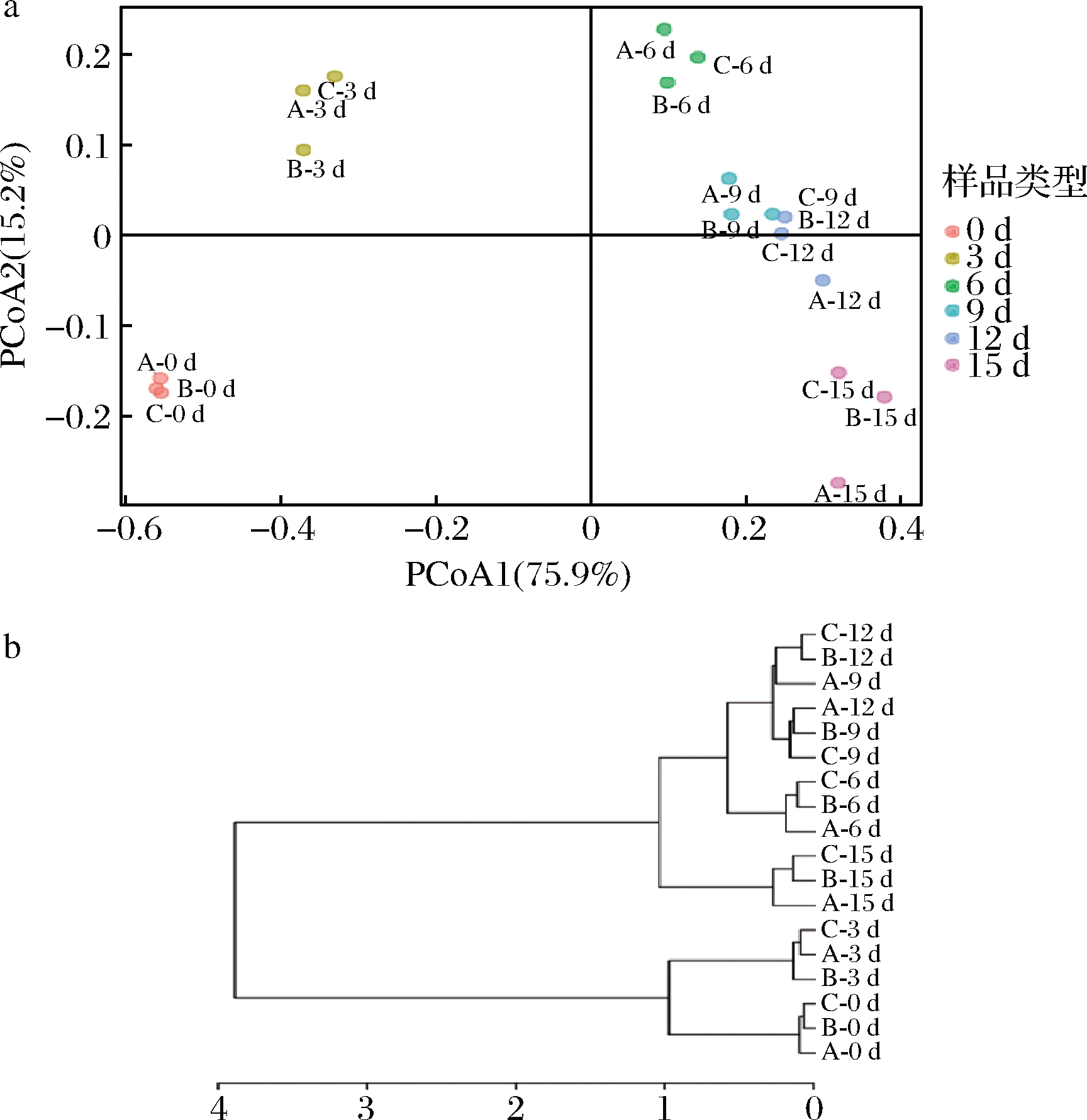
a-PCoA;b-CA
图4 醋醅中原核微生物群落的PCoA及CA
Fig.4 PCoA and CA of prokaryotic microbial community in Cupei
2.4 基础理化指标变化
固态发酵食醋醋酸发酵过程中,醋酸菌将乙醇转化为乙酸,乳酸菌将葡萄糖等小分子糖类转化为乳酸,同时伴随着一系列其他生化反应的发生,这些反应共同赋予了食醋独特的风味[23]。从图5可以看出,乙醇由5.78%下降到0.33%,而总酸由0.61%(以乙酸计)上升至5.35%(以乙酸计),两者变化趋势基本相反。而乳酸和还原糖含量的变化趋势都是先上升后下降,前期麸皮中的部分大分子糖类由于酸水解作用导致还原糖含量上升,随后由于乳酸菌等微生物利用还原糖转化为其他物质而下降;另一方面,前期乳酸菌利用还原糖产生乳酸,但后期醋酸菌等能利用乳酸生成乙偶姻,导致乳酸含量下降[24]。
2.5 相关性分析
冗余分析(redundancy analysis,RDA)主要反映菌群与环境因子之间的关系,环境因子箭头所处的象限表示环境因子与排序轴间的正负相关性,箭头连线的长度代表着某个环境因子与群落分布和种类分布间相关程度的大小,连线越长,说有相关性越大,反之越小。箭头连线和排序轴的夹角代表着某个环境因子与排序轴的相关性大小,夹角越小,相关性越高;反之越低[25]。由图6可知,乳杆菌属主要与乳酸和还原糖含量呈正相关,醋杆菌属、柠檬乳杆菌属(Limosilactobacillus)、葡糖醋杆菌属和醋酸乳杆菌属主要与酸度呈正相关,而农杆菌属、黄单胞菌属和甲基杆菌属等菌属主要与酸度呈负相关,且与乙醇含量呈正相关。

a-总酸、乙酸;b-还原糖、乳酸
图5 醋酸发酵过程理化指标变化
Fig.5 Changes in physical and chemical indicators during acetic acid fermentation
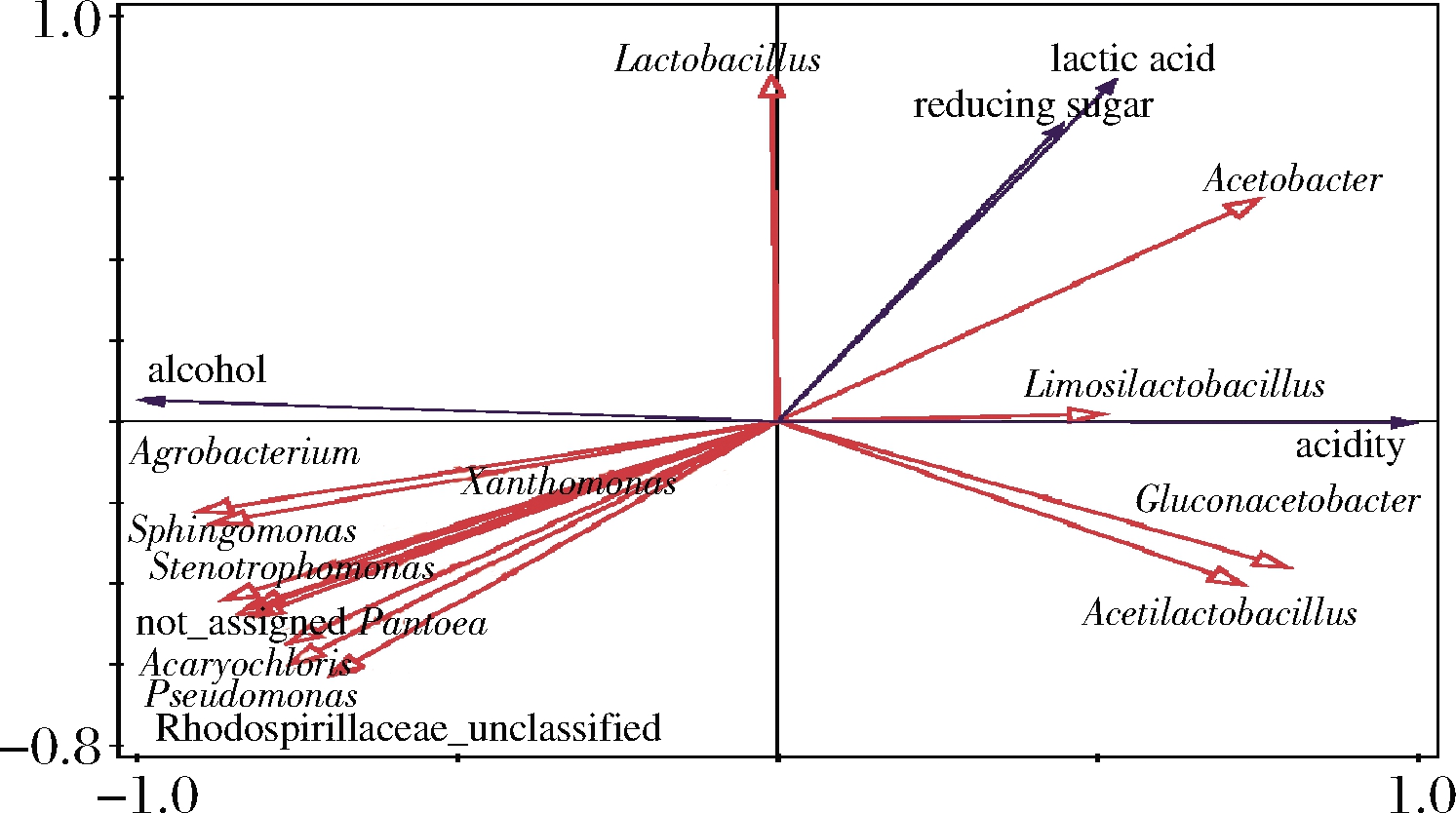
图6 醋酸发酵过程原核微生物属与环境因子的冗余分析
Fig.6 Redundancy analysis of prokaryotic microbial genus and environmental factors during acetic acid fermentation
3 结论
赤水晒醋醋酸发酵过程的OTU数和物种数均呈先略微上升后下降的趋势。原核微生物群落在门水平上可分为变形菌门(41.7%)、厚壁菌门(43.25%)、蓝细菌门(14.29%)、拟杆菌门(0.48%)和放线菌门(0.28%);在属水平上,醋酸乳杆菌属和葡糖醋杆菌属的相对丰度随着醋酸发酵的进行一直上升,乳杆菌属和醋杆菌属相对丰度随着醋酸发酵的进行先上升后下降;蓝藻细菌属、未分类的红螺菌属、农杆菌属、鞘氨醇单胞菌属等原核微生物属的相对丰度随着醋酸发酵的进行呈下降的趋势。主坐标分析表明:醋醅样品按照醋酸发酵天数聚在一起,并随着发酵的进行在第一主坐标轴上移动;通过聚类分析可将醋酸发酵过程分为前期(第0~3天)、中期(第6~12天)和末期(第15天)这3个阶段。总酸由0.61%(以乙酸计)上升至5.35%(以乙酸计),乙醇由5.78%下降至0.33%;还原糖和乳酸含量均呈先上升后下降的趋势。此外,相关性分析表明乳杆菌属主要与乳酸和还原糖含量呈正相关,醋杆菌属、柠檬乳杆菌属、葡糖醋杆菌属和醋酸乳杆菌属主要与酸度呈正相关,而农杆菌属、黄单胞菌属和甲基杆菌属等菌属主要与酸度呈负相关,且与乙醇含量呈正相关。
[1] 郑宇, 程程, 刘静, 等.中国传统固态发酵食醋主要特征风味物质组成分析[J].中国食品学报, 2020, 20(8):237-247.
ZHENG Y, CHENG C, LIU J, et al.Analysis of main characteristic flavor components in Chinese traditional solid-state fermented vinegars[J].Journal of Chinese Institute of Food Science and Technology, 2020, 20(8):237-247.
[2] 王兴洁, 刘爱平, 敖晓琳, 等.中国传统食醋微生物污染及控制措施研究进展[J].食品与发酵工业, 2019, 45(10):260-265.
WANG X J, LIU A P, AO X L, et al.Microbial contamination and control measures during traditional Chinese vinegar fermentation[J].Food and Fermentation Industries, 2019, 45(10):260-265.
[3] 邝格灵, 张洁, 孔德华, 等.植物乳杆菌与解淀粉芽孢杆菌对食醋风味的影响[J].中国酿造, 2018, 37(6):25-29.
KUANG G L, ZHANG J, KONG D H, et al.Effect of Lactobacillus plantarum and Bacillus amyloliquefaciens on vinegar flavor[J].China Brewing, 2018, 37(6):25-29.
[4] 唐杰. 赤水晒醋醋曲品质、微生物多样性及制曲工艺的探究[D].重庆:西南大学, 2018.
TANG J.Research on the quality, microbial diversity and production process of vinegar koji in Chishui[D].Chongqing:Southwest University, 2018.
[5] 卢叶, 张季, 向丽萍, 等.HPLC法同时测定赤水晒醋中9种有机酸[J].现代食品, 2020(13):149-153;160.
LU Y, ZHANG J, XIANG L P, et al.Simultaneous determination of nine organic acids in Chishui sun vinegar by HPLC[J].Modern Food, 2020(13):149-153;160.
[6] 石庆叠, 何星, 卢红梅, 等.赤水晒醋醋醅中醋酸菌的筛选和发酵特性研究[J].食品与发酵工业, 2021, 47(14):223-228.
SHI Q D, HE X, LU H M, et al.Screening of acetic acid bacteria in Chishui Sun-dried vinegar Pei and its fermentation characteristics[J].Food and Fermentation Industries, 2021, 47(14):223-228.
[7] 唐杰, 龙晓琴, 何银, 等.赤水晒醋的香气成分分析研究[J].中国调味品, 2017, 42(10):135-139.
TANG J, LONG X Q, HE Y, et al.Analysis of aroma components in Chishui Sun vineger[J].China Condiment, 2017, 42(10):135-139.
[8] 吴震, 卢红梅, 陈莉, 等.赤水晒醋生产过程中品质形成的探究[J].中国酿造, 2020, 39(2):56-62.
WU Z, LYU H M, CHEN L, et al.Formation of Chishui Sun vinegar quality in the production process[J].China Brewing, 2020, 39(2):56-62.
[9] 何银, 唐杰, 张惟广.赤水晒醋中腐败微生物的生长特性研究[J].中国调味品, 2018, 43(2):17-22.
HE Y, TANG J, ZHANG W G.Study on the growth characteristics of spoilage microorganisms in Chishui Sun vinegar[J].China Condiment, 2018, 43(2):17-22.
[10] 杨新, 陈莉, 卢红梅, 等.赤水晒醋中增黏微生物的分离鉴定及特性研究[J].中国酿造, 2018, 37(5):54-59.
YANG X, CHEN L, LU H M, et al.Isolation, identification and characteristics of microorganism with viscosity increment from Chishui Sun vinegar[J].China Brewing, 2018, 37(5):54-59.
[11] 杨新, 卢红梅, 白成松, 等.赤水晒醋中污染杂菌的防治[J].中国调味品, 2018, 43(7):102-109.
YANG X, LYU H M, BAI C S, et al.Prevention and control of contaminated bacteria in Chishui Sun vinegar[J].China Condiment, 2018, 43(7):102-109.
[12] 李荣源, 卢红梅, 秦兴, 等.高通量测序分析赤水晒醋各生产阶段微生物群落结构变化[J].食品科学, 2020, 41(24):78-86.
LI R Y, LU H M, QIN X, et al.High-throughput sequencing study on changes in microbial community structure in different production stages of Chishui Sun-dried vinegar[J].Food Science, 2020, 41(24):78-86.
[13] 邓永建. 酿醋微生物群落结构变化与醋醅氧含量的相关性研究[D].无锡:江南大学, 2020.
DENG Y J.Study on correlation between microbial community structure change of vinegar brewing and oxygen content of Cupei[D].Wuxi:Jiangnan University, 2020.
[14] GUIA U R C, GUIA
U R C, GUIA U S.Conditional and weighted measures of ecological diversity[J].International Journal of Uncertainty Fuzziness and Knowledge-Based Systems, 2003, 11(3):283-300.
U S.Conditional and weighted measures of ecological diversity[J].International Journal of Uncertainty Fuzziness and Knowledge-Based Systems, 2003, 11(3):283-300.
[15] 王宗敏. 镇江香醋醋酸发酵阶段菌群结构变化与风味物质组成之间的相关性研究[D].无锡:江南大学, 2016.
WANG Z M.Correlation between structural shifts of microbiota and compositions of flavor during the stage of acetic acid fermentation of Zhenjiang aromatic vinegar[D].Wuxi:Jiangnan University, 2016.
[16] 李敏. 夏冬两季食醋微生物群落演替特征及其特征微生物研究[D].太原:太原理工大学, 2019.
LI M.Succession characteristics of microbiota and characteristic microbes in vinegar brewing during summer and winter[D].Taiyuan:Taiyuan University of Technology, 2019.
[17] 赵风琴. 凉州熏醋传统酿造过程主要功能微生物群落演替对四甲基吡嗪的影响[D].兰州:甘肃农业大学, 2016.
ZHAO F Q.The effects of major functional microbial succession on tetramethylpyrazine during the traditional brewing process of Liangzhou fumigated vinegar[D].Lanzhou:Gansu Agricultural University, 2016.
[18] 方冠宇, 穆晓静, 蒋予箭.浙江玫瑰醋发酵过程中细菌菌群结构变化与有机酸形成相关性分析[J].食品科学, 2019, 40(22):177-184.
FANG G Y, MU X J, JIANG Y J.Correlation between structural shifts of bacterial community and organic acid formation during the fermentation of Zhejiang rosy vinegar[J].Food Science, 2019, 40(22):177-184.
[19] 王俊奇, 黄卫红, 李双彤, 等.永春老醋不同生产阶段细菌和真菌多样性动态变化特征分析[J].食品与发酵工业, 2021, 47(2):38-44.
WANG J Q, HUANG W H, LI S T, et al.Dynamic changes of bacterial and fungal diversities in Yongchun aged vinegar during production and storage[J].Food and Fermentation Industries, 2021, 47(2):38-44.
[20] YU Y J, LI X, ZHANG J H, et al.Lactobacillus jinshani sp. nov., isolated from solid-state vinegar culture of Zhenjiang aromatic vinegar[J].Antonie Van Leeuwenhoek, 2020, 113(1):43-54.
[21] ZHENG J S, WITTOUCK S, SALVETTI E, et al.A taxonomic note on the genus Lactobacillus:Description of 23 novel genera, emended description of the genus Lactobacillus Beijerinck 1901, and union of Lactobacillaceae and Leuconostocaceae[J].International Journal of Systematic and Evolutionary Microbiology, 2020, 70(4):2 782-2 858.
[22] LISDIYANTI P, NAVARRO R R, UCHIMURA T, et al.Reclassification of Gluconacetobacter hansenii strains and proposals of Gluconacetobacter saccharivorans sp.nov. and Gluconacetobacter nataicola sp.nov[J].International Journal of Systematic and Evolutionary Microbiology, 2006, 56(Pt9):2 101-2 111.
[23] LU Z M, WANG Z M, ZHANG X J, et al.Microbial ecology of cereal vinegar fermentation:Insights for driving the ecosystem function[J].Current Opinion in Biotechnology, 2018, 49:88-93.
[24] 赵玲. 食醋中乙偶姻转化及巴氏醋杆菌乙偶姻还原酶研究[D].东营:中国石油大学(华东), 2018.
ZHAO L.Transformation of acetoin in vinegar and the acetoin reductase from Acetobacter pasteurii[D].Dongying:China University of Petroleum (East China), 2018.
[25] LAZRAQ A, CLÉROUX R.Testing the significance of the successive components in redundancy analysis[J].Psychometrika, 2002, 67(3):411-419.