大西洋鲑鱼(Salmo salar)是一种洄游性深海鱼类,其肉质鲜美,口感滑嫩,被誉为“鱼中至尊”。其富含二十碳五烯酸(eicosapentaenoic acid,EPA)和二十二碳六烯酸(docosahexaenoic acid,DHA)等多种生物活性物质[1],对高血压、肥胖等疾病均有益处,深受消费者的青睐。
大西洋鲑鱼常以刺身的形式出现在餐桌上,除此之外,还被加工成烟熏鲑鱼、油炸鲑鱼等不同风味的产品。然而,由于加工导致大西洋鲑鱼外形特征受损,以至于无法通过肉眼对鱼肉加工制品进行物种判别。在这种情况下,一些不法商贩利用低价的淡水虹鳟鱼等其他鱼类假冒替代高价的大西洋鲑鱼[2]。这种以假乱真、掺假造假的行为不仅严重损害了消费者的经济利益,更可能引发食品安全问题。因此,建立快速有效的大西洋鲑鱼鉴定方法迫在眉睫。
基于DNA的检测方法已经成为水产品中鱼类品种鉴定的重要手段[3]。常见的方法有DNA条形码[4]、荧光PCR(real-time PCR)[5]、多重PCR(multiplex PCR)[6]和环介导等温扩增(loop-mediated isothermal amplification,LAMP)[7]。其中,LAMP技术具有特异性强、灵敏度高等特点,且不需要昂贵的设备,仅需1台水浴锅即可在1 h内完成扩增[8],可以满足现场检测的需要。目前,LAMP技术已被用于欧洲鳗鱼[9]、金枪鱼[10]、美洲大赤鱿[11]等多种水产品的真伪鉴定研究。
高效快速的DNA提取有助于现场检测,而获得高质量的DNA更是保证LAMP反应顺利进行的关键。十二烷基磺酸钠(sodium dodecyl sulfonate,SDS)法是从动物肌肉组织中提取DNA的常用方法,联合利用蛋白酶K对细胞进行消化,可获得高纯度的DNA[12]。但提取过程操作繁琐、耗时长。为此,许多研究致力于研发更简便高效的提取方法。ANDREA等[13]在含有SDS的鱼肉组织裂解液中加入玻璃珠,通过振荡研磨加速细胞裂解,同时降低了成本。XIONG等[14]通过煮沸裂解液,加速了十六烷基三甲基溴化铵(cetyltrimethylammonium bromide,CTAB)对鱼肉组织的消化,将整个提取耗时缩短至15 min。TAGLIAVIA等[15]提出的碱裂解法简化了提取的步骤,通过使用氯化钾裂解细胞,同时加入TritonX-100以增强裂解效果,最终获得的DNA满足PCR扩增及后续分析。此外,使用商品试剂盒也能获得高纯度的DNA,但DNA损失量大,得率相对较低。
本研究以大西洋鲑鱼为对象,模拟设计了4种前处理方式,并用SDS、市售试剂盒等5种常见提取方法进行DNA提取(图1)。通过对比DNA得率及纯度,分析不同提取方法的优劣。同时,对5种提取方法所得DNA进行LAMP,结合琼脂糖凝胶电泳及荧光可视化检测,进一步验证这5种提取方法对LAMP技术的适用性。
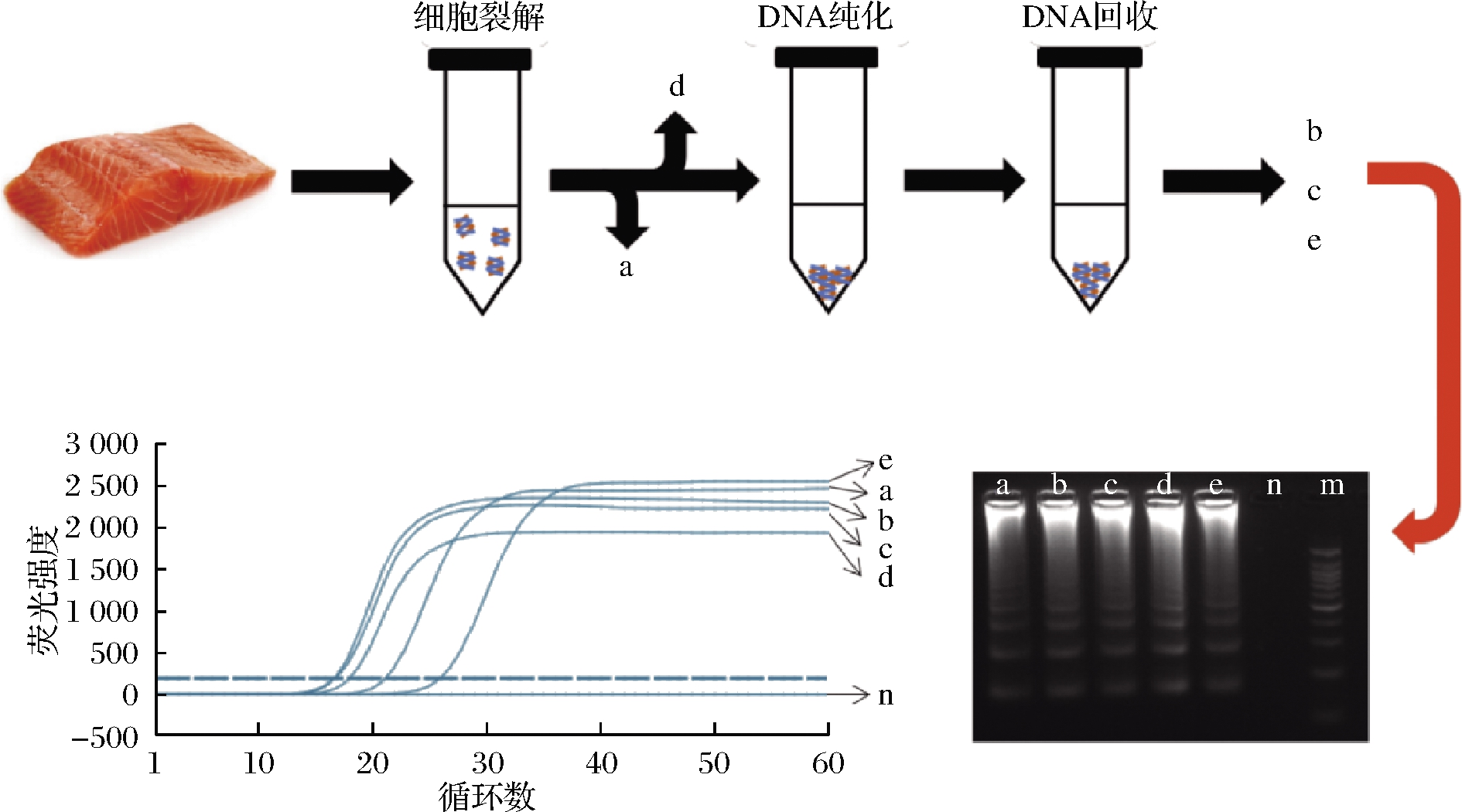
a-Takara;b-Tiangen;c-SDS;d-Tagliavia;e-Heating; n-无菌水;m-DL 100 marker
图1 不同DNA提取方法的比较研究示意图
Fig.1 Schematic procedure for the comparative study of different DNA extraction methods
1 材料与方法
1.1 材料与设备
1.1.1 样品采集
从南京市浦口区永辉超市采购1份新鲜的大西洋鲑鱼,样品抵达实验室后进行标记和拍照,按照样品制备方案进行处理。
1.1.2 实验试剂
细胞/血液/组织基因组DNA提取试剂盒(离心柱型)、100 bp DNA Ladder,天根生化科技(北京)有限公司;Takara细胞/血液/组织基因组DNA提取试剂盒,宝生物工程(大连)有限公司;2×Lamp PCR Master Mix(Universal)、引物合成、蛋白酶K、TritonX-100,生工生物工程(上海)股份有限公司;6×Loading Buffer,宝日医生物技术(北京)有限公司;琼脂糖凝胶,西班牙Biowest公司;GelRedTM核酸凝胶染料,美国Biotium公司;SYTOTM 9 green fluorescent nucleic acid stain,赛默飞世尔科技公司;SYBR Green Ⅰ 染料(10 000×),北京索莱宝科技有限公司;NaCl、NaH2PO4、Na2EDTA、异丙醇,西陇科学股份有限公司;SDS、Tris、KOH、CH3COONa,国药集团化学试剂有限公司;乙醇,无锡市亚盛化工有限公司;Tris-HCl,阿拉丁试剂(上海)有限公司。
1.1.3 仪器与设备
BioPhotometer D30核酸蛋白测定仪,德国艾本德股份有限公司;Sigma 1-14小型台式高速离心机,北京维欣仪奥科技发展有限公司;Tannon-1600凝胶成像仪,上海天能科技有限公司;JY300C凝胶电泳仪、JY-SPFT电泳槽,北京君意东方电泳设备有限公司;GWA-UNI-F40纯水/超纯水器,北京普析通用仪器有限责任公司;MS-100恒温混匀仪,杭州奥盛仪器有限公司;全自动医用PCR分析系统(Gentier 96E),西安天隆科技有限公司。
1.2 实验方法
1.2.1 样品制备
将待测鱼肉组织样本清洗后进行以下4种前处理:
(1)新鲜处理(Ax)为清洗后直接进行DNA提取;(2)冻藏处理(Bd)为-20 ℃贮藏6个月;(3)冷冻处理(Cl)为-20 ℃贮藏1周;(4)煎炸处理(Dy)为30 min盐腌后,煎炸5 min。
1.2.2 DNA提取
称取50 mg样本至1.5 mL离心管中,将样品剪碎后采用以下5种方法进行DNA提取。
1.2.2.1 Takara细胞/血液/组织基因组DNA提取试剂盒
按照试剂盒说明书提取DNA,最后移取200 μL上层清液至新的离心管并保存于-20 ℃。
1.2.2.2 细胞/血液/组织基因组DNA提取试剂盒
按照试剂盒说明书提取总DNA,最后用100 μL TE洗脱液洗脱DNA,室温放置5 min后以12 000 r/min 离心2 min,将溶液收集到离心管中并置于-20 ℃保存。
1.2.2.3 SDS法提取
根据ANDREA等[13]的提取方法并加以改进。在装有样品的离心管中加入200 μL裂解液I 20 g/L SDS,0.1 mol/L NaCl,0.1 mol/L EDTA,0.5 mol/L Tris,pH=8.0)、200 μL磷酸缓冲液(0.3 mol/L NaH2PO4,pH=8.0)和20 μL蛋白酶K(20 mg/mL),将其置于恒温混匀仪中,60 ℃处理60 min。14 000 r/min离心5 min后移取上清液转移至新的离心管,并加入0.5倍体积CH3COONa溶液(4 mol/L,pH=8.2)。混匀后14 000 r/min离心5 min。移取上层清液至新管,并加入0.6倍体积异丙醇,静置10 min。14 000 r/min离心10 min后,用800 μL 70%乙醇和无水乙醇分别对DNA清洗2次和1次。将装有DNA的离心管置于65 ℃烘箱中干燥20 min。最后,加入50 μL无菌水溶解DNA,置于-20 ℃保存。
1.2.2.4 Tagliavia法提取
参照TAGLIAVIA等[15]的提取方法。在装有样品的离心管中加入100 μL裂解液Ⅱ(200 mmol/L KOH,2 mmol/L Na2EDTA,2 g/L TritonX-100),置于恒温混匀仪中,60 ℃处理10 min。加入3倍体积的100 mmol/L Tris-HCl溶液,振荡混匀后置于恒温混匀仪中,85 ℃处理10 min。最后,移取200 μL上层清液至新管,置于-20 ℃保存。
1.2.2.5 Heating法提取
根据XIONG等[16]的提取方法并加以改进。加入200 μL裂解液Ⅲ(20 g/L SDS,2 mol/L NaCl,200 mmol/L KOH,0.5 mol/L Tris,pH=8,使用前需置于56 ℃烘箱加热至澄清)、200 μL磷酸缓冲液(0.3 mol/L NaH2PO4,pH=8.0),置于恒温混匀仪中,100 ℃ 处理10 min。14 000 r/min离心5 min后小心吸取200 μL上层水相至新管。随后加入等体积CH3COONa溶液(4 mol/L,pH=8.2),振荡混匀。14 000 r/min离心5 min,取200 μL上清液至新管,置于-20 ℃保存。
1.2.3 DNA浓度及纯度测定
采用BioPhotometer D30核酸蛋白测定仪检测DNA浓度、A260/A280和A260/A230的比值,以评估提取的DNA的质量和杂质去除情况。
1.2.4 LAMP反应体系的建立
本研究所用的大西洋鲑鱼特异性引物为本课题组前期研究成果[16]。
LAMP反应体系共计25 μL,各成分含量为:内引物Sal1-FIP、Sal1-BIP各0.8 μmol/L,外引物Sal1-F3、Sal1-B3各0.2 μmol/L,12.5 μL的2×Lamp PCR Master Mix,0.16 U/μL DNA聚合酶,0.2 μmol/L SYTO 9染料,50 ng模板DNA,无菌水补足至25 μL。LAMP扩增条件为65 ℃恒温扩增60 min,80 ℃保温10 min进行DNA聚合酶灭活,1 min收集1次荧光信号进行实时荧光观察。以10倍的浓度将DNA进行浓度梯度稀释(50 ng~5 pg),通过LAMP反应进行灵敏度评估。
LAMP扩增反应产物通过2%琼脂糖凝胶电泳进行分析。此外,通过添加0.1 μL SYBR Green Ⅰ(10 000×)在紫外下进行可视化检测。
1.2.5 数据分析
DNA的得率及纯度的结果采用平均值±标准差的形式表示。所有数据使用SPSS统计学软件进行统计分析,并采用单因素方差分析(one way analysis of variance)对DNA得率、纯度及Ct值进行显著性检验。P<0.05表示差异具有统计学意义。
2 结果与分析
2.1 DNA的浓度与纯度
2.1.1 DNA得率
5种不同提取方法的DNA得率如表1和图2所示。5种提取方法的DNA得率为(0.08±0.0.1)~(3.26±0.98) ng/g。在相同的前处理方式下,采用5种提取方法所得DNA得率均存在着极显著的差异(P<0.01)。其中,Tagliavia的得率最高[(3.26±0.98)ng/g],其次是Takara、Heating、SDS、Tiangen。而采用同一提取方法对不同前处理的样品进行DNA提取,除了Takara外,其他4种提取方法的DNA得率均存在着极显著的差异。
总体来看,Tiangen、SDS以及Heating这3种提取方法的DNA得率较低,这主要归因于它们都具备一定的DNA纯化步骤。纯化洗脱次数越多,DNA的损失越大。研究发现,DNA得率受样品加工处理的影响。对于煎炸处理的样品,DNA得率从为(0.10±0.03)~(3.26±0.98) ng/g,得率相差近30倍。导致该现象的原因主要有2个,一是高温处理导致DNA降解,从而对含量测定造成误差[17];二是盐腌及煎炸过程中残留的有机物对含量测定造成影响[18]。
表1 五种提取方法的DNA得率 单位:ng/g
Table 1 Yield of DNA extracted by five methods

编号TakaraTiangenSDSTagliaviaHeatingP值Ax1.43±0.51ab0.12±0.02Bc0.84±0.13ABbc1.66±0.47BCa0.75±0.22ABbc0.006∗∗Bd1.10±0.06a0.08±0.01Cc0.10±0.07Cc1.18±0.14Ca0.41±0.06Bb0.000∗∗Cl1.33±0.15b0.19±0.01Ad0.63±0.04Bcd2.30±0.60Ba1.03±0.03Abc0.000∗∗Dy1.10±0.13b0.10±0.03BCd1.02±0.19Abc3.26±0.98Aa0.72±0.25ABc0.000∗∗P值n.s.0.001∗∗0.000∗∗0.004∗∗0.039∗
注:最后一列及最后一行中,根据邓肯方差分析(Duncan)列出了P值,分别用于不同提取方法在同一前处理间的比较以及同一提取方法在不同处理间的比较;**差异极显著(P<0.01);*存在差异(P<0.05);n.s.表示无明显差异;上标大写字母(A~D)和小写字母(a~e)分别表示同一列和同一行中样品各指标的差异(下同)
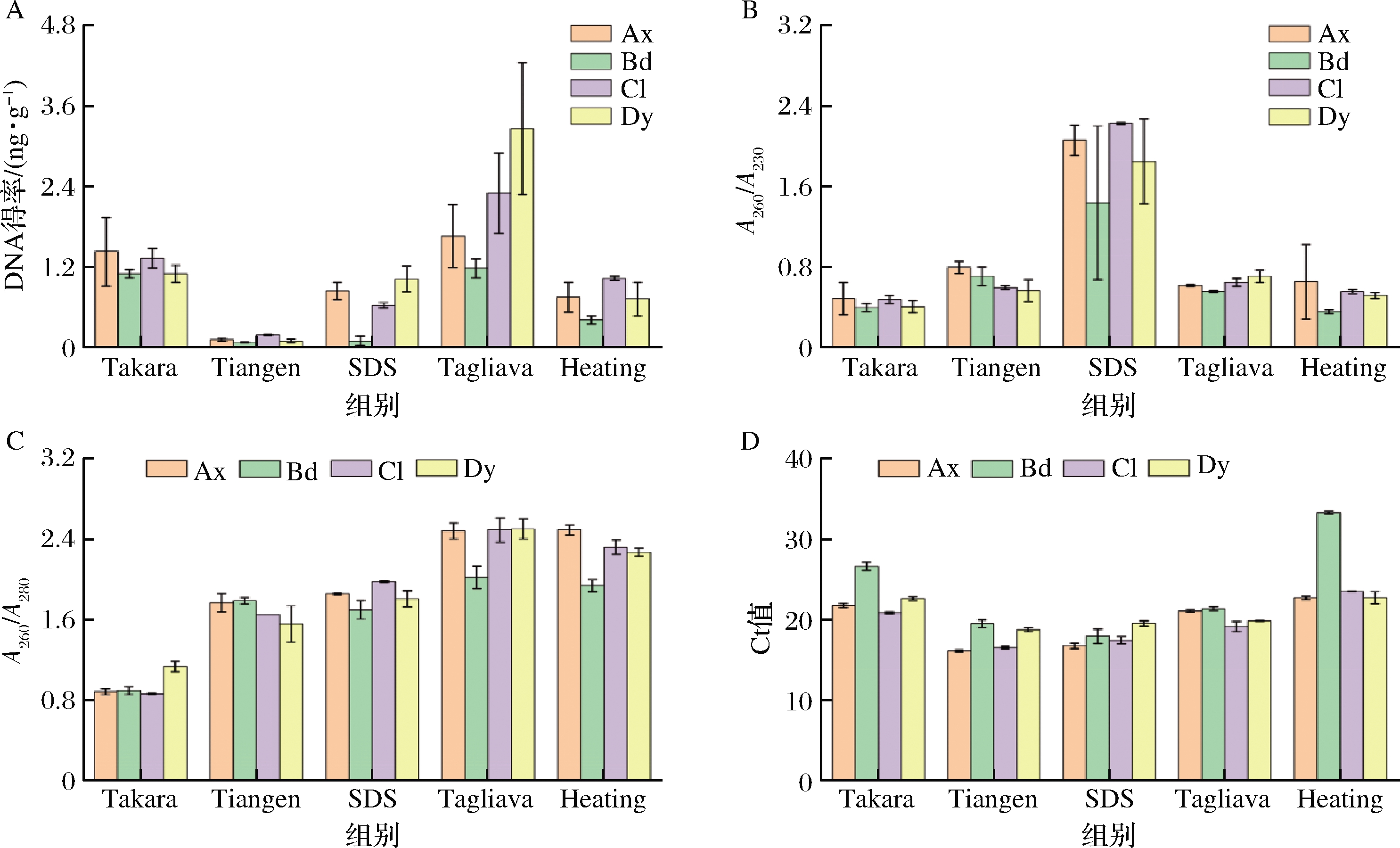
A-DNA得率;B-A260/A230;C-A260/A280;D-Ct值
图2 五种提取方法的DNA得率、纯度及LAMP反应的Ct值
Fig.2 Yield and purity of DNA with five extraction methods and Ct value of LAMP amplification
2.1.2 DNA纯度
5种不同提取方法的纯度测定结果如表2、表3和图2所示。在相同的前处理方式下,各种提取方法所得A260/A280值均存在着极显著的差异(P<0.01);而在5种提取方法中,仅有Tiangen提取DNA的A260/A280值在不同前处理间无显著差异。
已有研究表明,A260/A280为1.6~1.9的DNA粗提取液可被用于肉制品鉴定[19]。由表2可知,Tiangen、SDS等具有纯化步骤的提取方法所得DNA的A260/A280值在该范围内,由此说明了这2种提取方法均可获得高质量的DNA,更适合用于后期的检验工作。Heating及Tagliavia提取所得DNA的A260/A280值超出该范围,表明这2种方法导致残留较多的游离核苷酸、单链DNA和RNA,这些物质在260 nm处产生较高的吸收值。Takara的A260/A280值最小,表明了提取混合物中可能残留大量的蛋白质及其他杂质。
表2 五种提取方法所得DNA的A260/A280值
Table 2 A260/A280 values of DNA extracted by fiver methods

编号TakaraTiangenSDSTagliaviaHeatingP值Ax0.89±0.03Bc1.77±0.09b1.86±0.01Bb2.48±0.08Aa2.49±0.05Aa0.000∗∗Bd0.90±0.04Bc1.79±0.03b1.70±0.09Cb2.02±0.11Ba1.94±0.06Ca0.000∗∗Cl0.87±0.01Be1.65±0.00d1.98±0.01Ac2.49±0.12Aa2.32±0.07Bb0.000∗∗Dy1.14±0.05Ae1.56±0.18d1.81±0.08Bc2.50±0.10Aa2.27±0.04Bb0.000∗∗P值0.008∗∗n.s.0.002∗∗0.001∗∗0.000∗∗
表3 五种提取方法所得DNA的A260/A230值
Table 3 A260/A230 values of DNA extracted by fiver methods

编号TakaraTiangenSDSTagliaviaHeatingP值Ax0.49±0.16b0.80±0.06b2.06±0.15a0.62±0.01Bb0.66±0.37b0.000∗∗Bd0.40±0.040.71±0.091.44±0.760.56±0.01C0.36±0.02n.s.Cl0.48±0.04d0.60±0.02bc2.23±0.01a0.65±0.04ABb0.56±0.02c0.000∗∗Dy0.41±0.06c0.57±0.11bc1.85±0.42a0.71±0.06Ab0.52±0.03bc0.000∗∗P值n.s.n.s.n.s.0.003∗∗n.s.
A260/A230比值(一般范围在2.0~2.2)被用来反映DNA样品中是否存在碳水化合物、多肽或盐类等物质[20]。由图2可知,仅有SDS提取的样本DNA的A260/A230值接近该范围。这说明了SDS方法可消除大量的碳水化合物。而其他4种提取方法,不论采用何种前处理方式,所得DNA的A260/A230值都远低于正常范围,最高只有0.8±0.06。由此可知,加工程度及提取方法对DNA的A260/A230值都具有较大影响。
2.2 LAMP扩增
由图3可知,所有提取方法得到的DNA均能用于LAMP扩增,琼脂糖凝胶电泳结果均呈现出典型的梯形条带,荧光可视化检测结果也证实了LAMP反应成功地扩增了靶标。对于不具有纯化步骤的提取方法(Takara和Tagliavia),所得DNA同样可以通过LAMP反应进行扩增。这可能与DNA聚合酶的热稳定性以及LAMP技术对反应抑制物有着更高耐受性有关[21]。
在LAMP反应中添加SYTO 9染料可实现实时荧光检测。LAMP扩增曲线直观地展示出了纯化类及非纯化类的提取方法的差异,通过分析各样本的Ct值,发现该值在4种前处理及5种提取方法间均存在着极显著的差异(表4、图2)。
表4 五种提取方法的Ct值
Table 4 Ct value extracted by five methods
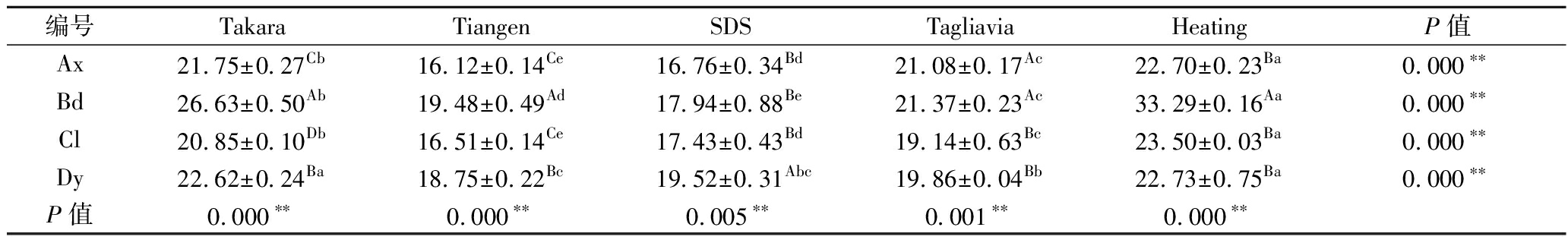
编号TakaraTiangenSDSTagliaviaHeatingP值Ax21.75±0.27Cb16.12±0.14Ce16.76±0.34Bd21.08±0.17Ac22.70±0.23Ba0.000∗∗Bd26.63±0.50Ab19.48±0.49Ad17.94±0.88Be21.37±0.23Ac33.29±0.16Aa0.000∗∗Cl20.85±0.10Db16.51±0.14Ce17.43±0.43Bd19.14±0.63Bc23.50±0.03Ba0.000∗∗Dy22.62±0.24Ba18.75±0.22Bc19.52±0.31Abc19.86±0.04Bb22.73±0.75Ba0.000∗∗P值0.000∗∗0.000∗∗0.005∗∗0.001∗∗0.000∗∗

a-Takara;b-Tiangen;c-SDS;d-Tagliavia;e-Heating;n-无菌水;m-DL 100 marker A-新鲜处理(Ax);B-冻藏(6个月)处理(Bd);C-冷冻(1周)处理(Cl);D-煎炸处理(Dy)
图3 用5种方法提取4种不同前处理样品的DNA进行LAMP扩增、琼脂糖凝胶电泳及可视化检测的结果
Fig.3 Results of LAMP amplification, agarose gel electrophoresis and visual detection of DNA form four different pretreatment samples extracted by five methods
在多数提取方法中,新鲜程度越高的样品可获得较小的Ct值。其中,Tiangen和SDS获得的DNA在LAMP反应中的Ct值最小。即这2种方法所得的DNA在LAMP反应中最早出现扩增信号,印证了这2种提取方法具有较好的纯化效果。其他3种提取方法的扩增曲线后移可能是DNA提取中使用的化学物质残留导致信号延迟。
2.3 不同提取方法的灵敏度测定
选择前处理为冻藏的DNA样品进行浓度梯度稀释,通过LAMP反应测定灵敏度。已有研究表明,熔解曲线分析可被用于物种鉴定分析[22]。因此,可结合实时荧光曲线和熔解峰曲线对不同提取方法用于LAMP反应的适用性进行评价。
由图4可知,Tiangen和SDS提取所得DNA最低检测可达5 pg;Heating可达0.05 ng;Tagliava可达0.5 ng;Takara可达5 ng。Tiangen和SDS的灵敏度高,熔解峰趋于重合,进一步展现了这2种提取方法的优势。同时,由图4可得大部分的熔解峰峰值在84.5~85 ℃。Heating、Tagliavia和Takara所得的熔解曲线出现偏离的峰,这很有可能是发生了非特异性扩增[23]。
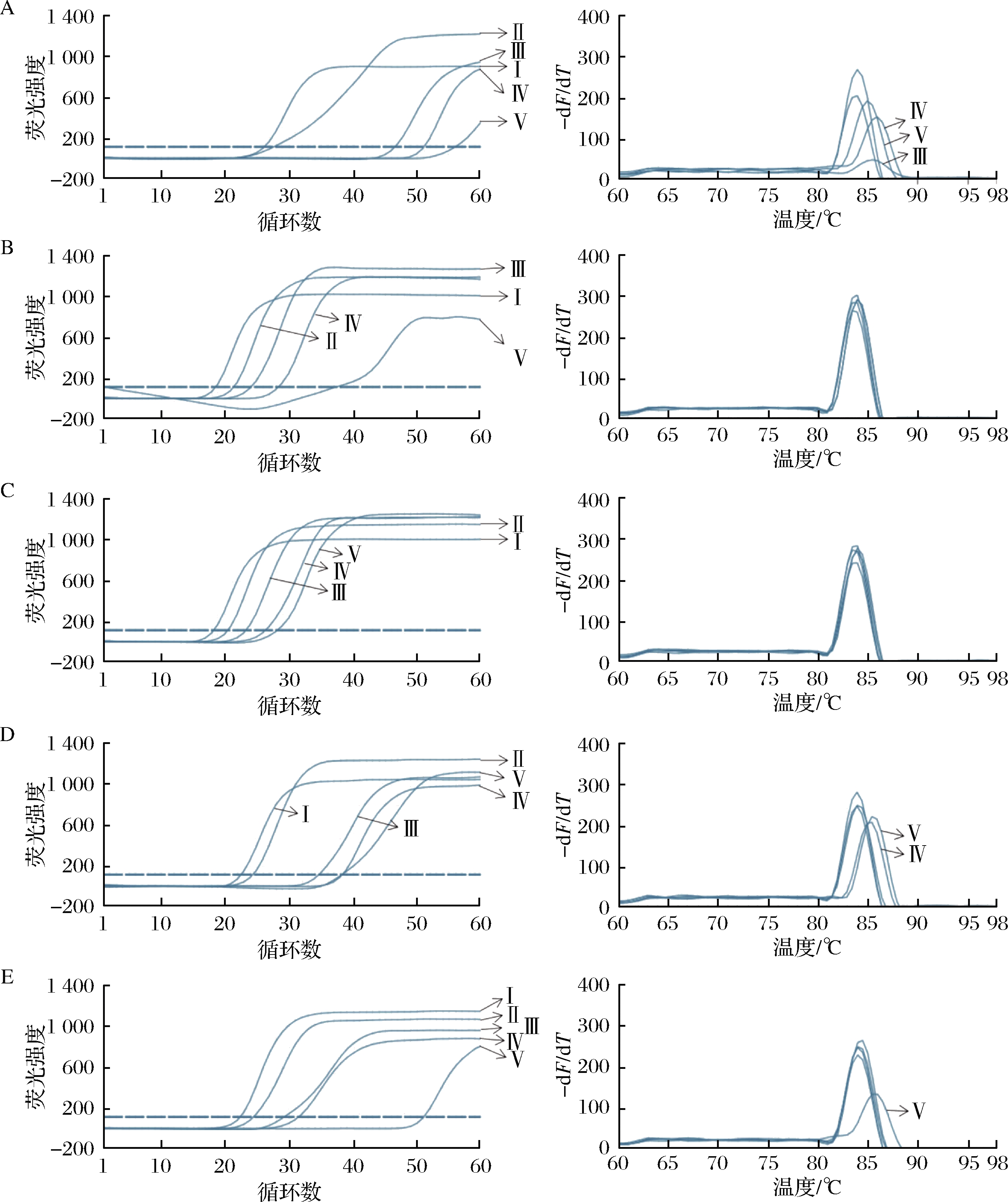
Ⅰ:50 ng/μL,Ⅱ:5 ng/μL,Ⅲ:0.5 ng/μL,Ⅳ:0.05 ng/μL,Ⅴ:5 pg/μL A-Takara;B-Tiangen;C-SDS;D-Tagliavia;E-Heating
图4 用LAMP反应对5种方法提取的冻藏处理样品的DNA进行灵敏度测定
Fig.4 Sensitivity determination of frozen sample DNA extracted by five methods using LAMP reaction 注:熔解曲线分析中,仅标注出84.5~85℃以外的峰
3 结论
本研究从DNA得率、纯度及LAMP反应等方面综合评价了5种核酸提取方法。总的来看,提取效果为Tiangen/SDS>Tagliavia>Takara>Heating。这5种提取方法从不同前处理的样品中获得的DNA均可用于LAMP反应,采用琼脂糖凝胶电泳及荧光可视化检测都可观测到阳性结果。根据Ct值进行比较分析,不同提取方法和不同的样品前处理会造成Ct值的显著性差异。Tiangen和SDS虽然DNA得率较低,但可为LAMP反应提供更高纯度和灵敏度的DNA,在LAMP反应中最早出现扩增信号。
[1] STONEHOUSE W, PAUGA M R, KRUGER R, et al.Consumption of salmon v.salmon oil capsules:Effects on n-3 PUFA and selenium status[J].The British Journal of Nutrition, 2011, 106(8):1 231-1 239.
[2] CAWTHORN D M, STEINMAN H A, WITTHUHN R C.DNA barcoding reveals a high incidence of fish species misrepresentation and substitution on the South African market[J].Food Research International, 2012, 46(1):30-40.
[3] KOTSANOPOULOS K V, EXADACTYLOS A, GKAFAS G A, et al.The use of molecular markers in the verification of fish and seafood authenticity and the detection of adulteration[J].Comprehensive Reviews in Food Science and Food Safety, 2021, 20(2):1 584-1 654.
[4] 王楠. 基于DNA条码技术的食品中鲑科鱼物种成分鉴别研究[D].泰安:山东农业大学, 2019.
WANG N.Identification of salmonid (Salmonidae) species in food using DNA barcoding[D].Taian:Shandong Agricultural University, 2019.
[5] 吴佳辉, 林霖, 朱成杰, 等.一种基于荧光PCR法检测虹鳟鱼源性成分方法的建立[J].食品科技, 2021, 46(3):283-287.
WU J H, LIN L, ZHU C J, et al.Detection of rainbow trout ingredients by fluorescence-based quantitative real-time PCR[J].Food Science and Technology, 2021, 46(3):283-287.
[6] XU W J, FU M Y, HUANG M H, et al.Duplex real-time PCR combined with melting curve analysis for rapid detection of Atlantic salmon (Salmo salar) and rainbow trout (Oncorhynchus mykiss)[J].Journal of Food Composition and Analysis, 2021, 97:103765.
[7] XIONG X, HUANG M H, XU W J, et al.Using real time fluorescence loop-mediated isothermal amplification for rapid species authentication of Atlantic salmon (Salmo salar) [J].Journal of Food Composition and Analysis, 2021, 95:103659.
[8] NOTOMI T, OKAYAMA H, MASUBUCHI H, et al.Loop-mediated isothermal amplification of DNA[J].Nucleic Acids Research, 2000, 28(12):e63.
[9] SPIELMANN G, ZIEGLER S, HASZPRUNAR G, et al.Using loop-mediated isothermal amplification for fast species delimitation in eels (genus Anguilla), with special reference to the European eel (Anguilla anguilla)[J].Food Control, 2019, 101:156-162.
[10] XIONG X, XU W J, GUO L Q, et al.Development of loop-mediated isothermal amplification (LAMP) assay for rapid screening of skipjack tuna (Katsuwonus pelamis) in processed fish products[J].Journal of Food Composition and Analysis, 2021, 102:104038.
[11] YE J, FENG J L, DAI Z Y, et al.Application of loop-mediated isothermal amplification (LAMP) for rapid detection of jumbo flying squid Dosidicus gigas (D′Orbigny, 1835)[J].Food Analytical Methods, 2017, 10(5):1 452-1 459.
[12] 许文杰, 熊雄, 黄曼虹, 等.金枪鱼制品中几种DNA提取方法的效果比较[J].现代食品科技, 2021, 37(7):57-65.
XU W J, XIONG X, HUANG M H, et al.Comparison of extraction effect of several DNA extraction methods in tuna products[J].Modern Food Science and Technology, 2021, 37(7):57-65.
[13] ANDREA A, LARA T, XIONG X, et al.Development of a simple and cost-effective bead-milling method for DNA extraction from fish muscles[J].Food Analytical Methods, 2014, 7(4):946-955.
[14] XIONG X, HUANG M H, YUAN F Y, et al.Development and validation of a fast DNA extraction protocol for fish products[J].Food Analytical Methods, 2019, 12(9):1 998-2 008.
[15] TAGLIAVIA M, NICOSIA A, SALAMONE M, et al.Development of a fast DNA extraction method for sea food and marine species identification[J].Food Chemistry, 2016, 203:375-378.
[16] XIONG X, HUANG M H, XU W J, et al.Tracing Atlantic salmon (Salmo salar) in processed fish products using the novel loop-mediated isothermal amplification (LAMP) and PCR assays[J].Food Analytical Methods, 2020, 13(6):1 235-1 245.
[17] ASLAN O, HAMILL R M, SWEENEY T, et al.Integrity of nuclear genomic deoxyribonucleic acid in cooked meat:Implications for food traceability[J].Journal of Animal Science, 2009, 87(1):57-61.
[18] YALÇ NKAYA B, YUMBUL E,
NKAYA B, YUMBUL E, ![]() E, et al.Comparison of DNA extraction methods for meat analysis[J].Food Chemistry, 2017, 221:1 253-1 257.
E, et al.Comparison of DNA extraction methods for meat analysis[J].Food Chemistry, 2017, 221:1 253-1 257.
[19] GIRISH P S, BARBUDDHE S B, KUMARI A, et al.Rapid detection of pork using alkaline lysis-Loop Mediated Isothermal Amplification (AL-LAMP) technique[J].Food Control, 2020, 110:107015.
[20] COSTA J, MELO V S, SANTOS C G, et al.Tracing tree nut allergens in chocolate:A comparison of DNA extraction protocols[J].Food Chemistry, 2015, 187:469-476.
[21] KANEKO H, KAWANA T, FUKUSHIMA E, et al.Tolerance of loop-mediated isothermal amplification to a culture medium and biological substances[J].Journal of Biochemical and Biophysical Methods, 2006, 70(3):499-501.
[22] BUT G W C, WU H Y, SHAO K T, et al.Rapid detection of CITES-listed shark fin species by loop-mediated isothermal amplification assay with potential for field use[J].Scientific Reports, 2020, 10(1):4 455.
[23] ROLANDO J C, JUE E, BARLOW J T, et al.Real-time kinetics and high-resolution melt curves in single-molecule digital LAMP to differentiate and study specific and non-specific amplification[J].Nucleic Acids Research, 2020, 48(7):e42.