重庆市是著名的泡菜之乡,很多家庭保持了使用泡菜坛和传统方法自制泡菜的习俗。传统泡菜制作的特点是在室温下发酵,由于泡菜坛有坛沿和水封结构而实现厌氧发酵,并且盐水一直留存于坛内持续发酵蔬菜数年甚至超过十年[1-2]。不同家庭由于食用的喜好而泡制的蔬菜品种存在差异,再加上具体的操作习惯、地域、环境等因素,自制泡菜富有多样性。通常衡量泡菜好坏的一个重要因素是盐水。优质盐水清彻透明,颜色淡黄,散发蔬菜自然的清香,可持续发酵产出浓厚地道风味的泡菜。劣质盐水反复出现“生花”现象,表现为盐水浑浊,有白色膜、团块或者颗粒漂浮于液面,有可能引起泡菜的异味或者变质。
目前关于生花现象的研究多集中于分离鉴定相关的微生物种类,主要的微生物是真菌,包括Pichia、Candida、Debaryomyces和Galactomyces属[3-7],生花是一种生物膜的现象,可能是酵母细胞彼此黏附,聚集在气液交界面,形成膜或者颗粒[6]。目前很少研究从盐水微生物群落结构的整体角度进行分析。盐水质量的好坏可能与其中的微生物群落结构有关。重庆泡菜的“老”盐水一般经历过数年的持续发酵,其间由于不断添加蔬菜或者环境等因素引入微生物,其中的微生物种类非常丰富,在长期发酵过程中形成具有一定动态平衡和稳定性的微生物群落结构,抵抗新进入的微生物,维持原有的菌种构成和丰度。因而优质的盐水能够持续保持优良的发酵性能,而“生花”的盐水则反复出现生花的现象。本论文采用Illumina MiSeq测序技术解析生花和不生花的优质盐水的真菌群落结构,对比它们之间的差异,分析相关的微生物信息。
1 材料与方法
1.1 实验材料
1.1.1 实验原料
采集重庆市涪陵区、渝北区、长寿区、垫江县、梁平区、武隆区连续发酵10年以上的家庭自制泡菜老盐水。取有明显的生花现象的盐水样品8份,记为生花组(P组);8份无生花现象盐水样品,记为不生花组(non-pellicle,NP组),部分样品照片如图1所示。

a-生花;b-不生花
图1 生花和不生花泡菜盐水样品照片
Fig.1 Pictures of Paocai brine samples with and without pellicle phenomenon
将样品装入已灭菌的样品瓶,置于有冰袋的保温箱中运回实验室。
1.1.2 药品试剂
硝酸银标准溶液(0.100 0 mol/L),天津渤化化学试剂有限公司;Premix Taq、TB Green Premix Ex Taq Ⅱ、RNase-free Water、pMD19-T Vecter Cloning Kit、DL2000DNA Ladder Marker、E.coli Competent Cells JM109,宝生物工程(大连)有限公司;DNeasy mericon Food Kit (69514),德国Qiagen公司。引物序列如表1所示,委托生工生物工程(上海)股份有限公司合成。
表1 引物序列
Table 1 The sequence of primers
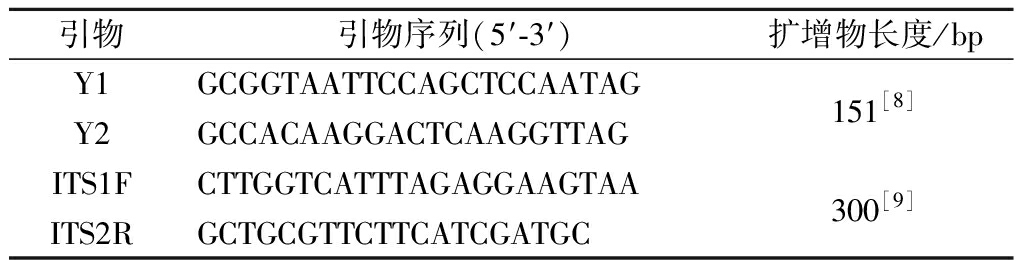
引物引物序列(5′-3′)扩增物长度/bpY1GCGGTAATTCCAGCTCCAATAGY2GCCACAAGGACTCAAGGTTAG151[8]ITS1FCTTGGTCATTTAGAGGAAGTAAITS2RGCTGCGTTCTTCATCGATGC300[9]
1.1.3 仪器设备
HBM-400D拍击式均质仪,天津市恒奥科技发展有限公司;CFX96 Real-time PCR仪、T100 PCR仪、Power pacbasic电泳仪,美国Bio-Rad公司;G:BOXEF凝胶成像系统,英国Syngene公司;P100/P100+超微量分光光度计,美国Pultton公司;Mini-Q超级纯水仪,法国Synergy公司。
1.2 实验方法
1.2.1 理化分析
泡菜盐水的pH值、可滴定酸、盐浓度、有机酸采用本实验室建立的方法检测[8]。亚硝酸盐浓度采用GB 5009.33—2016《食品安全国家标准 食品中亚硝酸盐与硝酸盐的测定》检测。
1.2.2 DNA的提取
将泡菜水均质,取20 mL以7 000×g转速离心15 min收集微生物沉淀颗粒,用1ⅹTE buffer洗涤沉淀颗粒,然后用DNeasy mericon food kit试剂盒按照说明书的步骤提取微生物的宏基因组DNA[8,10]。用P100/P100+超微量分光光度计检测DNA的浓度和纯度,用1%琼脂糖凝胶电泳检测DNA的质量。
1.2.3 qPCR法测定真菌的数量
用real-time PCR方法检测泡菜水中真菌数量。将标准菌株Saccharomyces cerevisiae ATCC 9763的ITS基因片段与质粒载体pMD19-T连接,构建标准质粒和标准曲线。qPCR反应体系组成是:12.5 μL premix buffer,0.5 μL Y1和 Y2引物 (10 mmol/L),2 μL DNA,补水至25 μL。qPCR反应程序是:94 ℃预变性45 s;94 ℃ 15 s、60 ℃ 15 s、72 ℃ 15 s,45次循环;最后72 ℃延伸5 min[8,11]。每个检测做3次平行。
1.2.4 微生物群落结构
提取的微生物宏基因组DNA送上海美吉生物医药科技有限公司,运用Illumina MiSeq PE300平台、ITS1F和ITS2R引物对真菌的ITS1区测序[8]。原始序列使用Trimmomatic软件质控[12],用FLASH软件进行拼接[13]:(1)设置50 bp的窗口,如果窗口内的平均质量值低于20,从窗口开始截去后端碱基,去除质控后长度低于50 bp的序列;(2)barcode需精确匹配,引物允许2个碱基的错配,去除模糊碱基;(3)根据重叠碱基overlap将两端序列进行拼接,overlap需>10 bp。去除无法拼接的序列。将获得的高质量序列采用QIIME包(quantitative insights into microbial ecology,定量研究微生物群落,version 1.9.1)[14]做生物信息分析。用UCLUST对序列在97%的相似度水平下进行聚类,获得分类操作单元(operational taxonomic units,OTUs)[15],将ITS序列基于Unite(unite7.0/its_fungi,Release 7.0,http://unite.ut.ee/index.php)[16]真菌数据库进行比对,比对阈值均为70%,得到每个OTU对应的物种分类信息,生成不同分类水平上的物种丰度表。用QIIME计算α多样性指数Sobs、Shannon、Simpson、Chao1、coverage。用R语言绘制Shannon指数图、物种构成图(门和属水平)、偏最小二乘判别分析(partial least squares discriminant analysis,PLS-DA)图,用Kruskal Wallis检验两组样品在属水平的物种差异并且绘制箱线图。用R语言的ggcorrplot包计算两组样品真菌群落结构与理化数据之间的相关性系数(Pearson相关)和绘制热图。部分数据分析在上海美吉生物医药科技有限公司的I-Sanger免费在线云平台中完成(https://www.i-sanger.com/)。
1.2.5 NCBI序列号
本研究中真菌的ITS1序列被存入NCBI的Sequence Read Archive(SRA)数据库(https://www.ncbi.nlm.nih.gov/sra),序列注册号为:No.PRJNA551794。
2 结果与分析
2.1 样品的信息、理化数据和生物量
表2列出了样品的采样信息及理化数据(pH值、可滴定酸、盐、亚硝酸盐和有机酸含量)和真菌生物量,分别计算了P和NP组数据的范围、平均值,采用独立样本t检验比较两组数据的差异,理化数据的差异不显著,真菌生物量的差异显著,P组样品真菌的平均拷贝数几乎是NP组的20倍,由此推测某些种类的真菌大量繁殖引起生花现象。赵慧君等[17]采用PCR-DGGE和培养法研究了襄阳大头菜正常和长膜醭腌制液中的真菌,发现2种腌制液酵母菌的种类相同且单一,均为鲁氏酵母和汉逊德巴利酵母,在有膜醭的大头菜腌制液中,鲁氏酵母和汉逊德巴利酵母的生物量显著增加,推断襄阳大头菜中的膜醭可能是由于鲁氏酵母和汉逊德巴利酵母的过度繁殖造成的,与本研究的结果和分析相似。MOON等[18]用平板计数法检测了韩国泡菜(kimchi)的生物膜形成过程中酵母的菌落总数,无论是粗糙型(R)还是光滑型(S)的菌落,其数量都呈明显的上升趋势,也能映证真菌数量的增加与生花现象有关。
表2 泡菜盐水样品的采样信息、生化指标和真菌生物量检测结果
Table 2 The information, chemical characteristics and fungal biomass of Paocai brine samples

分组样品采样地点老盐水连续发酵时间/年pH值可滴定酸/(g·L-1)盐质量浓度/(NaCl g·L-1)亚硝酸盐质量浓度/(mg·L-1)有机酸质量浓度/(g·L-1)乳酸醋酸琥珀酸真菌生物量/[lg (copies μL-1)]∗P组P1涪陵区>103.4311.784.891.273.550.515.514.75P2渝北区153.8010.367.703.653.400.673.384.52P3长寿区203.6110.4712.36 3.487.110.984.644.82P4垫江县203.3213.826.793.227.250.870.644.68P5垫江县403.2616.197.681.222.657.670.166.09P6垫江县303.3115.086.590.850.239.800.255.21P7梁平区153.488.209.614.121.753.780.525.66P8梁平区>103.4710.068.561.820.159.300.465.17平均值±标准差3.46±0.1812.00±2.778.02±2.242.45±1.303.26±2.734.20±4.091.95±2.205.11±0.54范围3.26~3.808.20~16.194.89~12.360.85~4.120.15~7.250.51~9.800.16~5.514.52~6.09NP组NP1涪陵区>203.3910.744.032.283.350.143.694.68NP2武隆区303.2813.933.993.054.160.551.964.48NP3渝北区183.3613.578.343.554.150.53.833.72NP4渝北区>103.3411.858.802.292.570.784.263.71NP5长寿区103.558.918.983.032.830.183.373.47NP6长寿区>103.4612.247.211.610.883.941.573.88NP7垫江县503.3315.246.051.900.5610.280.223.41NP8垫江县133.4111.068.281.390.175.420.924.05平均值±标准差3.39±0.0812.19±2.026.96±2.052.39±0.762.33±1.602.72±3.632.48±1.513.92±0.46范围3.28~3.558.91~15.243.99~8.981.39~3.550.17~4.160.14~10.280.22~4.263.41~4.68
*表示P和NP组之间真菌生物量存在显著性差异(独立样本t-检验)* P<0.05
2.2 Illumina MiSeq测序的基本数据和α多样性指数
采用Illumina MiSeq对泡菜盐水样品中的真菌多样性测序,测序的基本信息和α多样性指数如表3所示。所有样品的coverage均达到1.000,表示测序数据几乎覆盖了样品中所有的物种信息。采用独立样本t检验,P和NP组之间的4个α多样性指标(Sobs、Shannon、Simpson、Chao1)不存在显著性差异。
表3 泡菜盐水样品真菌多样性测序的基本信息和α多样性指数
Table 3 The basic information and α diversity indices of fungal community structure in Paocai brine samples with the use of Illumina MiSeq sequencing technology
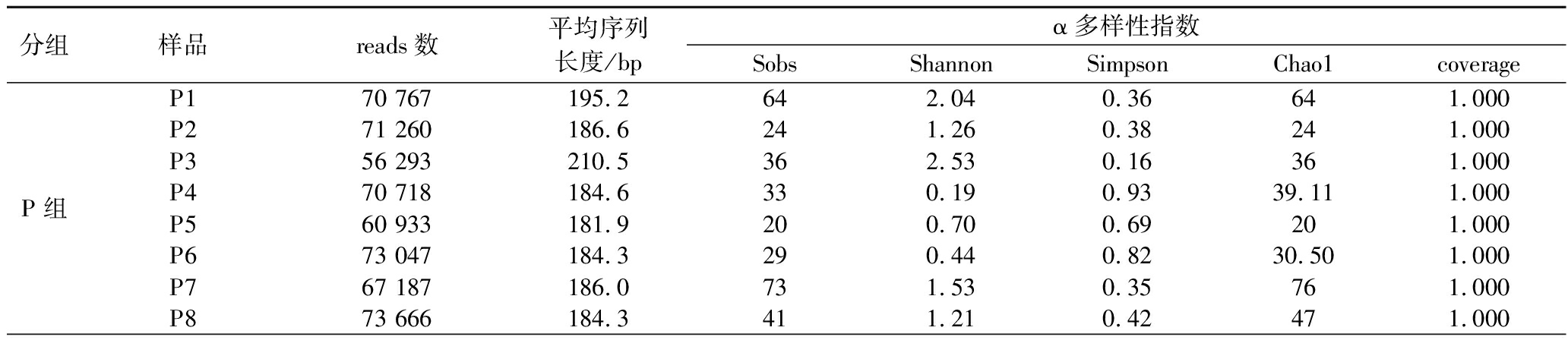
分组样品reads数平均序列长度/bpα多样性指数SobsShannonSimpsonChao1coverageP组P170 767195.2642.040.36641.000P271 260186.6241.260.38241.000P356 293210.5362.530.16361.000P470 718184.6330.190.9339.111.000P560 933181.9200.700.69201.000P673 047184.3290.440.8230.501.000P767 187186.0731.530.35761.000P873 666184.3411.210.42471.000
续表3

分组样品reads数平均序列长度/bpα多样性指数SobsShannonSimpsonChao1coverageP组平均值±标准差67 983.9±6 221.0189.2±9.540±18.931.24±0.790.51±0.2742.08±19.46范围56 293~73 666181.9~210.520~730.19~2.530.16~0.9320~76NP组NP163 562188.2541.280.56541.000NP250 401282.5352.880.08351.000NP370 253183.2580.900.66601.000NP437 919282.1371.940.28521.000NP569 684182.5430.740.69431.000NP674 283183.0501.010.57521.000NP740 860279.5462.090.26461.000NP860 299295.31681.460.42171.241.000平均值±标准差58 407.6±13 843.2234.5±54.061.38±43.801.54±0.720.44±0.2264.15±43.93范围37 919~74 283182.5~295.335~1680.74~2.880.08~0.6935~171.24
图2为盐水样品的真菌群落在OTU水平的Shannon指数曲线图,当reads数达到1 600 以上时,Shannon曲线达到了饱和状态,提示真菌的测序深度是足够的。

图2 泡菜盐水样品中真菌群落的Shannon 多样性指数曲线图
Fig.2 The Shannon diversity index curve of fungal community in Paocai brine samples
2.3 P和NP组真菌的群落结构
本研究采用Illumina MiSeq测序获得了P和NP组样品的真菌群落结构信息。总的来说,16个样品的序列被划分为386个OTU,鉴定为5个门和161个属,其中P组有177个OTU、5个门、74个属,NP组有286个OTU、5个门、140个属,两个组共有77个OTU、5个门和53个属。
P组中优势的门(平均相对丰度>1%,图3-a)有:Ascomycota(96.38%)、Basidiomycota(1.83%)。NP组优势的门有:Ascomycota(98.07%)、Basidiomycota(1.44%)。Ascomycota在15个样品中的相对丰度都超过90%,在样品P3中的丰度也高达83.98%,因此它是最优势的门。
P组优势的属(平均相对丰度>1%,图3-b)包括Pichia(85.22%)、Candida(2.07%)、Aspergillus (1.54%)、Fusarium(1.25%)。NP组优势的属有Pichia(46.04%)、Debaryomyces(21.75%)、Aspergillus (9.57%)、Kazachstania(5.31%)、Simplicillium(2.72%)、Penicillium(1.43%)、Wickerhamomyces(1.32%)、Fusarium(1.15%)、Candida(1.04%)、Schwanniomyces(1.03%)。Pichia是P组8个样品中相对丰度最高的属,它在P1和P3中的丰度分别是68.17%、41.47%,在其余6个样品中的丰度都超过了90%。NP组不同样品的优势菌属差异比较大,Pichia是样品NP1、NP3、NP5、NP6中最优势的菌属,其百分比分别为84.06%、93.10%、96.97%、94.19%。Debaryomyces是样品NP4、NP7和NP8中最优势的属,相对丰度分别为50.06%、48.06%、59.53%。样品NP2的优势属是Aspergillus(32.47%)。除了Trichosporon和未鉴定的属, 从P组中检出的属在NP组中也存在。

a-门水平;b-属水平
图3 P和NP组泡菜盐水样品中真菌群落在门、 属水平上的种类构成图
Fig.3 The composition of fungal community at phylum and genus levels in Paocai brine samples of groups P and NP 注:others,将平均相对丰度小于1%的门、属的 相对丰度归并为others;unclassified,未鉴定的门、属
2.4 P和NP组真菌群落结构在属水平的差异
如PLS-DA图(图4)所示,P与NP组的点呈现明显的分别聚类现象,提示两组的真菌群落结构有显著的差异。进一步用Kruskal Wallis检验分析得出两组样品中存在显著性差异的属为Debaryomyces和Kazachstania,如图5所示。从P组样品中没有检出Debar-yomyces和Kazachstania属,而在NP组样品中,Debaryomyces是最主要的属,Kazachstania的丰度也比较高。

图4 P和NP组泡菜盐水样品中真菌群落在属 水平上的PLS-DA图
Fig.4 PLS-DA graphs of fungal community in Paocai brine samples of groups P and NP at genus level
Debaryomyces属的菌株能够产生一种毒素,抑制成膜酵母菌的生长[19],可能是一个潜在的阻止“花”形成的因素。
2.5 P和NP组真菌群落结构与理化指标的相关性分析
图6为P和NP组真菌群落结构与理化指标的相关性热图。在P组中,盐浓度与Fusarium、Candida显著正相关,琥珀酸与Penicillium、Simplicillium、Aspergillus显著正相关,与Pichia显著负相关。在NP组中,真菌生物量与盐浓度之间显著负相关,Aspergillus、Simplicillium、Penicillium与pH值显著负相关。

图5 P和NP组泡菜盐水样品的真菌群落中存在 显著性差异的属
Fig.5 The genera with significant differences in the fungal community between P and NP groups of Paocai brine samples
盐浓度和酸(pH值、可滴定酸)通常是对泡菜盐水中微生物影响比较大的理化指标。根据图6,在NP组中,盐浓度与真菌生物量呈显著负相关,pH值与Aspergillus、Simplicillium、Penicillium相对丰度显著负相关,而在P组中,盐浓度与真菌生物量没有显著的相关性,而是与Fusarium、Candida显著正相关,pH值与真菌属之间也没有显著相关性,提示盐浓度和酸对真菌丰度的影响降低。现有文献报道从盐厂的高浓度盐水中分离出的Candida属菌株具有较强的耐盐性[20],这可能是Candida的相对丰度与盐浓度呈正相关的原因。

图6 P和NP组泡菜盐水样品中主要的真菌菌属与理化数据的相关性热图(Pearson相关)
Fig.6 The correlation heat-map between major fungal genera and physicochemical data in P and NP groups of Paocai brine samples (Pearson correlation)
从P组样品中检出的大部分属在NP组中也存在,即两组样品中真菌的属大部分是相同的。另一方面,real-time PCR的结果表明P组(生花)真菌的平均生物量(拷贝数)几乎是NP组(不生花)的20倍。
3 结论
本研究采用Illumina MiSeq测序技术解析了生花(P组)和不生花(NP组)盐水样品中真菌的群落结构。P组样品中主要的属包括:Pichia(85.22%)、Candida(2.07%)、Aspergillus(1.54%)、Fusarium(1.25%),NP组主要的属包括:Pichia(46.04%)、Debaryomyces(21.75%)、Aspergillus(9.57%)、Kazachstania(5.31%)、Simplicillium(2.72%)、Penicillium(1.43%)、Wickerhamomyces(1.32%)、Fusarium(1.15%)、Candida(1.04%)、Schwanniomyces(1.03%)。P组真菌的平均生物量(拷贝数)几乎是NP组的20倍。Kruskal Wallis检验发现两组的Debaryomyces和Kazachstania相对丰度存在显著性差异。分析推测生花现象可能与真菌数量的增加有关,NP组样品中Debaryomyces的存在可能是一个潜在的阻止“花”形成的因素。
[1] 饶箐, 尼海峰, 涂雪令, 等.四川泡菜的产品特点及产业技术发展浅析[J].食品与发酵科技, 2011, 47(4):1-4;9.
RAO Q, NI H F, TU X L, et al.Brief analysis on features and development of industrial technique of Sichuan pickles[J].Food and Fermentation Technology, 2011, 47(4):1-4;9.
[2] 赵楠. 四川泡菜的主要特性及其成因分析[D].无锡:江南大学, 2017.
ZHAO N.Analysis of the main characteristics of Sichuan Paocai and its formation mechanism[D].Wuxi:Jiangnan University, 2017.
[3] 张瑜, 郑伟, 谢婷婷, 等.腌制蔬菜“生花”微生物研究进展[J].广州化工, 2016, 44(2):33-35;49.
ZHANG Y, ZHENG W, XIE T T, et al.The development of “Shenghua”Microorganisms in salted vegetables[J].Guangzhou Chemical Industry, 2016, 44(2):33-35;49.
[4] 敖晓琳, 蔡义民, 夏姣, 等.引起泡菜“生花”腐败微生物的分离鉴定[J].食品科学, 2013, 34(21):204-208.
AO X L, CAI Y M, XIA J, et al.Isolation and identification of spoilage microorganisms from Sichuan pickles[J].Food Science, 2013, 34(21):204-208.
[5] 钟小廷, 樊君, 罗红刚, 等.泡菜生花酵母的鉴定及生理特性研究分析[J].食品与发酵科技, 2014, 50(5):19-22.
ZHONG X T, FAN J, LUO H G, et al.Identification and physiological characteristic analysis of yeast from Sichuan pickle[J].Food and Fermentation Technology, 2014, 50(5):19-22.
[6] 饶瑜, 常伟, 龚丽, 等.四川泡菜生花酵母的分离与鉴定[J].食品与发酵科技, 2013, 49(3):19-22.
RAO Y, CHANG W, GONG L, et al.Isolation and identification of spoilage microorganisms from Sichuan pickle[J].Food and Fermentation Technology, 2013, 49(3):19-22.
[7] CEN Q, CHEN T, ZHENG W, et al.Isolation, identification and partial characterization of film-forming microorganisms from Chinese homemade pickle, a traditional fermented vegetable product[J].Chiang Mai Journal of Science, 2018, 45(6):2 283-2 293.
[8] YANG J X, LI F Z, ZHANG Y L, et al.Metagenomic analysis of microbial community succession during the pickling process of Zhacai (preserved mustard tuber) and its correlation with Zhacai biochemical indices[J].Journal of the Science of Food and Agriculture, 2021, 101(4):1 646-1 658.
[9] ADAMS R I, MILETTO M, TAYLOR J W, et al.Dispersal in microbes:Fungi in indoor air are dominated by outdoor air and show dispersal limitation at short distances[J].The ISME Journal, 2013, 7(7):1 262-1 273.
[10] YANG J X, CAO J L, XU H Y, et al.Bacterial diversity and community structure in Chongqing radish Paocai brines revealed using PacBio single-molecule real-time sequencing technology[J].Journal of the Science of Food and Agriculture, 2018, 98(9):3 234-3 245.
[11] 李凤珠, 张玉礼, 杨吉霞.运用实时荧光定量PCR法研究榨菜腌制过程中细菌和真菌数量变化[J].食品与发酵工业, 2019, 45(18):58-64.
LI F Z, ZHANG Y L, YANG J X.Evaluation of quantity dynamics of bacteria and fungi during curing process of Zhacai by real-time quantitative PCR[J].Food and Fermentation Industries, 2019, 45(18):58-64.
[12] BOLGER A M, LOHSE M, USADEL B.Trimmomatic:A flexible trimmer for Illumina sequence data[J].Bioinformatics, 2014, 30(15):2 114-2 120.
[13] MAGO T, SALZBERG S L.FLASH:fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics (Oxford, England), 2011, 27(21):2 957-2 963.
T, SALZBERG S L.FLASH:fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics (Oxford, England), 2011, 27(21):2 957-2 963.
[14] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods, 2010, 7(5):335-336.
[15] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics, 2010, 26(19):2 460-2 461.
[16] ABARENKOV K, HENRIK NILSSON R, LARSSON K H, et al.The UNITE database for molecular identification of fungi-Recent updates and future perspectives[J].New Phytologist, 2010, 186(2):281-285.
[17] 赵慧君, 葛东颖, 沈馨, 等.襄阳大头菜腌制液中产膜醭酵母菌的多样性分析[J].中国酿造, 2018, 37(5):33-36.
ZHAO H J, GE D Y, SHEN X, et al.Diversity analysis of film-forming yeast in Xiangyang mustard root brine[J].China Brewing, 2018, 37(5):33-36.
[18] MOON S H, CHANG M, KIM H Y, et al.Pichia kudriavzevii is the major yeast involved in film-formation, off-odor production, and texture-softening in over-ripened Kimchi[J].Food Science and Biotechnology, 2014, 23(2):489-497.
[19] YOKOI K, NAKASHIMA M, SUZUKI C, et al.Inhibition of growth of pellicle forming yeasts by a killer toxin produced by a Debaryomyces hansenii KYT-1[J].Nippon Shokuhin Kagaku Kogaku Kaishi, 1996, 43(10):1 152-1 157.
[20] SEPCIC K, ZALAR P, GUNDE-CIMERMAN N.Low water activity induces the production of bioactive metabolites in halophilic and halotolerant fungi[J].Marine Drugs, 2010, 9(1):43-58.