头孢氨苄是一种β-内酰胺类抗生素,具有广谱抗菌性和低毒性,广泛应用于治疗由细菌引起的呼吸道、皮肤、泌尿道以及胃肠道等感染性疾病[1]。然而由于抗生素的广泛使用或过度使用,使其在治疗过程中对细菌产生一定的耐药性,造成一些公共安全问题。目前有关抗生素耐药性研究大多集中在致病菌,一旦致病菌获得耐药性就会造成临床治疗的失败[2]。随着人们对细菌耐药性研究的逐渐深入,益生菌耐药性的研究已受到人们的关注[3],当益生菌获得耐药性后,也许会对其益生效果产生积极作用,尤其是当益生菌与抗生素联合应用时,更有利于益生菌发挥益生作用。据报道,通过抗生素和益生菌的联合应用能协同治疗肠道腹泻、结肠炎、炎症性肠病等相关疾病[4]。已有研究将植物乳杆菌、鼠李糖乳杆菌、嗜酸乳杆菌和双歧杆菌与甲硝唑或万古霉素联合应用在治疗肠道疾病中,也取得一定的效果[5],这进一步促进了益生菌的应用。
植物乳杆菌是人体胃肠道中的一种固有益生菌,具有改善肠道菌群,抑制致病菌生长繁殖,促进肠道营养物质吸收利用的作用[6],广泛应用于各种发酵食品及保健食品中[7]。要发挥共生菌的益生效果,益生菌必须经过胃液的酸性环境和肠道的高胆盐环境,并能在肠道中定植[3],大多数益生菌对抗生素是敏感的[7],当有抗生素存在时,抗生素在杀死致病菌的同时也会导致益生菌的死亡,从而影响益生菌的效果。通过驯化可有效提高益生菌的耐药性[8],然而益生菌在获得耐药性的同时,其益生特性也许会发生相应改变,从而影响其益生效果。因此,本研究通过实验室进化方法将植物乳杆菌在头孢氨苄中进行耐药性驯化,并对进化菌株的益生特性进行评价,同时应用全基因组测序技术检测进化过程中菌株的基因突变,为植物乳杆菌与抗生素的联合应用提供基础。
1 材料与方法
1.1 实验材料
植物乳杆菌(ATCC 14917),中国普通微生物菌种保藏管理中心;金黄色葡萄球菌(ATCC 6538)、大肠杆菌(ATCC 8739)、沙门氏菌(CMCC 50071)、单核细胞增生李斯特菌(ATCC 15313),中国工业微生物菌种保藏管理中心;头孢氨苄,美国Sigma-Aldrich公司;de Man Rogosa Sharpe(MRS)培养基、Luria-Bertani(LB)培养基,北京奥博星生物技术有限责任公司;ISO(ISO-SENSITEST BROTH)培养基,英国oxoid公司;LSM培养基由90% ISO培养基和10% MRS培养基组成;牛胆盐、氯仿、二甲苯和乙酸乙酯均为分析纯,天津市天力化学试剂有限公司。
MRS培养基(g/L):蛋白胨10、葡萄糖20、乙酸钠5、硫酸镁0.2、硫酸锰0.05、牛肉粉5、酵母粉4、磷酸氢二钾2、柠檬酸三铵2、吐温80 1 mL,pH 6.2±0.2。
LB培养基(g/L):胰蛋白胨10、酵母提取物5、氯化钠10,pH 7.0。
ISO培养基(g/L):水解酪蛋白11、蛋白胨3、葡萄糖2、氯化钠3、可溶性淀粉1、磷酸氢二钠2、乙酸钠1、甘油磷酸镁0.2、葡萄糖酸钙0.1、硫酸钴0.001、硫酸铜0.001、硫酸锌0.001、硫酸亚铁0.001、氯化锰0.002、甲萘醌0.001、氰钴胺0.001、L-半胱氨酸盐酸盐0.02、L-色氨酸0.02、吡哆醇0.01,pH 7.4±0.2。
1.2 菌株的培养条件
菌株的培养条件按照ISO 10932/IDF223[9]的标准方法进行。菌株以1%接种量加入MRS培养基中经37 ℃,24 h培养进行活化,活化后的菌株以1%接种量在LSM培养基中经37 ℃,24 h传代培养,进行菌株的耐药性测定和实验室进化。
在进行菌株益生特性的测定时,将活化后的菌株在5 000×g下离心10 min,然后用0.9%(质量分数)的灭菌生理盐水将沉淀洗涤3次,再重悬于0.9%的灭菌生理盐水中,调整菌液浓度为109 CFU/mL后进行测定。
1.3 菌株耐药性的测定
菌株耐药性测定按照ISO 10932/IDF223[9]中的标准方法进行,菌株的耐药性用最低抑制浓度(minimum inhibitory concentration,MIC)表示。首先用LSM培养基配制头孢氨苄质量浓度为8 192 μg/mL的储备液,用0.22 μm的滤膜过滤,头孢氨苄质量浓度的检测范围为1~256 μg/mL,储备液用LSM培养基进行2倍梯度稀释。将活化后的菌株用LSM培养基稀释500倍,按照1%接种量接入含不同浓度头孢氨苄的稀释液中,在37 ℃下培养48 h后观察无肉眼可见细菌生长对应的最低抗生素浓度作为菌株的MIC值。
1.4 实验室进化
为了获得高耐药性的菌株,在实验室进化中选用半抑制浓度(IC50)作为菌株传代时抗生素的初始浓度,IC50是指能够抑制50%菌株生长的抗生素浓度[10]。本实验中,菌株在MRS培养基中经37 ℃活化24 h后,按1%接种量接入含8 μg/mL头孢氨苄的LSM培养基中,在37 ℃培养24 h,连续培养5 d,然后每5 d将LSM培养基中抗生素浓度增加1.5倍,继续培养进化,直到MIC值保持稳定,其中每5 d测定1次MIC值。为了探究菌株耐药性的稳定性,将获得的耐药性稳定的菌株在不含头孢氨苄的LSM培养基中继续传代培养,并再次测定其MIC值直到稳定。
1.5 菌株益生特性的测定
1.5.1 菌株耐酸和耐胆盐能力的测定
菌株耐酸能力的测定按照SUI等[11]的方法进行。用6 mol/L的盐酸将MRS培养基pH调整为2.0、3.0、4.0,将活化后的菌株以1%的接种量接种于不同pH的MRS培养基中,在37 ℃培养3 h,用MRS琼脂培养基在37 ℃培养24 h后进行菌落计数。
菌株耐胆盐能力的测定按照SUI等[11]的方法进行。在MRS培养基中加入牛胆盐,使胆盐质量浓度达到0、1、3、5 g/L,将活化后的菌株以1%的接种量接种于不同牛胆盐浓度的MRS培养基中,37 ℃培养3 h,用MRS琼脂培养基在37 ℃培养24 h后进行菌落计数。
1.5.2 菌株黏附能力的测定
菌株的黏附能力用细胞疏水性和自凝集能力评价[12]。
细胞疏水性的测定采用PATEL等[12]的方法。将活化后的菌株按1%接种量接入PBS缓冲液(0.1 mol/L,pH 7.2)中,测定菌悬液在600 nm处的吸光度作为A0,然后将菌悬液与等体积的氯仿、二甲苯和乙酸乙酯混合,涡旋振荡5 min,然后静置30 min,直至混合物分层,在600 nm处测量水相吸光度作为A30。细胞疏水率计算如公式(1)所示:
疏水率![]()
(1)
菌株自凝集能力的测定采用COLLADO等[13]的方法。将活化后的菌株悬浮在PBS缓冲液(0.1 mol/L,pH 7.2)中,用PBS缓冲液将吸光度(OD600nm)调节至0.25±0.05,然后在37 ℃静置,在静置2、16、20、24 h时,测量其在600 nm处的吸光度。自凝集率计算如公式(2)所示:
自凝集率![]()
(2)
式中:At代表2、16、20、24 h的OD600nm值;A0代表0 h的OD600nm值。
1.5.3 菌株抑菌能力的测定
采用牛津杯法测定菌株的抑菌能力[14]。将活化后的菌液在5 000×g离心10 min,上清液用0.22 μm滤膜过滤得到无细胞上清液。指示菌活化后在5 000×g离心10 min,重悬于无菌生理盐水中,调节指示菌浓度至106 CFU/mL,分别涂布于LB琼脂培养基中,室温下放置10 min后用牛津杯[内径(5.1±0.1)mm,外径(7.1±0.1)mm]均匀打孔。将无细胞上清液100 μL 加入打好的孔中,37 ℃培养24 h,利用游标卡尺测量抑菌圈的直径。
1.6 菌株全基因组测序分析
通过对实验室进化后的菌株进行全基因组测序,并与原始菌株基因相比,检测与耐药性和益生特性相关的突变基因。细菌DNA提取试剂盒(Qiagen,Valencia,CA,美国)提取样品的基因组DNA,采用微量紫外分光光度计和琼脂糖凝胶电泳法测定基因组DNA的浓度和纯度。提取的DNA样品在Illumina HiSeq 2500平台上进行测序,使用CLC Genomics Workbench v12.0软件进行质量控制、修剪、参考序列匹配、去重复、突变检测和产物注释,得到的数据用于生物信息学分析[15]。
1.7 数据统计分析
所有实验重复测定3次,所得数据用Excel软件进行统计分析,并利用DPS数据处理系统进行差异显著性分析,用Origin Pro 8.5软件进行绘图,数据表示为平均值±标准差。
2 结果与分析
2.1 植物乳杆菌对头孢氨苄耐药性的进化
植物乳杆菌在有头孢氨苄和无头孢氨苄条件下的耐药性进化结果见图1。
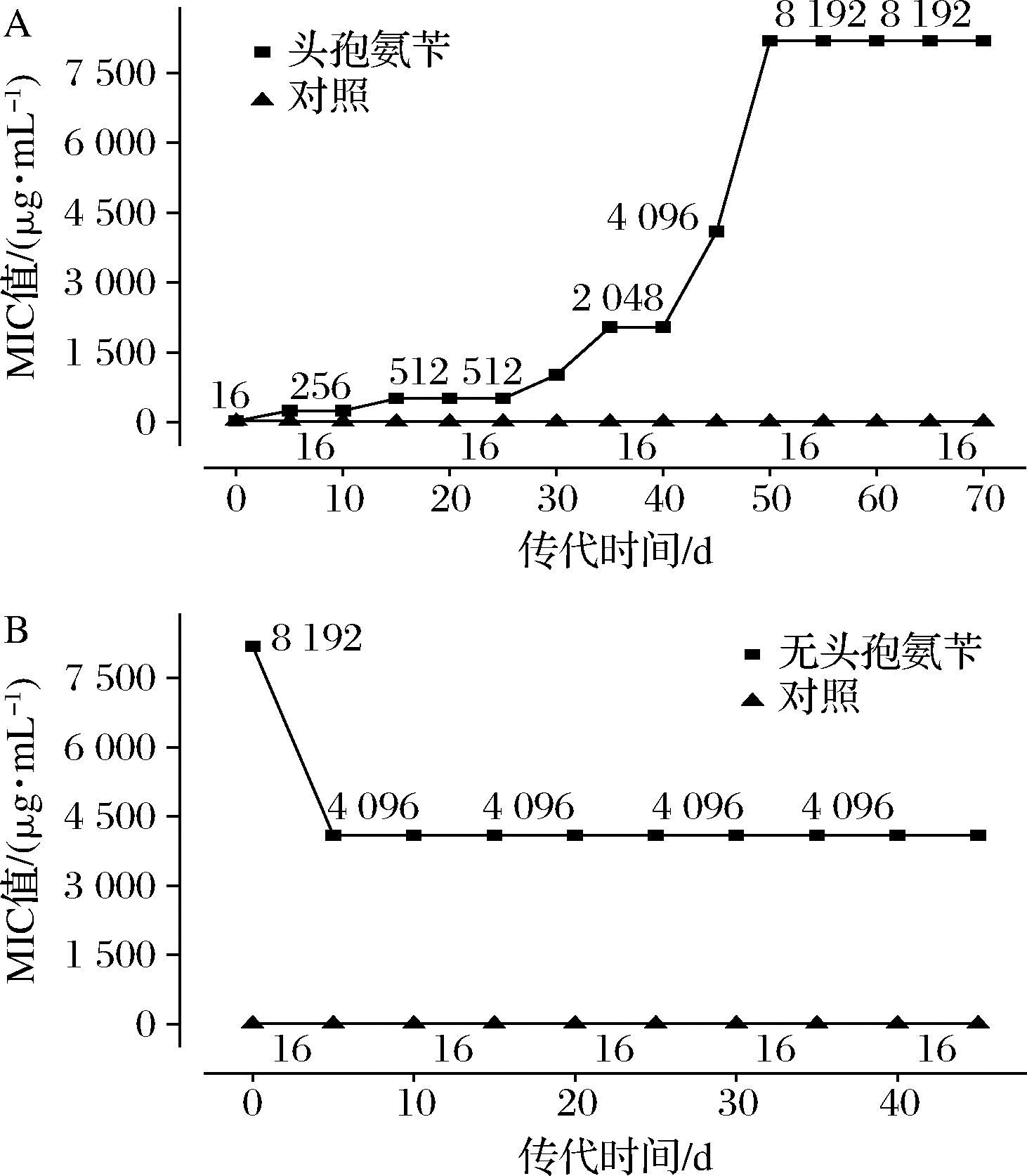
A-有头孢氨苄;B-无头孢氨苄
图1 植物乳杆菌在有、无头孢氨苄的培养基中的耐药性
Fig.1 The resistance of Lactobacillus plantarum in medium with or without cephalexin
注:对照为在无头孢氨苄的LSM培养基中传代的原始菌株
由图1可知,头孢氨苄对植物乳杆菌的初始MIC值为16 μg/mL,在头孢氨苄的存在下植物乳杆菌的MIC值逐渐提高,在进化50 d后植物乳杆菌的MIC值达到最大,为8 192 μg/mL,继续传代时MIC值保持稳定。为探究高耐药菌株的稳定性,将在头孢氨苄中传代后得到的菌株在去除头孢氨苄的培养基中继续传代培养(图1-B),发现菌株的MIC值在传代初期显著降低,经过5 d适应后,菌株的MIC值降至4 096 μg/mL,但在此后保持稳定。原始菌株(对照)在传代过程中MIC值为16 μg/mL,一直保持稳定。与原始菌株的MIC值相比,在有头孢氨苄条件下植物乳杆菌的MIC值提高了512倍,而在无头孢氨苄条件下植物乳杆菌的MIC值虽然有所降低,但比原始菌株也提高了256倍,这表明通过实验室进化植物乳杆菌对头孢氨苄耐药性的获得是不可逆的。
对植物乳杆菌进化期间的耐药性检测(表1)发现植物乳杆菌原始菌株为敏感型菌株,进化后的LpR′和LpR菌株都表现出高的耐药性,这也进一步说明通过实验室进化可获得高耐药型菌株。
表1 植物乳杆菌进化期间对头孢氨苄的耐药性
Table 1 The resistance of Lactobacillus plantarum to cephalexin during evolution

菌株MIC值/(μg·mL-1)临界值/(μg·mL-1)耐药性Lp01616SLpR′8 19216RLpR4 09616R
注:Lp0为原始菌株;LpR′为在头孢氨苄条件下传代的高耐药性菌株;LpR为在去除头孢氨苄后传代的高耐药性菌株;临界值采用EFSA标准(EFSA-Q-2004-079 Opinion of the Scientific Panel on Additives and Products or Substances used in Animal Feed on the updating of the criteria used in the assessment of bacteria for resistance to antibiotics of human or veterinary importance),MIC值>临界值时为耐药型(R),MIC值≤临界值为敏感型(S)
2.2 植物乳杆菌进化过程中益生特性的变化
2.2.1 植物乳杆菌的耐酸能力
植物乳杆菌进化过程中耐酸能力变化见图2。随着pH的降低,菌株活菌数有逐渐降低的趋势,尤其是当pH为2时活菌数显著降低。然而在相同pH条件下,原始菌株(Lp0)与在有头孢氨苄传代的高耐药性菌株(LpR′)和无头孢氨苄传代的高耐药性菌株(LpR)之间无显著差异(P>0.05),这表明植物乳杆菌在进化期间的耐酸能力保持稳定,不受进化过程的影响。
2.2.2 植物乳杆菌的耐胆盐能力
植物乳杆菌进化过程中耐胆盐能力变化见图3。随着培养基中胆盐浓度的增加,菌株的存活率逐渐降低,但在相同胆盐浓度的培养基中,植物乳杆菌原始菌株(Lp0)与在有头孢氨苄传代的高耐药性菌株(LpR′)和无头孢氨苄传代的高耐药性菌株(LpR)之间的存活率无显著性差异(P>0.05),表明植物乳杆菌在进化后的耐胆盐能力稳定。
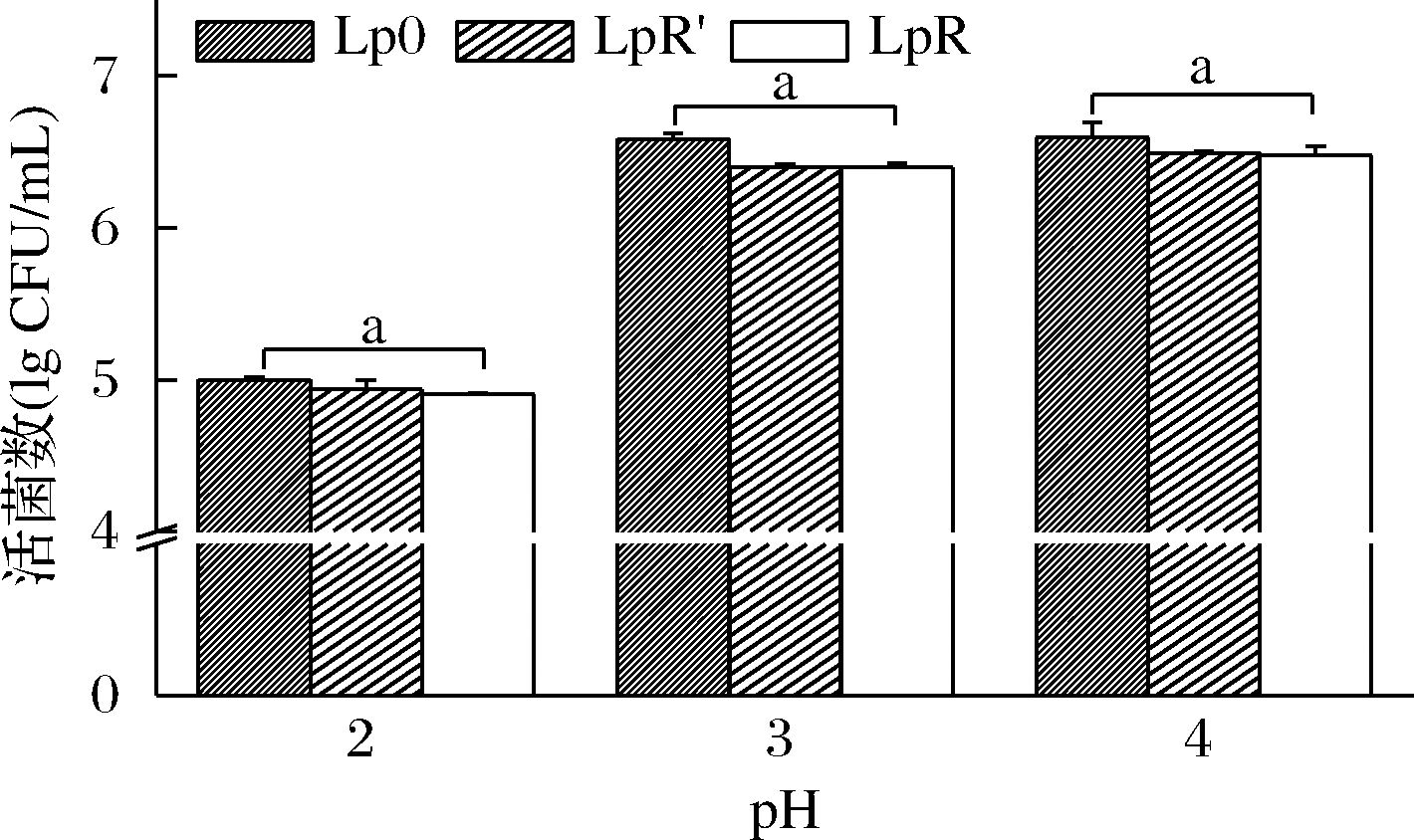
图2 植物乳杆菌在进化期间耐酸能力的变化
Fig.2 Changes in acid tolerance of Lactobacillus plantarum during evolution
注:Lp0为原始菌株;LpR′为在头孢氨苄条件下传代的高耐药性菌株; LpR为在去除头孢氨苄后传代的高耐药性菌株;a表示差异不显著 (P>0.05)(下同)
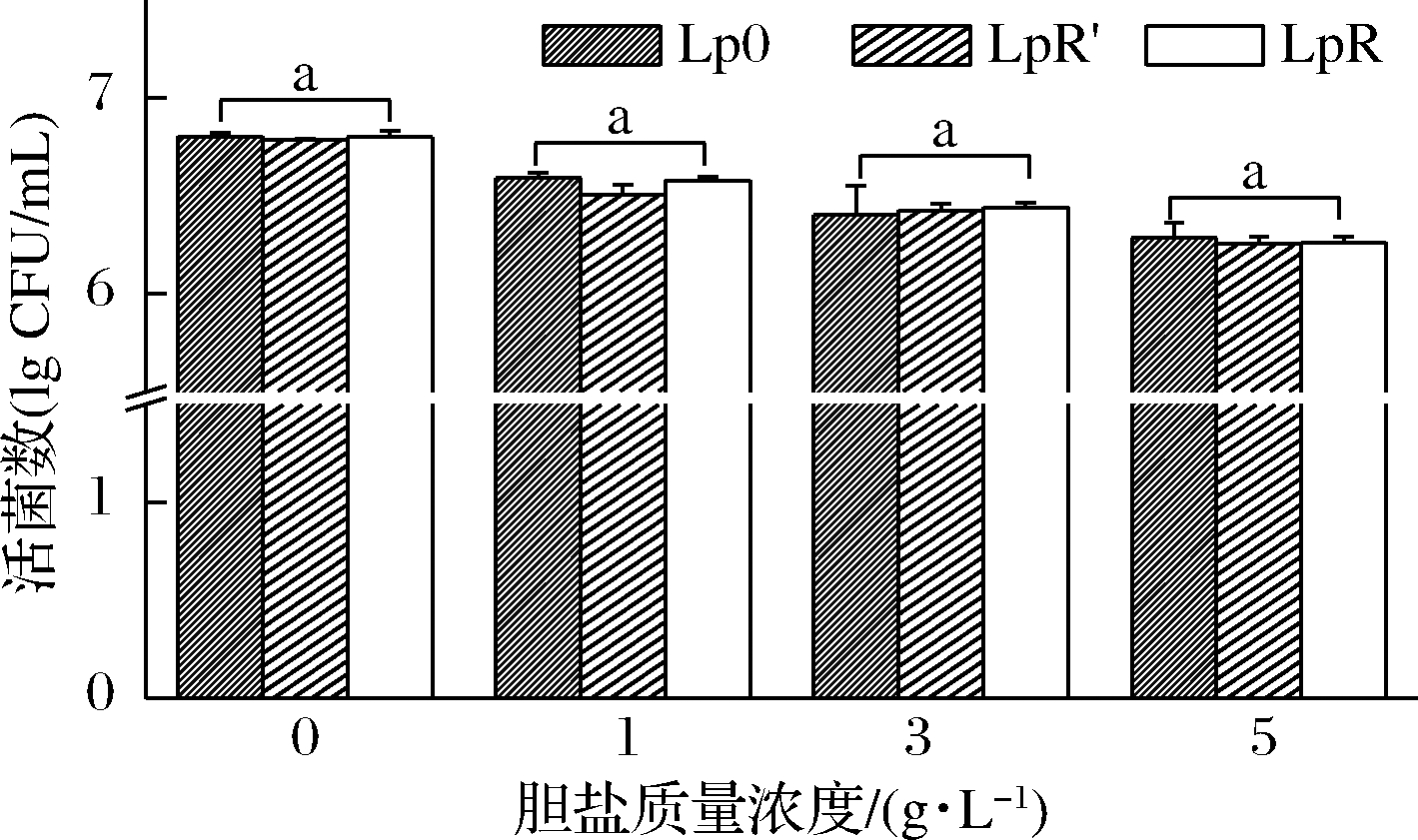
图3 植物乳杆菌在进化期间耐胆盐能力的变化
Fig.3 Changes in bile salts tolerance of Lactobacillus plantarum during evolution
2.2.3 植物乳杆菌的黏附能力
植物乳杆菌进化期间菌株的黏附特性用菌株的疏水性和自凝集能力来评价[12]。由表2可以看出,原始菌株(Lp0)和进化菌株(LpR′和LpR)的自凝集率在培养不同时间有一定的变化,在培养2 h时,Lp0、LpR′和LpR菌株的自凝集率较低,但在培养16、20、24 h后,菌株的自凝集率均有不同程度的提高。但在培养相同时间时,Lp0、LpR′和LpR菌株之间无显著差别(P>0.05),表明植物乳杆菌在进化后自凝集能力保持稳定。菌株在二甲苯、氯仿和乙酸乙酯溶剂中的疏水率有一定的差别,原始菌株的疏水率较低,但通过进化后的LpR′和LpR菌株的疏水率均有不同程度提高(P<0.05),且进化后的LpR′和LpR菌株间无显著差异(P>0.05),这表明植物乳杆菌在进化后的疏水性有所提高。
表2 植物乳杆菌进化期间的细胞疏水性和自凝集能力 单位:%
Table 2 Cell hydrophobicity and self-aggregation ability of Lactobacillus plantarum during evolution
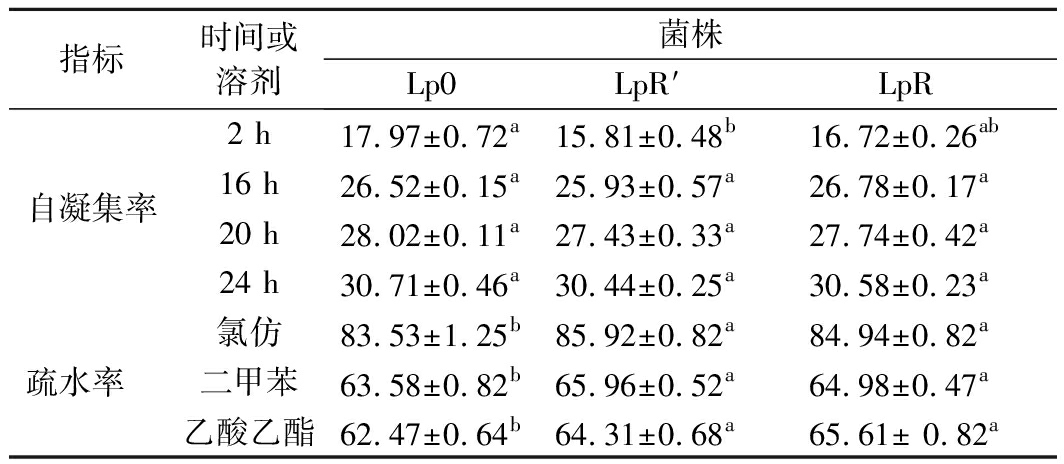
指标时间或溶剂菌株Lp0LpR′LpR2 h17.97±0.72a15.81±0.48b16.72±0.26ab自凝集率16 h26.52±0.15a25.93±0.57a26.78±0.17a20 h28.02±0.11a27.43±0.33a27.74±0.42a24 h30.71±0.46a30.44±0.25a30.58±0.23a氯仿83.53±1.25b85.92±0.82a84.94±0.82a疏水率 二甲苯63.58±0.82b65.96±0.52a64.98±0.47a乙酸乙酯62.47±0.64b64.31±0.68a65.61± 0.82a
注:同行不同小写字母表示差异显著(P<0.05)(下同)
2.2.4 植物乳杆菌的抑菌能力
植物乳杆菌进化期间菌株的抑菌能力见表3。植物乳杆菌对2种革兰氏阳性(G+)菌和2种革兰氏阴性(G-)菌均有一定的抑制作用。植物乳杆菌原始菌株(Lp0)的抑菌圈直径较大,具有较强的抑菌能力。通过进化后的LpR′和LpR菌株对4种致病菌也具有较好的抑菌性。与原始菌株(Lp0)相比,进化后的LpR′和LpR菌株对金黄色葡萄球菌(G+)抑制效果有所提高,而对大肠杆菌(G-)的抑制效果有轻微降低。总体而言,植物乳杆菌在进化后对单核细胞增生李斯特菌、金黄色葡萄球菌、大肠杆菌和沙门氏菌4种致病菌的抑制效果基本稳定。
表3 植物乳杆菌进化期间的抑菌能力
Table 3 Antimicrobial properties of Lactobacillus plantarum during evolution
指示菌抑菌圈直径/mmLp0LpR′LpR单核细胞增生李斯特菌(G+)13.44±0.44a14.11±0.10a14.09±0.07a金黄色葡萄球菌(G+)11.27±0.21b13.12±0.11a13.08±0.09a大肠杆菌(G-)13.20±0.08a12.16±0.10b12.13±0.12b沙门氏菌(G-)12.08±0.05a12.10±0.03a12.09±0.01a
2.3 植物乳杆菌进化菌株突变基因的分析
通过对植物乳杆菌进化后获得的高耐药性菌株LpR进行全基因组测序,并与原始菌株的基因序列比较,检出与耐药性和益生特性相关的突变基因及突变产物(表4)。在LpR菌株中共检测到3种与头孢氨苄相关的基因突变,其突变产物分别为耐药性MFS转运体(drug resistance MFS transporter)、丝氨酸型D-Ala-D-Ala羧肽酶(serine-type D-Ala-D-Ala carboxypeptidase)和推定折叠酶蛋白PrsA2(putative foldase protein PrsA2)。同时,在LpR菌株中也检测到2种与益生特性相关的基因突变,其突变产物分别为YibE/F样蛋白(YibE/F-like protein)和推定折叠酶蛋白PrsA2(putative foldase protein PrsA2)。这些突变基因编码的突变产物在植物乳杆菌进化过程中对其耐药性和益生特性起至关重要的作用。
表4 植物乳杆菌进化中与耐药性和益生特性相关的基因突变
Table 4 Gene mutations related to resistance and probiotic properties in the evolution of Lactobacillus plantarum
相关性染色体突变位置突变类型基因/基因标识突变产物GL379762355219..355237SVlmrBdrug resistance MFS transporter, drug:H+ antiporter-2 family耐药性 GL379764324360..352924SVdacASerine-type D-Ala-D-Ala carboxypeptidaseGL379764324360..352924SVPrsAputative foldase protein PrsA2益生特性GL379762336765..355219SVHMPREF 0531_11168YibE/F-like proteinGL379764324360..352924SVPrsAputative foldase protein PrsA2
注:SV为结构突变
3 讨论
细菌的耐药性通常通过自然进化产生,这种进化过程具有相当大的随机性和不可控性[16-17]。当细菌形成耐药性后可使一些抗生素的治疗失败,然而益生菌获得耐药性后也许对益生作用产生积极影响,尤其是当益生菌与抗生素联合使用时效果更加显著[18]。因此,有关益生菌耐药性驯化已日益受到人们的关注[19]。为了进行可控的益生菌耐药性驯化,实验室进化已广泛应用,GEORGE等[19]对植物乳杆菌在氨基糖苷类抗生素中进行进化,获得高耐药性菌株,我们从前也对植物乳杆菌在氨基糖苷类抗生素中进行驯化,获得了高耐药性的菌株[20]。本研究中植物乳杆菌(ATCC 14917)原始菌株对头孢氨苄表现为敏感型,通过在头孢氨苄存在下进行实验室进化,其耐药性提高了512倍,但这种耐药性不稳定,在去除头孢氨苄条件后进一步进行进化时,其耐药性有所降低,经过一段时间适应后,其耐药性达到4 096 μg/mL,并在此后进化中保持稳定,耐药性比原始菌株提高了256倍,表明通过实验室进化后可获得高耐药性的植物乳杆菌菌株,并且这种耐药性的获得是不可逆的。
益生菌产生益生作用的前提是能够通过胃液的酸性环境和肠道的高胆盐环境,并以活菌的状态通过胃肠道并定植在肠道中,这样才能发挥益生作用[3],当益生菌获得耐药性后,其益生性状可能发生改变,因此,对益生菌的益生特性评价十分必要。本研究中,对获得高耐药性的植物乳杆菌菌株在酸性环境(pH 2、3、4)及高胆盐环境(1、3、5 g/L)中进行培养,与原始菌株相比,其菌株的存活率无明显变化(P>0.05)。在菌株的黏附特性中,细胞疏水性对比原始菌株有一定的提高。这表明当植物乳杆菌获得耐药性后,其主要益生特性保持稳定,一些性状如疏水性有所提高,这为高耐药性植物乳杆菌的应用提供了一定的基础。
益生菌进化过程中的表型特性(包括耐药性和益生特性)的改变本质上与进化过程中菌株的基因突变有关。通过对进化过程中菌株突变基因的检测,可进一步从分子水平阐明表型特性变化的机制。我们从前对植物乳杆菌在庆大霉素进化后的耐药性菌株的基因检测发现lmrB、HMPREF0531_10299和HMPREF0531_11866基因是提高植物乳杆菌对庆大霉素耐药性的主要因素[20]。本研究中,应用全基因组测序技术对植物乳杆菌进化后获得的高耐药性菌株基因突变检测发现,突变基因lmrB、dacA和PrsA可能是增强植物乳杆菌耐药性的主要原因,lmrB编码的突变产物耐药性MFS转运体可以刺激细菌产生多药转运蛋白,在渗透压促进裂解物的跨膜运动中可以排出抗生素,以保护细胞免受损伤[21],为植物乳杆菌在进化过程中耐药性的提高发挥了重要作用。dacA编码的丝氨酸型D-Ala-D-Ala羧肽酶是一种外肽酶,这种酶从肽聚糖胞壁五肽的侧链取代末端的D-丙氨酸,使其不能与抗生素结合从而产生耐药性[22]。PrsA编码的推定折叠酶蛋白PrsA2,已被证明可调节β-内酰胺类抗生素的易感性[23],该蛋白可能通过调节分泌蛋白维持耐药性菌株在传代后期耐药性的稳定。同时检测到与益生特性相关的突变基因,HMPREF 0531_11168和PrsA。HMPREF 0531_11168编码的YibE/F样蛋白(YibE/F-like protein)家族中的序列与乳酸菌表达的2种蛋白质YibE和YibF相似,这些序列包含高比例的疏水性残基[24],这对菌株在进化过程中保持一定水平的疏水性起着重要作用。PrsA编码的推定折叠酶蛋白PrsA2会改变细胞壁或膜蛋白谱以及与细胞表面相关的表型[25],可以推测该突变产物在进化过程中与菌株的细胞表面疏水性的提高有关。
4 结论
通过实验室进化方法对植物乳杆菌在头孢氨苄中进行进化,获得了具有高耐药性的植物乳杆菌,并对其主要益生特性进行评价,发现获得的高耐药性植物乳杆菌具有稳定的耐酸,耐胆盐能力以及较好的黏附能力,并对一些常见致病菌具有稳定的抑菌效果,其益生性状稳定。同时应用全基因组测序技术对进化过程中突变基因检测,检测到了与耐药性和益生特性相关的突变基因,这为植物乳杆菌与抗生素联合应用的治疗提供基础。
[1] 王永红, 冉茂娟, 龙晓莉.莫西沙星联合头孢氨苄治疗老年患者下呼吸道细菌感染的临床疗效[J].中华医院感染学杂志, 2015,25(3):572-574.
WANG Y H, RAN M J, LONG X L.The clinical effect of moxifloxacin combined cefalexin in the treatment of lower respiratory tract bacterial infections in elderly patients[J].Chinese Journal of Nosocomiology, 2015,25(3):572-574.
[2] ZAMAN S B, HUSSAIN M A, NYE R, et al.A review on antibiotic resistance:Alarm bells are ringing[J].Cureus, 2017, 9(6):e1403.
[3] MARCO M L, PAVAN S, KLEEREBEZEM M.Towards understanding molecular modes of probiotic action[J].Current Opinion in Biotechnology, 2006, 17(2):204-210.
[4] PREIDIS G A, VERSALOVIC J.Targeting the human microbiome with antibiotics, probiotics, and prebiotics:Gastroenterology enters the metagenomics era[J].Gastroenterology, 2009, 136(6):2 015-2 031.
[5] BANERJEE P, MERKEl G J, BHUNIA A K.Lactobacillus delbrueckii ssp.bulgaricus B-30892 can inhibit cytotoxic effects and adhesion of pathogenic Clostridium difficile to Caco-2 cells[J].Gut Pathogens, 2009, 1(1):8.
[6] DE VRIES M C, VAUGHAN E E, KLEEREBEZEM M, et al.Lactobacillus plantarum-survival, functional and potential probiotic properties in the human intestinal tract[J].International Dairy Journal, 2006, 16(9):1 018-1 028.
[7] GUEIMONDE M, S NCHEZ B, DE LOS REYES-GAVIL
NCHEZ B, DE LOS REYES-GAVIL N C G, et al.Antibiotic resistance in probiotic bacteria[J].Frontiers in Microbiology, 2013, 4:202.
N C G, et al.Antibiotic resistance in probiotic bacteria[J].Frontiers in Microbiology, 2013, 4:202.
[8] HORINOUCHI T, FURUSAWA C.Understanding metabolic adaptation by using bacterial laboratory evolution and trans-omics analysis[J].Biophysical Reviews, 2020, 12(3):677-682.
[9] GENEVA I O F S, BRUSSELS I D F.Milk and milk products.Determination of the minimal inhibitory concentration (MIC) of antibiotics applicable to bifidobacteria and non-enterococcal lactic acid bacteria (LAB)[S].2010.
[10] L Z
Z R V, PAL SINGH G, SPOHN R, et al.Bacterial evolution of antibiotic hypersensitivity[J].Molecular Systems Biology, 2013, 9(1):700.
R V, PAL SINGH G, SPOHN R, et al.Bacterial evolution of antibiotic hypersensitivity[J].Molecular Systems Biology, 2013, 9(1):700.
[11] SUI L, ZHU X M, WU D Y, et al.In vitro assessment of probiotic and functional properties of Bacillus coagulans T242[J].Food Bioscience, 2020, 36(1):100675.
[12] PATEL A K, AHIRE J J, PAWAR S P, et al.Comparative accounts of probiotic characteristics of Bacillus spp.isolated from food wastes[J].Food Research International, 2009, 42(4):505-510.
[13] COLLADO M C, MERILUOTO J, SALMINEN S.Adhesion and aggregation properties of probiotic and pathogen strains[J].European Food Research and Technology, 2008, 226(5):1 065-1 073.
[14] HUANG R H, TAO X Y, WAN C X, et al.In vitro probiotic characteristics of Lactobacillus plantarum ZDY 2013 and its modulatory effect on gut microbiota of mice[J].Journal of Dairy Science, 2015, 98(9):5 850-5 861.
[15] FENG C J, ZHANG F X, WANG B N, et al.Genome-wide analysis of fermentation and probiotic trait stability in Lactobacillus plantarum during continuous culture[J].Journal of Dairy Science, 2020, 103(1):117-127.
[16] JANG S, KIM M, HWANG J, et al.Tools and systems for evolutionary engineering of biomolecules and microorganisms[J].Journal of Industrial Microbiology & Biotechnology, 2019, 46(9-10):1 313-1 326.
[17] ARNOLD F H, WINTRODE P L, MIYAZAKI K, et al.How enzymes adapt:Lessons from directed evolution[J].Trends in Biochemical Sciences, 2001, 26(2):100-106.
[18] SOLEYMANZADEH MOGHADAM S, KHODAII Z, FATHI ZADEH S, et al.Synergistic or antagonistic effects of probiotics and antibiotics-alone or in combination-on antimicrobial-resistant Pseudomonas aeruginosa isolated from burn wounds[J].Archives of Clinical Infectious Diseases, 2018, 13(3):e63121.
[19] GEORGE J, HALAMI P M.Subinhibitory concentrations of gentamicin triggers the expression of aac (6′)Ie- aph (2″)Ia, chaperons and biofilm related genes in Lactobacillus plantarum MCC 3011[J].Research in Microbiology, 2017, 168(8):722-731.
[20] DONG Y S, ZHANG F X, WANG B N, et al.Laboratory evolution assays and whole-genome sequencing for the development and safety evaluation of Lactobacillus plantarum with stable resistance to gentamicin[J].Frontiers in Microbiology, 2019, 10:1 235.
[21] FLUMAN N, BIBI E T.Bacterial multidrug transport through the lens of the major facilitator superfamily[J].Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 2009, 1 794(5):738-747.
[22] SEKIGUCHI J I, MIYOSHI-AKIYAMA T, ![]() E, et al.Detection of multidrug resistance in Mycobacterium tuberculosis[J].Journal of Clinical Microbiology, 2007, 45(1):179-192.
E, et al.Detection of multidrug resistance in Mycobacterium tuberculosis[J].Journal of Clinical Microbiology, 2007, 45(1):179-192.
[23] NCBI (National Center for Biotechnology Information).prsA2-Foldase protein PrsA 2 precursor-Streptococcus pyogenes serotype M1-prsA2 gene & protein[DB].2021.
[24] NCBI (National Center for Biotechnology Information).CDD Conserved Protein Domain Family:YibE-F[DB].Accessed Nov.19, 2015.
[25] GUO L, WU T, HU W, et al.Phenotypic characterization of the foldase homologue PrsA in Streptococcus mutans[J].Molecular Oral Microbiology, 2013, 28(2):154-165.