在一定条件下微生物细胞团聚在一起形成稳定、连续的多细胞联结现象称为微生物聚集[1]。微生物的聚集现象在自然界中广泛存在,如酿造行业中的酵母细胞的絮凝现象[2],废水处理中以絮体污泥、生物被膜和颗粒污泥3种形态存在的微生物聚集体[3],某些原生动物和单细胞藻类也具有絮凝或聚集现象[4]。生理上,聚集是微生物对不利环境采取的策略,如酿酒酵母形成絮凝后提高了对乙醇的耐受性[5],丁香假单胞菌细胞聚集体更能耐受环境胁迫[6],在干燥表面形成聚集(生物膜)的鲍曼不动杆菌存活时间更长[7],聚集的金黄色葡萄球菌对紫外线的耐受性显著高于非聚集型菌株[8]。
微生物聚集对其抗逆过程有着重要影响,但在微生物计数方面却带来了困难,尤其是微生物的直接计数过程,如平板计数法、显微镜计数法、图像计数法、流式细胞仪法等,导致对微生物细胞计数的低估[9]。所以探索有效的促进微生物在水溶液中的分散方法,对微生物计数有着重要意义。
本文以酿酒酵母为研究对象,从分散指数和分布类型角度,研究了不同分散方法对酿酒酵母细胞在水溶液中分散效果的影响,并进行正交优化实验,得到优化的分散条件,旨在为微生物细胞在溶液中的分散提供参考。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
酿酒酵母(Saccharomyces cerevisiae)ATCC 9080,本实验室保藏菌株。
1.1.2 试剂
0.01 mol/L PBS(pH=7.0)、乙二胺四乙酸(ethylene diamine tetraacetic acid,EDTA),上海生工生物工程有限公司;吐温-20,国药集团化学试剂有限公司;酵母膏胨葡萄糖(yeast peptone dextrose,YPD)液体培养基、马铃薯葡萄糖琼脂培养基(potato dextrose agar,PDA),青岛海博生物技术有限公司。
1.2 仪器与设备
BM1000生物显微镜,南京江南永新光学有限公司;SHP-100智能生化培养箱,上海三发科学仪器有限公司。
1.3 试验方法
1.3.1 酿酒酵母培养过程中的生长及分散情况
种子液的培养:从平板中挑取酿酒酵母单菌落,接种于100 mL YPD培养液中,于30 ℃、160 r/min培养16 h后,得到种子液。
酿酒酵母生长变化:接种0.5 mL种子液于50 mL YPD培养液的150 mL锥形瓶,于30 ℃、160 r/min培养18 h。从0 h开始,每隔3 h取培养液,1 500 r/min离心5 min,用同体积无菌PBS重悬后,在光学显微镜下观察酿酒酵母形态及分散情况,并用血球计数板测定细胞浓度。
1.3.2 酿酒酵母菌体的分散
1.3.2.1 酿酒酵母细胞分散指数测定
取培养至16 h的酿酒酵母菌液,在1 500 r/min离心5 min,菌泥用PBS洗涤1次后,将细胞浓度稀释至106个/mL,再次离心后,1份菌泥用0.01 mol/L PBS重悬作为对照组,另一份进行分散处理,用血球计数板分别测定分散处理前后的酿酒酵母细胞数量。细胞分散指数的计算如公式(1)所示:
(1)
式中:I,细胞分散指数;T1,加入分散剂酿酒酵母血球计数板5个中格细胞总数,T0,对照组酿酒酵母血球计数板5个中格细胞总数。
1.3.2.2 酿酒酵母分散处理单因素实验
(1)吐温-20对酿酒酵母分散性的影响
在无菌PBS中分别加入体积分数为0.2%、0.4%、0.6%、0.8%、1.0%的吐温-20,经0.22 μm滤膜过滤后得到不同浓度的PBST2(PBS-吐温-20)溶液。取培养至16 h的酿酒酵母菌液,用0.01 mol/L PBS稀释至细胞浓度为106个/mL,1 500 r/min离心5 min后,菌泥以不同浓度的PBST2重悬,放置15 min后,用血球计数板对不同处理组的酿酒酵母进行计数,计算不同浓度吐温-20处理的酿酒酵母分散指数。
(2)EDTA对酿酒酵母分散性的影响
取培养至16 h的酿酒酵母菌液,1 500 r/min离心5 min,用无菌PBS洗涤并稀释至细胞浓度为106个/mL,再次离心后,以2、4、6、7、8、9、10、12 mmol/L的无菌PBS-EDTA缓冲液重悬,用血球计数板对不同处理组的酿酒酵母进行计数,计算不同浓度EDTA处理的酿酒酵母分散指数。
(3)离子强度对酿酒酵母分散性的影响
取培养至16 h的酿酒酵母菌液,于1 500 r/min离心5 min,用无菌PBS洗涤并稀释至细胞浓度为106个/mL,再次离心后分别以0.01、0.02、0.03、0.04、0.05、0.06、0.07、0.08、0.09、0.1 mol/L的无菌PBS重悬后,用血球计数板对不同处理组的酿酒酵母进行计数,以0.01 mol/L PBS处理组为对照组,计算不同离子强度条件下的酿酒酵母分散指数。
(4)pH对酿酒酵母分散性的影响
配制pH为4.0、5.0、6.0、7.0、8.0、9.0的无菌PBS。取培养至16 h的酿酒酵母菌液,用无菌PBS洗涤并稀释至细胞浓度为106个/mL,以不同pH的PBS重悬后,用血球计数板对不同处理组的酿酒酵母进行计数,pH 7.0为对照组,计算不同pH条件下的酿酒酵母分散指数。
(5)超声波处理对酿酒酵母分散性的影响
取培养至16 h的酿酒酵母菌液,用无菌PBS洗涤并稀释至细胞浓度为106个/mL,置于低频超声波(50 Hz)中分别处理5、10、15、20、30、40、45、50、60 s后,用血球计数板对不同处理组的酿酒酵母进行计数,未处理组为对照组,计算不同超声波处理时间条件下的酿酒酵母分散指数。
1.3.3 酿酒酵母活性测定
对分散处理后的酿酒酵母菌液依据GB 4789.15—2016《食品安全国家标准 食品微生物学检验 霉菌和酵母计数》进行浓度测定,分析不同处理对酿酒酵母活性的影响。
1.3.4 酿酒酵母分散效果正交优化试验
根据单因素试验的结果,选择对酿酒酵母分散影响显著的因素,确定正交试验及其水平,进行正交试验设计,考查其对酿酒酵母细胞分散的影响,对其结果进行优化,比较分析后得出最优分散组合。
1.3.5 酿酒酵母分散处理前后在溶液中分布类型分析
对不同分散处理前后的酿酒酵母菌液,在显微镜下随机计数20个视野内的细胞数[10],分析分散处理对酿酒酵母在溶液中分布类型的影响。
1.4 数据分析方法
采用Excel 2019进行数据整理,利用Origin 2021分析与作图,利用SPSS 26对数据进行显著性分析,P<0.05表示差异显著。每个处理组设置3个平行,结果以平均值±标准差表示。
2 结果与分析
2.1 酿酒酵母的培养过程观察
由图1可知,随着培养时间的增加,酿酒酵母细胞浓度逐渐变大,细胞之间的聚集越来越多,当培养时间达到9 h,细胞浓度为1.7×108个/mL时,观察发现酿酒酵母细胞聚集在一起(图1-c),表明随着培养时间延长,细胞浓度增大,细胞聚集加重。在YPD中进行好氧培养时,酿酒酵母以出芽的方式繁殖。由于营养充分,酿酒酵母细胞连续出芽形成呈藕节状的假菌丝(图1-b);若出现不同方向的出芽繁殖,会形成分支假菌丝(图1-d)。
通过对不同培养阶段中假菌丝的观察,在培养时间为0~12 h,营养充足的条件下,酿酒酵母细胞由单细胞出芽繁殖形成假菌丝形态,并且随着培养时间的增加,酵母细胞增殖速度加快,组成假菌丝细胞串中的细胞数越来越多。当培养进入稳定期(12~18 h),由于培养液中营养成分消耗以及细胞密度过大(图1-f),酿酒酵母细胞增殖速度减缓,细胞逐渐从假菌丝串脱落。当培养时间为15 h时(图1-e),观察到视野中假菌丝基本消失,虽是稀释10倍条件下,但细胞聚集仍然存在。在分析酿酒酵母分散性的过程中,为排除假菌丝形成的聚集对结果判断的影响,选用培养时间为16 h的细胞进行实验。
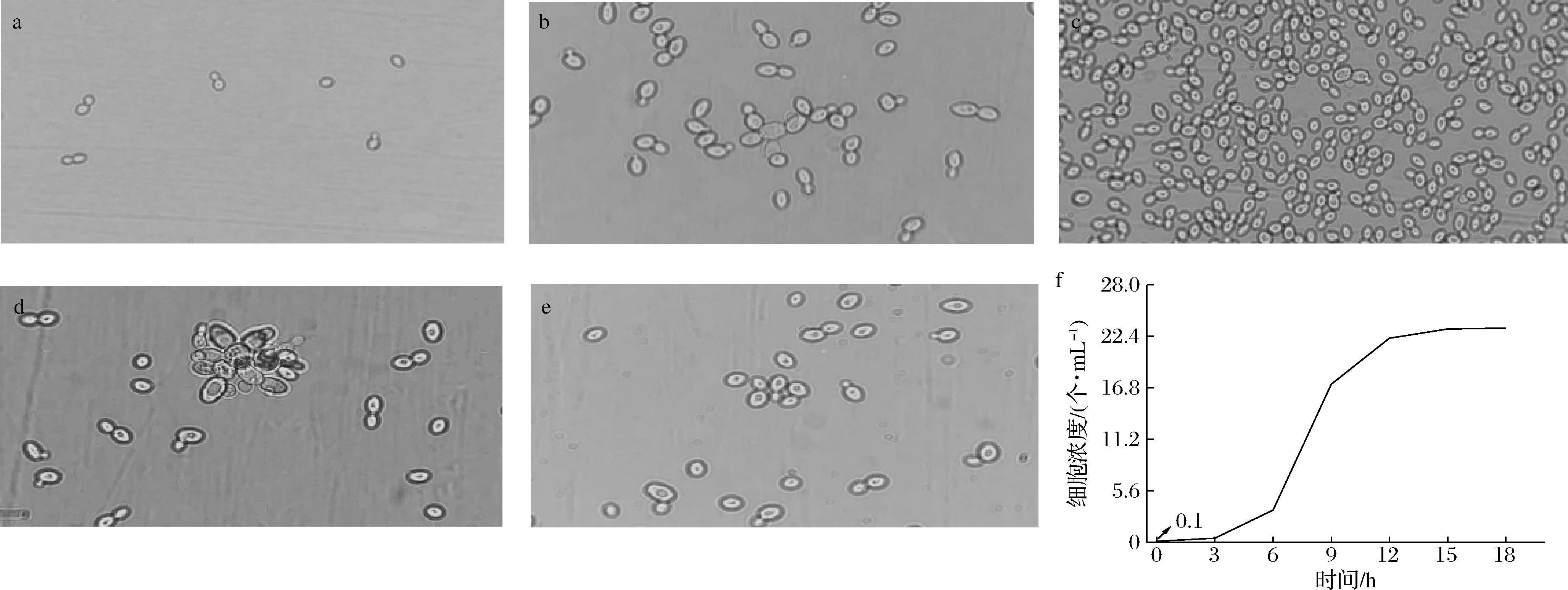
a-培养0 h形态;b-培养6 h形态;c-培养9 h形态;d-培养9 h稀释10倍形态;e-培养15 h稀释10倍形态;f-酿酒酵母细胞浓度
图1 YPD培养基中酿酒酵母好氧培养形态及细胞浓度
Fig.1 Morphology and cell density of S.cerevisiae by YPD broth in aerobic culture
2.2 酵母细胞分散单因素实验
2.2.1 不同浓度吐温-20对酿酒酵母分散影响
由图2可得,与对照组相比,吐温-20处理的酿酒酵母分散指数增大,说明加入吐温-20后酿酒酵母细胞的聚集得到了改善。当吐温-20浓度为0.4%时,酿酒酵母分散指数为0.12,此后随着吐温-20浓度增大,分散指数无显著性差异(P>0.05),即浓度为0.4%~1%的吐温-20对酿酒酵母均有较好促分散效果。实验中发现增大吐温-20浓度会产生较多泡沫,因此选择0.4% 吐温-20用于酿酒酵母促分散。
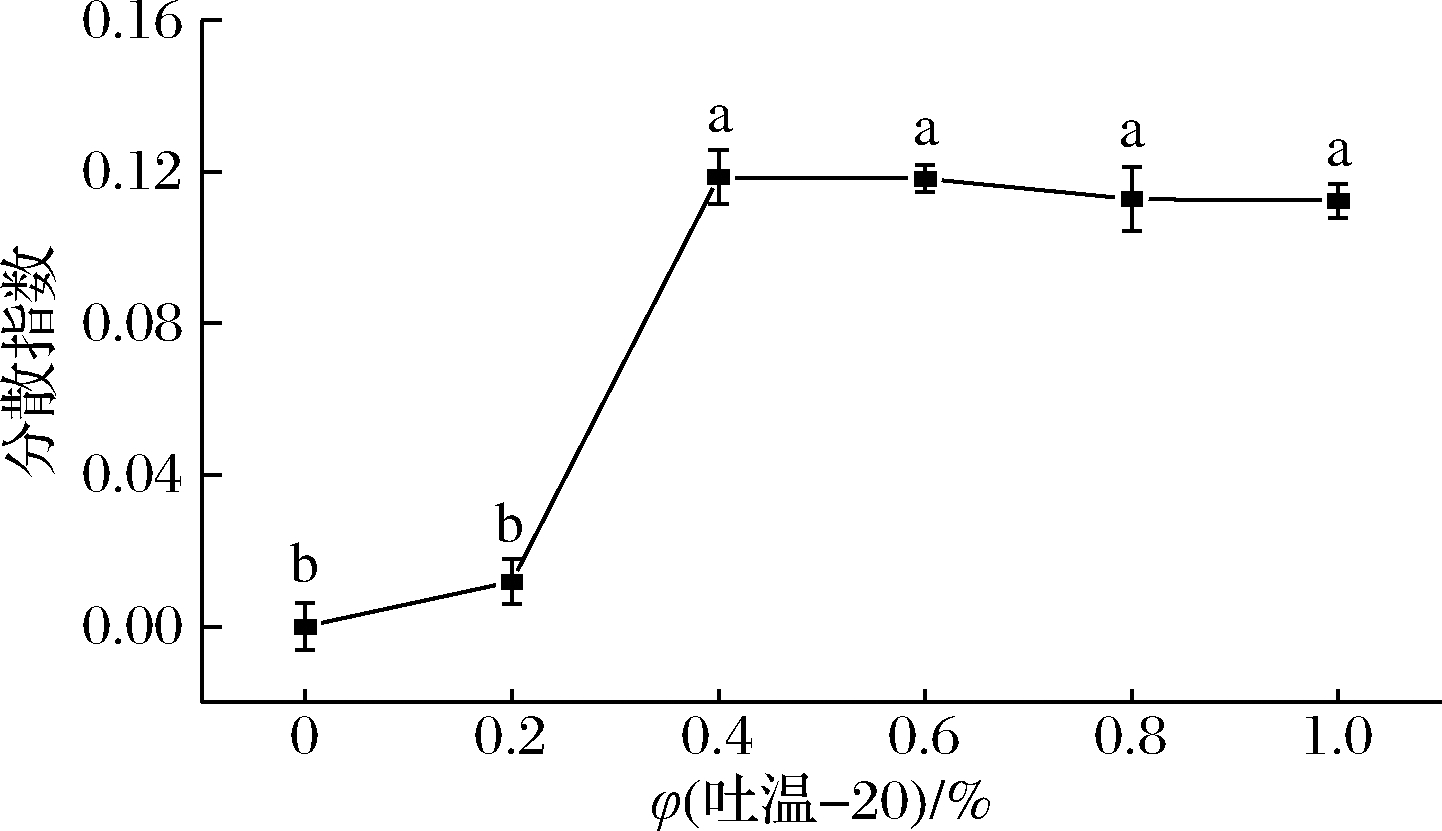
图2 不同浓度吐温-20对酿酒酵母分散指数的影响
Fig.2 Effect of different concentrations of tween 20 on the dispersion index of S.cerevisiae
注:不同字母表示有显著性差异(P<0.05)(下同)
酿酒酵母细胞壁由β-葡聚糖、甲壳素和甘露糖蛋白的纤维外层组成,这些成分的性质决定了的酿酒酵母的絮凝性[11],尤其是细胞壁中的黏附分子影响细胞表面的疏水性[12-13],酿酒酵母絮凝能力与细胞表面疏水性呈正相关[14]。吐温-20作为一种非离子型表面活性剂,分子中含有较多的亲水性基团,所以加入吐温-20可能影响酿酒酵母细胞的表面疏水性,从而促进聚集细胞的分散。废水的生物处理过程中,会使用TX-30、曲拉通X-100等表面活性剂从活性污泥中剥离微生物,表面活性剂将影响微生物分散性的胞外聚合物(extracellular polymeric substances, EPS)分散到水相中,从而将活性污泥中的微生物分散出来[15]。而酵母菌的EPS相对于细菌合成产量更高[16],并且参与凝胶状网络(生物膜)的形成,使细胞聚集在一起从而发挥保护功能[17]。所以,加入吐温-20可能也会减少酿酒酵母细胞壁表面的EPS含量,从而达到分散细胞的效果。
2.2.2 不同浓度EDTA对酿酒酵母分散影响
由图3可知,随着EDTA浓度的增加,酿酒酵母的分散指数先呈现明显的增加趋势(P<0.05),在EDTA浓度为8 mmol/L时分散指数为0.16,此后变化不显著(P>0.05)。与对照组相比,EDTA对酿酒酵母细胞的分散有显著效果(P<0.05),且当EDTA浓度达到8~12 mmol/L,分散效果达到最佳。

图3 不同浓度EDTA对酿酒酵母分散指数的影响
Fig.3 Effect of EDTA concentration on the dispersion index of S.cerevisiae
酿酒酵母中,促进絮凝的一系列蛋白统称为絮凝素,其中有一类蛋白由Flo1、Flo5、Flo9和Flo10组成,该蛋白与氨基酸序列有较高相似性,并能识别细胞表面的甘露糖残基以促进絮凝[18-19]。阳离子在酿酒酵母絮凝过程中起着核心作用,其中Ca2+是影响较显著的离子,且能促进絮凝素形成活性构象。活性絮凝素与相邻细胞壁上的甘露糖残基识别并相互作用形成聚集[20],随着细胞聚集导致絮凝现象。EDTA是结合大多数二价金属离子的螯合剂,与体系中的游离Ca2+形成络合物,减少了游离Ca2+对絮凝素的活化作用,从而减少了酵母细胞之间的聚集。
2.2.3 离子强度对酿酒酵母分散影响
由图4可得,随着体系中离子强度的增加,分散指数呈现先增加后下降的趋势,当PBS的缓冲液离子浓度为0.07 mol/L时,分散指数达到最大(I=0.09),说明离子强度对酿酒酵母分散有显著影响(P<0.05),且分散效果最佳为0.07 mol/L。
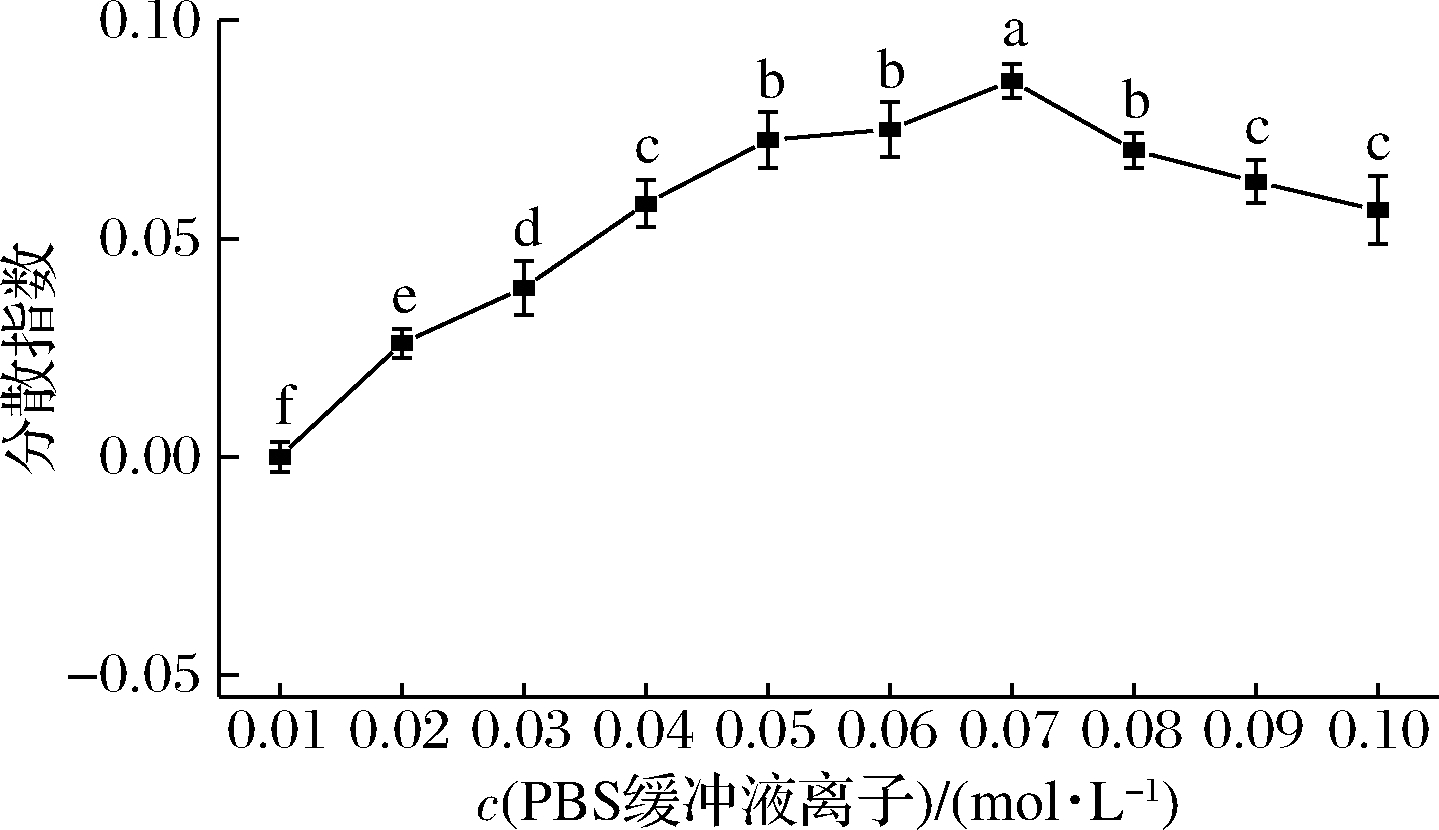
图4 离子强度对酿酒酵母分散指数的影响
Fig.4 Effect of ionic strength on the dispersion index of S.cerevisiae
有研究表明提高悬浮缓冲液的离子强度(0.01~0.20 mol/L),含2种表型蛋白(Flo1或NewFlo)的酿酒酵母菌株的絮凝性会随之降低[21]。本实验中酿酒酵母在离子强度为0.1 mol/L时相比低离子强度表现出聚集性降低的现象,可能是离子强度影响了絮凝素的性质。STRATFORD[22]认为高浓度的Na+和K+会引起酿酒酵母凝集素的扭曲,并拮抗Ca2+诱导的絮凝。高浓度的Na+和Cl-也会降低蛋白质分子疏水基团的溶解度,增强蛋白质周围的水团簇,使疏水分子更加致密,从而增强絮凝作用[23]。
2.2.4 pH对酿酒酵母分散影响
由图5可知,随着缓冲液pH的升高,分散指数呈现先减少后基本不变的趋势,说明低的pH环境(pH<7.0)下有助酿酒酵母细胞的分散。当pH>7.0,对酵母细胞的分散性影响不显著(P>0.05)。
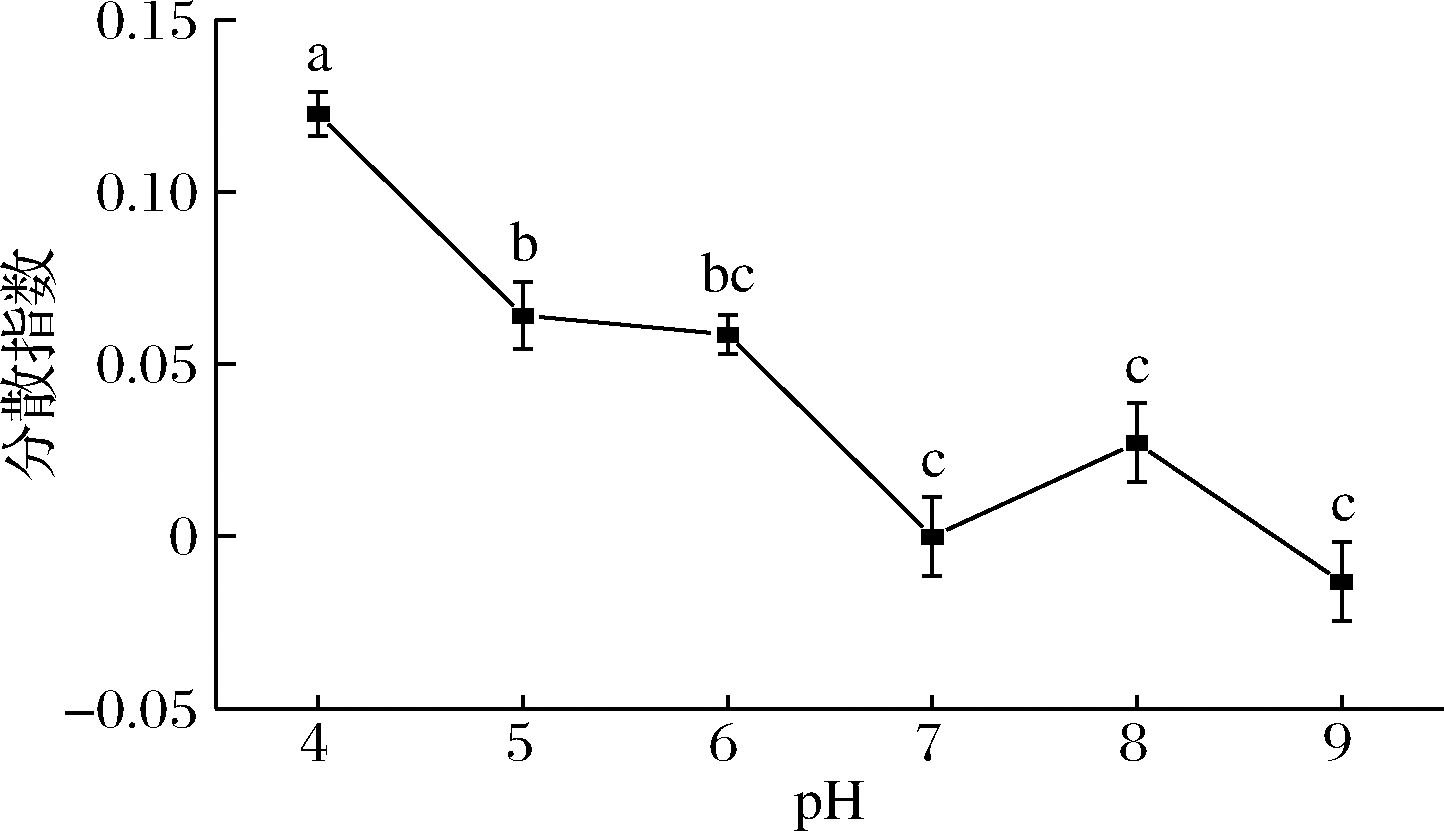
图5 pH对酿酒酵母分散性的影响
Fig.5 Effect of different pH on the dispersion index of S.cerevisiae
因为细胞壁中蛋白质和甘露聚糖的羧基和磷酸二酯基的电离,所以酿酒酵母细胞壁具有净负电荷[20]。体系pH越高,细胞表面带的负电荷则越强,静电排斥力也就越强,细胞在高pH值的环境中应该更分散,这与本实验结果不同。JIN等[21]同样发现了这种矛盾的现象,而他认为高pH环境除增加了净电荷外,也可能改变酿酒酵母细胞表面蛋白基团(如酵母凝集素分子)的电离,导致局部酵母凝集素构象的变化,使酿酒酵母细胞发生絮凝。CILLIERS等[24]也发现降低啤酒酵母悬浮液的pH值可以降低其絮凝度。
2.2.5 超声波对酿酒酵母分散影响
如图6所示,随着超声波时间的增加(0~60 s),分散指数在短暂增加后开始减少,但除60 s外,其他处理组(5~50 s)分散指数均大于对照组,说明超声波5~50 s对酿酒酵母的分散有显著促进(P<0.05)作用,而超声波处理60 s后酿酒酵母的分散指数小于对照组,说明超声波处理60 s后的酿酒酵母细胞分散性反而不如对照组,原因有待研究。
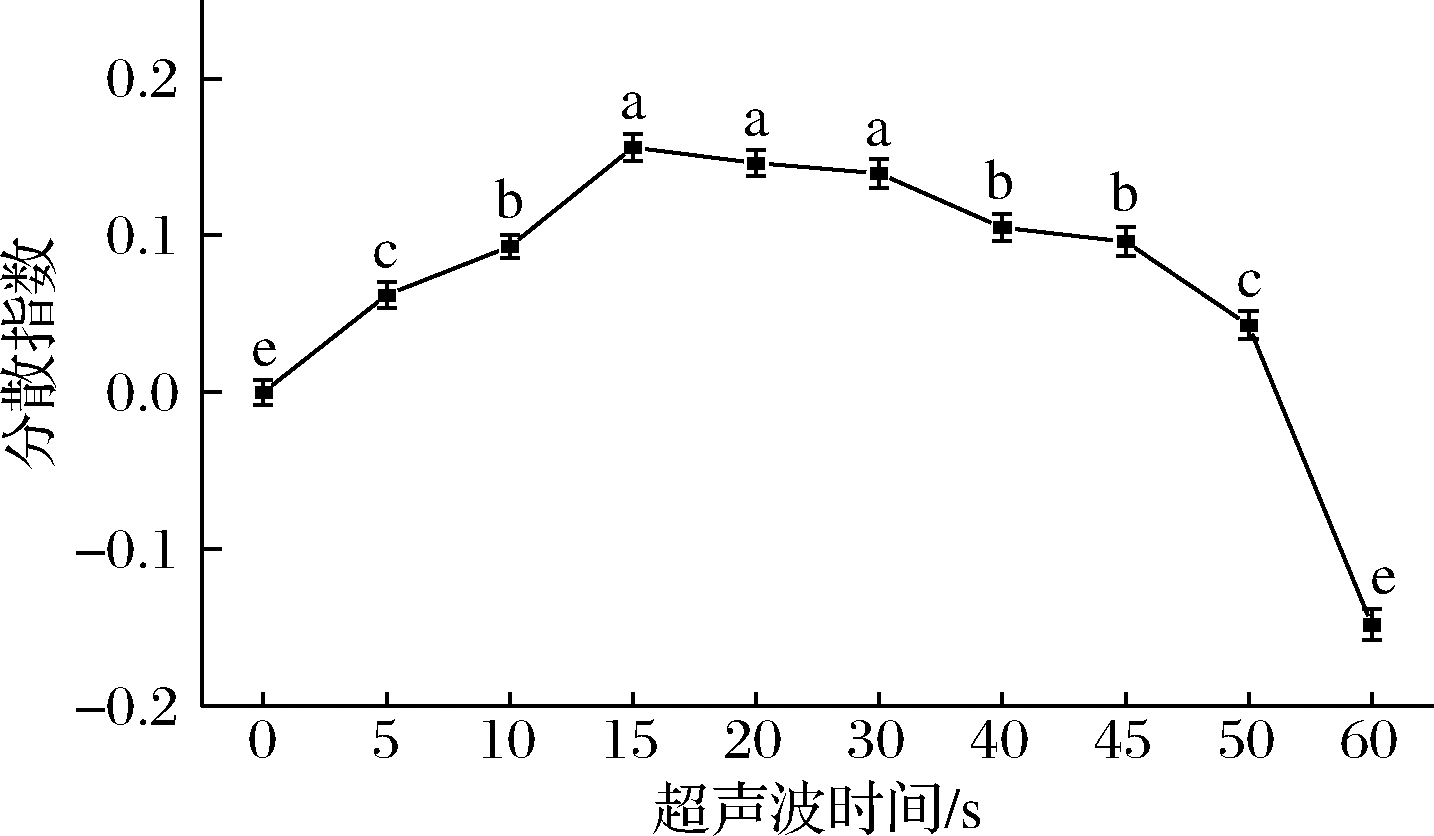
图6 超声波时间对酿酒酵母分散指数的影响
Fig.6 Effect of different ultrasound time on dispersion index of S.cerevisiae
2.3 不同分散处理对酿酒酵母活性影响
如图7所示,不同浓度吐温-20、EDTA以及不同离子强度处理组,处理前后平板法得到的菌落总数变化不明显,说明吐温-20(0.2%~1.0%)、EDTA(2~12 mmol/L)、离子强度(0.01~0.1 mol/L)不影响酵母细胞活性。从图7-c可知,经pH>7.0的缓冲液处理后的酿酒酵母菌液,随着pH的增大,菌落总数呈现逐渐下降的趋势,这是因为碱性处理后的酿酒酵母细胞活性受到影响,导致菌落总数减少。图7-e中超声波处理后的菌落总数相比对照组增加的原因,可能是超声波改善了酿酒酵母细胞的分散性,减少了细胞的黏连,使得平板计数结果增大,同时从图7-e中也可以看出超声波处理后对细胞活性无影响。
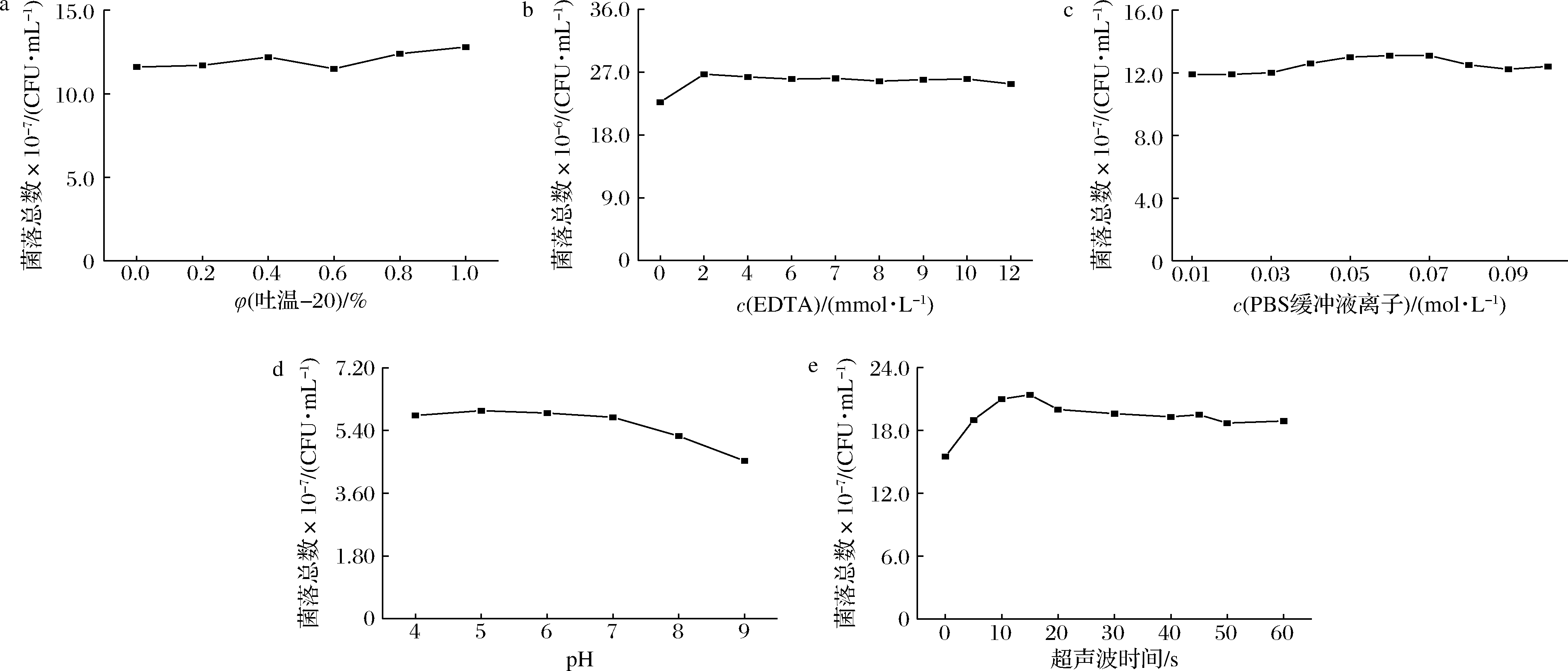
a-吐温-20;b-EDTA;c-离子强度;d-pH;e-超声波时间
图7 分散处理对酿酒酵母细胞活性的影响
Fig.7 Effect of dispersion treatment on cell viability of S.cerevisiae
2.4 酿酒酵母分散条件正交实验优化结果
在单因素试验的基础上,确定影响酿酒酵母细胞分散的主要因素及水平,以分散指数为考查指标,设计L9(33)正交试验。试验因素与水平设计见表1,结果见表2,采用SPSS 26.0对正交试验结果进行分析,方差分析结果如表3所示。
表1 正交试验因素水平表
Table 1 Factors and levels of orthogonal test

水平因素A(吐温-20)/%B(超声波时间)/sC(EDTA)/(mmol·L-1)10.210720.415830.6209
表2 正交试验结果
Table 2 Results of orthogonal test

因素ABCI11110.0921220.1931330.1442130.1052210.1162320.1573120.1483230.1693310.10K10.430.330.30K20.360.460.48K30.400.390.40R0.020.040.06
表3 正交试验方差分析结果
Table 3 Results of variance analysis of orthogonal test
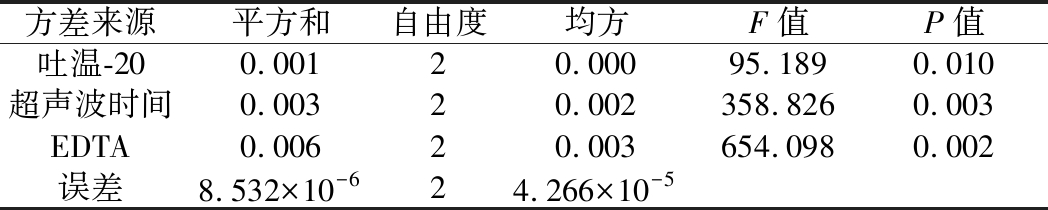
方差来源平方和自由度均方F值P值吐温-200.00120.00095.1890.010超声波时间0.00320.002358.8260.003EDTA0.00620.003654.0980.002误差8.532×10-624.266×10-5
由表2及表3可知,各因素对酿酒酵母细胞分散影响的主次为C>B>A,且因素C(EDTA)和因素B(超声波时间)对实验结果的影响极显著(P<0.01),因素A(吐温-20)对实验结果的影响显著(P<0.05)。正交优化分散条件为A1B2C2(0.2% 吐温-20、超声波时间15 s、8 mmol/L EDTA)。经3次重复试验验证,该条件下的酿酒酵母细胞的分散指数为0.24,证明优化的分散条件是适宜的。
2.5 酿酒酵母分散处理前后溶液中细胞分布类型分析
微生物群体细胞在空间上的分布分为随机分布、规则分布、集群分布3种基本分布类型。不同分布类型中微生物细胞的分散度不同,而这个微生物群体的分散度决定了方差(σ2)和均值(μ)的关系,由此可以用模拟方差和均值之间关系的多种数学模型来描述微生物群体的分布类型,泊松分布(σ2=μ)是随机分布的合适数学模型,二项分布(σ2<μ)为规则分布的近似模型,负二项分布(σ2>μ)为描述集群分布的数学模型之一[10]。
计算不同分散处理的酿酒酵母菌液,数据方差和均值的比值(σ2/μ),结果见图8。未经吐温-20处理的酿酒酵母细胞分布类型为集群分布;不同浓度吐温-20处理后,分布类型变为规则分布,表明吐温-20处理后的酿酒酵母分散性更好。不同浓度EDTA处理后酿酒酵母分布类型也由集群分布变成了规则分布;随着缓冲液离子强度的增加,酿酒酵母分布类型也有类似变化。酿酒酵母在不同pH条件下的分布类型也不相同,当pH<7.0时为规则分布,而pH>7.0时为集群分布。超声波处理组中,当经过超声波处理5~50 s后,酿酒酵母由未超声波处理时的集群分布变为处理后规则分布,而当超声波时间为60 s,分布类型则为集群分布。细胞分布类型的变化表明适宜的分散处理可以将集群分布的酿酒酵母在一定程度变为规则分布,即可以改变酿酒酵母的分散性。
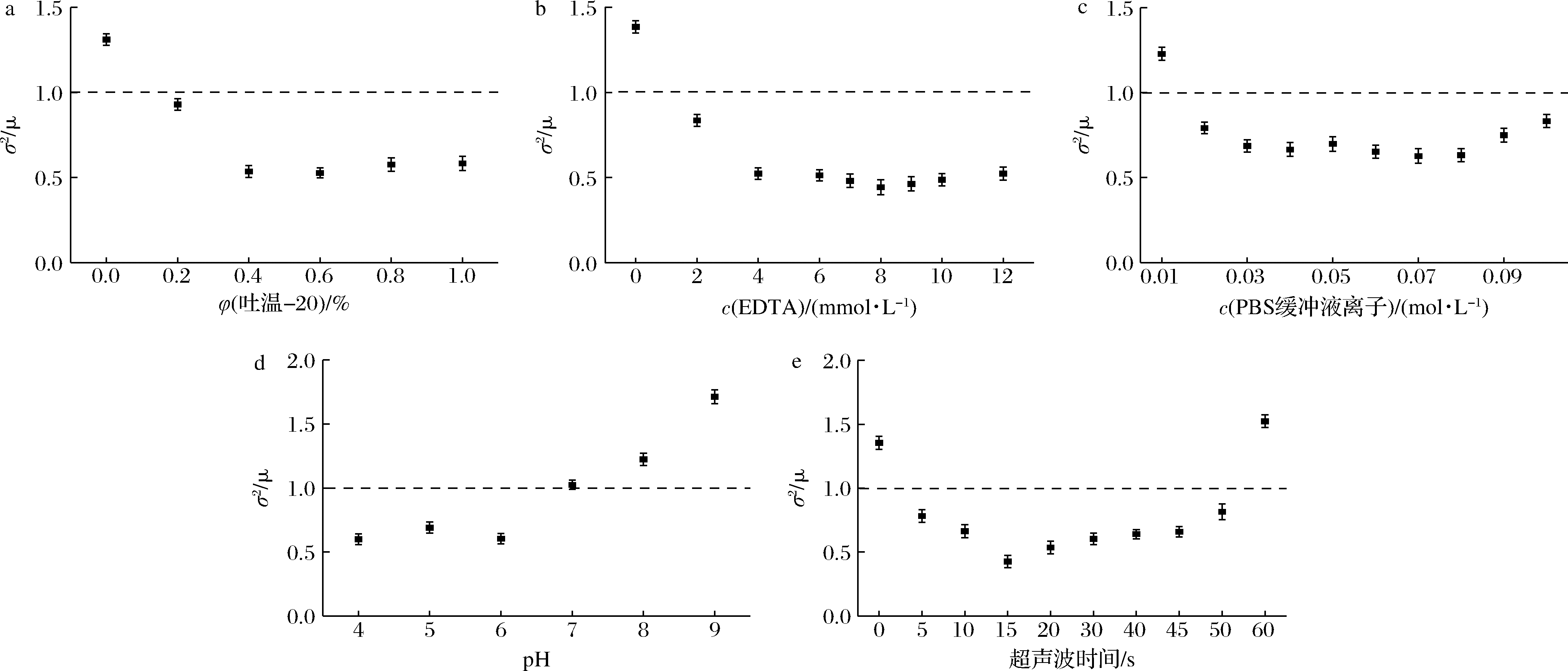
a-吐温-20;b-EDTA;c-离子强度;d-pH;e-超声波时间
图8 不同分散处理对酿酒酵母细胞分布类型的影响
Fig.8 Effect of different dispersion treatment on distribution types of S.cerevisiae
3 结论
本文以酿酒酵母为研究对象,针对其细胞聚集现象,提出了有效的分散方法,在优化条件下可使其分散指数提高0.24,即比对照组细胞浓度提高了24%,也证明了对照组所测定的细胞浓度偏低。从溶液中细胞分布类型的变化上,表明所采用的分散方法对酿酒细胞分散有作用。同时,本研究所实验的分散方法处理的酿酒酵母,通过平板计数分析对酿酒酵母细胞活性无影响,也表明对细胞分散的同时未影响其生长。本研究的分散处理可用于微生物计数方法的前处理中,更真实、准确反映样品中微生物细胞的浓度。本研究仅针对酿酒酵母进行水溶液中分散研究,但对于其他微生物的分散,以及食品、药品等基质中的分散效果,仍需进一步的研究。
在利用流式细胞仪分析微生物浓度中,为避免细胞聚集影响,有学者在样品前处理的过程中也涉及到了微生物分散,如HICKEY等[25]在利用流式细胞仪分析奶酪中的瑞士乳杆菌活力时,为避免细胞聚集,把回收的菌重悬在含0.02%吐温-20和1 mmol/L EDTA的缓冲液中,并进行适当稀释后再进行分析;BROWN等[26]在利用流式细胞仪计数活性污泥中细菌数量时,先用5%吐温-80和10 mmol/L焦磷酸钠破坏并分散活性污泥中的细菌。在计数污泥中病毒数量时[27],除用5%吐温-80、10 mmol/L焦磷酸钠处理活性污泥之外,还使用含1 mmol/L EDTA的缓冲液稀释样本,实验结果均表明,经处理后的样本经流式细胞仪分析后,呈现更大的计数结果。由此可见,对微生物细胞进行有效分散有着重要意义,也将为研究其他微生物分散提供参考价值。
本文研究了吐温-20、EDTA、离子强度、pH、超声波时间对酿酒酵母细胞分散的影响,结果表明,EDTA和超声波时间对酿酒酵母细胞的分散有极显著影响(P<0.01),吐温-20对酿酒酵母细胞分散性影响显著(P<0.05),并经正交优化得到适宜酿酒酵母的分散条件为体积分数0.2%的吐温-20、超声波处理15 s、8 mmol/L EDTA,可使酿酒酵母细胞分散指数达到0.24;对于分布类型为集群分布的酿酒酵母细胞,经有效分散处理后,其分布类型由集群分布转为规则分布,且适宜的分散处理不影响酿酒酵母细胞活性。
[1] CALLEJA G B.Microbial Aggregation[M].Boca Raton:CRC Press, 1984.
[2] MEHTA D V, CURTIS S J, RUDOLPH A B, et al.A mini review:The history of yeast flocculation with an emphasis on measurement techniques[J].Journal of the American Society of Brewing Chemists, 2021, 79(4):333-339.
[3] 刘晓猛. 微生物聚集体的相互作用及形成机制[D].合肥:中国科学技术大学, 2008.
LIU X M.Interactions and formation mechanisms of microbial aggregates[D].Hefei:University of Science and Technology of China, 2008.
[4] 马放,杨基先, 魏利.环境微生物图谱[M].北京:中国环境科学出版社, 2010.
MA F,YANG J X WEI L.Environmental Microbiology Atlas[M].Beijing:China Environmental Science Press, 2010.
[5] WESTMAN J O, MAPELLI V, TAHERZADEH M J, et al.Flocculation causes inhibitor tolerance in Saccharomyces cerevisiae for second-generation bioethanol production[J].Applied and Environmental Microbiology, 2014, 80(22):6 908-6 918.
[6] MONIER J M, LINDOW S E.Differential survival of solitary and aggregated bacterial cells promotes aggregate formation on leaf surfaces[J].Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(26):15 977-15 982.
[7] ESPINAL P, MART S, VILA J.Effect of biofilm formation on the survival of Acinetobacter baumannii on dry surfaces[J].Journal of Hospital Infection, 2012, 80(1):56-60.
S, VILA J.Effect of biofilm formation on the survival of Acinetobacter baumannii on dry surfaces[J].Journal of Hospital Infection, 2012, 80(1):56-60.
[8] VITZILAIOU E, KURIA A M, SIEGUMFELDT H, et al.The impact of bacterial cell aggregation on UV inactivation kinetics[J].Water Research, 2021, 204:117593.
[9] GUNASEKERA T S, ATTFIELD P V, VEAL D A.A flow cytometry method for rapid detection and enumeration of total bacteria in milk[J].Applied and Environmental Microbiology, 2000, 66(3):1 228-1 232.
[10] JARVIS B.Chapter 4-The Distribution of Microorganisms in Foods in Relation to Sampling[M].3rd ed.Cambridge:Academic Press, 2016.
[11] MAEKAWA H, TAKEGAWA K.Yeast flocculin:Methods for quantitative analysis of flocculation in yeast cells[J].Methods in Molecular Biology (Clifton, N.J.), 2020, 2132:437-444.
[12] GOVENDER P, DOMINGO J L, BESTER M C, et al.Controlled expression of the dominant flocculation genes FLO1, FLO5, and FLO11 in Saccharomyces cerevisiae[J].Applied and Environmental Microbiology, 2008, 74(19):6 041-6 052.
[13] VAN MULDERS S E, CHRISTIANEN E, SAERENS S M G, et al.Phenotypic diversity of Flo protein family-mediated adhesion in Saccharomyces cerevisiae[J].FEMS Yeast Research, 2009, 9(2):178-190.
[14] SMIT G, STRAVER M H, LUGTENBERG B J, et al.Flocculence of Saccharomyces cerevisiae cells is induced by nutrient limitation, with cell surface hydrophobicity as a major determinant[J].Applied and Environmental Microbiology, 1992, 58(11):3 709-3 714.
[15] 陈超. 微生物分散技术研究[D].南京:南京理工大学, 2012.
CHEN C.Study on dispersion of microorganisms[D].Nanjing:Nanjing University of Science and Technology, 2012.
[16] RAHBAR SAADAT Y, YARI KHOSROUSHAHI A, POURGHASSEM GARGARI B.Yeast exopolysaccharides and their physiological functions[J].Folia Microbiologica, 2021, 66(2):171-182.
[17] GIENTKA I, B A
A EJAK S, STASIAK-R
EJAK S, STASIAK-R
 A
A SKA L, et al.Exopolysaccharides from yeast:Insight into optimal conditions for biosynthesis, chemical composition and functional properties-review[J].Acta Scientiarum Polonorum.Technologia Alimentaria, 2015, 14(4):283-292.
SKA L, et al.Exopolysaccharides from yeast:Insight into optimal conditions for biosynthesis, chemical composition and functional properties-review[J].Acta Scientiarum Polonorum.Technologia Alimentaria, 2015, 14(4):283-292.
[18] BONY M, THINES-SEMPOUX D, BARRE P, et al.Localization and cell surface anchoring of the Saccharomyces cerevisiae flocculation protein Flo1p[J].Journal of Bacteriology, 1997, 179(15):4 929-4 936.
[19] BONY M, BARRE P, BLONDIN B.Distribution of the flocculation protein, Flop, at the cell surface during yeast growth:The availability of Flop determines the flocculation level[J].Yeast, 1998, 14(1):25-35.
[20] SOARES E V.Flocculation in Saccharomyces cerevisiae:A review[J].Journal of Applied Microbiology, 2011, 110(1):1-18.
[21] JIN Y L, SPEERS R A.Effect of environmental conditions on the flocculation of Saccharomyces cerevisiae[J].Journal of the American Society of Brewing Chemists, 2000, 58(3):108-116.
[22] STRATFORD M.Yeast flocculation:A new perspective[J].Advances in Microbial Physiology, 1992, 33:1-71.
[23] JIN Y L, RITCEY L L, SPEERS R A, et al.Effect of cell surface hydrophobicity, charge, and zymolectin density on the flocculation of Saccharomyces cerevisiae[J].Journal of the American Society of Brewing Chemists, 2001, 59(1):1-9.
[24] CILLIERS J J, HARRISON S T L.Yeast flocculation aids the performance of yeast dewatering using mini-hydrocyclones[J].Separation and Purification Technology, 2019, 209:159-163.
[25] HICKEY C D, FALLICO V, WILKINSON M G, et al.Redefining the effect of salt on thermophilic starter cell viability, culturability and metabolic activity in cheese[J].Food Microbiology, 2018, 69:219-231.
[26] BROWN M R, HANDS C L, COELLO-GARCIA T, et al.A flow cytometry method for bacterial quantification and biomass estimates in activated sludge[J].Journal of Microbiological Methods, 2019, 160:73-83.
[27] BROWN M R, CAMÉZULI S, DAVENPORT R J, et al.Flow cytometric quantification of viruses in activated sludge[J].Water Research, 2015, 68:414-422.