贻贝(Mytilus sp.)是一种营足丝附着生活的双壳类软体动物,属软体动物门(Mollusca),瓣鳃纲(Lanellibranchia),异柱目(Anisomyaria),贻贝科(Mytidea)[1]。作为贻贝养殖大国之一,我国2019年的贻贝养殖产量达87.07万t,占全球产量的42.09%,其中紫贻贝(Mytilus edulis)、厚壳贻贝(Mytilus coruscus)、加州贻贝(Mytilus californianus)等是我国常见的贻贝养殖品种[2]。贻贝本身具有很高的食用价值,其富含蛋白质、多聚不饱和脂肪酸、矿物质、维生素等营养物质,以及人体所需的8种必需氨基酸,素有“海中鸡蛋”的美称[3]。然而,目前除少量作为冻品、罐头和干品外,我国大部分贻贝产品仍作为活鲜品销售,产品单一,资源利用不足[4]。
近年来,对于贻贝来源的生物活性肽的研究越来越受到关注。目前已从贻贝蛋白制备获得了抗氧化肽[5]、血管紧张素转换酶(angiotensin converting enzyme, ACE)抑制肽[4]、抗血栓肽[6]、抗骨质疏松肽[7]、抗菌肽[8]、免疫调节肽[3]等。然而,从贻贝制备生物活性肽的过程通常依赖于天然蛋白的酶促水解结合多肽混合物的分离和鉴定,整个过程费时、费力、技术难度较大[9]。相比之下,计算机辅助酶解和多肽活性的生物信息学筛选能克服传统方法中的缺陷,并已逐步应用于指导生物活性多肽的制备和鉴定[10-11]。
基于此,本研究以我国常见的紫贻贝蛋白作为研究对象,使用计算机模拟蛋白消化过程并对消化物中的多肽活性进行了生物信息学分析,以期为紫贻贝蛋白生物活性的阐释及活性肽的研发提供重要理论依据。
1 材料与方法
1.1 紫贻贝蛋白的选择
从美国国家生物技术信息中心(NCBI,https://www.ncbi.nlm.nih.gov/)数据库搜索紫贻贝相关蛋白,选择其中4种主要蛋白质:前胶原蛋白P(Precollagen P,GenBank:AAB80 719.1)、前胶原蛋白D(Precollagen D,GenBank:AAB96 638.1)、肌球蛋白(Myosin heavy chain, partial,GenBank:AAA82 260.1)和肌动蛋白(Actin, partial,GenBank:AAD48 064.1)作为目标蛋白进行模拟消化。
1.2 模拟消化
通过BIOPEP-UWM数据库,选择胃蛋白酶和胰蛋白酶的组合对以上4种目标蛋白进行计算机模拟消化,对消化后的多肽序列进行后续分析,使用的胰蛋白酶(EC 3.4.21.4)和胃蛋白酶(pH 1.3,EC 3.4.23.1)的性质如表1所示[12]。
表1 所使用的蛋白酶的性质
Table 1 Properties of proteases
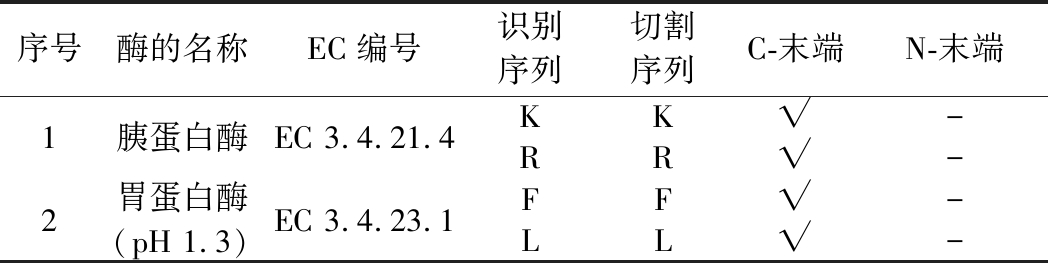
序号酶的名称EC编号识别序列切割序列C-末端N-末端1胰蛋白酶EC 3.4.21.4KK√-RR√-2胃蛋白酶(pH 1.3)EC 3.4.23.1FF√-LL√-
注:-表示数据未检出
1.3 消化产物中生物活性肽的筛选
使用PeptideRanker程序(https://bioware.ucd.ie/compass/biowareweb/Serverpages/peptideranker.php)对以上收集的多肽中的寡肽(二肽至五肽)进行生物活性预测,并以预测得分的形式表示其生物活性的相对大小[13]。以0.5作为活性预测的阈值,当多肽预测得分大于该值时,认为其具有潜在生物活性。
1.4 消化产物的理化性质评价
通过Peptide Property Calculator(http://www.innovagen.com/proteomics-tools),计算模拟消化后所得寡肽的分子质量、等电点、pH 7时的净电荷和水溶性。以水溶性强弱为依据,选择用于后续研究的肽。
1.5 消化产物的毒性分析
使用ToxinPred程序(http://crdd.osdd.net/raghava//toxinpred/)预测所得寡肽的潜在毒性[14]。
1.6 消化产物的ADMET评价
ADMET(absorption, distribution, metabolism, excretion, and toxicity;吸收、分配、代谢、排泄和毒性)评价是药物设计和筛选中的重要方法,它对识别具有强药代动力学谱的新型化合物至关重要[15]。本研究使用admetSAR(http://lmmd.ecust.edu.cn/admetsar2/)程序对所选寡肽进行在线评价,其中选择人体肠道吸收(human intestinal absorption, HIA)来确定吸收特性,选择血脑屏障(blood brain barrier, BBB)穿透率和细胞色素P450(CYP 450)2D6相互作用来分析其分布特征[16]。
1.7 消化产物的活性预测
使用BIOPEP-UWM(http://www.uwm.edu.pl/biochemia/index.php/pl/biopep)程序,预测消化后所产生寡肽的潜在生物活性。
1.8 ACE抑制肽的筛选
使用SwissDock(http://www.swissdock.ch/),将所选寡肽与ACE进行在线分子对接以比较这些寡肽的ACE抑制活性大小[17]。ACE蛋白的晶体结构(人血管紧张素转换酶与赖诺普利的复合物,1O86.pdb)得自RCSB蛋白数据库(PDB)。使用Chemdraw软件(19.0版)绘制多肽结构,并作为sybyl MOL2格式保存。通过比较寡肽与ACE蛋白对接的结合能大小,选择潜在的ACE抑制剂。
1.9 二肽基肽酶-IV(dipeptidyl peptidase-IV, DPP-IV)抑制肽的筛选
使用iDPPIV-SCM程序(http://camt.pythonanywhere.com/iDPPIV-SCM)预测DPP-IV抑制活性并对以上所选寡肽的相对DPP-IV抑制活性打分,其中当寡肽得分大于294时,认为该寡肽是潜在的DPP-IV抑制剂[18]。
1.10 分子对接
使用Discovery Studio(DS)2016软件进行分子对接研究。从RCSB蛋白数据库(PDB)获取ACE(PDB:1O86)和DPP-IV(PDB:5 J3 J)的蛋白晶体结构,并且在对接前除去晶体结构中的水分子并进行结构优化。使用Chemdraw软件(19.0版)绘制多肽结构,以结合能最低的对接构象显示对接结果。
2 结果与分析
2.1 In silico水解
本研究通过BIOPEP-UWM程序,使用胃蛋白酶(pH 1.3,EC 3.4.23.1)和胰蛋白酶(EC 3.4.21.4)的组合对4种紫贻贝蛋白进行模拟消化。经消化后,4种蛋白均能够被较好地水解,其中肌球蛋白和肌动蛋白水解度相对较高,分别达到30.85%和22.11%,而前胶原蛋白P、前胶原蛋白D水解度相对较低,均在10%左右。通过水解,前胶原蛋白P、前胶原蛋白D、肌球蛋白和肌动蛋白分别释放出87、97、64、43条片段(包括氨基酸),其中二肽至五肽总计分别为30、45、27、25条,占所释放的总多肽数的34.48%、46.39%、42.19%、58.14%(图1),表明经消化后,紫贻贝蛋白释放出大量短肽,因此对于提供生物活性肽具有良好潜力。
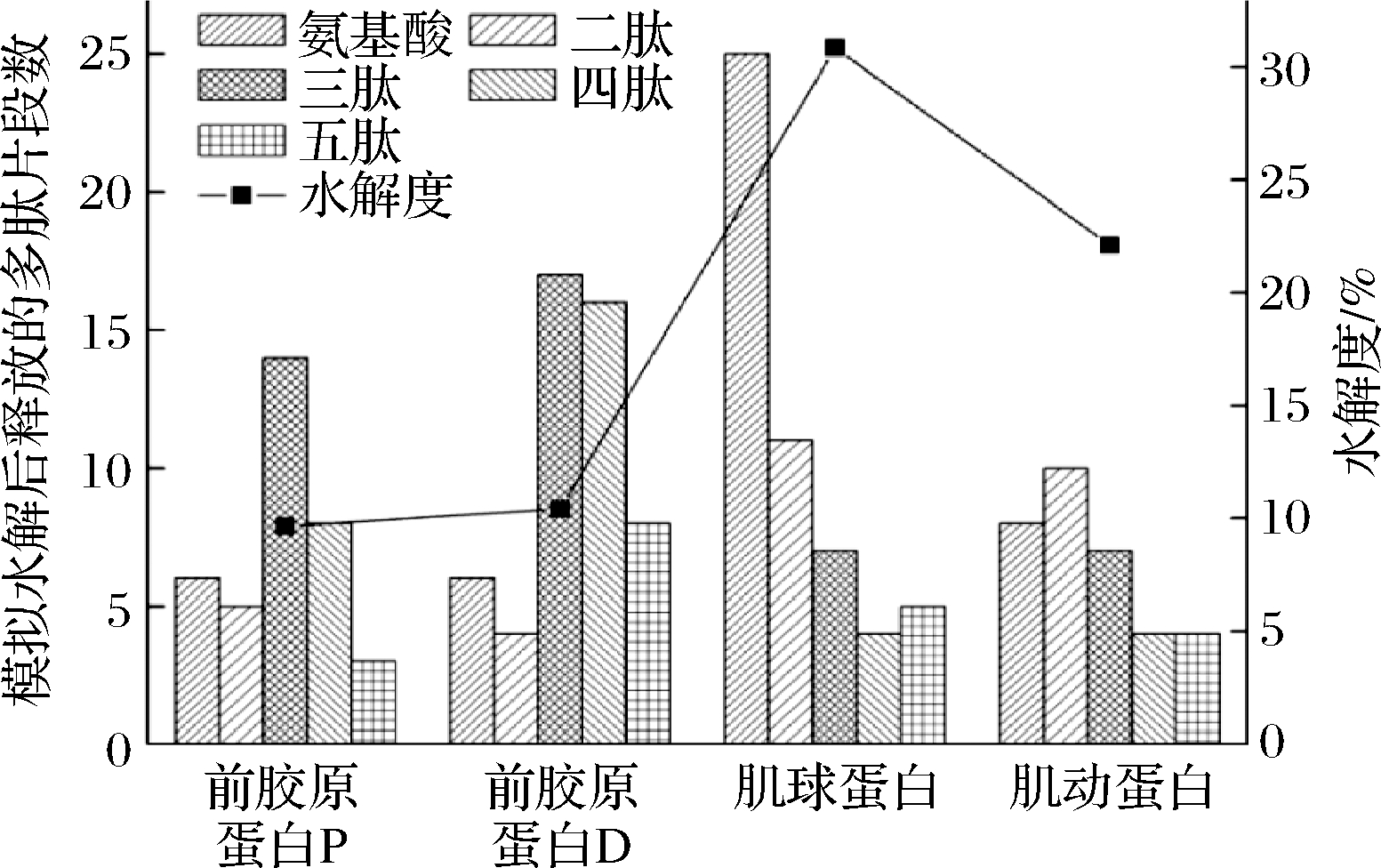
图1 紫贻贝蛋白模拟水解所释放的肽段数和水解度
Fig.1 Number of fragments released from M.edulis proteins after in silico digestion and the degree of hydrolysis
2.2 消化产物中寡肽的生物活性评价及理化性质分析
通过PeptideRanker程序对消化产生的小分子寡肽打分。除去重复序列后,得分大于0.5的二肽有5条,三肽、四肽均有7条,五肽有6条(表2),表明这些寡肽具有良好生物活性。随后,对这25条寡肽进行理化性质分析,发现这些多肽的分子质量较小,普遍分布在200~500,这使它们在胃肠道内能够被快速吸收,并进一步有利于其生物活性的发挥。这些多肽的等电点在1.0~11.0广泛分布,这与它们所含的碱性氨基酸和酸性氨基酸的数量比例有关。
表2 紫贻贝蛋白水解肽的活性评价及理化性质分析
Table 2 Activity evaluation and physical and chemical property analysis of the selected oligopeptides

多肽序列来源蛋白PeptideRanker得分分子质量等电点pH 7时的净电荷预测水溶性二肽PFa0.99262.3 4.150差DFd0.94280.280.76-1优GLa0.81188.223.630差GRb0.77231.2510.841优YRc0.53337.379.571优三肽GGFa, b0.99279.293.700差PGFa0.99319.364.150差GPRa, b0.87328.3710.840优GGLa, b0.84245.283.630差PGYa, b0.83335.353.850差HMRc0.64442.5410.591.1优GILd0.61301.383.630差四肽GGGFa0.98336.343.700差GPGFa0.98376.413.700差GYSFd0.88472.493.640差PGPRb0.87425.4811.291优GGGLb0.83302.333.630差PGPKa0.66397.4710.571优PGNRa0.60442.4711.291优五肽GSPGFb0.95463.483.700差AGAGFb0.83421.453.770差GGFVFa0.70515.567.810.1差GAGGLb0.69373.413.630差YGNGLb0.61522.553.340差QYGGRb0.55579.619.551优
注:a-前胶原蛋白P;b-前胶原蛋白D;c-肌球蛋白;d-肌动蛋白
水溶性是药物在机体内转运分布的基础,并且是多肽在生物、食品等领域中应用时需要考虑的重要指标。因此,本研究进一步预测了所得25条寡肽的水溶性情况。在所分析的寡肽中,共获得9条水溶性寡肽,即YR、DF、GR、GPR、HMR、PGNR、PGPK、PGPR、QYGGR。从这些寡肽的氨基酸序列来看,大部分水溶性寡肽的C末端包含碱性氨基酸赖氨酸(K)和精氨酸(R),表明C末端碱性氨基酸的存在对多肽水溶性具有重要影响。基于此,对上述9条具有良好生物活性的水溶性寡肽进行下一步分析。
2.3 消化产物的毒性和ADMET评价
本研究首先通过toxinPred程序对上述9条水溶性寡肽进行毒性评价,结果表明其SVM得分大都在-0.8左右,表明其无毒,因而具有良好的应用安全性。
在此基础上,本研究进一步使用admetSAR2程序预测了这些多肽的ADMET性质(表3)。HIA预测结果表明9条水溶性寡肽的HIA值分布在0.4~0.8,表明这些寡肽均具有较好的肠道吸收,其中四肽PGPK(HIA值:0.816 4)、PGPR(HIA值:0.787 0)和二肽GR(HIA值:0.728 4)的肠道吸收概率相对较大。除PGPK外,这些寡肽的BBB渗透性值均大于0.9,表现出良好的BBB渗透性,这可能与所选寡肽的分子质量较小有关。CYP450酶显著影响包括活性肽和其他膳食产品在内的多种内源性和外源性化合物的代谢。因此,本研究进一步比较了这些寡肽与CYP450 2D6之间的相互作用。预测结果表明这些寡肽均不是CYP450 2D6的底物或抑制剂,与该酶发生相互作用的概率较低。此外,其他CYP450酶(CYP450 2C9、CYP450 3A4和CYP450 1A2)也表现出类似的预测结果。
表3 所筛选的寡肽的ADMET分析和活性预测
Table 3 ADMET analysis and activity prediction of the selected oligopeptides
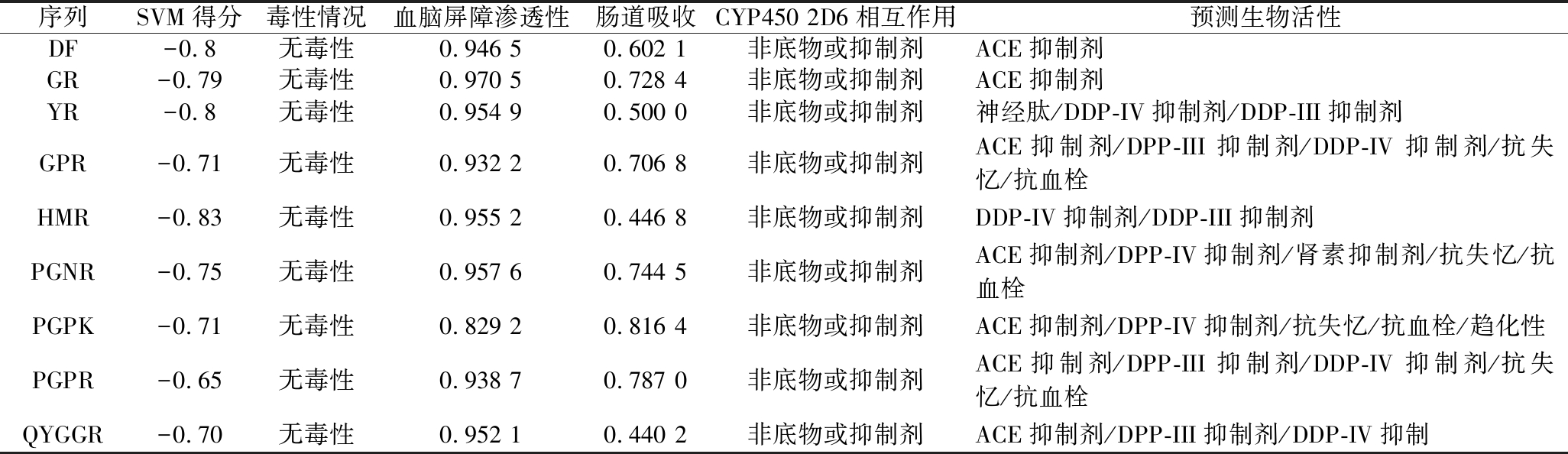
序列SVM得分毒性情况血脑屏障渗透性肠道吸收CYP450 2D6相互作用预测生物活性DF-0.8无毒性0.946 50.602 1非底物或抑制剂ACE抑制剂GR-0.79无毒性0.970 50.728 4非底物或抑制剂ACE抑制剂YR-0.8无毒性0.954 90.500 0非底物或抑制剂神经肽/DDP-IV抑制剂/DDP-III抑制剂GPR-0.71无毒性0.932 20.706 8非底物或抑制剂ACE抑制剂/DPP-III抑制剂/DDP-IV抑制剂/抗失忆/抗血栓HMR-0.83无毒性0.955 20.446 8非底物或抑制剂DDP-IV抑制剂/DDP-III抑制剂PGNR-0.75无毒性0.957 60.744 5非底物或抑制剂ACE抑制剂/DPP-IV抑制剂/肾素抑制剂/抗失忆/抗血栓PGPK-0.71无毒性0.829 20.816 4非底物或抑制剂ACE抑制剂/DPP-IV抑制剂/抗失忆/抗血栓/趋化性PGPR-0.65无毒性0.938 70.787 0非底物或抑制剂ACE抑制剂/DPP-III抑制剂/DDP-IV抑制剂/抗失忆/抗血栓QYGGR-0.70无毒性0.952 10.440 2非底物或抑制剂ACE抑制剂/DPP-III抑制剂/DDP-IV抑制
2.4 消化产物的活性预测
本研究使用BIOPEP程序进一步预测了9种寡肽的具体生物活性(表3)。结果表明,在这些寡肽中,DF、GR、GPR、PGNR、PGPK、PGPR、QYGGR显示出ACE抑制活性,YR、HMR、GPR、PGNR、PGPK、PGPR、QYGGR显示出DDP-IV抑制活性,YR、GPR、HMR、PGPR、QYGGR显示出DDP-III抑制活性,GPR、PGNR、PGPK、PGPR显示出抗血栓和抗失忆活性,PGNR显示出肾素抑制活性,YR显示出神经肽活性等。这些结果表明,紫贻贝蛋白在消化后产生了大量生物活性肽,因而具有良好的营养价值和生物学功能。
2.5 ACE抑制肽的筛选
本研究使用SwissDock分子对接比较了7条预测具有ACE抑制活性寡肽的抑制能力(表4)。7条寡肽与ACE蛋白对接时的吉布斯自由能均为负值,表明这些寡肽均能与ACE发生良好相互作用,从而发挥抑制活性。根据文献检索,发现其中GR已有报道,其IC50值为3 110 μmol/L,而GPR和DF作为寡肽序列的N末端(GPRGF)或C末端(KPAGDF)部分报道,其余4条寡肽均为尚未报道的新型ACE抑制肽。在这4条新型寡肽中,五肽QYGGR的ΔG值最小(-11.19 kcal/mol),该对接结果优于大西洋鲑胶原蛋白(ΔG值最小为四肽GPEG,ΔG:-9.87 kcal/mol)[19]和黄花鱼肌联蛋白(ΔG值最小为三肽WAR,ΔG:-10.40 kcal/mol)[20]模拟水解多肽中ACE抑制肽的筛选结果,表明QYGGR具有良好的ACE抑制能力和开发潜力。
表4 ACE抑制肽的筛选
Table 4 Screening of ACE inhibitory peptides

多肽序列预测的ΔG/(kcal·mol-1)IC50/(μmol·L-1)参考文献QYGGR-11.19/PGPR-10.51/PGNR-9.60/GPR-9.48200.91(GPRGF)[23]PGPK-9.15/DF-8.4177.45(KPAGDF)[24]GR-7.743 110[25]
在所筛选出的7条潜在的ACE抑制肽中,QYGGR、PGPR和GPR来源于前胶原蛋白D,PGNR、GPR、PGPK和GR来源于前胶原蛋白P,而DF来源于肌动蛋白,由此可以看出胶原蛋白在获得新型ACE抑制肽方面具有重要研究价值。从另一方面考虑,紫贻贝中胶原蛋白含量约为干重的8%[21],而肌球蛋白和肌动蛋白分别约占干重的20%和30%[22]。因此,对于单一多肽,肌动蛋白来源的多肽丰度可以大致估算为胶原蛋白来源多肽的4倍,表明尽管通过模拟消化从肌动蛋白所获得的ACE抑制肽的条数较少,但由于其相对高的丰度,因而在紫贻贝消化物的ACE抑制活性方面仍具有一定作用。
2.6 DPP-IV抑制肽的筛选
DPP-IV参与肠促胰岛素激素加工过程并在血糖调节中起关键作用[26]。基于此,本研究根据表5中的筛选结果,使用iDPPIV-SCM程序对具有潜在DPP-IV抑制活性的7条寡肽打分。除PGPR和QYGGR外,5条寡肽的预测得分大于阈值294,表明其具有DPP-IV抑制活性,其中HMR的抑制活性最高,其预测得分达到380.5,优于已报到的大部分植物来源(大豆、羽扇豆、藜麦)的DPP-IV抑制肽[27]。同时,通过文献检索发现以上5条寡肽均无相关文献报道,表明它们是具有良好研究价值的新型DPP-IV抑制肽。由于HMR的预测打分最高,因此将其用作潜在的DPP-IV抑制剂进行后续研究。
表5 DPP-IV抑制肽的筛选
Table 5 Screening of DPP-IV inhibitory peptides
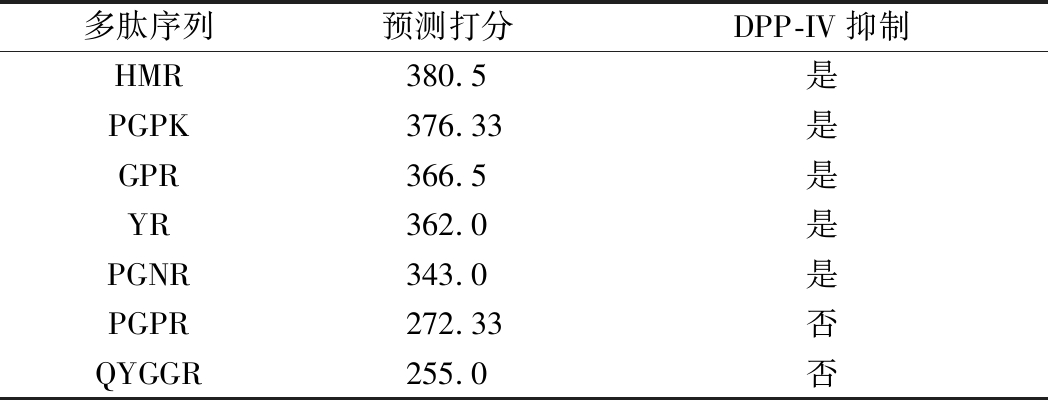
多肽序列预测打分DPP-IV抑制HMR380.5是PGPK376.33是GPR366.5是YR362.0是PGNR343.0是PGPR272.33否QYGGR255.0否
在所筛选出的5条潜在的DPP-IV抑制肽中,GPR来源于前胶原蛋白D,PGPK、GPR和PGNR来源于前胶原蛋白P,而HMR、YR来源于肌球蛋白,由此可见胶原蛋白和肌球蛋白在获得新型DPP-IV抑制肽方面具有重要价值。同样,从多肽丰度角度考虑,胶原蛋白和肌球蛋白来源的多肽均在紫贻贝消化物的DPP-IV抑制活性方面发挥着重要作用。
2.7 分子对接
为了进一步阐明多肽与目标蛋白之间的相互作用,本研究进一步将QYGGR(ACE抑制活性)和HMR(DPP-IV抑制活性)分别与ACE蛋白和DPP-IV蛋白进行分子对接。
在QYGGR与ACE的分子对接中,QYGGR能够结合ACE的活性残基,形成稳定复合物,其-CDOCKER能值为-156.078 kcal/mol,表明两者之间具有良好的结合亲和力(图2-a)。对接结果显示QYGGR主要通过与ACE蛋白分子的Tyr523、Glu411、Ala356、Ala354、Ser355、His513、His353、Glu376、Gln281、Asn277、Thr166形成11个氢键,与Trp357、Glu384、Asp415、Glu376、Glu162形成静电吸引来形成稳定复合物(图2-b)。ACE蛋白分子的活性位点主要包括12个氨基酸残基,即Gln281、Glu411、His513、His383、Glu384、His387、Tyr523、His353、Glu162、Tyr520、Lys511和Ala354[17]。QYGGR通过与Tyr523、Glu411、Ala354、His513、His353、Gln281、Glu384、Glu162发生相互作用而产生抑制。
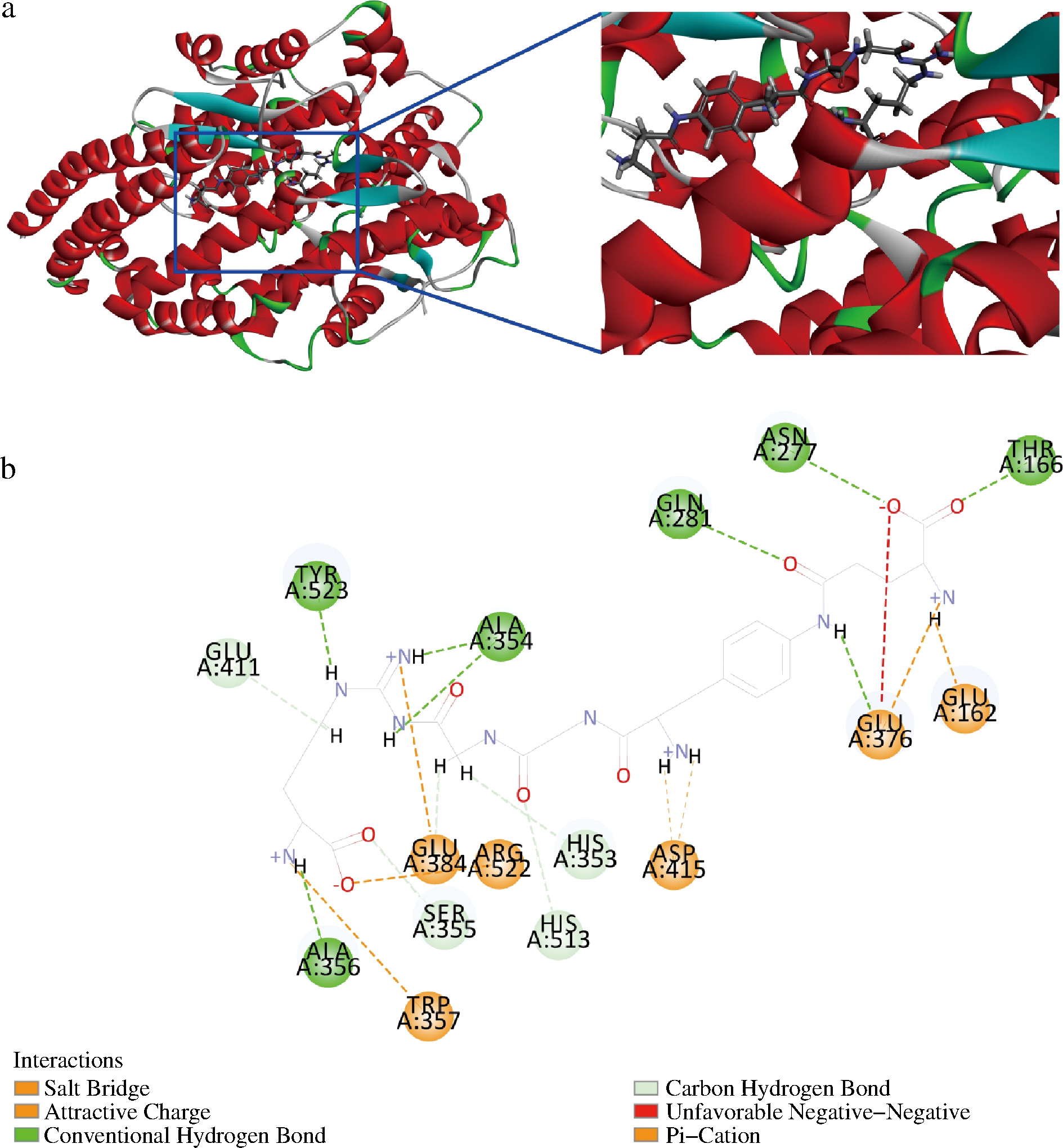
a-ACE-QYGGR复合物的预测3D结构;b-ACE-QYGGR分子相互作用的2D图
图2 ACE-QYGGR的分子对接图
Fig.2 Molecular docking diagram of ACE-QYGGR
在HMR与DPP-IV的分子对接中,HMR能够结合DPP-IV的活性残基,形成稳定复合物,其-CDOCKER能值为-92.410 kcal/mol,表明两者之间具有良好的结合亲和力(图3-a)。HMR主要通过与DPP-IV蛋白分子中的Tyr547、Tyr662和Glu205形成氢键、与His740形成π-烷基键、与Ser630、Tyr666和Tyr662形成π-π或酰胺-π键、与Tyr666、Asp663、Glu205、Glu206、Arg669和Arg358形成电荷吸引进行结合(图3-b)。DPP-IV分子包含3个活性口袋:S1、S2和S3,其中S1含有Tyr547、Ser630、Tyr631、Val656、Trp659、Tyr662、Tyr666、Asn710、Val711和His740;S2含有Glu205、Glu206和Tyr662;S3含有Ser209、Arg358和Phe357[18]。因此,HMR通过与S1中的Tyr547、Tyr662、His740、Ser630和Tyr666、S2中的Glu205、Glu206和Tyr662以及S3中的Arg358发生相互作用而产生抑制。
尽管本研究未对预测结果进行体外实验验证,但先前研究表明通过生物信息学方法对多肽分子生物活性的预测具有较好的可信性。例如,YU等[28]在对来源于卵转铁蛋白的ACE抑制三肽的研究中比较了分子对接过程中-CDOCKER能值最大的2种三肽EWL(106.486 kcal/mol)和KDF(103.876 kcal/mol)的体外抑制活性,结果显示-CDOCKER能值更高的三肽EWL同时也具有更强的体外抑制活性,表明分子对接方法能够在一定程度上指示小分子配体对目标蛋白的抑制效果。同时,本研究中所发现的ACE抑制肽QYGGR与ACE的-CDOCKER能值达到156.078 kcal/mol,远大于上述研究中的EWL,表明其具有较好的潜在ACE抑制活性和研究价值。同时,本研究中所发现的DPP-IV抑制肽HMR与DPP-IV的-CDOCKER能值达到92.410 kcal/mol,其高于鸡蛋来源的水溶性DPP-IV抑制肽CDR(-CDOCKER能值为81.371 3 kcal/mol,IC50=24.49 mmol/L)[29],表明其具有较好的潜在DPP-IV抑制活性。另一方面,尽管体外、体内和随机对照试验能够直接指示食源性多肽的生物活性,但这些肽对健康的有益作用仍需研究证实,而本研究提供了对紫贻贝源多肽活性的快速筛选,这为其长期研究提供了参考[30]。

a-DPP-IV-HMR复合物的预测3D结构;b-DPP-IV-HMR分子相互作用的2D图
图3 DPP-IV-HMR的分子对接图
Fig.3 Molecular docking diagram of DPP-IV-HMR
另外,本研究使用胃蛋白酶和胰蛋白酶的组合对紫贻贝蛋白进行模拟消化,获得了具有高ACE抑制活性的五肽QYGGR和高DPP-IV抑制活性的三肽HMR。然而,胰凝乳蛋白酶同样是人肠道中存在的一种重要的丝氨酸蛋白酶,因此对本研究中所筛选出的多肽存在潜在的酶切可能。基于上述考虑,使用胰凝乳蛋白酶(EC 3.4.21.1)分别对QYGGR和HMR进行模拟水解,发现两者均能被其进一步酶切。这提示当在人体中使用时,应进一步确认2种功能性多肽在体内的实际消化稳定性,从而为其广泛应用提供更完备的依据。
3 结论
本论文研究了紫贻贝主要蛋白的模拟消化情况,其中肌球蛋白和肌动蛋白水解度相对较高,分别达到30.85%和22.11%,而前胶原蛋白P、前胶原蛋白D水解度相对较低,均在10%左右,消化后4种蛋白均释放出大量寡肽(二肽至五肽)。通过PeptideRanker活性评分以及理化性质分析,从这些寡肽中筛选出9条具有潜在研究价值的多肽。通过ADMET分析和生物活性预测,发现9条寡肽中,DF、GR、GPR、PGNR、PGPK、PGPR、QYGGR具有ACE抑制活性,YR、HMR、GPR、PGNR、PGPK、PGPR、QYGGR具有DPP-IV抑制活性。对具有潜在ACE抑制活性和DPP-IV抑制活性的寡肽进行筛选,将QYGGR和HMR分别确定为具有潜在研究价值的ACE抑制肽和DPP-IV抑制肽。通过分子对接发现,QYGGR和ACE蛋白以及HMR和DPP-IV蛋白能够形成稳定复合物,其-CDOCKER能值分别为-156.078和-92.410 kcal/mol,其中QYGGR通过与ACE分子中的Tyr523、Glu411、Ala354、His513、His353、Gln281、Glu384、Glu162发生相互作用而产生抑制,而HMR通过与DPP-IV的S1中的Tyr547、Tyr662、His740、Ser630和Tyr666、S2中的Glu205、Glu206和Tyr662以及S3中的Arg358发生相互作用而产生抑制。本研究为紫贻贝营养价值的阐释以及紫贻贝来源的生物活性肽的开发提供了理论依据。
[1] 郭玉华, 李钰金, 吴新颖.贻贝食品的研究进展[J].肉类研究, 2009, 23(5):84-87.
GUO Y H, LI Y J, WU X Y.Advances on the research of food originated from mussel[J].Meat Research, 2009, 23(5):84-87.
[2] 金晶. 中国贻贝出口现状及发展对策研究[J].对外经贸实务, 2022(1):60-64.
JIN J.Research on the status quo and development countermeasures of China’s mussel exports[J].Practice in Foreign Economic Relations and Trade, 2022(1):60-64.
[3] 于莹, 宿小杰, 周德庆, 等.响应面法优化紫贻贝免疫活性肽的制备工艺[J].中国海洋药物, 2021, 40(6):21-29.
YU Y, SU X J, ZHOU D Q, et al.Optimization of preparation technology of immunomodulatory peptides from Mytilus edulis by response surface methodology[J].Chinese Journal of Marine Drugs, 2021, 40(6):21-29.
[4] 乔美玲, 刘汉雄, 樊凤娇, 等.贻贝盐溶蛋白特性分析及其ACE抑制肽的酶法制备[J].食品科学, 2018, 39(22):51-56.
QIAO M L, LIU H X, FAN F J, et al.Characterization of salt-soluble protein from Mytilus edulis and enzymatic preparation of ACE inhibitory peptides[J].Food Science, 2018, 39(22):51-56.
[5] JUNG W K, QIAN Z J, LEE S H, et al.Free radical scavenging activity of a novel antioxidative peptide isolated from in vitro gastrointestinal digests of Mytilus coruscus[J].Journal of Medicinal Food, 2007, 10(1):197-202.
[6] FENG L T, TU M L, QIAO M L, et al.Thrombin inhibitory peptides derived from Mytilus edulis proteins:Identification, molecular docking and in silico prediction of toxicity[J].European Food Research and Technology, 2018, 244(2):207-217.
[7] OH Y, AHN C B, CHO W H, et al.Anti-osteoporotic effects of antioxidant peptides PIISVYWK and FSVVPSPK from Mytilus edulis on ovariectomized mice[J].Antioxidants (Basel, Switzerland), 2020, 9(9):866.
[8] SHAN Y, DONG Y, JIANG D.Recombinant expression of a novel antimicrobial peptide consisting of human α-defensin 5 and Mytilus coruscus mytilin-1 in Escherichia coli[J].Journal of the Korean Society for Applied Biological Chemistry, 2015, 58(6):807-812.
[9] JO C, KHAN F F, KHAN M I, et al.Marine bioactive peptides:Types, structures, and physiological functions[J].Food Reviews International, 2017, 33(1):44-61.
[10] QIAO L S, LI B, CHEN Y K, et al.Discovery of anti-hypertensive oligopeptides from adlay based on in silico proteolysis and virtual screening[J].International Journal of Molecular Sciences, 2016, 17(12):2099.
[11] ABDELHEDI O, NASRI R, MORA L, et al.In silico analysis and molecular docking study of angiotensin I-converting enzyme inhibitory peptides from smooth-hound viscera protein hydrolysates fractionated by ultrafiltration[J].Food Chemistry, 2018, 239:453-463.
[12] MINKIEWICZ P, DZIUBA J, IWANIAK A, et al.BIOPEP database and other programs for processing bioactive peptide sequences[J].Journal of AOAC International, 2019, 91(4):965-980.
[13] MOONEY C, HASLAM N J, POLLASTRI G, et al.Towards the improved discovery and design of functional peptides:Common features of diverse classes permit generalized prediction of bioactivity[J].PLoS One, 2012, 7(10):e45012.
[14] GUPTA S, KAPOOR P, CHAUDHARY K, et al.In silico approach for predicting toxicity of peptides and proteins[J].PLoS One, 2013, 8(9):e73957.
[15] KAUTHALE S, TEKALE S, DAMALE M, et al.Synthesis, antioxidant, antifungal, molecular docking and ADMET studies of some thiazolyl hydrazones[J].Bioorganic & Medicinal Chemistry Letters, 2017, 27(16):3 891-3 896.
[16] LIU W Y, FENG X W, CHENG Q L, et al.Identification and action mechanism of low-molecular-weight peptides derived from Atlantic salmon (Salmo salar L.) skin inhibiting angiotensin I-converting enzyme[J].LWT, 2021, 150:111911.
[17] GROSDIDIER A, ZOETE V, MICHIELIN O.SwissDock, a protein-small molecule docking web service based on EADock DSS[J].Nucleic Acids Research, 2011, 39(suppl_2):W270-W277.
[18] CHAROENKWAN P, KANTHAWONG S, NANTASENAMAT C, et al.iDPPIV-SCM:A sequence-based predictor for identifying and analyzing dipeptidyl peptidase IV (DPP-IV) inhibitory peptides using a scoring card method[J].Journal of Proteome Research, 2020, 19(10):4 125-4 136.
[19] YU Z P, CHEN Y, ZHAO W Z, et al.Identification and molecular docking study of novel angiotensin-converting enzyme inhibitory peptides from Salmo salar using in silico methods[J].Journal of the Science of Food and Agriculture, 2018, 98(10):3 907-3 914.
[20] FAN Y, YU Z P, ZHAO W Z, et al.Identification and molecular mechanism of angiotensin-converting enzyme inhibitory peptides from Larimichthys crocea titin[J].Food Science and Human Wellness, 2020, 9(3):257-263.
[21] CIANCIOSI S C, HIRD F J R.The collagen content of selected animals[J].Comparative Biochemistry and Physiology Part B: Comparative Biochemistry, 1986, 85(2):295-298.
[22] 宋扬. 贝类肌肉副肌球蛋白的分布及理化性质[D].大连:大连海洋大学, 2018.
SONG Y.Paramyosin distribution of mollusks muscle protein in different commercial species[D].Dalian:Dalian Ocean University, 2018.
[23] FU Y, YOUNG J F, LØKKE M M, et al.Revalorisation of bovine collagen as a potential precursor of angiotensin I-converting enzyme (ACE) inhibitory peptides based on in silico and in vitro protein digestions[J].Journal of Functional Foods, 2016, 24:196-206.
[24] LI J Q, ZHAO J J, WANG X D, et al.Novel angiotensin-converting enzyme-inhibitory peptides from fermented bovine milk started by Lactobacillus helveticus KLDS.31 and Lactobacillus casei KLDS.105:Purification, identification, and interaction mechanisms[J].Frontiers in Microbiology, 2019, 10:2643.
[25] MOHD SALIM M A S, GAN C Y.Dual-function peptides derived from egg white ovalbumin:Bioinformatics identification with validation using in vitro assay[J].Journal of Functional Foods, 2020, 64:103618.
[26] NONGONIERMA A B, FITZGERALD R J.Features of dipeptidyl peptidase IV (DPP-IV) inhibitory peptides from dietary proteins[J].Journal of Food Biochemistry, 2019, 43(1):e12451.
[27] RIVERO-PINO F, ESPEJO-CARPIO F J, GUADIX E M.Identification of dipeptidyl peptidase IV (DPP-IV) inhibitory peptides from vegetable protein sources[J].Food Chemistry, 2021, 354:129473.
[28] YU Z P, CHEN Y, ZHAO W Z, et al.Novel ACE inhibitory tripeptides from ovotransferrin using bioinformatics and peptidomics approaches[J].Scientific Reports, 2019, 9:17434.
[29] ZHAO W Z, ZHANG D, YU Z P, et al.Novel membrane peptidase inhibitory peptides with activity against angiotensin converting enzyme and dipeptidyl peptidase IV identified from hen eggs[J].Journal of Functional Foods, 2020, 64:103649.
[30] BARATI M, JAVANMARDI F, JABBARI M, et al.An in silico model to predict and estimate digestion-resistant and bioactive peptide content of dairy products:A primarily study of a time-saving and affordable method for practical research purposes[J].LWT, 2020, 130:109616.