泥窖生香发酵是我国浓香型白酒的独特生产方式,窖泥在浓香型白酒中发挥着不可或缺的作用,测序技术显示窖泥微生物群落主要由梭菌纲和产甲烷古菌组成[1-3]。梭菌属(Clostridium)是梭菌纲中的一个优势属,约占细菌总丰度的0.54%~4.42%[1,4-6]。与退化窖泥相比,正常和优质窖泥中梭菌的相对丰度明显更高[4],而且梭菌属的相对丰度会随着窖龄的增加而增加[6]。梭菌属不仅是窖泥群落中的一个丰富类群,在浓香型白酒典型风味物质的形成中也发挥着重要作用[7]。目前已经通过分离培养和高通量测序方法证明梭菌属微生物是窖泥中短/中链脂肪酸合成的重要属之一[2,8-10]。此外,高通量测序结果显示,不同窖泥样本中互营细菌(包括互营球菌、互营单胞菌等)的丰度占总细菌的1.35%~7.64%[1,2,11],互营细菌能够与其他微生物进行互营代谢,但其在白酒酿造体系中与其他微生物的互作报道较少。
传统发酵食品大多在开放环境中进行生产,发酵过程中会形成复杂的微生物群落。微生物间相互作用方式有互利共生、偏利共生和竞争等多种类型,微生物间的互作在驱动微生物群落组装[12]、维持群落结构稳定[13]和调节群落代谢功能[14]方面起着至关重要的作用。到目前为止,关于窖泥微生物群相互作用关系的研究主要基于高通量测序数据的相关性分析[4,15]。例如,通过共现网络分析发现梭菌纲(Clostridia)、拟杆菌纲(Bacteroidia)、甲烷杆菌纲(Methanobacteria)和甲烷微菌纲(Methanomicrobia)之间存在大量显著的正相关或负相关[4,11,16]。然而,基于共现性网络等相关性分析方法研究的窖泥微生物群相互作用受到不同算法的影响[17],潜在的互作关系需要通过其他方法进一步验证。尽管目前已经从窖泥中分离出许多产脂肪酸的梭菌属微生物,但是关于梭菌属与其他物种之间相互作用对脂肪酸代谢的影响鲜有研究,这限制了对窖泥微生物功能的全面理解[8,9,18-19]。
实验室前期从窖泥中分离筛选到1株互营细菌,经系统发育分析其属于毛螺菌科(Lachnospiraceae),命名为Novisyntrophococcus fermenticellae JN500902T,与互营球菌Syntrophococcus sucromutans DSM 3224T具有最高的序列相似性(92.8%)[20]。在前期对窖泥梭菌分离筛选时发现,互营球菌与部分梭菌共培养时存在显著的协同效应,当前者存在时,这些梭菌菌株产丁酸、己酸等脂肪酸的能力可显著提高。己酸和丁酸是浓香型白酒主体风味物质己酸乙酯和丁酸乙酯形成的重要前体物质。因此,为进一步探究梭菌和互营球菌之间的互作规律,本研究以从窖泥中筛选出的1株互营球菌和7株梭菌属微生物为研究对象,结合比较基因组分析和共培养的方法,分析不同梭菌属微生物的短链脂肪酸代谢途径,以及与互营球菌共培养对生长和短链脂肪酸产生的影响。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
互营球菌Novisyntrophococcus fermenticellae JN500902(N.902)、老窖梭菌Clostridium fermenticellae JN500901(C.901)、酪丁酸梭菌C.tyrobutyricum Clt01、粪味梭菌C.scatologenes Cls01、路氏梭菌C.luticellarii Clu07、嗜酸梭菌C.aciditolerans Claci01、广西梭菌C.guangxiense Clgx01和拜式梭菌C.beijerinckii Clb01。以上菌株均分离自浓香型白酒窖泥,其中,N.902和C.901保藏于中国工业微生物菌种保藏管理中心(China Center of Industrial Culture Collection,CICC),编号分别为CICC 24502T和CICC 24501T,其他菌株保存于本实验室。
1.1.2 培养基
强化梭菌培养基(RCM)(g/L):蛋白胨10,酵母粉3,葡萄糖5,可溶性淀粉1,乙酸钠3,半胱氨酸盐酸盐0.5,琼脂粉20。添加1 mL/L(1.0 g/L)的刃天青作为厌氧指示剂。pH=6.28,121 ℃灭菌20 min。
1.1.3 主要仪器
Whitley DG250厌氧箱,英国Don Whitley Scientific;Tecan Spark多模微孔板阅读器,瑞士Tecan;S22型微量pH计,梅特勒-托利多(上海)仪器有限公司;SBA-40D生物传感分析仪,山东省科学院生物研究所;Waters e2695高效液相色谱,美国Waters公司。
1.1.4 主要试剂
RCM培养基原料,国药集团化学试剂有限公司;乙酸、丁酸和戊酸,Sigma-Aldrich公司;其他化学试剂,国药集团化学试剂有限公司。
1.2 实验方法
1.2.1 微生物系统进化树分析
基于7株菌的16S rRNA基因全长序列,利用MEGA X软件构建进化树[21]。分析方法为邻位相连法(neighbor-joining method),模型选择Kimura 2-parameter model,进化树的检验方法为bootstrap method,设置重复抽样次数为1 000,其他参数使用默认值[22]。
1.2.2 梭菌泛基因组分析
从NCBI上下载所研究梭菌物种的模式种或相似度最高的菌株基因组序列文件,基因组具体信息如表1所示。利用Prokka v1.14.6软件预测梭菌基因组的开放阅读框(open reading frame,ORF)[23]。使用eggNOG-Mapper对7个梭菌基因组的ORF进行功能注释,E-value设为1e-3[24]。采用PanGP软件的幂律模型,拟合梭菌的基因组数量与泛基因组大小和核心基因组数量的曲线[25]。使用在线网站Venn diagrams(http://bioinformatics.psb.ugent.be/webtools/Venn/)统计7株梭菌属微生物的核心基因组和菌株特异性基因进行可视化。根据KEGG(Kyoto Encyclopedia of Genes and Genomes)注释结果,统计梭菌属微生物短链脂肪酸代谢通路中基因的数量差异,绘制代谢通路图。
表1 模式菌株或代表菌株的基因组信息
Table 1 Genomic information of type strains or representative strains
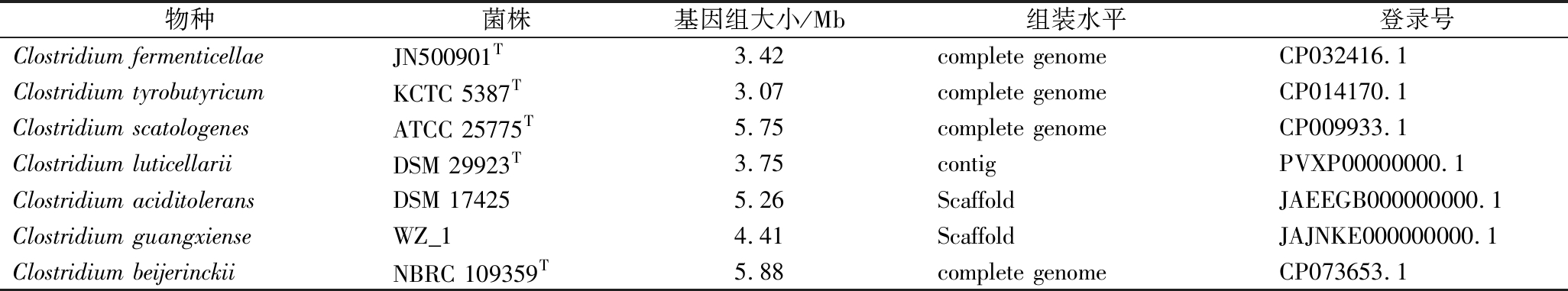
物种菌株基因组大小/Mb组装水平登录号Clostridium fermenticellaeJN500901T3.42complete genomeCP032416.1Clostridium tyrobutyricumKCTC 5387T3.07complete genomeCP014170.1Clostridium scatologenesATCC 25775T5.75complete genomeCP009933.1Clostridium luticellariiDSM 29923T3.75contigPVXP00000000.1Clostridium aciditoleransDSM 174255.26ScaffoldJAEEGB000000000.1Clostridium guangxienseWZ_14.41ScaffoldJAJNKE000000000.1Clostridium beijerinckiiNBRC 109359T5.88complete genomeCP073653.1
1.2.3 微生物共培养
种子液的制备:取出于-80 ℃冰箱中保藏的互营球菌N.902和7株梭菌,连续培养传代3次,经划线分离后获得纯菌株,用RCM液体培养基培养至对数生长期,制备活力良好的种子液。
共培养:互营球菌N.902和不同梭菌在装有100 mL的250 mL三角瓶中进行纯培养和共培养实验。收集对数生长中期活力良好的互营球菌N.902和梭菌种子液,测量种子液的OD600值后,参考CHARUBIN等[26]的研究计算种子液的接种量,使得接种后纯培养体系的理论初始OD600值为0.01,共培养体系理论初始OD600值为0.02。其中,共培养中互营球菌和梭菌的接种量保持一致。所有培养都在厌氧培养箱中进行,培养温度为37 ℃,培养时间为48 h,每隔6 h取样测定菌株生长和脂肪酸代谢情况。
1.2.4 微生物生长的检测
每隔6 h从上述纯培养和共培养的三角瓶中吸取200 μL发酵液至96孔板中,采用Tecan Spark多模微孔板阅读器测量OD600值的变化情况。
1.2.5 葡萄糖和pH值的检测
取发酵过程菌液0.5 mL,经适当稀释后离心(12 000 r/min,5 min)获得上清液,过滤膜除杂后,使用生物传感仪测量上清液中葡萄糖含量。另取1 mL发酵液用微量pH计测定pH值。
1.2.6 短链脂肪酸的检测
使用色谱柱为Sapphire C18(4.6 mm×250 mm,5 μm)测定短链脂肪酸;流动相:1 mL/min 70% NaH2PO4和30%的乙腈,用磷酸调节pH=2.7;进样量为10 μL;流速为1 mL/min;柱温为40 ℃;紫外检测波长设置为210 nm;每个样本的运行程序为20 min。
1.3 数据处理与可视化
采用在线网站(http://www.bioinformatics.com.cn)绘制花瓣图,Graphpad Prism 8软件绘制热图,Adobe Illustrator软件绘制短链脂肪酸代谢通路。采用OriginPro 2021软件对纯培养和共培养的OD600、pH和葡萄糖变化进行数据处理与绘图。所有图片的组合使用Adobe Illustrator软件。
2 结果与分析
2.1 系统发育树和泛基因组分析
将互营球菌N.902和7株梭菌的16S rRNA基因全长序列在EzBioCloud数据库和NCBI数据库中进行Blast,互营球菌N.902和老窖梭菌C.901为实验室前期从窖泥中筛选出的新种,全基因组测序结果已经提交至GenBank,其比对结果相似度都为100%。另外6株细菌与数据库的Blast结果也都在97%以上,符合种水平序列相似度标准,具体的比对结果如表2所示。
表2 不同菌株的16S rRNA比对结果
Table 2 Alignment results of different strains based on 16S rRNA gene sequences

菌株编号数据库比对结果相似度/%N.902Novisyntrophococcus fermenticellae JN500902100C.901Clostridium fermenticellae JN500901100Clt01Clostridium tyrobutyricum DSM 263798.65Cls01Clostridium scatologenes ATCC 2577599.93Clu07Clostridium luticellarii FW43197.31Claci01Clostridium aciditolerans JW/YJL-B399.56Clgx01Clostridium guangxiense ZGM21198.50Clb01Clostridium beijerinckii DSM 79198.58
以大肠杆菌Escherichia coli ATCC 11775T为外群构建最大似然法系统发育树,结果表明互营球菌N.902单独聚为一簇,梭菌属微生物分成两大支,嗜酸梭菌Claci01、粪味Cls01、老窖梭菌C.901、酪丁酸梭菌Clt01和路氏梭菌Clu07聚为一大支,拜式梭菌Clb01和广西梭菌Clgx01进化距离较近,两者单独聚为一簇(图1-a)。
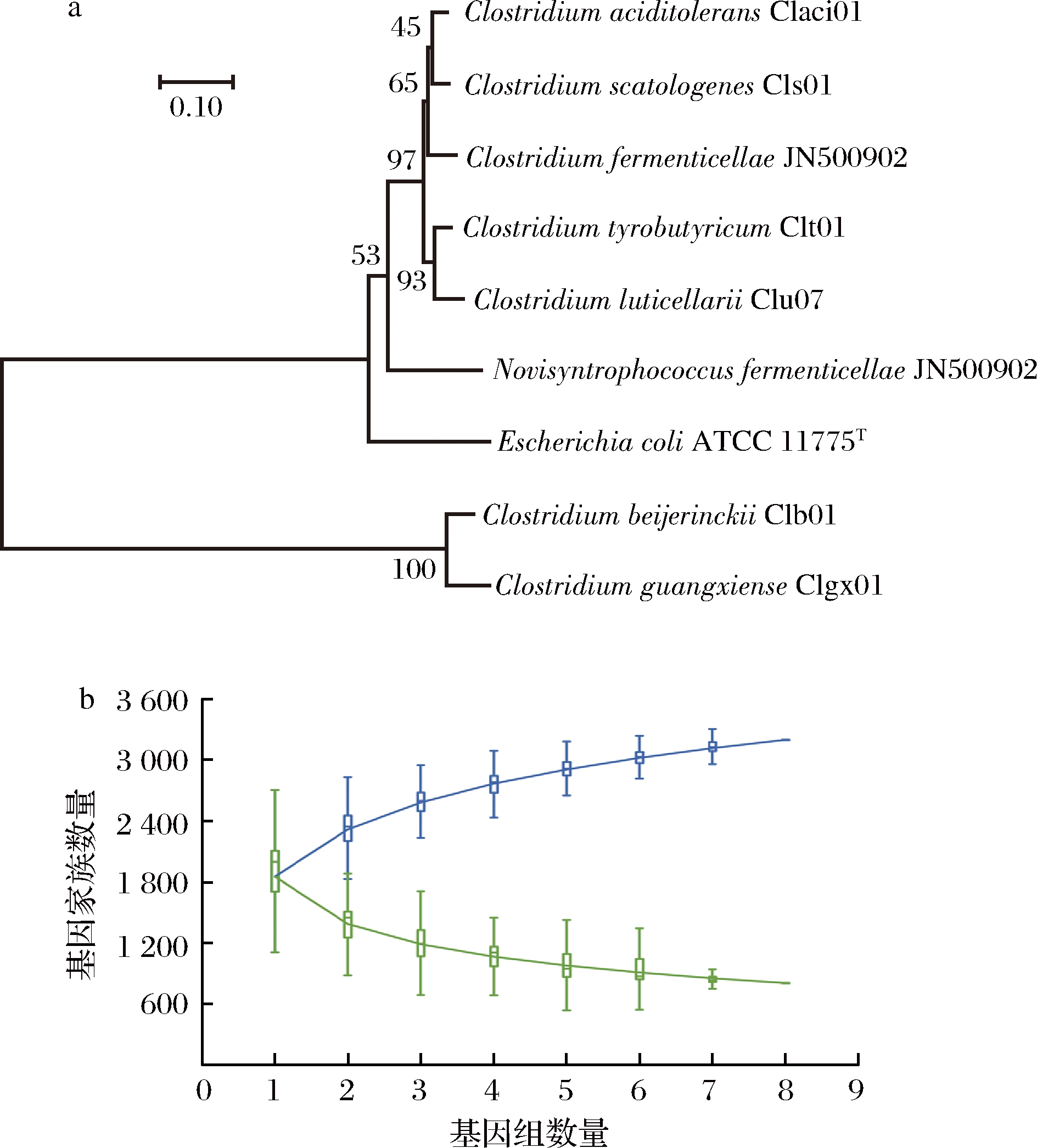
a-基于16S rRNA基因序列的系统发育树;b-7株梭菌的泛基因组积累曲线
图1 系统进化分析和泛基因组分析
Fig.1 Phylogenetic analysis and pan-genome analysis
梭菌属微生物是浓香型白酒中重要的产香功能菌群,为了进一步研究窖泥中梭菌属微生物的生理和代谢差异,我们从NCBI数据库中下载了7株梭菌属细菌的全基因组进行比较分析。如图1-b所示,泛基因组积累曲线表明梭菌属的核心基因组随着基因组数目的增多逐渐减小,而泛基因组呈现线性增加的趋势,由此可以推断梭菌具有开放型的泛基因组。这一结果与对其他梭菌属成员的研究一致,如C.butyricum[27]和C.beijerinckii[28]等,但是值得注意的是,泛基因组积累曲线受到分析的基因组数量的限制[29]。梭菌属微生物基因组的异质性,可能会影响互营球菌与不同梭菌的互作类型。
2.2 梭菌属微生物基因组的KEGG功能注释
泛基因组结果表明,7株梭菌的核心基因组由1 029个基因家族组成,占泛基因组的34%左右。特有基因由604个基因家族组成,占泛基因组的20%左右(图2-a)。对核心基因组进行功能注释,可以注释到细胞过程(cellular processes)、环境信息处理(environmental information processing)、基因信息处理(genetic information processing)、代谢(metabolism)和有机体系统(organismal systems)5个大类。其中,注释到代谢大类的基因数量最多,老窖梭菌C.901、嗜酸梭菌Claci01、拜式梭菌Clb01、广西梭菌Clgx01、路氏梭菌Clu07、粪味梭菌Cls01和酪丁酸梭菌Clt01分别注释到445、528、651、672、588、590、702和581个(图2-b)。7株梭菌都含有特异基因,其中拜式梭菌Clb01的特有基因最多,含有180个,其次为嗜酸梭菌Claci01(125个)、广西梭菌Clgx01(104个)、粪味梭菌Cls01(89个)、路氏梭菌Clu07(68个)、酪丁酸梭菌Clt01(28个)和老窖梭菌C.901(10个)。拜式梭菌Clb01中的特异基因主要集中在碳水化合物代谢(carbohydrate metabolism)和能量代谢(energy metabolism)方面,分别有32和23个。老窖梭菌C.901注释到的特异基因最少,均匀分布在信号转导(signal transduction)、脂质代谢(lipid metabolism)和辅因子与维生素代谢(metabolism of cofactors and vitamins)等方面。其余梭菌的特异基因则主要分布在碳水化合物代谢(carbohydrate metabolism)和氨基酸代谢(amino acid metabolism)方面(图2-c)。上述注释结果表明,梭菌属微生物在代谢方面可能存在一定的种间差异。
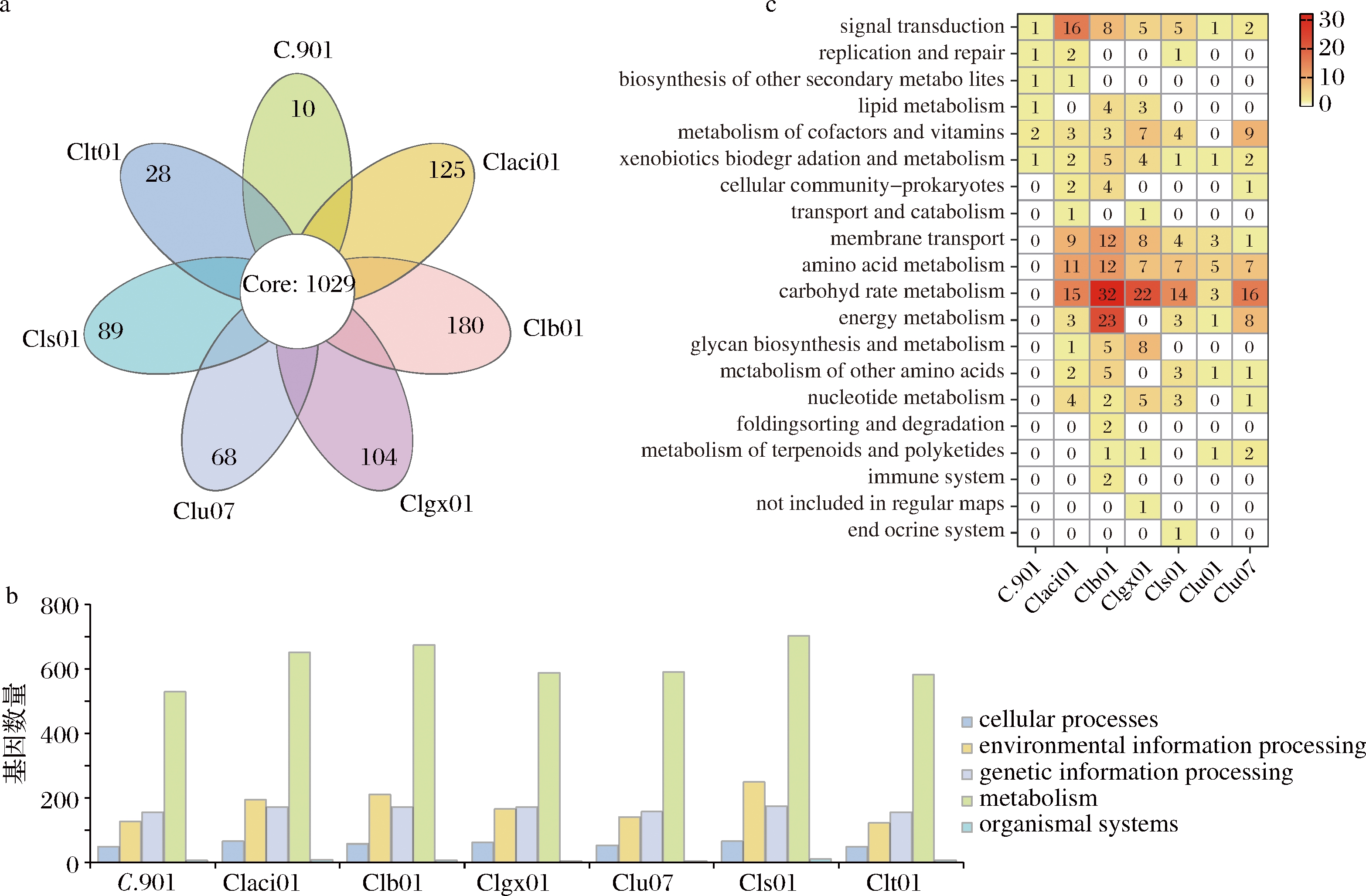
a-核心基因组与特异基因;b-核心基因组功能注释;c-特异基因功能注释
图2 梭菌属细菌基因组的功能注释
Fig.2 Functional annotation of Clostridium strains’ genomes
2.3 梭菌短链脂肪酸代谢通路分析
前期研究表明厌氧微生物可以通过反向β-氧化途径利用乙酸进行碳链延长,乳酸和乙醇是有效电子供体[30]。在浓香白酒的发酵体系中,存在糖、乙酸、乳酸和乙醇等多种底物[10]。KEGG代谢通路结果显示,这7株从窖泥中筛选出的梭菌属细菌都存在与葡萄糖、乙酸、乳酸和乙醇利用相关的基因,但是短链脂肪酸的代谢途径存在差异(图3)。

图3 七株梭菌属微生物的短链脂肪酸代谢通路
Fig.3 Metabolic pathways of short-chain fatty acids in seven strains of the genus Clostridium
7株梭菌属细菌的基因组中都存在较完整的丁酸和己酸产生相关的基因,但是路氏梭菌Clu07和酪丁酸梭菌Clt01中未注释到编码丁酰磷酸转移酶(phosphate butyryltransferase,EC:2.3.1.19)和丁酸激酶(butyrate kinase,EC:2.7.2.7)相关的基因(buk途径),仅存在but途径产生丁酸。这和CHAI等[8]通过克隆文库对窖泥中产丁酸梭菌的研究结果一致,窖泥中梭菌属的产丁酸途径具有种间特异性。此外,全基因组的注释结果显示互营球菌N.902可以通过丙酮酸氧化途径、Wood-Ljungdahl乙酰化途径产乙酸、乙醇等代谢物,但不具备逆β-氧化途径,无法进行碳链的进一步延伸(图4)。

图4 互营球菌基因组中的产乙酸代谢途径
Fig.4 Metabolic pathway of acetic acid production in the genome of N.902
这和下文中N.902纯培养中主要检测到乙酸,检测不到2C以上的羧酸的结果一致。基因组的注释结果中互营球菌N.902和梭菌的代谢途径存在一定互补,N.902产生的乙酸、乙醇等是梭菌碳链延伸途径中的底物。7株梭菌都具有编码乙酰CoA(acetyl-CoA)生成丙二酰CoA(malonyl-CoA),进一步生成丙酰CoA(propionyl-CoA)的相关基因。丙酰CoA作为重要的前体物质,可以进一步代谢生成丙酸、戊酸等奇数链脂肪酸。除上述途径外,嗜酸梭菌Claci01、路氏梭菌Clu07和粪味梭菌Cls01也可以利用葡萄糖产生甘油磷酸(glycerone phosphate),然后代谢产生丙酰CoA进入逆β-氧化途径。综合基因组注释结果来看,互营球菌N.902和梭菌都可以利用葡萄糖产生短链脂肪酸,这可能导致共培养中互营球菌和梭菌对葡萄糖的竞争,进而影响短链脂肪酸的产生。
2.4 互营球菌和梭菌纯培养和共培养中的生长代谢差异
窖泥中的微生物不是独立存在的,它们之间会发生复杂的交互作用。实验室前期在分离筛选窖泥微生物时发现互营球菌N.902具有促进梭菌属微生物短链脂肪酸产生的潜力[20]。为了进一步研究互营球菌N.902和梭菌之间的交互作用,我们将互营球菌N.902与7株梭菌属微生物进行共培养。如图5所示,互营球菌N.902和7株梭菌共培养中的生长、葡萄糖代谢和pH的变化存在较大差异。

a-N.902和C.901共培养;b-N.902和Claci01共培养;c-N.902和Clb01共培养;d-N.902和Clgx01共培养;e-N.902和Clu07共培养;f-N.902和Cls01共培养;g-N.902和Clt01共培养
图5 互营球菌和梭菌纯培养和共培养中的生长代谢差异
Fig.5 Differences in growth and metabolism of N.902 and Clostridium in mono- and co-culture
互营球菌N.902和老窖梭菌C.901共培养显著促进生长,生长延滞期缩短,OD值的最大变化速率也高于纯培养,发酵结束时共培养的OD值(0.80)高于C.901(0.39)和N.902(0.39)纯培养的OD值之和。共培养促进了葡萄糖的消耗,48 h利用了3.38 g/L。此外,共培养48 h后pH值为5.58,介于C.901(7.09)和N.902(4.95)之间(图5-a)。互营球菌N.902和嗜酸梭菌Claci01共培养对生长、葡萄糖的消耗及pH的影响不大,但是共培养OD值的最大变化速率高于Claci01纯培养。这可能和Claci01的生长速度快,在共培养中占据优势地位有关(图5-b)。互营球菌N.902和拜式梭菌Clb01共培养前期(12 h之前)对生长有一定的抑制作用,但是12 h之后随着N.902在共培养中逐渐开始生长,共培养的OD值开始超过Clb01纯培养。发酵终点时,纯培养和共培养的OD值都为1.0左右,但生长的最大变化速率远高于Clb01纯培养,此外,共培养的葡萄糖消耗量也更高(图5-c)。互营球菌N.902和广西梭菌Clgx01共培养的延滞期比Clgx01纯培养提前6 h,发酵结束时共培养(1.1)中OD值高于纯培养(0.9)。此外,共培养的葡萄糖最大消耗速率和pH最大变化速率出现时间比Clgx01纯培养更早(图5-d)。
互营球菌N.902和路氏梭菌Clu07共培养显著促进了生长,48 h时OD值为0.7左右,远高于纯培养。共培养中葡萄糖的消耗量最高,发酵至42 h左右就已耗尽。Clu07纯培养中pH逐渐上升,在42 h左右达到最高点6.4左右,而N.902和共培养的pH均呈下降趋势,这可能是由于共培养中N.902大量产酸所致(图5-e)。互营球菌N.902和粪味梭菌Cls01共培养的生长曲线和葡萄糖消耗曲线与Cls01纯培养基本一致。发酵结束时,Cls01的pH维持在5.5左右,共培养的pH低至5.2左右(图5-f)。互营球菌N.902和酪丁酸梭菌Clt01共培养生长延滞期缩短至6 h,但是最大生长速率低于纯培养。N.902,Clt01和共培养三者的pH都呈现下降趋势,分别在发酵结束达到4.7、5.0和5.2左右(图5-g)。综上,N.902和不同梭菌共培养对生长产生不同的影响,这可能和多种因素有关。YAO等[31]研究表明不同葡萄糖浓度下,C.bovifaecis的代谢流会发生变化,进一步影响生长。共培养中两株菌对葡萄糖的竞争性利用可能会改变代谢流。除此之外,微生物代谢产生的酸类也会改变环境pH造成菌株的生长压力[32]。
2.5 互营球菌和梭菌纯培养和共培养中的短链脂肪酸代谢差异
进一步研究了互营球菌N.902和不同梭菌互作对短链脂肪酸积累的影响。除嗜酸梭菌Claci01外,6株梭菌都可以产生丁酸,只有老窖梭菌C.901中检测到己酸存在(表3)。此外,嗜酸梭菌Claci01、路氏梭菌Clu07和粪味梭菌Cls01中都检测到奇数碳戊酸的积累,这和基因组的注释结果一致,它们都具备利用葡萄糖代谢产生的甘油醛三磷酸途径生成丙酰CoA的能力,丙酰CoA可以进入逆β-氧化途径产生奇数碳脂肪酸。比较纯培养和共培养中短链脂肪酸的代谢差异,结果表明互营球菌N.902与老窖梭菌C.901、路氏梭菌Clu07和粪味梭菌Cls01共培养能够促进中短链脂肪酸的积累。互营球菌N.902与老窖梭菌C.901共培养对培养基中乙酸的利用减少,但是丁酸和己酸的积累量分别增加0.44倍和0.63倍左右;互营球菌N.902与路氏梭菌Clu07共培养后,丁酸和戊酸的产量分别增加了0.77倍和2.63倍;互营球菌N.902和粪味梭菌Cls01共培养丁酸的产量变化不大,但戊酸的积累量略有增加0.12倍。互营球菌N.902的主要代谢产物是乙酸和乙醇,前期的研究表明乙醇和乙酸可以作为逆β-氧化途径的底物,因此共培养中的代谢物的交叉喂养可能是促进短链脂肪酸积累的原因之一[29]。共培养也会对短链脂肪酸的积累产生负作用,互营球菌N.902和嗜酸梭菌Claci01共培养中戊酸的积累量比纯培养中降低0.63倍左右。互营球菌N.902和3株主要产丁酸的拜式梭菌Clb01、广西梭菌Clgx01和酪丁酸梭菌Clt01共培养中丁酸的积累量都发生不同程度的下调。互营球菌N.902与梭菌共培养对丁酸代谢影响不一致可能与生长、碳源的利用、葡萄糖的消耗及中间代谢物等多种因素有关[33-34]。
表3 互营球菌和梭菌纯培养和共培养中短链脂肪酸代谢差异
Table 3 Difference of short-chain fatty acid metabolism between mono-and co-culture of N.902 and Clostridium
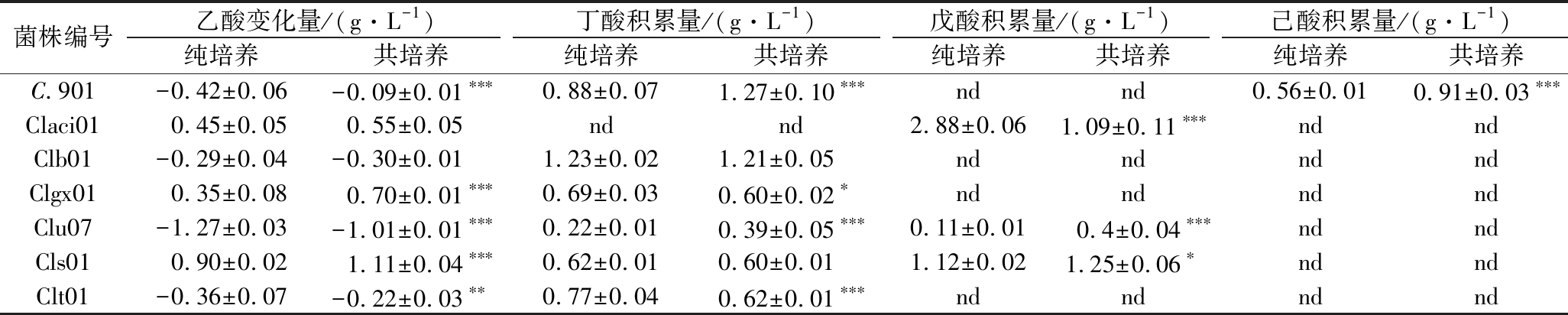
菌株编号乙酸变化量/(g·L-1)丁酸积累量/(g·L-1)戊酸积累量/(g·L-1)己酸积累量/(g·L-1)纯培养共培养纯培养共培养纯培养共培养纯培养共培养C.901-0.42±0.06-0.09±0.01∗∗∗0.88±0.071.27±0.10∗∗∗ndnd0.56±0.010.91±0.03∗∗∗Claci010.45±0.050.55±0.05ndnd2.88±0.061.09±0.11∗∗∗ndndClb01-0.29±0.04-0.30±0.011.23±0.021.21±0.05ndndndndClgx010.35±0.080.70±0.01∗∗∗0.69±0.030.60±0.02∗ndndndndClu07-1.27±0.03-1.01±0.01∗∗∗0.22±0.010.39±0.05∗∗∗0.11±0.010.4±0.04∗∗∗ndndCls010.90±0.021.11±0.04∗∗∗0.62±0.010.60±0.011.12±0.021.25±0.06∗ndndClt01-0.36±0.07-0.22±0.03∗∗0.77±0.040.62±0.01∗∗∗ndndndnd
注:培养基中乙酸的初始测量值为2.7 g/L,乙酸的变化量=发酵终点测量值-2.7,其中“-”代表消耗了乙酸,“+”代表积累了乙酸,“nd”代表未检测到对应短链脂肪酸;*代表纯培养和共培养之间差异显著性,*,P<0.05;**,P<0.01;***,P<0.001
3 结论
本研究通过比较基因组和培养的方法,证明了窖泥中不同梭菌属微生物产短链脂肪酸的多样性。通过两两菌株共培养体系表明,窖泥中的互营球菌和不同梭菌属微生物之间存在正、负相互作用(图6)。N.902与老窖梭菌C.901或路氏梭菌Clu07共培养对生长和短链脂肪酸的代谢都表现出显著的促进作用。N.902与Cls01共培养仅对单一短链脂肪酸的积累有促进作用。N.902与嗜酸梭菌Claci01、拜式梭菌Clb01、广西梭菌Clgx01或酪丁酸梭菌Clt01共培养时对生长的影响表现出多样化,但都对短链脂肪酸的积累产生抑制作用。本研究加深了对窖泥中梭菌属功能多样性的理解,对互营球菌和梭菌互作关系的研究拓宽了对窖泥微生物群落复杂互作网络的认识。
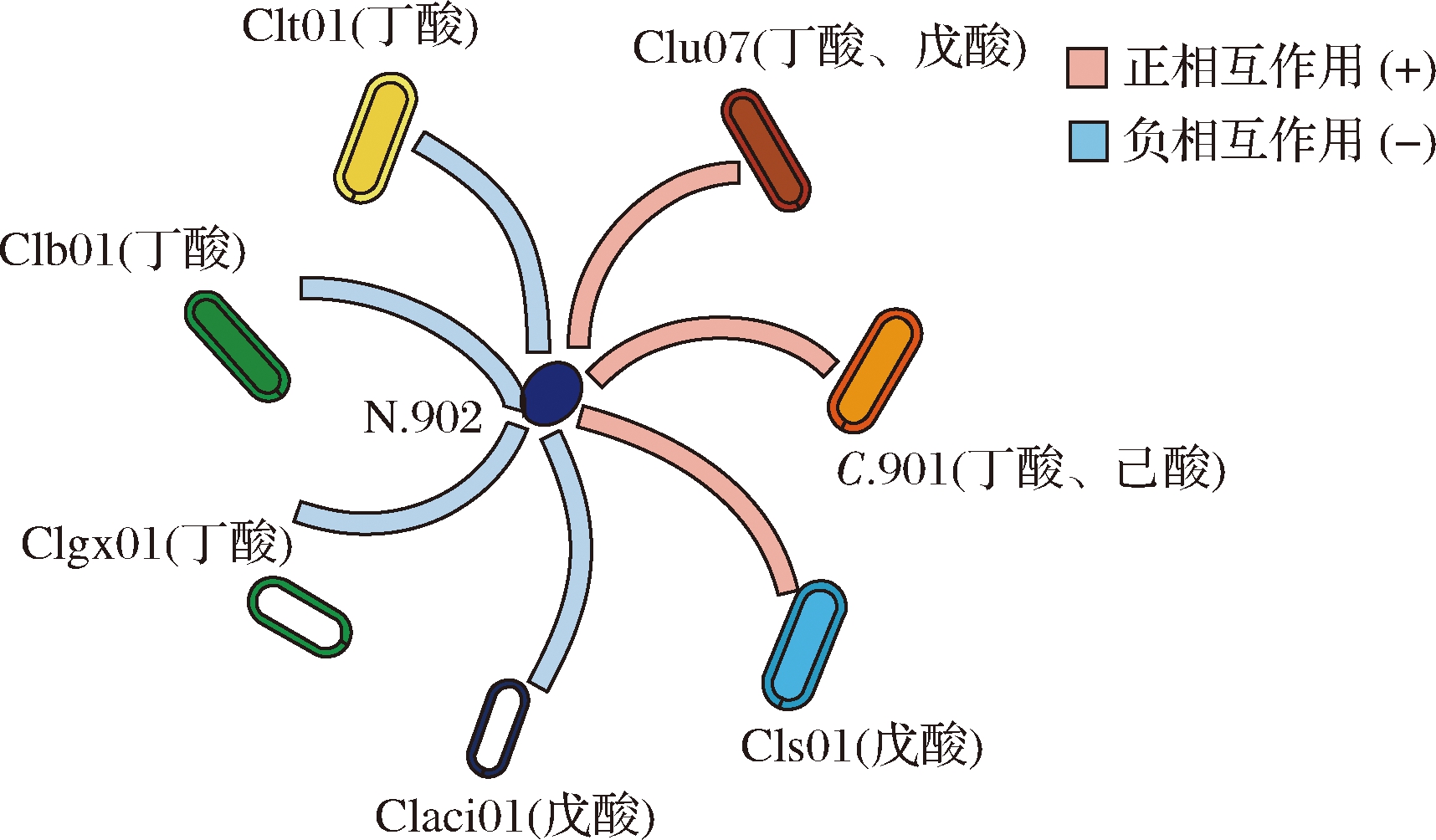
图6 互营球菌和不同梭菌的交互作用类型
Fig.6 Types of interaction between N.902 and different Clostridium species
[1] CHAI L J, QIAN W, ZHONG X Z, et al.Mining the factors driving the evolution of the pit mud microbiome under the impact of long-term production of strong-flavor Baijiu[J].Applied and Environmental Microbiology, 2021, 87(17):e0088521.
[2] FU J X, CHEN L, YANG S Z, et al.Metagenome and analysis of metabolic potential of the microbial community in pit mud used for Chinese strong-flavor liquor production[J].Food Research International, 2021, 143:110294.
[3] LIU M K, TANG Y M, GUO X J, et al.Deep sequencing reveals high bacterial diversity and phylogenetic novelty in pit mud from Luzhou Laojiao cellars for Chinese strong-flavor Baijiu[J].Food Research International, 2017, 102:68-76.
[4] HU X L, DU H, REN C, et al.Illuminating anaerobic microbial community and cooccurrence patterns across a quality gradient in Chinese liquor fermentation pit muds[J].Applied and Environmental Microbiology, 2016, 82(8):2 506-2 515.
[5] LIU M K, TANG Y M, ZHAO K, et al.Contrasting bacterial community structure in artificial pit mud-starter cultures of different qualities:A complex biological mixture for Chinese strong-flavor Baijiu production[J].3 Biotech, 2019, 9(3):89.
[6] TAN G X, ZHOU R, ZHANG W Q, et al.Detection of viable and total bacterial community in the pit mud of Chinese strong-flavor liquor using propidium monoazide combined with quantitative PCR and 16S rRNA gene sequencing[J].Frontiers in Microbiology, 2020, 11:896.
[7] ZHENG X W, HAN B Z.Baijiu, Chinese liquor:History, classification and manufacture[J].Journal of Ethnic Foods, 2016, 3(1):19-25.
[8] CHAI L J, XU P X, QIAN W, et al.Profiling the Clostridia with butyrate-producing potential in the mud of Chinese liquor fermentation cellar[J].International Journal of Food Microbiology, 2019, 297:41-50.
[9] 钱玮, 陆震鸣, 柴丽娟, 等.泸型酒酒醅与窖泥中梭菌群落结构、演替和功能差异[J].生物工程学报, 2020, 36(6):1 190-1 197.
QIAN W, LU Z M, CHAI L J, et al.Differences of the structure, succession and function of Clostridial communities between Jiupei and pit mud during Luzhou-flavour Baijiu fermentation[J].Chinese Journal of Biotechnology, 2020, 36(6):1 190-1 197.
[10] ZOU W, YE G B, ZHANG K Z.Diversity, function, and application of Clostridium in Chinese strong flavor Baijiu ecosystem:A review[J].Journal of Food Science, 2018, 83(5):1 193-1 199.
[11] ZHENG Y, HU X L, JIA Z J, et al.Co-occurrence patterns among prokaryotes across an age gradient in pit mud of Chinese strong-flavor liquor[J].Canadian Journal of Microbiology, 2020, 66(9):495-504.
[12] ZHANG Y C, KASTMAN E K, GUASTO J S, et al.Fungal networks shape dynamics of bacterial dispersal and community assembly in cheese rind microbiomes[J].Nature Communications, 2018, 9:336.
[13] BLASCHE S, KIM Y, MARS R A T, et al.Metabolic cooperation and spatiotemporal niche partitioning in a kefir microbial community[J].Nature Microbiology, 2021, 6(2):196-208.
[14] WANG S J, TANG H Z, PENG F, et al.Metabolite-based mutualism enhances hydrogen production in a two-species microbial consortium[J].Communications Biology, 2019, 2:82.
[15] 柴丽娟, 钱玮, 钟小忠, 等.浓香型白酒发酵过程中窖内古菌群落分布特征[J].生物工程学报, 2020, 36(12):2 635-2 643.
CHAI L J, QIAN W, ZHONG X Z, et al.Distribution of archaeal community in the mud pit during strong-flavor Baijiu fermentation[J].Chinese Journal of Biotechnology, 2020, 36(12):2 635-2 643.
[16] DU H, LIU B, WANG X S, et al.Exploring the microbial origins of p-cresol and its co-occurrence pattern in the Chinese liquor-making process[J].International Journal of Food Microbiology, 2017, 260:27-35.
[17] RÖTTJERS L, FAUST K.From hairballs to hypotheses-biological insights from microbial networks[J].FEMS Microbiology Reviews, 2018, 42(6):761-780.
[18] HU X L, DU H, XU Y.Identification and quantification of the caproic acid-producing bacterium Clostridium kluyveri in the fermentation of pit mud used for Chinese strong-aroma type liquor production[J].International Journal of Food Microbiology, 2015, 214:116-122.
[19] XU J L, SUN L P, XING X, et al.Culturing bacteria from fermentation pit muds of Baijiu with culturomics and amplicon-based metagenomic approaches[J].Frontiers in Microbiology, 2020, 11:1223.
[20] CHAI L J, FANG G Y, XU P X, et al.Novisyntrophococcus fermenticellae gen.nov., sp.nov., isolated from an anaerobic fermentation cellar of Chinese strong-flavour Baijiu[J].International Journal of Systematic and Evolutionary Microbiology, 2021, 71(9):DOI:10.1099/ijsem.0.004991.
[21] KUMAR S, STECHER G, LI M, et al.MEGA X:Molecular evolutionary genetics analysis across computing platforms[J].Molecular Biology and Evolution, 2018, 35(6):1 547-1 549.
[22] FELSENSTEIN J.Confidence limits on phylogenies:An approach using the bootstrap[J].Evolution;International Journal of Organic Evolution, 1985, 39(4):783-791.
[23] SEEMANN T.Prokka:rapid prokaryotic genome annotation[J].Bioinformatics, 2014, 30(14):2 068-2 069.
[24] HUERTA-CEPAS J, FORSLUND K, COELHO L P, et al.Fast genome-wide functional annotation through orthology assignment by eggNOG-mapper[J].Molecular Biology and Evolution, 2017, 34(8):2 115-2 122.
[25] ZHAO Y B, JIA X M, YANG J H, et al.PanGP:A tool for quickly analyzing bacterial pan-genome profile[J].Bioinformatics, 2014, 30(9):1 297-1 299.
[26] CHARUBIN K, PAPOUTSAKIS E T.Direct cell-to-cell exchange of matter in a synthetic Clostridium syntrophy enables CO2 fixation, superior metabolite yields, and an expanded metabolic space[J].Metabolic Engineering, 2019, 52:9-19.
[27] ZOU W, YE G B, ZHANG K Z, et al.Analysis of the core genome and pangenome of Clostridium butyricum[J].Genome, 2021, 64(1):51-61.
[28] ZOU W, YE G B, LIU C J, et al.Comparative genome analysis of Clostridium beijerinckii strains isolated from pit mud of Chinese strong flavor Baijiu ecosystem[J].G3 Genes|Genomes|Genetics, 2021, 11(11):jkab317.
[29] UDAONDO Z, DUQUE E, RAMOS J L.The pangenome of the genus Clostridium[J].Environmental Microbiology, 2017, 19(7):2 588-2 603.
[30] 刘昊鹏, 刘超, 王雯, 等.基于厌氧微生物的碳链延长合成高价值化学品反应机理及研究进展:不同电子供体[J].北京化工大学学报(自然科学版), 2020, 47(5):1-17.
LIU H P, LIU C, WANG W, et al.Advances in understanding the mechanism of chain elongation with anaerobic microbes for the synthesis of high value-added chemicals:The effect of different electron donors[J].Journal of Beijing University of Chemical Technology (Natural Science Edition), 2020, 47(5):1-17.
[31] YAO Y, FU B, HAN D F, et al.Formate-dependent acetogenic utilization of glucose by the fecal acetogen Clostridium bovifaecis[J].Applied and Environmental Microbiology, 2020, 86(23):e01870-e01820.
[32] SAN-VALERO P, ABUBACKAR H N, VEIGA M C, et al.Effect of pH, yeast extract and inorganic carbon on chain elongation for hexanoic acid production[J].Bioresource Technology, 2020, 300:122659.
[33] SAN-VALERO P, FERN NDEZ-NAVEIRA
NDEZ-NAVEIRA  , VEIGA M C, et al.Influence of electron acceptors on hexanoic acid production by Clostridium kluyveri[J].Journal of Environmental Management, 2019, 242:515-521.
, VEIGA M C, et al.Influence of electron acceptors on hexanoic acid production by Clostridium kluyveri[J].Journal of Environmental Management, 2019, 242:515-521.
[34] DUBER A, ZAGRODNIK R, CHWIALKOWSKA J, et al.Evaluation of the feed composition for an effective medium chain carboxylic acid production in an open culture fermentation[J].Science of the Total Environment, 2020, 728:138814.