白酒作为我国传统的蒸馏酒,历史悠久,其作为一种嗜好性饮品,白酒的品质及对健康的影响一直受到广泛关注。风味是影响白酒品质的重要属性之一[1],高级醇作为重要的呈香呈味物质,其含量的多少影响着白酒的品质。从1906年至今,高级醇的研究已经历了110多年的发展。高级醇是含3个及以上碳链骨架的一价醇类的总称[2],主要包括正丙醇、异丁醇、异戊醇、2-甲基丁醇、2-苯乙醇等[3]。不同酒中高级醇的种类和含量各不相同,每种高级醇都具有其独特的呈味特征(表1),共同作用形成各式风味独特的白酒。适量且适宜比例的高级醇可以赋予酒体丰富的口感,但当高级醇超过一定限度,酒体会呈现苦涩、冲辣的现象,产生不愉悦的杂味[4]。
表1 主要高级醇的呈味特征
Table 1 Flavor characteristics of main higher alcohols

高级醇适量时气味过量时气味正丙醇有乙醚气味,类似于乙醇易产生似醚臭、有苦味异丁醇有脂肪香产生苦味、杂醇油臭味异戊醇微带甜苦产生不愉悦苦味2-甲基-1-丁醇略带花香腐败味和汗臭味2-苯乙醇蜜香、玫瑰香有酯类的酸味
此外,研究发现,高级醇在人体内过多、较长时间的停留会导致人的神经系统受到损伤,不利于饮用者的身体健康[5],因此,合理控制白酒中高级醇的含量显得尤为重要。目前国标对白酒中高级醇的含量并没有统一的规定,小曲液态发酵白酒中高级醇的含量一般为600~2 500 mg/L,固态发酵白酒中高级醇的含量一般为500~1 800 mg/L[6]。在白酒酿造过程中,酿酒酵母通过繁殖代谢生成相应的高级醇,随着发酵时间的延长,高级醇可与酸类物质反应生成酯类物质,进一步改善白酒口感,提高酒体的品质。但与此同时,传统白酒发酵工艺存在发酵周期长、酒损大、效率低、成本高等问题,不利于白酒的工业化生产。因此,为缩短发酵周期、降低工业能耗、减少生产成本、提高产品质量、探明酿酒酵母中高级醇的形成机制、合理调控白酒中高级醇的含量具有十分重要的意义。本文对酿酒酵母中高级醇的形成机制、关键调控基因和白酒酿造中适产高级醇酿酒酵母菌株的选育方式进行了归纳阐述,以期为合理控制白酒中高级醇含量,进一步为推动白酒行业的健康发展提供参考。
1 酿酒酵母中高级醇的生成途径
在白酒酿造过程的主发酵时期,酿酒酵母发酵产生的中间代谢产物α-酮酸可经过脱羧、脱氢生成高级醇。根据α-酮酸来源的不同,高级醇的生成途径可分为氨基酸分解代谢途径和糖合成代谢途径。
1.1 氨基酸分解代谢途径
酿酒酵母在生长繁殖过程中,利用发酵原料中游离的氨基酸合成自身所需蛋白质,当氨基酸中的氨基被利用后,残余的α-酮酸会通过脱羧、还原过程生成相应的高级醇[7]。1907年,德国化学家Felix Ehrlich首次提出了该代谢途径,因此也被称作Ehrlich途径[2, 8]。特定的氨基酸会经过分解代谢途径生成相应的高级醇,如缬氨酸、亮氨酸、苯丙氨酸、异亮氨酸可在此途径下经代谢分别生成异丁醇、异戊醇、2-苯乙醇、活性戊醇。
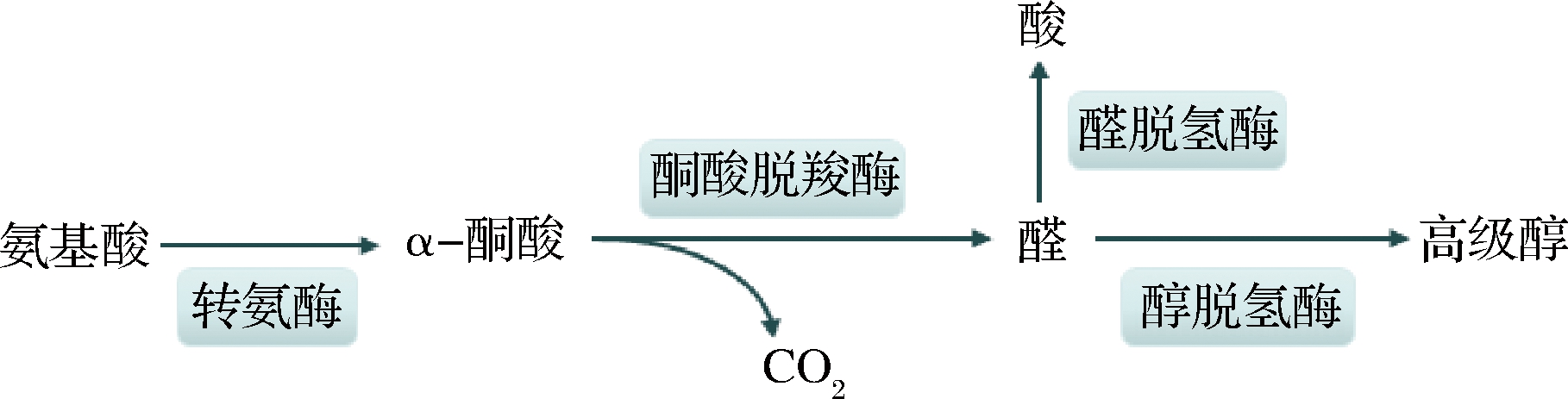
图1 氨基酸分解代谢途径
Fig.1 The amino acids catabolic pathways
1.2 糖合成代谢途径
葡萄糖可通过酿酒酵母的糖酵解途径生成丙酮酸,丙酮酸进一步生成相应的α-酮酸,也可由三羧酸(tricarboxylic acid,TCA)循环途径直接生成相应的α-酮酸,α-酮酸会在相应酶的参与下,脱羧、还原生成相应的高级醇,即为糖合成代谢途径[9]。同时,α-酮酸也可通过转氨作用生成相应的氨基酸,若该过程中氨基供应不足,过量的α-酮酸会进一步生成相应的高级醇。该途径最初由Harris提出,因此也被称作Harris途径。
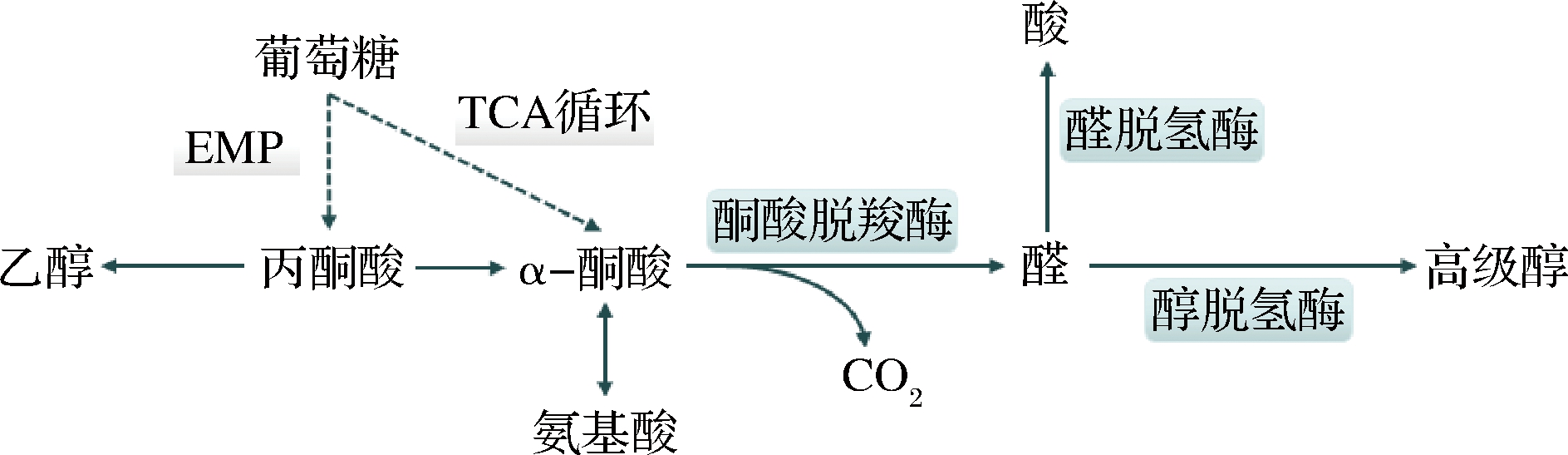
图2 糖合成代谢途径
Fig.2 The carbohydrates biosynthetic pathways
在白酒发酵早期,发酵液中氮源充足,酿酒酵母通过氨基酸分解代谢途径生成的高级醇明显增加。随着发酵的进行,环境中游离氨基酸的含量逐渐减少,此时,酿酒酵母会利用糖合成代谢途径生成自身所需氨基酸;若环境中氮源不足,酿酒酵母形成的α-酮酸则无法顺利合成氨基酸,导致大量α-酮酸积累,此时,糖合成代谢途径会被激活[10],致使过量的α-酮酸经脱羧、还原生成相应的高级醇。在白酒生产过程中,正丙醇、异戊醇、异丁醇和活性戊醇等主要在糖代谢生成氨基酸的过程中产生,2-苯乙醇、酪醇、色醇等高级醇则主要来自氨基酸分解代谢途径中相应氨基酸的降解代谢。研究发现,高级醇中异戊醇、异丁醇和活性戊醇的含量有75%来自糖代谢合成途径,25%来自亮氨酸、缬氨酸、异亮氨酸参与的氨基酸分解代谢途径[11]。
2 酿酒酵母中高级醇代谢调控关键基因
利用酿酒酵母生产饮料酒的方式已有八千多年的历史[12],同时高级醇的研究也经历了百余年的发展,人们对酿酒酵母中高级醇代谢调控机制的研究正不断深入。高级醇的形成受多种基因调控,各基因独立且相互联系,共同调控高级醇的生成(图3)。现对高级醇代谢调控关键基因的研究多集中在氨基酸代谢相关基因、α-酮酸合成相关基因、α-酮酸分解相关基因及乙酸酯代谢相关基因中。
2.1 氨基酸代谢相关基因
氨基酸是影响高级醇生成的重要因素,其中,氨基酸渗透酶和转氨酶对酿酒酵母氨基酸的代谢至关重要,相关基因的表达水平影响着酿酒酵母对氨基酸的代谢能力。
在酿酒酵母中已发现16种氨基酸渗透酶[13],根据酿酒酵母自身繁殖代谢的需要,氨基酸渗透酶可将底物输送到细胞中,参与相关的代谢过程,其中,BAP2(编码支链氨基酸渗透酶)和GAP1(编码氨基酸转运蛋白)基因的研究更为深入。BAP2基因编码的支链氨基酸渗透酶参与亮氨酸、异亮氨酸和缬氨酸3种支链氨基酸的转运,其表达水平影响着酿酒酵母对这3种氨基酸的摄取量。有研究表明,BAP2基因的缺失会导致亮氨酸、异亮氨酸和缬氨酸的摄取量减少20%~50%[14]。同时,BAP2基因表现出一定的温度依赖性,2001年,KODAMA等[15]研究了酿酒酵母BH-225中BAP2基因对高级醇生成量的影响,发现升高温度可促进BAP2基因的表达,进而提高缬氨酸、亮氨酸和异亮氨酸的吸收率。另外,环境中可利用氨基酸含量的变化诱导BAP2基因的转录,研究发现,微摩尔量的亮氨酸是调控发酵环境中支链氨基酸吸收的主要信号[16]。通过控制BAP2基因的表达以及环境中温度和可利用氨基酸的含量,可实现对酿酒酵母氨基酸利用率的调控,进而影响高级醇的生成量。另外,GAP1基因编码的氨基酸转运蛋白Gap1可快速激活培养基诱导的发酵生长(fermentable-growth-medium-induced,FGM)信号通路,控制蛋白激酶A(protein kinase A,PKA)靶点,研究发现,Gap1可将原料中常见的L型和D型氨基酸转运到酵母细胞中,进而提供酿酒酵母生长代谢所需的氨基酸[17]。酿酒酵母对氨基酸的利用能力影响着高级醇的最终生成量,同时,氨基酸的积累也会影响酿酒酵母中氨基酸的分解代谢途径,最终影响高级醇的形成[18],而当发酵环境中缺乏氨基酸时,酿酒酵母将利用糖代谢产生的酮酸合成自身所需氨基酸,同时影响乙醇、高级醇、酯和其他风味物质的生成[19]。

图3 酿酒酵母中高级醇的代谢途径
Fig.3 The higher alcohols metabolic pathway in Saccharomyces cerevisiaeBAP2-支链氨基酸渗透酶编码基因;BAT1、BAT2-支链氨基酸转氨酶编码基因;LEU1-异丙基苹果酸合成酶编码基因;LEU2-β-丙基苹果酸脱氢酶编码基因;ILV1-苏氨酸脱氨酶编码基因;ILV2-乙酰乳酸合成酶编码基因;ILV3-二羟酸脱水酶编码基因;ILV5-乙酰羟基酸还原异构酶和mtDNA结合蛋白编码基因;ILV6-乙酰乳酸合成酶调节亚基编码基因;PDCs、THI3、ARO10-脱羧酶编码基因;ADHs-乙醇脱氢酶编码基因;SFA1-双功能醇脱氢酶和甲醛脱氢酶编码基因;ALDs-醛脱氢酶编码基因;ATF1、ATF2-乙醇乙酰基转移酶编码基因。
蓝色箭头代表糖合成代谢途径,红色箭头代表氨基酸分解代谢途径。
转氨酶是Ehrlich途径中的第一步反应酶,BAT1、BAT2、ARO8和ARO9基因的表达水平影响着氨基酸脱去氨基生成α-酮酸的过程。BAT1和BAT2基因编码的支链氨基酸转氨酶Bat1p和Bat2p分别位于线粒体和细胞质中,催化支链氨基酸和α-酮酸之间氨基的转移[20]。2002年,YOSHIMOTO等[21]通过对BAT2基因研究发现,与野生型相比,BAT2基因缺失突变体KY1058发酵后所生成的异戊醇和异丁醇的含量分别减少了40%和72%。ARO8和ARO9基因编码的芳香族氨基酸转氨酶促进芳香族氨基酸的利用,ARO8基因编码的芳香族转氨酶I负责苯丙氨酸和酪氨酸的生物合成,其表达水平影响酿酒酵母中苯丙氨酸和酪氨酸的形成[22],ARO9基因编码的芳香族转氨酶II主要参与色氨酸的降解。相关研究发现,过量表达ARO8或敲除ARO8基因,2-苯乙醇的产量均有所提升[23],这可能是由于ARO8基因主要参与苯丙氨酸的脱氨反应[24],过量表达可提高2-苯乙醇的合成水平,另外,由于各基因间的相互协调作用,使得敲除ARO8基因后诱导了ARO9基因的表达[22],进而提高了2-苯乙醇的合成量。有研究证实,敲除酿酒酵母单倍体菌株的ARO8基因,可有效提高酵母菌株2-苯乙醇合成水平,在此基础上过量表达ARO10(编码脱羧酶)基因,2-苯乙醇的生成量可提高约13倍[25]。BAT1和BAT2基因的敲除阻碍了酿酒酵母对支链氨基酸中氨基的利用,导致高级醇生成量显著降低;ARO8和ARO9基因可协同作用来调控酿酒酵母中2-苯乙醇的合成量。
2.2 α-酮酸合成相关基因
α-酮酸是酿酒酵母合成高级醇的关键中间代谢产物,LEUs和ILVs基因的表达水平影响着α-酮酸的合成过程。LEUs基因主要参与亮氨酸的合成[26],ILVs基因主要参与缬氨酸及异亮氨酸的合成[27],通过调控α-酮酸合成代谢途径的关键基因及调控因子的表达,可有效控制高级醇的生成量。2011年,佐一含等[28]敲除酿酒酵母S-6中的LEU2(编码β-丙基苹果酸脱氢酶)基因后,将改造菌株与出发菌株一同进行发酵实验来测定高级醇的生成量,结果发现,与出发菌株相比,改造菌株发酵液中高级醇的总含量降低了9.97%,发酵性能并无明显变化。2013年,AVALOS等[29]同时过量表达酿酒酵母JAy1中的ILV2(编码乙酰乳酸合成酶)、ILV5(编码乙酰羟基酸还原异构酶)、ILV3(编码二羟酸脱水酶)基因,结果显示,与初始菌株相比,高级醇生成量提高了约2.3倍,其中异丁醇生成量提高3.8倍,提升幅度最大。LEUs和ILVs基因的表达水平皆可显著影响酿酒酵母高级醇的合成能力,从而有效实现高级醇的合理化调控。
2.3 α-酮酸分解相关基因
α-酮酸在脱羧酶的作用下,脱羧形成醛类物质并产生CO2,形成的醛类物质可在醇脱氢酶的作用下进一步还原生成相应的高级醇。与该过程相关的脱羧酶编码基因主要有PDC1、PDC5、PDC6、ARO10,YDL080c等,催化α-酮酸的脱羧过程,ADHs、SFA1基因编码的醇脱氢酶进一步促进高级醇的生成。醛类物质也可以在ALDs基因编码的醛脱氢酶的作用下脱氢生成相应的酸(图3)。2012年,KONDO等[30]构建了ADH6(编码NADPH依赖性中链醇脱氢酶)过表达菌株YTD16,与出发菌株相比,该菌株的异丁醇提高了2.6倍,为进一步改变乙醇向异丁醇生物合成的碳通量方向,实验者在PDC1(编码丙酮酸脱羧酶)基因缺失菌株的基础上,构建ADH6、ILV2基因过表达菌株YTD306,与YTD106相比,YTD306的异丁醇产量高出1.8倍,且乙醇生成量明显降低。2020年,ZHENG等[31]以酿酒酵母HJ-1为出发菌株构建了ALD6(编码醛脱氢酶)过表达菌株,结果表明,改造菌株的高级醇生成量下降39%以上。上述研究发现,改变α-酮酸分解相关基因的表达水平可调控高级醇的代谢通路,相关数据可为酿酒酵母菌株的高级醇合理化调控提供理论参考。
2.4 乙酸酯代谢相关基因
高级醇一般在白酒酿造过程中的3~7天内形成,随着发酵的进行,其绝大部分仍以高级醇的形式存在,而少部分会和乙酰辅酶A反应生成相应的乙酸酯类化合物,进一步改善白酒的品质。ATF1、ATF2、Lg-ATF1基因负责编码的相应乙醇乙酰基转移酶(Atf1p、Atf2p、Lg-Atf1p)可促进上述催化反应的进行。在发酵过程中,Atf1p和Atf2p参与乙酸乙酯和乙酸异酰胺的生成[32],为研究ATF1和ATF2基因对高级醇及酯类物质含量的影响,2003年,VERSTREPEN等[33]对酿酒酵母BY4742进行ATF1和ATF2基因的敲除,发现ATF1基因的缺失使得菌株的乙酸乙酯生成量降低37.5%,乙酸异戊酯的生成量减少83.9%,但高级醇生成总量并无明显变化;敲除ATF2基因后,乙酸乙酯生成量和乙酸异戊酯生成量分别减少12.5%、17.5%,高级醇总量无显著变化。与其他乙醇乙酰基转移酶相比,Atf1p活性最强,其对高级醇及乙酸酯的生成量影响最大[34],同时,ATF1、ATF2等基因的表达对乙酸酯生成量的影响更为显著,对高级醇生成量的影响存在不确定性。此外,IAH1基因编码的乙酸异戊酯水解酯酶可通过水解酯类物质进而生成相应的醇[35-36]。
3 白酒酿造中适产高级醇酿酒酵母菌株的选育
在酿造过程中,原料和发酵工艺的改良是调控酒中高级醇含量的一种快捷、简便的方法,但由于原料的差异性及发酵环境的复杂性,使得简单通过改善工艺来控制高级醇的含量变得更加困难。白酒发酵液中的高级醇主要由酿酒酵母代谢生成[37],通过调控酿酒酵母中高级醇的代谢途径,选育优良的酿酒酵母,可为高级醇的有效控制提供参考方向。目前,在白酒酿造中,选育适产高级醇酿酒酵母菌株的方式主要有诱变育种和基因工程育种。
3.1 诱变育种
诱变育种是通过物理、化学等因素对实验菌株进行诱变,筛选、鉴定并最终选育出符合预期的优良菌株的方法,其中高通量筛选是诱变育种选育优良菌株的关键。常见的酿酒酵母诱变育种方法有紫外诱变、微波诱变、室温常压等离子体等,目前已有学者通过诱变的方式实现了白酒酵母高级醇合成的有效调控。
在诱变育种中,离子注入诱变是一种新型的复合诱变方法,具有生理损伤小、突变频率高而广的特点。2008年,王鹏银等[38]通过低能氮离子注入诱变法获得一株具有亮氨酸营养缺陷特性的酿酒酵母AY-15突变株,利用玉米糖化醪进行模拟液态白酒发酵实验,并测定发酵液中高级醇的最终生成量,结果表明,与亲本菌株相比,该突变株的异戊醇生成量降低39.85%,高级醇总含量降低33.6%,且发酵性能无明显变化。室温常压等离子体诱变具有突变率高、处理温度较低等优点,非常适合酿酒酵母菌株的诱变。2015年,王国正等[39]对从酒曲中筛选得到的酵母菌株CF4进行常温常压等离子体诱变,筛选得到一株低产高级醇的酿酒酵母菌株ARTP5,通过模拟发酵测定高级醇生成量,结果表明,与亲本菌株CF4相比,诱变菌株的高级醇产量降低20.0%。诱变育种可以在短时间内提高菌株突变率,进而筛选获得适产高级醇的酿酒酵母。
3.2 基因工程育种
利用基因工程育种的方法对酿酒酵母合成高级醇的关键基因进行定向改造,可有效实现高级醇含量的调控。
3.2.1 氨基酸代谢相关基因的调控
为探究氨基酸代谢相关基因的表达对白酒高级醇生成量的影响,2013年,张翠英等[40]利用低产高级醇酿酒酵母工程菌株AYΔbat2进行小曲半固态白酒发酵实验。结果发现,与纯小曲发酵相比,工程菌株发酵得到的正丙醇、异丁醇和异戊醇含量分别降低20.6%、32.3%、50.8%,高级醇总量下降37.2%,BAT2基因的敲除可显著降低半固态白酒发酵中酿酒酵母高级醇的生成量。2017年,LI等[41]以α5为出发菌株构建BAT2缺失菌株,通过模拟白酒发酵进一步测定高级醇合成量,研究发现,与α5相比,BAT2缺失菌株的高级醇总量降低了22.01%。2020年,WANG等[42]为探究GDH1(编码谷氨酸脱氢酶)、CAN1(编码精氨酸渗透酶)、GAT1(编码氮分解抑制机制的转录调节蛋白)基因的敲除对白酒高级醇含量的影响,以酿酒酵母α5为出发菌株,进行模拟白酒发酵实验,通过对比出发菌株及改造菌株的高级醇变化,发现α5Δgdh1菌株的高级醇含量为348.68 mg/L,降低了27.31%,α5Δcan1和α5Δgat1菌株的高级醇含量则分别提高211.44%和28.36%,实验结果表明,氨基酸代谢对酿酒酵母高级醇的生成具有重要意义。
3.2.2 α-酮酸代谢相关基因的调控
在白酒高级醇的含量调控中,α-酮酸代谢相关基因起着重要作用。为研究LEU1(编码异丙基苹果酸合成酶)基因对白酒酿造过程中高级醇含量的影响,2015年,石钰等[43]以酿酒酵母AY15单倍体A8为出发菌株,利用醋酸锂转化法和同源重组技术得到LEU1基因缺失突变株A-L9,随后对出发菌株及突变菌株分别进行模拟酒精发酵和玉米糖化醪(添加30 mg/L亮氨酸)发酵实验,测定最终高级醇的生成量,结果发现,与出发菌株相比,2种发酵条件下A-L9的正丙醇生成量分别提高18%、47%,异丁醇的生成量分别提高52%、158%,异戊醇的生成量则分别降低29%、51%,2种发酵方式均可在提高正丙醇、异丁醇的同时,降低异戊醇生成量,且突变株A-L9发酵性能无明显变化。2018年,LI等[44]以酿酒酵母AY15 CICC 32315为出发菌株进行了ILV1、LEU1、LEU2基因的敲除,并通过白酒模拟发酵实验对各菌株的高级醇最终生成量进行对比,与亲本二倍体菌株相比,ILV1双等位基因缺失菌株生成的正丙醇、活性戊醇和2-苯乙醇的浓度分别下降了30.33%、35.58%、11.71%,异丁醇和异戊醇产量分别升高326.39%、57.6%;LEU1双等位基因缺失菌株的正丙醇和异丁醇合成量分别升高14.09%、41.72%,异戊醇和2-苯乙醇生成量分别下降33.74%、13.21%;LEU2双等位基因缺失突变体的异丁醇合成量提高52.18%。α-酮酸在高级醇的代谢过程中至关重要,对α-酮酸代谢相关基因的合理调控,可在改变高级醇形成比例的同时,合理调控高级醇的最终生成量。此外,通过调控α-酮酸在线粒体和细胞质中的含量,可在不显著改变高级醇总量的同时,实现各类高级醇合成量的差异化调控,对酒体风味的改善具有积极的指导作用[45],同时,这些研究也为阐明酿酒酵母中α-酮酸的代谢机制提供了理论依据。
3.2.3 乙酸酯代谢相关基因的调控
乙醇乙酰基转移酶催化乙酸酯类化合物的合成,影响高级醇和乙酸酯的最终生成量。2017年,LI等[46]在白酒发酵实验中发现,以酿酒酵母α5为出发菌株,ATF1基因过表达、BAT2基因敲除菌株生成的乙酸乙酯含量是α5的54.03倍,与此同时,与α5相比,该改造菌株的高级醇含量降低了57.02%。此外,2017年,LI等[41]以BAT2敲除突变体为出发菌株进行ATF1、ATF2、Lg-ATF1基因过表达的研究,结果显示,ATF2过表达菌株产生的高级醇与出发菌株相似,Lg-ATF1过表达菌株所产生的高级醇及酯类物质含量与出发菌株无明显差异,而ATF1过表达菌株的高级醇合成量则降低48.17%,ATF1基因的表达水平对高级醇及乙酸酯生成量的影响更为显著,而Lg-ATF1基因的影响程度最小。2018年,CUI等[47]为调节白酒中高级醇及酯的浓度,通过易错PCR构建PGK1启动子库,以BAT2敲除酿酒酵母a45为出发菌株,利用7个不同的PGK1突变启动子进行ATF1的过表达,结果表明所有构建菌株的酯及高级醇含量均明显改变,其中乙酸乙酯的含量提高了1.66~27.22倍,异丁醇含量降低16.11%~30.97%,异戊醇含量降低1.78%~45.95%,2-甲基丁醇含量降低17.2%~42.94%,正丙醇和2-苯乙醇浓度无明显变化。ATF1基因的过表达增加了乙酰辅酶A活性,促进了乙酸酯的生成[48],同时BAT2基因的敲除可显著降低高级醇含量[49],进而有效调控白酒中的醇酯比。2019年,LI等[19]成功构建了BAT2敲除且ATF1过表达的工程菌株,该菌株可有效降低白酒发酵后高级醇的生成量,提高酯类物质的合成水平,进而更贴合工业生产的需求。此外,由于代谢途径中涉及多个基因的相互作用及制约,因此高级醇关键基因的调控存在一定的复杂性及不确定性。2010年,郝欣等[50]在探究THI3基因缺失对白酒酿酒酵母AY-15的a型和α型单倍体菌株中高级醇生成水平的影响时,发现其最终高级醇的合成能力均未发生明显改变。
表2 不同类型基因对酿酒酵母菌株高级醇的影响
Table 2 Effects of different types of genes on higher alcohols in S.cerevisiae
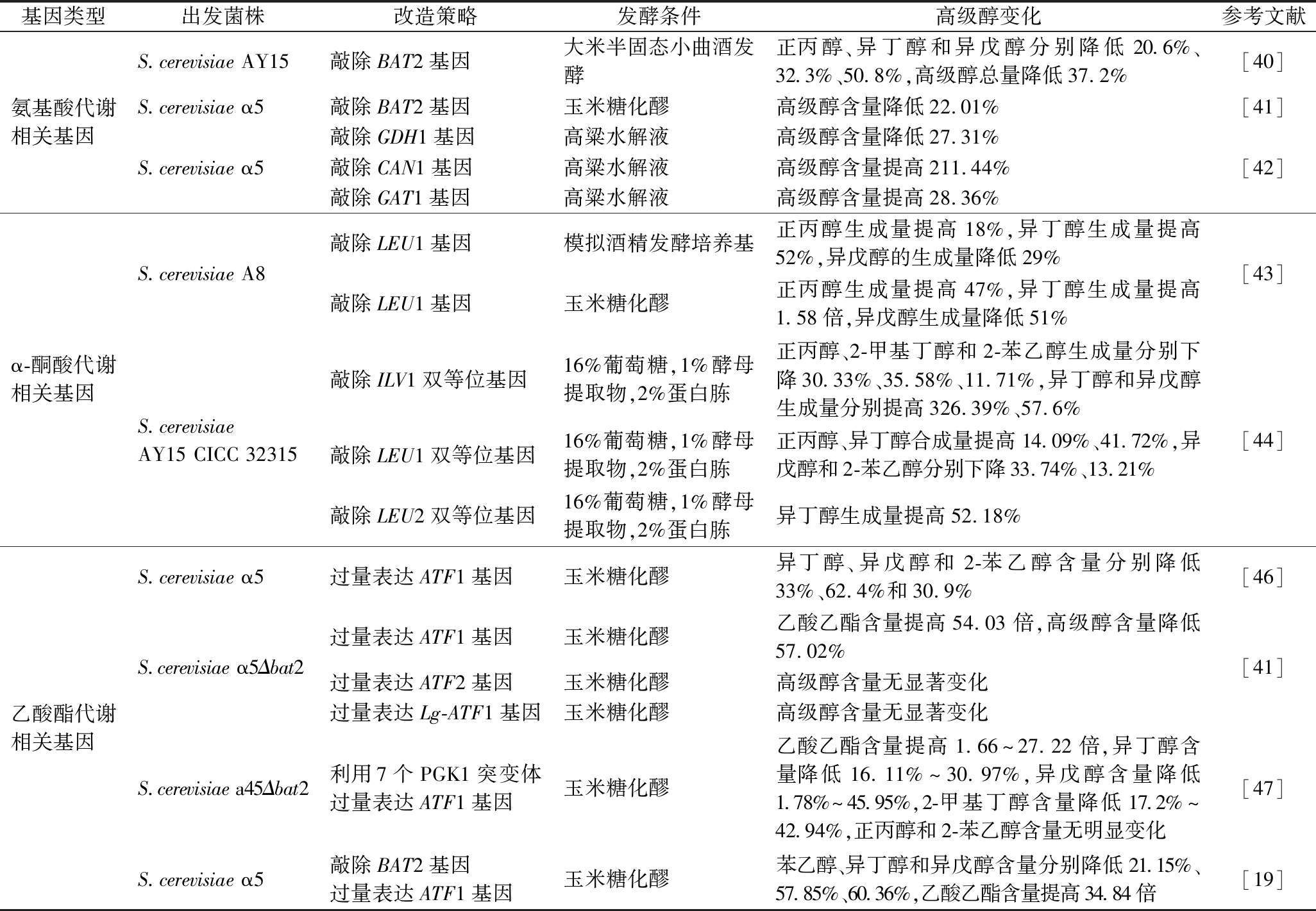
基因类型出发菌株改造策略发酵条件高级醇变化参考文献氨基酸代谢相关基因S.cerevisiae AY15S.cerevisiae α5S.cerevisiae α5敲除BAT2基因大米半固态小曲酒发酵正丙醇、异丁醇和异戊醇分别降低20.6%、32.3%、50.8%,高级醇总量降低37.2%敲除BAT2基因玉米糖化醪高级醇含量降低22.01%敲除GDH1基因高粱水解液高级醇含量降低27.31%敲除CAN1基因高粱水解液高级醇含量提高211.44%敲除GAT1基因高粱水解液高级醇含量提高28.36%[40][41][42]α-酮酸代谢相关基因S.cerevisiae A8S.cerevisiae AY15 CICC 32315敲除LEU1基因模拟酒精发酵培养基正丙醇生成量提高18%,异丁醇生成量提高52%,异戊醇的生成量降低29%敲除LEU1基因玉米糖化醪正丙醇生成量提高47%,异丁醇生成量提高1.58倍,异戊醇生成量降低51%敲除ILV1双等位基因16%葡萄糖,1%酵母提取物,2%蛋白胨正丙醇、2-甲基丁醇和2-苯乙醇生成量分别下降30.33%、35.58%、11.71%,异丁醇和异戊醇生成量分别提高326.39%、57.6%敲除LEU1双等位基因16%葡萄糖,1%酵母提取物,2%蛋白胨正丙醇、异丁醇合成量提高14.09%、41.72%,异戊醇和2-苯乙醇分别下降33.74%、13.21%敲除LEU2双等位基因16%葡萄糖,1%酵母提取物,2%蛋白胨异丁醇生成量提高52.18%[43][44]乙酸酯代谢相关基因S.cerevisiae α5S.cerevisiae α5Δbat2S.cerevisiae a45Δbat2S.cerevisiae α5过量表达ATF1基因玉米糖化醪异丁醇、异戊醇和2-苯乙醇含量分别降低33%、62.4%和30.9%过量表达ATF1基因玉米糖化醪乙酸乙酯含量提高54.03倍,高级醇含量降低57.02%过量表达ATF2基因玉米糖化醪高级醇含量无显著变化过量表达Lg-ATF1基因玉米糖化醪高级醇含量无显著变化利用7个PGK1突变体过量表达ATF1基因玉米糖化醪乙酸乙酯含量提高1.66~27.22倍,异丁醇含量降低16.11%~30.97%,异戊醇含量降低1.78%~45.95%,2-甲基丁醇含量降低17.2%~42.94%,正丙醇和2-苯乙醇含量无明显变化敲除BAT2基因过量表达ATF1基因玉米糖化醪苯乙醇、异丁醇和异戊醇含量分别降低21.15%、57.85%、60.36%,乙酸乙酯含量提高34.84倍[46][41][47][19]
4 总结和展望
白酒中的高级醇主要由酿酒酵母生成,利用诱变育种和基因工程育种的方式调控酿酒酵母中高级醇的代谢途径,选育适产高级醇的酿酒酵母菌株对改善白酒的酒体和风味具有重要意义。目前,研究者们已利用诱变育种筛选出了大量适产高级醇的优质酿酒酵母。但同时诱变育种也存在突变方向难控制、正向突变率低等问题,因此,提高诱变育种效率、探索定向诱变育种技术以及将诱变育种技术与其他生物技术相结合筛选适产高级醇酿酒酵母是未来研究的重要方向。利用基因工程育种可实现酿酒酵母中高级醇的定向调控,研究者们通过基因工程育种技术对高级醇代谢关键基因进行深入研究已经取得了显著的成果,如对BAT2、ILVs、LEUs、ATF1等基因的定向改造,在保证酿酒酵母发酵性能不发生明显变化的同时,实现了高级醇含量的有效控制。同时,无痕改造技术使通过基因工程育种获得的工程菌株中不包含任何外源DNA片段,降低了安全隐患,相信未来基因工程菌株将逐步应用于白酒的工业化生产中。预测未来高级醇代谢相关基因的定向改造首先是更精细、全面地研究各基因对高级醇代谢的影响;其次是深度挖掘高级醇代谢网络中各基因之间的互作关系,再者是在选育适产高级醇酿酒酵母菌株的同时,需充分考虑白酒整体的风味和口感,保证各风味物质之间的协调,这才能进一步为推动白酒行业的健康发展提供参考。
[1] 孙宝国, 黄明泉, 王娟.白酒风味化学与健康功效研究进展[J].中国食品学报, 2021, 21(5):1-13.
SUN B G, HUANG M Q, WANG J.Research progress on flavor chemistry and healthy function of Baijiu[J].Journal of Chinese Institute of Food Science and Technology, 2021, 21(5):1-13.
[2] PIRES E J, TEIXEIRA J A, BR NYIK T, et al.Yeast:The soul of beer’s aroma--A review of flavour-active esters and higher alcohols produced by the brewing yeast[J].Applied Microbiology and Biotechnology, 2014, 98(5):1 937-1 949.
NYIK T, et al.Yeast:The soul of beer’s aroma--A review of flavour-active esters and higher alcohols produced by the brewing yeast[J].Applied Microbiology and Biotechnology, 2014, 98(5):1 937-1 949.
[3] 苟静瑜, 贾智勇, 闫宗科, 等.降低白酒中高级醇含量的研究进展[J].酿酒, 2016, 43(4):25-29.
GOU J Y, JIA Z Y, YAN Z K, et al.Research progress in decreasing the contents of higher alcohols in Baijiu(liquor)[J].Liquor Making, 2016, 43(4):25-29.
[4] LONGO R, CAREW A, SAWYER S, et al.A review on the aroma composition of Vitis vinifera L.Pinot noir wines:Origins and influencing factors[J].Critical Reviews in Food Science and Nutrition, 2021, 61(10):1 589-1 604.
[5] FANG C, DU H, JIA W, et al.Compositional differences and similarities between typical Chinese Baijiu and western liquor as revealed by mass spectrometry-based metabolomics[J].Metabolites, 2018, 9(1):2.
[6] 孙中贯, 刘琳, 王亚平, 等.酿酒酵母高级醇代谢研究进展[J].生物工程学报, 2021, 37(2):429-447.
SUN Z G, LIU L, WANG Y P, et al.Higher alcohols metabolism by Saccharomyces cerevisiae:A mini review[J].Chinese Journal of Biotechnology, 2021, 37(2):429-447.
[7] EL-DALATONY M M, SAHA S, GOVINDWAR S P, et al.Biological conversion of amino acids to higher alcohols[J].Trends in Biotechnology, 2019, 37(8):855-869.
[8] CHOI Y J, LEE J, JANG Y S, et al.Metabolic engineering of microorganisms for the production of higher alcohols[J].mBio, 2014, 5(5):e01524-e01514.
[9] CHEN E C H.The relative contribution of Ehrlich and biosynthetic pathways to the formation of fusel alcohols[J].Journal of the American Society of Brewing Chemists, 1978, 36(1):39-43.
[10] LEI H J, ZHENG L Y, WANG C X, et al.Effects of worts treated with proteases on the assimilation of free amino acids and fermentation performance of lager yeast[J].International Journal of Food Microbiology, 2013, 161(2):76-83.
[11] 秦伟帅. 葡萄酒酵母遗传操作构建高级醇低产菌株的研究[D].泰安:山东农业大学, 2013.
QIN W S.Construction of wine yeast strain with low-yield higher alcohols by genetic manipulation[D].Taian:Shandong Agricultural University, 2013.
[12] MCGOVERN P E, ZHANG J Z, TANG J G, et al.Fermented beverages of pre- and proto-historic China[J].Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(51):17 593-17 598.
[13] KUMARI S, KUMAR M, GAUR N A, et al.Multiple roles of ABC transporters in yeast[J].Fungal Genetics and Biology, 2021, 150:103550.
[14] GRAUSLUND M, DIDION T, KIELLAND-BRANDT M C, et al.BAP2, a gene encoding a permease for branched-chain amino acids in Saccharomyces cerevisiae[J].Biochimica et Biophysica Acta, 1995, 1269(3):275-280.
[15] KODAMA Y, OMURA F, MIYAJIMA K, et al.Control of higher alcohol production by manipulation of the BAP2 gene in brewing yeast[J].Journal of the American Society of Brewing Chemists, 2001, 59(4):157-162.
[16] DIDION T, GRAUSLUND M, KIELLAND-BRANDT M C, et al.Amino acids induce expression of BAP2, a branched-chain amino acid permease gene in Saccharomyces cerevisiae[J].Journal of Bacteriology, 1996, 178(7):2 025-2 029.
[17] DONATON M C V, HOLSBEEKS I, LAGATIE O, et al.The Gap1 general amino acid permease acts as an amino acid sensor for activation of protein kinase A targets in the yeast Saccharomyces cerevisiae[J].Molecular Microbiology, 2003, 50(3):911-929.
[18] SAIKI R K, SCHARF S, FALOONA F, et al.Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell Anemia[J].Science, 1985, 230(4732):1 350-1 354.
[19] LI P, GE J L, GAO Y Y, et al.A seamless gene deletion method and its application for regulation of higher alcohols and ester in Baijiu Saccharomyces cerevisiae[J].Biomed Research International, 2019:6723849.
[20] COL N M, HERN
N M, HERN NDEZ F, L
NDEZ F, L PEZ K, et al.Saccharomyces cerevisiae Bat1 and Bat2 aminotransferases have functionally diverged from the ancestral-like Kluyveromyces lactis orthologous enzyme[J].PLoS One, 2011, 6(1):e16099.
PEZ K, et al.Saccharomyces cerevisiae Bat1 and Bat2 aminotransferases have functionally diverged from the ancestral-like Kluyveromyces lactis orthologous enzyme[J].PLoS One, 2011, 6(1):e16099.
[21] YOSHIMOTO H, FUKUSHIGE T, YONEZAWA T, et al.Genetic and physiological analysis of branched-chain alcohols and isoamyl acetate production in Saccharomyces cerevisiae[J].Applied Microbiology and Biotechnology, 2002, 59(4-5):501-508.
[22] URRESTARAZU A, VISSERS S, IRAQUI I, et al.Phenylalanine- and tyrosine-auxotrophic mutants of Saccharomyces cerevisiae impaired in transamination[J].Molecular and General Genetics, 1998, 257(2):230-237.
[23] YIN S, ZHOU H, XIAO X, et al.Improving 2-phenylethanol production via Ehrlich pathway using genetic engineered Saccharomyces cerevisiae strains[J].Current Microbiology, 2015, 70(5):762-767.
[24] IRAQUI I, VISSERS S, CARTIAUX M, et al.Characterisation of Saccharomyces cerevisiae ARO8 and ARO9 genes encoding aromatic aminotransferases I and II reveals a new aminotransferase subfamily[J].Molecular and General Genetics, 1998, 257(2):238-248.
[25] SHEN L, NISHIMURA Y, MATSUDA F, et al.Overexpressing enzymes of the Ehrlich pathway and deleting genes of the competing pathway in Saccharomyces cerevisiae for increasing 2-phenylethanol production from glucose[J].Journal of Bioscience and Bioengineering, 2016, 122(1):34-39.
[26] BAICHWAL V R, CUNNINGHAM T S, GATZEK P R, et al.Leucine biosynthesis in yeast:Identification of two genes (LEU4, LEU5) that affect α-isopropylmalate synthase activity and evidence that LEU1 and LEU2 gene expression is controlled by α-isopropylmalate and the product of a regulatory gene[J].Current Genetics, 1983, 7(5):369-377.
[27] HOLMBERG S, PETERSEN J G.Regulation of isoleucine-valine biosynthesis in Saccharomyces cerevisiae[J].Current Genetics, 1988, 13(3):207-217.
[28] 佐一含, 朱旭东, 陈叶福, 等.LEU2基因敲除对工业啤酒酵母高级醇生成量的影响[J].中国酿造, 2011, 30(3):27-30.
ZUO Y H, ZHU X D, CHEN Y F, et al.Effect of LEU2 gene knockout on higher alcohols production in industrial Saccharomyces cerevisiae[J].China Brewing, 2011, 30(3):27-30.
[29] AVALOS J L, FINK G R, STEPHANOPOULOS G.Compartmentalization of metabolic pathways in yeast mitochondria improves the production of branched-chain alcohols[J].Nature Biotechnology, 2013, 31(4):335-341.
[30] KONDO T, TEZUKA H, ISHII J, et al.Genetic engineering to enhance the Ehrlich pathway and alter carbon flux for increased isobutanol production from glucose by Saccharomyces cerevisiae[J].Journal of Biotechnology, 2012, 159(1-2):32-37.
[31] ZHENG N, JIANG S, HE Y H, et al.Production of low-alcohol Huangjiu with improved acidity and reduced levels of higher alcohols by fermentation with scarless ALD6 overexpression yeast[J].Food Chemistry, 2020, 321:126691.
[32] THURSTON P A, TAYLOR R, AHVENAINEN J.Effects of linoleic acid supplements on the synthesis by yeast of lipids and acetate esters[J].Journal of the Institute of Brewing, 1981, 87(2):92-95.
[33] VERSTREPEN K J, VAN LAERE S D M, VANDERHAEGEN B M P, et al.Expression levels of the yeast alcohol acetyltransferase genes ATF1, Lg-ATF1, and ATF2 control the formation of a broad range of volatile esters[J].Applied and Environmental Microbiology, 2003, 69(9):5 228-5 237.
[34] SCHREIER P.Flavor composition of wines:A review[J].Critical Reviews in Food Science and Nutrition, 1979, 12(1):59-111.
[35] ZHANG J W, ZHANG C Y, DAI L H, et al.Effects of overexpression of the alcohol acetyltransferase-encoding gene ATF1 and disruption of the esterase-encoding gene IAH1 on the flavour profiles of Chinese yellow rice wine[J].International Journal of Food Science and Technology, 2012, 47(12):2 590-2 596.
[36] DONG J, XU H Y, ZHAO L B, et al.Enhanced acetate ester production of Chinese liquor yeast by overexpressing ATF1 through precise and seamless insertion of PGK1 promoter[J].Journal of Industrial Microbiology and Biotechnology, 2014, 41(12):1 823-1 828.
[37] PROCOPIO S, QIAN F, BECKER T.Function and regulation of yeast genes involved in higher alcohol and ester metabolism during beverage fermentation[J].European Food Research and Technology, 2011, 233(5):721-729.
[38] 王鹏银, 郝欣, 郭学武, 等.离子注入诱变选育低产高级醇酿酒酵母菌株[J].酿酒科技, 2008(2):17-21;26.
WANG P Y, HAO X, GUO X W, et al.Screening of Saccharomyces cerevisiae strains with low yield of higher alcohols by ion implantation[J].Liquor-Making Science & Technology, 2008(2):17-21;26.
[39] 王国正, 吴群, 徐岩.低产高级醇酿酒酵母突变菌株的差异蛋白组分析及高级醇合成相关蛋白的差异表达[J].微生物学通报, 2015, 42(12):2 407-2 416.
WANG G Z, WU Q, XU Y.Comparative intracellular proteomics analysis of a low higher alcohols producing Saccharomyces cerevisiae mutant and different expression of higher alcohols synthesis related proteins[J].Microbiology China, 2015, 42(12):2 407-2 416.
[40] 张翠英, 张艳英, 齐亚楠, 等.低产高级醇酿酒酵母工程菌株在小曲酒酿造中的应用[J].酿酒科技, 2013, (7):62-64.
ZHANG C Y, ZHANG Y Y, QI Y N, et al.Application of a Saccharomyces cerevisiae engineering strain with low-yield of higher alcohols in the production of Xiaoqu liquor[J].Liquor-Making Science&Technology, 2013, (7):62-64.
[41] LI W, CUI D Y, WANG J H, et al.Overexpression of different alcohol acetyltransferase genes with BAT2 deletion in Saccharomyces cerevisiae affects acetate esters and higher alcohols[J].European Food Research and Technology, 2018, 244(3):555-564.
[42] WANG Y P, WEI X Q, GUO X W, et al.Effect of the deletion of genes related to amino acid metabolism on the production of higher alcohols by Saccharomyces cerevisiae[J].BioMed Research International, 2020, 2020:6802512.
[43] 石钰, 陈叶福, 肖冬光.LEU1基因缺失对酿酒酵母高级醇生成量的影响[J].酿酒科技, 2015, (2):12-16.
SHI Y, CHEN Y F, XIAO D G.Effects of LEU1 gene deletion on higher alcohols yield of S.cerevisiae[J].Liquor-Making Science&Technology, 2015, (2):12-16.
[44] LI W, CHEN S J, WANG J H, et al.Genetic engineering to alter carbon flux for various higher alcohol productions by Saccharomyces cerevisiae for Chinese Baijiu fermentation[J].Applied Microbiology and Biotechnology, 2018, 102(4):1 783-1 795.
[45] YUAN J F, MISHRA P, CHING C B.Engineering the leucine biosynthetic pathway for isoamyl alcohol overproduction in Saccharomyces cerevisiae[J].Journal of Industrial Microbiology and Biotechnology, 2017, 44(1):107-117.
[46] LI W, WANG J H, ZHANG C Y, et al.Regulation of Saccharomyces cerevisiae genetic engineering on the production of acetate esters and higher alcohols during Chinese Baijiu fermentation[J].Journal of Industrial Microbiology and Biotechnology, 2017, 44(6):949-960.
[47] CUI D Y, ZHANG Y, XU J, et al.PGK1 promoter library for the regulation of acetate ester production in Saccharomyces cerevisiae during Chinese Baijiu fermentation[J].Journal of Agricultural and Food Chemistry, 2018, 66(28):7 417-7 427.
[48] KRUIS A J, LEVISSON M, MARS A E, et al.Ethyl acetate production by the elusive alcohol acetyltransferase from yeast[J].Metabolic Engineering, 2017, 41:92-101.
[49] MA L J, HUANG S Y, DU L P, et al.Reduced production of higher alcohols by Saccharomyces cerevisiae in red wine fermentation by simultaneously overexpressing BAT1 and deleting BAT2[J].Journal of Agricultural and Food Chemistry, 2017, 65(32):6 936-6 942.
[50] 郝欣, 肖冬光, 张翠英.酿酒酵母类丙酮酸脱羧酶基因缺失对高级醇生成量的影响[J].微生物学报, 2010, 50(8):1 030-1 035.
HAO X, XIAO D G,ZHANG C Y.Effect of YDL080C gene deletion on higher alcohols production in Saccharomyces cerevisiae haploids[J].Acta Microbiologica Sinica, 2010, 50(8):1 030-1 035.