小白链霉菌(Streptomyces albulus)是从土壤中分离得到的一株ε-聚赖氨酸(ε-poly-L-lysine,ε-PL)产生菌,目前它成为ε-PL主要生产菌,其最大合成能力已经突破70 g/L[1]。因ε-PL对革兰氏阳性菌、革兰氏阴性菌、霉菌以及酵母均具有很好的抑制作用,故被日本、韩国、美国和中国批准用于饮料、肉制品、乳制品等食品的防腐与保鲜,是天然食品防腐剂取代化学食品防腐剂的关键品种[2-6]。
pH值是影响小白链霉菌高效合成ε-PL的最关键因素,控制发酵过程pH值保持在4.0左右是实现小白链霉菌大量合成ε-PL的先决条件。然而,链霉菌合成次级代谢产物的最适pH值一般为中性,例如子囊霉素、达托霉素合成的最适pH值为6.5和7.0[7-8]。已有的研究结果显示,低pH值促进S.albulus积累ε-PL主要有两方面原因:一是低pH值环境能有效抑制ε-PL分解酶活性。分离纯化S.albulus的ε-PL分解酶,其降解活性在≤pH 4.0环境下几乎完全丧失[9],而ε-PL合成酶在该低pH环境下依旧能保持约20%的催化活性(最适pH 8.5)[10]。二是低pH环境有利于S.albulus胞内积累ATP。ε-PL合成酶催化活性发挥依赖胞内较高浓度ATP积累,而YAMANAKA等[11]发现在低pH环境下S.albulus NBRC14147胞内ATP浓度明显高于较高pH值。因此,他们认为低pH环境会帮助细胞积累ATP到ε-PL合成酶启动ε-PL合成所需的临界值,进而实现ε-PL合成起始。令人遗憾的是,已有的研究仅关注低pH值对ε-PL合成起始与降解问题,却忽略了小白链霉菌为何能在低pH值条件下进行生长和代谢?以及低pH值的环境胁迫对小白链霉菌造成了何种生理代谢改变?
近年来,随着系统生物学技术发展,微生物响应低pH和酸胁迫的生理机制逐渐被揭示。研究发现,微生物面对低pH和酸胁迫环境时,首先是保持胞内pH值稳态(限制氢质子内流、提高质子泵活性、产生碱性物质消耗氢质子等方式),其次是改变细胞膜组分以增强其流动性,三是改变细胞内代谢,四是产生保护和修复大分子等生理变化。由此可见,低pH或酸胁迫往往会对微生物造成全局性的生理影响。为此,本文借助比较转录组学研究方法,考察S.albulus M-Z18在恒化培养方式下响应低pH胁迫所做出的全局基因转录变化,以期揭示低pH环境胁迫对S.albulus造成的生理代谢影响。
1 材料与方法
1.1 菌种及培养基
S.albulus M-Z18由本实验室保藏。MS固体培养基(g/L):甘露醇20,大豆粉20,琼脂粉20。种子培养基(g/L):葡萄糖 50,酵母粉 5,(NH4)2SO4 10,KH2PO4 1.36,K2HPO4·3H2O 0.8,ZnSO4·7H2O 0.04,MgSO4·7H2O 0.5,FeSO4·7H2O 0.05,pH 6.8。分批发酵培养基(g/L):葡萄糖 60,酵母粉 10,(NH4)2SO4 10,MgSO4·7H2O 0.8,FeSO4·7H2O 0.05,KH2PO4 4,pH 6.8。
1.2 试剂和仪器
TaKaRa MiniBEST Universal RNA Extraction Kit RNA提取试剂盒,宝日医生物技术(北京)有限公司;UV-2100分光光度计,优尼科仪器有限公司;T&J-Miniskid迷你平行生物反应器(1 L×4),迪必尔生物工程(上海)有限公司。
1.3 分批发酵
使用迪必尔1 L生物反应器进行分批发酵。接种量为8%,装液量0.7 L,发酵过程中通过调节通气量和转速使溶氧维持在30%。接种前,分别将温度、通气量以及搅拌转速设为30 ℃,1 m3/(m3·h),300 r/min。随着发酵的进行发酵液pH会自发下降,用体积分数为25%的氨水调节培养基至所需pH。发酵过程中每隔6 h取1次样,测定发酵液中ε-PL浓度、pH、菌体量及残余葡萄糖浓度。
1.4 样品制备
在pH 4.0、5.5环境中对S.albulus M-Z18进行恒化培养,其中具体的实验方法参考本实验室之前的报道[12]。待菌株达到稳定状态时,从发酵罐中取出20 mL发酵液,离心去上清液后用9 g/L KCl洗涤2次,最后用液氮处理,保存于-80 ℃冰箱备用。使用TaKaRa MiniBEST Universal RNA Extraction Kit提取样品总RNA,质检合格RNA样品寄送至华大基因生物有限公司完成转录组的测序。
1.5 分析方法
菌体干重(dry cell weight, DCW)测定:取10 mL发酵液,经10 000 r/min离心8 min后弃去上清液并全部抽滤至滤纸上,于105 ℃烘干12 h。随后,放置在干燥器中冷却称取其质量,通过计算其与滤纸质量之差计算菌体干重。
ε-PL浓度测定:采用甲基橙比色法[13]。
2 结果与分析
2.1 不同pH值对S.albulus M-Z18菌体生长和ε-PL合成的影响
S.albulus M-Z18作为ε-PL生产菌,在摇瓶发酵过程中pH值会从6.8自动下降至3.0左右,极低的pH严重影响菌株的生长。当在发酵罐中控制培养基的pH时,S.albulus M-Z18在pH值3.5以上能很好地维持细胞生长,而且细胞的生物量随着pH值的增加而增加(图1-a)。但ε-PL的合成受到pH值的严格调控,只有在pH≤5时才会积累ε-PL,而且随着pH的降低ε-PL产量逐渐升高(图1-b)。因此,S.albulus M-Z18发酵ε-PL过程需要严格的控制在低pH环境中。
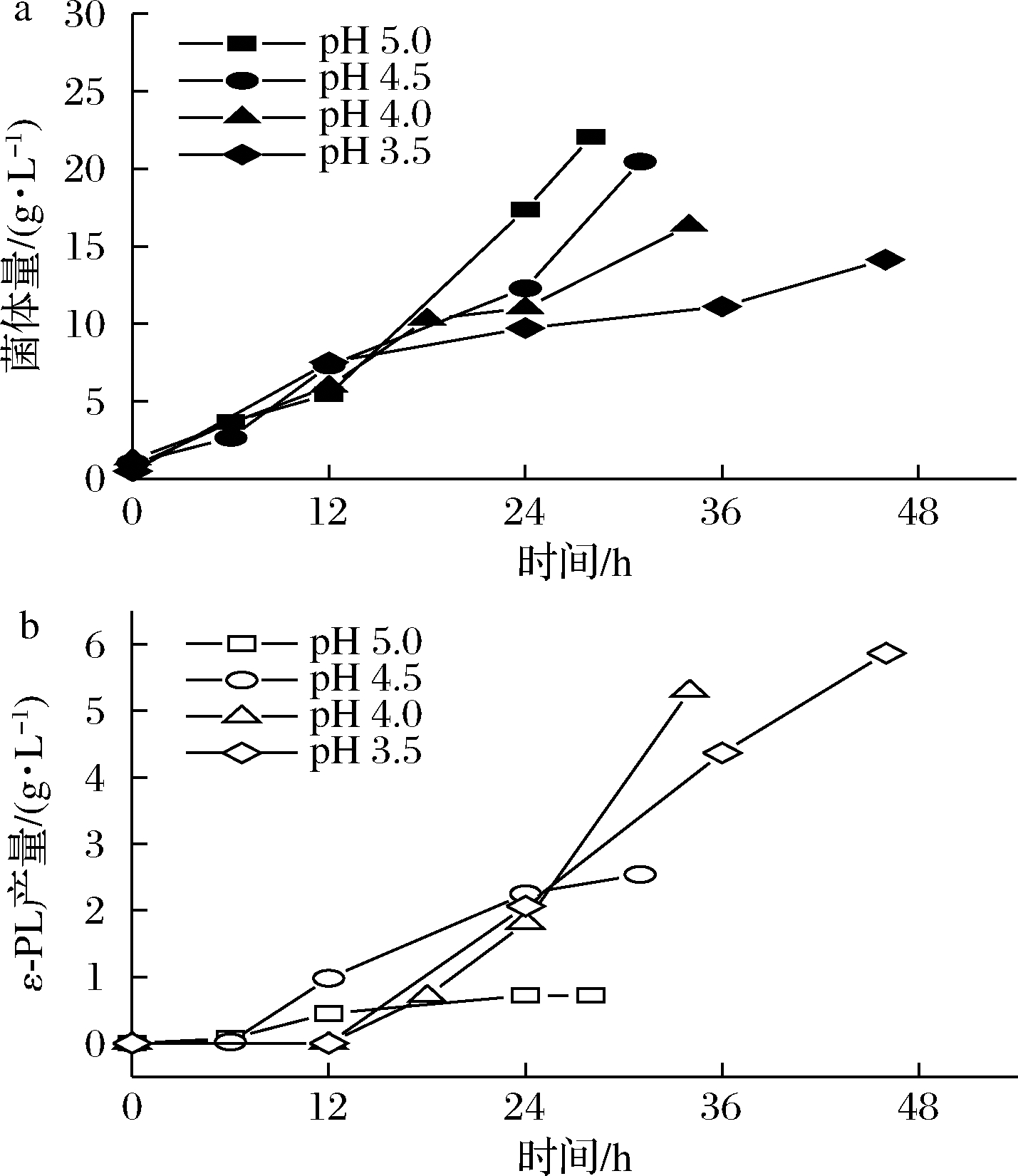
a-菌体量;b-ε-PL产量
图1 不同pH分批发酵下S.albulus M-Z18的菌体量和ε-PL浓度
Fig.1 Biomass and ε-PL concentration of S.albulus M-Z18 under batch fermentation at different pH
2.2 低pH胁迫条件下S.albulus M-Z18基因表达差异与分析
本研究通过恒化培养控制S.albulus M-Z18在不同pH值(5.5、4.0)条件下达到稳态[溶解氧(dissolved oxygen,DO)、DCW、ε-PL浓度、残余葡萄糖浓度等基本不变]。这将消除菌株在不同pH值(5.5、4.0)条件下的生长差异,更好的比较菌株之间生理差异。取稳态时的样品提取RNA进行全基因转录组测序及分析。
以Q<0.05且基因差异表达量倍数在2倍以上为筛选条件,筛选显著差异表达基因。结果如图2所示,共有3 893个基因显著差异表达,其中1 786 个上调显著基因,2 107个下调显著基因,显著差异基因数目占检测到基因数目的53.97%,说明S.albulus M-Z18基因表达发生了显著的变化以应对低pH胁迫。
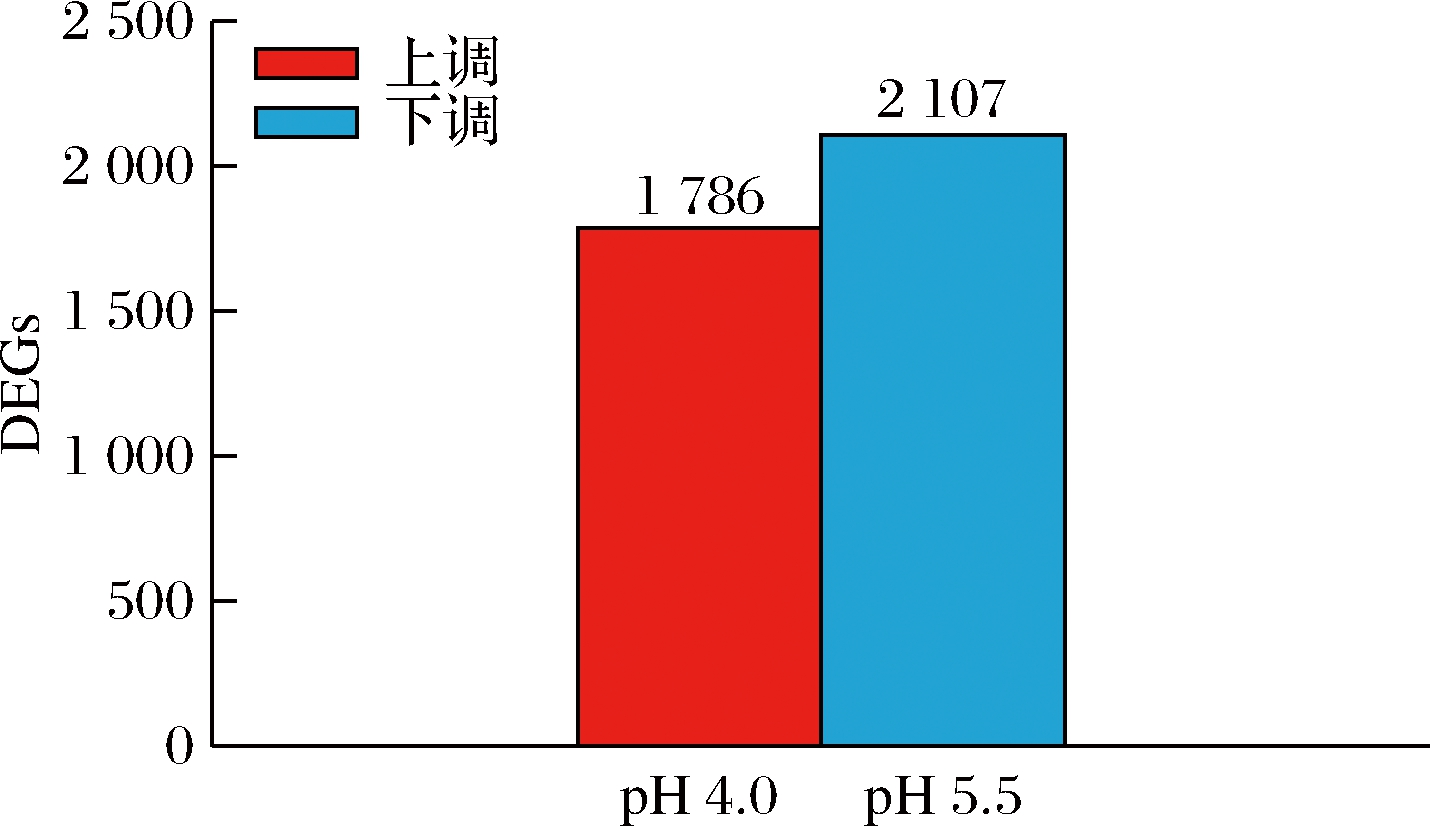
图2 显著差异表达基因数目统计图
Fig.2 Statistic of significantly differently expressed genes
pH 5.5和pH 4.0两个样本的GO功能富集分析结果如图3所示,差异表达基因主要集中于生物过程(biological process),此外分子功能(molecular function)和细胞成分(cellular components)也富集了大量的差异表达基因。生物过程显著富集的条目是代谢过程(metabolic process)、细胞过程(cellular process)和单生物代谢过程(single-orgnism process)。对pH 5.5和pH 4.0两个样本进行KEGG富集分析。由图4可知差异基因主要集中于代谢(metabolism)这一大类中,其中多数基因注释到碳代谢(carbohydrate metabolism)、氨基酸代谢(amino acid metabolism)和萜类化合物和聚酮化合物的代谢(metabolism of terpenoids and polyketides)通路上。此外,跨膜运输(membrane transport)和信号转导(signal transduction)通路中多数基因也发生了显著差异表达。总之,S.albulus M-Z18的代谢、物质转运和信息交互在低pH胁迫环境中发生了显著的差异变化。对这些代谢通路展开具体分析以探究S.albulus M-Z18响应酸胁迫的具体分子机制。
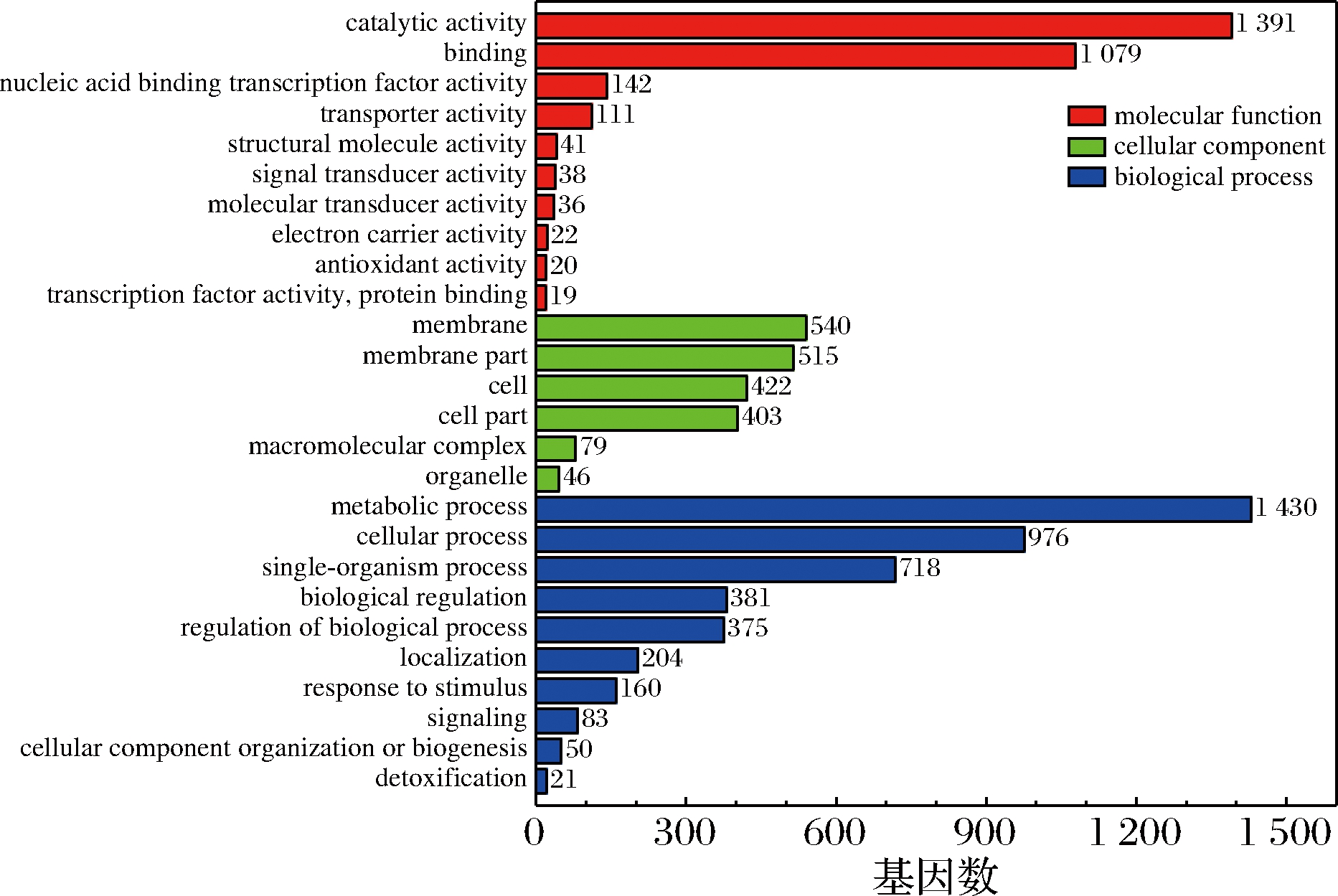
图3 显著差异表达基因的GO功能分类图
Fig.3 GO functional classification of significantly differentially expressed gene

图4 显著差异表达基因KEGG Pathway分类图
Fig.4 KEGG pathway classification of significantly differentially expressed genes
2.3 S.albulus M-Z18响应低pH胁迫生理机制
2.3.1 糖代谢途径
糖代谢过程主要包括糖酵解途径、三羧酸循环和戊糖磷酸途径。葡萄糖分子活化阶段、葡萄糖降解阶段和氧化产能阶段为糖酵解途径的三个阶段。如图5所示,pH 4.0条件下,糖酵解第一、二阶段过程中所有的酶都没有显著上调,而且己糖激酶[(glk,SAZ_RS 25070(-5.86倍),SAZ_RS14035(-5.21倍)]显著下调,糖酵解途径的第一、二阶段是受到抑制的。在糖酵解的第三阶段,即从3-磷酸甘油醛到丙酮酸的过程需要4个酶催化,其中3个酶的表达显著上调[gapA(2.83倍)、eno(6.96倍)、pyk(2.83倍)],糖酵解的第三阶段增强。另外,从丙酮酸到乙酰-CoA途径的相关酶基因均显著上调。这表明在低pH环境中丙酮酸的代谢加速,S.albulus M-Z18可能需要大量的ATP供能。
三羧酸循环作为能量供应和物质氧化的主要途径,如图5所示,柠檬酸合酶(gltA, 3.03倍)和α-酮戊二酸脱氢酶复合体[(sucA(2.14倍)、sucB(2.3倍)和(old, 3.03倍)]这两个关键酶显著上调。此外,琥珀酸脱氢酶[frdB(3.73倍)、 frdC(2.3倍)]也显著上调,这表明低pH环境下三羧酸循环增强,这有助于菌株获得大量能量抵御酸胁迫。
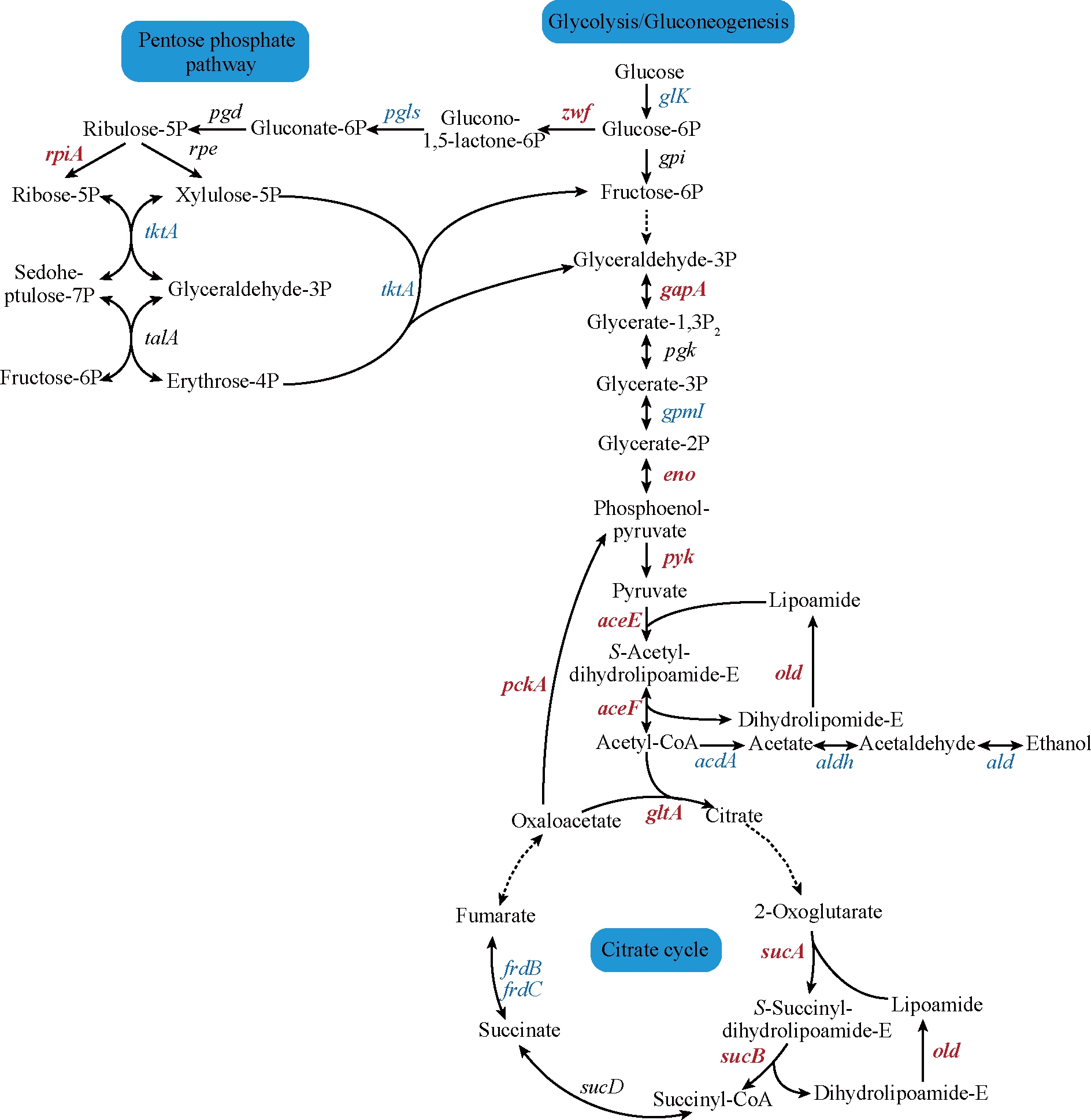
图5 糖代谢途径
Fig.5 Glucose metabolic pathway
注:红色字体表示该基因上调;蓝色字体表示该基因下调;黑色字体表示基因没有显著差异(见电子版)(下同)
戊糖磷酸途径是NADPH和核糖合成的主要途径[14]。氧化阶段和非氧化阶段为戊糖磷酸途径的两个阶段。在pH 4.0环境中,葡萄糖-6-磷酸脱氢酶基因(zwf, 2.3倍)表达上调,葡萄糖-6-磷酸内脂酶基因(pglS, -13.93倍)却显著下调,推测低pH条件下S.albulus M-Z18的NADPH合成增加。在非氧化阶段,核酮糖-5-磷酸异构酶基因(rpiA, 6.32倍)显著上调、转酮酶(tktA, -17.03倍)基因大幅下调,其余基因没有显著变化,说明戊糖磷酸途径的非氧化阶段在低pH环境下是减弱的。综上,低pH条件下S.albulus M-Z18通过增强糖酵解途径第三阶段、戊糖磷酸途径中NADPH合成、减弱戊糖磷酸途径非氧化阶段,使丙酮酸更多的进入柠檬酸循环以增加糖代谢产能来应对低pH胁迫。
2.3.2 氧化磷酸化途径
需氧微生物主要通过氧化磷酸化产生ATP来进行胞内的代谢反应。完成这一过程的是电子传递链,其主要由4种蛋白质复合物组成,分别是复合体Ⅰ(NADH-Q还原酶)、复合体Ⅱ(琥珀酸-Q还原酶)、复合体Ⅲ(泛醌-细胞色素c还原酶)和复合体Ⅳ(细胞色素c氧化酶)。pH 4.0条件下,S.albulus M-Z18呼吸链复合体Ⅱ和复合体Ⅳ显著上调,这说明菌株的呼吸强度增强。pH 4.0条件下ATP合酶的δ亚基(atpH, -6.32倍)、B亚基(atpF, -2.51倍)和C亚基(atpE, -2.71倍)下调,推测这些基因的下调有可能限制酸性条件下H+流入胞内防止胞内环境酸化。但总体上来说,低pH条件下氧化磷酸化增强,呼吸产能增加,这将有助于菌株抵御低pH胁迫(表1)。
表1 氧化磷酸化相关的差异表达基因
Table 1 Differently expressed genes related to pentose oxidative phosphorylation
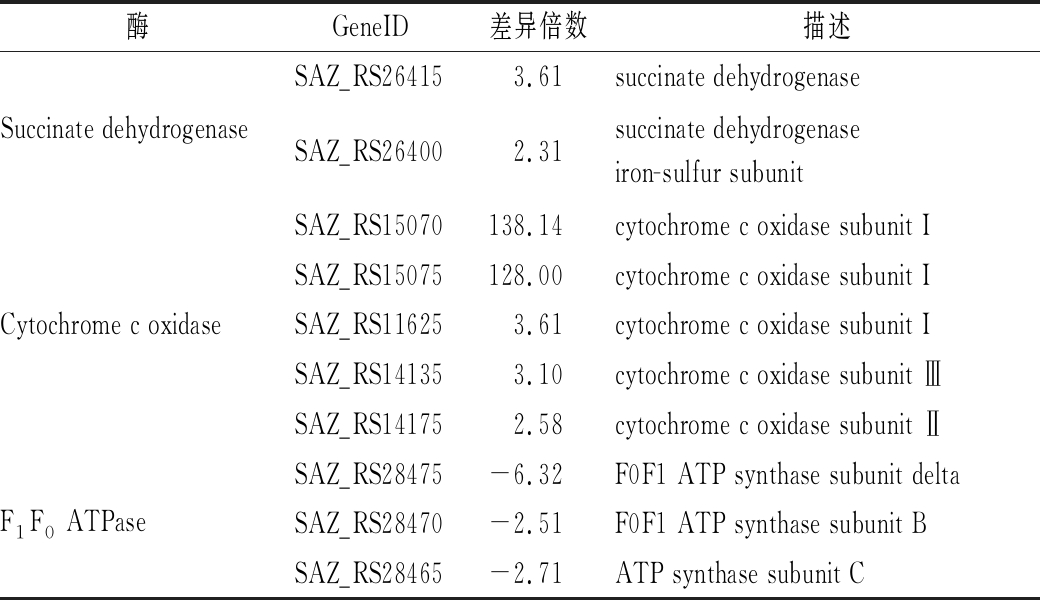
酶GeneID差异倍数描述SAZ_RS264153.61succinate dehydrogenaseSuccinate dehydrogenaseSAZ_RS264002.31succinate dehydrogenase iron-sulfur subunitSAZ_RS15070138.14cytochrome c oxidase subunit ISAZ_RS15075128.00cytochrome c oxidase subunit ICytochrome c oxidaseSAZ_RS116253.61cytochrome c oxidase subunit ISAZ_RS141353.10cytochrome c oxidase subunit ⅢSAZ_RS141752.58cytochrome c oxidase subunit ⅡSAZ_RS28475-6.32F0F1 ATP synthase subunit deltaF1F0 ATPaseSAZ_RS28470-2.51F0F1 ATP synthase subunit BSAZ_RS28465-2.71ATP synthase subunit C
2.3.3 氨基酸合成途径
低pH环境对细胞内氨基酸合成相关基因转录的影响如图6所示。组氨酸是一种碱性氨基酸,它的合成起始物是5-磷酸核糖-1-焦磷酸盐(phosphoribosyl pyrophosphate,PRPP)。由于戊糖磷酸途径中的转酮酶(tktA, -17.03倍)基因表达下调,非氧化阶段主要合成5-磷酸核糖,因此PRPP的合成可能是增强的。从PRPP到组氨酸的合成途径中4个基因发生显著变化,磷酸核糖-ATP二磷酸酶(hisE, 9.2倍)显著上调、磷酸核糖甲酰胺-5-氨基咪唑甲酰胺核糖异构酶[(hisA, -2.46倍)、转氨酶(hisC, SAZ_RS43525(-4.6倍)、SAZ_RS12845(-2.64倍)]和组氨酸脱氢酶(hisD, -2.46倍)都显著下调。后续基因显著下调可能是合成组氨酸的量太大造成了反馈抑制,组氨酸合成最终可能是增强的。羟基丙酮酸和3P-D-甘油酸是丝氨酸氨基酸家族的合成起始物。由图6可知,丙氨酸乙醛转氨酶(agxT, -4.2倍)和D-3-磷酸甘氨酸脱氢酶(serA,-15.14倍)都显著下调,这将整体限制了丝氨酸、甘氨酸和半胱氨酸的合成。4-磷酸赤藓糖是芳香族氨基酸(苯丙氨酸、酪氨酸和色氨酸)的前体物质。前文的分析显示,pH 4.0条件下,S.albulus M-Z18的戊糖磷酸途径非氧化阶段整体减弱,而且从4-磷酸赤藓糖到苯丙氨酸、酪氨酸和色氨酸合成途径的多个基因显著下调,这说明整个芳香族氨基酸合成整体减弱。乙酰乳酸合成酶、丙酮酸是丙酮酸族氨基酸(丙氨酸、缬氨酸、异亮氨酸)生物合成的限速酶[15]和前体。经前文分析,pH 4.0条件下丙酮酸更多的流向糖酵解途径,而乙酰乳酸合酶(livB, -3.78倍)和2-异丙基苹果酸合酶(leuA, -4.0倍)也显著下调,这使得丙酮酸族氨基酸合成整体上被削弱。虽然后续合成途径中酮醇酸还原异构酶(livC, 2.2倍)、3-异丙基苹果酸脱水酶小亚基(leuD, 2.2倍)显著上调,但是合成途径的最后一步,支链氨基酸转氨酶[livE, SAZ_RS38250(-3.03倍)、SAZ_RS38655(-3.03倍)和SAZ_RS10355(-2.46倍)]和亮氨酸脱氢酶(leu, -4.92倍)显著下调。总之,低pH条件下丙酮酸族氨基酸的生物合成也会减弱。天冬氨酸族氨基酸的合成起始物是草酰乙酸,从图6可知天冬氨酸氧化酶(nadB, -4.59倍)显著下调,这将限制了天冬氨酸族氨基酸的合成。天冬氨酸激酶(lysC, 3.71倍)、苏氨酸合酶(thrC, 3.25倍)和5-甲基四氢叶酸-同型半胱氨酸甲基转移酶(metH, 3.73倍)显著上调,天冬氨酸更多的流向苏氨酸和甲硫氨酸,天冬氨酸的含量可能减少。令人惊讶的是pH 4.0条件下天冬氨酸β-半醛脱氢酶基因(asd)上调了2 164.77倍,而后续赖氨酸合成途径的基因几乎都发生下调,考虑到S.albulus M-Z18只有在低pH条件下大量合成ε-PL,所以赖氨酸的合成一定是增强的。推测赖氨酸合成途径酶基因的下调可能是低pH条件下赖氨酸的大量合成,反馈抑制赖氨酸合成途径中酶的合成。α-酮戊二酸是谷氨酸族氨基酸(谷氨酸、谷氨酰胺、脯氨酸和精氨酸)的前体,低pH条件下谷氨酸合酶[gltB, SAZ_RS13535(4.6倍)、SAZ_RS13540(2.46倍)]和谷氨酸脱氢酶(gdhA, 2.64倍)基因表达上调,证明谷氨酸的合成可能增强。而谷氨酰胺酶(glsA, 5.28倍)和谷氨酰胺合酶(glnA, 8倍)也表达上调,证明谷氨酰胺与谷氨酸之间转换强度增加,谷氨酰胺合酶的合成也可能增强。谷氨酸、谷氨酰胺是耐酸系统(谷氨酸脱羧酶系统和谷氨酸-谷氨酸转运蛋白系统)的底物,其含量增加有助于S.albulus M-Z18在低pH条件下存活。另外,pH 4.0条件下从谷氨酸到精氨酸和脯氨酸的合成途径多个基因显著下调,精氨酸和脯氨酸的合成可能减弱。综上,低pH条件下,丝氨酸家族氨基酸、芳香族氨基酸、丙酮酸族氨基酸、天冬氨酸、脯氨酸和精氨酸的合成被削弱,而组氨酸、甲硫氨酸、苏氨酸、赖氨酸、谷氨酸和谷氨酰胺的合成可能增强。总之,低pH条件下大部分氨基酸合成削弱了,但是碱性氨基酸组氨酸、赖氨酸的合成增强,这将有助于菌株抵御酸胁迫。

图6 氨基酸合成途径
Fig.6 Amino acid synthesis pathway
2.3.4 细胞壁肽聚糖合成途径
细胞壁作为细胞最外层的保护屏障,对于维持菌体的正常形态和抵抗渗透压具有重要作用。在低pH条件下,细胞壁可以通过限制H+或酸性物质进入细胞来抵御酸胁迫。小白链霉菌细胞壁主要由多层肽聚糖囊状结构和嵌插、镶嵌其中的磷壁酸(teichoic acid,TA)、蛋白质等物质组成。作为小白链霉菌细胞壁主要成分——肽聚糖的生物合成可大致分为三个阶段:细胞质中UDP-N-乙酰壁酰五肽(UDP-N-acetylmuramic acid-pentapeptide,UDP-MurNAc-pentapeptide)的合成、细胞膜中双糖肽亚单位的形成和胞外肽聚糖链的交联[16-17]。6-磷酸果糖是肽聚糖合成起始物,UDP-N-乙酰葡糖胺(UDP-N-acetylglucosamine,UDP-GlcNAc)和UDP-N-乙酰胞壁酸(UDP-MurNAc)是肽聚糖聚糖链的基本组成单位,在低pH条件下,果糖磷酸激酶[scrk, SAZ_RS13190(-2.83倍)、SAZ_RS15195(-4.29倍)]和UDP-N-乙酰壁酸脱氢酶(glmM, -2.3倍)下调,使得肽聚糖合成整体受限。相较于pH 5.5,UDP-N-乙酰壁氨酰丙氨酸-D-谷氨酸连接酶(murD,-3.89倍)和UDP-N-乙酰壁氨酰-L-丙氨酰-D-谷氨酸-2,6-二氨基吡啶连接酶[murE, SAZ_RS37110(-2.64倍)、SAZ_RS13835(-2.14倍)]下调,这会导致UDP-MurNAc-五肽的肽尾合成削弱。此外,低pH时,磷酸-N-乙酰壁氨酰五肽转移酶(mraY, 2.3倍)和甘氨酸转移酶[femX, SAZ_RS00170(4.93倍)和SAZ_RS41770(4.93倍)],揭示肽聚糖合成第二阶段的增强。令人惊讶的是,关于肽聚糖交联的糖基转移酶、DD-转肽酶和DD-羧肽酶多个同工酶表现出显著上调,说明低pH条件下肽聚糖的初级交联、次级交联的速度增加(图7)。
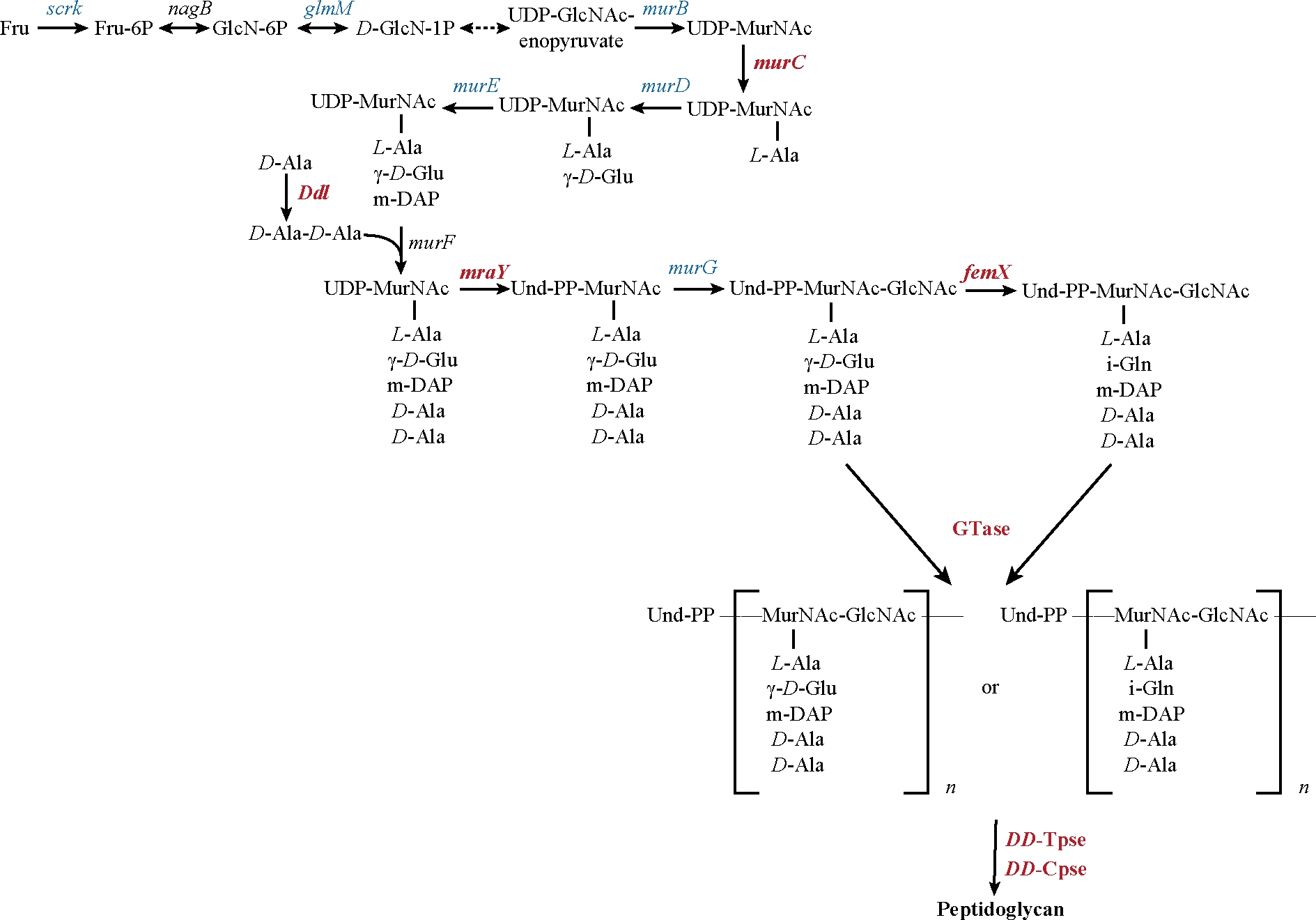
图7 细胞壁肽聚糖合成途径
Fig.7 Cell wall peptidoglycan synthesis pathway
2.3.5 细胞膜脂肪酸合成途径
脂肪酸的合成可分为两个阶段:初始反应和循环反应。在起始反应中,乙酰-CoA羧化酶和丙二酰转移酶首先催化乙酰-CoA形成丙二酸单酰ACP,然后β-酮酰基ACP合成酶Ⅲ催化丙二酸单酰ACP和乙酰ACP缩合形成β-酮酰基ACP,这些反应完成后,脂肪酸的合成进入循环反应阶段[18]。循环反应阶段是在在Ⅱ型脂肪酸合酶的催化下经缩合、β还原、脱水、烯酰还原4个反应重复进行脂肪酸的合成,每进行1次循环脂肪酸碳链长度增加2个碳原子,形成的脂肪酸有4~18个碳不等。而乙酰-CoA羧化酶(acetyl-CoA carboxylase,ACC)是脂肪酸合成的第一个限速酶[19]。如图8所示,pH 4.0条件下,乙酰-CoA羧化酶基因下调(acc, -2.07倍),表明在低pH环境下脂肪酸合成起始反应阶段受到限制。此外参与脂肪酸循环反应阶段的酶也出现不同程度下调,表明脂肪酸循环反应阶段也受到限制。然而酰基载体蛋白基因[acp, SAZ_RS36395(10.27倍)、SAZ_RS15400(10.06倍)和SAZ_RS03525(7.73倍)]在pH 4.0条件下显著上调,在脂肪酸合成受限的情况下这将有助于增加脂肪酸的合成速度。环丙烷脂肪酸有助于微生物抵御酸胁迫[20],在pH 4.0时,环丙烷脂肪酰磷脂合酶(cfa, 4.99倍)显著上调,表明S.albulus M-Z18可能通过增加细胞膜中环丙烷脂肪酸含量抵御酸胁迫。此外,在pH 4.0条件下,脂肪酸去饱和酶(desA, 3.23倍)基因上调,推测细胞膜脂肪酸的不饱和度增加。综上,S.albulus M-Z18脂肪酸合成整体减弱,但是细胞膜中不饱和脂肪酸的比例、环丙烷脂肪酸的含量可能增加。
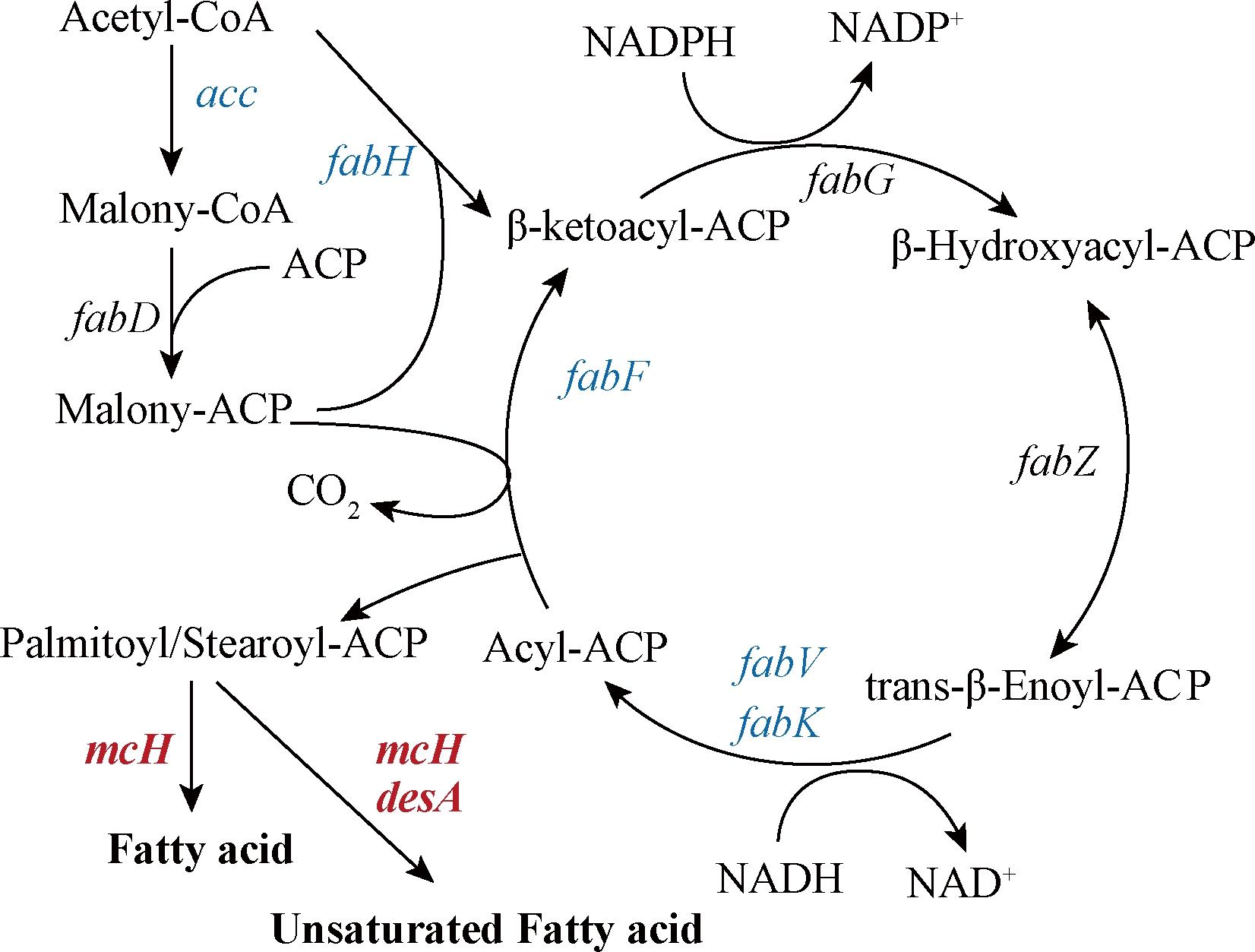
图8 细胞膜脂肪酸合成途径
Fig.8 Cell wall peptidoglycan synthesis pathway
2.3.6 蛋白质和核苷酸修复分子
分子伴侣对细胞中的蛋白质在生理和压力条件下的正确折叠至关重要,其可以防止未折叠多肽的聚集和变性多肽的正确再折叠[21]。在低pH环境中,分子伴侣蛋白DnaK[SAZ_RS39120(8.57倍)、SAZ_RS21050(2.41倍)和SAZ_RS43735(2.04倍)]、DnaJ(2.95倍)和GroES(2.31倍)均表达上调,而水解错误折叠错误蛋白的Clp蛋白酶基因全部上调,表明低pH环境中S.albulus M-Z18修复折叠错误蛋白质能力的增强(表2)。
在酸胁迫环境中细菌DNA通常会受到损伤,因此细菌通常有DNA的修复机制以保持基因组的完整性。当DNA结构有较大损伤或DNA链多处严重损伤会诱导核苷酸的切除修复。在低pH环境中,参与核苷酸片段切除修复的Uvr A(3.07倍)、UvrB(5.28倍)、DNA聚合酶I(4.5倍)和连接酶(2.81倍)基因表达上调,S.albulus M-Z18的核苷酸切除修复能力增强。而当DNA双链发生断裂,则需要非同源末端连接的修复及基因的同源重组。pH 4.0时,Ku蛋白(11.24倍)、DNA聚合酶(6.11倍)RecA蛋白(6.77倍)这些参与非同源末端连接修复和同源重组的基因显著上调(表2)。综上,在低pH环境中S.albulus M-Z18通过增强蛋白质和DNA修复能力以应对低pH胁迫造成的蛋白质和DNA损伤。
表2 蛋白质、DNA保护与修复相关的差异表达基因
Table 2 Differently expressed genes related to protein and DNA protection and repair
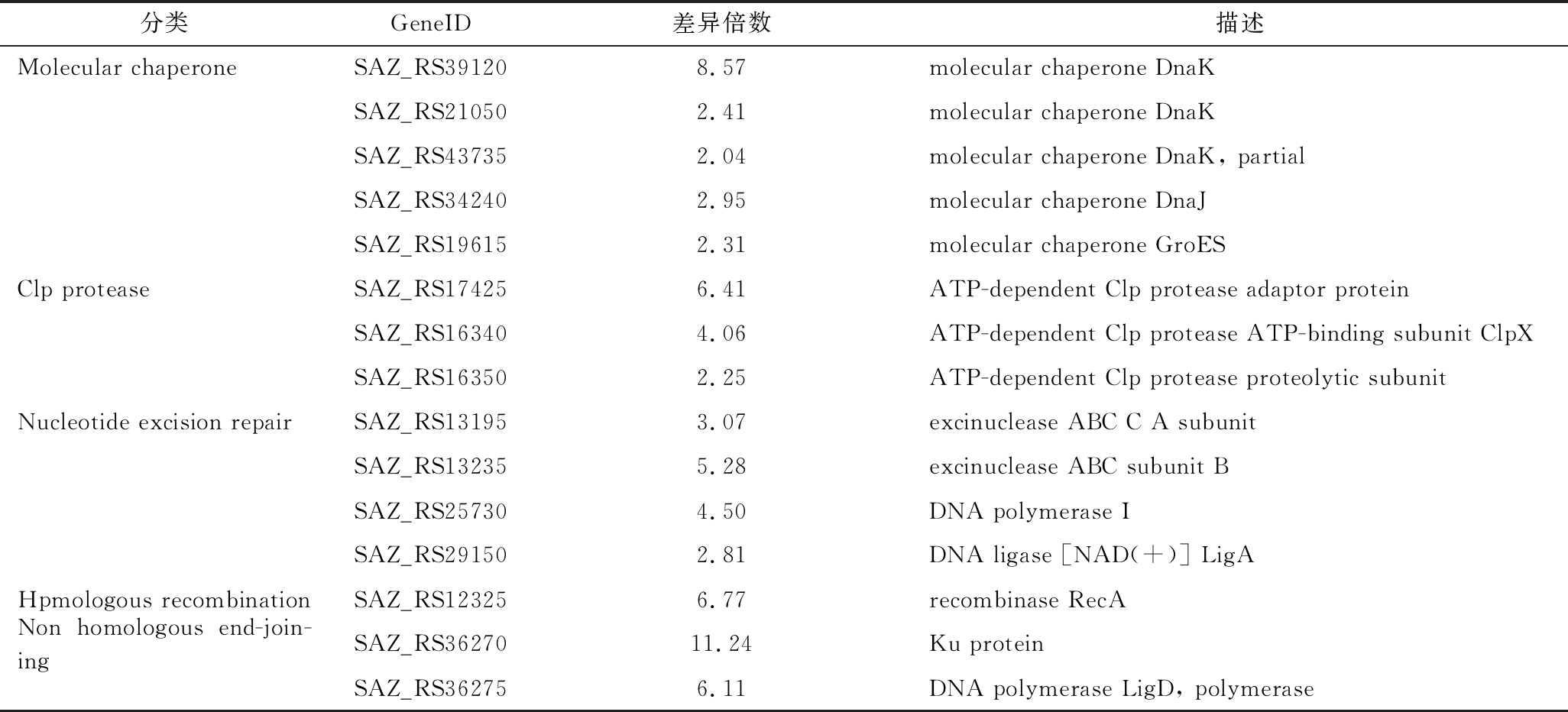
分类GeneID差异倍数描述Molecular chaperoneSAZ_RS391208.57molecular chaperone DnaKSAZ_RS210502.41molecular chaperone DnaKSAZ_RS437352.04molecular chaperone DnaK, partialSAZ_RS342402.95molecular chaperone DnaJSAZ_RS196152.31molecular chaperone GroESClp proteaseSAZ_RS174256.41ATP-dependent Clp protease adaptor proteinSAZ_RS163404.06ATP-dependent Clp protease ATP-binding subunit ClpXSAZ_RS163502.25ATP-dependent Clp protease proteolytic subunitNucleotide excision repairSAZ_RS131953.07excinuclease ABC C A subunitSAZ_RS132355.28excinuclease ABC subunit BSAZ_RS257304.50DNA polymerase ISAZ_RS291502.81DNA ligase [NAD(+)] LigAHpmologous recombinationSAZ_RS123256.77recombinase RecANon homologous end-join-ingSAZ_RS3627011.24Ku proteinSAZ_RS362756.11DNA polymerase LigD, polymerase
2.3.7 耐酸系统
为了维持胞内环境中pH的稳态,微生物主要通过阻碍细胞外H+的进入、增强细胞内酸性物质的排出和消耗来实现。微生物细胞可以通过氨基酸脱羧、脱氨反应、尿素水解反应来消耗、中和胞内的H+。经转录组数据的分析,在S.albulus M-Z18中存在完整的谷氨酸脱羧酶系统、谷氨酸-谷氨酰胺转运蛋白系统、不完整的赖氨酸脱羧酶系统、胍基丁胺脱亚氨酶系统、尿素酶系统和精氨酸脱氨酶系统。如图9所示,低pH环境中尿素酶系统相关基因显著上调,其中尿素酶亚基α(urease subunit alpha ,UreC)、镍转运蛋白(nickel permease,NixA)上调了84.64倍和47.97 倍。在低pH环境中,尿素酶系统可能对维持胞内环境中pH的稳态起着重要作用。
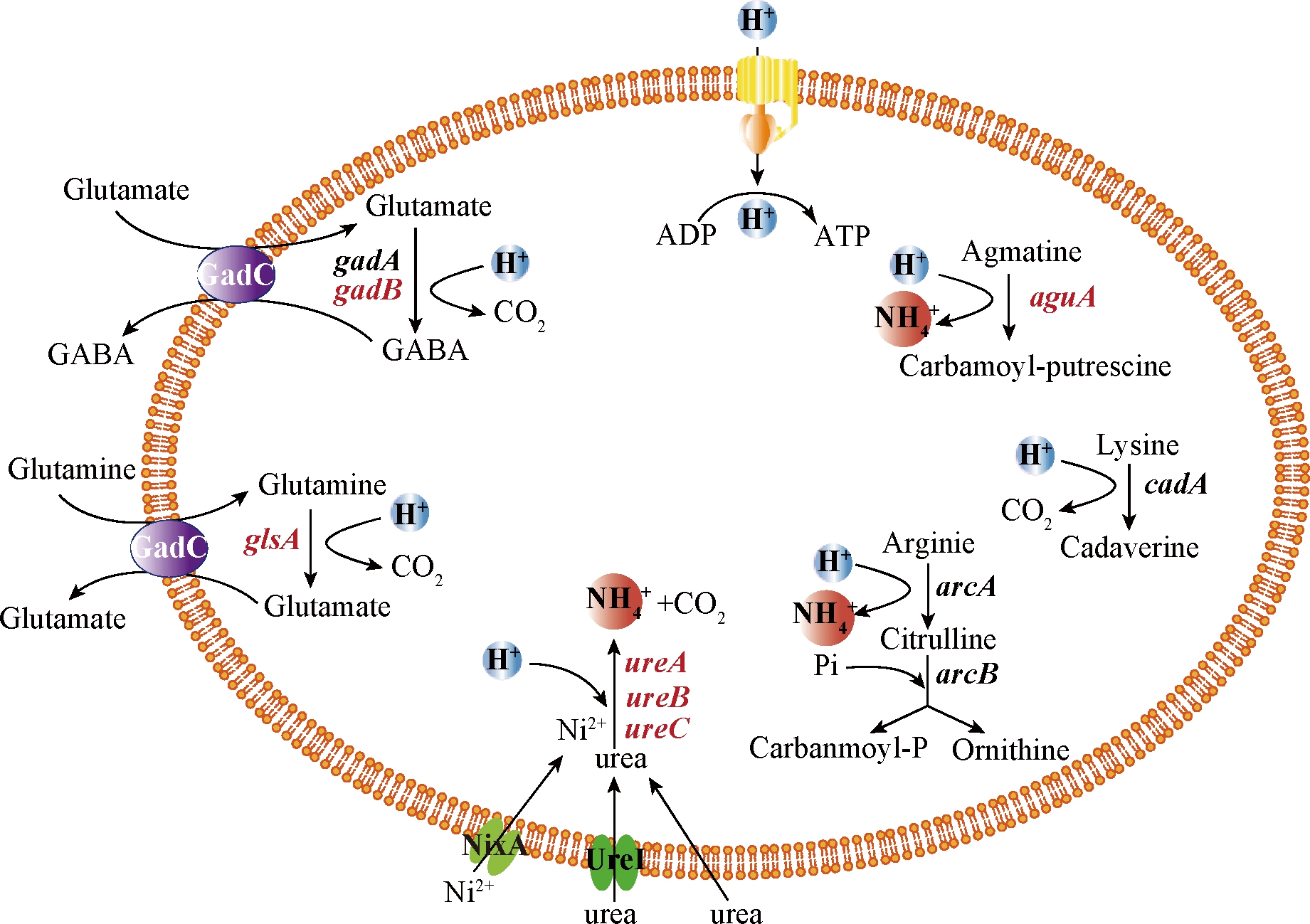
图9 耐酸系统组分
Fig.9 Acid resistant system components
此外,谷氨酸脱羧酶系统中的谷氨酸脱羧酶(gadB, 2.85倍)、谷氨酸-GABA反向转运体[gadC, 3.68倍)、SAZ_RS03735(5.28倍)],谷氨酸-谷氨酰胺转运蛋白系统中的谷氨酰胺酶(glsA, 5.39倍),胍基丁胺脱亚氨酶系统中的胍基丁胺脱亚氨酶[augA, SAZ_RS39900(7.89倍)、SAZ_RS39905(2.75倍)]在pH 4.0环境中都显著上调。综上,在低pH环境中S.albulus M-Z18可能通过增强多种耐酸系统来抵御低pH的胁迫。
3 结论与讨论
小白链霉菌是工业生产ε-PL的主要生产菌,其发酵过程中pH会自发下降至3.0左右,需要流加氨水维持发酵过程中pH的稳定。通过不同pH的分批发酵培养,其可以在pH 3.5及以上的培养基中正常生长,这证明S.albulus M-Z18具有较强的酸耐受性。
通过恒化培养,使得菌体达到稳定状态以消除S.albulus M-Z18在不同pH环境中因比生长速率造成的生长差异。取此时的样品用于分析菌株在不同pH值(4.0和5.5)下基因转录的差异。pH 4.0条件下,共有3 893个基因差异表达达到显著,其中显著上调的基因有1 786个,显著下调的基因有2 107个。GO分析显示显著差异表达基因主要集中于生物过程,而KEGG分析显著差异表达基因集中在代谢途径。具体分析显著差异表达基因富集的代谢途径,低pH环境中S.albulus M-Z18通过增强糖代谢中糖酵解第三阶段、三羧酸循环途径、氧化磷酸化途径和戊糖磷酸途径的氧化阶段来增强能量的合成,这将有助于菌株在低pH条件下存活;pH 4.0条件下大部分氨基酸的合成被削弱,但碱性氨基酸组氨酸和赖氨酸的合成增加了。细胞壁作为阻碍胞外H+的第一道屏障,据报道酸胁迫中细菌细胞壁的厚度会减小[22-23]。转录组分析显示pH 4.0条件下S.albulus M-Z18肽聚糖的合成整体上削弱,这与文献报道一致,但是催化肽聚糖交联的酶的基因几乎全部上调,推测菌株可能是通过增加肽聚糖的组装速度以维持酸胁迫环境中完整的细胞形态。除了细胞壁,细胞膜也是阻碍胞外H+进入胞内的外界屏障,很多研究报道显示微生物在面对酸胁迫时,细胞膜脂肪酸会发生相应的改变以适应不利环境,这包括增加脂肪酸的不饱和度、平均碳链长度和环丙烷脂肪酸的含量[24-25]。本文研究发现,低pH环境中S.albulus M-Z18关于合成不饱和脂肪酸、环丙烷脂肪酸相关基因显著上调,说明低pH环境中S.albulus M-Z18可能改变细胞膜脂肪酸成分,以适应酸胁迫环境中和外界之间物质和信息的交互。低pH胁迫时往往会造成微生物中蛋白质折叠错误、变性,DNA碱基错配、插入或缺失等损伤。同样发现pH 4.0条件下S.albulus M-Z18中分子伴侣、Clp蛋白酶上调、核苷酸切除修复和非同源末端连接相关基因也表达上调,说明低pH环境下蛋白质和DNA损伤的修复增强。胞内pH稳态对于耐酸微生物在低pH环境中维持正常生理生化反应具有重要意义。耐酸系统通过消耗胞内的H+、产生碱性物质来维持胞内pH稳态。革兰氏阳性菌中已报道的多数耐酸系统基因在S.albulus M-Z18基因组中都可以找到。pH 4.0条件下,谷氨酸脱羧酶系统、谷氨酰胺酶-谷氨酰胺转运蛋白系统、胍基丁胺脱亚氨酶系统、尿素酶系统和精氨酸脱氨酶系统都发生不同程度的上调,其中尿素酶系统相关基因上调最为显著,尿素酶系统可能起着重要的作用。通过以上研究,确定S.albulus M-Z18有着不同于一般链霉菌的酸耐受能力。发现在低pH环境中,S.albulus M-Z18可能通过增强胞内代谢产能、碱性氨基酸的合成,改变细胞膜成分,维持细胞壁完整性,加强蛋白质、DNA损伤修复的能力,上调耐酸系统相关基因表达以应对低酸胁迫。然而,关于S.albulus M-Z18的耐酸分子机制还需要进一步分子生物学水平的验证。
[1] WANG L, LI S, ZHAO J, et al.Efficiently activated ε-poly-L-lysine production by multiple antibiotic-resistance mutations and acidic pH shock optimization in Streptomyces albulus[J].Microbiologyopen, 2019, 8(5):e00728.
[2] SHIMA S, SAKAI H.Polylysine produced by Streptomyces[J].Agricultural and Biological Chemistry, 1977, 41(9):1 807-1 809.
[3] SHIMA S, SAKAI H.Poly-L-lysine produced by Streptomyces.Part Ⅲ.chemical studies[J].Agricultural and Biological Chemistry, 1981, 45(11):2 503-2 508.
[4] XU Z X, XU Z, FENG X H, et al.Recent advances in the biotechnological production of microbial poly(ε-L-lysine) and understanding of its biosynthetic mechanism[J].Applied Microbiology and Biotechnology, 2016, 100(15):6 619-6 630.
[5] HIRAKI J, ICHIKAWA T, NINOMIYA S I, et al.Use of ADME studies to confirm the safety of ε-polylysine as a preservative in food[J].Regulatory Toxicology and Pharmacology, 2003, 37(2):328-340.
[6] CHEN X S, WANG K F, ZHENG G C, et al.Preparation, characterization and antimicrobial activity of ε-poly-L-lysine with short chain length produced from glycerol by Streptomyces albulus[J].Process Biochemistry, 2018, 68:22-29.
[7] YU Z, SHEN X, WU Y, et al.Enhancement of ascomycin production via a combination of atmospheric and room temperature plasma mutagenesis in Streptomyces hygroscopicus and medium optimization[J].Amb Express, 2019, 9(1):25.
[8] LU W Y, FAN J H, WEN J P, et al.Kinetic analysis and modeling of daptomycin batch fermentation by Streptomyces roseosporus[J].Applied Biochemistry and Biotechnology, 2011, 163(4):453-462.
[9] KITO M, TAKIMOTO R, YOSHIDA T, et al.Purification and characterization of an ε-poly-L-lysine-degrading enzyme from an ε-poly-L-lysine-producing strain of Streptomyces albulus[J].Archives of Microbiology, 2002, 178(5):325-330.
[10] YAMANAKA K, MARUYAMA C, TAKAGI H, et al.ε-poly-L-lysine dispersity is controlled by a highly unusual nonribosomal peptide synthetase[J].Nature Chemical Biology, 2008, 4(12):766-772.
[11] YAMANAKA K, KITO N, IMOKAWA Y, et al.Mechanism of ε-poly-L-lysine production and accumulation revealed by identification and analysis of an ε-poly-L-lysine-degrading enzyme[J].Applied and Environmental Microbiology, 2010, 76(17):5 669-5 675.
[12] 王开方, 潘龙, 刁文娇, 等.pH值和比生长速率协同调控Streptomyces albulus合成ε-聚赖氨酸[J].食品与发酵工业, 2019, 45(23):8-14.
WANG K F, PAN L, DIAO W J, et al.Effects of pH and specific growth rates on ε-poly-L-lysine biosynthesis in Streptomyces albulus[J].Food and Fermentation Industries, 2019, 45(23):8-14.
[13] ITZHAKI R F.Colorimetric method for estimating polylysine and polyarginine[J].Analytical Biochemistry, 1972, 50(2):569-574.
[14] BESSELL E M, THOMAS P.The effect of substitution at C-2 of D-glucose 6-phosphate on the rate of dehydrogenation by glucose 6-phosphate dehydrogenase (from yeast and from rat liver)[J].The Biochemical Journal, 1973, 131(1):83-89.
[15] ZHENG P Z, SUN X M, GUO L L, et al.Cloning, expression, and characterization of an acetolactate synthase (ALS) gene from Anabaena azotica[J].Process Biochemistry, 2015, 50(9):1 349-1 356.
[16] BARRETEAU H, ![]() A, BONIFACE A, et al.Cytoplasmic steps of peptidoglycan biosynthesis[J].FEMS Microbiology Reviews, 2008, 32(2):168-207.
A, BONIFACE A, et al.Cytoplasmic steps of peptidoglycan biosynthesis[J].FEMS Microbiology Reviews, 2008, 32(2):168-207.
[17] SHAM L T, BUTLER E K, LEBAR M D, et al.Bacterial cell wall.MurJ is the flippase of lipid-linked precursors for peptidoglycan biogenesis[J].Science, 2014, 345(6 193):220-222.
[18] 余永红, 马建荣, 王海洪.细菌脂肪酸合成多样性的研究进展[J].微生物学杂志, 2016, 36(4):76-83.
YU Y H, MA J R, WANG H H.Advances in fatty acid biosynthetic diversity in bacteria[J].Journal of Microbiology, 2016, 36(4):76-83.
[19] BIANCHI A, EVANS J L, IVERSON A J, et al.Identification of an isozymic form of acetyl-CoA carboxylase[J].Journal of Biological Chemistry, 1990, 265(3):1 502-1 509.
[20] CHEN Y Y, G NZLE M G.Influence of cyclopropane fatty acids on heat, high pressure, acid and oxidative resistance in Escherichia coli[J].International Journal of Food Microbiology, 2016, 222:16-22.
NZLE M G.Influence of cyclopropane fatty acids on heat, high pressure, acid and oxidative resistance in Escherichia coli[J].International Journal of Food Microbiology, 2016, 222:16-22.
[21] BEN-ZVI A P, GOLOUBINOFF P.Review:Mechanisms of disaggregation and refolding of stable protein aggregates by molecular chaperones[J].Journal of Structural Biology, 2001, 135(2):84-93.
[22] JIN J H, QIN Q, GUO H Y, et al.Effect of pre-stressing on the acid-stress response in bifidobacterium revealed using proteomic and physiological approaches[J].PLoS One, 2015, 10(2):e0117702.
[23] XU N, LYU H F, WEI L, et al.Impaired oxidative stress and sulfur assimilation contribute to acid tolerance of Corynebacterium glutamicum[J].Applied Microbiology Biotechnology, 2019, 103(4):1 877-1 891.
[24] BROADBENT J R, LARSEN R L, DEIBEL V, et al.Physiological and transcriptional response of Lactobacillus casei ATCC 334 to acid stress[J].Journal of bacteriology, 2010, 192(9):2 445-2 458.
[25] HUANG R, PAN M, WAN C, et al.Physiological and transcriptional responses and cross protection of Lactobacillus plantarum ZDY2013 under acid stress[J].Journal of Dairy Science, 2016, 99(2):1 002-1 010.