植物与人类有特别密切的关系,尤其是具有重要经济、文化、科学价值的植物,如优质作物食品、地方特色植物食品、药食同源食品等,常有很高的食用价值。然而,由于检测技术发展的局限性,人们常难以严格区分地方特色植物食品、高价值植物性食品,甚至出现在不知情的情况下误食致敏性植物成分而对身体造成不同程度的损害,从而面临着植物掺杂、掺假的高通量检测评估、监管执法带来严重的问题。
随着人类基因组测序工作的完成和人类基因组草图的公布,生物信息学的研究走向了一个高潮[1-2]。2003年,加拿大分类学家HEBERT首次提出DNA条形码概念,是生物体一段公认的能够代表该物种的标准的、有足够变异的、易扩增且相对较短的DNA片段[3-4]。基于DNA 条形码技术,根据样品类型选择基因条形码引物,扩增基因条码序列并进行测序分析,通过数据库比对确定目标物种,成为物种鉴定的有效方法[5-7]。例如,HAMB CK等[8]采用DNA条形码技术对波罗的海海岸周边的蜘蛛摄食进行了筛查,白文明等[9]基于DNA条形码技术鉴别有毒鹅膏菌属物种。目前,国际上比较权威的核酸数据库为GenBank[10]、EMBL[11]以及DDBJ[12]。叶绿体基因组中的RbcL[13-15]基因是植物源性分子系统学研究中使用最为广泛的分子指标之一。高等植物RbcL基因在结构上和原核生物基因相似,由5′非编码区、编码区和3′非编区3部分组成。5′非编码区具有可以和叶绿体16S rRNA 3′端附近互补的S-D序到,3′非编码区具反向重复序列,能形成典型的茎环结构作为转录终止信号[16]。目前,RbcL基因在很多植物鉴定及其多样性的研究中获得了较好的结果。姚丹丹等[17]利用RbcL基因序列分析马尾藻属遗传进化关系,FAWLEY等[18]通过RbcL基因序列分析,揭示了藻类的广泛多样性,陈玉花等[19]建立了蜀葵花HPLC指纹图谱及RbcL序列DNA条形码分子鉴定方法,林晓霞等[20]基于RbcL序列对石斛属植物亲缘关系进行了研究。
CK等[8]采用DNA条形码技术对波罗的海海岸周边的蜘蛛摄食进行了筛查,白文明等[9]基于DNA条形码技术鉴别有毒鹅膏菌属物种。目前,国际上比较权威的核酸数据库为GenBank[10]、EMBL[11]以及DDBJ[12]。叶绿体基因组中的RbcL[13-15]基因是植物源性分子系统学研究中使用最为广泛的分子指标之一。高等植物RbcL基因在结构上和原核生物基因相似,由5′非编码区、编码区和3′非编区3部分组成。5′非编码区具有可以和叶绿体16S rRNA 3′端附近互补的S-D序到,3′非编码区具反向重复序列,能形成典型的茎环结构作为转录终止信号[16]。目前,RbcL基因在很多植物鉴定及其多样性的研究中获得了较好的结果。姚丹丹等[17]利用RbcL基因序列分析马尾藻属遗传进化关系,FAWLEY等[18]通过RbcL基因序列分析,揭示了藻类的广泛多样性,陈玉花等[19]建立了蜀葵花HPLC指纹图谱及RbcL序列DNA条形码分子鉴定方法,林晓霞等[20]基于RbcL序列对石斛属植物亲缘关系进行了研究。
为进一步解决食品中基于扩增子测序的植物掺假非定向筛查问题,本研究基于植物RbcL基因通用引物PCR扩增检测和测序技术,通过反复实验,开发了食品中基于扩增子测序的27种植物掺假非定向筛查方法。
1 材料与方法
1.1 植物RbcL基因通用引物序列
由于二代测序平台测序长度的限制,以及考虑大部分食品为深加工产品,导致基因组发生一定程度的降解,小片段条形码更加适用。因此本研究采用430 bp 左右的RbcL[21]片段作为条形码,引物序列如下:
5′端引物:5′-AATCTTCTACTGGTACATGGAC-3′
3′端引物:5′-TCATCATCTTTGGTAAAATCAAG-3′
1.2 试剂配制
1.2.1 PBS缓冲液
800 mL水中加入8.0 g氯化钠,0.2 g氯化钾,2.98 g磷酸氢二钠和0.22 g磷酸二氢钠,充分溶解后用盐酸调pH至7.4,加水定容至1 000 mL,在103.4 kPa,121 ℃条件下,灭菌15 min后使用。
1.2.2 裂解缓冲液Ⅰ
500 mL水中加人117.0 g氯化钠、20 g十六烷基三甲基溴化铵,充分溶解后,加入200 mL三羟甲基氨基甲烷-盐酸溶液,100 mL乙二胺四乙酸二钠溶液,加水定容至1 000 mL,在103.4 kPa,121 ℃条件下,灭菌15 min后使用。
1.2.3 裂解缓冲液Ⅱ
800 mL水中加入50 g十二烷基肌氨酸钠,充分溶解后,加水定容至1 000 mL,在103.4 kPa、121 ℃条件下,灭菌15 min后使用。
1.2.4 3 mol/L乙酸钾溶液(pH 5.2)
60 mL水中加入29.4 g乙酸钾,充分溶解,用冰乙酸调pH至5.2,加水定容至100 mL。
1.3 仪器与设备
NanoDrop 2000c紫外可见分光光度计,美国Thermo公司;ProFlex PCR仪,美国ABI公司;5424R小型台式冷冻离心机,德国Eppendorf公司;DYCP-31A电泳仪,北京六一仪器厂;EC3Darkroom凝胶成像系统,美国Spring Scientific公司。
1.4 样品制备及基因组DNA提取
1.4.1 独立样品的制备及DNA提取
称取0.5 g待测样品,充分研磨后转移至离心管中。加入1.0 mL裂解缓冲液I和0.4 mL裂解缓冲液Ⅱ,充分混匀,65 ℃温浴40 min,20 ℃、12 000×g离心15 min,吸取上清液到另一新的离心管中。加入等体积平衡酚-氯仿溶液,轻轻混匀,20 ℃、12 000×g离心10 min,吸取上清液到另一新的离心管中。加入等体积氯仿,轻缓混匀,20 ℃、12 000×g离心10 min,吸取上清液到另一新的离心管中。加入0.6倍体积异丙醇、0.1倍体积的乙酸钾溶液,轻轻颠倒混匀,-20 ℃静置2 h以上,20 000×g离心10 min,弃上清液。加入0.5 mL 1.0 mL 70%(体积分数)乙醇溶液,颠倒混合。12 000×g离心10 min,弃上清液。干燥DNA沉淀。加100 μL水或TE缓冲液溶解DNA。DNA的浓度和质量采用紫外分光光度法测定。
1.4.2 混合样品的制备及DNA提取
将1.4.1提取的DNA选择4~6个物种,将其按照等比例浓度进行混合形成混合样本A1~A4,具体成分见表1。
表1 混合植物源性样本成分
Table 1 Mixed plant-derived sample components

样本名称样本物种成分A1杏仁、花生、大豆、核桃、腰果、绿豆A2小米、玉米、水稻、绿豆、大豆A3大豆、水稻、小麦、腰果A4木薯、绿豆、豌豆、红薯、土豆、大豆
1.5 PCR扩增与电泳
反应体系总体积为25 μL,其中10×PCR缓冲液5 μL,正反向引物(10 μmol/L)各1 μL,dNTPs(10 μmol/L)2 μL,Taq DNA聚合酶(2.5U)0.2 μL,DNA模板(10~100 ng/μL)2 μL,用灭菌去离子水补足至总体积25 μL。可使用相同效果的商品化DNA聚合酶预混液进行PCR扩增。
反应参数:95 ℃(5 min)→95 ℃(30 s)→56 ℃(30 s)→72 ℃(30 s),35个循环;72 ℃延伸10 min;4℃保存。
电泳采用琼脂糖凝胶电泳法检测。
1.6 测序及分析
单一物种样品检测时,将PCR扩增产物进行Sanger法DNA测序。混合物种样品检测时,将PCR扩增产物进行二代测序分析。本研究测序由生工生物工程(上海)股份有限公司测序平台完成。
采用Cutadapt(v1.18)、PRINSEQ(0.20.4)、Usearch(11.0.667)软件对测序原始数据进行优化处理;采用Usearch(11.0.667)、gplots(3.0.1.1)、RDPclassifier(2.12)、ETE3(3.1.1)软件对所有序列进行操作分类单元(operational taxonomic units,OTU)划分并进行生物信息统计分析。
2 结果与分析
2.1 引物通用性、特异性验证
以市售红小豆、大豆、绿豆、马铃薯、桃核、芝麻等27种蔬果作物的基因组DNA和10种动物基因组DNA为模板,利用RbcL通用引物进行PCR扩增后凝胶电泳检测(图1),验证引物的通用适用性与植物特异性。

M-Marker(DL 2000);1~27-以红小豆、大豆、绿豆、红薯、马铃薯、木薯、薏米、小麦、玉米、小米、水稻、杏、腰果、榛子、芝麻、巴旦木、核桃、桃、香蕉、梨、小番茄、南瓜、胡萝卜、苹果、花生、开心果、芸豆基因组为模板的电泳结果;28~37-以猪、牛、羊、鸡、鸭、鹅、马、驴、猫、狗基因组为模板的电泳结果;38-阴性对照
图1 PCR扩增结果电泳图
Fig.1 Electrophoresis of PCR amplification results
实验结果表明,以27种植物基因组DNA为模板,利用通用引物进行PCR扩增,均可获得430 bp左右大小的条带,而以10种动物基因组DNA为模板,利用通用引物进行PCR扩增,均未扩增出目标片段。通用引物对该27种植物成分检测特异性、通用性较好。PCR产物经过Sanger法DNA测序后,将得到的碱基序列通过DNAMAN软件进行比对(图2),由图2可见,经过本实验涉及的引物扩增,27种不同物种的基因序列存在不同,可以利用数据库进行比对。经过NCBI数据库(https://blast.ncbi.nlm.nih.gov)序列比对,综合考虑Score值、Coverage值、Evalue值,相似度与Genbank序列号见表2,相似度均>99%。通常要求同种植物序列的相似度≥99%[22]。因此,植物通用引物RbcL可以有效地将实验所涉及的27种不同物种的植物区分开来。

图2 27种植物测序碱基序列比对
Fig.2 Sequence comparison of 27 plants
表2 PCR产物测序NCBI比对结果
Table 2 Sequencing comparison results of PCR products from NCBI
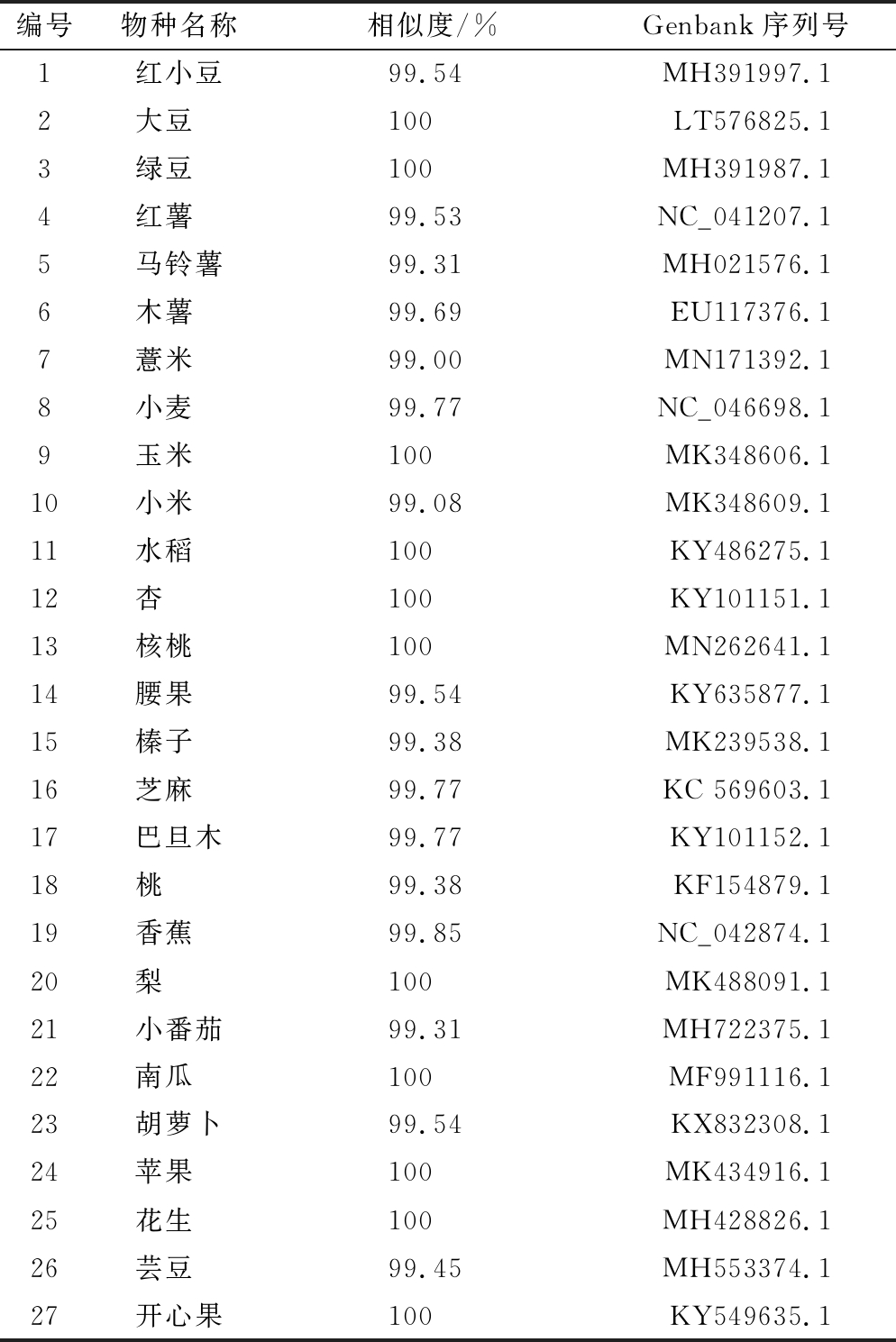
编号物种名称相似度/%Genbank序列号1红小豆99.54MH391997.12大豆 100LT576825.13绿豆 100MH391987.14红薯 99.53NC_041207.15马铃薯99.31MH021576.16木薯 99.69EU117376.17薏米 99.00MN171392.18小麦 99.77NC_046698.19玉米 100MK348606.110小米 99.08MK348609.111水稻 100KY486275.112杏 100KY101151.113核桃 100MN262641.114腰果 99.54KY635877.115榛子 99.38MK239538.116芝麻 99.77KC 569603.117巴旦木99.77KY101152.118桃 99.38KF154879.119香蕉 99.85NC_042874.120梨 100MK488091.121小番茄99.31MH722375.122南瓜 100MF991116.123胡萝卜99.54KX832308.124苹果 100MK434916.125花生 100MH428826.126芸豆 99.45MH553374.127开心果100KY549635.1
2.2 检测方法灵敏度验证
10倍系列梯度稀释目标植物源性成分基因组DNA,PCR反应体系内分别加入100、10、1、0.1 ng/μL 基因组DNA,进行PCR扩增,图3为玉米基因组DNA 10倍系列梯度稀释PCR扩增后电泳检测图。
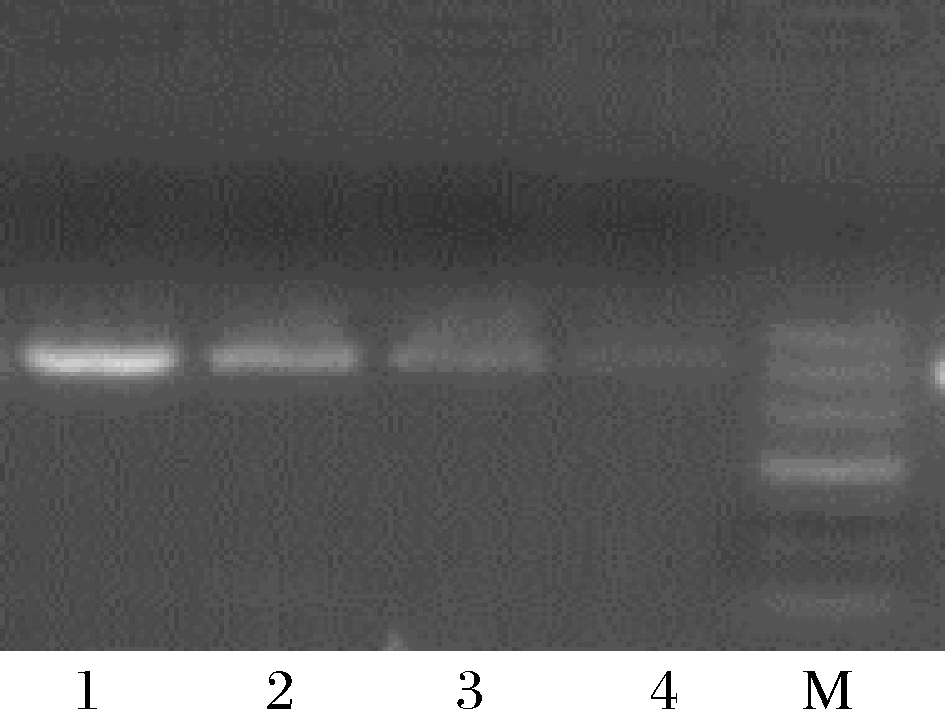
1-100 ng/μL基因组DNA;2-10 ng/μL基因组DNA;3-1 ng/μL基因组DNA;4-0.1 ng/μL基因组DNA;M-Marker(DL 500)
图3 玉米基因组梯度稀释电泳检测图
Fig.3 Electrophoretogram of maize genomic by gradient dilution
实验结果表明,随着模板DNA浓度的递减,PCR扩增条带呈现梯度变弱趋势。当基因组模板DNA质量浓度为0.1 ng/μL时,扩增条带良好。回收电泳条带进行测序,结果见表3。当基因组模板DNA质量浓度为0.1 ng/μL时,回收失败,因此该方法的检测灵敏度为1 ng/μL。
表3 玉米基因组梯度稀释PCR产物测序NCBI比对结果
Table 3 Sequencing comparison results of PCR products of maize genomic by gradient dilution from NCBI

编号玉米基因组稀释度/(ng·μL-1)相似度/%Genbank序列号1100100MK348606.1210100MK348606.131100MK348606.140.1条带回收失败
2.3 混合植物源性成分样本分析
2.3.1 测序数据统计分析
4个样本提取基因组DNA,PCR扩增后将扩增产物进行二代测序,对测序原始数据进行数据量和测序质量的统计,A1、A2、A32、A4样本的数据产出的有效序列条数分别为69 693、68 267、73 099、97 713(表4)。
表4 测序数据信息
Table 4 Sequencing data information

样本名称有效序列条数碱基数平均长度最短序列长度最长序列长度A169 69327 100 782388.86305444A268 26726 429 476387.15279430A373 09928 423 318388.83261451A497 71337 997 011388.86339441
2.3.2 OTU聚类分析及物种注释
OTU是在系统发生学或群体遗传学研究中,为了便于进行分析,人为给某一个分类单元(品系,属,种、分组等)设置的统一标志。通过聚类操作,将序列按照彼此的相似性分归为许多OTU。通常对97%相似水平下的OTU进行生物信息统计分析[23]。对4个样本进行OTU聚类分析,共产生13个OTU,4个样本OTU中序列数统计以及OTU对应的物种注释信息见表5,表中仅展示了丰度前5的OTU。
表5 样本OTU中序列数统计及物种注释
Table 5 Sequence numbers tatistics and species annotation of samples OTU

OTU编号A1A2A3A4物种注释信息OTU522 115327581616d__Viridiplantae;p__Streptophyta;c__norank_Streptophyta;o__Rosales;f__Rosaceae;g__Prunus;s__Prunus_pedunculata;OTU115 00168451 2031 950d__Viridiplantae;p__Streptophyta;c__norank_Streptophyta;o__Fabales;f__Fabaceae;g__Glycine;s__Glycine_soja;OTU21 2367101 18035 565d__Viridiplantae;p__Streptophyta;c__norank_Streptophyta;o__Fabales;f__Fabaceae;g__Vigna;s__Vigna_radiata;OTU67 941129372219d__Viridiplantae;p__Streptophyta;c__norank_Streptophyta;o__Fabales;f__Fabaceae;g__Arachis;s__Arachis_hypogaea;OTU442728557323 305d__Viridiplantae;p__Streptophyta;c__norank_Streptophyta;o__Solanales;f__Solanaceae;g__Solanum;s__Solanum_tarijense;
为得到每个OTU对应的物种分类信息,利用RDPclassifier进行物种分类注释。根据每个样本的分类学比对结果,选出优势物种的分类,从整个分类系统上了解测序的样本中优势物种和丰度差异。使用python的ete3 package绘制每个样本分类系统组成树状图。4个样本在不同分类水平下(域,门,纲,目,科,属,种)的物种种类数目统计如图4所示,种水平下物种类别与样本信息基本一致。

a-A1分类系统组成树;b-A2分类系统组成树;c-A3分类系统组成树;d-A4分类系统组成树
图4 分类学系统组成树状
Fig.4 Taxonomic system composition tree
3 结论
高通量测序使核酸测序成本大幅度下降,从而推动了生命科学各学科的发展[23]。检测植物掺假问题的方法中,分子生物学方法是较为重要的手段,包括酶联免疫技术、DNA条形码技术、实时荧光定量PCR技术[24]等。酶联免疫技术具有检测速度快、费用低廉、仪器简单易携等优点,广泛应用于现场检测,但只是检测的辅助手段,特异性和灵敏性有待提高。实时荧光定量PCR技术特异性强、灵敏度高、重复性好、全封闭反应、减少了产物的污染,同时也存在无法高通量筛查样本的问题,面对未知成分样本,检测过程复杂。本研究基于扩增子测序检测手段,发展针对食品中常见27种植物源性成分的高通量筛查技术,通过反复实验和大量实际样品验证,利用一代测序分析技术、二代测序分析技术对单一物种食品、混合物种食品中常见植物源性进行高通量、非定向筛查,从而解决植物源性食品掺假快速检测的问题。然而,本实验之外的其他物种的RbcL基因序列在该引物的扩增区域有无完全相同的现象,是否会导致难以区分某两种或几种物种还有待验证,同时为了规范植物食品样本信息,有效抓取海量数据,需要不断完善参考序列数据库的质量和完整性,为宏基因组技术在植物源性成分食品的掺假非定向筛查研究中广泛应用提供更加坚实的基础。
[1] 孙善峰, 黄晓宁, 韩北忠, 等.扩增子测序分析助力传统发酵食品微生物群落研究[J].中国酿造, 2019, 38(5):1-5.
SUN S F, HUANG X N, HAN B Z, et al.Amplicon sequencing analysis enhancing the understanding on microbial diversity of traditional Chinese fermented foods[J].China Brewing, 2019, 38(5):1-5.
[2] 刘伟, 张纪阳, 谢红卫.生物信息学[M].北京:电子工业出版社, 2014:5-15.
LIU W, ZHANG J Y, XIE H W.Bioinformations[M].Beijing:Publishing House of Electronics Industry, 2014:5-15.
[3] 颜亨梅, 钟文涛.动物捕食性天敌摄食分析方法的研究进展[J].生命科学研究, 2021, 25(1):1-8.
YAN H M, ZHONG W T.Research progress of feeding analysis methods for predatory animals[J].Life Science Research, 2021, 25(1):1-8.
[4] BUGLIONE M, MASELLI V, RIPPA D, et al. A pilot study on the application of DNA metabarcoding for non -invasive diet analysis in the Italian hare[J].Mammalian Biology, 2018, 88:31-42.
[5] JENSEN-VARGAS E, MARIZZI C.DNA barcoding for identification of consumer-relevant fungi sold in New York:A powerful tool for citizen scientists?[J].Foods(Basel, Switzerland), 2018, 7(6):87.
[6] XING B P, LIN H S, ZHANG Z L, et al.DNA barcoding for identification of fish species in the Taiwan Strait[J].PLoS One, 2018, 13(6):0198109.
[7] QASIM M, WANG B H, ZOU H S, et al.Phylogenetic relationship and genetic diversity of citrus psyllid populations from China and Pakistan and their associated Candidatus bacterium[J].Molecular Phylogenetics and Evolution, 2018, 126:173-180.
[8] HAMB CK P A, WEINGARTNER E, DALÉN L, et al.Spatial subsidies in spider diets vary with shoreline structure:Complementary evidence from molecular diet analysis and stable isotopes[J].Ecology and Evolution, 2016, 6(23):8 431-8 439.
CK P A, WEINGARTNER E, DALÉN L, et al.Spatial subsidies in spider diets vary with shoreline structure:Complementary evidence from molecular diet analysis and stable isotopes[J].Ecology and Evolution, 2016, 6(23):8 431-8 439.
[9] 白文明, 邢冉冉, 陈丽萍, 等.基于DNA条形码技术鉴别有毒鹅膏菌属物种[J].食品科学, 2021, 42(4):278-286.
BAI W M, XING R R, CHEN L P, et al.DNA barcoding for identification of toxic Amanita species[J].Food Science, 2021, 42(4):278-286.
[10] BENSON D A, CAVANAUGH M, CLARK K, et al.GenBank[J].Nucleic Acids Research, 2013, 41:D36-D42.
[11] COCHRANE G, ALDEBERT P, ALTHORPE N, et al.EMBL nucleotide sequence database:Developments in 2005[J].Nucleic Acids Research, 2006, 34(S1):D10-D15.
[12] MASHIMA J, KODAMA Y, KOSUGE T, et al.DNA data bank of Japan (DDBJ) progress report[J].Nucleic Acids Research, 2015, 44(D1):D51-D57.
[13] 张稚兰, 邢炳鹏.小球藻属DNA条形码的鉴定研究[J].海洋开发与管理, 2021, 38(2):69-76.
ZHANG Z L, XING B P.Assessment of candidate DNA sequences for barcoding of the genus Chlorellla[J].Ocean Development and Management, 2021, 38(2):69-76.
[14] LI X L, HONG M H.Aqueous extract of Dendrobium officinale confers neuroprotection against hypoxic-ischemic brain damage in neonatal rats[J].The Kaohsiung Journal of Medical Sciences, 2020, 36(1):43-53.
[15] JIN W T, JIN X H, SCHUITEMAN A, et al.Molecular systematics of subtribe Orchidinae and Asian taxa of Habenariinae (Orchideae, Orchidaceae) based on plastid matK, rbcL and nuclear ITS[J].Molecular Phylogenetics and Evolution, 2014, 77:41-53.
[16] 杨荣武. 分子生物学[M].南京:南京大学出版社, 2017:636-637.
YANG R W.Molecular Biology[M].Nanjing:Nanjing University Press, 2017:636-637.
[17] 姚丹丹, 陈佐, 朱军, 等.基于coxⅠ、ITS和rbcL基因序列的马尾藻系统进化分析[J].分子植物育种, 2020,20(4):1 153-1 162.
YAO D D, CHEN Z, ZHU J, et al.Genetic evolution analysis of Sargassum based on coxⅠ, ITS and rbcL gene sequences[J].Molecular Plant Breeding, 2020,20(4):1 153-1 162.
[18] FAWLEY M W, FAWLEY K P, CAHOON A B.Finding needles in a haystack-Extensive diversity in the eustigmatophyceae revealed by community metabarcode analysis targeting the rbcL gene using lineage-directed primers[J].Journal of Phycology, 2021, 57(5):1 636-1 647.
[19] 陈玉花, 撒切尔, 肖田梅, 等.蒙药蜀葵花HPLC指纹图谱及rbcL序列分子鉴定研究[J].中草药, 2020, 51(21):5 607-5 612.
CHEN Y H, SA Q E, XIAO T M, et al.Fingerprint and molecular identification of rbcL sequence of Mongolian medicine Althaeae Roseae Flos by HPLC[J].Chinese Traditional and Herbal Drugs, 2020, 51(21):5 607-5 612.
[20] 林晓霞, 鹿炎, 李会丽, 等.基于matK和rbcL序列的石斛属植物亲缘关系研究[J].生物学杂志, 2021, 38(1):46-50.
LIN X X, LU Y, LI H L, et al.Genetic relationship analysis of Dendrobium plants based on chloroplast matK and rbcL gene[J].Journal of Biology, 2021, 38(1):46-50.
[21] 赵琳娜, 胡凤月, 吴孝槐, 等.用于转基因检测的番木瓜基因组DNA提取方法的比较[J].现代食品科技, 2010, 26(2):188-191.
ZHAO L N, HU F Y, WU X H, et al.Study on DNA extraction from papaya for detection of genetically modified organism[J].Modern Food Science and Technology, 2010, 26(2):188-191.
[22] LANDEWEERT R, LEEFLANG P, KUYPER T W, et al.Molecular identification of ectomycorrhizal mycelium in soil horizons[J].Applied Environmental Microbiology, 2003, 69(1):327-333.
[23] 夏梦雷, 杨帆, 陆锴, 等.高通量测序技术在传统发酵食品微生物群落中的应用研究[J].中国酿造, 2021, 40(5):1-7.
XIA M L, YANG F, LU K, et al.Advances in the application of high-throughput sequencing technology in traditional fermented foods[J].China Brewing, 2021, 40(5):1-7.
[24] 翟清燕, 任易婕, 郑世超, 等.三种植物源性成分阳性质粒的构建及检测方法的验证[J].食品与发酵工业, 2020, 46(17):237-241.
ZHAI Q Y, REN Y J, ZHENG S C, et al.Verification of detection method and construction of plasmid molecule for three plant-derived components[J].Food and Fermentation Industries, 2020, 46(17):237-241.