生胞梭菌(Clostridium sporogenes)源于1908年METCHNIKOFF提出的Bacillus sporogenes[1],由BERGEY等[2]于1923年正式命名为Clostridium sporogenes,收录于1980年细菌名称的批准名录(approed lists 1980)中[3]。生胞梭菌(C.sporogenes)种名sporogenes(sporos-genes)即spore-producing,译为产芽胞,故又名产芽胞梭状芽胞杆菌、产胞梭菌。
生胞梭菌在食品、医药等行业应用广泛。生胞梭菌可在食品行业检测中作为对照菌株。生胞梭菌是生物危害程度4类的菌种,在通常情况下不会引起人类或者动物疾病,其从表型和基因型方面都与肉毒梭菌(Clostridium botulinum)亲缘关系最近,可用做肉毒梭菌(C.botulinum)检测试验中培养基验证、方法学验证和样品增菌阶段的阳性对照菌株[4]。生胞梭菌在医药等行业可作为药品控制菌在微生物污染定性检测中起重要作用[5]。我国药典控制菌检查类群包括生胞梭菌CICC 10385(=CMCC 64941)等[6],用于无菌检查法通过微生物培养检查药品是否无菌,其他如欧洲药典、美国药典、日本药局方也有用到生胞梭菌[7]。我国2020版药典《1101 无菌检查法》和《1106 非无菌产品微生物限度检查:控制菌检查法》中继续使用生胞梭菌CICC 10385(=CMCC 64941)[8]。生胞梭菌亦可用作生物指示剂,如验证一个灭菌工艺系统能否使产品灭菌后达到无菌要求,可采用生胞梭菌进行相应的物理性验证及生物指示剂验证[9-10]。我国2020版药典《1421 灭菌法》微生物挑战试验中根据灭菌工艺选择适宜的生物指示剂,对于热不稳定性物品灭菌常用的生物指示剂为生胞梭菌。生胞梭菌CICC 10385(=CMCC 64941)应用广泛,对其进行全基因组测序(whole genome sequencing,WGS)及鉴定具有重要意义。
本研究针对梭菌属中的生胞梭菌,以中国工业微生物菌种保藏管理中心(China Center of Industrial Culture Collection,CICC)保藏菌株CICC 10385(=CMCC 64941)为研究对象,进行基于WGS的平均核苷酸一致性(aerage nucleotide identity,ANI)种水平鉴定,为明确菌种分类学地位、确保使用安全等方面提供依据,同时为研究其生物学功能奠定基础,为梭菌属(Clostridium sp.)相关菌种的鉴定提供理论依据。
1 材料与方法
1.1 材料与试剂
1.1.1 实验菌株
实验菌株CICC 10385为CICC保藏菌株,CICC 10385菌株等同编号为CMCC 64941。
1.1.2 试剂
卵黄琼脂培养基,北京陆桥技术有限责任公司;细菌基因组DNA提取试剂盒(离心柱型),天根生化科技(北京)有限公司;2×PCR Mix、DNA Marker,北京全式金生物技术有限公司;API 20 A检验试剂条、ITEK ANC鉴定卡,法国梅里埃公司;其他化学试剂均为进口分析纯产品。
1.2 仪器与设备
AC2-4S1生物安全柜,新加坡ESCO公司;5415D离心机,德国Eppendorf公司;professional TRIO 48型PCR仪,德国Biometra公司;BioDrop μLite微量核酸蛋白分析仪,英国Biochrom公司;Gel Doc EZ凝胶成像分析仪,美国Bio-Rad公司;BGISEQ-500全基因组测序仪,深圳华大智造科技有限公司(MGI Tech Co.Ltd.)。
1.3 实验方法
1.3.1 菌株转接、活化与保藏
采用平板涂布法对冻干管菌种CICC 10385进行转接活化。在生物安全柜中使用无菌枪头吸取无菌水置于冻干管中混匀,涂布卵黄琼脂培养基平板,置于加装厌氧袋的厌氧罐后密封,35 ℃培养48 h。待单菌落出现后,划线法转接至卵黄琼脂培养基斜面培养,置于4 ℃冰箱保藏。同时将活化的菌株以甘油管形式保藏于CICC备用。
1.3.2 形态学观察
将CICC 10385接种于卵黄琼脂培养基平板,置于加装厌氧袋的厌氧罐后密封,35 ℃培养48 h,观察其在卵黄琼脂培养基上的宏观形态特征,并利用光学显微镜及电子显微镜观察菌体微观形态特征[11]。
1.3.3 生理生化特征检测
采用API 20A检测试剂条及ITEK 2 Compact鉴定系统中的ITEK ANC鉴定卡,依据其说明书中操作步骤对CICC 10385的酶活性、碳源利用等生理生化特征进行检测。
1.3.4 WGS和ANI分析
1.3.4.1 WGS和组装
采用细菌基因组DNA提取试剂盒提取CICC 10385基因组DNA,并采用微量核酸蛋白分析仪检测基因组DNA质量和浓度,确保符合文库构建要求。检测合格的基因组DNA进行测序文库构建与上机测序。获得测序数据后,对序列数据进行预处理,过滤掉外源序列以及质量较低的序列信息,获得较高质量的序列信息,将质控后的数据使用SPAdes 3.11.0软件进行拼接组装获得基因组序列及基因组序列总长度、contigs数量、(G+C) mol%含量等参数值。
1.3.4.2 16S rRNA基因预测及序列系统发育分析
使用RNAmmer 1.2软件进行全基因组中16S rRNA基因预测,获得序列通过模式菌株序列数据库进行序列比对,获得同近缘种序列相似性[12-14]。采用MEGA 5.0软件中的p-distance模型计算进化距离,邻接法(neighbor-joining)构建16S rRNA基因序列系统发育树[15],自展值(bootstrap alues)设为1 000分析评估进化树的稳定性,进行菌株CICC 10385的16S rRNA基因序列系统发育分析。
1.3.4.3 ANI分析
进行CICC 10385全基因组ANI分析[16],将16S rRNA基因序列相似性>98.65%的模式菌株作为ANI分析比对参考模式菌株,从NCBI数据库下载参考模式菌株全基因组序列。利用ANI Calculator(https://www.ezbiocloud.net/tools/ani)进行CICC 10385全基因组序列和参考模式菌株序列比对,若实验菌株仅与1个参考模式菌株序列ANI值大于95%~96%[17-18],则判定实验菌株与此参考模式菌株为同1个种。通过核心基因系统发育分析(core-genome multi-locus sequence typing,cgMLST),获得CICC 10385与近缘模式菌株全基因组序列的共有基因,采用TreeBeST 1.9.2中PHYML基于最大似然法(maximum likelihood)构建系统发育树,自展值设为100。采用Genome-to-Genome Distance Calculator(GGDC 3.0,http://ggdc.dsmz.de/ggdc.php)计算CICC 10385与近缘模式菌株的DNA-DNA杂交(DNA-DNA hybridization,DDH)值,70%可作为原核生物种水平判定阈值[19]。
2 结果与分析
2.1 菌株CICC 10385的转接活化与形态学观察
经过冻干管打管转接活化实验菌株CICC 10385,在卵黄琼脂培养基上35 ℃厌氧培养48 h,菌落直径为3~6 mm,菌落白色、近圆形、扁平、边缘不规则,菌落珍珠层样,周围窄虹彩样薄层(图1-a);光学显微镜观察显示菌体直杆状或稍弯曲,(0.5~0.6) μm×(1.1~4.1) μm,单个或成对排列,芽胞椭圆形,近端生,胞囊膨大,革兰氏阳性(图1-b)。电子显微镜菌体形态见图1-c。结果显示CICC 10385的形态特征符合伯杰氏系统细菌学手册中对生胞梭菌的基本特征描述[11]。

a-宏观形态学观察;b-光学显微镜观察;c-扫描电镜观察
图1 CICC 10385的形态学观察结果
Fig.1 Morphological obseration result of CICC 10385
2.2 菌株CICC 10385生理生化特征
经API 20A检测试剂条和ITEK 2 Compact鉴定系统中的ITEK ANC鉴定卡对菌株CICC 10385的酶活性、利用碳源产酸等生理生化特征进行检测。API 20A检测试剂条鉴定系列有21个测定指标,能快速、简便地对厌氧菌进行生化鉴定。API 20A检测结果显示,菌株CICC 10385能水解明胶与七叶灵,能利用麦芽糖和海藻糖产酸,不能利用葡萄糖、甘露醇、乳糖、蔗糖、麦芽糖、柳醇、木糖、阿拉伯糖、甘油、纤维二糖、甘露糖、松叁糖、棉子糖、山梨醇、鼠李糖、海藻糖等产酸。采用API 20A鉴定检测与生胞梭菌和肉毒梭菌相似率均为99.9%。补充实验显示具有牛奶不凝固、蛋白胨-酵母膏-葡萄糖+胆碱培养基不生长、淀粉不水解等特征。API 20A检测结果详见表1。
表1 CICC 10385的API 20A检测结果
Table 1 API 20A test result of CICC 10385

检测项目结果检测项目结果检测项目结果检测项目结果吲哚产生-脲酶-葡萄糖-甘露醇-乳糖-蔗糖-麦芽糖+柳醇-木糖-阿拉伯糖-明胶+七叶灵+甘油-纤维二糖-甘露糖-松叁糖-棉子糖-山梨醇-鼠李糖-海藻糖+接触酶-牛奶(凝固)-蛋白胨-酵母膏-葡萄糖+胆碱培养基生长-淀粉水解-
注:“+”,阳性;“-”,阴性(下同)
ITEK 2 Compact鉴定系统中的ITEK ANC厌氧菌鉴定卡基于已建立的生化方法并采用新开发的底物,共有36种检测碳源利用和酶活性的生化试验,能快速、简便地对厌氧菌进行生化鉴定[20]。ITEK 2 Compact鉴定系统检测结果显示,CICC 10385能产L-脯氨酸芳胺酶、L-吡咯烷酮芳胺酶,能利用麦芽三糖,不能利用D-半乳糖、D-葡萄糖、D-甘露糖、D-麦芽糖等,与生胞梭菌鉴定概率百分比[21]为99%,ITEK 2 Compact检测结果详见表2。
表2 CICC 10385的ITEK 2 Compact检测结果
Table 2 ITEK 2 Compact test result of CICC 10385

检测项目结果检测项目结果检测项目结果检测项目结果D-半乳糖-L-脯氨酸芳胺酶+丙氨酸-苯丙氨酸-脯氨酸芳胺酶-蔗糖-尿素酶-5-溴-4-氯-3-吲哚-α-D-半 乳糖苷-麦芽三糖+5-溴-4-氯-3-吲哚-α-D-甘 露糖苷-d-核糖-亮氨酸芳胺酶-L-吡咯烷酮芳胺酶+D-葡萄糖-熊果苷-5-溴-4-氯-3-吲哚-β-D- 葡萄糖苷酸-β-甘露糖苷酶-七叶苷水解-α-L-岩藻糖苷酶-苯基磷酸盐-ELLMAN试剂-D-纤维二糖-D-甘露糖-N-乙酰-D-氨基葡萄糖-β-半乳糖吡喃糖苷酶吲 哚酚-精氨酸 GP-β-D-岩藻糖苷酶-磷酸酶-α-L-阿拉伯糖苷-苯丙氨酸芳胺酶-酪氨酸芳胺酶-D-麦芽糖-5-溴-4-氯-3-吲哚-β-D-葡 萄糖苷-α-阿拉伯糖苷酶-丙酮酸盐-5-溴-4-氯-3-羟基吲哚-β- N-乙酰-葡糖胺-L-阿拉伯糖-D-木糖-
2.3 WGS和ANI分析
2.3.1 WGS和组装
采用全基因测序仪对CICC 10385进行全基因组从头测序,测得全基因组大小为4 252 276 bp,含有58个contigs,基因组(G+C)mol%为27.9%。NCBI登录号为GCA_013867655.1,序列片段归档数据库(sequence read archie,SRA)中登录号为SRR9967484。
2.3.2 16S rRNA基因预测及序列系统发育分析
通过RNAmmer 1.2预测CICC 10385全基因组中16S rRNA基因序列为1 500 bp,将该序列通过模式菌株序列数据库进行序列比对,同生胞梭菌(C.sporogenes)、肉毒梭菌(C.botulinum)、孔氏梭菌(C.combesii)、C.tepidum模式菌株序列相似性分别为99.86%、99.72%、99.72%、99.02%,同其余参考模式菌株序列相似性均低于98.65%。以Oxobacter pfennigii DSM 3222T(X77838)序列为外群,邻接法(neighbor-joining)构建CICC 10385与其近缘模式菌株的系统发育树(图2)。结果显示通过16S rRNA基因序列系统发育分析可以将菌株CICC 10385鉴定为梭菌属(Clostridium sp.)。

图2 邻接法构建CICC 10385和近缘种16S rRNA 基因序列系统发育树
Fig.2 Neighbor-joining phylogenetic tree based on 16S rRNA gene sequences of CICC 10385 and related species
2.3.3 ANI分析
采用ANI Calculator(https://www.ezbiocloud.net/tools/ani)进行CICC 10385和梭菌属(Clostridium sp.)内近缘模式菌株的全基因组序列比对分析[22]。结果显示CICC 10385同生胞梭菌DSM 795T(GCA_001020205.1)模式菌株序列ANI值为99.39%,高于ANI用于细菌种水平鉴定的判定阈值95%~96%[17-18],同肉毒梭菌(C.botulinum)ATCC 25763T(GCA_001276985.1)、孔氏梭菌(C.combesii)DSM 20696T(GCA_002749945.1)、C.tepidum IEH 97212T(GCA_002008345.1)模式菌株ANI值分别为91.88%、91.87%、88.16%,与梭菌属其余模式菌株ANI值均低于95%,判定CICC 10385与生胞梭菌为同1个种。通过cgMLST分析,获得CICC 10385与近缘模式菌株885个核心基因序列系统发育树(图3),在cgMLST系统发育树中,CICC 10385与生胞梭菌以100%支持率聚在同一分支,且与梭菌属内其他菌种明显区分开。采用GGDC 3.0计算获得CICC 10385同生胞梭菌DSM 795T(GCA_001020205.1)模式菌株序列DDH值为94.8%,同其余参考模式菌株DDH值均低于70%,符合种水平判定标准。综上,基于WGS的ANI分析,菌株CICC 10385被鉴定为生胞梭菌。

图3 最大似然法构建CICC 10385和近缘种核心 基因序列系统发育树
Fig.3 Maximum likelihood phylogenetic tree based on core-genome sequences of CICC 10385 and related species
3 结论与讨论
本研究采用基于WGS的ANI分析对菌株CICC 10385进行种水平鉴定并首次获得生胞梭菌CICC 10385(=CMCC 64941)的全基因组序列。采用微生物学传统分离培养方法对35 ℃条件下的CICC 10385进行转接活化,并通过形态学和生理生化特征分析获得其基础信息。采用BGISEQ-500全基因测序仪进行WGS,SPAdes软件进行序列拼接获得全基因组序列。采用RNAmmer软件进行16S rRNA基因预测,并对其进行序列比对分析,获得与CICC 10385亲缘关系较近菌种信息。采用ANI Calculator软件对CICC 10385和近缘菌种进行全基因组ANI等分析。获得CICC 10385的全基因组大小为4 252 276 bp,含有58个contigs,基因组(G+C)mol%为27.9%。16S rRNA基因序列分析与生胞梭菌(C.sporogenes)、肉毒梭菌(C.botulinum)、孔氏梭菌(C.combesii)和C.tepidum最为近缘,ANI分析与生胞梭菌DSM 795T(GCA_001020205.1)的ANI值为99.39%,与梭菌属其余菌种ANI值均低于95%,通过cgMLST系统发育分析与生胞梭菌以100%支持率聚在同一分支,且与其他菌种明显区分开,同生胞梭菌DDH值为94.8%,同其余参考模式菌株DDH值均低于70%,符合种水平判定标准,可明确将CICC 10385鉴定为生胞梭菌。经研究表明基于WGS的ANI分析是准确有效的生胞梭菌种水平鉴定方法。
菌种鉴定通常基于表型和基因型的多相分类鉴定技术方法[23],随着测序技术的发展,分子DNA鉴定技术广泛应用,WGS也变得越来越普遍。自2018年1月起,International Journal of Systematic and Eolutionary Microbiology要求发表微生物新种需提供基因组测序数据,并在分类中增加这部分的描述。基于WGS的ANI值95%~96%与DDH值70%、16S rRNA基因序列相似性98.65%相对应,可作为原核生物种水平的判定阈值[17-18]。全基因组序列数据不仅能够提供菌株的分类鉴定信息,还能提供菌株毒力因子、临床抗微生物药物产生或耐受以及已知有毒代谢物产生等其他安全特性信息。随着微生物在各领域的广泛应用,菌株安全性评价的重要性会愈加凸显,基于WGS的鉴定方法将提供重要数据信息,其应用会愈加广泛。
生胞梭菌(C.sporogenes)在食品、医药等行业应用广泛,采用基于WGS的ANI分析对其进行种水平鉴定研究,明确其菌种分类学地位具有重要意义。本研究对CICC 10385的分类地位进行基于全基因组测序平均核苷酸一致性的准确鉴定,为针对该菌及梭菌属(Clostridium sp.)等相关菌种鉴定研究提供理论依据,为开展其生物学功能的相关研究奠定基础。
[1] HELLER H H.Certain genera of the Clostridiaceae:Studies in pathogenic anaerobes.[J].Journal of Bacteriology, 1922, 7(1):1-38.
[2] BERGEY D H, HARRISON F C, BREED R S, et al.Bergey’s Manual of Determinatie Bacteriology[M].Baltimore:Williams & Wilkins Company, 1923.
[3] SKERMAN B D, SNEATH P H A, MCGOWAN .Approed lists of bacterial names[J].International Journal of Systematic and Eolutionary Microbiology, 1980, 30(1):225-420.
[4] 中国食品发酵工业研究院, 中国工业微生物菌种保藏管理中心.食品安全国家标准 食品微生物检验标准菌株图鉴[M].北京:中国轻工业出版社, 2014.
China National Research Institute of Food & Fermentation Industries, China Center of Industrial Culture Collection.National Food Safety Standard Food Microbiological Inspection Standard Strain Illustration[M].Beijing:China Light Industry Press, 2014.
[5] 谢文, 赵宏大, 纪绍梅.关于我国药品无菌试验用培养基质量的现状分析及改进建议[J].中国药事, 2010, 24(1):67-68.
XIE W, ZHAO H D, JI S M.Analysis on the current situation of the quality of media with sterility test and recommendations for improement[J].Chinese Pharmaceutical Affairs, 2010, 24(1):67-68.
[6] 郑小玲, 杨丹燕, 李珏, 等.现代菌种鉴定技术在药品控制菌检查中的比较[J].中国卫生检验杂志, 2015, 25(5):658-660.
ZHENG X L, YANG D Y, LI J, et al.Comparison of modern strain identification technologies in drug control bacteria test[J].Chinese Journal of Health Laboratory Technology, 2015, 25(5):658-660.
[7] 顾兆姝. 欧、美、日、中现行版药典无菌试验的差异[J].中国药学杂志, 2005, 40(5):390-392.
GU Z S.Differences of sterility test in current editions of pharmacopoeias in Europe, America, Japan and China[J].Chinese Pharmaceutical Journal, 2005, 40(5):390-392.
[8] 国家药典委员会编. 中华人民共和国药典:2020年版[M].四部.北京:中国医药科技出版社, 2020.
Chinese Pharmacopoeia Commission.Pharmacopoeia of the People’s Republic of China 2020 edition[M].4th ed.Beijing:China Medical Science Press, 2020.
[9] 熊海元, 崔强.生孢梭菌孢子作为生物指示剂的研究[J].中国医药工业杂志, 2003, 34(7):354-356.
XIONG H Y, CUI Q.Study on Clostridium sporogenes spores used as a biological indicator[J].Chinese Journal of Pharmaceuticals, 2003, 34(7):354-356.
[10] 熊海元, 潘友文, 崔强, 等.生孢梭菌孢子作为生物指示剂用于残存概率灭菌工艺验证的可行性[J].中国药事, 2003, 17(9):577-579.
XIONG H Y, PAN Y W, CUI Q, et al.Use of Clostridium sporogenes spores as biological indicator during sterilization cycle alidations[J].Chinese Pharmaceutical Affairs, 2003, 17(9):577-579.
[11] BRENNER D J, KRIEG N R, STALEY J T, et al.Bergey’s Manual of Systematic Bacteriology[M].Boston:Springer US, 2005.
[12] SULAIMAN I M, MIRANDA N, SIMPSON S.MALDI-TOF mass spectrometry and 16S rRNA gene sequence analysis for the identification of foodborne Clostridium spp.[J].Journal of AOAC International, 2021, 104(5):1 381-1 388.
[13] SEO B, JEON K, BAEK I, et al.Clostridium fessum sp.no., isolated from human faeces[J].International Journal of Systematic and Eolutionary Microbiology, 2021, 71(1):004579.
[14] DOBRITSA A P, KUTUMBAKA K K, WERNER K, et al.Clostridium tepidum sp.no., a close relatie of Clostridium sporogenes and Clostridium botulinum group I[J].International Journal of Systematic and Eolutionary Microbiology, 2017, 67(7):2 317-2 322.
[15] TAMURA K, PETERSON D, PETERSON N, et al.MEGA5:Molecular eolutionary genetics analysis using maximum likelihood, eolutionary distance, and maximum parsimony methods[J].Molecular Biology and Eolution, 2011, 28(10):2 731-2 739.
[16] LI X, LI Y F, ZANG M Y, et al.Complete chloroplast genome sequence and phylogenetic analysis of Quercus acutissima[J].International Journal of Molecular Sciences, 2018, 19(8):2 443.
[17] RICHTER M, ROSSELL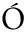 -M
-M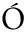 RA R.Shifting the genomic gold standard for the prokaryotic species definition[J].Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(45):19 126-19 131.
RA R.Shifting the genomic gold standard for the prokaryotic species definition[J].Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(45):19 126-19 131.
[18] KIM M, OH H S, PARK S C, et al.Towards a taxonomic coherence between aerage nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J].International Journal of Systematic and Eolutionary Microbiology, 2014, 64(Pt 2):346-351.
[19] MEIER-KOLTHOFF J P, CARBASSE J S, PEINADO-OLARTE R L, et al.TYGS and LPSN:A database tandem for fast and reliable genome-based classification and nomenclature of prokaryotes[J].Nucleic Acids Research, 2022, 50(D1):D801-D807.
[20] SANCHEZ RAMOS L, RODLOFF A C.Identification of Clostridium species using the ITEK® MS[J].Anaerobe, 2018, 54:217-223.
[21] 刘杰, 赵满仓, 周海峰.ITEK Ⅱ全自动微生物分析系统应用问题分析[J].中国医疗设备, 2013, 28(9):119-121.
LIU J, ZHAO M C, ZHOU H F.Analysis of application problems of ITEK Ⅱ automatic microbe identification system[J].China Medical Deices, 2013, 28(9):119-121.
[22] YOON S H, HA S M, LIM J M, et al.A large-scale ealuation of algorithms to calculate aerage nucleotide identity[J].Antonie an Leeuwenhoek, 2017, 110(10):1 281-1 286.
[23] TINDALL B J, ROSSELL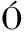 -M
-M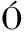 RA R, BUSSE H J, et al.Notes on the characterization of prokaryote strains for taxonomic purposes[J].International Journal of Systematic and Eolutionary Microbiology, 2010, 60(Pt 1):249-266.
RA R, BUSSE H J, et al.Notes on the characterization of prokaryote strains for taxonomic purposes[J].International Journal of Systematic and Eolutionary Microbiology, 2010, 60(Pt 1):249-266.