浓香型白酒是我国公认的五大主要香型白酒之一[1],具有芳香浓郁、绵柔甘冽和尾净余长等特点,深受消费者青睐,产量和市场份额在全国白酒行业中占有主导地位[2]。酒醅作为一种特定的发酵基质,是酿造微生物的生长繁殖地,也是其代谢产物形成的重要场所[3]。
白酒酿造过程中营养物质的利用和代谢产物的积累使得窖内微环境发生变化,如pH值降低、酒精浓度增加、氧气含量下降等,会使酿造微生物受到多种胁迫,进而限制其生长,但是仍然有少部分微生物适应了这种发酵环境而大量生长繁殖[4]。目前,利用传统可培养技术和免培养技术(PCR-DGGE、高通量测序技术)研究浓香型白酒发酵过程中微生物的群落结构,发现糟醅中优势细菌主要是乳杆菌和乙酸菌[5]。在窖池发酵3 d后,含有乳杆菌属等9种优势菌属,发酵15 d后,乳杆菌为主要优势菌[6],在夏天和冬天发酵糟醅中也都发现乳杆菌为优势菌,且耐酸乳杆菌(Lactobacillus acetotolerans)为糟醅中的优势种[7],耐酸乳杆菌在上、中、下层窖泥中所占比例分别为20.6%、23.8%和36.6%[8],耐酸乳杆菌也是窖泥中乳酸菌的优势种。WANG等[9]采用PCR-DGGE和16S rRNA基因克隆文库,分析白酒的细菌群落结构,发现耐酸乳杆菌为发酵后期的优势菌种;LI等[10]研究发现在白酒发酵9~40 d过程中,耐酸乳杆菌在细菌菌群中占比高达80%以上。耐酸乳杆菌最早由ENTANI等[11]从米醋中分离得到,2015年TOH等[12]首次公开了一株从日本清酒中分离鉴定出L.acetotolerans RIB 9124(NBRC 13120)菌株的全基因组序列。在酸胁迫下耐酸乳杆菌能够生长代谢,且相对于其他微生物具有更好的酸耐受性,其可能存在某些应答酸胁迫的机制及一些未知功能。
此外,耐酸乳杆菌以乳酸、乙酸等有机酸为糖酵解终产物,这些有机酸直接或间接地影响着白酒的品质和风味[13-14]。各种有机酸是浓香型白酒主体香味的酯类物质(丁酸乙酯、乳酸乙酯、己酸乙酯、乙酸乙酯)的前体物,对白酒的香气、滋味和口感都有重要的影响,适量的有机酸使酒体丰满协调、回味悠长,并且有利于白酒中苦味物质的消除和促进新酒的陈化[15]。刘文容[16]发现耐酸乳杆菌是造成黄酒酸败的主要微生物,能使黄酒中的乳酸含量提高403.9%,乙酸含量提高382.9%,说明耐酸乳杆菌具有良好的产酸特性。因此,分离筛选浓香型白酒中耐酸乳杆菌,研究其耐酸和产酸特性对浓香型白酒发酵生产白酒具有重要意义。本研究对浓香型白酒酒醅中耐酸乳杆菌进行分离鉴定和分析其耐酸、产酸特性,以期为浓香型白酒风味和品质的提高以及后期酸胁迫应答机制的研究提供理论基础。
1 材料与方法
1.1 试验样品
本试验样品为浓香型白酒窖池发酵出窖酒醅,从四川省某浓香型白酒酒厂采集,采集后放置于无菌封口袋中,4 ℃保存。
1.2 培养基与试剂
MRS液体培养基(g/L):牛肉膏10.0,蛋白胨10.0,酵母浸出粉5.0,葡萄糖20.0,乙酸钠5.0,柠檬酸氢二铵2.0,KH2PO4 2.0,MgSO4·7H2O 0.58,MnSO4·4H2O 0.25,吐温-80 1.0 mL,121 ℃灭菌20 min。
MRS固体培养基:MRS培养基中添加20 g/L琼脂。
细菌基因组DNA提取试剂盒,北京索莱宝科技有限公司;PCR扩增试剂与合成扩增引物27F和1492R、rpoA-21-F和rpoA-22-R、pheS-21-F和pheS-22-R,生工生物工程(上海)股份有限公司。
1.3 仪器与设备
FA2004 N电子天平,上海菁海仪器有限公司;ZQP-75G台式全温振荡培养箱,天津市莱玻特瑞仪器设备有限公司;JJ-CJ-1FD洁净工作台,苏州市金净净化设备科技有限公司;YXQ-100A立式压力蒸汽灭菌锅,上海博迅实业有限公司医疗设备厂;E-201-C-9型pH计,上海鸿盖仪器有限公司;Analytic jena FlexCycler型PCR基因扩增仪,美国贝克曼库尔特公司;722 N可见分光光度计,上海仪电分析仪器有限公司;1260高效液相色谱仪,安捷伦科技有限公司;S-3400 N扫描电子显微镜,HITACHI(日立)公司。
1.4 乳酸菌的分离和纯化
称取酒醅样品10 g,加至100 mL生理盐水中,30 ℃、160 r/min摇床振荡30 min。将菌液进行梯度稀释,分别取10-1、10-2、10-3、10-4梯度稀释液100 μL涂布于MRS固体培养基(pH 5,下同)上,分别在30 ℃厌氧条件下静置培养6 d。待菌落长出后,根据菌落的形态学特征,挑取菌落形态不同的菌株在MRS固体培养基上划线分离纯化,直至固体培养基上呈现无其他杂菌的菌落。将纯化后的乳酸菌菌株编号,然后挑取单菌落接种于20 mL MRS液体培养基中,30 ℃厌氧160 r/min振荡培养,待培养基浑浊后于25%(体积分数)甘油中-80 ℃保存。
1.5 菌株形态学鉴定
参考《伯杰氏细菌鉴定手册》[17],观察单菌落的大小、颜色、形状、边缘和湿润程度等菌落特征。挑选分离纯化后的单菌落进行革兰氏染色,利用光学显微镜和扫描电子显微镜,观察菌体形态特征。扫描电子显微镜样品处理方法[18]:收集菌体,加入2.5%戊二醛在室温下固定2~4 h,8 000 r/min离心15 min,弃上清液。用磷酸盐缓冲液(pH 7.2)漂洗2次,8 000 r/min离心15 min,弃上清液。30%、50%、70%、80%、90%、95%、100%乙醇梯度离心脱水,每次脱水时间为8~10 min,8 000 r/min离心15 min,弃上清液,脱水后对菌泥进行CO2临界点干燥、粘样、镀膜,最后上镜观察。
1.6 菌株生理生化鉴定
使用乳酸菌生化鉴定条对乳酸菌进行生化鉴定。挑取单菌落制成菌悬液,将100 μL菌悬液分别接种在七叶苷、纤维二糖、麦芽糖、甘露醇、麦芽糖、水杨苷、山梨醇、蔗糖、棉子糖、菊糖和乳糖培养基中,37 ℃培养24~48 h后,观察不同反应的颜色变化,七叶苷培养基中颜色变黑为阳性,其余培养基中颜色变黄为阳性。过氧化氢酶试验中,取一环菌涂于干净的载玻片上,滴加1~2滴3%(体积分数)H2O2观察起泡现象。
1.7 菌株分子生物学鉴定
使用细菌基因组DNA提取试剂盒,按照操作手册提取细菌DNA,以提取DNA为模板,利用细菌16S rDNA的一对通用扩增引物27F(5′-AGAGTTTGATCATGGCTCAG-3′)和1492R(5′-TACGGTTACCTTGTTACGACTT-3′),对菌株的16S rDNA基因PCR扩增[19]。以及两对管家基因rpoA-21-F (5′-ATGATYGARTTTGAAAAACC-3′)和rpoA-22-R (5′-ACYTTATCATNTCWGYTC-3′);pheS-21-F(5′-CAYCCNGCHCGYGAYATGC-3′)和pheS-22-R(5′-CCWARCCRAARGCAAARCC-3′),对菌株的rpoA和pheS基因PCR扩增[20]。PCR扩增体系为25 μL:10.5 μL ddH2O,12.5 μL 2×Taq PCR Master Mix,上下游引物(10 μmol/L)各0.5 μL,DNA模板1 mL。16S rDNA 基因PCR扩增程序:95 ℃预变性10 min;94 ℃ 30 s,55 ℃ 45 s,72 ℃ 1 min,33个循环;72 ℃延伸10 min。rpoA和pheS基因PCR扩增程序:95 ℃预变性5 min;95 ℃ 60 s,46 ℃ 135 s,72 ℃ 75 s,3个循环;95 ℃ 35 s,46 ℃ 75 s,72 ℃ 75 s,30个循环;72 ℃延伸7 min。PCR扩增产物取1 μL在1%(质量分数)的琼脂糖凝胶中电泳检测,具有明亮条带的PCR产物送上海生工生物工程有限公司测序,将扩增片段的测定序列与NCBI(http://www.ncbi.nlm.nih.go/)中已知菌株序列进行BLAST同源性对比。
1.8 修正的Gompertz生长模型分析
将筛选出的耐酸乳杆菌平板划线活化2次后接入MRS液体培养基,30 ℃厌氧条件下160 r/min振荡培养至生长对数中期(OD600=1)。以10%的接种量把种子液接入pH 4的MRS液体培养基,30 ℃厌氧条件下160 r/min振荡培养,每隔12 h取样,测定其在在600 nm下的吸光度值。设置空白对照(灭菌并未接种的MRS液体培养基),利用修正的Gompertz模型来拟合耐酸乳杆菌的生长曲线,修正的Gompertz模型[21]如公式(1)所示:
Yt=y0+aexp{-exp[-k(x-xc)]}
(1)
式中:Yt,t时刻菌体OD600值;y0,初始菌体OD600值;k,耐酸乳杆菌最大生长速率;xc,达到最大生长速率所需时间,该值越小,耐酸乳杆菌的耐酸性越强;a,稳定期与初始值之间的差距。
1.9 生长曲线和产酸曲线测定
对筛选出耐酸性强的耐酸乳杆菌,经平板划线活化2次后接入MRS液体培养基,30 ℃厌氧条件下160 r/min摇床振荡培养至生长对数中期(OD600=1)。分别用盐酸、乳酸和乙酸调节MRS液体培养基pH至3、4、5、6,按10%(体积分数,下同)接种量接种于50 mL MRS液体培养基中,30 ℃厌氧条件下160 r/min摇床振荡培养,每隔12 h取样,以对应pH并未接种的培养基为空白对照,测定600 nm下的吸光度值,同时测定发酵液中的pH,以发酵时间为横坐标,OD600和pH为纵坐标分别绘制生长曲线和产酸曲线。
1.10 最适生长pH测定
乳酸菌平板划线活化2次后接入MRS液体培养基,30 ℃厌氧培养至生长对数中期(OD600=1)后作为种子液,按2%的接种量分别接入利用乳酸调pH为4、5、6、7、8的新鲜MRS液体培养基中,30 ℃厌氧条件下160 r/min摇床振荡培养48 h,测定培养液在600 nm下的吸光度值。
1.11 有机酸组成和含量的测定
收集菌株在盐酸、乳酸和乙酸酸化的MRS液体培养基中生长至稳定期的发酵液,采用高效液相色谱法测定有机酸的种类和含量,参考文献[22]的方法,并略作修改。
样品前处理:取1 mL发酵液于离心管中10 000 r/min离心2 min,取0.1 mL上清液于2 mL离心管中,用0.1%(体积分数)的磷酸水溶液稀释至1 mL,振荡均匀,最后用0.22 μm滤膜过滤后作为待测样品进行分析。
色谱柱:Agilent ZORBAX SB-Aq(250 mm×4.6 mm,5 μm);色谱条件:流动相为0.1%的磷酸水溶液,流速1 mL/min,紫外线检测波长214 nm,柱温30 ℃。
1.12 数据处理
所有试验均进行3次重复。采用Excel 2016对数据进行统计和制表,Origin 2019用于图形的绘制和修正的Gompertz模型拟合。SPSS 25.0进行单因素方差分析、新复极差法多重比较。
2 结果与分析
2.1 酒醅中耐酸乳杆菌的分离及鉴定
2.1.1 菌株分离与形态学鉴定
从浓香型白酒酒醅中初步分离出3株疑似耐酸乳杆菌,其中FBKL1.0203在MRS培养基平板上的单菌落呈乳白色、圆形、表面湿润、有皱褶、中间有突起,菌落直径约为2.46 mm;FBKL1.0204的单菌落呈乳白色、圆形、边缘不整齐、表面湿润粗糙、有皱褶、中间有突起,菌落直径约为3.26 mm;FBKL1.0205的单菌落呈白色、边缘半透明、不规则圆形、表面粗糙、有皱褶、中间突起,菌落直径约为2.34 mm。镜检结果均为杆状或短杆状、单个排列,革兰氏染色阳性,但从扫描电镜结果可知FBKL1.0203的大小为(2.2~2.6)μm×(0.4~0.5)μm,FBKL1.0204的大小为(1.6~2.1)μm×(0.5~0.6)μm,FBKL1.0205的大小为(1.4~1.9)μm×(0.5~0.6)μm,差异较为明显(图1)。

a-菌落形态;b-光学显微镜下细胞形态; c-扫描电子显微镜下细胞形态
图1 菌株的形态学特征
Fig.1 Morphological characteristics of strains
2.1.2 菌株生理生化鉴定
通过分析利用单糖、二塘、多糖、糖醇、七叶苷情况,以及检测过氧化氢酶活性,可初步认为这3株菌为耐酸乳杆菌。FBKL1.0203特征与前人[23-24]研究基本一致,而FBKL1.0204和FBKL1.0205对利用纤维二糖存在差异,3株耐酸乳杆菌对利用纤维二糖、甘露醇和菊糖存在差异(表1),表明不同耐酸乳杆菌菌株间的生理生化特性存在一定的差异。
表1 生理生化试验特征分析
Table 1 Physiological and biochemical analysis of L.acetotolerans

实验项目结果FBKL1.0203FBKL1.0204FBKL1.0205耐酸乳杆菌描述[23-24]七叶苷++++纤维二糖-+--麦芽糖++++甘露醇-++-水杨苷++++山梨醇----蔗糖----棉子糖----菊糖-++-乳糖----过氧化氢酶----
注:+阳性;-阴性
2.1.3 菌株分离与形态学鉴定分子生物学鉴定
对分离的3株乳酸菌进行16S rDNA鉴定,琼脂糖凝胶电泳结果见图2,均为1 500 bp左右的单一片段,且条带清晰。
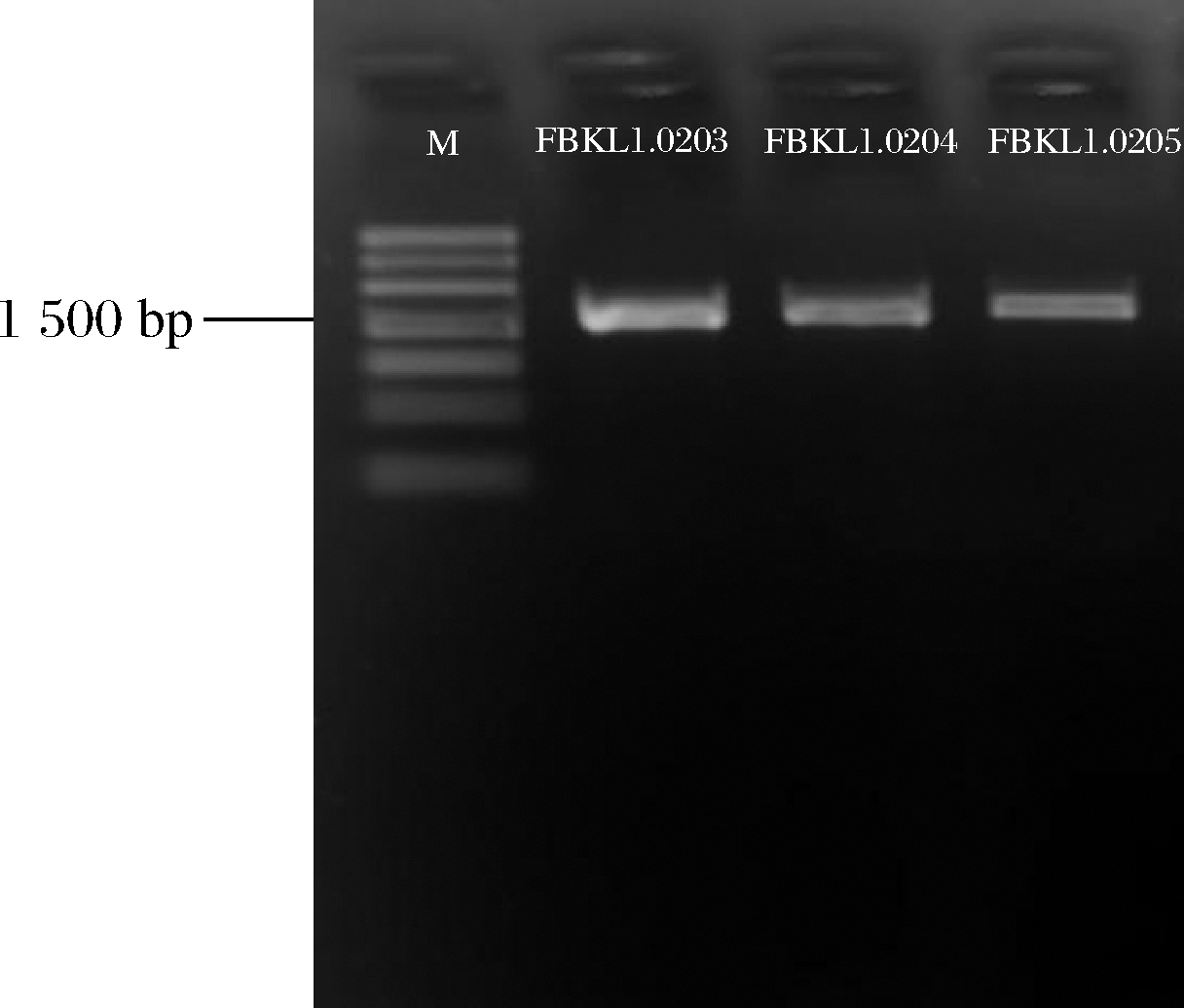
图2 琼脂糖凝胶电泳条带
Fig.2 Agarose gel electrophoresis bands
16S rDNA测定序列经BLAST与GenBank数据库中已知序列进行比对,FBKL1.0203、FBKL1.0204、FBKL1.0205与Lactobacillus acetotolerans LC202658.1的16S rDNA基因同源性分别为99%、100%和99%。利用MEGA 7.0的邻接法绘制系统发育树,菌株FBKL1.0203、FBKL1.0204、FBKL1.0205与L.acetotolerans属于同一分支,表明FBKL1.0203、FBKL1.0204、FBKL1.0205与L.acetotolerans的菌株亲缘关系最近(图3)。乳酸菌16S rDNA序列不能鉴别种间差异,因此,我们扩增3株菌的管家基因rpoA和pheS进行同源性分析,根据rpoA基因序列测序结果,菌株FBKL1.0204、FBKL1.0203和FBKL1.0205与Lactobacillus acetotolerans CP046528.1的rpoA基因序列同源性分别为99%、98%和99%;根据pheS基因序列测序结果,FBKL1.0204与Lactobacillus acetotolerans CP051649.1的pheS基因序列同源性分别为96%;FBKL1.0203和FBKL1.0205与Lactobacillus acetotolerans CP044496.1的pheS基因序列同源性分别为98%和99%,确定这3株菌均为耐酸乳杆菌。

图3 基于16S rDNA序列基因构建的系统发育树
Fig.3 Phylogenetic tree constructed based on 16S rDNA sequence genes
2.2 菌株耐酸性筛选
浓香型白酒酒醅窖池发酵中后期酸度增加,pH值约为3~4[25],酸胁迫是乳酸菌在发酵过程中遭受的重要环境压力[26]。为了筛选出具有优良耐酸性的耐酸乳杆菌,将3株耐酸乳杆菌接种到pH 4的MRS培养基中,3株耐酸乳杆菌的生长曲线均呈“S”型,均具有典型的延滞期、对数期和稳定期,并且生长曲线趋势一致。但3株耐酸乳杆菌生长速率,生长延滞期、对数期和稳定期时间和生物量有差异(图4),FBKL1.0204的延滞期最短为12 h,培养至108 h后进入稳定期,同时达到稳定期时生物量最大,OD600值为2.28;FBKL1.0203在培养20 h时进入对数期,120 h后进入稳定期,此时OD600值为2.08;FBKL1.0205生长速度最慢,延滞期、对数期和进入稳定期时间最长,稳定期时OD600值最小,为1.98,说明不同菌株耐酸性能不同。

图4 耐酸乳杆菌发酵过程生物量
Fig.4 Fermentation process biomass of L.acetotolerans
利用修正的Gompertz模型来拟合3株耐酸乳杆菌的生长曲线,拟合度较高(R2>0.99),生长修正Gompertz模型参数见表2。在3株耐酸乳杆菌中,FBKL1.0204的最大生长速率最高,为0.035,且达到最大生长速率所需要的时间最短,为42 h左右。综上,选择耐酸性最强的FBKL1.0204研究耐酸乳杆菌的耐酸及产酸特性。
表2 耐酸乳杆菌的生长动力学参数
Table 2 Growth kinetics parameters of L.acetotolerans
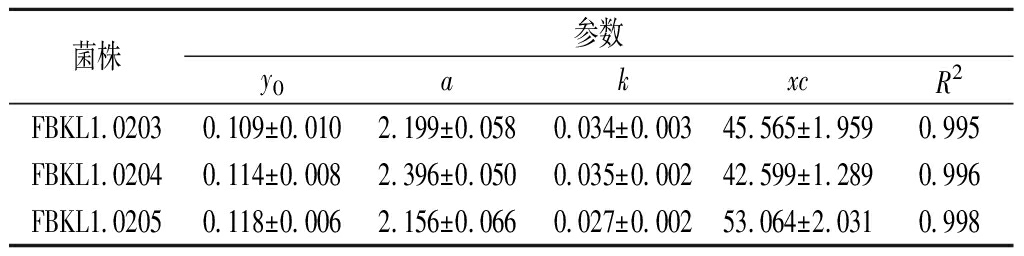
菌株参数y0akxcR2FBKL1.02030.109±0.0102.199±0.0580.034±0.00345.565±1.9590.995FBKL1.02040.114±0.0082.396±0.0500.035±0.00242.599±1.2890.996FBKL1.02050.118±0.0062.156±0.0660.027±0.00253.064±2.0310.998
2.3 耐酸乳杆菌耐酸特性分析
由于白酒窖池发酵酸性环境主要是乳酸和乙酸,二者均为弱酸,在发酵过程中,乳酸和乙酸胁迫下,耐酸乳杆菌在pH 4、pH 5和pH 6的酸性条件下生物量相同,当发酵初始pH为4时,乳杆菌的生物量明显受到抑制,稳定期时间延长2.6~3.6倍,但生物量OD600值约为2.3。当发酵初pH为3时,乳杆菌不增殖(图5-a和图5-b)。

a-乳酸;b-乙酸;c-盐酸;d-最适生长pH
图5 FBKL1.0204在乳酸、乙酸和盐酸胁迫下的生物量以及最适生长pH
Fig.5 Biomass of FBKL1.0204 under lactic acid, acetic acid and hydrochloric acid stress and optimum growth pH
TANAKA等[23]发现L.acetotolerans HT在含有0~40 g/L乙酸的MRS培养基中,耐酸乳杆菌的生长速率随乙酸浓度的增加而降低,与本试验结果相似。乳酸和乙酸都是弱酸,调pH值到4后,乳酸和乙酸质量浓度达到13.5 g/L和24.5 g/L,调pH值到3后,乳酸和乙酸质量浓度达到55.5 g/L和233.5 g/L,高浓度的乳酸和乙酸可能影响乳杆菌增殖,有研究表明,有机酸可以通过扩散作用进入细胞,在细胞内解离成H+,使细胞质的pH值下降,造成细胞内酸化[27]。因此,我们考虑用强酸调节培养基的初始pH,利用盐酸调节培养基初始pH为4后,乳杆菌增殖显著受到抑制,但比乳酸和乙酸的抑制能力弱,达到稳定期时的生物量相同。此外,当利用盐酸调节培养基初始pH为3后,乳杆菌也不增殖,达到极端生存pH(图5-c),说明H+抑制耐酸乳杆菌增殖。乳杆菌对酸的敏感性主要在于胞内pH,H+的大量存在使细胞内pH降低,从而影响乳杆菌的生长、代谢以及相关酶的活性等[28]。虽然酸胁迫严重影响乳杆菌的存活能力,但这种抑制作用随酸度的下降而减小,各种微生物都有其生长的最适、最低和最高pH值。为了探索耐酸乳杆菌的最佳生长pH,将乳杆菌接种到不同初始pH的培养基中,发现初始pH=6时耐酸乳杆菌增殖最快(图5-d)。
2.4 耐酸乳杆菌产酸能力分析
pH值可以用来表征乳酸菌的产酸能力[29],耐酸乳杆菌在纯种发酵过程中代谢产酸,随着发酵过程的延长,大量有机酸积累,pH值下降,当初始pH=5和pH=6时,增殖36 h后pH降低到4(图6),此时的生物量达到稳定期(图5),随着发酵时间延长,酸度缓慢积累,pH达到3.5左右;当初始pH=4时,酸度也在发酵过程中降低,最终pH达到3.5左右。盐酸调节培养基初始pH为5和4时,发酵后pH分别下降了1.55和1.02,与黄桂东[30]的研究相比,FBKL1.0204的产酸特性良好。当将初始pH调到3,乳杆菌不增殖,酸度几乎没有降低(图4),说明高浓度酸性环境使FBKL1.0204生长受到显著抑制,同时菌株产酸也停滞。
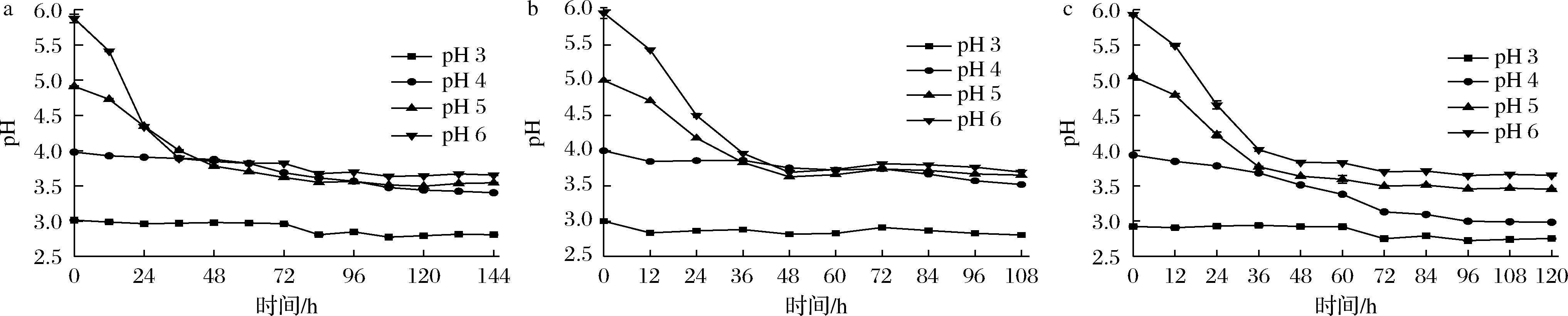
a-乳酸;b-乙酸;c-盐酸
图6 酸胁迫下FBKL1.0204产酸特性
Fig.6 Acid production characteristics of FBKL1.0204 under acid stress
乳杆菌有同型乳酸发酵和异型乳酸发酵代谢产生乳酸和乙酸,为了探索耐酸乳杆菌的产有机酸特性,检测耐酸乳杆菌发酵液中有机酸组成,发现耐酸乳杆菌在乳酸、乙酸和盐酸胁迫下都能产生乳酸和乙酸,且乳酸和乙酸比例为24∶1.7(图7),说明耐酸乳杆菌在酸胁迫下是异型乳酸发酵,主要将葡萄糖代谢为乳酸。此外,随着pH降低,乳酸和乙酸的含量逐渐降低,说明酸度影响耐酸乳杆菌乳酸和乙酸代谢。FBKL1.0204在乙酸pH=6胁迫下合成乳酸和乙酸含量最高,分别为(28.32±0.54)g/L和(2.73±0.52)g/L,比pH=4胁迫酸含量提高了34.24%和434.98%。杨帆[31]发现白酒中Lactobacillus panis在最佳产酸环境下(培养基乳酸添加量10 g/L),乳酸产量为12.3 g/L。综上,FBKL1.0204产酸能力较强。
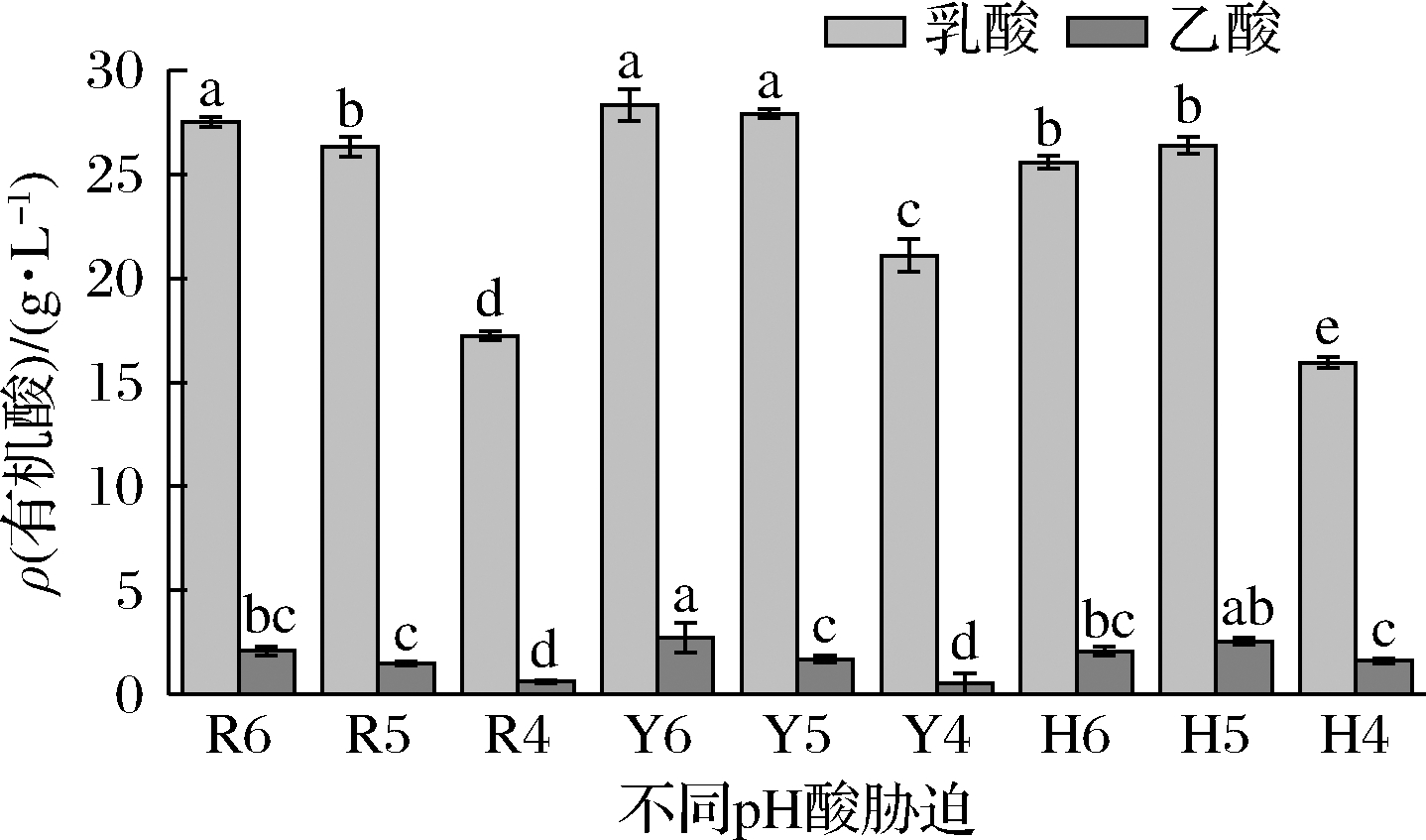
图7 不同pH酸胁迫下FBKL1.0204代谢产生有机酸的含量
Fig.7 Contents of organic acids produced by FBKL1.0204 metabolism under acid stress of different pH 注:R6、R5、R4、Y6、Y5、Y4、H6、H5、H4表示乳酸、乙酸和 盐酸分别调节pH为6、5、4的MRS液体培养基,组间 不同小写字母代表存在显著性差异(P<0.05)
3 结论
本试验利用可培养方法,根据菌落形态差异、生理生化指标和分子生物学鉴定从浓香型酒醅中分离鉴定出3株耐酸乳杆菌,其中菌株FBKL1.0204在pH 4的初始酸性环境中能生长代谢,具有较强的耐酸性。FBKL1.0204主要代谢生成乳酸和乙酸,并发现H+是酸胁迫的主要原因,具体酸胁迫抑制菌体生长的原因还需要进一步探究。该菌株在有机酸和强酸胁迫下都可产生有机酸,且发酵液pH最终降至3.5左右,具有良好的产酸特性。本研究有助于耐酸乳杆菌发酵产酸提高浓香型白酒的风味和品质,以期为酸胁迫响应机制的研究提供一定的理论基础。
[1] 徐军. 浓香型枝江白酒香味成分的分析研究[D]. 武汉: 华中农业大学, 2019.
XU J. Study on aroma components of strong-aroma Zhijiang Baijiu[D]. Wuhan: Huazhong Agricultural Uniersity, 2019.
[2] HU X L, DU H, XU Y. Identification and quantification of the caproic acid-producing bacterium Clostridium kluyeri in the fermentation of pit mud used for Chinese strong-aroma type liquor production[J]. International Journal of Food Microbiology, 2015, 214:116-122.
[3] ZHENG J, WU C, HUANNG J, et al. Spatial distribution of bacterial communities and related biochemical properties in Luzhou-flaor liquor-fermented grains[J]. Journal of Food Science, 2014, 79(12): M2 491-M2 498.
[4] DI MARTINO C, TESTA B, LETIZIA F, et al. Effect of exogenous proline on the ethanolic tolerance and malolactic performance of Oenococcus oeni[J]. Journal of Food Science and Technology, 2020, 57(11): 3 973-3 979.
[5] ZOU W, ZHAO C Q, LUO H B. Diersity and function of microbial community in Chinese strong-flaor Baijiu ecosystem: A reiew[J]. Frontiers in Microbiology, 2018, 9:671.
[6] WANG X S, DU H, XU Y. Source tracking of prokaryotic communities in fermented grain of Chinese strong-flaor liquor[J]. International Journal of Food Microbiology, 2017, 244:27-35.
[7] SUN W N, XIAO H Z, PENG Q, et al. Analysis of bacterial diersity of Chinese Luzhou-flaor liquor brewed in different seasons by Illumina Miseq sequencing[J]. Annals of Microbiology, 2016, 66(3):1 293-1 301.
[8] 刘森, 李林光, 李可, 等. 中国浓香型白酒窖池窖泥中原核微生物群落的空间异质性[J]. 食品科学, 2013, 34(21):221-226.
LIU S, LI L G, LI K, et al. Spatial heterogeneity of prokaryotic microbial communities in Luzhou-flaor liquor pit mud[J]. Food Science, 2013, 34(21):221-226.
[9] WANG H Y, ZHANG X J, ZHAO L P, et al. Analysis and comparison of the bacterial community in fermented grains during the fermentation for two different styles of Chinese liquor[J]. Journal of Industrial Microbiology and Biotechnology, 2008, 35(6):603-609.
[10] LI X R, MA E B, YAN L Z, et al. Bacterial and fungal diersity in the traditional Chinese liquor fermentation process[J]. International Journal of Food Microbiology, 2011, 146(1):31-37.
[11] ENTANI E, MASAI H, SUZUKI K I. Lactobacillus acetotolerans, a new species from fermented inegar broth[J]. International Journal of Systematic Bacteriology, 1986, 36(4):544-549.
[12] TOH H, MORITA H, TSUJI H, et al. Complete genome sequence of Lactobacillus acetotolerans RIB 9124 (NBRC 13120) isolated from putrefied (hiochi) Japanese sake[J]. Journal of Biotechnology, 2015, 214:214-215.
[13] YANG X P, TENG K L, ZHANG J, et al. Transcriptome responses of Lactobacillus acetotolerans F28 to a short and long term ethanol stress[J]. Scientific Reports, 2017, 7:2650.
[14] LIU J Y, DENG Y, PETERS B M, et al. Transcriptomic analysis on the formation of the iable putatie non-culturable state of beer-spoilage Lactobacillus acetotolerans[J]. Scientific Reports, 2016, 6:36753.
[15] LI H, HE R Y, XIONG X M, et al. Dynamic diersification of bacterial functional groups in the Baiyunbian liquor stacking fermentation process[J]. Annals of Microbiology, 2016, 66(3):1 229-1 237.
[16] 刘文容. 黄酒陈酿过程中酸败乳酸菌的分离鉴定及其特性研究[D]. 无锡:江南大学, 2017.
LIU W R. Studies on the isolation, identification and characteristics of spoilage lactic acid bacteria in storage of Chinese rice wine[D]. Wuxi: Jiangnan Uniersity, 2017.
[17] 布坎南·吉本斯. 伯杰氏细菌鉴定手册[M]. 第九版.北京: 科学出版社, 1984.
BUCHANAN GIBBONS. Berger’s Bacteria Identification Handbook[M]. 9th edition. Beijing: Science Press, 1984.
[18] 袁峥, 赵瑞香, 牛生洋, 等. 酸胁迫下嗜酸乳杆菌菌体形态的扫描电镜观察[J]. 食品工业科技, 2012, 33(24):199-201.
YUAN Z, ZHAO R X, NIU S Y, et al. Obseration of the mycelia morphology of Lactobacillus acidophilus with scanning electron microscope under acid stress[J]. Science and Technology of Food Industry, 2012, 33(24):199-201.
[19] 陈笑语, 张力元, 纪海玉, 等. 渤海虾酱中益消细菌的分离和鉴定[J]. 中国食品学报, 2020, 20(3):258-265.
CHEN X Y, ZHANG L Y, JI H Y, et al. Isolation and identification of beneficial bacteria in Bohai shrimp paste[J]. Journal of Chinese Institute of Food Science and Technology, 2020, 20(3):258-265.
[20] ENDO A, TANIZAWA Y, ARITA M. Isolation and Identification of Lactic Acid Bacteria from Enironmental Samples[M]. New York: Springer New York, 2019.
[21] 韩之皓, 郭帅, 黄天, 等. 复合益生菌在乳酸菌饮料中的发酵特性和功能性质研究[J]. 中国食品学报, 2020, 20(11):129-138.
HAN Z H, GUO S, HUANG T, et al. Studies on fermentation characteristics and functional properties of compound probiotics in lactic acid bacteria beerage[J]. Journal of Chinese Institute of Food Science and Technology, 2020, 20(11):129-138.
[22] 王芳, 廖勤俭, 安明哲, 等. 高效液相色谱同时检测白酒中四大酸的方法[J]. 酿酒科技, 2018(5):114-116.
WANG F, LIAO Q J, AN M Z, et al. HPLC for detecting the content of four main acids in Baijiu simultaneously[J]. Liquor-Making Science & Technology, 2018(5):114-116.
[23] TANAKA K, TAJIRI S, SAWADA R, et al. Acid-toleracnt lactic acid bacterium isolated from rice inegar[J].Impact Journals, 2015, 10(3): 2 347-4 580.
[24] 翟磊, 程宵宵, 苏姣姣, 等. 一株食醋污染菌CICC 10774的鉴定及其生长代谢特性[J]. 微生物学通报, 2016, 43(7):1 524-1 531.
ZHAI L, CHENG X X, SU J J, et al. Identification and characterization of strain CICC 10774 causing inegar spoilage[J]. Microbiology China, 2016, 43(7):1 524-1 531.
[25] 柴丽娟, 钱玮, 钟小忠, 等. 浓香型白酒发酵过程中窖内古菌群落分布特征[J]. 生物工程学报, 2020, 36(12):2 635-2 643.
CHAI L J, QIAN W, ZHONG X Z, et al. Distribution of archaeal community in the mud pit during strong-flaor Baijiu fermentation[J]. Chinese Journal of Biotechnology, 2020, 36(12):2 635-2 643.
[26] DE ANGELIS M, GOBBETTI M. Enironmental stress responses in Lactobacillus: A reiew[J]. Proteomics, 2004, 4(1):106-122.
[27] CARPENTER C E, BROADBENT J R. External concentration of organic acid anions and pH: Key independentariables for studying how organic acids inhibit growth of bacteria in mildly acidic foods[J]. Journal of Food Science,2009, 74(1): R12-R15.
[28] 张明阳, 张娟, 刘龙, 等. 精氨酸代谢途径抗酸关键基因对乳酸乳球菌Lactococcus lactis NZ9000胁迫抗性的影响[J]. 微生物学通报, 2017,44(2): 314-324.
ZHANG M Y, ZHANG J, LIU L, et al. Influence of key acid-resistant genes in arginine metabolism on stress tolerance in Lactococcus lactis NZ9000[J]. Microbiology China, 2017, 44(2): 314-324.
[29] 孟掉琴, 吴霞, 岳田利, 等. 混菌发酵苹果浊汁的益生菌筛选及其发酵动力学模型构建[J]. 食品科学, 2019, 40(12):153-159.
MENG D Q, WU X, YUE T L, et al. Screening of probiotics for mixed culture fermentation of cloudy apple juice and construction of kinetic models[J]. Food Science, 2019, 40(12):153-159.
[30] 黄桂东. Lactobacillus breis NCL912的耐酸特性及其酸胁迫下差异表达蛋白的研究[D]. 南昌: 南昌大学, 2011.
HUANG G D. Acid tolerance of lactobaillus breis NCL912 and its differentially expressed protein under acid strese[D]. Nanchang: Nanchang Uniersity, 2011.
[31] 杨帆. 酱香型白酒中乳酸代谢机理及调控策略的研究[D]. 无锡: 江南大学, 2020.
YANG F. Lactic acid metabolic mechanism inestigation and control strategy deelopment during Chinese Jiang-flaor liquor making[D]. Wuxi: Jiangnan Uniersity, 2020.