三孢布拉霉是目前大规模生产β-胡萝卜素的工业化菌株[1],在该菌株中发现了多个类胡萝卜素合成调控基因,如crgA[2]、wc-1c[3]。在卷枝毛霉中类胡萝卜素光诱导合成的效应主要由wc-1c基因介导[4],通过将三孢布拉霉来源的btwc-1c导入到卷枝毛霉相应功能缺失菌中,初步证明了btwc-1c参与光诱导的类胡萝卜素合成[3]。然而,目前缺乏针对三孢布拉霉的高效遗传转化方法[5],难以对类胡萝卜素合成关键调控基因的作用机制进行深入研究。
目前文献已报道的关于三孢布拉霉的转化方法包括聚乙二醇介导的原生质体转化[6]、农杆菌侵染原生质体[5]与电击转化原生质体[7]等。原生质体转化通常使用聚乙二醇介导将核酸导入原生质体中,巩尊洋等[6]采用该方法筛选到阳性转化子,然而受限于三孢布拉霉细胞壁结构的复杂及对细胞壁水解酶的了解使该方法转化效率较低[8]。农杆菌侵染原生质体是依靠T-DNA在诱导条件下转移基因到宿主基因组中的一种转化方法[9],广泛应用于丝状真菌和植物的转化过程。李娜等[5]采用该方法筛选到了转化子,然而该方法操作复杂耗时且后期农杆菌难以完全去除,筛选出的转化子即使在含有头孢噻肟钠的培养基上继代多次仍然有可能携带农杆菌[10]。相对于前两种方法,电击转化法可使外源核酸立即转移到细胞内[8],转化耗时短,另外还能导入双质粒。WANG等[7]采用该方法成功筛选到了转化子,但其转化效率依然不高,可能的原因包括影响真菌原生质体制备的因素,如菌龄、酶解时间与温度、渗透压稳定剂的种类等会影响原生质体的数量与质量[11],而电击转化时的电压大小与电脉冲时间,再生培养时摇床转速及培养时间等会影响转化子的获得。
本研究基于电击转化原生质体存在的问题,利用可以提高转化效率的含有AMA1自主复制元件[12]的复制型质粒pFC332进行电转化,优化了电击转化三孢布拉霉原生质体多个参数。同时,以转化含有btwc-1c的重组载体为例,成功构建了btwc-1c过表达菌株。本研究为在三孢布拉霉中进行代谢工程和解析类胡萝卜素合成的代谢调控机制奠定了一定的基础。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
菌株三孢布拉霉(Blakeslea trispora)负菌NRRL2896、大肠杆菌JM109(Escherichia coli JM109)由本实验室保藏。质粒pFC332和pUC57-AMA1-pgpdA-cas9-TrpC-hph由本实验室保藏。
1.1.2 培养基与溶液
麦汁培养基 (1 L):5°麦汁,25 g琼脂;
种子培养基 (g/L):玉米粉30,大豆粉50,KH2PO4 1.5,MgSO4·7H2O 0.5;
再生培养基 (g/L):蔗糖200,马铃薯200,葡萄糖20,KH2PO41、MgSO4·7H2O 0.1;
LB培养基(g/L):NaCl 10、蛋白胨10、酵母提取物5。如需配制LB固体平板,再加入2%的琼脂粉后灭菌。
溶液 (mol/L):稳渗液:NaCl 0.6;电脉冲缓冲液:蔗糖0.6、磷酸缓冲液0.005、山梨醇1,pH 7。
1.1.3 主要试剂
Prime STAR® Max DNA Polymerase,宝日医生物技术(北京)有限公司;溶菌酶、溶壁酶、蜗牛酶、氨苄青霉素、潮霉素B,无锡特达生物技术有限公司;DNA小量抽提试剂盒,天根生化科技(北京)有限公司;RNApure Plant Kit,北京康为世纪生物科技有限公司;MolPure® Plant DNA Kit植物DNA提取试剂盒、Hifair® Ⅲ 1 st Strand cDNA Synthesis SuperMix for qPCR (gDNA digester plus)、Hieff® qPCR SYBR Green Master Mix (High Rox Plus),翌圣生物科技(上海)股份有限公司;ClonExpress® Ⅱ One Step Cloning Kit,南京诺唯赞生物科技股份有限公司;引物合成与测序等服务,苏州金唯智生物科技有限公司或上海生物工程有限公司。
1.2 实验方法
1.2.1 三孢布拉霉潮霉素最低抑制浓度试验
利用pFC332载体上的潮霉素抗性作为抗性筛选标记,对三孢布拉霉NRRL2896野生型进行潮霉素敏感性测试。将利用酶解制备的三孢布拉霉原生质体通过血球计数板计数后在含有不同浓度潮霉素的培养基中进行培养,确定潮霉素对三孢布拉霉的最低抑菌浓度。
1.2.2 电转化三孢布拉霉原生质体条件的确立及优化
图1为电击转化三孢布拉霉原生质体的基本流程,初始转化条件如下:
(1)三孢布拉霉原生质体的制备
将三孢布拉霉按1%接种量接种于50 mL种子液中,28 ℃培养48 h,8 000 r/min离心5 min,收集菌丝体,无菌水水洗2次,添加10 mL由0.6 mol/L的蔗糖溶液配制的酶解液(3 g/100mL溶壁酶+1.5 g/100mL溶菌酶+1.5 g/100mL蜗牛酶),避光孵育1.5 h,使用4层擦镜纸过滤后用0.6 mol/L的蔗糖溶液清洗2次,4 000 r/min,10 min离心,调整原生质体个数为1×107个/mL后备用。
(2)电转化三孢布拉霉原生质体
将200 μL的原生质体与0.5 μg的线性质粒充分混合后移入规格为0.2 cm的电转杯,设置4 kV/cm的条件,电转1 ms。电转3次(第1次间隔2 s,第2次间隔3 s)后迅速加入940 μL的再生培养基。
(3)再生与培养
在28 ℃,180 r/min培养1.5 h进行再生,然后以9 000 r/min离心1 min,取100 μL转移至含80 μg/mL 的潮霉素固体麦汁培养基中。
需要进行的优化参数如表1所示。

图1 电转化三孢布拉霉原生质体的基本流程
Fig.1 The basic process of electrotransformation for protoplasts of Blakeslea trispora
表1 三孢布拉霉电击转化原生质体的单因素优化
Table 1 Single factor optimization of electroshock transformation of protoplasts by Blakeslea trispora

过程参数水平设置酶解的时间/h1、2、3、4、5电压的大小/kV0.2、0.4、0.6、0.8、1、1.5电脉冲时间/ms1、2、3核酸的质量浓度/(μg·mL-1)1、2.5、5、7.5、10、12.5电脉冲缓冲液种类/(mol·L-1)磷酸缓冲液0.005、山梨醇1、蔗糖0.6、蔗糖0.6+10%(体积分数)甘油再生时间/h0、1、2、3、4、5
1.2.3 再生率及转化效率的计算
计算原生质体再生率步骤如下:在0.6 mol/L NaCl的酶解液(溶壁酶3 g/100mL+溶菌酶1.5 g/100mL+蜗牛酶1.5 g/100mL)中于28 ℃,75 r/min 下避光酶解1~5 h后将酶解后的液体采用4层擦镜纸进行过滤后再用0.6 mol/L NaCl溶液洗涤2遍后使用血球计数板计数将溶液稀释至1×107个/mL,取100 μL后涂布在含200 g/L蔗糖的高渗麦汁固体培养基中,培养2 d 左右后记下菌落数,计为Y1,另外取100 μL的原生质体涂步在不含200 g/L蔗糖的低渗麦汁固体培养基中,培养2 d左右后记下菌落数,计为Y2。
计算电击后的再生率的步骤如下:取200 μL的原生质体在不同的电压大小与电脉冲时间下进行电转(电击3次:一次间隔2 s,一次间隔3 s),涂布在含200 g/L蔗糖的高渗麦汁固体培养基中,培养2 d左右后记下菌落数,计为Y1,另外在同样条件下处理后涂步在不含200 g/L蔗糖的低渗麦汁固体培养基中,培养2 d左右后记下菌落数,计为Y2。
原生质体再生率按照公式(1)和公式(2)进行计算:
再生率![]()
(1)
转化效率![]()
(2)
1.2.4 btwc-1c过表达载体的构建
采用RNApure Plant Kit试剂盒提取野生型三孢布拉霉RNA,然后使用Hifair® Ⅲ 1 st Strand cDNA Synthesis SuperMix for qPCR试剂盒反转录成cDNA,以cDNA为模板,使用引物W-F与W-R得到btwc-1c片段,以质粒pUC57-AMA1-pgpdA-cas9-TrpC-hph为模板采用P-F与P-R反向PCR扩增得到pUC57-AMA1-pgpdA-TrpC片段。使用ClonExpress®Ⅱ One Step Cloning Kit试剂盒将pUC57-AMA1-pgpdA-TrpC与btwc-1c连接构建表达载体pUC57-AMA1-pgpdA-btwc-1c-TrpC-hph。
构建的流程如图2所示,引物见表2。
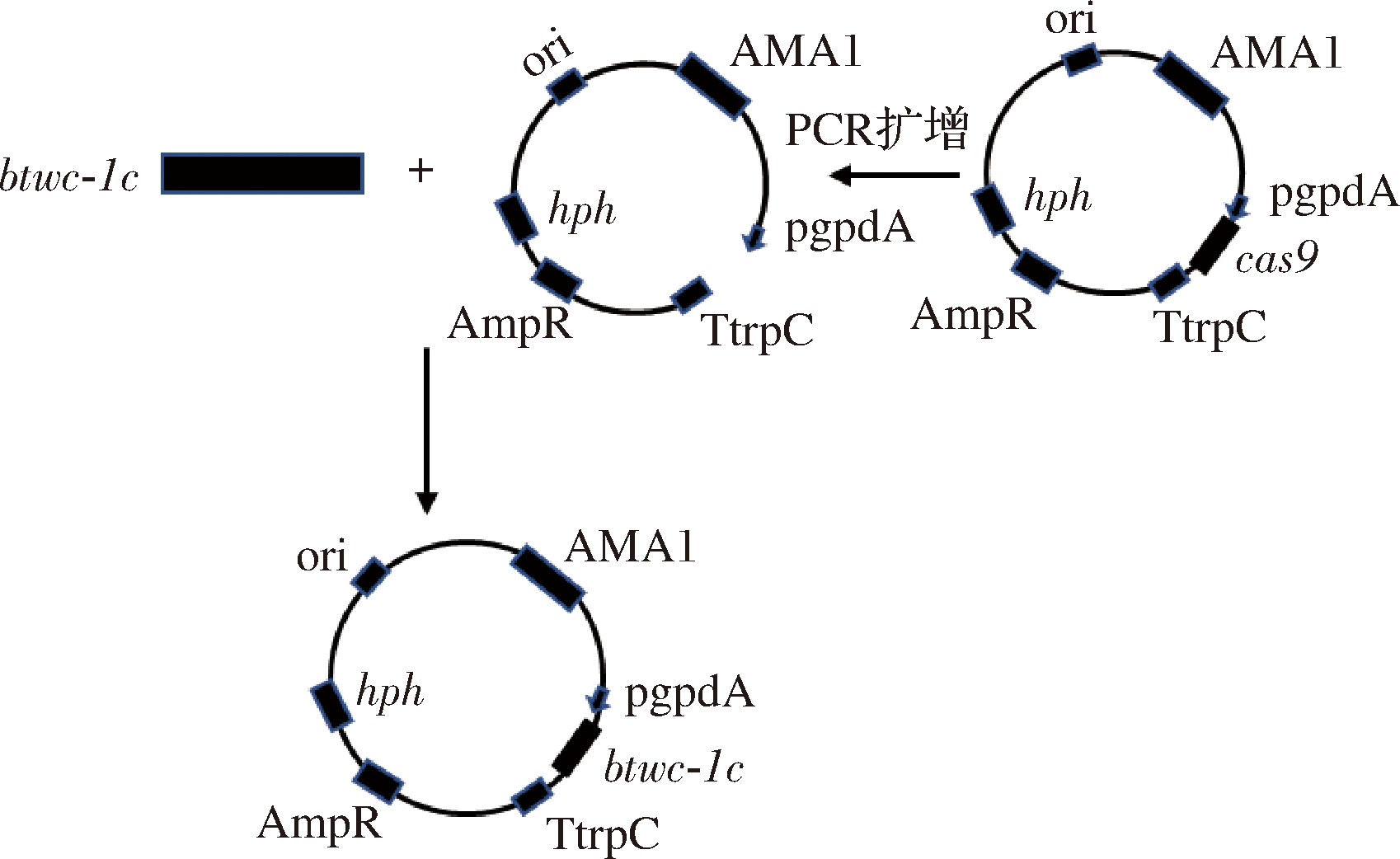
图2 过表达载体pUC57-AMA1-pgpdA-btwc-1c-TrpC-hph的 构建
Fig.2 Construction of overexpression vector pUC57-AMA1-pgpdA-btwc-1c-TrpC-hph
表2 本研究所用到的引物对
Table 2 Primer pairs used in this study

引物名称引物序列5’-3’W-FATGAATAATTCCTCAGCGTCTTATTTACW-RTTATTTTTTGCCTTGCGCAP-FGTGCGCAAGGCAAAAAATAACACTTAACGTTACTGAAATCATCAAACP-RGACGCTGAGGAATTATTCATGGTGATGTCTGCTCAAGCGG-FAAACAGCTATGACCATGATTACGG-RCTGAGTTATGTTCGGGTTGACTqwc-1c-FCAGCATCACCTACCAGCAATGqwc-1c-RGGATTTGAGGATTGGGTCTATGAACtef1-FAACTCGGTAAGGGTTCCTTCAAGtef1-RCGGGAGCATCAATAACGGTAAC
注:下划线为用于一步克隆组装的同源臂序列
1.2.5 转化子鉴定及转录水平分析
将初筛的转化子菌苔转接至含80 μg/mL的潮霉素固体麦汁培养基平板上进行复筛,培养4~5 d后提取基因组,采用MolPure® Plant DNA Kit DNA提取试剂盒进行基因组的提取,采用G-F与G-R验证过表达的转化子,鉴定引物如表2所示。采用Heffi® qPCR SYBR Green Master Mix试剂盒及实时荧光定量PCR仪(StepOnePlus,美国应用生物系统公司)检测,实时荧光定量PCR反应程序为95 ℃预变性5 min;40个循环:95 ℃变性10 s,60 ℃退火/延伸30 s;溶解曲线仪器默认。采用相对定量2-ΔΔCt法计算基因的相对转录水平。2-ΔΔCt法具体步骤如下:
(1)目的基因Ct值归一化:
ΔCt实验组=Ct实验组目的基因-Ct实验组内参基因
ΔCt对照组=Ct对照组目的基因-Ct对照组内参基因
(2)实验组ΔCt值归一化:
ΔΔCt=ΔCt实验组-ΔCt对照组
(3)基因表达差异倍数=(1+n)-ΔΔCt;n为引物的扩增效率。
1.2.6 生物量和β-胡萝卜素含量的测定
在固体培养基上培养三孢布拉霉,然后在红光照射下采用一次性涂布器刮取菌苔迅速称量菌体湿重。测定β-胡萝卜素含量需制定其标准曲线。称取1 mg β-胡萝卜素标准品,用乙酸乙酯配制成10 μg/mL的标准溶液,添加20 g/L 2,6-二叔丁基对甲酚(butylated hydroxy toluene,BHT)防止β-胡萝卜素氧化。分别取标准溶液1、2、3、4、5、6 mL,用乙酸乙酯定容于10 mL棕色容量瓶中。分别取200 μL不同浓度的标准品溶液,用酶标仪测定450 nm下的吸光值。以标准品浓度为横坐标,OD450值为纵坐标,绘制标准曲线。标准曲线计算公式为y=0.129x-0.027 4,y为OD450值,x为β-胡萝卜素含量(ng/μL)。红光环境下取培养的菌丝体测量其湿重,迅速加入10 mL棕色离心管中,用蒸馏水洗涤3次后,将干菌体置于研钵中,加入液氮将菌丝体研磨成粉末状,用含有6 mL 2%(质量分数)BHT的乙酸乙酯,4 ℃萃取1 h[13],4 ℃条件下10 000 r/min离心5 min,分离上清液与沉淀,继续用6 mL的乙酸乙酯进行萃取。将2次上清液进行混合,β-胡萝卜素含量分析采用酶标仪在450 nm下测定吸光值,待测样品的β-胡萝卜素含量根据标准曲线进行计算。
2 结果与分析
2.1 原生质体抗性分析及酶解孵育时间对原生质体再生的影响
选择潮霉素为抗性筛选标记,需首先对三孢布拉霉NRRL2896(-)野生型菌株进行潮霉素抗性分析。先将在平板中培养3~4 d的三孢布拉霉菌苔接种至种子培养基中培养48 h后,将菌丝体进行酶解(3 h),将原生质体数稀释为1.0×107个/mL,取100 μL分别涂布在潮霉素终质量浓度为40~120 μg/mL的麦汁固体平板上,培养4~5 d后观察菌丝体生长情况。由表3结果可知潮霉素质量浓度在80 μg/mL时三孢布拉霉原生质体无法长为菌丝体,因此后续的转化实验将潮霉素质量浓度控制在 80 μg/mL。
表3 三孢布拉霉潮霉素抗性敏感度测试
Table 3 Sensitivity test of Blakeslea trispora to hygromycin

潮霉素质量浓度/(μg·mL-1)三孢布拉霉40+60+80-100-120-
注:“+”代表菌丝体可以生长,“-”代表菌丝体无法生长
真菌具有复杂的、动态的细胞壁结构,在抵御外界刺激过程中发挥着重要作用[14]。丝状真菌细胞壁主要由葡聚糖、几丁质、壳聚糖、甘露聚糖、糖蛋白等组成[15-17],结构复杂、外界物质不易穿透,因而无法像原核生物那样不经处理或经过简单处理后即可进行电转化[18],因此需要制备高质量的原生质体。有研究报道[7]溶壁酶(3 g/100mL)、溶菌酶(1.5 g/100mL)和蜗牛酶(1.5 g/100mL)溶解在0.6 mol/L蔗糖中黑暗孵育1.5 h可以制备出原生质体用于电转,而李晔[19]指出与0.6 mol/L蔗糖相比采用0.6 mol/L NaCl作为稳渗液在28 ℃,75 r/min 避光酶解下更适合制备原生质体,故本研究直接采用0.6 mol/L NaCl作为稳渗液。酶解时间是影响原生质体制备的另一个关键因素,时间较短原生质体释放的不够,时间长原生质体则会易破裂死亡。从图3可知酶解3 h原生质体的再生率最高达到约51.6%,在酶解0~3 h时再生率逐渐升高,3 h后随着酶解时间的延长再生率下降。
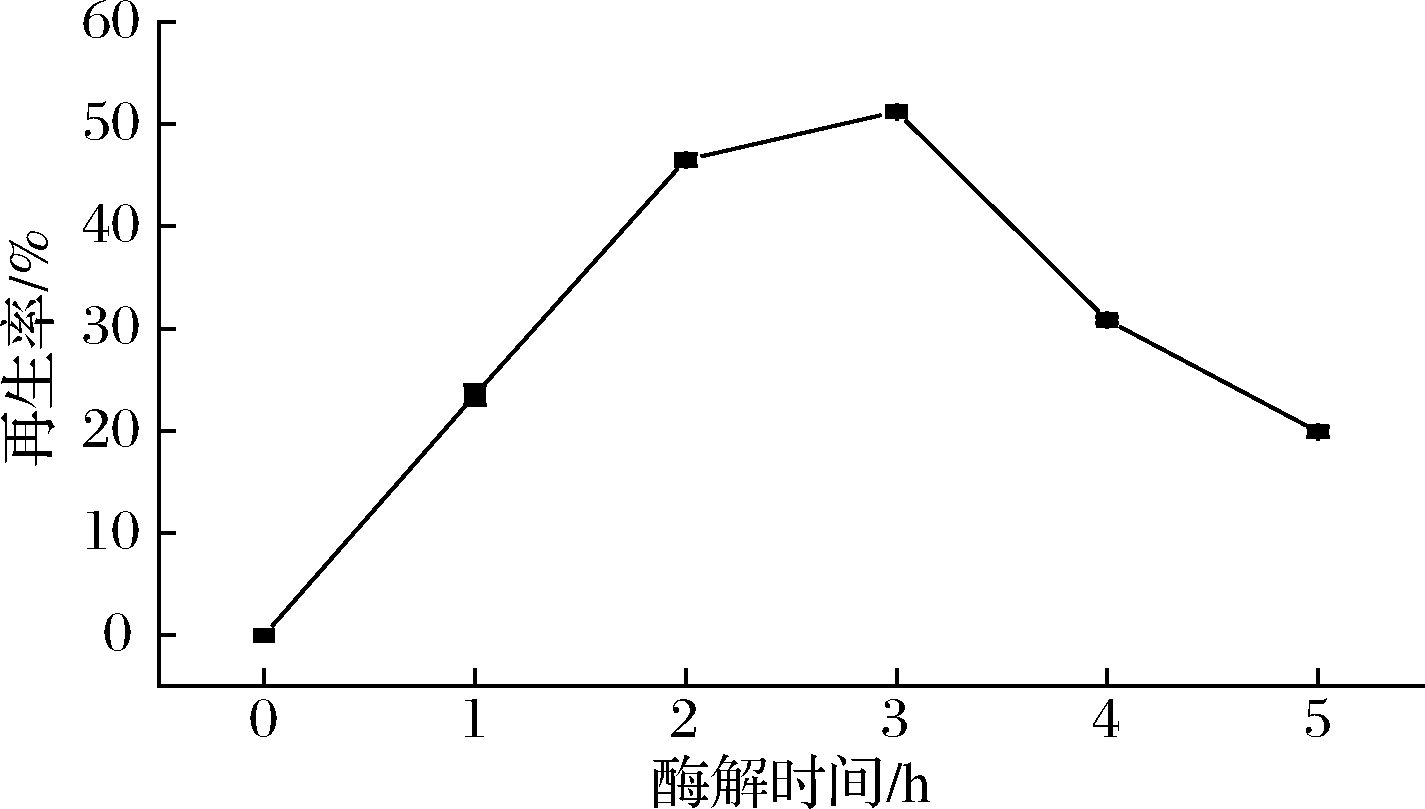
图3 酶解时间对再生率的影响
Fig.3 Effect of enzymatic hydrolysis time on regeneration rate
2.2 不同电场强度及温度对电转化效率的影响
电场强度是影响再生率与电转化效率的关键因素。电场强度没有达到合适的电转阈值时电转效率低,而过高的电场强度会对细胞膜造成不可逆的损伤,导致细胞死亡[20]。因此,本研究设置了不同的电压大小与电脉冲时间,考察其对电转化效率与再生率的影响。如图4-a所示,电脉冲时间为1 ms时的再生率高于3 ms时的,而与2 ms时的再生率相差不大。从图4-b可知电压大小与电脉冲时间对转化效率的影响也较大。当脉冲时间为2 ms时的转化效率比其他两种脉冲时间的转化效率要高。可能的原因是低的脉冲时间(1 ms)不足以将核酸稳定地导入细胞内,脉冲时间较长(3 ms)时对细胞的损伤增加导致再生率低。另外,转化效率受电压影响很大,且在不同脉冲时间下变化趋势一致。由此当电压为0.4 kV,脉冲时间为2 ms时,转化效率达到最大值(15.7 CFU/μg)。
2.3 核酸浓度、电击缓冲液的种类与再生时间对电击转化效率的影响
外源核酸浓度[21]、电击缓冲液的种类与再生时间也会影响电击转化效率,但对再生率的影响并不显著(数据未列出)。采用pUC57-AMA1-pgpdA-btwc-1c-TrpC-hph线性质粒(Sac I进行单酶切)进行转化。由图5-a可知核酸浓度明显影响了转化效率,核酸浓度越高转化效率越低;当核酸质量浓度在2.5 μg/mL时的转化效率最高(约16 CFU/μg),因此选择2.5 μg/mL作为转化时的核酸质量浓度。电转化时,电转缓冲液为其提供了重要的环境。目前常用的介质有蔗糖、甘油蔗糖溶液[22]、磷酸盐缓冲液[22]或山梨醇溶液[23]等,这些介质均能用于电转化。由图5-b可知采用10%甘油+0.6 mol/L的蔗糖溶液或者磷酸盐缓冲液作为电脉冲缓冲液转化效率较低,而1 mol/L 的山梨醇转化效率和蔗糖溶液效果均较好,当选择山梨醇作为电脉冲缓冲液,其转化效率达到30 CFU/μg。另外,电转后原生质体再生时间也会影响转化效率,电转后细胞受到损伤,需要合适的环境与时间进行修复。再生时间太长会影响野生型与转化子生长的比例,再生时间过短细胞壁再生效果不好影响转化效率。由图5-c可知,再生时间为3 h时转化效率达到最大值。
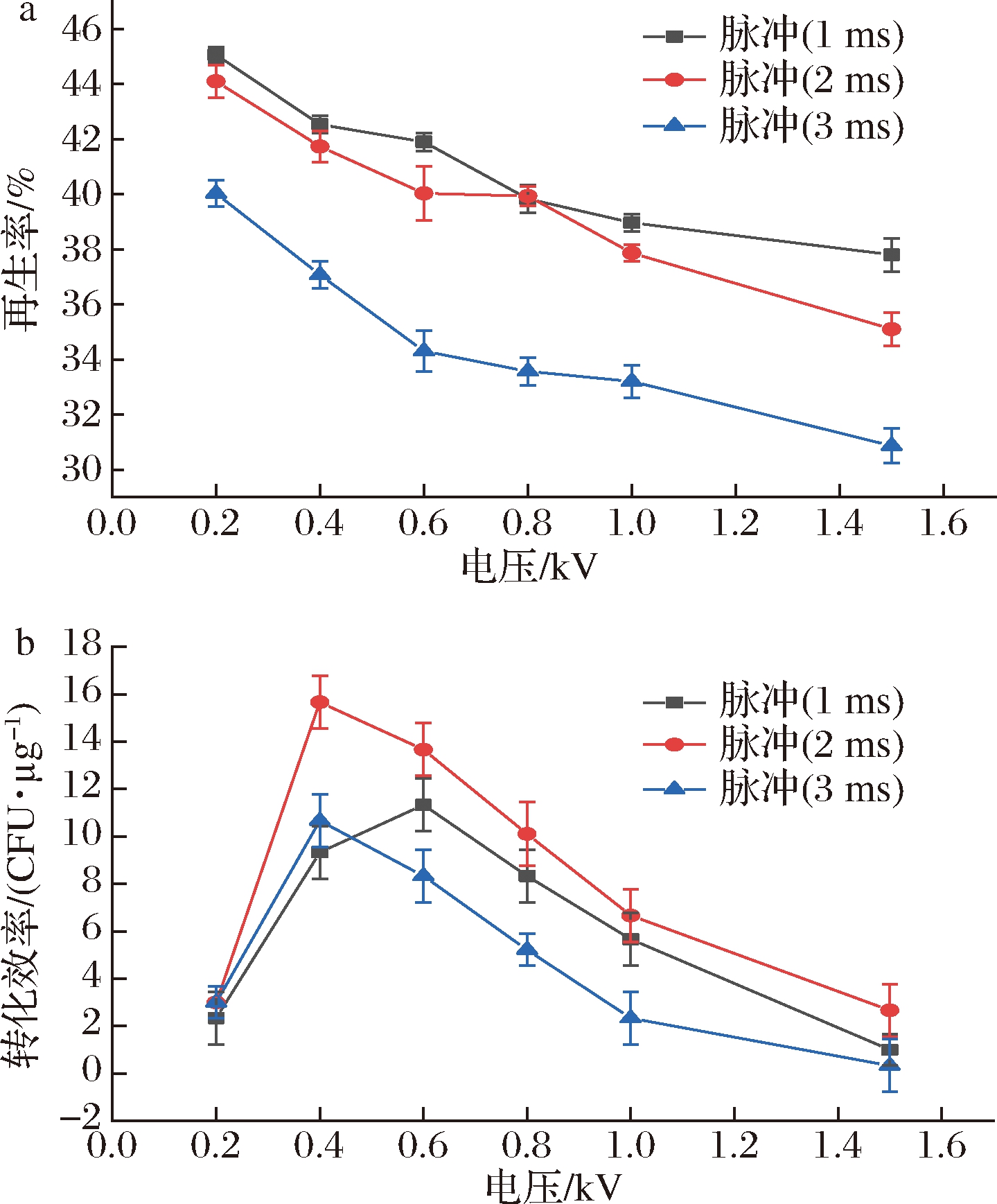
a-电压与电脉冲对再生率的影响; b-电压与电脉冲对电转化效率的影响
图4 三孢布拉霉电击转化参数的优化
Fig.4 Optimization of electric shock conversion parameters of Blakeslea trispora
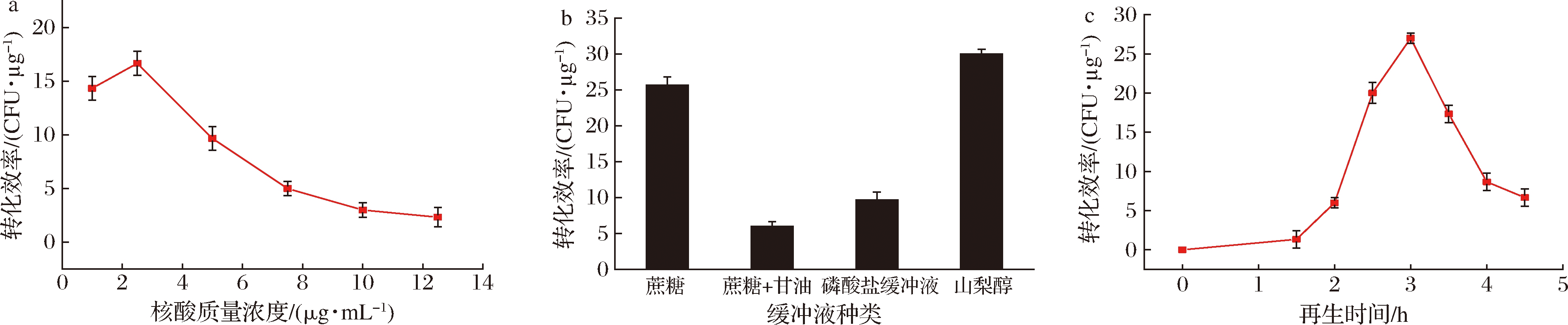
a-核酸浓度对转化效率的影响;b-电脉冲缓冲液种类对转化效率的影响;c-再生时间对转化效率的影响
图5 核酸浓度、电脉冲缓冲液种类与再生时间对电转化效率的影响
Fig.5 The influence of the nucleic acid concentration, the type of electric pulse buffer and the regeneration time on the electric conversion efficiency
2.4 电击转化效率的正交试验分析
在之前单因素试验的基础上,选择电压、电脉冲时间、核酸浓度及再生时间等因素,设计正交试验考察它们对电击原生质体转化效率的影响。每个因素设置4个水平,每个水平进行3个处理,设置一个空白对照。具体参数如下:电压(0.2、0.3、0.4、0.5 kV)、电脉冲时间(1、2、3、4 ms)、核酸质量浓度(1、1.5、2、2.5 μg/mL)及再生时间(2、2.5、3、3.5 h)。
通过软件正交设计助手VII 3.1设计标准正交表,各个因素的离合平方差及极差结果分析如表4所示,根据每个因素不同水平的离合平方差大小,A3B2C4D3为最优组合,转化效率约35.1 CFU/μg。从表5方差分析可知,再生时间偏差平方和小于误差平方和,因此再生时间归入误差,不考虑显著性,而电压大小在不同水平上存在显著差异(P<0.05)。因此选择最优组合电压0.4 kV,电脉冲2 ms,核酸质量浓度2.5 μg/mL,再生时间3 h作为转化最优条件。
2.5 基于优化的电转化策略在三孢布拉霉中的应用
将线性化质粒pUC57-AMA1-pgpdA-btwc-1c-TrpC在上述最优电转条件下转化至野生型三孢布拉霉中,在25 ℃条件下培养4~5 d,转化效率为30 CFU/μg。在80 μg/mL的麦汁固体培养基中传代3次后提取基因组以确定为阳性转化子,随后以pgpdA-btwc-1c框为筛选序列,采用引物C-F与C-R进行PCR共筛选到8个稳定的阳性转化子(图6-a)。将btwc-1c过表达菌株与野生型菌株培养在光照培养箱中,以tef1[24](转录因子编码基因)作为内源参照基因。实时荧光定量PCR分析结果表明转化菌中btwc-1c的基因相对表达水平约为野生型菌株的2.5倍(图6-b),显示了电转化的重组载体在三孢布拉霉中表达了btwc-1c;表型分析结果表明转化菌中β-胡萝卜素含量相对于野生菌提高了约2.1倍,证明了转化菌中表达的btwc-1c能够诱导β-胡萝卜素的合成。
表4 正交分析表
Table 4 Orthogonal analysis table
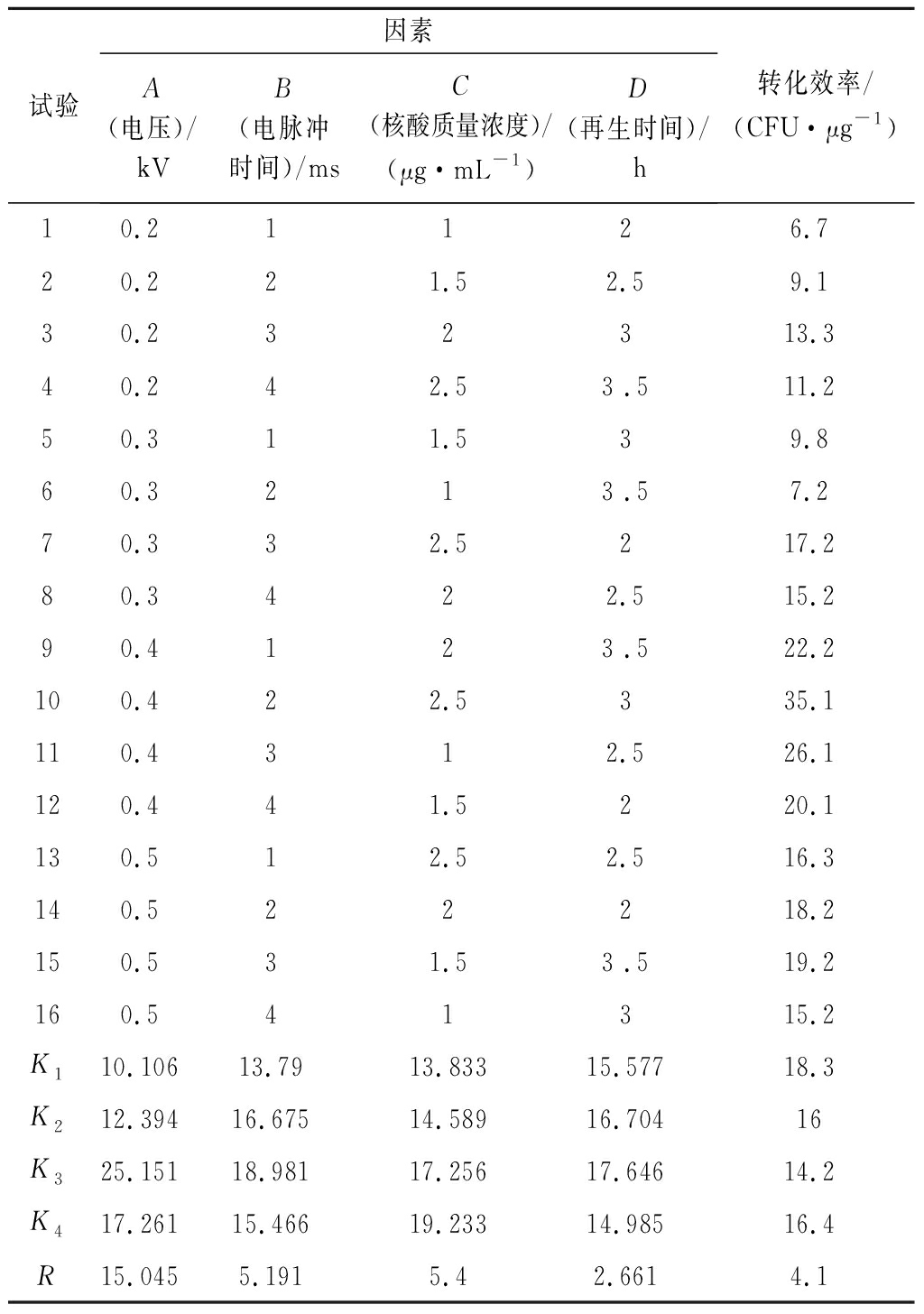
试验因素A(电压)/kVB(电脉冲时间)/msC(核酸质量浓度)/(μg·mL-1)D(再生时间)/h转化效率/(CFU·μg-1)10.21126.720.221.52.59.130.232313.340.242.53 .511.250.311.539.860.3213 .57.270.332.5217.280.3422.515.290.4123 .522.2100.422.5335.1110.4312.526.1120.441.5220.1130.512.52.516.3140.522218.2150.531.53 .519.2160.541315.2K110.10613.7913.83315.57718.3K212.39416.67514.58916.70416K325.15118.98117.25617.64614.2K417.26115.46619.23314.98516.4R15.0455.1915.42.6614.1
注:K1、K2、K3表示离合平方差;R表示极差
表5 方差分析表
Table 5 Table of variance analysis
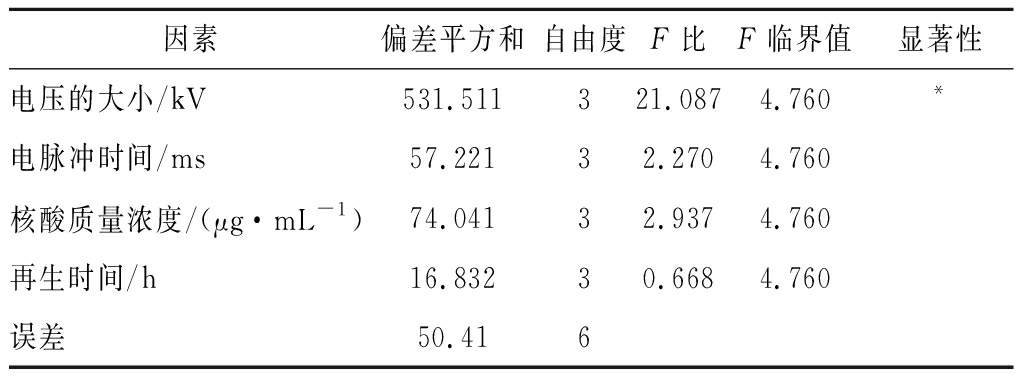
因素偏差平方和自由度F比F临界值显著性电压的大小/kV531.511321.0874.760*电脉冲时间/ms57.22132.2704.760核酸质量浓度/(μg·mL-1)74.04132.9374.760再生时间/h16.83230.6684.760误差50.416
注:*表示差异显著(P<0.05)

M-DNA maker;1-阴性对照,以野生型为模板的PCR结果; 2-阳性对照,pUC57-AMA1-pgpdA-btwc-1c-TrpC-hph 为模板的PCR扩增结果;3~8-阳性转化子的PCR扩增结果; WT-野生型三孢布拉霉;btwc-1c+-btwc-1c基因过表达的三孢布拉霉 a-pgpdA-btwc-1c框的电泳图;b-btwc-1c基因过表达菌株与 野生型菌株相对表达水平及β-胡萝卜素含量的比较
图6 btwc-1c基因过表达菌株的检测
Fig.6 Detection of btwc-1c gene overexpression strain
3 结论
目前对于三孢布拉霉进行遗传转化方法有农杆菌侵染原生质体[5]、原生质体转化[6]及电转原生质体[7]等,因转化效率低造成它们的敲除效率仅在1%~10%。CRISPR/Cas9是最近几年比较热门的对微生物进行分子改造的方法,成功实现了对粗糙脉孢菌[25]、黄青霉[26]及米曲霉[27]等丝状真菌的分子改造。为了提高编辑效率,一般对这些丝状真菌采用双质粒进行同时导入。然而,由于农杆菌侵染操作繁琐、不能同时进行多质粒转化,且对三孢布拉霉原生质体侵染效率较低导致开发出高效率的编辑策略受到了限制。而电转化原生质体能够克服上述多种问题,从而提供了一个潜在的解决方案。为了构建高效的转化方法从而提升三孢布拉霉的分子改造效率,本研究对三孢布拉霉电击转化原生质体进行了优化,包括制备原生质体的酶解时间、电压大小与电脉冲时间、缓冲液种类、核酸浓度及再生时间等。通过单因素试验及正交试验分析最终确定了对培养48 h的三孢布拉霉在0.6 mol/L NaCl酶解液中避光酶解3 h,在1 mol/L的山梨醇电脉冲缓冲液中,核酸添加量为2.5 μg/mL,在0.4 kV、2 ms 的电转条件下进行电转,电转后再生培养3 h,最后涂布在含80 μg/mL潮霉素的麦汁固体培养基中,转化效率达到了35.1 CFU/μg。最后,以电转化btwc-1c基因为例,转化菌中btwc-1c基因表达水平和β-胡萝卜素合成水平分别提高了2.5倍和2.1倍,证明了本研究建立的转化方法能够有效地进行基因转化,为研究三孢布拉霉中类胡萝卜素合成调控基因的功能奠定了坚实的基础。
[1] PAPAIOANNOU E H, LIAKOPOULOU K M.Substrate contribution on carotenoids production in Blakeslea trispora cultivations[J].Food Bioprod Process, 2010, 88(C2-3):305-311.
[2] LUO W, GONG Z Y, LI N, et al.A negative regulator of carotenogenesis in Blakeslea trispora[J].Applied and Environmental Microbiology,2020, 86(6):e02462-e02419.
[3] LUO W, XUE C, ZHAO Y, et al.Blakeslea trispora photoreceptors:Identification and functional analysis[J].Applied and Environmental Microbiology, 2020, 86(8):e02962-e02919.
[4] SILVA F, TORRES MART NEZ S, GARRE V.Distinct white collar-1 genes control specific light responses in Mucor circinelloides[J].Molecular Microbiology, 2006, 61(4):1 023-1 037.
NEZ S, GARRE V.Distinct white collar-1 genes control specific light responses in Mucor circinelloides[J].Molecular Microbiology, 2006, 61(4):1 023-1 037.
[5] 李娜, 曲音波, 杨培龙, 等.三孢布拉霉遗传转化体系的构建及应用[J].食品与生物技术学报, 2021, 40(9):33-39.
LI N, QU Y B,YANG P L, et al.Construction and application of genetic transformation system in Blakeslea trispora[J].Journal of Food Science and Biotechnology, 2021,40(9):33-39.
[6] 巩尊洋, 罗玮, 杜瑶, 等.crgA调控三孢布拉霉合成类胡萝卜素[J].微生物学报, 2017, 57(10):1 527-1 535.
GONG Z Y, LUO W, DU Y et al.Regulation effect of crgA on carotenoid production in Blakeslea trispora[J].Acta Microbiologica Sinica, 2017, 57(10):1 527-1 535.
[7] WANG Y L, PANG J, ZHENG Y M, et al.Genetic manipulation of the bifunctional gene, carRA, to enhance lycopene content in Blakeslea trispora[J].Biochemical Engineering Journal, 2017, 119(15):27-33.
[8] LI D D, TANG Y, LIN J, et al.Methods for genetic transformation of filamentous fungi[J].Microbial Cell Factories, 2017, 16(1):168-181.
[9] HAN G M, SHAO Q, LI C, et al.An efficient Agrobacterium-mediated transformation method for aflatoxin generation fungus Aspergillus flavus[J].The Journal of Microbiology, 2018, 56(5):356-364.
[10] 刘建雨, 张美彦, 张丹, 等.电击法转化金针菇菌丝碎片表达报告基因eGFP及GUS的研究[J].食用菌学报, 2018, 25(2):23-28.
LIU J Y, ZHANG M Y, ZHANG D, et al.Electroporation of mycelial fragments to express reporter genes eGFP and GUS in Flammulina velutipes[J].Acta Edulis Fungi, 2018, 25(2):23-28.
[11] 沈慧敏, 李超, 高利 等.原生质体法介导真菌遗传转化的研究进展[J].植物保护, 2017, 43(2):25-28;42.
SHEN H M, LI C, GAO L, et al.Research progress in transformation of fungi mediated by protoplasts[J].Plant Protection, 2017, 43(2):25-28;42.
[12] KATAYAMA T, NAKAMURA H, ZHANG Y, et al.Forced recycling of an AMA1-based genome-editing plasmid allows for efficient multiple gene deletion/integration in the industrial filamentous fungus Aspergillus oryzae[J].Applied and Environmental Microbiology, 2019, 85(3):e01896-e01818.
[13] 董雪田. 三孢布拉霉中光照对类胡萝卜素生物合成的影响及其部分调控元件的初步挖掘[D].无锡:江南大学, 2022.
DONG X T.The effect of light on carotenoid biosynthesis and the preliminary exploration of some regulatory elements in Blakeslea trispora[D].Wuxi:Jiangnan University, 2022.
[14] YOSHIMI A, MIYAZAWA K, ABE K.Function and biosynthesis of cell wall alpha-1,3-glucan in Fungi[J].J Fungi, 2017, 3(4):E63.
[15] ARANDA M A, LOPEZ M F, LOPEZ L L V.Cell wall composition plays a key role on sensitivity of filamentous fungi to chitosan[J].Journal of Basic Microbiol, 2016, 56(10):1 059-1 070.
[16] RUIZ HERRERA J, ORTIZ CASTELLANOS L.Cell wall glucans of fungi.A review[J].The Cell Surface, 2019, 5:100022.
[17] GARCIA -RUBIO R, DE OLIVEIRA H C, RIVERA J, et al.The fungal cell wall:Candida, Cryptococcus, and Aspergillus species[J].Frontiers in Microbiology, 2020, 10(13):2993.
[18] 董雪田, 朱恺丽, 曲音波, 等.一种快速提取三孢布拉霉基因组的方法[J].食品与生物技术学报, 2021, 40(10):63-71.
DONG X T, ZHU K L, QU Y B, et al.Rapid method of genome extraction of Blakeslea trispora[J].Journal of Food Science and Biotechnology, 2021, 40(10):63-71.
[19] 李晔. RNA干扰三孢布拉氏霉菌番茄红素环化酶基因的研究[D].北京:北京化工大学, 2009.
LI Y.Study on silencing of the carra gene by RNA interfetrnce in filamentous fungus Blakeslea trispora[D].Beijing:Beijing University of Chemical Technology, 2009.
[20] WEAVER J C.Electroporation of biological membranes from multicellular to nano scales[J].IEEE Transactions on Dielectrics and Electrical Insulation, 2003, 10(5):754-768.
[21] PALOMINO M M, ALLIEVI M C, PRADO A M, et al.New method for electroporation of Lactobacillus species grown in high salt[J].Journal of Microbiol Methods, 2010, 83(2):164-167.
[22] 于建宁, 宋小敬, 王公金, 等.乳酸乳球菌电转化方法的优化[J].江苏农业科学, 2013, 41(8):34-36.
YU J N, SONG X J, WANG G J, et al.Optimization of electrotransformation method of Lactococcus lactis[J].Jiangsu Agricultural Sciences, 2013, 41(8):34-36.
[23] ZHOU J L, LIU X B, YUAN F W, et al.Biocatalysis of heterogenously-expressed chitosanase for the preparation of desirable chitosan oligosaccharides applied against phytopathogenic fungi[J].ACS Sustainable Chemistry & Engineering, 2020, 8(12):4 781-4 791.
[24] SCHMIDT A D, HEINEKAMP T, MATUSCHEK M, et al.Analysis of mating-dependent transcription of Blakeslea trispora carotenoid biosynthesis genes carB and carRA by quantitative real-time PCR[J].Applied Microbiology and Biotechnology, 2005, 67(4):549-555.
[25] MATSU URA T, BAEK M, KWON J, et al.Efficient gene editing in Neurospora crassa with CRISPR technology[J].Fungal Biology and Biotechnology, 2015, 2(4).DOI:10.1186/S40694-015-0015-1.
[26] POHL C, KIEL J A K W, DRIESSEN A J M, et al.CRISPR/Cas9 Based genome editing of Penicillium chrysogenum[J].ACS Synthetic Biology, 2016, 5(7):754-764.
[27] ZHENG Y M, LIN F L, GAO H, et al.Development of a versatile and conventional technique for gene disruption in filamentous fungi based on CRISPR-Cas9 technology[J].Scientific Reports, 2017, 7(1):9250.