巴氏杀菌乳是采用低温杀菌的一种鲜奶,适度的热加工可以灭活生乳中的致病菌和部分腐败菌,同时最大程度地保留了生乳天然的营养成分和优良的感官品质[1]。我国巴氏杀菌乳的消费量约占乳品市场的20%,对标乳业发达国家的90%,国内巴氏杀菌乳仍具有可观的发展空间[2-3]。巴氏杀菌工艺加工强度较低,终产品中仍有部分耐热细菌或芽胞存活[4],因此巴氏杀菌乳常需要结合冷链进行储运,且更低的储运温度能延长其货架期[5]。贮藏过程中温度的失控可能会导致热杀菌后存活的微生物细胞数量快速增加,并最终导致产品的变质。巴氏杀菌乳的菌群结构与其品质密切相关,而菌群结构易受到环境温度的影响[6-7],因此,了解贮藏过程的细菌多样性变化对产品的货架期预测和品质判断具有重要作用。
目前,细菌群落结构和细菌多样性研究中应用较为广泛的是16S rRNA基因高通量测序,其具有高通量、高准确度和高覆盖等优点[8],能迅速准确揭示样本微生物群落组成等信息。闫艳华等[9]应用高通量测序技术分析了奶牛养殖场采奶环节中4个不同的操作对生鲜乳细菌多样性的影响,发现生鲜乳的品质与牛乳头和采奶设备的洁净程度密切相关。PORCELLATO等[10]对不同乳品厂生产的巴氏杀菌乳微生物多样性进行研究,发现细菌群落的组成主要受生产月份和贮藏温度的影响。可以看出,高通量测序技术的应用为当前的乳品中微生物多样性研究提供了一个全面解析的手段。
本研究对温度失控条件下巴氏杀菌乳中微生物组成进行表征,通过细菌扩增子测序技术分析巴氏杀菌乳贮藏过程中细菌群落多样性和群落结构动态变化规律,分析其结构及功能演变,为巴氏杀菌乳贮藏过程中微生物动态变化研究及产品质量控制提供理论依据。
1 材料与方法
1.1 试剂与仪器
E.Z.N.A.Food DNA Kit食品基因组DNA提取试剂盒,美国Omega公司;DL 2 000 DNA分子质量标准、10×Loading Buffer,上海捷瑞生物工程有限公司;TransStart FastPfu超高保真PCR酶,上海生工生物工程有限公司;平板计数琼脂(PCA),青岛海博生物公司。
Fresco 17高速冷冻离心机、Nano Drop 2000微量核酸蛋白定量仪,美国ThermoFisher公司;Gel Doc XR凝胶成像仪,美国Bio-Rad公司;Biometra TOne 96 G PCR仪,德国Analytik Jena公司;FC-B2V恒温培养箱,德国MMM集团。
1.2 实验方法
1.2.1 样品
巴氏杀菌乳购自上海本地超市,产品标注的蛋白质和脂肪含量分别为3.1 g/100mL和3.5 g/100mL,2~6 ℃下保质期为7 d。购买后放置于有冰袋的冷藏箱内并立即运回实验室,无菌分装为3份,分别置于10、15、25 ℃贮藏。
1.2.2 基因组DNA提取及细菌总数测定
在10、15、25 ℃贮藏条件下,分别间隔24、12、4 h进行牛奶样品无菌取样,分别获得10、10、9个时期样品。使用试剂盒对上述样品进行细菌基因组DNA提取,经细菌收集、裂解,DNA分离、纯化等步骤获得细菌基因组DNA,并测定其浓度和纯度。同时参照GB 4789.2—2016《食品安全国家标准 食品微生物学检验 菌落总数测定》使用PCA培养基对菌落总数进行计数。
1.2.3 16S rRNA基因扩增及高通量测序
利用已提取的DNA作为模板,选择特异性引物338F(5’-ACTCCTACGGGAGGCAGCAG-3’)和806R(5’-GGACTACHVGGGTWTCTAAT-3’),对细菌16S rRNA基因的V3~V4区进行扩增。20 μL反应体系:5×FastPfu Buffer 4 μL、2.5 mmol/L dNTPs 2 μL、Forward Primer(5 μmol/L)0.8 μL、Reverse Primer(5 μmol/L)0.8 μL、FastPfu Polymerase 0.4 μL、Template DNA 10 ng,补ddH2O至20 μL。PCR扩增条件为:95 ℃预变性3 min,95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,共29个循环,最后72 ℃延伸10 min[11]。测序由上海美吉生物公司使用Illumina Miseq PE300测序仪完成。
1.2.4 生物信息学分析
采用FLASH(v1.2)软件[12]对测序的原始数据进行拼接。使用QIIME(v1.9)分析软件[13]对低质量的拼接序列进行过滤,并去除嵌合体,得到优质序列。根据Uparse(v7.0)以97%相似性水平上对优质序列进行聚类,得到可操作分类单元(operational taxonomic unit,OTU),以各OTU中丰度最高的序列作为代表序列,采用核糖体数据库项目(ribosomal database project,RDP)工具classifier贝叶斯算法对OTU代表序列进行分类学比对,物种分类数据库使用的是Silva 138。在OTU水平上计算不同贮存温度、时间样品、Alpha多样性、Beta多样性。用稀疏曲线(rarefaction curve)和香农指数曲线(Shannon index curve)评估不同测序深度时群落的多样性,以评价各样本的测序量是否合理。超1指数(Chao1 index)和辛普森指数(Simpson index)用于评估样品贮藏过程中细菌群落的丰度和多样性的变化。使用UniFrac距离对样品之间进行非加权的主坐标分析(principal coordinate analysis,PCoA)。
同时利用R软件[14]生成不同分类水平上(门、属)的物种分布图。使用Tax4Fun[15]对16S扩增子数据进行基因组功能预测,其通过构建Silva数据库的16S分类谱系与KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库中原核生物的分类谱系的线性关系,实现对菌群基因功能的预测。本研究中涉及的序列数据已提交至NCBI(National Center of Biotechnology Information)数据库,登录号为PRJNA796711。
2 结果与分析
2.1 样品16S rRNA基因序列质量评估
通过Illumina MiSeq高通量测序,29个样品共产生了1 475 026条高质量16S rRNA基因序列,10、15、25 ℃贮藏温度下每个样品平均分别产生44 730、68 753、50 728条。根据序列的97%相似性进行OTU划分,并按照最小样本序列数进行抽平,共得到2 258个OTU进行进一步分析。
从稀疏曲线(图1-a)可以看出,随着测序量的增加,新增OTU的速度变缓,同时Shannon指数曲线逐渐平稳(图1-b)。说明现有的测序量比较充足,能够反映样品中绝大部分的细菌物种信息。因此当前测序量满足进行生物信息分析的条件。
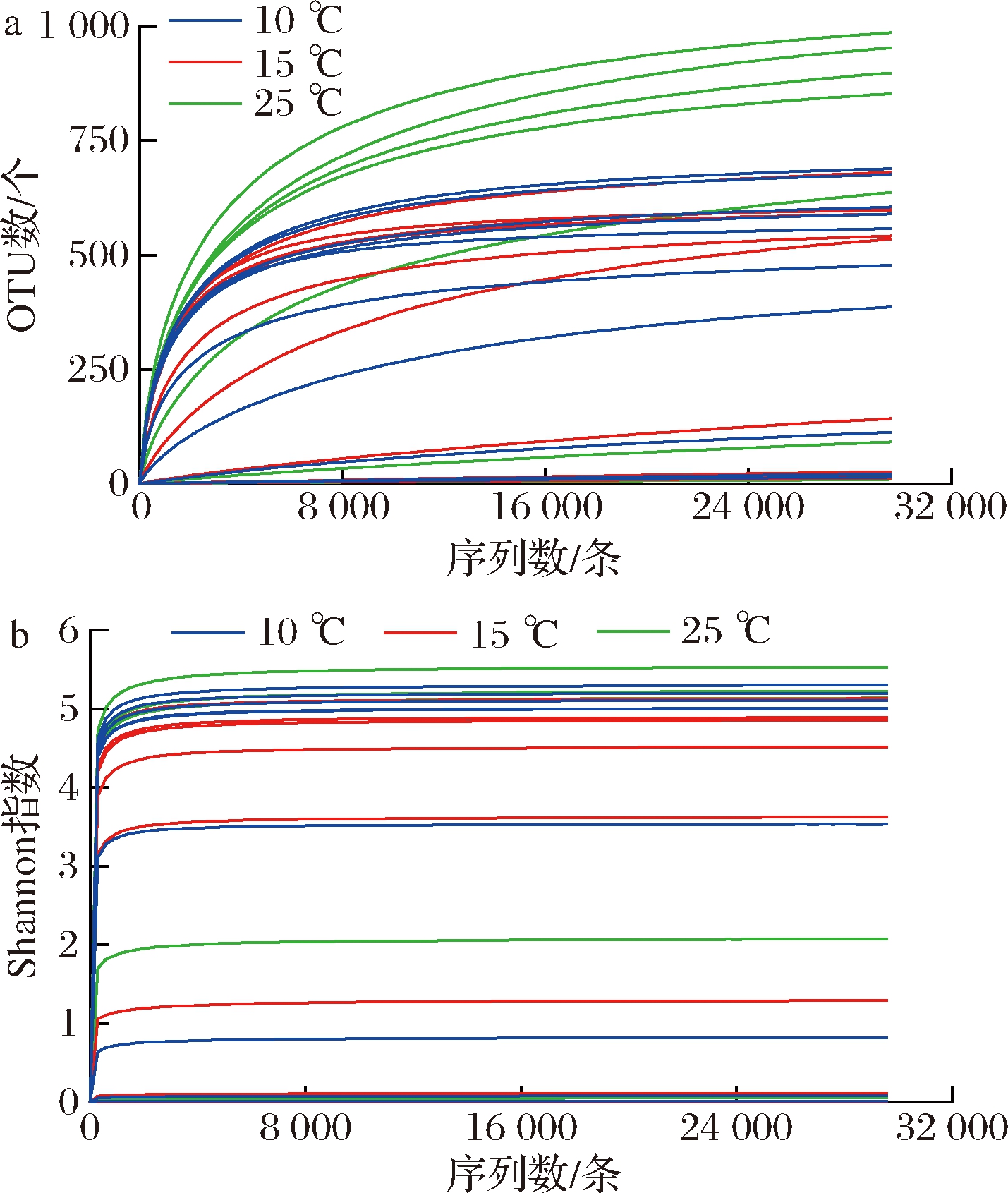
a-稀疏曲线;b-Shannon指数曲线
图1 巴氏杀菌乳的稀疏曲线和Shannon指数曲线
Fig.1 Rarefaction curves and Shannon index curves of pasteurized milk samples
2.2 巴氏杀菌乳细菌群落多样性分析
在生态学研究中Chao1指数和Simpson指数是反映细菌群落Alpha多样性的重要指标,Chao1指数数值与细菌群落丰富度呈正相关,而Simpson指数数值越大细菌群落多样性越低。在贮藏过程中巴氏杀菌乳的细菌菌群丰度随时间变化(图2)。3个温度下的贮藏初期,细菌丰度波动较小;10 ℃下贮藏到第5~7天细菌多样性急剧下降,15、25 ℃条件下,多样性骤降的区间分别发生在48~72 h和12~20 h;在贮藏后期巴氏杀菌乳中的细菌群落多样性降低到较低的水平,说明体系中出现了优势菌种。Chao1指数与Simpson指数变化规律表明3个贮藏温度下巴氏杀菌乳中细菌多样性都是随着贮藏时间增加而降低,但是降低发生的时间不同,而且贮藏温度越低,细菌群落结构变化越慢。丁瑞雪等[16]也发现巴氏杀菌乳在0 ℃贮藏能保持长时间的细菌多样性,而在10 ℃的贮藏过程中群落的物种丰度下降较快。
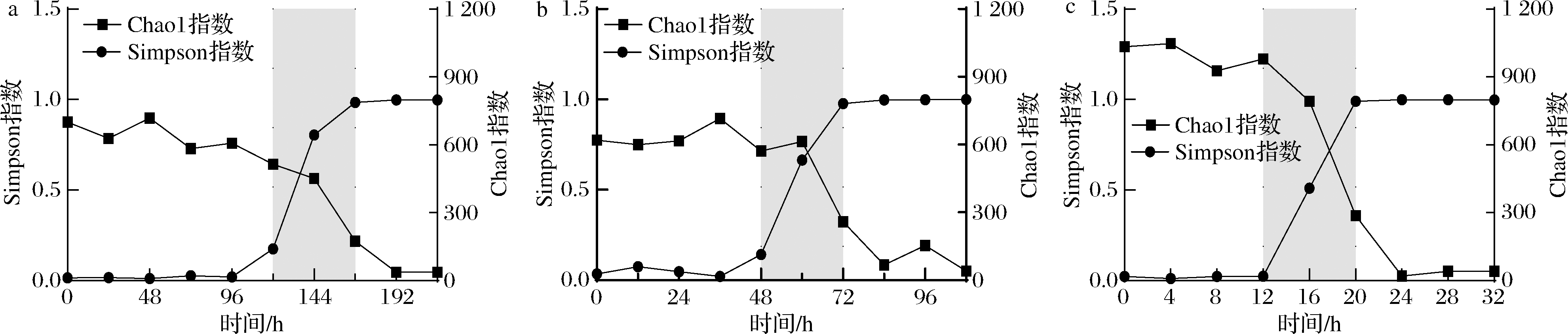
a-10 ℃;b-15 ℃;c-25 ℃
图2 巴氏杀菌乳贮藏过程中Chao 1指数和Simpson指数变化
Fig.2 Changes of Chao1 index and Simpson index during storage of pasteurized milk
使用Beta多样性分析呈现不同温度贮藏的巴氏杀菌乳间的细菌群落结构的相似性和差异性,在OTU水平上进行UniFrac的非加权PCoA,结果如图3所示,第一主坐标和第二主坐标的贡献率分别为33.20%和10.65%。15 ℃及25 ℃贮藏的样品呈现出一定的重叠现象,说明这两个温度贮藏的巴氏杀菌乳的细菌群落结构具有一定的相似性。而10 ℃贮藏的巴氏杀菌乳样品呈现出一定的聚类趋势,说明该温度下贮藏的样品相比于另外两个温度组具有特殊的群落结构。
综合以上多样性分析,发现贮藏温度会影响巴氏杀菌乳贮藏过程中细菌群落结构,并且控制其中物种的演替速度,进而影响产品的货架期。

图3 巴氏杀菌乳细菌群落的UniFrac非加权PCoA
Fig.3 PCoA of bacterial community in pasteurized milk based on unweighted UniFrac distance
2.3 巴氏杀菌乳细菌群落构成分析
基于OTU的物种分类分析,在相似性为97%水平下,不同样品的细菌种类共注释到32个门,其中含量最多的前5位依次为硬壁菌门(Firmicutes)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)和螺旋菌门(Spirochaetae)。由图4可知,在3个恒定温度保藏的巴氏杀菌乳,在贮藏过程中样品的各细菌门的变化趋势基本相同。硬壁菌门在贮藏始终占主导地位,在贮藏前期占比约为40%,贮藏后期硬壁菌门在菌群中进一步扩大优势成为绝对优势细菌门,杨雪[17]对巴氏杀菌乳模拟自然腐败过程也得到相同的结果。贮藏初期的次优势细菌门是变形菌门,随着贮藏时间的延长,其序列数量变化呈现出与硬壁菌门相反的趋势,表明在贮藏过程中两者之间可能存在相互作用[18]。

a-10 ℃;b-15 ℃;c-25 ℃
图4 基于门水平的巴氏杀菌乳中细菌丰度变化
Fig.4 Changes of bacterial abundance at the phylum level in pasteurized milk
注:相对丰度<0.01的合并为其他(图5同)
进一步对巴氏杀菌乳细菌群落组成进行分析,注释到的物种丰度共计766个属,其中丰度高于1%的属共计7个,分别为芽胞杆菌属(Bacillus)、肠球菌属(Enterococcus)、不动杆菌属(Acinetobacter)、螺菌科UCG-005属(Oscillospiraceae)、棒状杆菌属(Corynebacterium)、克雷伯氏菌属(Klebsiella)和罗氏斯通氏菌属(Ralstonia)。由图5可知,10 ℃贮藏的巴氏杀菌乳在0~96 h内其微生物多样性保持恒定,样品的细菌总数无明显变化;贮藏96 h后,肠球菌属和不动杆菌属等非优势菌属的丰度逐渐下降,而芽胞杆菌属的占比快速上升,与此同时菌落总数也在快速增加,芽胞杆菌属占据优势,细菌群落多样性下降。15 ℃贮藏的样品在前36 h内,肠球菌属先增加后减少,其余大部分菌属的丰度在保持一个动态恒定的阶段,但是传统平板的计数上,可培养的微生物数量从2.1 lgCFU/mL 增加到4.0 lgCFU/mL;在48~108 h内芽胞杆菌属的丰度快速上升,成为贮藏后期的优势菌属,菌落总数也在不断增加。巴氏杀菌乳在25 ℃贮藏的细菌菌落结构变化更加快速,贮藏4 h后肠球菌属的丰度明显下降,贮藏到12 h,微生物的数量增加了1个数量级,继续贮藏12 h,细菌总数达到7.3 lgCFU/mL,超出限量标准,细菌组成与另外两个温度结果相似,芽胞杆菌属仍是贮藏后期的优势菌属。
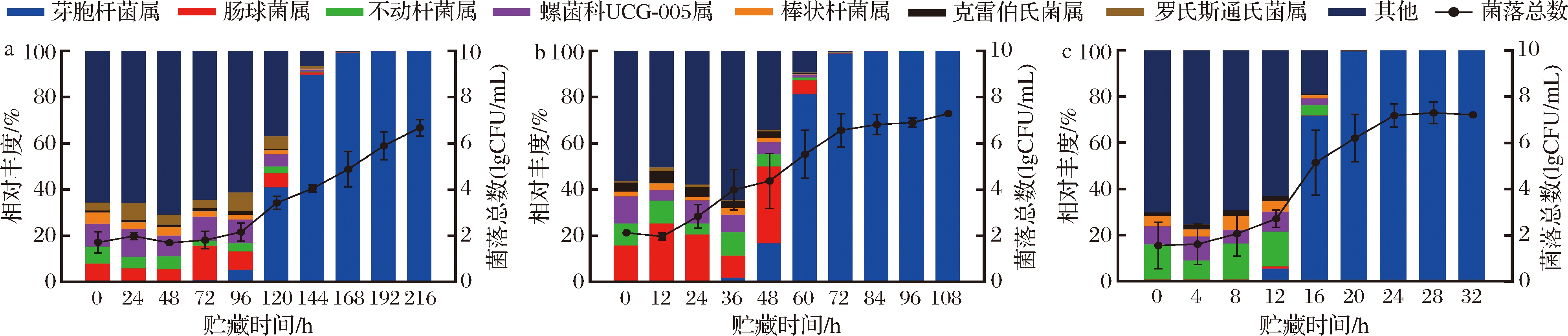
a-10 ℃;b-15 ℃;c-25 ℃
图5 巴氏杀菌乳细菌属水平上菌落丰度及菌落总数
Fig.5 Bacterial abundance at the genus level and aerobic bacterial count in pasteurized milk
2.4 巴氏杀菌乳细菌群落功能预测分析
基于Tax4Fun方法对巴氏杀菌乳中细菌群落进行基因组功能预测,该方法主要是通过构建SILVA分类与KEGG数据库中原核生物分类间的线性关系,实现对微生物群落功能的预测。本研究中获得了264个预测的通路功能,其中丰度前15的功能如图6所示。其中涉及氨基酸、核苷酸和糖类等代谢通路的有8项,3个温度下随着贮藏时间的推移,嘧啶、嘌呤等核苷酸代谢功能通路丰度下降,精氨酸、脯氨酸、半胱氨酸、蛋氨酸代谢通路丰度增加,丝氨酸和苏氨酸代谢通路丰度基本保持不变,此外,在贮藏过程中淀粉和蔗糖等糖类代谢通路丰度下降,发挥膜运输及信号传导等环境信息处理功能的通路丰度下降,氨酰合成及核糖体合成通路丰度下降,氨基酸和核苷酸糖代谢通路丰度在10 ℃和15 ℃贮藏过程中保持恒定,而在25 ℃贮藏过程中逐渐增加。由图6可知3个温度贮藏下的牛奶整体功能类型变化趋势相似,个别功能通路存在一定差异。

a-10 ℃;b-15 ℃;c-25 ℃
图6 巴氏杀菌乳中细菌功能预测分布
Fig.6 Function prediction distribution of bacteria in pasteurized milk
3 讨论
巴氏杀菌乳经过适度的热加工处理,保留了生乳中大量的营养成分及风味物质,深受消费者的喜爱。研究表明,巴氏杀菌乳的变质主要是热加工后残留细菌及后污染的细菌生长和繁殖所引起[19],在巴氏杀菌工艺中,生乳内大部分微生物被杀灭,部分耐热性细菌及芽胞存活并残留。这些微生物在后期的贮藏过程中缓慢复苏并生长,合适的贮藏条件下货架期内其品质和安全是可以保证的,但是在贮藏温度失控的情况下,残留细菌的生长代谢加快,造成产品腐败变质。因此,探索温度失控条件下巴氏杀菌乳贮藏过程中微生物的动态变化规律,对于产品质量控制及货架期预测,具有重要的理论意义和应用价值。
通过16S rRNA基因扩增子高通量测序技术对不同贮藏条件下巴氏杀菌乳的微生物多样性进行分析。研究结果表明,在测定3个温度的贮藏过程中,巴氏杀菌乳中细菌多样性都逐渐降低,芽胞杆菌属是引起巴氏杀菌乳自然腐败变质最主要的细菌属。此结果与MUGADZA等[20]分析延长货架期(extended shelf life,ESL)牛奶贮藏过程中细菌群落演变的研究结果一致。芽胞杆菌属在自然界中分布广泛,其形成的高耐热芽胞能抵抗生乳的热加工环节,部分细菌还会产生稳定的毒素[21],对乳品安全和消费者健康产生威胁。RASOLOFO等[22]通过微滤及添加CO2来抑制牛奶贮藏过程中嗜湿性腐败细菌的生长;LI等[23]使用二次热加工,第一次加热将芽胞杆菌减少到可接受的范围,第二次则是灭活萌发的芽胞,以延长牛奶的保质期。这些措施进一步保证了牛奶的安全,但过度加热会降低营养价值、影响风味。平衡乳品营养质量和微生物安全以及经济成本的关系,仍然是巴氏奶加工需要持续探索的问题。
非培养方法的分子生物学手段能检测出样本内部的活菌和死菌,传统的菌落总数检测方法可以定量食品内可培养细菌的数量,这两种方法各有优势,又互相补充[24]。在本研究中这两种方法显现出某些值得关注的共同特征:第一,牛奶贮存初期,可培养的细菌处在延滞期,这个阶段细菌多样性基本恒定;第二,细菌总数的对数增长期与物种多样性指数骤降的时间区间基本重合,但前者往往持续的更长,说明在牛奶贮藏过程的中后期,只有单一的物种或者菌属在快速生长,这点在细菌菌落组成上得到证实。这说明基于16S rRNA基因的细菌多样性分析方法可辅助判断巴氏杀菌乳的货架期,而腐败初期占比上升的芽胞杆菌属可作为品质下降的指示菌属。
在Tax4Fun功能预测分析中,样品中存在丰富的细胞代谢及环境信息处理功能相关基因,这些基因丰度的演变与乳品的品质相关[25]。在3个温度下的贮藏初期,牛奶中游离氨基酸的含量较少,氨基酸代谢通路的丰度较低,随着贮藏时间的延长,牛奶中蛋白质水解成氨基酸,微生物利用精氨酸、脯氨酸、半胱氨酸、蛋氨酸等氨基酸为生命活动提供能量,此类氨基酸代谢通路丰度上升。该功能分析结论与李蕊等[26]实测巴氏杀菌乳几种常见的氨基酸含量变化的结果一致。NIYAZBEKOVA等[27]对山羊奶进行微生物群落组成及功能预测分析,发现成熟乳相比于初乳,其细胞代谢、遗传信息处理及环境信息处理通路的丰度上升,与本文的结果相同。有研究表明异源生物降解和代谢、脂类代谢相关的途径与乳品风味之间存在相关性,因此这些基因可作为乳品感官质量的一个指标[28]。本研究中,巴氏杀菌乳在3个温度的贮存过程中功能通路丰度变化基本相同,可能与微生物菌群变化相同有关。
4 结论
本研究通过高通量测序技术揭示了不同贮藏温度下巴氏杀菌乳贮藏过程中细菌群落组成结构及功能演变规律,检测到32个细菌门,766个细菌菌属,多样性指数和丰富度指数表明贮藏过程中细菌结构组成逐渐单一化,是巴氏杀菌乳品质下降在菌相分析上的特征。芽胞杆菌属贮藏初期尽管相对丰度较低,但在温度失控的贮藏条件下仍对巴氏杀菌乳的细菌群落结构和营养品质有较大影响,且温度越高影响越大。本研究结果有助于了解温度对巴氏杀菌乳细菌群落组成的影响,为巴氏杀菌乳的品质控制提供理论依据。
[1] BARBANO D M, MA Y, SANTOS M V.Influence of raw milk quality on fluid milk shelf life[J].Journal of Dairy Science, 2006, 89:E15-E19.
[2] 王慧, 杨永龙, 张杰, 等.浅析巴氏杀菌奶在中国的发展前景[J].饮料工业, 2010, 13(11):4-7.
WANG H, YANG Y L, ZHANG J, et al.Analysis on prospects of pasteurized milk in China[J].Beverage Industry, 2010, 13(11):4-7.
[3] 杨茜茜. 消费者对进口牛奶的偏好和支付意愿研究[D].无锡:江南大学, 2021.
YANG Q Q.Research on consumers′ preference and willingness to pay for imported milk[D].Wuxi:Jiangnan University, 2021.
[4] BURGESS S A, LINDSAY D, FLINT S H.Thermophilic bacilli and their importance in dairy processing[J].International Journal of Food Microbiology, 2010, 144(2):215-225.
[5] ELWELL M W, BARBANO D M.Use of microfiltration to improve fluid milk quality[J].Journal of Dairy Science, 2006, 89:E20-E30.
[6] VITHANAGE N R, DISSANAYAKE M, BOLGE G, et al.Biodiversity of culturable psychrotrophic microbiota in raw milk attributable to refrigeration conditions, seasonality and their spoilage potential[J].International Dairy Journal, 2016, 57:80-90.
[7] KABLE M E, SRISENGFA Y, LAIRD M, et al.The core and seasonal microbiota of raw bovine milk in tanker trucks and the impact of transfer to a milk processing facility[J].mBio, 2016, 7(4):e00836-e00816.
[8] DERAKHSHANI H, TUN H M, KHAFIPOUR E.An extended single-index multiplexed 16S rRNA sequencing for microbial community analysis on MiSeq illumina platforms[J].Journal of Basic Microbiology, 2016, 56(3):321-326.
[9] 闫艳华, 曹慧慧, 董李学, 等.高通量测序技术对不同采奶环节细菌多样性的研究[J].中国乳品工业, 2021, 49(3):24-28.
YAN Y H, CAO H H, DONG L X, et al.Study on the diversity of bacteria in different milk collection stages by high throughput sequencing technology[J].China Dairy Industry, 2021, 49(3):24-28.
[10] PORCELLATO D, ASPHOLM M, SKEIE S B, et al.Microbial diversity of consumption milk during processing and storage[J].International Journal of Food Microbiology, 2018, 266:21-30.
[11] 郑钰倩, 喻勇新, 赵海霞, 等.上海市市售低温酸奶中细菌多样性的初步研究[J].食品与发酵工业, 2022,48(6):58-63.
ZHENG Y Q, YU Y X, ZHAO H X, et al.Preliminary study on bacterial diversity in low-temperature yogurt from Shanghai supermarket[J].Food and Fermentation Industries, 2022,48(6):58-63.
[12] ![]() T, SALZBERG S L.FLASH:Fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics, 2011, 27(21):2 957-2 963.
T, SALZBERG S L.FLASH:Fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics, 2011, 27(21):2 957-2 963.
[13] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods, 2010, 7(5):335-336.
[14] OLSON N D, SHAH N, KANCHERLA J, et al.Metagenome Features:An R package for working with 16S rRNA reference databases and marker-gene survey feature data[J].Bioinformatics, 2019, 35(19):3 870-3 872.
[15] ASSHAUER K P, WEMHEUER B, DANIEL R, et al.Tax4Fun:Predicting functional profiles from metagenomic 16S rRNA data[J].Bioinformatics, 2015, 31(17):2 882-2 884.
[16] 丁瑞雪, 耿丽娟, 张铁华, 等.基于下一代测序技术分析巴氏杀菌乳中残留细菌在贮藏期间的动态变化[J].食品科学, 2019, 40(14):77-83.
DING R X, GENG L J, ZHANG T H, et al.Dynamic analysis of changes in residual bacteria in pasteurized milk during storage based on next-generation sequencing[J].Food Science, 2019, 40(14):77-83.
[17] 杨雪. 巴氏奶腐败过程中微生物群落及腐败微生物溯源[D].昆明:昆明理工大学, 2018.
YANG X.Microorganism during pasteurized milk corruption process and spoilage microorganism origin[D].Kunming:Kunming University of Science and Technology, 2018.
[18] 布仁其其格, 高雅罕, 任秀娟, 等.不同发酵时期酸马奶细菌群落结构[J].食品科学, 2016, 37(11):108-113.
BURENQIQIGE, GAO Y H, REN X J, et al.Dynamic changes of bacteria community structure during koumiss fermentation[J].Food Science, 2016, 37(11):108-113.
[19] HE H F, DONG J, LEE C N, et al.Molecular analysis of spoilage-related bacteria in pasteurized milk during refrigeration by PCR and denaturing gradient gel electrophoresis[J].Journal of Food Protection, 2009, 72(3):572-577.
[20] MUGADZA D T, BUYS E.Bacillus and Paenibacillus species associated with extended shelf life milk during processing and storage[J].International Journal of Dairy Technology, 2018, 71(2):301-308.
[21] DOUGHARI H J, NDAKIDEMI P A, HUMAN I S, et al.The ecology, biology and pathogenesis of Acinetobacter spp.:An overview[J].Microbes and Environments, 2011, 26(2):101-112.
[22] RASOLOFO E A, ST-GELAIS D, LAPOINTE G, et al.Molecular analysis of bacterial population structure and dynamics during cold storage of untreated and treated milk[J].International Journal of Food Microbiology, 2010, 138(1-2):108-118.
[23] LI F, HUNT K, BUGGY A K, et al.The effects of sequential heat treatment on microbial reduction and spore inactivation during milk processing[J].International Dairy Journal, 2020, 104:104648.
[24] 李引强, 朱宝利, 吴俊, 等.16S rRNA的分子生物学方法分析牛奶中的细菌菌群[J].食品科学, 2013, 34(20):255-260.
LI Y Q,ZHU B L,WU J, et al.Molecular biological analysis of bacterial community in milk based on 16S rRNA gene[J].Food Science, 2013, 34(20):255-260.
[25] CHI F M, TAN Z K, GU X D, et al.Bacterial community diversity of yak milk dreg collected from Nyingchi region of Tibet, China[J].LWT-Food Science and Technology, 2021, 145:111308.
[26] 李蕊, 王一然, 刘丽云, 等.贮存温度对巴氏杀菌乳中游离氨基酸组成及品质的影响[J].中国乳品工业, 2020, 48(1):8-13.
LI R, WANG Y R, LIU L Y, et al.Effects of storage temperature on the composition and flavor of free amino acids in pasteurized milk[J].China Dairy Industry, 2020, 48(1):8-13.
[27] NIYAZBEKOVA Z, YAO X T, LIU M J, et al.Compositional and functional comparisons of the microbiota in the colostrum and mature milk of dairy goats[J].Animals, 2020, 10(11):1 955.
[28] YANG C C, ZHAO F Y, HOU Q C, et al.PacBio sequencing reveals bacterial community diversity in cheeses collected from different regions[J].Journal of Dairy Science, 2020, 103(2):1 238-1 249.