烟酰胺是一种辅助性药物,是烟酸的酰胺形式,众多研究中表明其参与抗炎和抗衰老过程[1],现已广泛用于治疗各种皮肤病,如高危人群的非黑色素瘤皮肤癌[2]。目前,工业上生物法产烟酰胺在渐渐取代化学合成法,主要是因为生物法大大提高了产物的纯度,降低了可导致血管舒张等副作用的副产物烟酸[3]的含量,节约了分离成本。
腈水合酶(nitrile hydratase,NHase,EC 4.2.1.84)是一种金属酶,在微生物的腈代谢过程中催化腈类化合物生成相应的酰胺。由于腈类及酰胺类化合物属于有机溶剂,其对酶蛋白的损害较大,或改变了酶蛋白的构象,或导致酶蛋白表面必需水分子的丢失[4],且水合反应是一个放热反应。因此,寻求高稳定性的腈水合酶一直是研究的热点。
本课题组于2020年在GenBank数据库中寻找到一种嗜热菌温泉热碱芽胞杆菌(Caldalkalibacillus thermarum) TA2.A1来源的腈水合酶基因[Cal.tNHase,GenBank:EGL84005.1 (α亚基), EGL84004.1(β亚基)],实现了其在大肠杆菌工程菌株内的异源表达,并初步研究了该酶的酶学性质和催化性能[5]。该酶稳定性已满足工业应用的需求,但其催化烟腈的活性尚有提升的空间。
在此之前,研究采用各种蛋白质工程的方法以提高NHase的催化活性,如结构域交换和亚基融合,然而,所获得的NHase的性质并不总是令人满意[6]。近年来,酶的底物通道受到越来越多的关注,其对酶的催化性能起着关键作用[7]。定制底物通道可以显著提高酶的活性、选择性和稳定性,是一种新的有效策略[8-9]。本文将通过此方法来提高Cal.tNHase的活性,以提高其应用潜力。
1 材料与方法
1.1 实验材料
1.1.1 菌株与质粒
大肠杆菌JM109感受态用于基因克隆,大肠杆菌BL21(DE3)感受态用于蛋白表达,均为实验室前期保藏菌株;质粒 pET-24a(+),由本实验室保藏。
1.1.2 酶与主要试剂
Prime STAR MAX 高保真酶、Quick Cut Dpn I 消化酶,大连宝生物工程有限公司;3-氰基吡啶、烟酰胺,上海麦克林生化科技有限公司;葡萄糖、氨水、培养基中所用各种盐类,国药化学试剂有限公司;胰蛋白胨、酵母提取物,英国Oxoid公司。
1.1.3 培养基
LB培养基(g/L):胰蛋白胨10.0, 酵母提取物5.0, NaCl 10.0。
2YT培养基(g/L):胰蛋白胨16.0, 酵母提取物10.0, NaCl 5.0, IPTG终浓度0.4 mmol/L, Co 0.1。
5 L发酵罐培养基(g/L):胰蛋白胨16.0, 酵母提取物10.0, 葡萄糖10.0, KH2PO4 4.6, (NH4)2PO4 34.0, 二水合柠檬酸三钠7.4, 微量元素母液10 mL。
补料培养基(g/L):葡萄糖500.0, MgSO4 7.4, 酵母提取物12.0, 胰蛋白胨12.0。
诱导剂母液(g/100 mL):IPTG 0.286, CoCl2·6H2O 1.8。
微量元素母液(g/L):FeSO4·7H2O 10.0,CuSO4·5H2O 3.0,MnSO4·H2O 0.38,ZnSO4·7H2O 5.25,(NH4)6Mo7O24 0.11,Na2B4O7·10H2O 0.23,CaCl2·2H2O 2.65,溶于1 mol/L HCl中。
1.2 实验方法
1.2.1 计算机半理性设计方法
Cal.tNHase的晶体结构目前尚未被解析。根据PSI-BLAST的检索结果,该酶α亚基与来源于Bacillus sp.RAPc8的腈水合酶(PDB ID:2DPP)的同源性为86%,其β亚基与来源于Bacillus smithii SC-J05-1的腈水合酶(PDB ID:1V29)同源性最高,达63.6%,于是以此为模板,使用trRosetta在线工具构建出Cal.tNHase的三维结构[10]。PROCHECK输出的拉式图[11](Ramachandran plot)表明所构建的模型是可靠的。随后三维模型经NAMD软件[12]进行50 ns的分子动力学模拟,继续对其结构进行进一步优化,所用力场为Charmm力场。底物通道的计算分析使用的是CAVER Analyst 2.0[13]软件,最小探针半径0.9 Å,检测深度4 Å,底物通道起始位点为腈水合酶活性中心的几何中心。
1.2.2 突变体库的构建
以温泉热碱芽胞杆菌来源的腈水合酶构建的重组质粒 pET-24a(+)-Cal.tNHase 为模板,采取全质粒PCR对预测的突变位点进行单点的代表性氨基酸(A、D、F、H、K)突变,将构建好的质粒转入E.coli JM109中,送测序,将测序结果正确的质粒重新转入E.coli BL21(DE3)中备用。
1.2.3 Cal.tNHase和突变体的表达与纯化
将上述构建成功的菌株, 分别在含有50 g/mL卡那霉素的LB平板上划线,37 ℃倒置培养过夜,挑取单菌落置于含有50 g/mL卡那霉素的5 mL 2YT液体培养基的试管中,37 ℃、200 r/min培养8~10 h将培养好的试管种子液以1%(体积分数,下同)的接种量转接到含有50 g/mL卡那霉素的100 mL 2YT培养基摇瓶中,37 ℃、200 r/min培养至OD600 为0.6~0.8时,添加终浓度为0.4 mmol/L IPTG、0.1 g/L CoCl2·6H2O的诱导剂,30 ℃、200 r/min诱导14 h。
表1 本文所用引物及其序列
Table 1 Primers used in this study

引物名称引物序列5′-3′αL89A-FTATTATGGTGCACAAGGTGAACATATCCGCGTGαL89D-FTATTATGGTGACCAAGGTGAACATATCCGCGTGGαL89F-FTATTATGGTTTTCAAGGTGAACATATCCGCGαL89H-FTATTATGGTCATCAAGGTGAACATATCCGCGTGGαL89K-FTATTATGGTAAACAAGGTGAACATATCCGCGTGβL37A-FTTTGGTTTAGCCATTGGTACCGCCGGTCAAGβL37D-FTTTGGTTTAGATATTGGTACCGCCGGTCAAGβL37F-FTTTGGTTTATTCATTGGTACCGCCGGβL37H-FTTTGGTTTACATATTGGTACCGCCGGTCAAGβL37K-FTTTGGTTTAAAAATTGGTACCGCCGGTCAAGβA41D-FTAATTGGTACCGATGGTCAAGGTCTGTACβA41F-FTAATTGGTACCTTCGGTCAAGGTCTGTACAATβA41H-FTAATTGGTACCCACGGTCAAGGTCTGTACAATβA41K-FTAATTGGTACCAAAGGTCAAGGTCTGTACAATTβG42A-FGGTACCGCCGACCAAGGTCTGTACAATTβG42D-FGTACCGCCGATCAAGGTCTGTACβG42F-FGGTACCGCCTTTCAAGGTCTGTACAATTTAGATβG42H-FGGTACCGCCCATCAAGGTCTGTACAATTTAGβG42K-FTGGTACCGCCAAACAAGGTCTGTACAATTTAGβY46A-FCAAGGTCTGGCCAATTTAGATGAGTTCCGCCβY46D-FCAAGGTCTGGATAATTTAGATGAGTTCCGCβY46F-FCAAGGTCTGTTCAATTTAGATGAGTTCCGβY46H-FCAAGGTCTGCACAATTTAGATGAGTTCCβY46K-FCAAGGTCTGAAAAATTTAGATGAGTTCCGCCβV121A-FCTGGAGCAAGACATTAAGGTGGGTGTTAGβV121D-FCTGGAGCAAGATATTAAGGTGGGTGβV121F-FCTGGAGCAATTTATTAAGGTGGGTGβV121H-FCTGGAGCAACATATTAAGGTGGGTGTTAGCβV121K-FCTGGAGCAAAAAATTAAGGTGGGTGTTAGCACβV126A-FCAAGTTATTAAGGTGGGTGACAGCACCGTGCβV126D-FTTATTAAGGTGGGTGATAGCACCGTGCβV126F-FGTTATTAAGGTGGGTTTTAGCACCGTGCβV126H-FCAAGTTATTAAGGTGGGTCATAGCACCGTGCβV126K-FCAAGTTATTAAGGTGGGTAAAAGCACCGTGCCommon-RTTTTGCCAGTTCTTCTTCGGTCAT
注:划线部分表示突变位点
将上述培养的菌液以12 000 r/min离心后收集菌体,再用结合缓冲液(20 mmol/L Na2HPO4、280 mmol/L NaCl、6 mmol/L KCl,pH 7.4)浓缩5倍后重悬浮,于超声波破碎仪中以开3 s停7 s破碎20 min,然后将破碎液12 000 r/min离心30 min,取上清液过0.22 μm膜后置于冰上备用。纯化使用strep亲和层析,先准备好除菌且除气泡的超纯水、结合缓冲液、洗涤缓冲液(20 mmol/L Na2HPO4、280 mmol/L NaCl、6 mmol/L KCl、2.5 mmol/L脱硫生物素,pH 7.4)。先用超纯水将柱子中保存的乙醇溶液至少以5倍体积冲洗干净;再用结合缓冲液平衡柱子,紫外吸收调零;上样10~15 mL, 再以结合缓冲液平衡柱子,使紫外吸收降到0;然后用洗涤缓冲液洗脱,收集目的蛋白。纯化结束后,将收集到的蛋白用Bradford法标定蛋白质量浓度,SDS-PAGE测定纯度,最后将蛋白质量浓度调整到0.05 mg/mL备用。
1.2.4 水合活性的测定
腈水合酶反应体系:10 μL质量浓度0.05 mg/mL的蛋白或OD600=1的细胞液加入490 μL含200 mmol/L的3-氰基吡啶的水溶液,25 ℃下反应10 min,再加入500 μL乙腈进行终止。将反应后的样品过0.22 μm的膜除菌,用HPLC过C18柱检测其酶活力,流动相为V(乙腈)∶V(水)=1∶2的混合液,检测波长215 nm,检测温度40 ℃,流动相流速0.6 mL/min,进样量10 μL,监测10 min,每组数据至少进行3次平行实验。
酶活力定义:25 ℃下1 min内生成1 μmol烟酰胺所需要的酶量(U)。比酶活力定义为1 mg蛋白所具有的酶活力(U/mg);细胞酶活力定义为1 mL发酵液细胞所具有的酶活力(U/mL)。
1.2.5 酶学性质研究
热稳定性:将NHase进行表达纯化,统一调整蛋白质量浓度到0.1 mg/mL,将酶液置于金属浴设备中,设置金属浴设备温度65 ℃,处理酶液0、0.2、0.5、1、2、4 h,取样进行酶活力测定,定义每个酶在处理时间为0 h时酶活力为100%,分析其热稳定性。
底物及产物耐受性:将NHase进行培养诱导,然后离心菌液收集菌体,将OD600=8的菌体分别置于3-氰基吡啶浓度为0、1 mol/L和烟酰胺浓度为0、2 mol/L的水溶液中,25 ℃孵育30 min,12 000 r/min离心1 min后去除上清液,再用10 mmol/L的磷酸缓冲液清洗菌体2次,测其菌体酶活力,定义每个酶用不含3-氰基吡啶和烟酰胺水溶液处理后酶活力为100%,分析其底物及产物耐受性情况。
1.2.6 5 L罐产酶发酵
从-80 ℃保存的甘油管内用接种环沾取菌液,在含有50 g/mL卡那霉素的LB平板上划线,37 ℃培养过夜,挑取单菌落与含有50 g/mL卡那霉素的30 mL LB液体培养基培养8~10 h,再以1%接种量转接于新鲜的含有50 g/mL卡那霉素的30 mL LB液体培养基中进行扩培7.5 h。最后将扩培后的菌液接入5 L发酵罐中,接种量为6%,卡那霉素添加量为0.1%(质量分数)。发酵罐的初始培养体积为2 L,pH 7.0,培养温度37 ℃,搅拌桨转速200 r/min,通气量3 L/min,此时将罐内的溶氧标定为100%。在接种后,发酵罐内的溶氧与转速偶联,维持溶氧在30%,最大通气量为7 L/min,最大搅拌桨转速1 000 r/min。发酵进行到6 h左右出现溶氧反弹现象,此时初始培养基中的碳源基本耗尽,开始以指数流加的方式进行补料,流加时控制菌体比生长速率μ=0.2 h-1。当细胞 OD600达到60左右时,降低温度为30 ℃,以15 mL/h的恒流速流加诱导剂进行诱导,直至100 mL诱导剂全部补完。整个发酵过程中流加氨水进行pH调节,维持罐内发酵液pH=7.0。发酵过程在开始补料后以2 h间隔取样,监测其生长状况,记录OD600和葡萄糖浓度,样品采用HPLC测定水合活性。
2 结果与分析
2.1 Cal.tNHase突变体的筛选
利用trRosetta在线建模工具构建了Cal.tNHase的三维结构模型,并使用PROCHECK获取了所构建模型的拉式图,拉式图结果表明所构建的模型是可靠的(图1)。

图1 Cal.tNHase三维结构拉式图
Fig.1 Main Ramachandran plot of the constructed model of Cal.tNHase
在此基础上,通过CAVER底物通道预测工具,预测定位了如图2所示的8个位于底物通道上的氨基酸残基αL89、βL37、βA41、βG42、βY46、βL48、βV121、βV126。
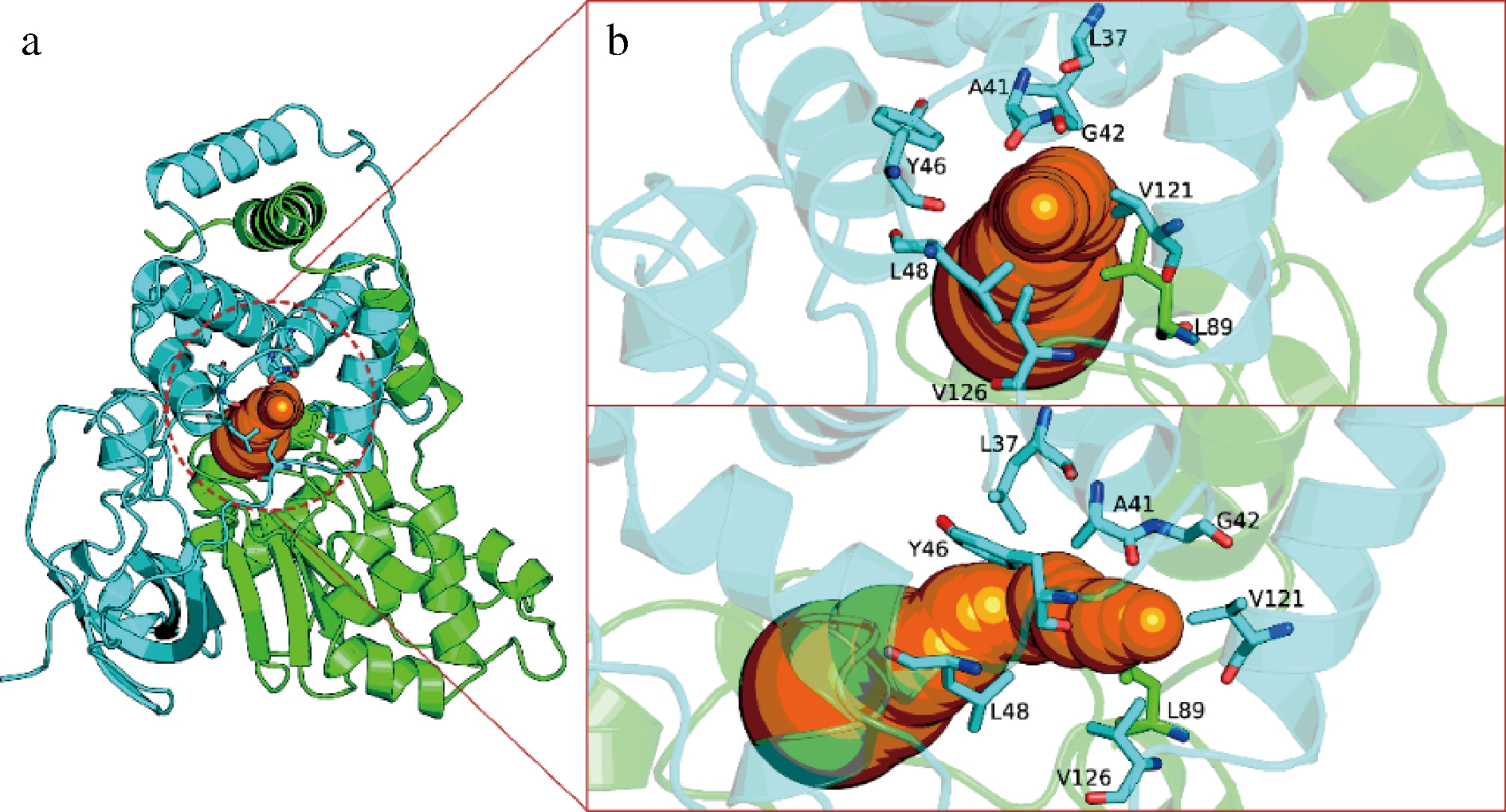
图2 Cal.tNHase结构模型及其底物通道模型
Fig.2 Cartoon model of Cal.tNHase and its substrate access tunnel
由于βLeu48位点已在前期研究中筛选,本研究对余下7个位点进行突变库的构建。为提高筛选效率且保证突变体库中氨基酸类型的全面性,将其分别突变为其他5种具有代表性氨基酸:侧链带负电的天冬氨酸(D)、带芳香族侧链的苯丙氨酸(F)、侧链带正电的赖氨酸、组氨酸(K、H)以及带非极性侧链的丙氨酸(A),共计33个突变体,研究底物通道上氨基酸残基的种类对腈水合酶活性的影响。首先,将野生型腈水合酶和突变体进行培养诱导,测其细胞酶活力。调整细胞液OD600=1,结果如图3所示,野生型Cal.tNHase酶活力为21.4 U/mL,在这33个突变体中,αL89H、βG42K的酶活力有一定的提高,分别为37.8 和42.2 U/mL。

图3 Cal.tNHase突变体细胞酶活力
Fig.3 Catalytic activity of cells harboring Cal.tNHase mutants
为了进一步促进腈水合酶活力的提高,遂将αL89H和βG42K进行组合突变。野生型以及这3个突变体经诱导表达后分离纯化,测得的比酶活力数据如图4所示。突变体βG42K比酶活力为553.0 U/mg,是野生型(220.3 U/mg)的2.5倍,但在与αL89H进行组合突变后,酶活力未得到进一步提高。
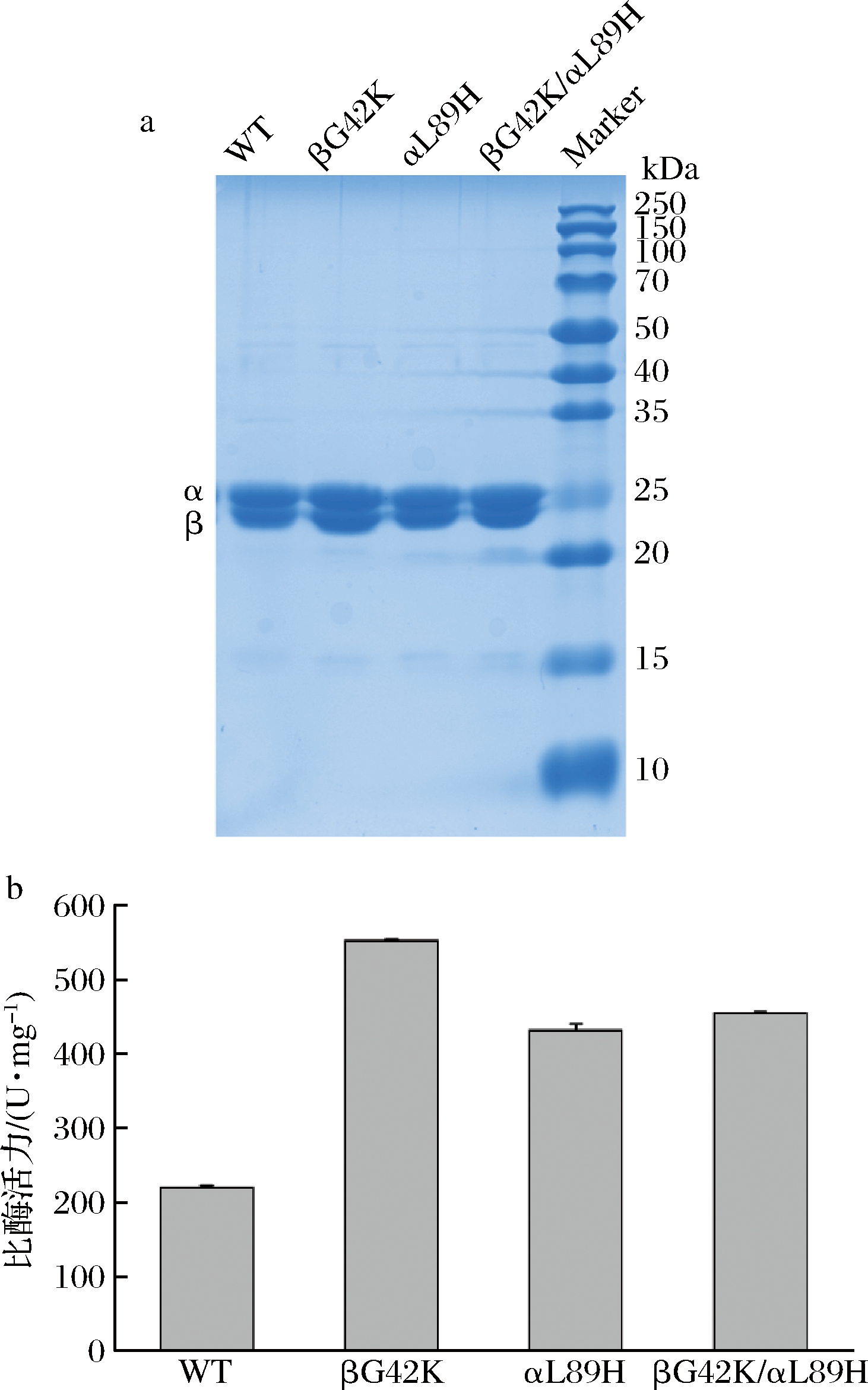
a-突变化的表达纯化;b-突变化比酶活力
图4 突变体表达纯化及比酶活力比较
Fig.4 Expression and purification of the mutants and their specific activities toward 3-cyanopyridine
注:WT-野生型
2.2 Cal.tNHase-βG42K突变体的酶学性质
经过筛选得到的突变体βG42K催化活性得到了一定的提高,但可能使其稳定性有所降低,所以从热稳定性、底物耐受性、产物耐受性方面与其野生型进行了比较,结果如图5-a所示。野生型酶在65 ℃孵育3 h后仍保持其初始活性的50%左右,而βG42K突变体在65 ℃热处理1.5 h后失去一半的活性,但在热处理的过程中,βG42K突变体的比酶活力一直高于野生型。据报道,现有热稳定性最优的腈水合酶来源于芽胞杆菌菌株RAPc8,该酶在50 ℃下热处理2.5 h后可保持一半活性,然而在60 ℃下热处理20 min后,活性丧失一半以上[14]。显然,研究所获得的突变体βG42K的热稳定性更具优势,具备一定的应用前景。
腈水合酶的水合过程能否持续进行与其本身对腈类底物或酰胺产品的耐受性有关。因此实验中用高浓度的3-氰基吡啶和烟酰胺来处理Cal.tNHase野生型和βG42K突变体,检测其残余酶活力,分析酶的底物及产物耐受性。如图5-b所示,突变体βG42K经1 mol/L 3-氰基吡啶处理30 min后,相对酶活力为48.2%,而Cal.tNHase野生型为59.3%。经2 mol/L烟酰胺处理30 min后,突变体βG42K保留了67.2%,野生型为63.3%(图5-c)。
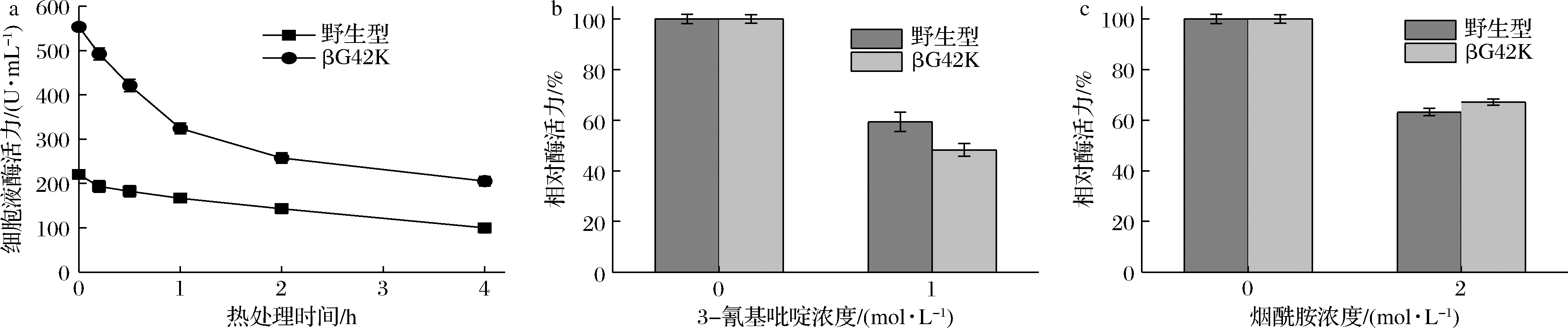
a-65 ℃热处理时间;b-3-氰基吡啶浓度;c-烟酰胺浓度
图5 Cal.tNHase βG42K酶学性质
Fig.5 Enzymatic properties of the Cal.tNHase bG42K
2.3 突变株5 L罐产酶发酵
经过上述验证,突变体βG42K的催化性能基本满足应用所需,遂将此菌株进行5 L罐的放大培养。采用分批补料的方式,在OD600=60时开始诱导,菌体前期培养温度37 ℃,诱导温度30 ℃。如图6所示,整个补料过程中葡萄糖积累量不超过1 g/L,在发酵时长为25 h时菌体OD600最高为110,此时细胞酶活力是5 539.0 U/mL,为已报道的WANG等[15]研究中细胞酶活力的2倍,说明突变体βG42K相较于此前的研究在发酵酶活力方面更具优势。
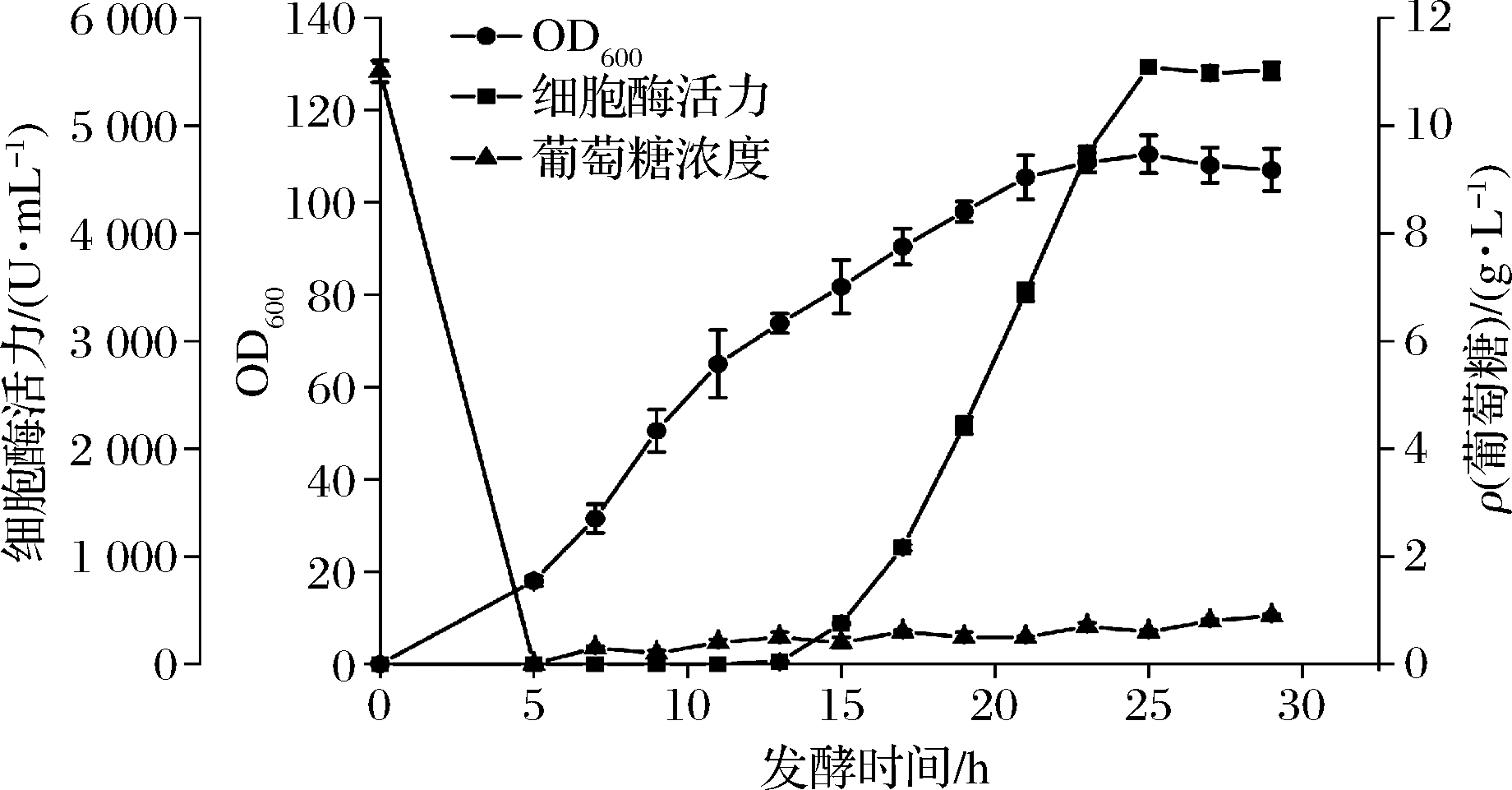
图6 突变株βG42K高密度发酵
Fig.6 High-density fermentation of mutant strain βG42K
3 结论
本研究成功通过定制底物通道提高了Cal.tNHase的活力,筛选出的突变体βG42K比酶活力是其野生型的2.5倍。该突变体在65 ℃下的半衰期仍有1.5 h,且对底物3-氰基吡啶和产品烟酰胺的耐受性并未大幅度降低,能满足工业生产的需要。通过对突变体βG42K进行放大发酵,在5 L罐上完成高密度发酵,细胞酶活力达到5 539.0 U/mL。
[1] YOSHINO J, BAUR J A, IMAI S I.NAD(+) intermediates:The biology and therapeutic potential of NMN and NR[J].Cell Metabolism, 2018, 27(3):513-528.
[2] SCATOZZA F, MOSCHELLA F, D’ARCANGELO D, et al.Nicotinamide inhibits melanoma in vitro and in vivo[J].Journal of Experimental & Clinical Cancer Research:CR, 2020, 39(1):211.
[3] RATNARAJAH K, ZARGHAM H, JAFARIAN F.Confusion among different forms of vitamin B3[J].Journal of Cutaneous Medicine and Surgery, 2020, 24(6):642-643.
[4] CUI H Y, ELTOUKHY L, ZHANG L L, et al.Less unfavorable salt bridges on the enzyme surface result in more organic cosolvent resistance[J].Angewandte Chemie (International Ed.in English), 2021, 60(20):11 448-11 456.
[5] 张赛兰, 李婷, 程中一, 等.新型耐热腈水合酶的异源表达及其催化工艺研究[J].食品与发酵工业, 2020, 46(14):108-113.
ZHANG S L, LI T, CHENG Z Y, et al.Heterologous expression of a novel thermostable nitrile hydratase and its catalytic process[J].Food and Fermentation Industries, 2020, 46(14):108-113.
[6] GUO J L, CHENG Z Y, BERDYCHOWSKA J, et al.Effect and mechanism analysis of different linkers on efficient catalysis of subunit-fused nitrile hydratase[J].International Journal of Biological Macromolecules, 2021, 181:444-451.
[7] GORA A, BREZOVSKY J, DAMBORSKY J.Gates of enzymes[J].Chemical Reviews, 2013, 113(8):5 871-5 923.
[8] KOKKONEN P, BEDNAR D, PINTO G, et al.Engineering enzyme access tunnels[J].Biotechnology Advances, 2019, 37(6):107386.
[9] SUBEDI B P, FITZPATRICK P F.Mutagenesis of an active-site loop in tryptophan hydroxylase dramatically slows the formation of an early intermediate in catalysis[J].Journal of the American Chemical Society, 2018, 140(15):5 185-5 192.
[10] YANG J, ANISHCHENKO I, PARK H, et al.Improved protein structure prediction using predicted interresidue orientations[J].Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(3):1 496-1 503.
[11] LASKOWSKI R A, MACARTHUR M W, MOSS D S, et al.PROCHECK:A program to check the stereochemical quality of protein structures[J].Journal of Applied Crystallography, 1993, 26(2):283-291.
[12] PHILLIPS J C, BRAUN R, WANG W, et al.Scalable molecular dynamics with NAMD[J].Journal of Computational Chemistry, 2005, 26(16):1 781-1 802.
[13] JURCIK A, BEDNAR D, BYSKA J, et al.CAVER Analyst 2.0:Analysis and visualization of channels and tunnels in protein structures and molecular dynamics trajectories[J].Bioinformatics, 2018, 34(20):3 586-3 588.
[14] PEREIRA R A, GRAHAM D, RAINEY F A, et al.A novel thermostable nitrile hydratase[J].Extremophiles:Life Under Extreme Conditions, 1998, 2(3):347-357.
[15] WANG Z, LIU Z M, CUI W J, et al.Establishment of bioprocess for synthesis of nicotinamide by recombinant Escherichia coli expressing high-molecular-mass nitrile hydratase[J].Applied Biochemistry and Biotechnology, 2017, 182(4):1 458-1 466.