食源性致病菌是影响食品安全最主要的因素之一。大肠埃希氏菌、金黄色葡萄球菌、沙门氏菌、溶藻弧菌、副溶血弧菌、铜绿假单胞菌、产气荚膜梭菌、单增李斯特菌和创伤弧菌都是即食食品中广受关注的食源性致病菌,常引起急性胃肠炎,并伴有恶心、头痛、低热,严重时可造成死亡[1-2]。由于这9种致病菌可同时长期存活于水体环境中,增加贝类产品在养殖过程中致病菌污染的可能性,进而在人群食用过程中产生食物中毒[3]。因此建立一种可靠的、针对这9种致病菌检测的快速方法,对于食品安全、健康和民生都有重要意义。
目前已有关于贝类产品中食源性病毒(如诺如病毒)快速检测的报道[4-5],但尚缺乏生蚝中大肠埃希氏菌、金黄色葡萄球菌、沙门氏菌、溶藻弧菌、副溶血弧菌、产气荚膜梭菌、铜绿假单胞菌、单增李斯特菌和创伤弧菌的高通量检测手段的研究。传统基于选择性培养基和生化分析的方法检测这9种致病菌,具有检测程序复杂、耗时长的缺点,并且可能会因方法的低灵敏度和低特异性导致大量的错检[6-7]。过去10年,基于传统PCR的方法发展迅速,该方法具有分析时间短,节省人力等优点[8]。
多重实时荧光串联PCR(multiplexed tandem PCR,MT-PCR)结合了多重PCR、巢式PCR和荧光定量PCR技术,可实现对食源性致病菌的高通量快速检测。该方法针对一个靶基因设计内、外2对引物,首先使用混合的外引物,进行一个循环数较少的高通量多重富集PCR,然后将第一轮多重富集PCR的产物用ddH2O稀释作为第二轮的模板,其次再使用内引物进行第二轮巢式荧光定量PCR,结果根据扩增曲线和熔解曲线分析。基于此原理,MT-PCR已被应用于检测临床样品中的病毒[9]、真菌[10]和转基因食品[11]。
本研究通过设计特性引物、优化MT-PCR参数,建立了一种可同时检测以上9种致病菌的方法,并将该方法用于检测人工染菌的生蚝样品,评价了该技术的定量能力和检测灵敏度,为快速检测贝类产品中食源性致病菌提供技术支持。
1 材料与方法
1.1 材料与试剂
1.1.1 样品与菌种
大肠埃希氏菌(Escherichia coli)、金黄色葡萄球菌(Staphylococcus aureus)、沙门氏菌(Salmonella)、溶藻弧菌(Vibrio alginolyticus)、副溶血弧菌(Vibrio parahaemolyticus)、铜绿假单胞菌(Pseudomonas aeruginosa)和单增李斯特菌(Listeria monocytogenes),暨南大学食品科学与工程系,创伤弧菌(Vibrio vulnificus)、产气荚膜梭菌(Clostridium perfringens),广东省微生物菌种保藏中心,9株目标菌株用于建立MT-PCR方法。
表皮葡萄球菌(Staphylococcus epidermidis)、化脓性链球菌(Staphylococcus pyogenes)、腐生葡萄球菌(Staphylococcus saprophyticus)、沃氏葡萄球菌(Staphylococcus warneri)、肠道沙门氏菌(Salmonella enteritidis)、霍利斯弧菌(Vibrio hollisae)、拟态弧菌(Vibrio mimicus)、长双歧杆菌(Bifidobacterium longum)、普雷沃氏菌(Prevotella melaninogenica)、嗜黏蛋白阿克曼菌(Akkermansia Muciniphila)、英诺克李斯特菌(Listeria innocua)、威尔氏李斯特菌(Listeria welshimeri)、肺炎克雷伯氏菌(Klebsiella pneumoniae)、荧光假单胞菌(Pseudomonas fluorescens)、粘质沙雷氏菌(Serratia marcescens)、溶血链球菌(Streptococcus haemolyticus)、嗜热链球菌(Streptococcus thermophilus)、弗氏柠檬酸杆菌(Citrobacter freundii)、奇异变形杆菌(Proteus mirabilis)保存于暨南大学食品科学与工程系,19株非目标菌株用于MT-PCR特异性检验。
实验采用的人工污染贝类产品为生蚝,广州水产市场。
1.1.2 试剂与仪器
营养琼脂(nutrient agar medium,NA)、脑心浸出液(brain heart infusion,BHI)、3% NaCl碱性蛋白胨水(alkaline peptone water,APW)、胰胨-亚硫酸盐-环丝氨酸琼脂基础(tryptone-sulphite-cycloserine,TSC)、液体硫乙醇酸盐培养基(fluid-thioglycollate,FT)、胰蛋白胨大豆琼脂(tryptose soya agar,TSA),广东环凯微生物科技有限公司;DP302细菌基因组DNA提取试剂盒、DP324海洋动物基因组DNA提取试剂盒,天根生化科技(北京)有限公司;Multiplex PCR Assay Kit Ver.2、TB Green Premix DimerEraser,大连宝生物科技有限公司。
C1000梯度PCR仪,德国Eppendorf公司;X3FR高速冷冻离心机,赛默飞世尔科技有限公司;Stepone Plus荧光定量PCR系统,美国应用生物系统有限公司;LS-400无菌均质器,上海比隆仪器有限公司。
1.2 实验方法
1.2.1 细菌的培养及模板DNA提取
弧菌菌株在含3%NaCl的TSA平板上活化,梭菌、厌氧菌株在TSC平板上活化,其他的好氧菌株在NA平板上活化,所有菌株活化后,挑单菌落接种于液体培养基,厌氧菌放置于厌氧盒中37 ℃过夜培养,好氧菌于37 ℃,120 r/min恒温振荡培养箱中过夜培养。9种目标菌株过夜培养后分别取1 mL菌液,采用细菌基因组DNA提取试剂盒提取DNA,按照说明书进行,将DNA模板冻存于-20 ℃备用。
1.2.2 内外引物的设计与合成
使用primer primmer V5.0,分别针对大肠埃希氏菌的uidA基因、金黄色葡萄球菌的Nuc基因、沙门氏菌的STM4497基因、溶藻弧菌的gyrB基因[12]、副溶血弧菌的collagenase基因[12]、铜绿假单胞菌的ecfX基因[13]、单增李斯特菌的ORF2819基因,创伤弧菌的vvhA基因[12]和产气荚膜梭菌的plc基因[14]进行内外引物筛选,部分序列参考已有文献,所有引物序列经NCBI-BLAST比对分析,以评估产生交叉反应的可能性。验证后的所有引物(表1)由擎科生物科技有限公司合成。
表1 MT-PCR的内引物和外引物
Table 1 Outer and inner primer of MT-PCR

目标菌株外引物序列产物大小/bp内引物序列产物大小/bpE.coliF:CGAAAACGGCAAGAAAAAGCR:GAACGCGTGGTTACAGTCTTG146F:GTCCTGTAGAAACCCCAACCR:ATCCAGACTGAATGCCCAC65S.aureusF:ATTGATGGTGATACGGTTAAR:ACTTGCTTCAGGACCATAT130F:CGATTGATGGTGATACGGTTAR:TAGGATGCTTTGTTTCAGGTG93SalmonellaF:CGACTTTCACTTCCTGCTGAR:TCGCGCACCTCAACATCTTT90F:GCGGTCAAATAACCCACGR:CGCACCTCAACATCTTTCAG62L.monocytogenesF:ACTAAAGCCTCCCATTGAGCTCR:ACCGGAAGATTTCCACGCAATA135F:TCCAGGACTTCACTTGTCATTCR:GCAATACTCCAAAAACTCCCTC72
续表1
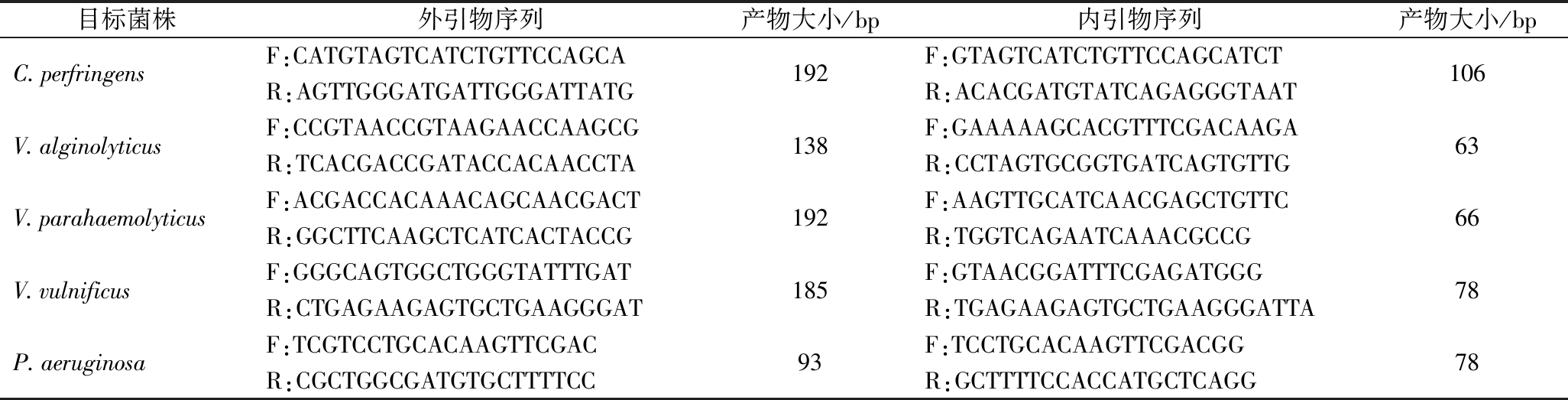
目标菌株外引物序列产物大小/bp内引物序列产物大小/bpC.perfringensF:CATGTAGTCATCTGTTCCAGCAR:AGTTGGGATGATTGGGATTATG192F:GTAGTCATCTGTTCCAGCATCTR:ACACGATGTATCAGAGGGTAAT106V.alginolyticusF:CCGTAACCGTAAGAACCAAGCGR:TCACGACCGATACCACAACCTA138F:GAAAAAGCACGTTTCGACAAGAR:CCTAGTGCGGTGATCAGTGTTG63V.parahaemolyticusF:ACGACCACAAACAGCAACGACTR:GGCTTCAAGCTCATCACTACCG192F:AAGTTGCATCAACGAGCTGTTCR:TGGTCAGAATCAAACGCCG66V.vulnificusF:GGGCAGTGGCTGGGTATTTGATR:CTGAGAAGAGTGCTGAAGGGAT185F:GTAACGGATTTCGAGATGGGR:TGAGAAGAGTGCTGAAGGGATTA78P.aeruginosaF:TCGTCCTGCACAAGTTCGACR:CGCTGGCGATGTGCTTTTCC93F:TCCTGCACAAGTTCGACGGR:GCTTTTCCACCATGCTCAGG78
1.2.3 MT-PCR反应体系的建立与优化
第一轮多重富集PCR反应体系:25 μL mix 2,1 μL mix 1,4.8 μL各基因外引物混合液和2 μL DNA模板混合液,用ddH2O调整体积至50 μL。反应条件为:95 ℃ 15 min;94 ℃ 30 s,58 ℃ 90 s,72 ℃ 90 s,10~20个循环;72 ℃ 10 min。
将第一轮多重富集PCR产物用ddH2O稀释(10~100倍)作为第二轮实时荧光定量PCR的模板,每孔体系为:10 μL TB mix,2 μL第一轮PCR产物稀释液,0.4 μL各基因的内引物,0.4 μL ROX,用ddH2O调整体积至20 μL。反应条件为:95 ℃ 30 s;95 ℃ 5 s,58 ℃ 30 s,72 ℃ 30 s,35个循环,每个循环在72 ℃结束后检测荧光信号;反应结束后进行熔解曲线分析:65~95 ℃,每0.5 ℃扫描荧光一次。
此外,分别对9种目标菌种的DNA进行单独的内引物实时荧光定量PCR(qPCR)扩增,作为对照组。qPCR的反应程序与MT-PCR第二轮程序及体系一致。
1.2.4 MT-PCR特异性与灵敏度评价
利用9对特异性引物与9种目标菌株和19种非目标菌株的基因组DNA分别进行正交测试,以验证MT-PCR体系的特异性。
分别培养9种目标菌株至同一数量级,等比例混匀后按照10倍梯度稀释,经试剂盒提取DNA后再分别采用MT-PCR和qPCR进行检测并建立标准曲线以对比2种方法的灵敏度。
1.2.5 MT-PCR检测人工污染贝类产品
将生蚝组织剪碎、均质,称取均质样品25 mg,紫外照射30 min,再将样品分别浸泡于10倍梯度稀释的混合菌液中,染菌后样品经海洋动物基因组DNA提取试剂盒提取模板DNA,按照1.2.3步骤进行检测,评价MT-PCR在检测水产样品中的定量能力和灵敏度。
2 结果与分析
2.1 多重MT-PCR的优化结果
第一轮循环数优化结果如图1-a所示,当第一轮循环数为10~15个循环,其阈值循环数(cycle threshold,Ct)值大于或接近qPCR所得Ct值,说明其灵敏度低于qPCR;当第一轮循环数为20个循环,MT-PCR针对9个目标菌的Ct值小于qPCR对应所得Ct值,且9个目标菌株所对应的Ct值为15左右,MT-PCR方法优于qPCR方法。

a-第一轮反应循环数的优化;b-第一轮产物稀释倍数的优化
图1 MT-PCR的优化
Fig.1 The optimization of MT-PCR
对第一轮反应的产物分别稀释10、50、100倍,用作于第二轮实时荧光定量PCR的模板。结果如图1-b所示,稀释倍数为50、100倍时,MT-PCR扩增所得Ct值高于或接近qPCR,熔解曲线为单峰,稀释倍数为10倍时,其Ct值低于qPCR,且9个目标菌株所对应的Ct值为15左右,溶解曲线为单峰,无非特异性扩增。
因此,在对MT-PCR的优化过程中,为保证MT-PCR的扩增优势,选择第一轮的循环数为20个循环,第一轮扩增产物的稀释倍数为10倍,作为MT-PCR反应的条件,并用于后续实验。
2.2 多重MT-PCR的特异性评价
28株菌株的DNA分别与9对引物的MT-PCR正交反应,特异性结果如表2所示,MT-PCR检测9种目标菌株时,只有其对应的目标基因经MT-PCR扩增,结果显示阳性,非目标基因检测结果显示阴性。
表2 MT-PCR 特异性分析
Table 2 The specificity of MT-PCR

实验菌株菌株来源MT-PCR特异性结果uidANucSTM4497gyrBcollagenaseplcecfXORF2819vvhAE.coliATCC 25922+--------S.aureusATCC 6538-+-------SalmonellaATCC 14028--+------V.alginolyticusCMCC 33787---+-----V.parahaemolyticusATCC 17802----+----C.perfringensATCC 13124-----+---P.aeruginosaATCC 27853------+--L.monocytogenesCMCC 54002-------+-V.vulnificusATCC 27562--------+S.epidermidisATCC 12228---------S.pyogenesCMCC 26103---------S.saprophyticusATCC 49907---------S.warneriATCC 27836---------S.enteritidisATCC 13076---------V.hollisaeATCC 33564---------V.mimicusATCC 33653---------B.longumATCC 15707---------P.melaninogenicaATCC 25845---------A.MuciniphilaATCC BAA-835---------L.innocuaATCC 33090---------L.welshimeriCICC 21672---------K.pneumoniaeATCC 4352---------P.fluorescensATCC 13525---------S.marcescensATCC 14040---------S.haemolyticusATCC 19194---------S.thermophilusCICC 6063---------C.freundiiATCC 10787---------P.mirabilisATCC 29906---------
注:+表示阳性,-表示阴性结果
其中19种非目标菌株的DNA分别与9对引物的MT-PCR反应,结果均显示阴性,未检测到扩增,表明MT-PCR反应体系中的引物序列针对非目标菌株时无非特异性扩增及自身无交叉反应,即本研究建立MT-PCR方法特异性强。
2.3 多重MT-PCR体系灵敏度评价
10倍梯度稀释9种食源性致病菌的混合液,提取相应的基因组DNA,同时进行MT-PCR和qPCR检测以对比2种方法的定量能力和灵敏度。结果如图2所示,2种方法的标准曲线线性关系良好,R2均>0.98,扩增效率在90%~110%。MT-PCR针对大肠埃希氏菌、金黄色葡萄球菌、产气荚膜梭菌、铜绿假单胞菌、单增李斯特菌和创伤弧菌的检测限为102~104 CFU/mL,相比qPCR的检测定量限均低一个数量级。而针对沙门氏菌、溶藻弧菌和副溶血弧菌,MT-PCR和qPCR的检测定量限一致,为102~103 CFU/mL,说明MT-PCR对DNA模板的预扩增具有一定优势。
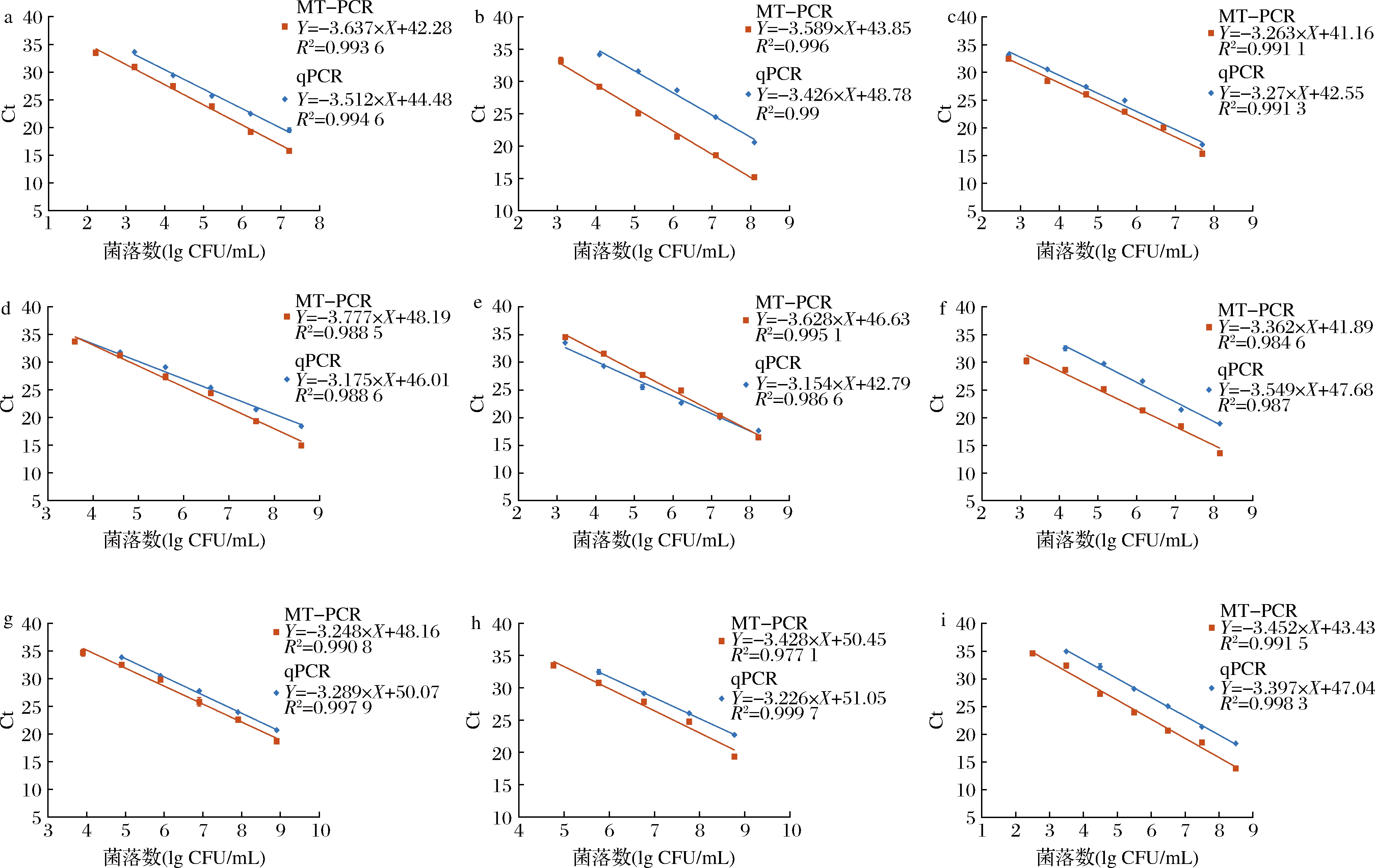
a-E.coli;b-S.aureus;c-Salmonella;d-V.alginolyticus;e-V.parahaemolyticus;f-C.perfringens;g-P.aeruginosa;h-L.monocytogenes;i-V.vulnificus
图2 MT-PCR和qPCR检测9种食源性致病菌在培养菌液中的灵敏度
Fig.2 The sensitivity of MT-PCR and qPCR for detecting 9 foodborne pathogens in pure culture
2.4 多重MT-PCR在人工染菌生蚝样品中的应用
10倍梯度稀释9种食源性致病菌的混合液,人工染菌至生蚝组织上,提取基因组DNA进行MT-PCR扩增。如图3所示,MT-PCR至少可检测该样品中1.65×102 CFU/g的大肠埃希氏菌、3.95×103 CFU/g的金黄色葡萄球菌、6.58×102 CFU/g的沙门氏菌、3.70×103 CFU/g的溶藻弧菌、1.62×103 CFU/g的副溶血弧菌、1.36×102 CFU/g的产气荚膜梭菌、8.24×102 CFU/g的铜绿假单胞菌、1.83×103 CFU/g的单增李斯特菌以及1.09×102 CFU/g的创伤弧菌。因此,MT-PCR在生蚝样品中应用的检测限范围为102~103CFU/g。
3 结论与讨论
MT-PCR方法作为一个开放的检测系统,可通过增加靶基因的数量来实现高通量检测,但这同时也会导致引物的非特异性结合,因此设计并筛选出合适的引物是保证方法特异性和灵敏度的关键。研究表明,实时荧光定量PCR产物长度小于200 bp有利于MT-PCR检测[15],因此本研究在设计内外引物时控制外引物长度在90~200 bp,内引物长度在60~110 bp。此外,所有的内外引物均针对本研究中9个目标菌株的特异性序列,MT-PCR针对目标菌株和非目标菌株的检测结果显示出了100%的选择性和特异性。
第一轮较少的循环数可以确保目标基因得到预扩增的同时有效避免引物的竞争和底物的消耗。在本研究中,第一轮反应循环数为20,稀释倍数为10倍时,MT-PCR针对9种目标菌株所得的Ct值最小,比qPCR有更强的检测信号。MT-PCR较高的灵敏度与其预扩增步骤相关性较大,而本研究建立的MT-PCR方法对6种菌株的检测定量限(102~104 CFU/mL)均低于qPCR(103~105 CFU/mL)也说明了这一点。COSTA等[16]的研究也出现了类似结果,其使用单管巢式实时荧光定量PCR检测榛子中的过敏原,结果显示该方法的灵敏度比普通qPCR高10倍。此外,本研究建立的MT-PCR技术的灵敏度与其他研究利用多重PCR检测致病菌所得的检测灵敏度具有一定的可比性。TAO等[17]基于通用引物建立的多重PCR检测单增李斯特菌、大肠杆菌O157∶H7、副溶血性弧菌、金黄色葡萄球菌和产气荚膜梭菌,灵敏度在103~104 CFU/mL。

a-E.coli;b-S. aureus;c-Salmonella;d-V.alginolyticus;e-V.parahaemolyticus;f-C.perfringens;g-P.aeruginosa;h-L.monocytogenes;i-V.vulnificus
图3 MT-PCR检测人工染菌生蚝样品中的9种食源性致病菌的灵敏度
Fig.3 The sensitivity of MT-PCR for simultaneously detecting 9 foodborne pathogens in oyster
D’SOUZA等[18]使用qPCR可检测含有102 CFU/g创伤弧菌的蛤蜊。ZHANG等[19]使用多重实时荧光定量PCR可检测含102 CFU/g副溶血弧菌、单增李斯特菌和沙门氏菌的对虾样品。本研究建立的MT-PCR方法可同时检测人工染菌生蚝中的9种致病菌,灵敏度为102~103 CFU/g。说明本研究建立的MT-PCR方法在实现高通量检测的同时具有较高灵敏度。
因此,本研究建立的MT-PCR方法可用于同时检测9种病原菌,具体包括大肠埃希氏菌、金黄色葡萄球菌、单增李斯特菌、沙门氏菌、溶藻弧菌、副溶血弧菌、产气荚膜梭菌、铜绿假单胞菌和创伤弧菌,该方法灵敏度高、特异性强,且拥有比qPCR更优或相当的定量能力。此外,MT-PCR还适用于同时检测生蚝样品中多种食源性致病菌,在高通量快速检测领域具有广阔的应用前景。
[1] MA B, LI J L, CHEN K, et al.Multiplex recombinase polymerase amplification assay for the simultaneous detection of three foodborne pathogens in seafood[J].Foods (Basel, Switzerland), 2020, 9(3):278.
[2] DUMEN E, EKICI G, ERGIN S, et al.Presence of foodborne pathogens in seafood and risk ranking for pathogens[J].Foodborne Pathogens and Disease, 2020, 17(9):541-546.
[3] LEHEL J, YAUCAT-GUENDI R, DARNAY L, et al.Possible food safety hazards of ready-to-eat raw fish containing product (sushi, sashimi)[J].Critical Reviews in Food Science and Nutrition, 2021, 61(5):867-888.
[4] 陈嘉茵, 方苓, 吴诗微, 等.一步法微滴数字PCR检测生菜中GII型诺如病毒[J].食品科学, 2019, 40(4):332-337.
CHEN J Y, FANG L, WU S W, et al.Detection of Norovirus genogroup II in lettuce by droplet digital PCR[J].Food Science, 2019, 40(4):332-337.
[5] 赵峰, 佟利惠, 杨敏, 等.牡蛎中诺如病毒的感染及其防控研究进展[J].南方水产科学, 2021, 17(4):133-140.
ZHAO F, TONG L H, YANG M, et al.Progress and prospects of infection, prevention and control of Norovirus in oyster[J].South China Fisheries Science, 2021, 17(4):133-140.
[6] FODDAI A C G, GRANT I R.Methods for detection of viable foodborne pathogens:Current state-of-art and future prospects[J].Applied Microbiology and Biotechnology, 2020, 104(10):4 281-4 288.
[7] MAILLET A, BOUJU-ALBERT A, ROBLIN S, et al.Impact of DNA extraction and sampling methods on bacterial communities monitored by 16S rDNA metabarcoding in cold-smoked salmon and processing plant surfaces[J].Food Microbiology, 2021, 95:103705.
[8] WEI C J, ZHONG J L, HU T, et al.Simultaneous detection of Escherichia coli O157:H7, Staphylococcus aureus and Salmonella by multiplex PCR in milk[J].3 Biotech, 2018, 8(1):1-7.
[9] ATTWOOD L O, FRANCIS M J, HAMBLIN J, et al.Clinical evaluation of AusDiagnostics SARS-CoV-2 multiplex tandem PCR assay[J].Journal of Clinical Virology, 2020, 128:104448.
[10] LAU A, STANLEY K, SORRELL T.Multiplex-tandem PCR for fungal diagnostics[J].Methods in Molecular Biology (Clifton, N.J.), 2013, 968:195-201.
[11] WEI S, WANG C G, ZHU P Y, et al.A high-throughput multiplex tandem PCR assay for the screening of genetically modified maize[J].LWT, 2018, 87:169-176.
[12] 魏霜, 汪天杰, 龙阳, 等.多重富集定量PCR(ME-qPCR)同时检测4种食源性病原弧菌[J].中国农业科学, 2016, 49(23):4 619-4 627.
WEI S, WANG T J, LONG Y, et al.Multiplex enrichment quantitative PCR assays for the detection of Vibrio alginolyticus, Vibrio parahaemolyticus, Vibrio vulnificus and Vibrio cholerae[J].Scientia Agricultura Sinica, 2016, 49(23):4 619-4 627.
[13] XIE G Y, YU S, LI W, et al.Simultaneous detection of Salmonella spp., Pseudomonas aeruginosa, Bacillus cereus, and Escherichia coli O157:H7 in environmental water using PMA combined with mPCR[J].Journal of Microbiology (Seoul, Korea), 2020, 58(8):668-674.
[14] NAGPAL R, OGATA K, TSUJI H, et al.Sensitive quantification of Clostridium perfringens in human feces by quantitative real-time PCR targeting alpha-toxin and enterotoxin genes[J].BMC Microbiology, 2015, 15:219.
[15] GRACIAS K S, MCKILLIP J L.A review of conventional detection and enumeration methods for pathogenic bacteria in food[J].Canadian Journal of Microbiology, 2004, 50(11):883-890.
[16] COSTA J, MAFRA I, KUCHTA T, et al.Single-tube nested real-time PCR as a new highly sensitive approach to trace hazelnut[J].Journal of Agricultural and Food Chemistry, 2012, 60(33):8 103-8 110.
[17] TAO J, LIU W W, DING W, et al.A multiplex PCR assay with a common primer for the detection of eleven foodborne pathogens[J].Journal of Food Science, 2020, 85(3):744-754.
[18] D’SOUZA C, KUMAR B K, RAI P, et al.Application of gyrB targeted SYBR green based qPCR assay for the specific and rapid detection of Vibrio vulnificus in seafood[J].Journal of Microbiological Methods, 2019, 166:105747.
[19] ZHANG Z H, XIAO L L, LOU Y, et al.Development of a multiplex real-time PCR method for simultaneous detection of Vibrio parahaemolyticus, Listeria monocytogenes and Salmonella spp.in raw shrimp[J].Food Control, 2015, 51:31-36.