豆瓣酱是一种以蚕豆和面粉为主要原料,在霉菌、酵母菌和乳酸菌等微生物的协同作用下,自然发酵而成的半固体粘稠状食品。其色泽红润、风味独特、营养丰富,已成为日常生活及餐饮行业不可缺少的调味品[1]。豆瓣酱发酵主要分为制曲和酱醅发酵两步,其中制曲主要依赖微生物分泌的丰富蛋白酶资源实现对蚕豆瓣中大分子物质的降解,为后期豆瓣酱发酵过程中微生物生长代谢与风味化合物形成提供了关键前体物质。因此,在制曲过程中提高成曲的蛋白酶活力是加快豆瓣酱发酵周期且改善豆瓣酱品质的关键。
米曲霉是一种丝状真菌,其通常作为常用发酵剂应用于亚洲传统豆类发酵食品的生产,被美国食品药品监督管理局和世界卫生组织认定为公认安全(Generally Recognized as Safe,GRAS)的微生物[2]。在制曲过程中,米曲霉会分泌大量蛋白酶,将蚕豆中的蛋白质降解成寡肽和氨基酸。这些酶解产物不仅可以为发酵过程中微生物的生长代谢提供必须的营养源,也作为豆瓣酱中的增味剂,促进风味化合物的积累[3]。因此,具有高蛋白酶活力生产能力的米曲霉被认为是豆瓣酱生产的理想菌种。米曲霉3.042是国内豆瓣酱发酵行业中常用的菌株,在制曲过程中具有较强的蛋白酶生产能力[4]。近年来,研究人员通过菌株诱变筛选及菌种改造策略获得了高产蛋白酶的米曲霉菌株,如紫外线诱变[5]、原生质体融合育种[6]和常温等离子体(atmospheric room temperature plasma,ARTP)诱变。GAO等[7]通过ARTP获得了米曲霉3.042突变体H8,其酸性、中性和碱性蛋白酶活力较出发菌提高7.56%、28.92%和26.23%。在前期研究过程中,本研究室从豆瓣酱酱醅中筛选得到1株具有较高蛋白酶产生能力的米曲霉BL18[8]。在此基础上,以目前行业常用酿造菌株米曲霉3.042为对照,分析了制曲过程米曲霉孢子数、蛋白酶活力以及成曲游离氨基酸含量,测定了后续发酵过程蛋白酶活力、理化参数,从制曲、发酵2个阶段探究了米曲霉BL18在实际生产中应用的可能,同时从基因组水平深入挖掘了米曲霉BL18表型产生差异的原因。
1 材料和试剂
1.1 材料
1.1.1 菌株及主要试剂
菌株:米曲霉(Aspergillus oryzae) BL18(保藏号CGMCC No.19628)筛选自豆瓣酱酱醅、米曲霉3.042为目前常用的豆瓣酱制曲微生物,保藏于本实验室。
主要试剂:H2SO4、NaCl、36%(体积分数,下同)甲醛水溶液、Na2CO3、三氯乙酸、95%乙醇、干酪素等。
1.1.2 培养基
马铃薯葡萄糖琼脂(potato dextrose agar, PDA)培养基:称取土豆200 g,煮沸30 min,过滤后定容至1 L,加入葡萄糖20 g,琼脂20 g,121 ℃,高压蒸汽灭菌20 min。
1.1.3 仪器和设备
超净工作台,苏州净化设备有限公司;HHS型数显式电热恒温水浴锅,上海博迅实业有限公司;全自动高压蒸汽灭菌锅,日本HIRAYAMA株式会社;恒温恒湿培养箱,上海博迅实业有限公司;紫外可见分光光度计,UNICO(上海)仪器有限公司;1100液相色谱仪,美国安捷伦科技有限公司;电子天平、pH计,METTLER TOLEDO公司。
1.2 实验方法
1.2.1 菌株活化及孢子液制备
米曲霉BL18与米曲霉3.042接种到PDA培养基活化后,接种至PDA固体平板,30 ℃培养3 d,用无菌生理盐水将孢子冲下经纱布过滤后完成孢子悬液制备。
1.2.2 制曲
干蚕豆泡发,经高温蒸煮并冷却到40 ℃左右拌入面粉(15%,质量分数),分别接种米曲霉BL18和3.042孢子液,接种量为1×106个/g。放置在30 ℃、相对湿度90%恒温恒湿培养箱,每隔8 h通过肉眼观察并通过照相机拍照,取样测定蛋白酶活力及孢子数,48 h后每隔16 h观察拍照及取样测定。
1.2.3 发酵
将成曲与170 g/L的盐水按质量比1∶1.1投入透明塑料小桶,搅拌均匀。置于30 ℃培养箱中静置发酵。发酵第一周,每天进行翻酱。发酵一周后每隔7 d进行翻酱取样。
1.2.4 理化指标测定
孢子数测定参照SB/T 10315—1999《孢子数测定法》;蛋白酶活力测定参照SB/T 10317—1999《蛋白酶活力测定法 福林法》,条件为酸性(pH 3.0)、中性(pH 7.2)和碱性(pH 10.0);氨基酸态氮测定参照GB 5009.235—2016《食品安全国家标准 食品中氨基酸态氮的测定 酸度计法》;可滴定酸测定参照GB 12456—2021《食品安全国家标准 食品中总酸的测定 pH计电位滴定法》。水分及pH测定参照文献[9]的方法。
1.2.5 游离氨基酸含量测定
称取样品1.0 g研磨均匀,用50 g/L三氯乙酸溶解并转移至25 mL容量瓶定容。常温超声20 min,静置2 h。经双层滤纸过滤后,取1 mL滤液于1.5 mL离心管中,12 000 r/min离心10 min,取上清液500 μL经0.22 μm水膜过滤于液相瓶,使用Agillent 1100高效液相色谱仪进行测定分析,条件如下:色谱柱:ODS柱4.6 mm×250 mm×5 μm;流动相A:醋酸钠溶液(20 mmol/L);B:V(醋酸钠溶液)∶V(甲醇)∶V(乙腈)=1∶2∶2;流速1.0 mL/min;检测波长338 nm(脯氨酸262 nm);洗脱方式等度洗脱;柱温40 ℃;进样量10 μL。
1.2.6 米曲霉BL18基因组测序分析
使用BGI定制的磁性植物基因组DNA试剂盒提取米曲霉BL18的基因组DNA。确认提取DNA浓度、纯度以及完整性后,在华大基因(中国深圳)的PacBio Sequel系统上进行全基因组测序。首先,使用g-TUBE装置将1 μg基因组DNA剪切成10~15 kb的片段。然后通过SMRTbell® Express Template Preparation Kit 2.0构建文库,最终使用Sequel (PacBio) Sequencing Kit 2.0对SMRTbell文库进行测序。
原始测序数据首先过滤包含测序接头、低质量碱基比率和高未知碱基比率的读数。对这些过滤后的高质量数据进行数据统计分析和下游生物信息学分析。结合多种软件对测序数据进行组装,经过Subreads纠正、Corrected Reads组装、组装结果纠正、构建Scaffold及补洞四步对基因组进行组装。从头注释所采用的软件包括Genewise[10]、SNAP[11]、Augustus[12]、Genemarkes[13];同时利用多种数据库进行功能注释,包括分析NR、KEGG、COG、GO数据库、EggNOG以及Swiss-Prot等数据库(无特殊说明的软件采用默认参数)。原始数据和基因组组装结果已上传至NCBI,注册号JAJGRD000000000 (BioProject PRJNA776566)。
对米曲霉BL18和米曲霉3.042的共有及特有基因进行分析,首先对菌株的蛋白质基因集进行CD-HIT(v4.6.6)[14]聚类分析,将聚类的最终基因集作为Pan基因集。提取聚类结果中每个样本共有的序列作为共有基因集。每个样品中特有的基因集为特有基因集,Pan基因集中去除共有基因集和特有基因集为非必要基因集。
1.3 数据分析
采用Excel进行数据统计,实验结果中的数据均为3个平行样品均值,相对标准偏差在表和图中分别以数值和误差棒表示,采用Origin 2022作图分析,显著性分析由SPSS 19.0软件完成。
2 结果与分析
2.1 制曲过程中的形态变化
将米曲霉BL18和米曲霉3.042在制曲过程中进行应用,并对制曲过程中曲块表面米曲霉菌丝生长和产孢现象进行跟踪观察。如图1所示,米曲霉BL18产孢提前8 h,在培养32 h时就能明显观察到菌丝与孢子产生,在培养40 h和48 h时曲块产孢现象已经比较明显。而接种米曲霉3.042的曲块在制曲40 h时才开始出现明显产孢,且在48 h和64 h成曲产孢程度明显不如米曲霉BL18制备成曲。

图1 制曲过程形态变化
Fig.1 Morphological changes during koji making
2.2 制曲过程中孢子数的变化
进一步对米曲霉BL18和米曲霉3.042在制曲过程中的孢子数进行了测定。如图2所示,在发酵前32 h米曲霉BL18和3.042的孢子数几乎相同(P>0.05)。
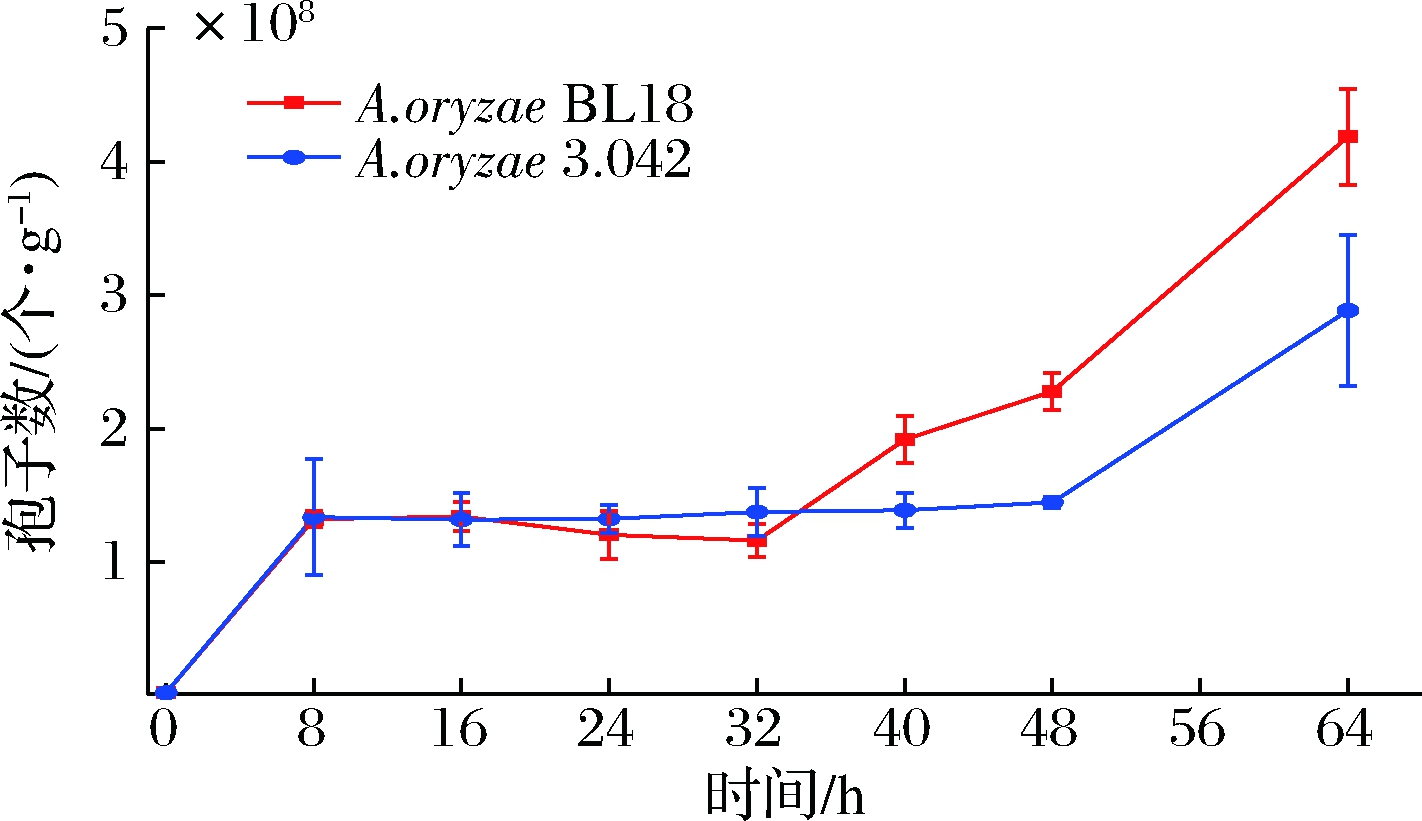
图2 制曲过程中的孢子数变化
Fig.2 Changes in the number of spores during koji making
在制曲40~48 h时,米曲霉BL18制曲的孢子数开始上升,而米曲霉3.042的孢子数则几乎不变。米曲霉BL18制备的曲样品在48 h和64 h的孢子量分别为2.28×108个/g干曲和4.19×108个/g干曲,显著高于(P<0.05)米曲霉3.042样品孢子数(1.25×108个孢子/g干曲和2.89×108个孢子/g干曲)。上述结果说明米曲霉制曲过程的孢子数测定结果与制曲状态观察结果一致。豆瓣曲表面绿色孢子的形成是曲成熟的指标之一[15],因此孢子形成速度越快,制曲时间越短。先前的研究还报道,米曲霉绿色孢子的形成伴随着蛋白酶的产生和分泌[16]。具有更多孢子的曲样品可能具有更高的蛋白酶活性。
2.3 制曲过程蛋白酶活力的变化
如图3所示,米曲霉BL18和米曲霉3.042产生酸性蛋白酶的速率相似,而米曲霉BL18中性和碱性蛋白酶的分泌速率明显快于米曲霉3.042。米曲霉BL18制曲样品在培养64 h后的酸性、中性和碱性蛋白酶活性分别达到542.36、1 828.41、2 283.58 U/g,和米曲霉3.042制备的曲样品(356.76、1 698.66、1 548.98 U/g)相比分别提高52%、7.6%和45.6%。米曲霉BL18制备的成曲总蛋白酶活性为4 654.35 U/g,比米曲霉3.042制备的成曲(3 604.40 U/g)提高29.1%。米曲霉在制曲过程发挥主要作用,米曲霉通过产生蛋白酶将原料中的蛋白质降解为寡肽和氨基酸,其生长和分泌蛋白酶的能力对成熟豆瓣曲的质量具有重要意义[17],在豆瓣酱制曲过程中,首选蛋白酶活性较高的米曲霉,因为通过分解蛋白质产生的氨基酸和寡肽越多,豆瓣酱产品的质量可能会更好。
2.4 制曲过程中游离氨基酸的含量
取制曲时间为64 h样品对17种游离氨基酸进行测定。如图4所示,相比于米曲霉3.042,除了甘氨酸、丙氨酸、缬氨酸、蛋氨酸外,其他13种氨基酸含量在米曲霉BL18所制曲中均有提升。其中天冬氨酸、谷氨酸、酪氨酸和苯丙氨酸分别提高了30.1%、8.9%、11.7%和1.2%;组氨酸、苏氨酸、精氨酸、半胱氨酸、赖氨酸分别提升了96.8%、83.6%、85.7%、114.1%、179.7%;游离氨基酸总量提升16.3%。游离氨基酸含量的提升依赖于蛋白质及多肽的降解,蛋白酶活力的高低将直接影响游离氨基酸产生,具有较高制曲酶活力的米曲霉BL18能有效提高多种氨基酸含量。
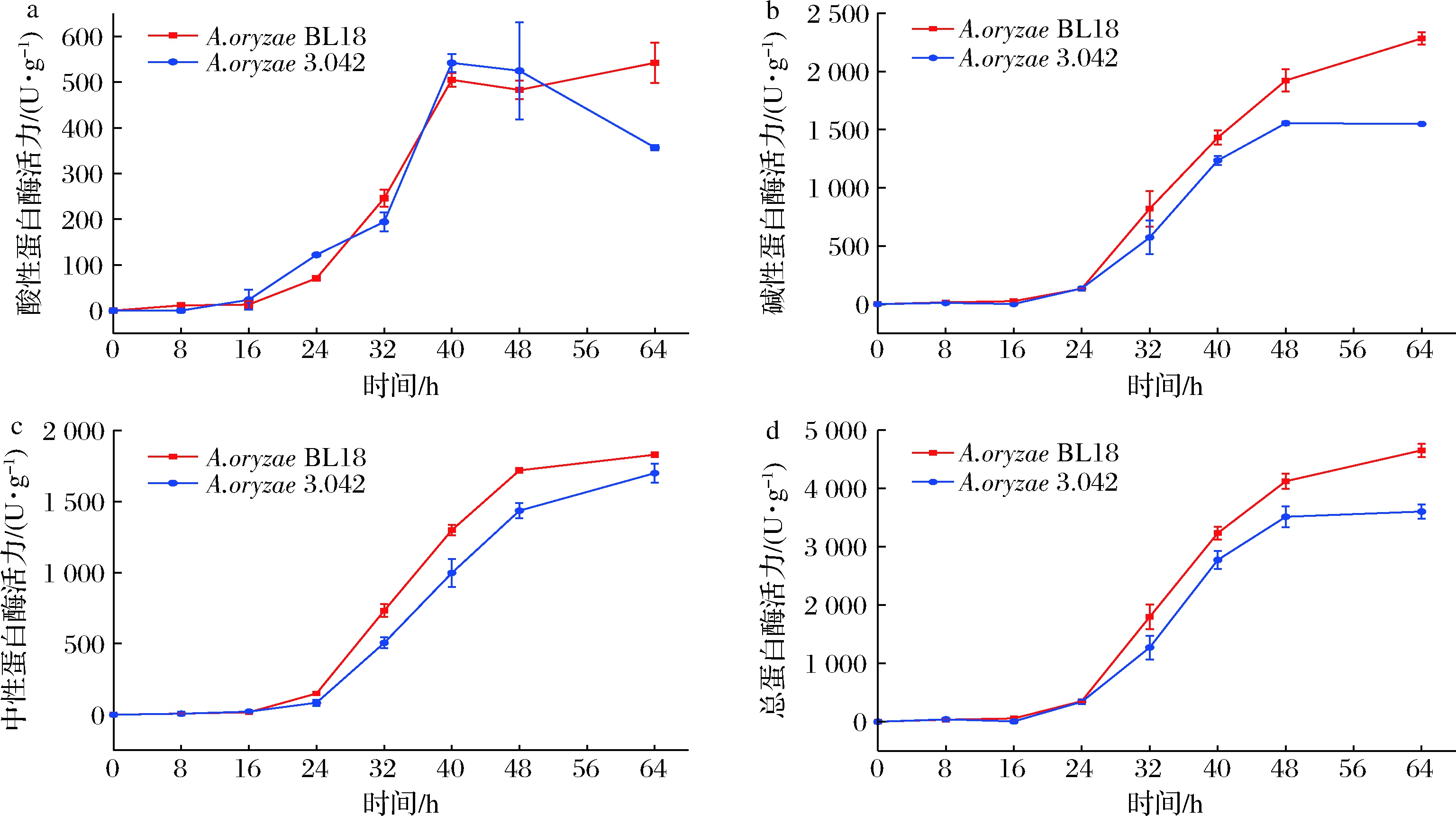
a-酸性蛋白酶;b-碱性蛋白酶;c-中性蛋白酶;d-总蛋白酶
图3 制曲过程蛋白酶活力的变化
Fig.3 Changes of protease activity during koji making
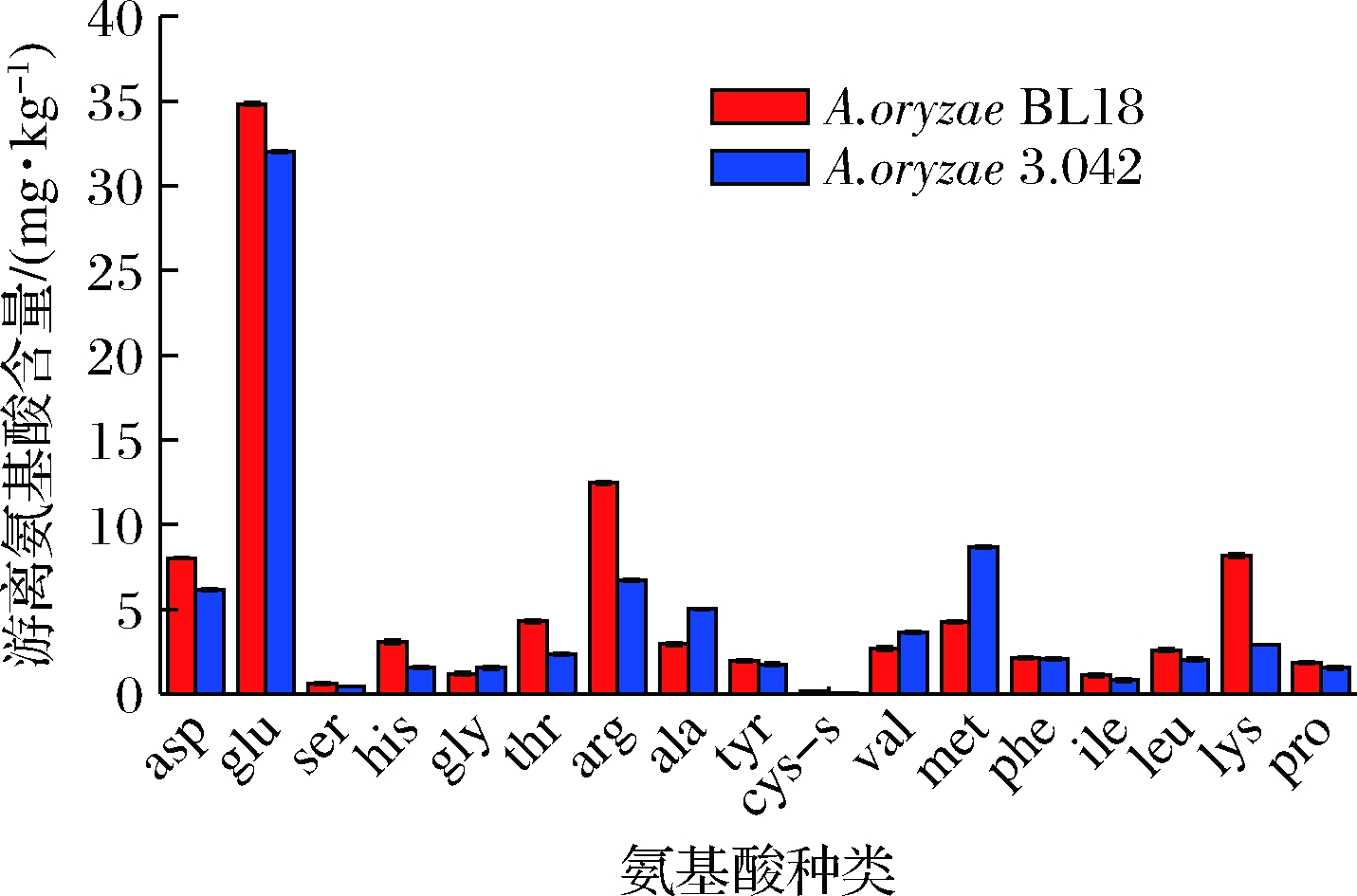
图4 制曲64 h游离氨基酸的含量
Fig.4 Free amino acid content at 64 h of koji making
2.5 发酵过程中可滴定酸和pH的变化
可滴定酸通常是检测发酵过程的一个重要参数,它的变化同微生物菌群的代谢情况有密切关系。在酱醅发酵的初始阶段,由于高盐环境的抑制作用,仅有少量的耐盐微生物可以在其中生长繁殖,其中占优势的主要是葡萄球菌属及魏斯氏菌属微生物。由于没有其他微生物的竞争抑制,它们的数量快速增加,并产生大量的乳酸等有机酸,导致了酱醅发酵初期的pH值迅速下降(图5-a)。随着发酵过程的继续,由于产酸的不断积累,pH值下降到 5.5左右,而过低的pH值又抑制了产酸微生物(如葡萄球菌属及魏斯氏菌属)的生长,此时,一些可以耐高盐和低pH值的酵母开始大量繁殖,并逐渐占优势[18]。
米曲霉BL18和3.042酱醅可滴定酸含量在发酵过程迅速积累(图5-b)。米曲霉BL18可滴定酸从0.64 g/100 g增加到了1.63 g/100 g,pH则从6.21下降到了5.81。米曲霉3.042可滴定酸从0.51 g/100 g增加到了1.63 g/100 g,pH则从6.22下降到了5.87。制曲过程米曲霉BL18具有更高的蛋白酶活力,高蛋白酶活力可使原料更充分地降解,使成曲氨基酸及寡肽等营养物质含量提高,这些营养物质为发酵初始阶段产酸微生物提供了快速增长条件,从而造成产酸以及pH等指标差异。
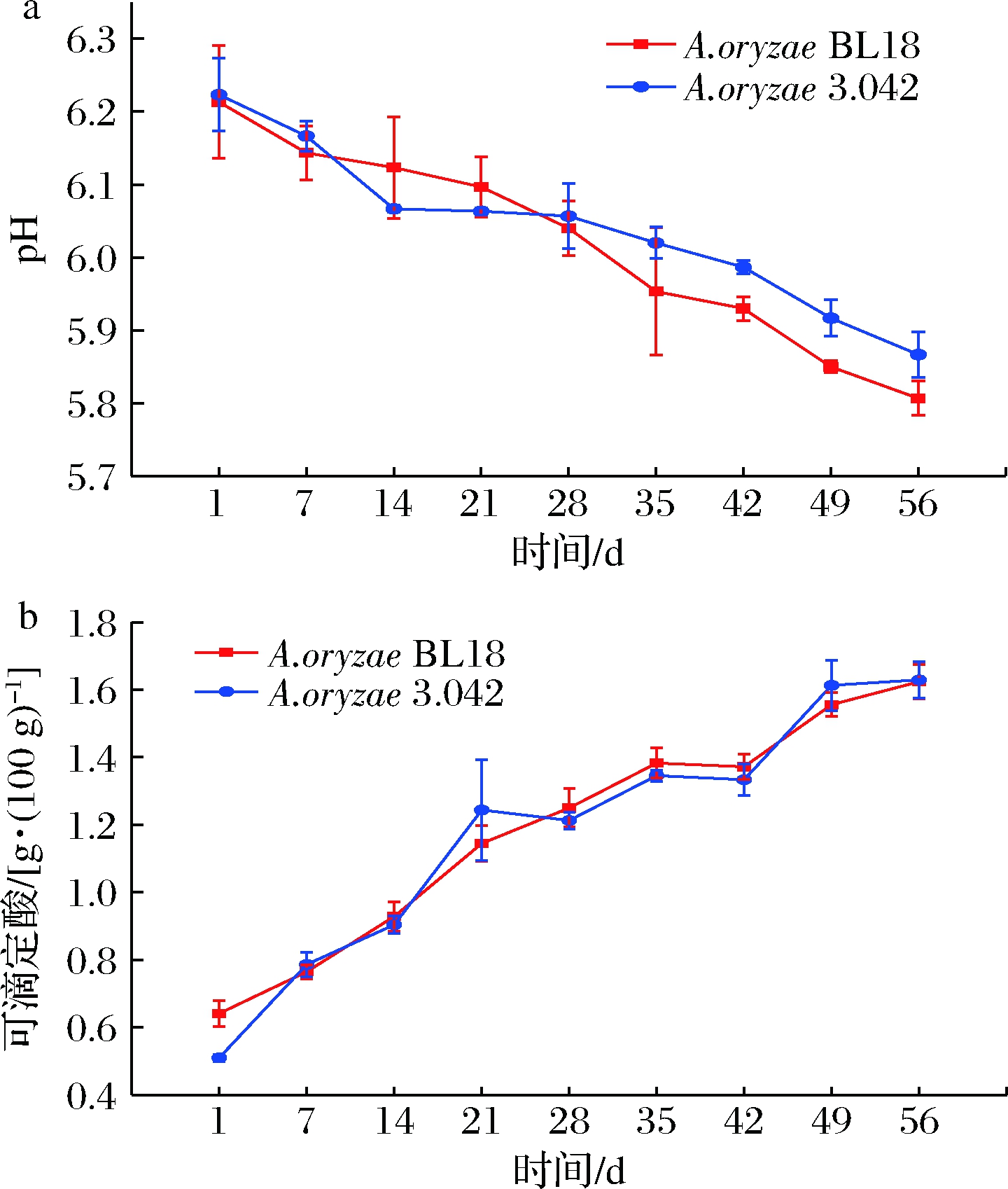
a-pH;b-可滴定酸
图5 发酵过程pH及可滴定酸的变化
Fig.5 Titratable acid and pH changes during fermentation
2.6 发酵过程中蛋白酶活力的变化
蛋白酶的活力和稳定性对豆瓣酱的原料利用率和品质有着重要的作用。豆瓣酱醅属于一种极端的生理环境,具有高盐度和低pH的特点[19]。蛋白酶的活力在其中会受到高盐度的抑制,从而导致发酵周期的延长,低 pH 值则会影响到蛋白酶的稳定性。如图6所示,酱醅蛋白酶活力在发酵过程呈现先上升后下降趋势,中性和碱性蛋白酶活力在发酵第7天达到峰值,然后呈现下降趋势;米曲霉BL18成曲所制酱醅发酵过程酸性蛋白酶活力在第28天达到最大,而米曲霉3.042则在第35天达到最大。米曲霉BL18成曲所制酱醅酸性、碱性及中性蛋白酶活力在发酵前28天均高于米曲霉3.042。以上结果说明米曲霉BL18发酵豆瓣酱在发酵前期的蛋白酶具有优势。之前的研究表明米曲霉在加入盐水发酵后生长受到抑制,在发酵前期逐渐消亡[20],但是米曲霉分泌的许多蛋白酶都具有一定的耐盐性,仍然能够继续在高盐酱醅体系里发挥作用,研究表明耐盐米曲霉蛋白酶在发酵相同时间后剩余蛋白酶活力更高,使发酵成品氨基酸及风味物质含量得到提升[21]。发酵前期,米曲霉BL18成曲所制酱醅蛋白酶活力优于米曲霉3.042,可能与它分泌的蛋白酶耐盐性具有一定关系,同时也有助于豆瓣酱品质提升。另一方面米曲霉BL18更高的蛋白酶活力能产生更丰富的寡肽氨基酸等营养物质,这些营养物质为其他产蛋白酶微生物如芽胞杆菌等的生长提供了条件[18]。
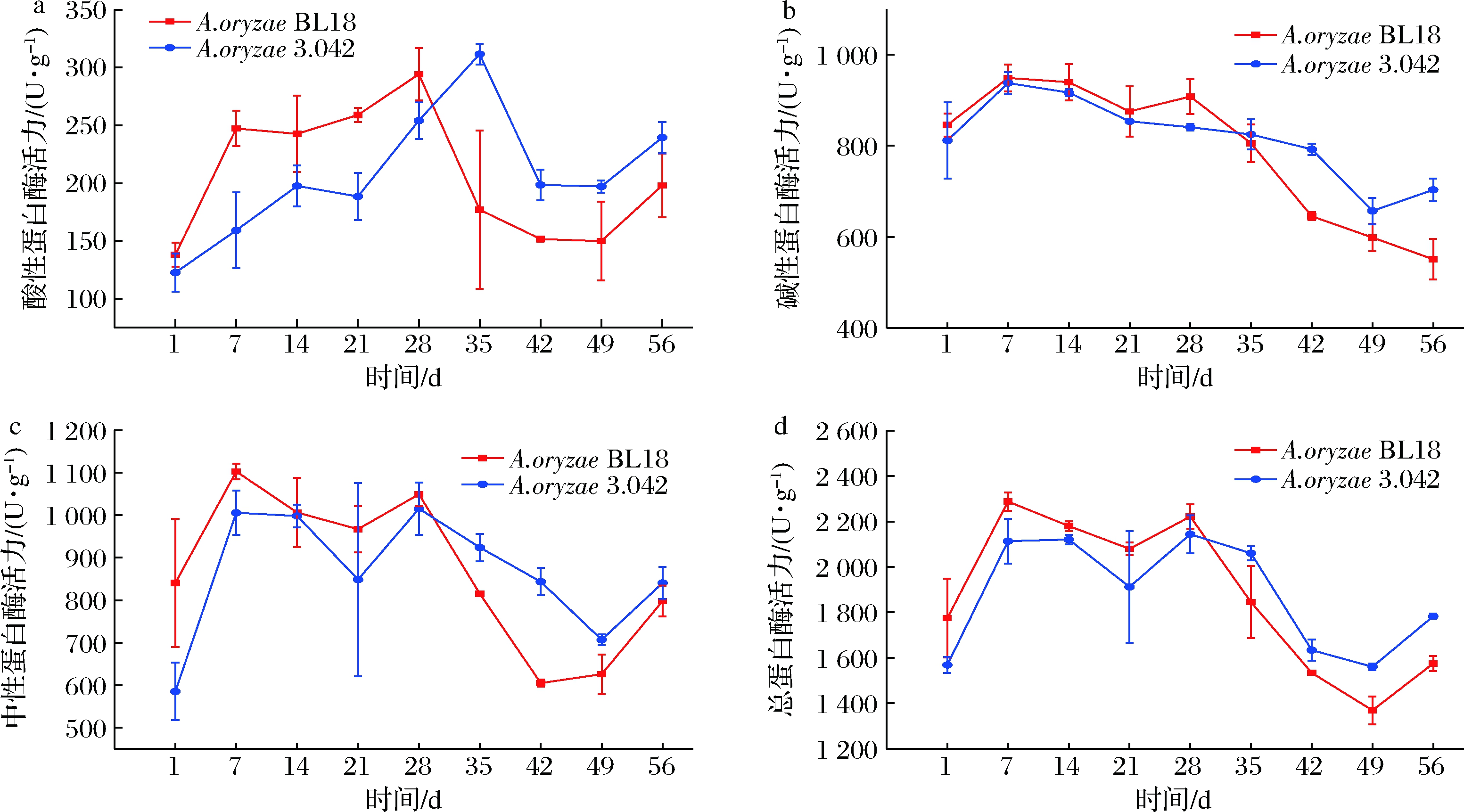
a-酸性蛋白酶;b-碱性蛋白酶;c-中性蛋白酶;d-总蛋白酶
图6 发酵过程蛋白酶活力的变化
Fig.6 Changes of protease activity during fermentation
2.7 发酵过程中氨基酸态氮的变化
氨基态氮是衡量豆瓣酱品质的一个重要指标,它的含量同蛋白酶活力有着密切关系。如图7所示,发酵前21天,米曲霉BL18酱醅的氨基酸态氮迅速积累,从开始的0.27 g/100 g增加到了0.90 g/100 g,米曲霉3.042则从0.27 g/100 g增加到了0.84 g/100 g。之后增速放缓,发酵结束时,氨基酸态氮含量分别达到1.03 g/100 g和0.99 g/100 g。氨基酸态氮含量变化与蛋白酶活力变化相反,发酵28 d后,酸性、中性和碱性蛋白酶活力都呈现下降趋势,而氨基酸态氮增速恰好放缓,这说明氨基态氮的产生主要集中在发酵前期,这与CHOU等[22]的研究一致,且和蛋白酶的逐渐失活密切相关。
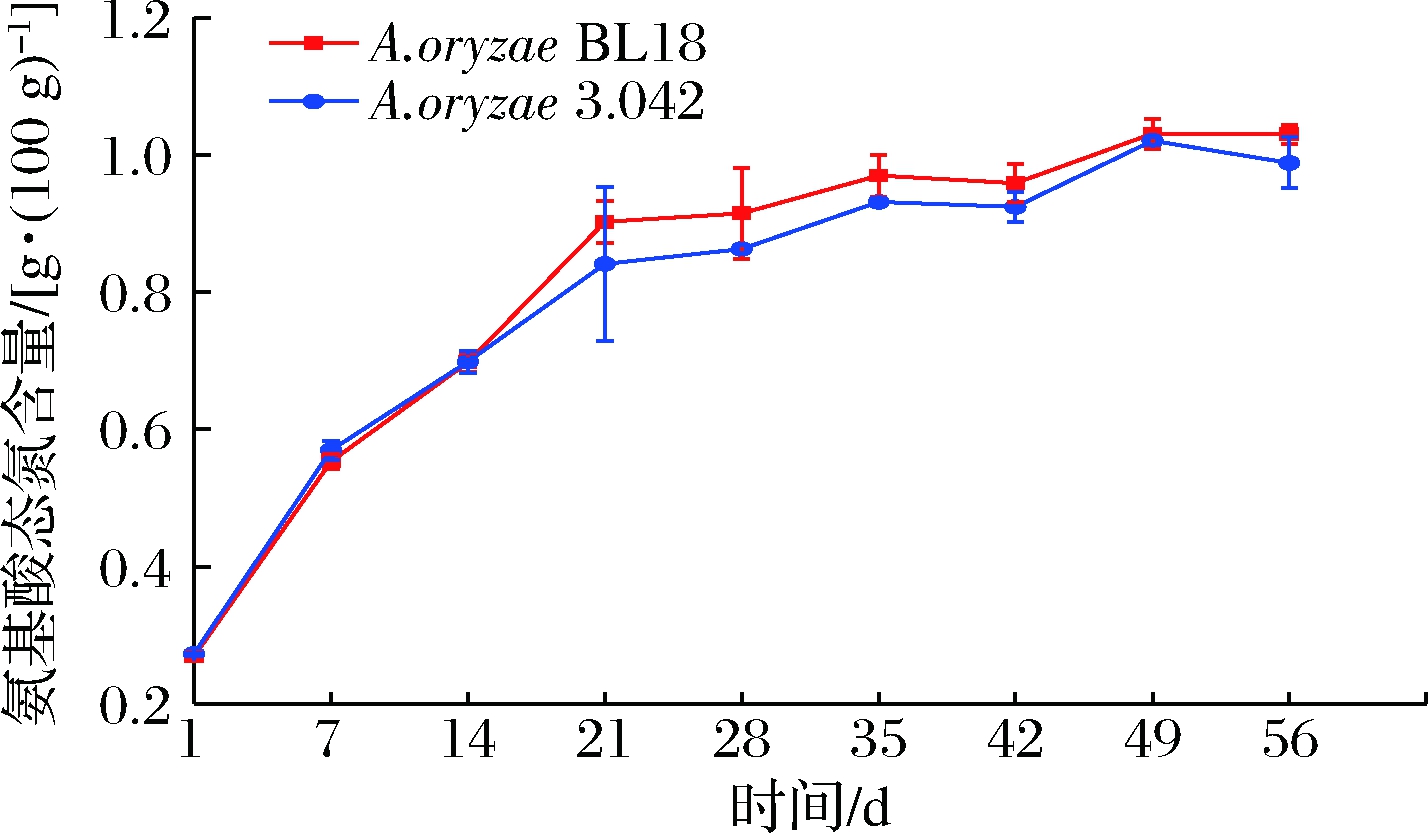
图7 发酵过程氨基酸态氮含量的变化
Fig.7 Changes of amino acid nitrogen content
during fermentation
2.8 发酵结束的游离氨基酸含量
食物的口感主要由低分子质量物质和非挥发性物质所决定。口味通常分5种,即甜味、苦味、咸味、酸味和鲜味[23]。豆瓣酱中的游离氨基酸主要是由多肽的进一步降解而产生,对豆瓣酱口感的形成具有重要的作用。不同的氨基酸可以提供不同的口味,谷氨酸具有强烈的鲜味,甘氨酸、丝氨酸、苏氨酸和丙氨酸具有甜味,而缬氨酸、苯丙氨酸和组氨酸具有苦味。如图8所示,相比于米曲霉3.042豆瓣样品,米曲霉BL18豆瓣样品中的鲜味氨基酸、甜味氨基酸总量分别提升33.3%和10.8%,必需氨基酸、苦味氨基酸总量略有提升(8.1%、9.4%),鲜味及甜味氨基酸的提升可能有助于提高豆瓣酱鲜甜口感。以上结果表明米曲霉BL18发酵的豆瓣酱在氨基酸组成方面优于3.042。
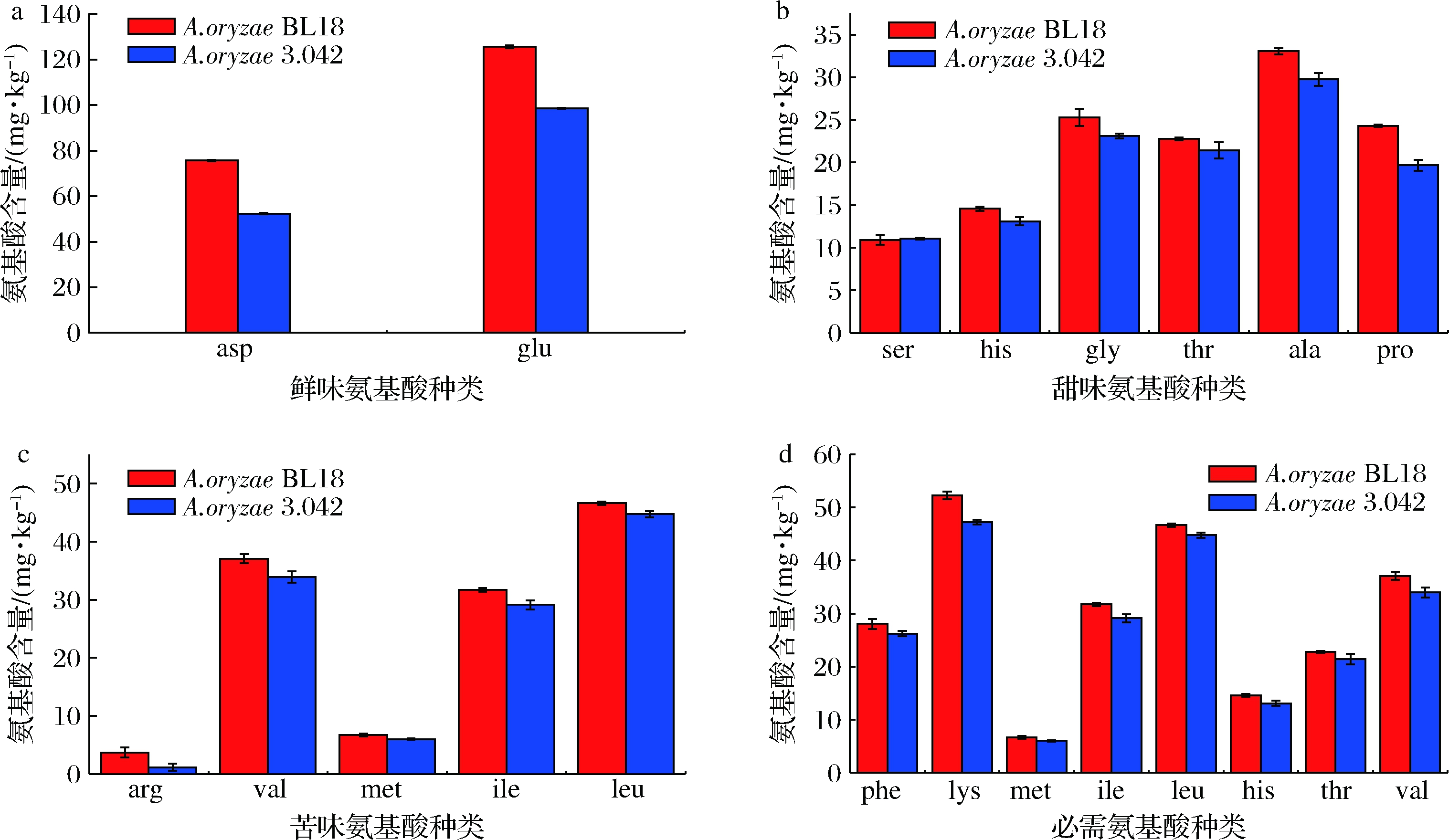
a-鲜味氨基酸;b-甜味氨基酸;c-苦味氨基酸;d-必需氨基酸
图8 发酵56 d游离氨基酸含量
Fig.8 Free amino acid content at day 56 of fermentation
2.9 米曲霉BL18基因组测序结果
为进一步分析米曲霉BL18遗传信息,挖掘其具有优良发酵性能的分子基础,对米曲霉BL18进行全基因组测序。基因组组装结果如表1及图9-a显示。
表1 米曲霉BL18基因统计
Table 1 Aspergillus oryzae BL18 gene statistics
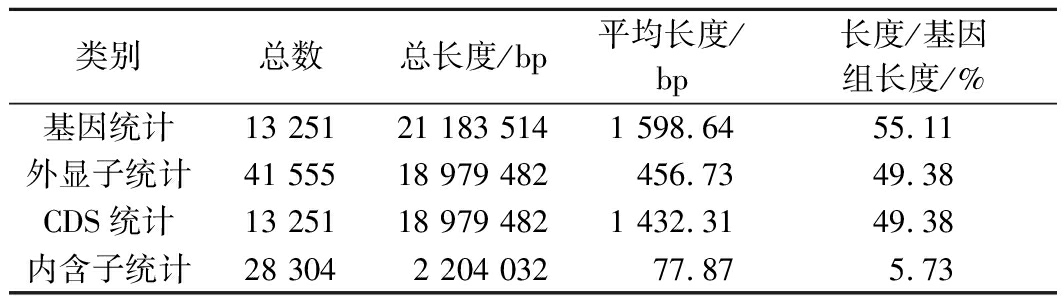
类别总数总长度/bp平均长度/bp长度/基因组长度/%基因统计13 25121 183 5141 598.6455.11外显子统计41 55518 979 482456.7349.38CDS统计13 25118 979 4821 432.3149.38内含子统计28 3042 204 03277.875.73
米曲霉BL18基因组共有9个Scaffold、17个Contig,全长38 438 172 bp,GC含量为47.18%。编码蛋白基因共13 251个,占全基因组长度的49.38%;外显子数41 555,占全基因组长度的49.38%;内含子数28 304,占全基因组长度的5.73%。米曲霉3.042则预测到11 379个基因,外显子区总长度为44%[24],基因数量和外显子长度比例的差异,一方面是测序技术和分析手段的进步引起的,另一方面也体现了米曲霉BL18优势酿造性能的分子基础。
对米曲霉 BL18 基因组注释的基因进行 KOG 功能注释(图10),其中以下功能类群富集了大量功能基因:氨基酸的运输与代谢(Amino acid transport and metabolism)、能量生产与转换(Energy production and conversion)、脂肪运输与代谢(Lipid transport and metabolism)、糖运输与代谢(Carbohydrate transport and metabolism)、次级代谢(Secondary metabolisms biosynthesis,transport and catabolism)、翻译、核糖体结构和生物发生(Translation, ribosomal structure and biogenesis)、辅酶运输和代谢(Coenzyme transport and metabolism)、翻译后修饰(Posttranslational modification,protein turnover, chaperones)。大量功能基因的富集,表明米曲霉具有强大的能量合成能力、蛋白质合成及转运的能力,这是其在发酵工业广泛应用的遗传基础[25]。类群次级代谢富集大量基因,表明米曲霉具有较强的次级代谢产物合成能力,这为米曲霉产生多种多样的风味功能物质提供了可能。
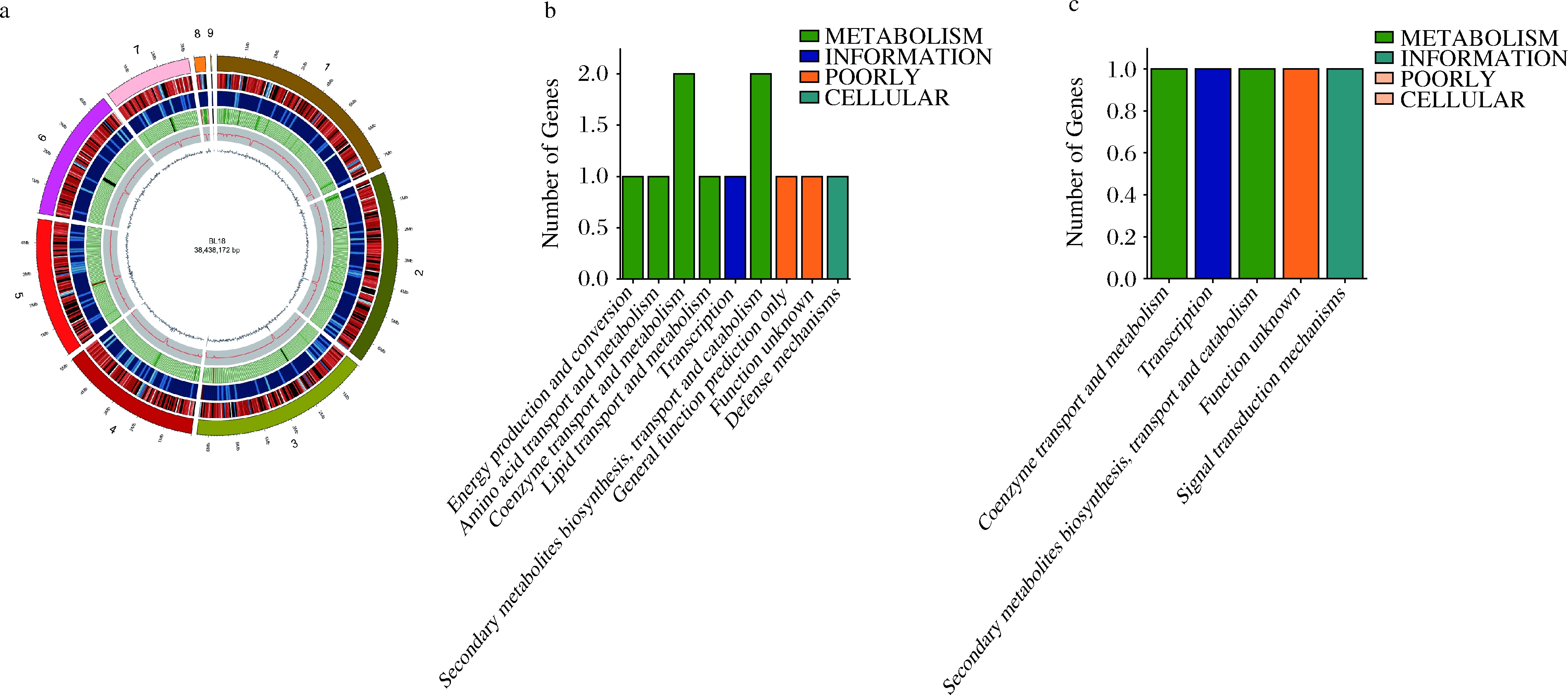
a-米曲霉BL18基因组圈图;b-米曲霉BL18特有基因注释结果;c-米曲霉3.042特有基因注释结果
图9 米曲霉BL18基因组及特有基因注释信息
Fig.9 A. oryzae BL18 genome and unique gene annotation information
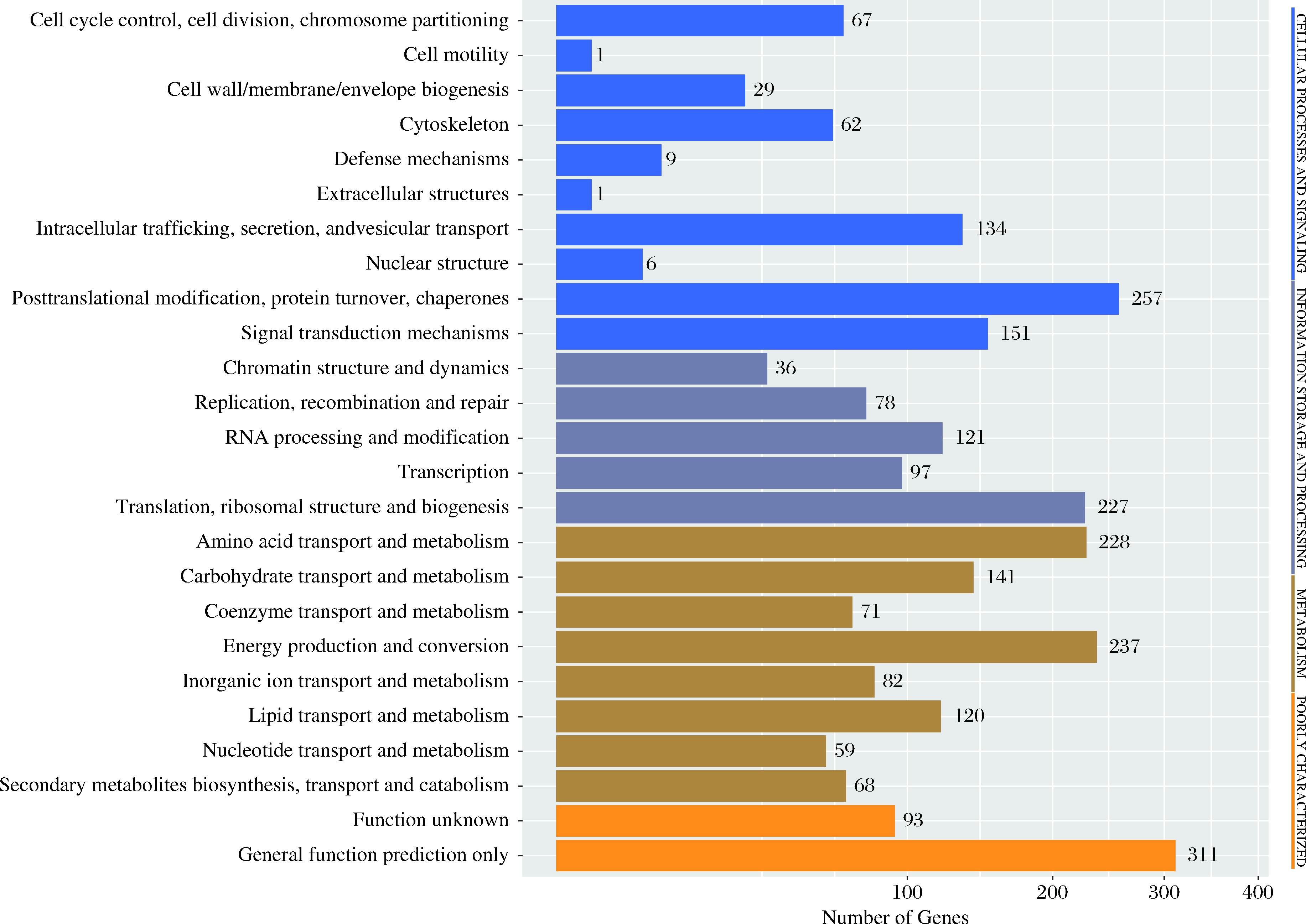
图10 米曲霉BL18基因KOG功能注释
Fig.10 A. oryzae BL18 gene KOG annotation results
对米曲霉BL18基因组注释的基因进行KEGG途径分析(图11),以下途径富集了大量功能基因:碳水化合物代谢(Carbohydrate metabolism)、氨基酸代谢(Amino acid metabolism)、运输和分解代谢(Transport and catabolism)、翻译(Translation)、脂质代谢(Lipid metabolism)、信号转导(Signal transduction)、折叠、排序与降解(Folding, sorting and degradation)、能量代谢(Energy metabolism)。这与KOG功能富集分析的结果是一致的,说明米曲霉BL18在碳代谢(与生长相关)、蛋白质合成、能量代谢方面具有遗传优势。此前关于米曲霉酿造模式菌株RIB40转录组研究的结果表明,与能量代谢、蛋白质合成等相关的基因在固体发酵条件下具有高的转录表达水平[26],也说明米曲霉在基因组上与能量代谢、蛋白质合成相关优势也能体现在实际的发酵应用中。

图11 米曲霉BL18基因的KEGG代谢途径注释
Fig.11 KEGG pathway analysis of annotated genes in the genome of A. oryzae BL18
同时分析比较了米曲霉 BL18和米曲霉3.042的基因序列,共同拥有的基因为共有基因(多数为菌株生长必须基因),而某个样品所特有的基因称为这个样品的特有基因。共有基因和特有基因的研究对不同样品基因组的功能差异性和相似性的研究有重要作用,为物种表型的相似性和差异性的研究提供分子依据。2株米曲霉共有基因为4 359个,米曲霉BL18中有647个特有基因,而米曲霉3.042仅为57个。进一步对特有基因在COG数据库进行注释,结果如图9-b及图9-c所示,米曲霉BL18注释到11个基因,而米曲霉3.042只注释到5个基因,而且米曲霉BL18特有基因注释到氨基酸转运及代谢、能量产生和转化以及脂质转运及代谢功能。
3 结论
本研究通过对米曲霉BL18进行豆瓣酱的制曲发酵,并对常规理化指标进行跟踪,发现米曲霉BL18在豆瓣酱的生产过程具有优势性能,同时对优势性能的分子基础进行了初步探究。在豆瓣酱制曲过程,米曲霉BL18能有效提高制曲过程孢子数以及成曲蛋白酶活力,米曲霉BL18成曲的孢子数达到4.19×108个/g干曲,酸性、中性和碱性蛋白酶活力分别提高52%、7.6%和45.6%。13种游离氨基酸包括天冬氨酸、谷氨酸、酪氨酸等含量也有所提高。米曲霉BL18蛋白酶在豆瓣酱发酵前期具有优势,由于可滴定酸的迅速增加,酱醅的pH值快速下降,导致蛋白酶活力的下降,氨基酸态氮的生成主要集中在发酵初期。对发酵后的豆瓣酱游离氨基酸进行分析,发现米曲霉BL18豆瓣样品中的鲜味氨基酸、甜味氨基酸总量分别提升33.3%和10.8%。与米曲霉3.042进行比较,发现从制曲过程蛋白酶的活力、孢子产生速率及数量、发酵豆瓣酱的蛋白酶活力及氨基酸组成等方面,米曲霉BL18均优于对照组。米曲霉BL18基因KOG和KEGG数据库注释结果表明米曲霉 BL18具有强大的能量合成、蛋白质合成及次级代谢产物合成能力。分析米曲霉BL18和3.042共性基因与特性基因时,发现米曲霉BL18中有647个特有基因,而米曲霉3.042仅为57个,特有基因功能集中在氨基酸转运及代谢、能量产生和转化以及脂质转运及代谢功能。米曲霉BL18在制曲发酵过程表现出的优势性能及大量功能基因的注释为其在豆瓣酱发酵应用中提供了理论基础。
[1] 冯宽, 孙群, 邓维琴, 等.一种同时测定豆瓣酱中的7种有机酸的HPLC方法[J].食品与发酵科技, 2019, 55(4):128-131;141.
FENG K, SUN Q, DENG W Q, et al.A HPLC method for determination of seven kinds of organic acids in broad bean paste with different fermentation year[J].Food and Fermentation Sciences & Technology, 2019, 55(4):128-131;141.
[2] HE B, TU Y Y, JIANG C M, et al.Functional genomics of Aspergillus oryzae:Strategies and progress[J].Microorganisms, 2019, 7(4):103.
[3] YUE X Q, LI M H, LIU Y M, et al.Microbial diversity and function of soybean paste in East Asia:What we know and what we don’t[J].Current Opinion in Food Science, 2021, 37:145-152.
[4] ZHU Y Y, LIANG X L, ZHANG H, et al.A comparative secretome analysis of industrial Aspergillus oryzae and its spontaneous mutant ZJGS-LZ-21[J].International Journal of Food Microbiology, 2017, 248:1-9.
[5] ZHAO G Z, HOU L H, LU M F, et al.Construction of the mutant strain in Aspergillus oryzae 3.042 for abundant proteinase production by the N+ ion implantation mutagenesis[J].International Journal of Food Science & Technology, 2012, 47(3):504-510.
[6] XU D F, PAN L, ZHAO H F, et al.Breeding and identification of novel koji molds with high activity of acid protease by genome recombination between Aspergillus oryzae and Aspergillus niger[J].Journal of Industrial Microbiology and Biotechnology, 2011, 38(9):1 255-1 265.
[7] GAO X L, LIU E M, YIN Y Y, et al.Enhancing activities of salt-tolerant proteases secreted by Aspergillus oryzae using atmospheric and room-temperature plasma mutagenesis[J].Journal of Agricultural and Food Chemistry, 2020, 68(9):2 757-2 764.
[8] 钮成拓, 赵帅, 杜姝颖, 等.一株产高蛋白酶活的米曲霉及其在豆酱发酵中的应用:CN111808757B[P].2022-04-29.
[9] 高秀芝, 王小芬, 李献梅, 等.天源酱园豆酱发酵过程中营养及理化指标动态[J].食品科学, 2008, 29(9):352-354.
GAO X Z, WANG X F, LI X M, et al.Dynamics of nutrients and physical and chemical parameters during fermentation of Tianyuan Jiangyuan soybean paste[J].Food Science, 2008, 29(9):352-354.
[10] BIRNEY E, CLAMP M, DURBIN R.GeneWise and genomewise[J].Genome Research, 2004, 14(5):988-995.
[11] JOHNSON A D, HANDSAKER R E, PULIT S L, et al.SNAP:A web-based tool for identification and annotation of proxy SNPs using HapMap[J].Bioinformatics, 2008, 24(24):2 938-2 939.
[12] STANKE M, DIEKHANS M, BAERTSCH R, et al.Using native and syntenically mapped cDNA alignments to improve de novo gene finding[J].Bioinformatics, 2008, 24(5):637-644.
[13] TER-HOVHANNISYAN V, LOMSADZE A, CHERNOFF Y O, et al.Gene prediction in novel fungal genomes using an ab initio algorithm with unsupervised training[J].Genome Research, 2008, 18(12):1 979-1 990.
[14] FU L M, NIU B F, ZHU Z W, et al.CD-HIT:Accelerated for clustering the next-generation sequencing data[J].Bioinformatics, 2012, 28(23):3 150-3 152.
[15] 林祖申. 米曲霉制曲过程中酶活性变化及其工艺优化[J].中国酿造, 2007, 26(5):56-59.
LIN Z S.Change of enzyme activity in Aspergillus oryzae cultivation and technical optimization[J].China Brewing, 2007, 26(5):56-59.
[16] BALTUSSEN T J H, ZOLL J, VERWEIJ P E, et al.Molecular mechanisms of conidial germination in Aspergillus spp[J].Microbiology and Molecular Biology Reviews:MMBR, 2019, 84(1):e00049-e00019.
[17] KUSUMOTO K I, YAMAGATA Y, TAZAWA R, et al.Japanese traditional miso and koji making[J].Journal of Fungi (Basel, Switzerland), 2021, 7(7):579.
[18] JIA Y, NIU C T, LU Z M, et al.A bottom-up approach to develop a synthetic microbial community model:Application for efficient reduced-salt broad bean paste fermentation[J].Applied and Environmental Microbiology, 2020, 86(12):e00306-e00320.
[19] HOANG N X, FERNG S, TING C H, et al.Optimizing the initial moromi fermentation conditions to improve the quality of soy sauce[J].LWT, 2016, 74:242-250.
[20] WU J R, TIAN T, LIU Y M, et al.The dynamic changes of chemical components and microbiota during the natural fermentation process in Da-Jiang, a Chinese popular traditional fermented condiment[J].Food Research International, 2018, 112:457-467.
[21] 王栋, 冯杰, 张丽敏, 等.耐盐米曲霉CICIM F0899在酱油酿造过程中的应用[J].食品与发酵工业, 2012, 38(5):1-6.
WANG D, FENG J, ZHANG L M, et al.Application of a salt-tolerant Aspergillus oryzae CICIM F0899 in soy sauce fermentation[J].Food and Fermentation Industries, 2012, 38(5):1-6.
[22] CHOU C C, LING M Y.Biochemical changes in soy sauce prepared with extruded and traditional raw materials[J].Food Research International, 1998, 31(6-7):487-492.
[23] HAN B Z, ROMBOUTS F M, NOUT M J R.Amino acid profiles of sufu, a Chinese fermented soybean food[J].Journal of Food Composition and Analysis, 2004, 17(6):689-698.
[24] ZHAO G Z, YAO Y P, QI W, et al.Draft genome sequence of Aspergillus oryzae strain 3.042[J].Eukaryotic Cell, 2012, 11(9):1178.
[25] KANG C, YU X W, XU Y.Cloning and expression of a novel prolyl endopeptidase from Aspergillus oryzae and its application in beer stabilization[J].Journal of Industrial Microbiology and Biotechnology, 2015, 42(2):263-272.
[26] WANG B, GUO G W, WANG C, et al.Survey of the transcriptome of Aspergillus oryzae via massively parallel mRNA sequencing[J].Nucleic Acids Research, 2010, 38(15):5 075-5 087.