如今,牛奶过敏、乳糖不耐症、高胆固醇血症以及对素食主义的青睐等影响了消费者对于牛乳替代品的选择[1]。尽管以乳制品为基础的功能性食品种类非常丰富,但植物源性乳作为乳制品的可持续替代品,因其丰富的营养价值以及能够缓解畜牧业对环境的巨大压力而受到广泛关注[2]。《2020~2025年中国植物蛋白饮料行业市场需求与投资规划分析报告》指出,在食品行业未来几年的发展中,植物乳制品将有希望保持20%以上的平均增长速度。植物基蛋白发酵酸奶是以大豆、椰子等植物原料作为基底,辅以代糖、柠檬酸等其他原料,经菌株发酵后得到的酸奶[3]。而作为一种优质植物源性原料,大豆具有高蛋白营养、植物甾醇和多不饱和脂肪酸以及低成本效益等特点[4],同时其所含的肽类已被证明具有抗氧化、抗癌、抗菌、免疫调节和胰岛素调节等特性,因此,大豆成为了近年来功能性食品研究的新热点。
对发酵乳制品中微生物多样性的研究,传统方法在多数情况下采用分离培养,但是在各种环境样品中仅有小于1%的微生物能获得纯培养。而如变性梯度凝胶电泳、荧光原位杂交等传统免培养技术能检测到的微生物种类有限且准确性低。基于高通量测序技术的宏基因组学技术则可对生态位中提取的所有微生物DNA进行深度测序和分析,鉴定其物种组成、菌群结构和表征群落丰度,具有读长高、精度高、通量高、无偏性等优势。同时,技术的发展和成熟也使得高通量测序更多地用于乳制品及其他领域微生物群落分析,如用高通量测序研究腌制麻竹笋中细菌群落的动态演替[5];分析新疆不同地区自然发酵辣椒酱的微生物群落多样性[6];分析西藏牦牛酸奶菌群多样性[7];评价不同饲料源附近生微生物群及其对纳皮尔草发酵品质和微生物多样性的影响[8]。
目前对酸奶微生物多样性的研究多数以牛乳为原料,对植物蛋白发酵酸奶与动物蛋白发酵酸奶进行对比的研究甚少,而进一步开发植物蛋白中特殊的微生物资源将逐渐成为研究的热点。植物蛋白酸奶虽然具有广阔的发展前景,但仍要解决营养不均衡、发酵体系不稳定等问题[1]。本研究通过对黄豆发酵酸奶和牛乳发酵酸奶中菌群组成、结构和群落差异进行分析,探究植物蛋白与动物蛋白发酵乳品中细菌多样性之间的联系,以期为开发利用植物蛋白中微生物资源奠定基础。
1 材料与方法
1.1 材料与试剂
乳酸菌酸奶发酵粉(经典5菌型:保加利亚乳杆菌、嗜热链球菌、嗜酸乳杆菌、植物乳杆菌、干酪乳杆菌),北京川秀科技有限公司;罗汉果糖,湖南华城生物资源股份有限公司;伊利无菌砖纯牛奶,长沙市沃尔玛超市;FastDNA® Spin Kit for Soi DNA抽提试剂盒,美国MP Biomedicals 公司;NEXTFLEX Rapid DNA-Seq Kit建库试剂盒,美国Bioo Scientific公司;biowest agArose琼脂糖,西班牙biowest公司;MiSeq Reagent Kit v3测序试剂盒,美国Illumina公司;FastPfu Polymerase,中国TransGen公司。
1.2 仪器与设备
N13462C移液器、5424R高速台式冷冻离心机,德国Eppendorf公司;ABSON MiFly-6小型离心机,合肥艾本森科学仪器有限公司;GeneAmp®9700 PCR仪,美国ABI公司;QuantusTM Fluorometer微型荧光计,美国Promega公司;Illumina MiSeq测序仪,美国Illumina公司;ELx800酶标仪,美国Biotek公司;TL-48R粉碎研磨仪,上海万柏生物科技有限公司;QL-901旋涡混合器,海门其林贝尔仪器制造有限公司;DYY-6C电泳仪,北京市六一仪器厂。
1.3 实验方法
1.3.1 纯黄豆、纯牛乳发酵酸奶的制备
以纯黄豆发酵酸奶为例,黄豆冷水浸泡10 h后热开水浸泡15 min,榨汁并加入1.5 g琼脂,过滤后加入13 g罗汉果糖,经25~30 MPa均质处理后95 ℃高温灭菌10 min,待混合物降温至40 ℃,接种1.0 g乳酸菌发酵粉进行发酵处理,于42 ℃恒温发酵6.5 h,经杀菌包装后于低温下冷藏保存。牛乳发酵酸奶原料采用牛乳替代黄豆汁,其余流程与纯黄豆发酵酸奶相同,无糖样本不添加罗汉果糖。黄豆发酵酸奶制备工艺流程如图1所示。取1~2 g酸奶样本置于无菌冻存管中,做3个平行,共12个样本于-80 ℃保存。
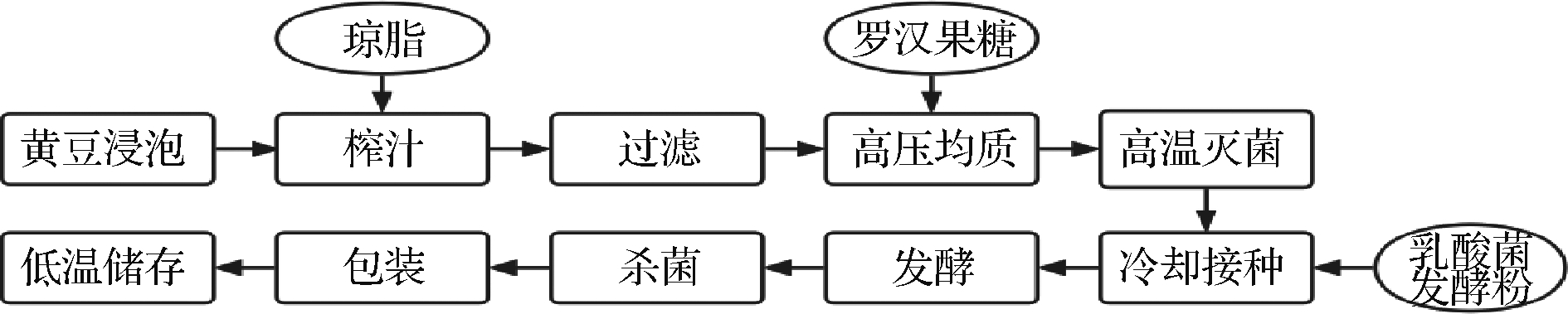
图1 纯黄豆发酵酸奶制备工艺流程图
Fig.1 The production process flow chart of soy fermented yoghurt
1.3.2 理化指标的测定
水分含量参照GB 5009.3—2016《食品安全国家标准 食品中水分的测定》中的直接干燥法测定;灰分含量参照GB 5009.4—2016《食品安全国家标准 食品中灰分的测定》;脂肪含量参照GB 5009.6—2016《食品安全国家标准 食品中脂肪的测定》索氏抽提法测定;蛋白质含量参照GB 5009.4—2016《食品安全国家标准 食品中蛋白质的测定》中的凯氏定氮法测定;碳水化合物含量通过组分总量减去水分、灰分、脂肪和蛋白质的组分含量测得;钠含量参照GB 5009.91—2017《食品安全国家标准 食品中钾、钠的测定》中的火焰原子吸收光谱法测定。
1.3.3 菌群DNA抽提和PCR扩增测序
酸奶菌群DNA提取参照李薇等[5]、戈子龙等[9]的方法稍作改良。以338F-806R(468 bp)为引物,扩增细菌16S rRNA V3-V4可变区。PCR扩增具体体系为5×FastPfu Buffer 4 μL、2.5 mmol/L dNTPs 2 μL、Forward Primer 0.8 μL、Reverse Primer 0.8 μL、FastPfu Polymerase 0.4 μL、BSA 0.2 μL、Template DNA 10 ng,补ddH2O至20 μL。PCR扩增具体程序为95 ℃预变性3 min,95 ℃变性30 s、55 ℃退火30 s、72 ℃延伸45 s重复进行27次,72 ℃延伸10 min。Picogreen染料荧光计对PCR产物进行定量均一化混匀,2%琼脂糖凝胶电泳检测,Miseq PE300平台测序。上海美吉生物医药科技有限公司完成PCR扩增及测序工作。
1.3.4 数据处理
使用SPSS Statistics 25.0软件对理化指标数据进行统计分析。基于美吉生物云平台对环境微生物群落进行交互式分析并对数据进行处理。质控采用FASTP软件[10],拼接在FLASH软件[11]上进行。根据97%[12-13]的相似度在UPARSE软件[13]上对序列进行操作分类单元(operational taxonomic units, OTUs)聚类、剔除嵌合体。通过RDP classifier[14]进行物种分类注释,比对阈值70%(Silva 16SrRNA数据库-v138)。
2 结果与分析
2.1 测序信息统计
通过测序12个样品共产生564 314个序列数。去除叶绿体、线粒体等背景污染、抽平序列后的有效序列数为557 197,平均长度为428 bp,其中纯黄豆发酵酸奶有271 452个序列数,纯牛乳发酵酸奶有285 745个序列数。通过OTU聚类,总共汇聚成116个OTU。详细数据如表1所示。
表1 测序数据结果统计表
Table 1 The statistical table of sequencing data results
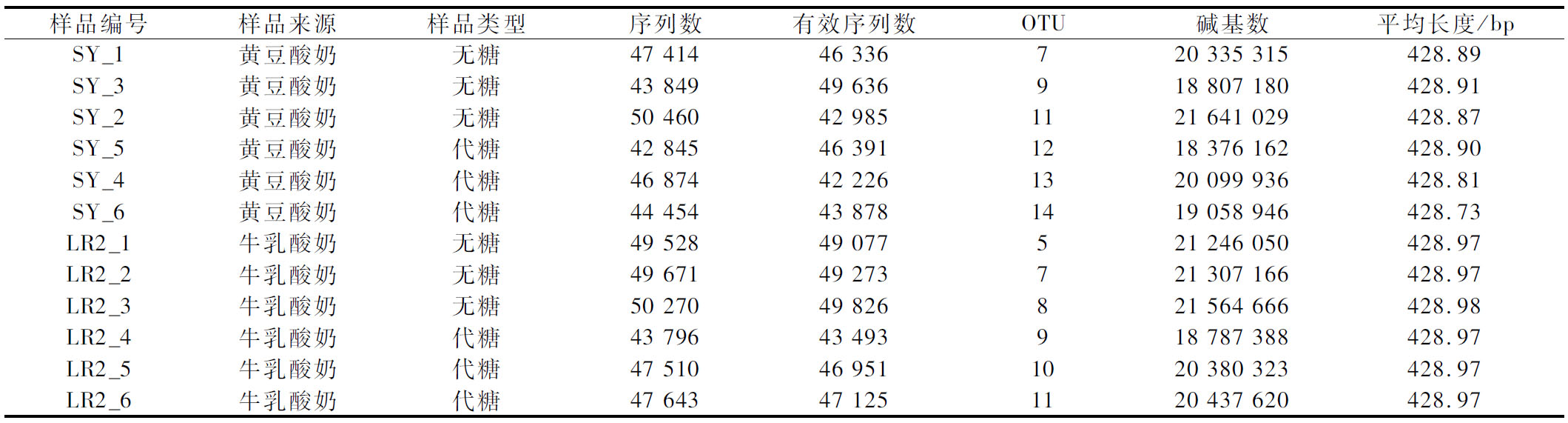
样品编号样品来源样品类型序列数有效序列数OTU碱基数平均长度/bpSY_1黄豆酸奶无糖47 41446 336720 335 315428.89SY_3黄豆酸奶无糖43 84949 636918 807 180428.91SY_2黄豆酸奶无糖50 46042 9851121 641 029428.87SY_5黄豆酸奶代糖42 84546 3911218 376 162428.90SY_4黄豆酸奶代糖46 87442 2261320 099 936428.81SY_6黄豆酸奶代糖44 45443 8781419 058 946428.73LR2_1牛乳酸奶无糖49 52849 077521 246 050428.97LR2_2牛乳酸奶无糖49 67149 273721 307 166428.97LR2_3牛乳酸奶无糖50 27049 826821 564 666428.98LR2_4牛乳酸奶代糖43 79643 493918 787 388428.97LR2_5牛乳酸奶代糖47 51046 9511020 380 323428.97LR2_6牛乳酸奶代糖47 64347 1251120 437 620428.97
2.2 纯黄豆、牛奶发酵酸奶菌群多样性分析
2.2.1 稀释曲线和α-多样性分析
当测序深度逐渐增加时,稀释曲线趋近于平缓(图2),2组发酵酸奶样本在测序覆盖度上没有显著差异,所有测序覆盖度高于99.99%以上,说明对纯黄豆、纯牛乳发酵酸奶微生物群落的检测率接近饱和,测序量能够覆盖酸奶中的绝大部分物种。
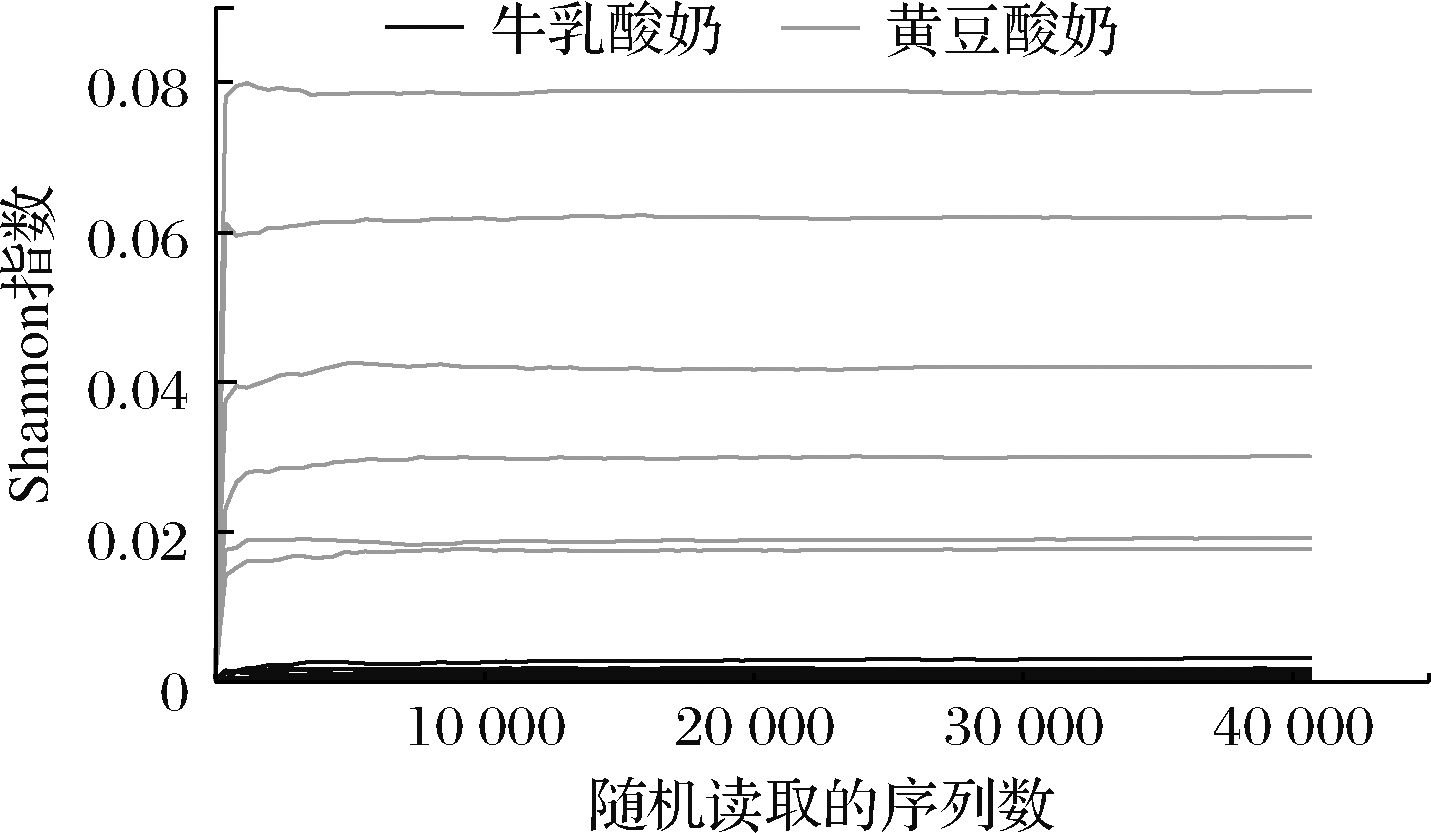
图2 Shannon指数稀释曲线
Fig.2 The Shannon exponential dilution curve
对纯黄豆发酵酸奶(SY)和纯牛乳发酵酸奶(LR2)微生物群落多样性指数进行分析(表2)。指数组间差异检验结果如图3所示。
表2 基于T检验的α-多样性指数表
Table 2 α-Diversity index table based on T test
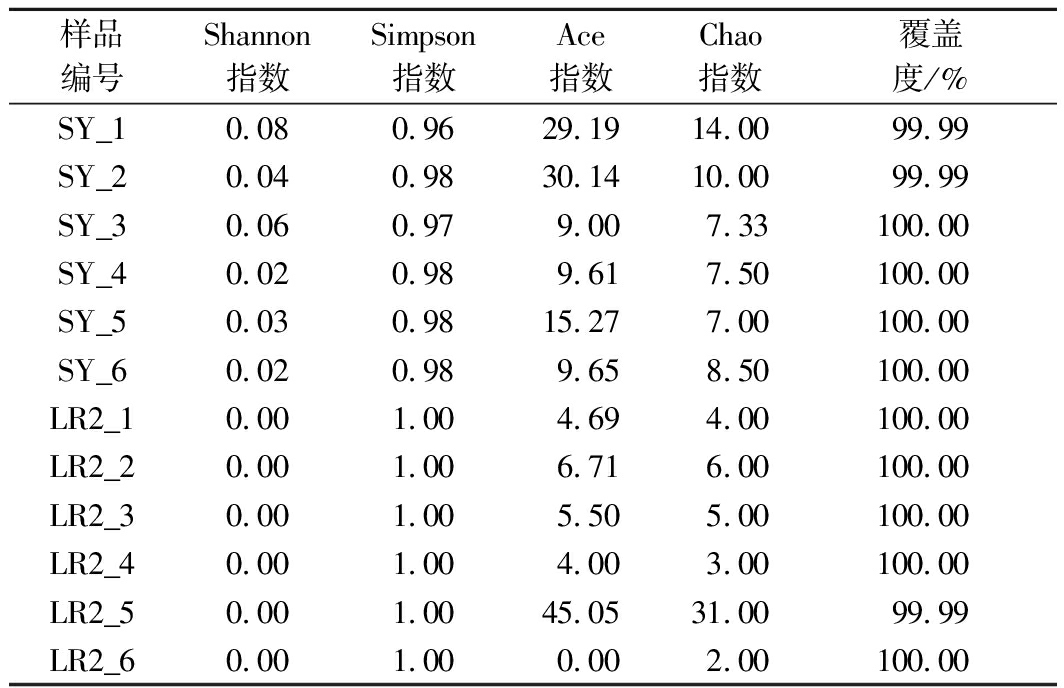
样品编号Shannon指数Simpson指数Ace指数Chao指数覆盖度/%SY_10.080.9629.1914.0099.99SY_20.040.9830.1410.0099.99SY_30.060.979.007.33100.00SY_40.020.989.617.50100.00SY_50.030.9815.277.00100.00SY_60.020.989.658.50100.00LR2_10.001.004.694.00100.00LR2_20.001.006.716.00100.00LR2_30.001.005.505.00100.00LR2_40.001.004.003.00100.00LR2_50.001.0045.0531.0099.99LR2_60.001.000.002.00100.00
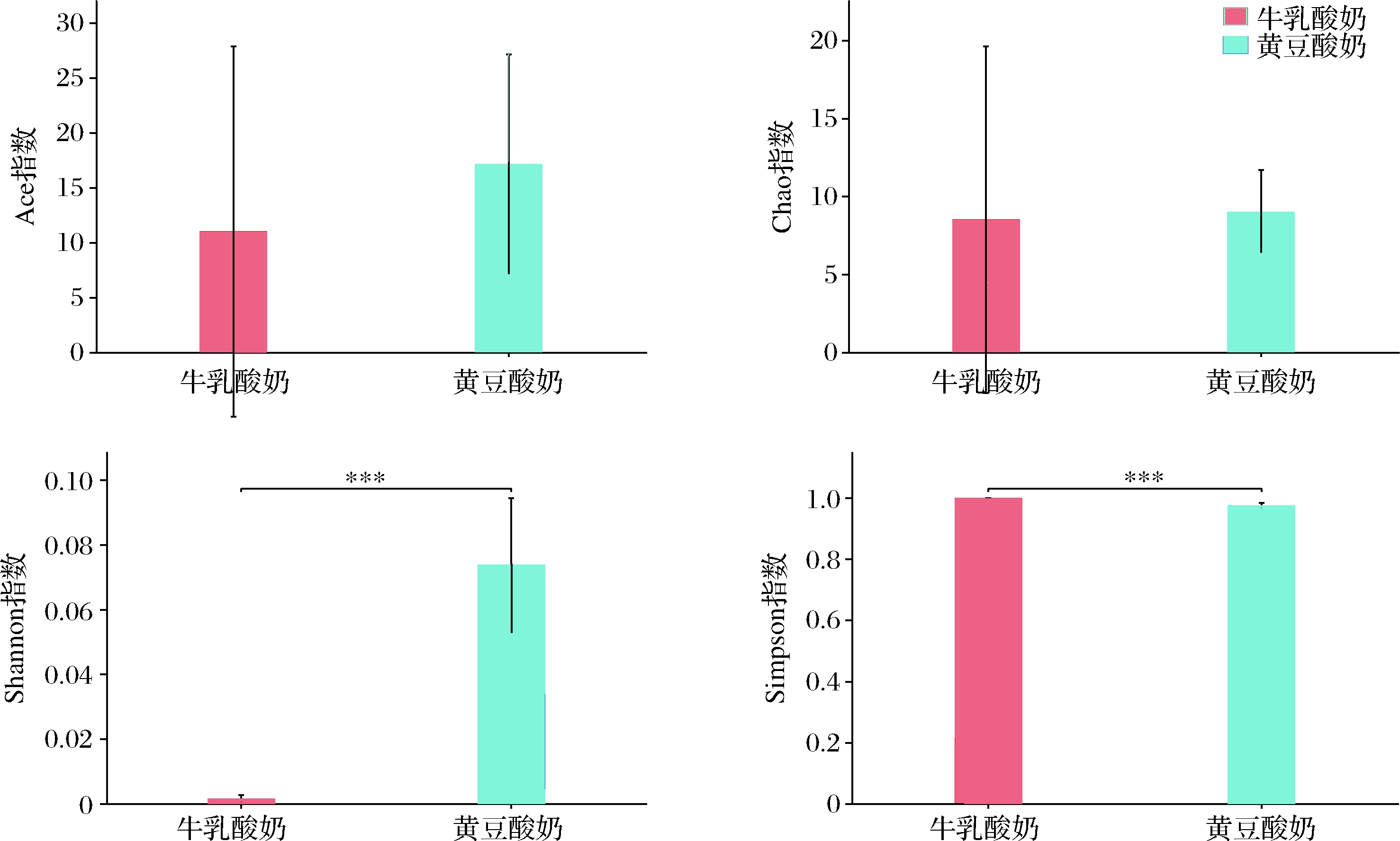
图3 指数组间差异检验柱形图
Fig.3 Histogram of difference test between index groups
注:***表示存在极显著性差异(P<0.01)
两组样本检测到包括Chao指数(P=0.907 6)和Ace指数(P=0.459 1)的群落OTU丰富度没有显著性差异。然而,两组样本的群落多样性指数,包括Shannon指数(P≤0.001)和Simpson指数(P≤0.001)具有极显著性差异,表明黄豆发酵酸奶细菌多样性高于牛乳发酵酸奶。
2.2.2 Beta多样性分析
纯黄豆、纯牛乳发酵酸奶通过置换多元方差分析(permutational multivariate analysis of variance,PERMANOVA),黄豆样本、牛乳样本组间菌群结构差异极显著大于组内差异(R=0.577 8,P=0.003<0.01)(图4),本次实验分组具有意义。
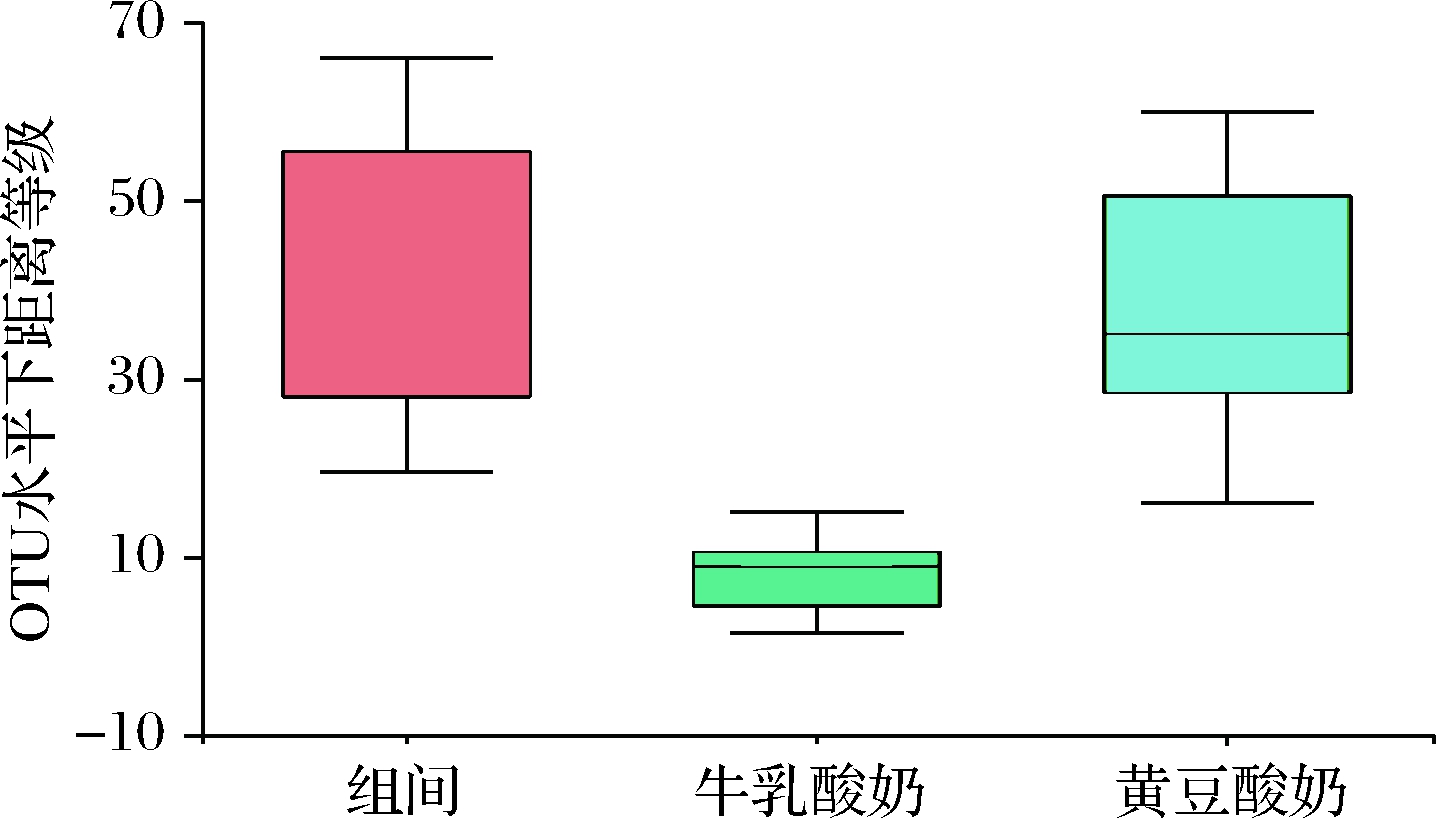
图4 基于Bray-Curtis的组间距离盒状图
Fig.4 The box plot of distance between groups based on Bray-Curtis
主坐标分析结果表明(图5),在OTU水平上,黄豆样本、牛乳样本分别聚集在一起(R2=0.207 5,P=0.039),第1主成分对样本菌群结构差异解释度所占比重为39.15%,第2、第3主成分分别为21.13%、14.37%,黄豆样本与牛乳样本组间群落组成差异极其显著(P<0.05)。
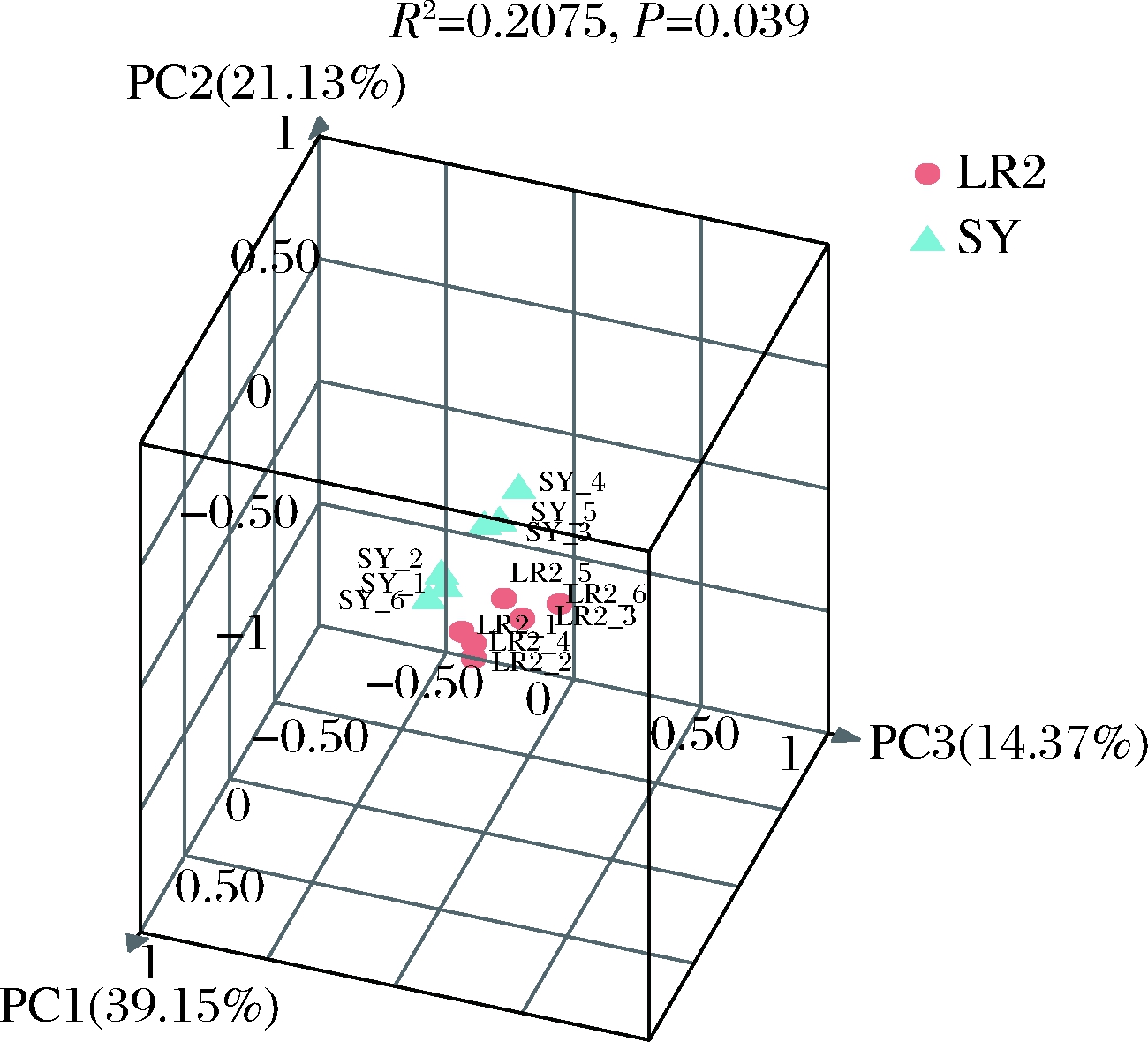
图5 基于unweighted_unifrac的细菌群落主坐标分析3D图
Fig.5 The PCoA-3D of bacterial community
based on unweighted_unifrac
2.3 纯黄豆、牛乳发酵酸奶中营养成分和细菌菌群的组成及差异分析
2.3.1 营养指标分析
如表3所示,水分、脂肪和碳水化合物的含量在黄豆样本和牛乳样本中有显著性的差异;黄豆无糖、牛乳无糖样本水分含量明显高于黄豆代糖、牛乳代糖样本;黄豆无糖、牛乳无糖样本碳水化合物含量远低于黄豆代糖、牛乳代糖样本。由此可知,原料不同对于发酵酸奶中主要营养组分的含量具有一定的影响。
表3 黄豆、牛乳发酵酸奶的营养指标分析 单位:g/100 g
Table 3 The nutritional index analysis of soybean and milk fermented yoghurt

样本水分灰分脂肪蛋白质碳水化合物钠牛乳无糖87.20±0.02a0.69±0.01a3.93±0.01a3.21±0.01a4.97±0.01a36.80±0.01a牛乳代糖83.10±0.02b0.67±0.01b3.84±0.01b3.03±0.01b9.36±0.01b35.70±0.01b黄豆无糖93.70±0.01c0.37±0.01c1.62±0.01c2.64±0.01c1.67±0.01c-黄豆代糖90.01±0.01d0.31±0.01d1.60±0.01d2.41±0.01d5.68±0.01d-
注:“-”表示未检出,检出限为3 mg/100 g
2.3.2 细菌菌群的组成及差异分析
通过菌群组成分析,属水平上共测得12个属(图6-a),链球菌属(Streptococcus)在黄豆样本和牛乳样本菌群中均占主要优势。除链球菌属外(图6-b),芽孢杆菌属(Bacillus)、肠杆菌属(Enterobacter)仅在黄豆样本中存在;棒状杆菌属(Corynebacterium)仅在黄豆无糖5(SY_5)样本中存在;伯克霍尔德氏菌属(Burkholderia)、丛毛单胞菌属(Comamonas)仅在牛乳样本中存在;摩根氏菌属(Morganella)、漫游球菌属(Vagococcus)、假红杆菌属(Pseudorhodobacter)、产碱杆菌属(Alcaligenes)、乳酸杆菌属(Lactobacillus)、泰式菌属(Tissierella)在黄豆样本和牛乳样本中均存在。链球菌属以及乳杆菌属在发酵过程中进行微弱的水解作用,有助于维持较高的发酵活性,经常用于混合发酵以制备大豆酸奶[15]。门水平上,共测得2个门:厚壁菌门(Firmicutes)和变形菌门(Proteobacteria),其中厚壁菌门在黄豆、牛乳样本中均占主要优势,这与张敏等[16]对酸奶中微生物多样性的研究结果相一致。
通过LEfSe多级物种层级分析显示(图7-a),从目水平来看,芽孢杆菌目(Bacillales)在黄豆样本显著富集,而在牛乳样本大量富集乳酸杆菌目(Lactobacillales);从种水平来看,枯草芽孢杆菌枯草亚种(Bacillus
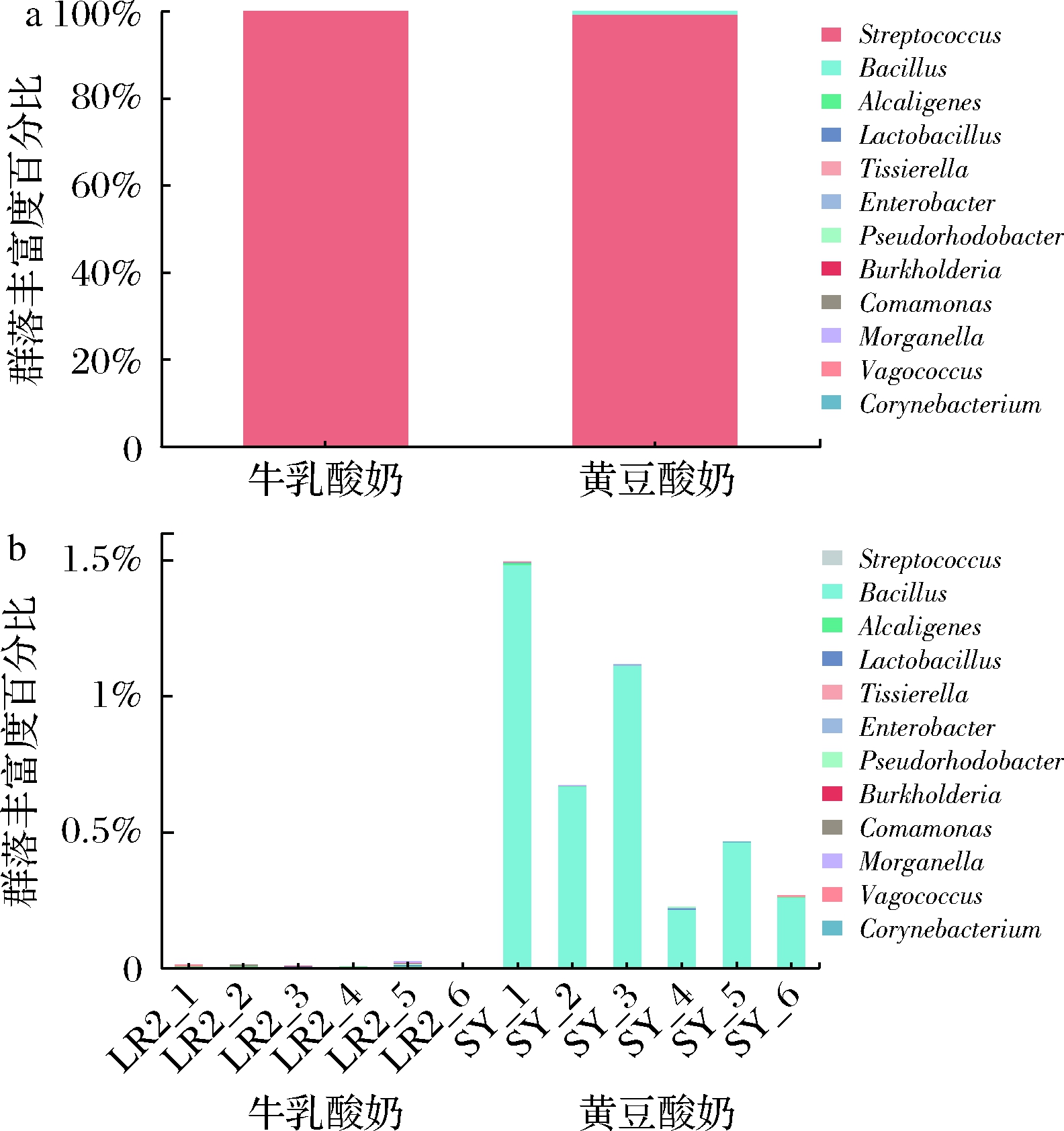
a-属水平上细菌群落;b-隐去链球菌属的细菌群落
图6 属水平下的群落柱状图
Fig.6 The histogram of the community at the genus level
subtilis subsp. subtilis)在黄豆样本显著富集,牛乳样本唾液链球菌嗜热亚种(Streptococcus salivarius subsp. thermophilus)显著富集。线性判别分析(linear discriminant analysis, LDA)(LDA阈值>1.0)(图7-b)表明,对牛乳、黄豆样本菌群结构差异具有显著影响的物种主要包括芽孢杆菌属(Bacillus)和链球菌属(Streptococcus)(P<0.01)。

a-LEfSe多级物种层级分析;b-LDA图
图7 纯黄豆、纯牛乳发酵酸奶菌群差异分析图
Fig.7 The difference analysis of bacterial flora between
pure soybean and pure milk fermented yoghurt
2.3.3 主要营养成分与优势菌属的相关性分析
通过SPSS 25.0和TB tools软件,将黄豆、牛乳发酵酸奶中主要营养成分含量与其菌群中主要优势菌属(链球菌属)丰度进行相关性分析(图8)。可知水分、脂肪和碳水化合物含量与菌群优势菌属丰度值呈极显著相关;蛋白质、钠含量与菌群优势菌属不相关(P>0.05)。结合营养指标分析数据(表3)可推测得,原料中有无添加植物代糖对黄豆、牛乳发酵酸奶中细菌菌群种类有一定的影响,后续对此结论进行了深入的分析与证实。
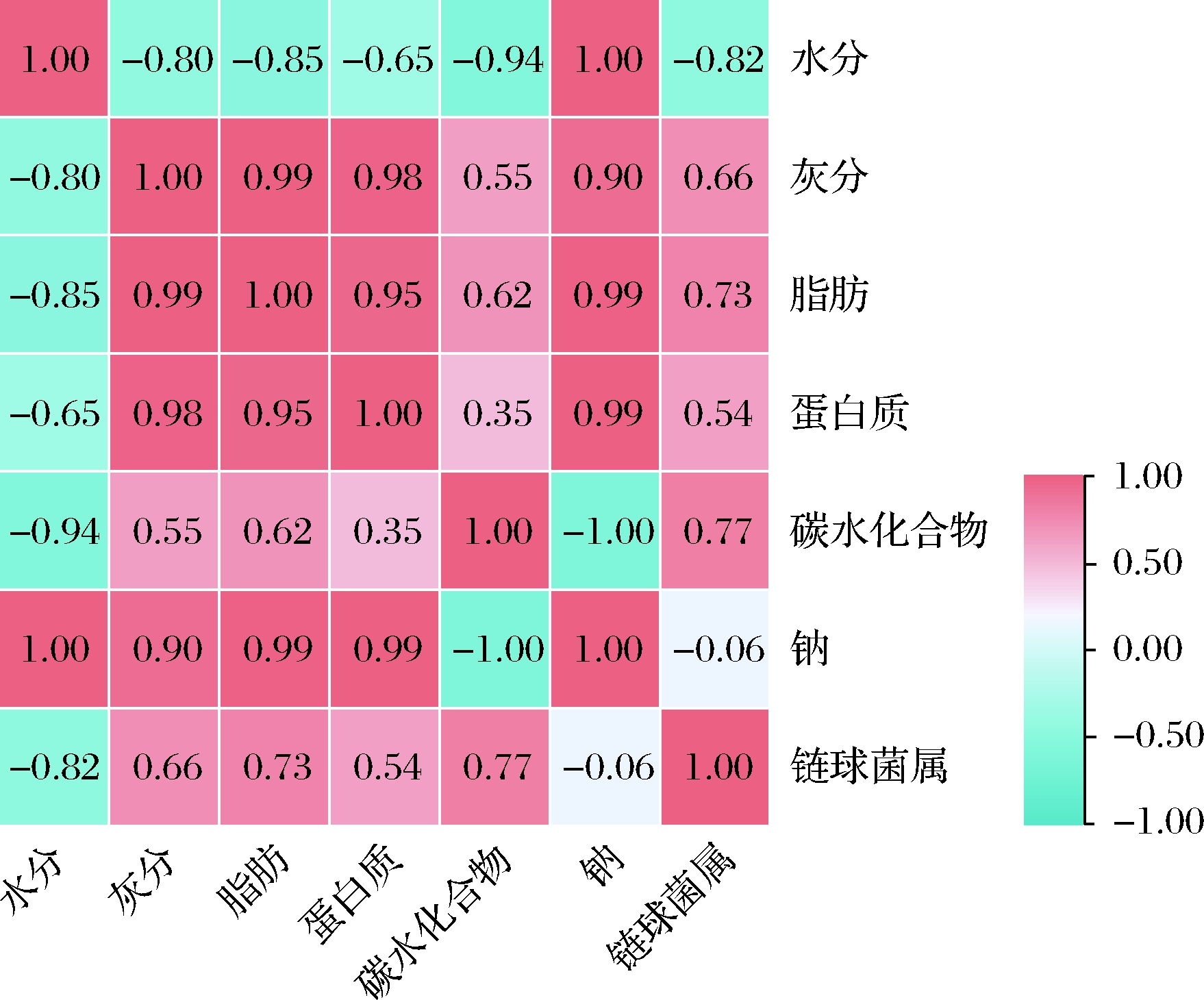
图8 酸奶组分与主要菌群菌属相关性热图
Fig.8 Heat map of correlation between yogurt
components and flora
2.4 植物代糖对黄豆、牛乳发酵酸奶中微生物菌群的影响
本研究从是否添加代糖的角度对2组样本进行层级聚类(图9),添加代糖样本(SY_4、SY_5、SY_6、LR2_4、LR2_5、LR2_6),未添加代糖样本(SY_1、SY_2、SY_3、LR2_1、LR2_2、LR2_3)。根据分析结果,共聚成两大分支,第一大分支为黄豆无糖样本,第二大分支为其他样本。第二大分支中,6个牛乳样本所属同一个小分支,不论有无植物代糖,6个样本分支距离皆小,说明样本特征值组成差异较小,即对于纯牛乳发酵酸奶,添加代糖与否对菌群差异的影响较小;有糖黄豆样本分别属于2个小分支,其中SY_2无糖与SY_5有糖黄豆样本属于同一小分支的原因还需要进一步实验探究。可初步判定,有无添加植物代糖对黄豆样本群落组成有一定的影响。因此,可从有无添加植物代糖的角度对豆类等植物源性发酵酸奶中植物代糖对人体肠道菌群的影响。
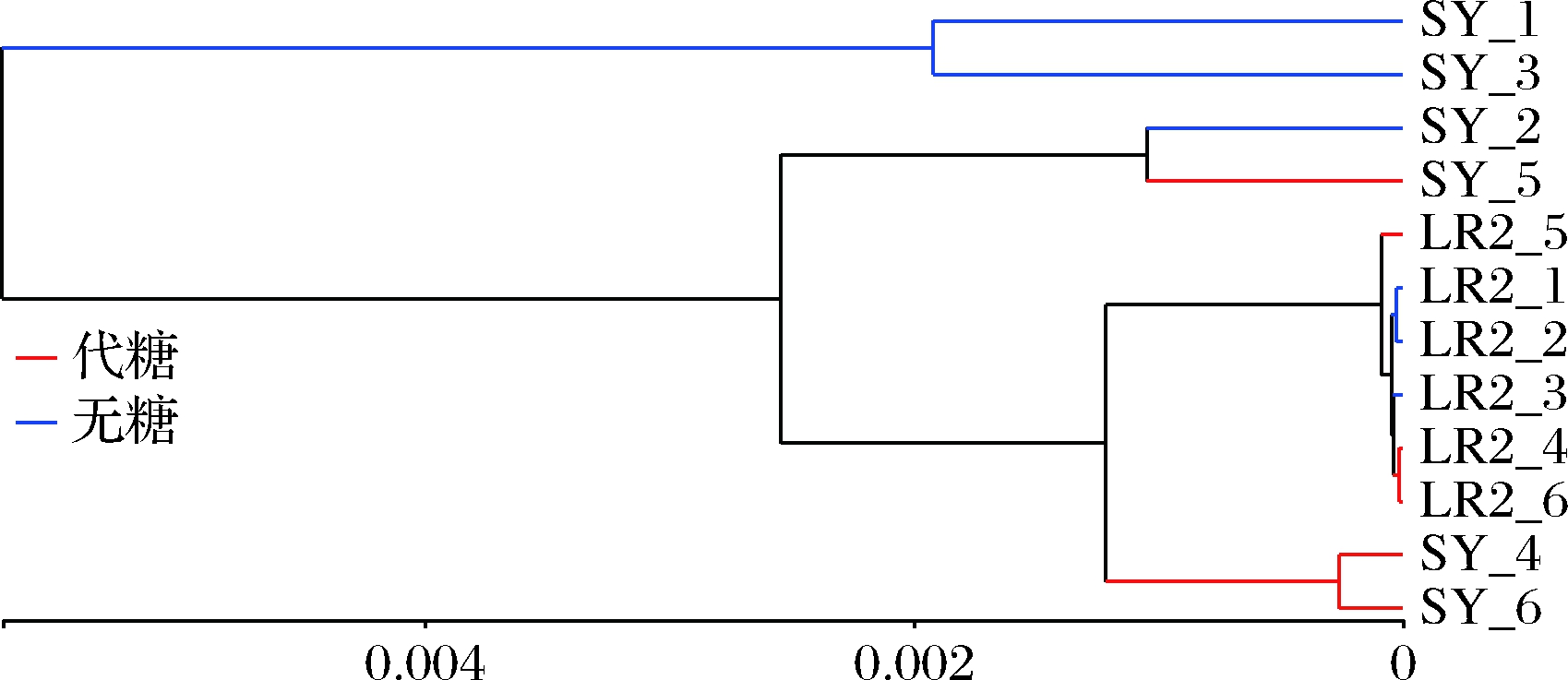
图9 基于Bray-Curtis的样本层级聚类树
Fig.9 The sample level clustering tree on Bray-Curtis
2.5 对黄豆、牛乳样本中微生物群落功能组成的预测
普通酸奶是牛奶经过乳酸菌发酵而成,有改善肠道微环境的作用[17],但以植物蛋白为原料的发酵酸奶对人体肠道菌群、健康功能的影响目前所知甚少。本研究对牛乳与黄豆样本菌群KEGG pathway进行功能组成分析,得到的KEGG功能预测信息组成基本相同(图10),12份样品的细菌群落组成虽存在明显差异(图6),但基因功能一致,只是部分功能丰度变化有所不同,原因可能是不同菌群所具有的基因功能具有相似性[18]。
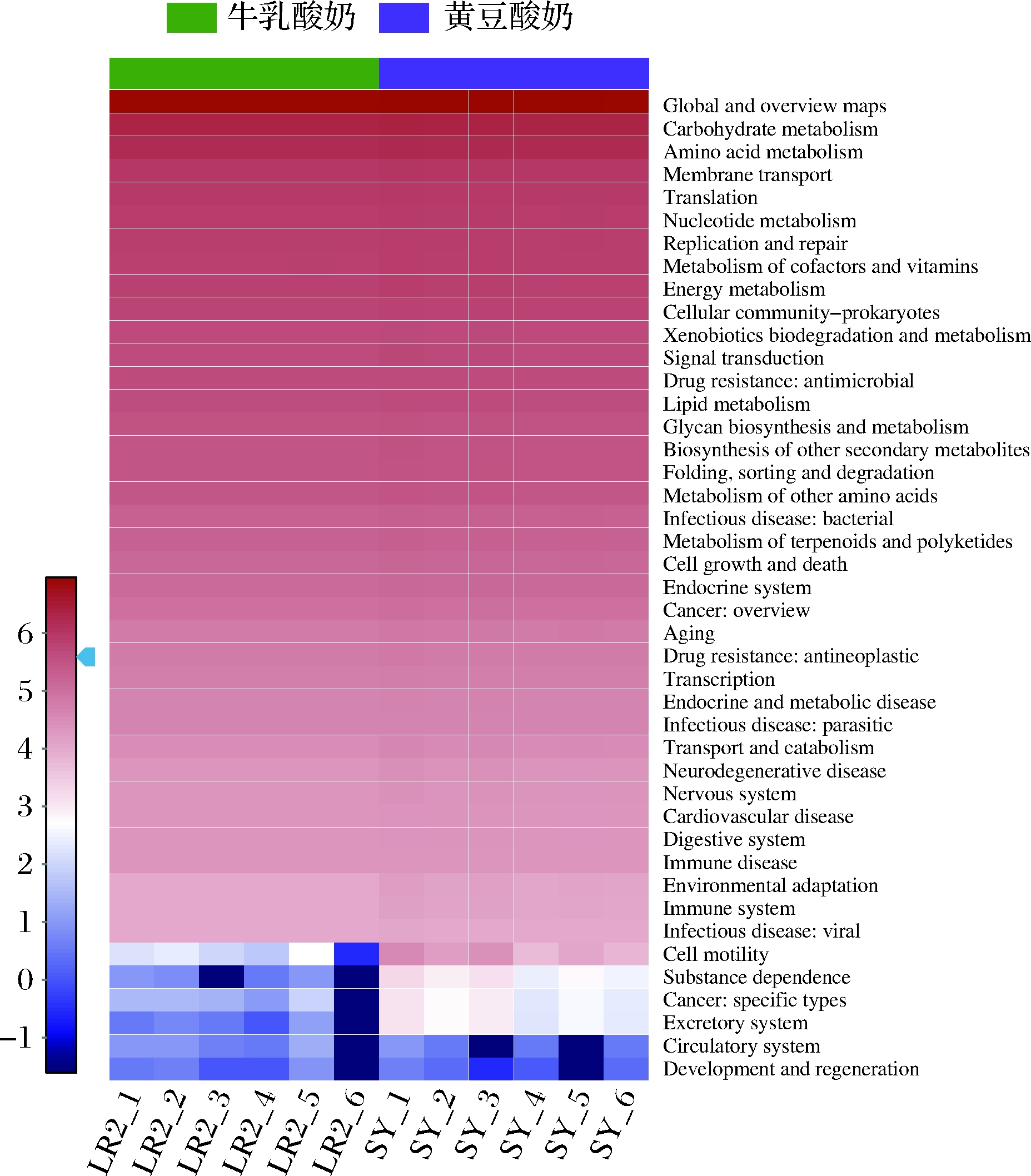
图10 KEGG功能热图
Fig.10 The heat map of KEGG functions
菌群基因大多与碳水化合物代谢(9.93%)、氨基酸代谢(7.43%)、翻译功能(3.91%)以及能量转换(3.04%)有关,这与前人的研究结果具有相似性[19],表明在发酵酸奶细菌基因组中包含大量参与蛋白质、碳水化合物代谢相关的基因。黄豆、牛乳样本具体在以下4个功能中丰度变化显著不同:细胞运动(cell motility)、癌症:特定类型(cancer:specific types)、排泄系统(excretory system)、物质依赖(substance dependence),且在黄豆酸奶样本中丰度值均远大于在牛乳酸奶样本中,这与前人的研究结果有一定的关联性,即食用豆制品被证明可以降低患心血管疾病[20]、癌症(乳腺癌和前列腺癌)和骨质疏松症的风险,但不能明确地表明作用机制,应该进一步研究验证植物蛋白发酵酸奶为肠道菌群变化而带来的健康益处及具体作用机制。
3 结论
黄豆发酵酸奶细菌多样性显著高于牛乳发酵酸奶,黄豆发酵酸奶与牛乳发酵酸奶组间菌群结构及群落组成差异极其显著。对黄豆、牛乳样本菌群结构差异有显著影响的物种主要包括链球菌属和芽孢杆菌属,链球菌属在黄豆、牛乳样本中均为优势属。初步判定,有无添加植物代糖对纯黄豆发酵酸奶群落组成有一定的影响,可从此方面对其进行更深入的探究。KEGG pathway功能组成分析发现,在以下4个功能中样本丰度变化显著不同:细胞运动、癌症:特定类型、排泄系统、物质依赖,且从丰度值来看,黄豆样本中均远大于牛乳样本,此发现可为后续对植物蛋白基因功能等方面的深入探究提供参考。因此,本研究通过对纯黄豆、牛乳发酵酸奶中细菌多样性进行分析,为探究植物蛋白与动物蛋白发酵乳品中细菌多样性之间的联系提供借鉴。植物基发酵酸奶有很大潜力成为一种新型的功能性食品,然而随着消费者偏好的变化和发酵过程的进步,需要对植物蛋白乳制品及其他发酵食品中的微生物资源进行更深层次研究,以优化植物蛋白酸奶发酵工艺条件以及菌种优良选育,改善人体肠道健康以及自然环境可持续发展。
[1] SETHI S, TYAGI S K, ANURAG R K.Plant-based milk alternatives an emerging segment of functional beverages:A review[J].Journal of Food Science and Technology, 2016, 53(9):3 408-3 423.
[2] WOODSIDE J V, BRENNAN S, CANTWELL M.Are soy-milk products viable alternatives to cow’s milk?[J].Springer International Publishing, 2016.DOI:10.1007/978-3-319-23672-810.
[3] 刘淑秋, 张楠, 朱文秀, 等.柚子汁豆奶粉发酵型植物酸奶的研制[J].中国果菜, 2021, 41(7):34-41.
LIU S Q, ZHANG N, ZHU W X, et al.Preparation of plants yogurt fermented by soybean milk powder and pomelo juice[J].China Fruit and Vegetable, 2021, 41(7):34-41.
[4] CHEN K N, ERH M H, SU N W, et al.Soyfoods and soybean products:From traditional use to modern applications[J].Applied Microbiology and Biotechnology, 2012, 96(1):9-22.
[5] 李薇, 吴良如, 索化夷, 等.基于高通量测序方法研究腌制麻竹笋发酵过程中细菌群落的动态演替[J].食品与发酵工业, 2020, 46(24):9-15.
LI W,WU L R,SUO H Y, et al.Bacterial community dynamic succession during fermentation of pickled Ma bamboo shoots based on high-throughput sequencing[J].Food and Fermentation Industries, 2020, 46(24):9-15.
[6] 武亚婷, 杜木英, 何欢欢, 等.基于高通量测序技术分析新疆不同地区自然发酵辣椒酱微生物群落多样性[J].食品与发酵工业, 2019, 45(21):221-228.
WU Y T, DU M Y, HE H H, et al.Microbial diversity analysis of natural fermented Chili Sauce from different regions in Xinjiang by high-throughput sequencing[J].Food and Fermentation Industries, 2019, 45(21):221-228.
[7] 刘怡萱, 许国琪, 曹鹏熙, 等.基于16S rRNA高通量测序的西藏农、牧区牦牛酸奶菌群多样性分析[J].食品科学, 2020, 41(18):92-97.
LIU Y X, XU G Q, CAO P X, et al.Microbial diversity of yak yogurt in agricultural and pastoral areas of Tibet analyzed based on high-throughput 16S rRNA gene sequencing[J].Food Science, 2020, 41(18):92-97.
[8] ALI N, WANG S R, ZHAO J, et al.Using a high-throughput sequencing technology to evaluate the various forage source epiphytic microbiota and their effect on fermentation quality and microbial diversity of Napier grass[J].Archives of Microbiology, 2021, 203(8):4 879-4 890.
[9] 戈子龙, 张泽金, 周爱梅, 等.基于高通量测序与培养方法分析新鲜佛手与老香黄中的细菌多样性[J].食品与发酵工业, 2020, 46(3):250-256.
GE Z L, ZHANG Z J, ZHOU A M, et al.Analysis of bacteria diversity in fresh fingered citron and Laoxianghuang based on high throughput sequencing and culture methodology[J].Food and Fermentation Industries, 2020, 46(3):250-256.
[10] CHEN S F, ZHOU Y Q, CHEN Y R, et al.Fastp:an ultra-fast all-in-one FASTQ preprocessor[J].Bioinformatics, 2018, 34(17):i884-i890.
[11] MAGO T, SALZBERG S L.FLASH:fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics, 2011, 27(21):2 957-2 963.
T, SALZBERG S L.FLASH:fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics, 2011, 27(21):2 957-2 963.
[12] STACKEBRANDT E, GOEBEL B M.Taxonomic note:A place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J].International Journal of Systematic and Evolutionary Microbiology, 1994, 44(4):846-849.
[13] EDGAR R C.UPARSE:Highly accurate OTU sequences from microbial amplicon reads[J].Nature Methods, 2013, 10(10):996-998.
[14] WANG Q, GARRITY G M, TIEDJE J M, et al.Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J].Applied and Environmental Microbiology, 2007, 73(16):5 261-5 267.
[15] ROGOSA M, MITCHELL J A, WISEMAN R F.A selective medium for the isolation and enumeration of oral and fecal lactobacilli[J].Journal of Bacteriology, 1951, 62(1):132-133.
[16] 张敏, 张艳, 黄丽丽, 等.基于16S rDNA高通量测序方法比较新疆西北部地区乳品中微生物的多样性[J].食品科学,2017,38(20):27-33.
ZHANG M, ZHANG Y, HUANG L L, et al.Application of 16S rDNA high-throughput sequencing for comparative study of the microbial diversity of dairy products from western and northern Xinjiang, China[J].Food Science, 2017, 38(20):27-33.
[17] 常嘉乐, 张婷, 袁亚宏, 等.藜麦酸奶混菌发酵工艺优化及品质与风味评价[J].食品工业科技, 2021, 42(18):197-208.
CHANG J L, ZHANG T, YUAN Y H, et al.Optimization of fermentation process and evaluation of quality and flavor of quinoa yogurt produced by mixed lactic acid bacteria[J].Science and Technology of Food Industry, 2021, 42(18):197-208.
[18] PRAGYA P, KAUR G, ALI S A, et al.High-resolution mass spectrometry-based global proteomic analysis of probiotic strains Lactobacillus fermentum NCDC 400 and RS2[J].Journal of Proteomics, 2017, 152:121-130.
[19] PORCELLATO D, SKEIE S B.Bacterial dynamics and functional analysis of microbial metagenomes during ripening of Dutch-type cheese[J].International Dairy Journal, 2016, 61:182-188.
[20] TORREJON C, JUNG U J, DECKELBAUM R J.N-3 Fatty acids and cardiovascular disease:Actions and molecular mechanisms[J].Prostaglandins, Leukotrienes and Essential Fatty Acids, 2007, 77(5-6):319-326.