肉类制品掺假造假现象颇为常见,在利益的驱使下,不法商贩们常用较低廉的鸭肉、鸡肉、猪肉等冒充牛肉、羊肉,掺入其肉类制品中,这不仅带来极大的食品安全隐患,危害人们的宗教信仰,而且还严重侵害了消费者的权益和食品的公平交易[1-2],因此,建立一种肉类及其制品的快速、精准检测方法,用于鉴别肉类的不同种属、及时发现掺假肉尤为重要。基于质谱的蛋白组学技术可以快速、精准的检测目标化合物,是目前在肉类掺假鉴别中最常用的检测方法之一。串联质谱的多反应监测(multiple reaction monitoring/selective reaction monitoring, MRM/SRM)定量方法,一直以来就是定量的“黄金标准”,实验的关键是选择出靶蛋白的典型肽段,要求肽段含有8~25个氨基酸、相对分子质量适于四极杆扫描的质核比范围、离子化效率高,稳定好、不易发生修饰、尽量不含有蛋氨酸(易发生氧化反应)和半胱氨酸(易发生脲甲基化和氧化反应)等残基以减少可能的误差[3]。同时,挑选的肽段应是唯一对应其蛋白质,以提高检测的特异性和减少对结果的干扰,由于生物样本的复杂性,每一个肽段往往需要选择3个离子对(母离子-子离子)所产生的信号。
四级杆-高分辨串联质谱技术(如四极杆飞行时间质谱仪、四极杆轨道阱质谱仪)是利用四极杆质量过滤器对母离子的高选择性和高特异性、高分辨率的质量分析器的准确质量检测能力,结合质量过滤器和质量分析器的优势实现准确鉴别与高通量筛查,且该技术的发展和扫描速度的提升为目标蛋白或肽段的定量测定也提供了新的途径。平行反应监测(parallel reaction monitoring,PRM)正是相对于传统SRM建立起来的高分辨离子监测技术[4-5],基于以四极杆静电场轨道阱(quadrupole orbitrap,Q-Orbitrap)为代表的四极杆-高分辨质谱平台,与SRM每次扫描一个离子对不同,PRM模式每次对一个母离子产生的所有离子对进行全扫描,即平行监测了一个母离子对应的所有离子对,监测精度高、无需事先确定离子对和优化碰撞能量、线性范围广、靶向定量灵敏度和通量更高,去除基质干扰能力强[6-7]。基于高分辨、高精度质谱的离子监视技术,能够对目标蛋白质、目标肽段(如发生翻译后修饰的肽段)进行选择性检测,从而实现对复杂样本中的目标蛋白质/肽段进行准确地特异性分析和绝对定量。本文采用超高效液相色谱-高分辨质谱法对肉类特征肽的检测方法进行探究,比较四极杆静电场轨道阱高分辨质谱的PRM模式和靶标单一离子监测/数据依赖扫描(targeted single ion monitoring/data-dependent MS/MS scans,tSIM/ddMS2)采集模式定量的差异,以期优化出各肉类特征肽测定的条件,为更加快速、精准的鉴别肉类及肉类制品的蛋白来源提供有力的技术支撑。
1 材料与方法
1.1 材料与试剂
质谱级乙腈、甲醇、甲酸(formic acid,FA)等,默克股份有限公司;正己烷(色谱级)、牛胰蛋白酶(Type I, 活力10 000 BAEE/mg蛋白)、二硫苏糖醇(dithiothreitol, DTT,≥99.5%),西格玛有限公司;硫脲(≥99%)、CaCl2·H2O(≥99%)、尿素、NH4HCO3、三羟甲基氨基甲烷(Tris≥99%)、碘代乙酰胺(iodoacetamide, IAA)等,生工生物工程(上海)股份有限公司;合成肽(SALAHAVQSSR、LADNLDTLGAAAAK、NALAHALQSAR、ALEDQLSELK、YTPVEAIEK、AAIAQAGYTDK、FISLLDELQK、TLAFLFAER),上海强耀生物科技有限公司;固相萃取柱(HLB柱、3 mL/200 mg),Waters公司;超纯水(电阻率≥18.2 MΩ·cm),实验室自制;牛肉及牛肉丸,市售。
1.2 仪器与设备
Vanquish Flex Binary超高效液相色谱、EASY-nLC 1200纳升级液相色谱、Q Extractive四极杆静电场轨道阱高分辨质谱仪,Thermo公司;ZX4漩涡振荡器,意大利VELP公司;ME204E/02电子天平,梅特勒-托利多仪器(上海)有限公司;Milli-Q超纯水机,密理博有限公司;2-16P高速离心机,西格玛有限公司;移液枪,赛默飞世尔科技(中国)有限公司;HH-30恒温水浴锅,江苏科析仪器有限公司;As系列超声波清洗机,天津奥特赛恩斯仪器有限公司。
1.3 实验方法
1.3.1 蛋白提取与酶解
分别称取0.5 g搅碎的原料肉及肉制品置于10 mL离心管中,加入2.5 mL正己烷,超声波处理3 min,涡旋5 min,4 500 r/min离心3 min,保留沉淀物。重复以上步骤2次,在N2的环境下使沉淀物干燥。加入4 mL提取液(6 mol/L尿素、2 mol/L硫脲、50 mmol/L Tris-HCl),旋涡混匀5 min,超声波处理3 min,4 ℃、12 000 r/min离心4 min,收集上清液,转移到10 mL离心管中并涡旋1 min。随后,将提取物在室温下1 315×g离心3 min,以消除先前步骤中可能产生的泡沫[8]。该上清液即为肉蛋白原样液,采用考马斯亮蓝染色法测定蛋白质的含量,4 ℃下保存。取0.1 mL肉蛋白提取液与0.9 mL水混合,涡旋30 s,取200 μL混合溶液加入装有150 μL NH4HCO3(0.5 mol/L)的2 mL离心管中,涡旋30 s,加入10 μL DTT溶液(0.5 mol/L),混匀后水浴处理(75 ℃,30 min),以使溶液中的蛋白质变性,提取物冷却至室温。添加150 mmol/L碘代乙酰胺使蛋白质烷基化,最终浓度为15 mmol/L,暗处反应30 min。随后,添加50 μL牛胰蛋白酶溶液(0.5 mg/L,用30 mmol/L NH4HCO3溶液配制)和10 μL CaCl2溶液(0.1 mol/L)。立即将酶解液涡旋1 min,37 ℃水浴酶解12 h。向酶解液中加入20 μL甲酸终止反应,混匀后置于室温中15 min,加水定容至1 mL。
1.3.2 脱盐
用HLB固相萃取柱进行脱盐处理,先用3 mL甲醇洗涤和活化柱,然后用3 mL体积分数1% FA平衡[9]。取酶解液1 mL过柱,并用0.5 mL体积分数 1%的甲酸冲洗空的离心管,将洗涤液装入小柱,以定量转移样品。然后用2 mL体积分数 1%的甲酸水洗涤小柱。最后,将肽用1 mL V(乙腈)∶V(0.1%甲酸水)=1∶1洗脱到10 mL的离心管中,加入洗脱液后将小柱浸泡10 min,使肽能完全洗脱。再加1 mL混合洗脱液重复洗脱1次。随后,将全部洗脱液在N2流中吹干,色谱分析前将提取物用0.5 mL的V(乙腈)∶V(0.1%甲酸水)=3∶97复溶,涡旋30 s,过0.22 μm有机系滤膜,上机检测。
1.3.3 纳流液相色谱-高分辨质谱法分析筛选特征肽
将肉蛋白酶解复溶液以80 MPa的压力直接上样到PepMap 100 C18分析柱上(75 μm×250 mm,2 μm,美国Thermo公司),再用300 nL/min的流速进行洗脱,流动相A为0.1%(体积分数)甲酸水溶液,流动相B为80%体积分数乙腈溶液(含0.1%甲酸),梯度洗脱程序:0~2 min,2% B;2~40 min,2%~32% B;40~45 min,32%~37% B;45~50 min,37%~100% B;50~55 min,100% B。离子源为nano-Flex纳喷雾离子源,正离子扫描模式,毛细管电压2.0 kV,离子传输管温度320 ℃,RF-lens为50%。数据依赖性质谱(data-dependent acquisition,DDA)采集模式,一级质谱分辨率70 000@m/z 200,自动增益目标值(automatic gain control,AGC)值为1e6,最大注入时间(maximum injection time,MIT)为100 ms,扫描范围为m/z 300~1 500。二级谱图分辨率设为17 500@m/z 200,最大注入时间为80 ms,扫描范围为m/z 150~2 000,隔离窗口设为2.0 Da,碎裂能量设为28,碎裂模式为HCD,AGC值为1e5,采集top N设为20,其余参数按照默认设置。
1.3.4 超高效液相色谱-高分辨质谱法测定特征肽的条件
分别取特征肽标准液或样品溶液涡旋30 s、过0.22 μm滤膜,置于样品瓶中,采用超高效液相色谱-四极杆静电场轨道阱高分辨质谱进行DDA采集模式检测。色谱条件:色谱柱:Hypersil GOLD C18选择性柱(2.1 mm×100 mm,3.0 μm,Thermo公司),柱温40 ℃,进样室温度10 ℃,流速0.3 mL/min;进样量5 μL;洗针进样,流动相A为0.1%甲酸水溶液,流动相B为0.1%甲酸乙腈;梯度洗脱程序为:0~1 min,5% B;1~20 min,5% B~30% B;20~25 min,30% B~80% B;25~28 min,80% B;28~28.1 min,80% B~5% B;28.1~30 min,5% B。质谱条件:正离子(ESI+)模式,鞘气流速30 arb,辅助气流速10 arb,毛细管电压3.8 kV,离子源温度320 ℃,S-lens RF 50%,辅助气温度350 ℃。一级母离子扫描范围m/z 300~2 000,二级碎片离子扫描范围m/z 50~2 000,前1 min和25 min后的流动相进废液,离子列表为376.203 63+、576.317 62+、554.785 32+、384.547 53+、525.279 32+、672.361 92+、573.306 12+、534.297 82+、603.342 42+、563.801 82+;一级全扫描质谱采集参数:分辨率70 000@m/z 200,AGC值为1e6,MIT为100 ms,分离窗口2.0 m/z;tSIM/ddMS2定量采集方法:一级质谱采集参数同前,二级质谱采集参数:分辨率35 000@m/z 200,AGC值为2e5,MIT为80 ms,环数为15,分离窗口2.0 m/z,各肽段碎片离子和碰撞能采用优化后的值,肽段匹配优选,动态排除15.0 s,其他参数采用默认值;PRM定量采集方法:分辨率35 000@m/z 200,AGC值为2e5,MIT为80 ms,环数为8,分离窗口2.0 m/z,各肽段碎片离子和碰撞能采用归一化能量(normalized collision energy, NCE)值28,预设时间(time-scheduled)窗口为2.0 min。
1.3.5 方法学验证
本研究对超高效液相色谱-高分辨质谱法测定特征肽的线性范围、检出限(limit of detection, LOD)、定量限(limit of quantification, LOQ)、基质效应、加标回收率、精密度等方法学参数进行分析。加标回收率采用在牛肉丸基质中添加低(4 μg/g)、中(20 μg/g)、高(100 μg/g)3个浓度的混合肽标准溶液,按照1.3.1和1.3.2方法酶解和过柱脱盐后进行检测分析,计算回收率和准确度。分别按照3倍信噪比(S/N=3)和10倍信噪比(S/N=10)为依据,计算该方法测定各种特征肽的LOD和LOQ。精密度实验分别在不同时间重复测定100 μg/L的特征肽标准溶液的含量,以相对标准偏差(relative standard deviation, RSD)表示。移取适量的特征肽混合标准溶液,按质量比加入到按1.3.1和1.3.2处理的肉丸蛋白酶解液中,配制成一定浓度梯度的混合工作液,涡旋30 s、过0.22 μm滤膜,按照1.3.4方法测定,绘制各特征肽的标准曲线,按照公式(1)计算基质效应:
基质效应![]()
(1)
式中:KS和KM,分别为纯溶剂和肉丸蛋白酶解液基质配制的特征肽工作液标准曲线的斜率。
1.4 数据检索及处理
质谱数据采集用Xcalibur 4.2.47软件(Thermo Scientific),肽段DDA数据使用Proteome Discoverer 2.4(Thermo Scientific)和MaxQuant软件进行搜库鉴定,检索UniProt_Bovine(包含876 759条蛋白序列)、_Chicken(包含709 277条蛋白序列)、_Pig(包含709 277条蛋白序列)、_Duck(包含842 876条蛋白序列)数据库,一级质谱中母离子质量误差为10×10-6,二级质谱中碎片离子质量精度:0.02 Da,酶为胰蛋白酶(trypsin),完全酶切,漏切数目为0个,固定修饰为半胱氨酸的烷基化(C,+57.021 50 Da),可变修饰为甲硫氨酸的氧化(M,+15.994 92 Da),鉴定结果作为谱图库进行特征肽段的挑选与鉴别,通过UniProt数据库中的BLAST算法验证其种属特异性。特征肽参数优化和谱图库离子挑选、定性确证等分析使用Skyline 20.1软件,参数设置:截止得分0.99;添加修饰:半胱氨酸碘乙酰化(carbamidomethyl)、氧化(oxidation);配置离子对设置:母离子电荷:2+、3+、4+,子离子电荷:1+、2+,离子类型:y、b、p;串联质谱筛选参数设置:采集方法:targeted,子离子质量分析仪:Orbitrap,分辨率:35000@200;导入fasta文件。tSIM/ddMS2和PRM数据使用Processing模板建立处理文件,导入Xcalibur采集序列表进行批处理,将处理后的序列文件用Quan Browser插件打开查看质谱图、标准曲线等。
2 结果与分析
2.1 特征肽的筛选
纳升级高效液相色谱-高分辨质谱能灵敏快速的鉴定多肽,结合数据库搜索,短时间内可获得大分子蛋白质的序列数据。采用高分辨率、高扫描速度的质谱对牛肉、猪肉、鸡肉和鸭肉等4个物种肉中的蛋白质进行了分析鉴定,极大丰富了分析结果中的蛋白质和多肽的信息,同时,也增加了质谱测定结果中的同源蛋白质和肽生物标志物的数量。图1展示的是牛属肌球蛋白-1来源的特征肽ALEDQLSELK(理论m/z 573.306 12+)和肌红蛋白来源的特征肽HPSDFGADAQAAMSK(理论m/z 766.843 52+)的母离子色谱质谱提取峰及其二级碎片离子质谱图,由于胰蛋白酶是在精氨酸(R)和赖氨酸(K)的羧基端将肽切断,大部分多肽段的母离子带有2个或3个正电荷,在低能碰撞诱导裂解产生的碎片离子主要是b-离子和y-离子,前者含有氨基末端、后者含有羧基末端,通常Q、R、K残基会丢失氨基(-17),S、T、E残基等会丢失水分子(-18),如特征肽ALEDQLSELK中y1(K-)和y6(-Q-L-S-E-L-K)分别脱氨形成m/z 130.086 6和m/z 700.387 0的碎片离子峰,特征肽ALEDQLSELK和HPSDFGADAQAAMSK中碎片离子y3(-E-L-K)和b8(-H-P-S-D-F-G-A-D-)失去水分子形成m/z 371.228 9和m/z 809.315 9的碎片离子峰。碎片离子的实测m/z与理论m/z偏差均在10×10-6范围以内的水平,谱图质量非常高,结果非常可信,可用于鉴别分析。
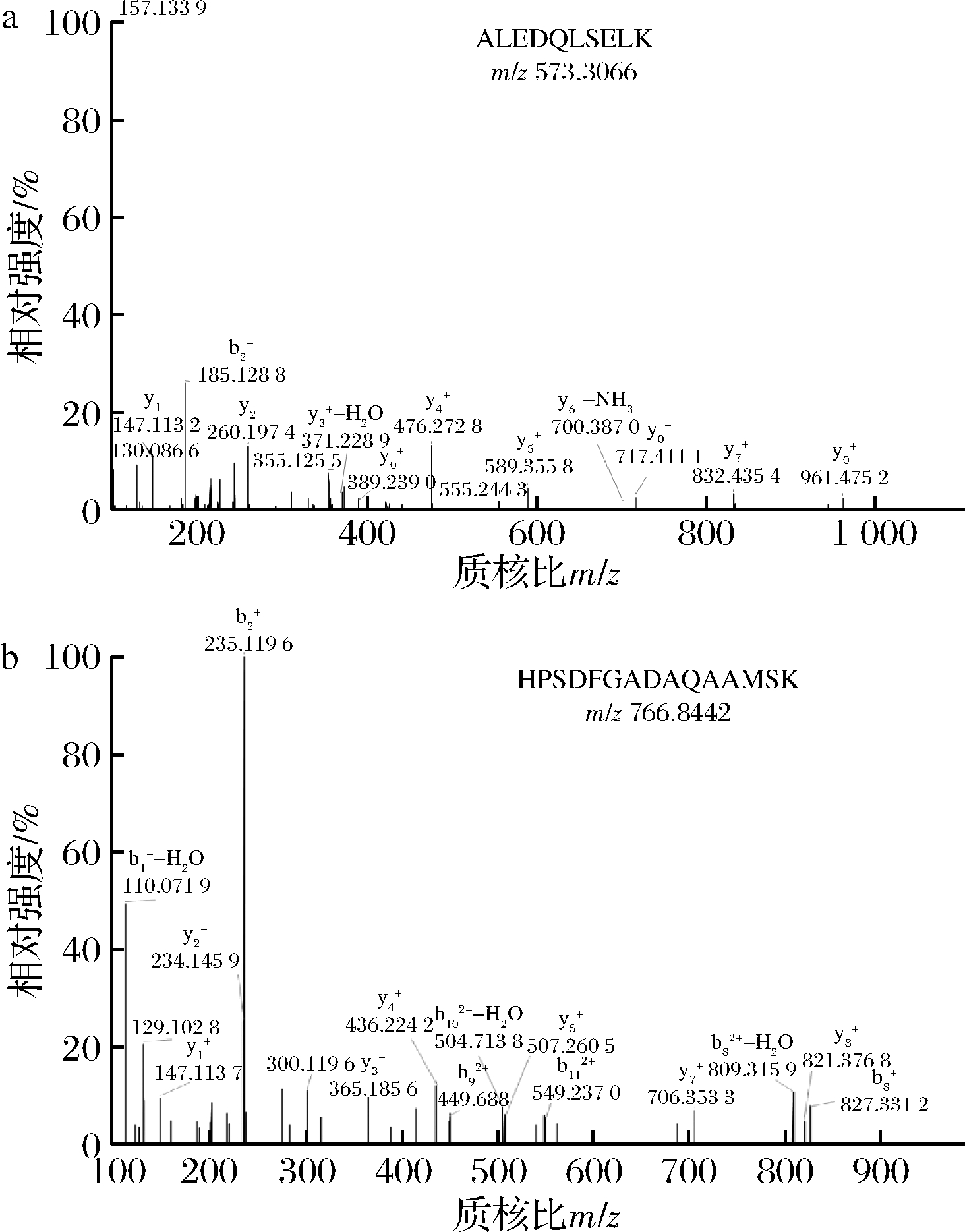
图1 特征肽ALEDQLSELK(a)和HPSDFGADAQAAMSK(b)
的二级碎片离子图
Fig.1 Fragment ion spectra of marker peptide
ALEDQLSELK (a) and HPSDFGADAQAAMSK (b)
根据测定到的y系列和b系列离子的匹配情况鉴别备选特征肽段,借助UniProt数据库的BLAST算法[10-11]可鉴定出特征肽的数量如下:鸡属19条、牛属15条、鸭属16条、猪属12条,而用于定量测定分析的特征肽选择常遵循以下原则[12]:含8~18个氨基酸、不易发生修饰而影响定量、易被质谱检测、色谱保留性适当、碎片离子响应较强、氨基酸序列中不含C或M,从中分别选择8条特征肽作为各种属肉类的鉴别,具体信息如表1所示。
2.2 色谱参数的优化
肽段氨基酸序列构成会影响其溶解性、亲水疏水性,从而影响其色谱保留行为和分离效果。极性较小的肽段通常需要更高比例的有机相才能洗脱出,部分性质相似的肽段需要更长时间的洗脱才能分离,但加长洗脱梯度又不可避免地会导致色谱峰变宽以及母离子信噪比降低。同时,较低的流速可以延长目标物的保留时间,但是过低的流速将引起峰展宽和拖尾,如图2所示,亲水性或疏水性较强的特征肽比较容易洗脱,且峰型较好,而特征肽1和8的拖尾现象非常明显,这与2种特征肽的平均疏水性值(表1)接近于0的结果一致,因此应当考虑提高流速以获得理想的梯度洗脱分离效果和良好的重现性,实验表明0.3 mL/min的流速非常适合分离特征肽与兼容质谱分析。采用同样的二元流动相进行梯度洗脱优化,根据前期研究[8]分别探讨了梯度洗脱时间25、30、42 min等对特征肽分离效果的影响,25 min的梯度洗脱效果较差,部分特征肽不能完全分开;30 min梯度洗脱能实现特征肽较好地分离,且减少了溶剂峰的干扰;42 min梯度洗脱时特征肽的分离度最好,但分析所需时间较长,溶剂消耗量较多;30 min梯度洗脱既可降低分析所需时间和溶剂消耗量,又能够达到快速、高效分离的要求,适合于质谱检测,优化后的梯度洗脱条件如方法1.3.4所示。

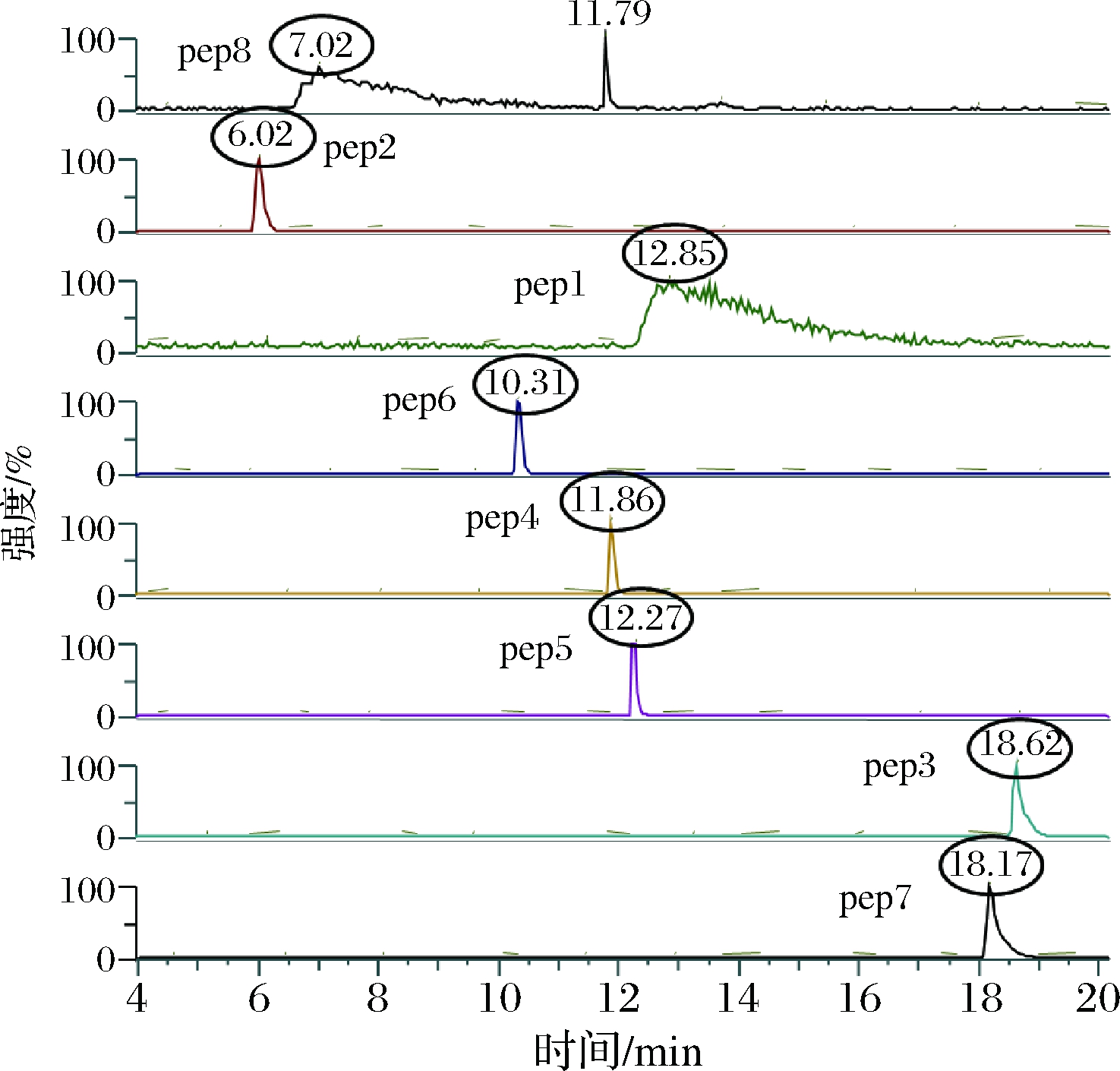
图2 低流速(0.2 mL/min)洗脱对色谱峰形状的影响
Fig.2 Effect of low flow rate (0.2 mL/min) elution on
shape of chromatographic peaks
2.3 质谱参数的优化
碰撞能量是影响质谱信号和测定结果的重要因素,过低的碰撞能量,母离子被轰击打碎的分子碎片较少,不利于目标物的鉴别;而过高的碰撞能量母离子被过度打碎,分子碎片增多且碎片离子较小,也不利于鉴别和定量。此外,较高碰撞能下二级定量碎片离子的信号强度较高,无其他碎片离子干扰、可提高定量分析的准确度。将选择的特征肽段导入Skyline 20.1软件,利用该软件也可自动优化出四极杆静电场轨道阱高分辨质谱平台上系列离子对、碰撞能量等参数以建立定量方法,软件优化的碰撞能如表2所示。为简化实验程序可直接采用Skyline软件优化的碰撞能进行测定,tSIM质谱采集模式测定结果也表明,用自动优化的碰撞能(collision energy, CE)值所测得的母离子和碎片离子的信号强度无显著差异(P>0.05)。同时,采用该高分辨质谱法可解决传统三重四极杆质谱仪中需要优化母离子与子离子对进行定量的问题,高分辨质谱仪可记录目标母离子的全部碎片离子图谱,且全是高分辨、高质量精度的子离子,这为准确鉴别干扰离子排除假阳性提供了非常好的帮助。PRM模式中无需优化碰撞能量,肽段测定中常用NCE值为28,其中NCE是一个无量纲数值,大致相当于质量数为500、电荷数为1的参比离子的碰撞能(单位eV),实际使用的能量是根据所选母离子的质量和电荷数实时自动计算的,这进一步简化了方法开发所需的时间。PRM模式中目标肽段预设时间窗口大小也是影响检测的主要参数,预设时间窗口大小直接受目标肽段保留时间偏移的影响,窗口设定太小,目标物保留时间出现偏移检测不出;窗口设定太大,则肽段检测比例及扫描点数明显减少[12]。较短的色谱梯度下肽段保留时间的偏移相对更低,且色谱表现更稳定,优化后的参数见实验方法部分。2种采集模式下,8条特征肽段的色谱-质谱图如图3所示,色谱峰形尖锐,分离效果好,可用于定量分析特征肽的含量。

a-tSIM/ddMS2;b-PRM
图3 不同质谱采集模式下特征肽段的色谱-质谱图
Fig.3 Chromatography-mass spectrometry of marker
peptides in different acquisition modes
2.4 方法学考察
将配制好的合成肽混合标准系列样品液按1.3的色谱-质谱条件进行超高效液相色谱-高分辨质谱测定,记录各特征肽出峰时间和母离子及碎片离子的峰面积,以各特征肽的峰面积为纵坐标,标准溶液浓度为横坐标绘制标准曲线,结果如表2所示。结果表明,8条特征肽在2种质谱采集模式(tSIM/ddMS2和PRM)下具有良好的线性关系,线性相关系数均>0.99,2种质谱采集模式下的相关性差异不大(P>0.05),2种采集模式下的检出限分别为0.04~0.19 μg/L、0.01~0.16 μg/L,PRM模式采用二级碎片离子加和方式表示出峰情况,其检测限更低。

由表3可知,牛肉丸样品提取液不影响特征肽pep2、pep5、pep6、pep7、pep8的测定,即不存在基质效应,而对特征肽pep1、pep3、pep4产生一定的抑制效应。各特征肽的加标回收率高,该回收率与多肽平均疏水性的相关系数为0.982,这主要是由于HLB为反相亲水性的改性苯乙烯聚合物,对蛋白质、多肽、氨基酸等具有较好的保留,表1中多肽平均疏水值<0表示多肽为疏水性,疏水值>0表示多肽为亲水性,pep5和pep6的疏水性较强,其保留效果较差、回收率偏低,应用中可根据多肽的平均疏水性值选择合适的固相萃取柱和洗脱溶液。
2.5 样品测定
为了验证所建立分析方法的适用性,对市场上购买的不同价位的12种牛肉丸样品进行检测,结果如表4所示。本次抽样筛查的牛肉丸样品中共检测出5条不同种属的特征肽,其种属来源分别为牛肉2条、猪肉1条、鸡肉1条、牛肉和鸡肉共有1条,在检测的样品中不含其他种属肉类的比例为41.7%,牛肉丸样品中标明混有其他肉类的比例为42.8%,这表明市售的牛肉丸中部分产品混有其他种类的肉,其混入量不等,且有的产品中猪肉混入的量非常高。
如图4所示,2种质谱采集模式下均测出强信号的猪肉源特征肽。

a-PRM;b-tSIM/ddMS2
图4 两种采集模式测定的样品2中特征肽的色谱-质谱图
Fig.4 Chromatography-mass spectrometry of marker
peptides in sample 2 determined by two acquisition modes
鸭肉中的1条特征肽(pep4)在部分样品的色谱-质谱图中也出峰,如图5-a所示,但与特征肽pep4的二级质谱(图5-b)对比可以看出碎片离子不同,出峰的应为干扰杂质,即鸭肉的2条特征肽均未测得,这表明牛肉丸样品中未混入鸭肉。从测定结果可以看出,本方法可以利用高分辨质谱的二级图谱有效排除假阳性结果,抽检的样品中S2和S7的测定结果与标识一致,这表明所建方法可靠,可用于肉及肉制品中蛋白种属来源的鉴别和测定,比已报道[13-14]的三重四极杆质谱法的鉴定能力更强。

a-干扰物的二级质谱图;b-特征肽pep4的二级质谱图
图5 样品8中干扰物与特征肽pep4的质谱图对比
Fig.5 Comparison of mass spectrograms of interfering
substances in the sample 8 with pep4
基于超高效液相色谱-四极杆静电场轨道阱高分辨质谱的tSIM/ddMS2和PRM两种采集模式建立的定量测定8条不同种属来源的肉类特征肽的方法检出限分别为9.84~33.13 μg/kg、2.46~27.90 μg/kg,远优于采用纳升级液相色谱-四极杆飞行时间质谱的数据依赖型采集模式测定5条特征肽的方法[15],且本文建立的方法重现性和测定所需时间也优于纳升级液相色谱法,这与文献[16]报道的鉴别出的蛋白数量少而够用的情况下采用超高效液相色谱分析以提高其重现性的结果一致。同时,基于高分辨质谱法的灵敏度远低于三重四极杆质谱法的灵敏度[13,17],这主要是由于三重四极杆质谱在靶向定量方面具有非常出色的检测能力,但在鉴别复杂基质中共洗脱的质核比相近的肽段方面的分辨率较低而出现假阳性结果[18],而采用四极杆静电场轨道阱高分辨质谱的tSIM/ddMS2和PRM两种采集模式可借助二级图谱的精确质量数进行鉴别有效排除假阳性结果。随着高分辨质谱技术的进步和设备升级、检测能力增强,特别是近年来报道的四极杆静电场轨道阱高分辨质谱的PRM模式在定量能力方面可以“媲美”高端三重四极杆[19],使其在定量检测领域得到广泛的应用,因而本文建立的采用tSIM/ddMS2模式的一级质谱图和PRM模式的二级质谱图定量测定特征肽的方法可为准确快速地鉴别肉制品中蛋白质种属来源和肉类掺假情况提供强有力的支撑。
3 结论
纳升级高效液相色谱-高分辨质谱法筛选出的8条特征肽碎片离子的实测质核比与理论值偏差小,谱图质量高,结果可信,可用于肉种属鉴别和分析。牛肉丸样品中蛋白质变性程度大,需用强提取缓冲液经超声辅助法提取,胰蛋白酶酶解后经固相柱净化去除基质中的盐类等干扰物,超高效液相色谱梯度洗脱分离,采用四极杆静电场轨道阱高分辨质谱的PRM和tSIM/ddMS2采集模式定量测定,较低的色谱流速对平均疏水性值接近0的2条特征肽洗脱难、拖尾严重,梯度洗脱流速增加至0.3 mL/min可有效减少峰展宽与拖尾,且采用前1 min的液相洗脱液直接进废液的方式可有效降低盐分对质谱仪的损害和目标肽段离子化的抑制效应。碰撞能是影响质谱测定结果的重要因素,PRM采集模式不需优化碰撞能可直接采用NCE为28的值,tSIM/ddMS2采集模式可采用Skyline软件优化出的最优CE值,从而简化优化CE的过程。PRM模式在测定较多目标物时需预设时间窗口,以提高目标物检测的通量,常用时间窗口为1.5~2.0 min,以尽量避免出峰时间波动对目标物检测的影响和窗口太大肽段检测比例及扫描点数明显减少的影响。不同采集模式下各特征肽标准溶液质量浓度的线性关系良好(相关系数≥0.99),tSIM/ddMS2和PRM采集模式下的检出限分别为0.04~0.19 μg/L、0.01~0.16 μg/L,在牛肉丸提取液中的基质效应介于81.3%~114.7%,3个添加浓度下的加标回收率在89%~117%且相对标准偏差≤10%(n=5)。2种采集模式均可利用二级碎片离子鉴别目标物以提高检测的准确性,且PRM采集模式应用二级质谱的加和峰进行定量检测限更低,12批次牛肉丸样品检测中共检出含有猪肉源或鸡肉源特征肽的比例为58.3%,其中有明确标识的为42.8%,部分产品中检测出猪肉源特征肽的信号强度非常高。
[1] MEDINA S, PERESTRELO R, SILVA P, et al.Current trends and recent advances on food authenticity technologies and chemometric approaches[J].Trends in Food Science & Technology, 2019, 85:163-176.
[2] BÖHME K, CALO-MATA P, BARROS-VEL ZQUEZ J, et al.Recent applications of omics-based technologies to main topics in food authentication[J].TrAC Trends in Analytical Chemistry, 2019, 110:221-232.
ZQUEZ J, et al.Recent applications of omics-based technologies to main topics in food authentication[J].TrAC Trends in Analytical Chemistry, 2019, 110:221-232.
[3] GALLIEN S, DURIEZ E, DOMON B.Selected reaction monitoring applied to proteomics [J].Journal of Mass Spectrometry, 2011, 46(3):298-312.
[4] VIDOVA V, SPACIL Z.A review on mass spectrometry-based quantitative proteomics:Targeted and data independent acquisition[J].Analytica Chimica Acta, 2017, 964:7-23.
[5] 张伟. 定量蛋白质组学质谱采集技术进展[J].分析化学, 2014, 42(12):1 859-1 868.
ZHANG W.Progress in mass spectrometry acquisition approach for quantitative proteomics[J].Chinese Journal of Analytical Chemistry, 2014, 42(12):1 859-1 868.
[6] WU W, DAI R T, BENDIXEN E.Comparing SRM and SWATH methods for quantitation of bovine muscle proteomes [J].Journal of Agricultural and Food Chemistry, 2019, 67(5):1 608-1 618.
[7] BONNET M, SOULAT J, BONS J, et al.Quantification of biomarkers for beef meat qualities using a combination of Parallel Reaction Monitoring- and antibody-based proteomics[J].Food Chemistry, 2020, 317:126376.
[8] 王忠合, 李晓婷, 胡文梅, 等.超高效液相色谱-高分辨质谱法测定肉类特征肽[J].食品与发酵工业, 2021, 47(18):258-266.
WANG Z H, LI X T, HU W M, et al.Determination of peptide markers of meat species by ultra-high pressure liquid chromatography coupled with high resolution mass spectrometry[J].Food and Fermentation Industries, 2021, 47(18):258-266.
[9] 古淑青, 詹丽娜, 赵超敏, 等.基于液相色谱-串联质谱法的肉类特征肽段鉴别及掺假测定[J].色谱, 2018, 36(12):1 269-1 278.
GU S Q, ZHAN L N, ZHAO C M, et al.Identification of meat marker peptides and detection of adulteration by liquid chromatography-tandem mass spectrometry[J].Chinese Journal of Chromatography, 2018, 36(12):1 269-1 278.
[10] H FNER L, KALKHOF S, JIRA W.Authentication of nine poultry species using high-performance liquid chromatography-tandem mass spectrometry[J].Food Control, 2021, 122:107803.
FNER L, KALKHOF S, JIRA W.Authentication of nine poultry species using high-performance liquid chromatography-tandem mass spectrometry[J].Food Control, 2021, 122:107803.
[11] MONTOWSKA M, FORNAL E.Detection of peptide markers of soy, milk and egg white allergenic proteins in poultry products by LC-Q-TOF-MS/MS[J].LWT, 2018, 87:310-317.
[12] 李恺, 宋雷, 石文昊, 等.双反相色谱串联平行反应监测靶向定量蛋白质组方法的建立[J].生物工程学报, 2017, 33(11):1 859-1 868.
LI K, SONG L, SHI W H, et al.Establishment of dual reverse phase chromatography combined parallel reaction monitoring for targeted quantitative proteomics [J].Chinese Journal of Biotechnology, 2017, 33(11):1 859-1 868.
[13] HOFFMANN B, MÜNCH S, SCHW GELE F, et al.A sensitive HPLC-MS/MS screening method for the simultaneous detection of lupine, pea, and soy proteins in meat products[J].Food Control, 2017, 71:200-209.
GELE F, et al.A sensitive HPLC-MS/MS screening method for the simultaneous detection of lupine, pea, and soy proteins in meat products[J].Food Control, 2017, 71:200-209.
[14] NALAZEK-RUDNICKA K, K OSOWSKA-CHOMICZEWSKA I, WASIK A, et al.MRM-MS of marker peptides and their abundance as a tool for authentication of meat species and meat cuts in single-cut meat products[J].Food Chemistry, 2019, 283:367-374.
OSOWSKA-CHOMICZEWSKA I, WASIK A, et al.MRM-MS of marker peptides and their abundance as a tool for authentication of meat species and meat cuts in single-cut meat products[J].Food Chemistry, 2019, 283:367-374.
[15] GALLEGO M, MORA L, ARISTOY M C, et al.Optimisation of a simple and reliable label-free methodology for the relative quantitation of raw pork meat proteins[J].Food Chemistry, 2015, 182:74-80.
[16] ZHANG H W, ZHANG X M, ZHAO X, et al.Discrimination of dried sea cucumber (Apostichopus japonicus) products from different geographical origins by sequential windowed acquisition of all theoretical fragment ion mass spectra (SWATH-MS)-based proteomic analysis and chemometrics[J].Food Chemistry, 2019, 274:592-602.
[17] FORNAL E, MONTOWSKA M.Species-specific peptide-based liquid chromatography-mass spectrometry monitoring of three poultry species in processed meat products[J].Food Chemistry, 2019, 283:489-498.
[18] BR CKER J, BROCKMEYER J.Characterization and detection of food allergens using high-resolution mass spectrometry:Current status and future perspective [J].Journal of Agricultural and Food Chemistry, 2018, 66(34):8 935-8 940.
CKER J, BROCKMEYER J.Characterization and detection of food allergens using high-resolution mass spectrometry:Current status and future perspective [J].Journal of Agricultural and Food Chemistry, 2018, 66(34):8 935-8 940.
[19] ZHOU J T, LIU H Y, LIU Y, et al.Development and evaluation of a parallel reaction monitoring strategy for large-scale targeted metabolomics quantification[J].Analytical Chemistry, 2016, 88(8):4 478-4 486.