肌苷是一种嘌呤核苷,参与机体物质代谢和能量代谢,具有重要的应用价值。在医药领域,肌苷常用于肝病、心脏病等疾病治疗药物的生产,也用于异丙肌苷和利巴韦林等抗病毒药物的合成。在食品领域,肌苷是复合鲜味剂“I+G”的重要原料。此外,肌苷还被广泛应用在农业、化工和营养保健等诸多领域。目前,主流的肌苷生产方式为微生物发酵法,该方法利用廉价的原材料,通过生物反应器的扩大培养得到发酵液,再经过分离纯化得到肌苷产品,具有高效、稳定、成本低等优点。除此之外,酶解法与化学分解法也曾用来生产肌苷,但因其存在较多弊端而被淘汰。
肌苷生物合成途径包含3个部分(图1):首先,葡萄糖磷酸化后形成6-磷酸葡萄糖,后者经磷酸戊糖途径(pentose phosphate pathway,PPP)生成5-磷酸核糖,在5-磷酸核糖-1-焦磷酸(5-phosphoribosyl 1-pyrophosphate,PRPP)合酶(PRPP synthetase,Prs)的催化下继续转化为PRPP。接着,以PRPP、谷氨酰胺(Gln)、甘氨酸(Gly)、天冬氨酸(Asp)和一碳单位等为前体物,经10步酶促反应生成肌苷酸(inosine monophosphate,IMP)。最后,IMP在5′-核苷酸酶的催化下生成肌苷。合成途径冗长复杂,还受到严格的反馈调控,是微生物发酵法生产肌苷的限制因素之一。另外,肌苷的直接前体物IMP存在另外两条分支途径,与肌苷支路竞争碳代谢流,是限制肌苷合成的另一关键因素。常见的肌苷工程菌株也是主要针对以上两个方面进行构建。首先是增强肌苷合成途径及解除限速酶所受反馈调控。ASAHARA等[1]通过敲除枯草芽孢杆菌KMBS436的purR基因和嘌呤操纵子核糖开关,解除了嘌呤合成中的反馈阻遏和转录衰减;又通过优化嘌呤操纵子的启动子序列,增强了嘌呤基因的表达,肌苷产量从1.8 g/L提升至6 g/L。SHIMAOKA等[2]在大肠杆菌I-9m中过表达了purFK326Q,P410W和prsD128A突变基因,解除了限速酶反馈抑制,使肌苷产量提高了3.6倍。另外是阻断肌苷合成过程的竞争代谢途径。LI等[3]通过敲除枯草芽胞杆菌BS017中的purA基因来阻断腺苷支路,肌苷产量从150 mg/L提升至7.6 g/L。PEIFER等[4]通过敲除谷氨酸棒杆菌ATCC 13032中的purA和guaB基因,同时阻断了腺苷和鸟苷支路,使IMP产量增加了45倍。然而直接阻断腺苷或鸟苷支路均会导致菌株营养缺陷[5],菌株生长缓慢,不利于产物生产。因此,适当调节腺苷和鸟苷合成支路通量,平衡菌株生长和产物合成,是肌苷工程菌株构建的重点和难点。
在早期研究中,主要通过诱变枯草芽孢杆菌的方式来选育肌苷工程菌株,但诱变后的菌株普遍生产稳定性偏低,且易发生负向突变,不利于工业化生产[6]。随着系统生物学的发展,运用代谢工程理论和基因编辑技术对野生型菌株进行从头遗传改造成为肌苷工程菌株构建的主要方法。相比芽孢杆菌,大肠杆菌遗传背景清晰,基因操作简便,发酵周期较短,是发酵法生产肌苷另一个优良的宿主。然而,目前已有肌苷生产菌株的生产效率仍然偏低,且普遍存在携带质粒、营养缺陷等问题,增加了生产的成本,无法满足工业化生产的要求。此外,现有菌株的构建策略多局限在对限速反应、分支途径等关键节点进行改造,少有对肌苷代谢网络的系统优化。针对这些问题,本研究选择大肠杆菌标准株作为出发菌株,采用代谢工程技术,通过阻断肌苷降解途径、强化肌苷合成途径、弱化腺苷合成支路、增强前体物供应及修饰肌苷转运系统,获得了一株无质粒、非缺陷型且可稳定生产的肌苷工程菌株。

图1 肌苷生物合成途径和本研究的改造策略
Fig.1 Biosynthetic pathway of inosine and the strategies used in this study
注:*表示突变体
1 材料与方法
1.1 菌株、质粒及试剂
表1列举了本研究所用到的菌株及质粒。
表1 菌株和质粒
Table 1 Strains and plasmids
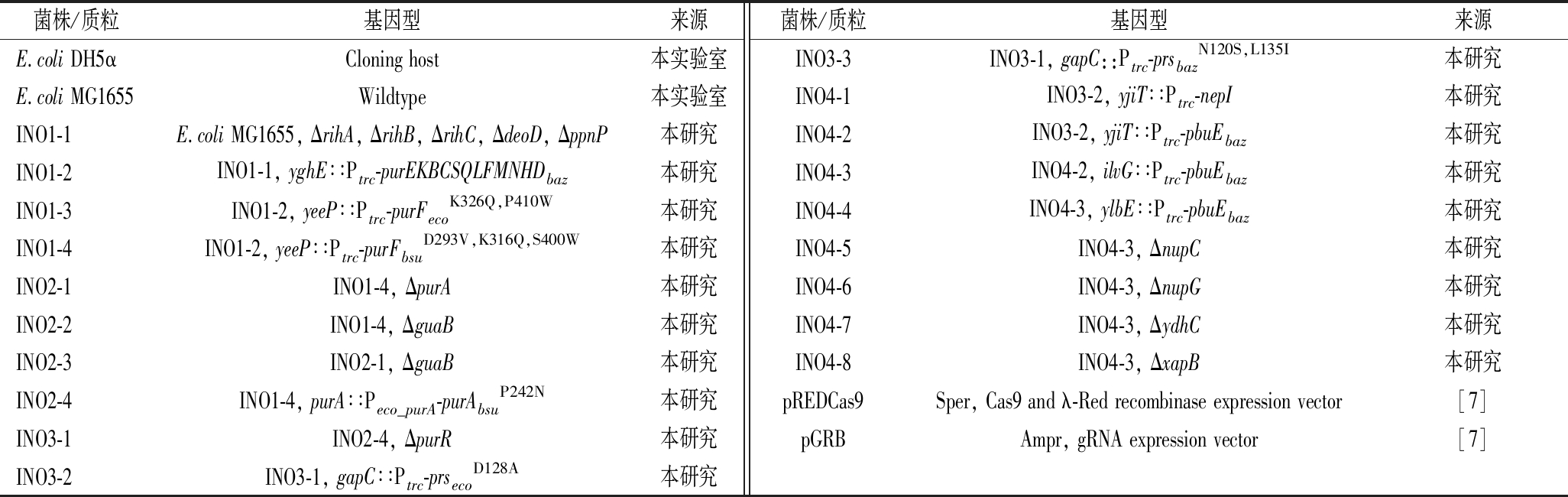
菌株/质粒基因型来源菌株/质粒基因型来源E.coli DH5αCloning host本实验室INO3-3INO3-1, gapC::Ptrc-prsbazN120S,L135I本研究E.coli MG1655 Wildtype本实验室INO4-1INO3-2, yjiT∶∶Ptrc-nepI本研究INO1-1E.coli MG1655, ΔrihA, ΔrihB, ΔrihC, ΔdeoD, ΔppnP本研究INO4-2INO3-2, yjiT∶∶Ptrc-pbuEbaz本研究INO1-2INO1-1, yghE∶∶Ptrc-purEKBCSQLFMNHDbaz本研究INO4-3INO4-2, ilvG∶∶Ptrc-pbuEbaz本研究INO1-3INO1-2, yeeP∶∶Ptrc-purFecoK326Q,P410W本研究INO4-4INO4-3, ylbE∶∶Ptrc-pbuEbaz本研究INO1-4INO1-2, yeeP∶∶Ptrc-purFbsuD293V,K316Q,S400W本研究INO4-5INO4-3, ΔnupC本研究INO2-1INO1-4, ΔpurA本研究INO4-6INO4-3, ΔnupG本研究INO2-2INO1-4, ΔguaB本研究INO4-7INO4-3, ΔydhC本研究INO2-3INO2-1, ΔguaB本研究INO4-8INO4-3, ΔxapB本研究INO2-4INO1-4, purA∶∶Peco_purA-purAbsuP242N本研究pREDCas9Sper, Cas9 and λ-Red recombinase expression vector[7]INO3-1INO2-4, ΔpurR本研究pGRBAmpr, gRNA expression vector[7]INO3-2INO3-1, gapC∶∶Ptrc-prsecoD128A本研究
引物,GENEWIZ公司;Primer STAR HS DNA、Ex taq DNA Polymerase,TaKaRa公司;2×Rapid Taq Mix、ClonExpress® Ⅱ One Step Cloning Kit,Vazyme公司;Plasmid Mini Kit I D6943、Gel & PCR Clean Up Kit D2000、Cycle Pure Kit D6492,Omega Bio-Tek公司。
1.2 引物
具体内容见附表1(https://kns.cnki.net/kcms/detail/11.1802.TS.20220509.1719.012.html)。
1.3 培养基
斜面培养基(g/L):葡萄糖5,蛋白胨10,酵母粉5,牛肉膏10,NaCl 5,琼脂20。
种子培养基(g/L):酵母粉5,KH2PO4·3H2O 1.2,蛋白胨3,MgSO4·7H2O 0.5,FeSO4·7H2O 0.01,MnSO4·H2O 0.01,维生素B1、维生素B3、维生素B5、维生素B12和维生素H各0.001。
发酵培养基(g/L):酵母粉4,蛋白胨5,柠檬酸钠2,KH2PO4·3H2O 2,MgSO4·7H2O 2,FeSO4·7H2O 0.02,MnSO4·H2O 0.02,维生素B1、维生素B3、维生素B5、维生素B12和维生素H各0.002。
在INO2-4基础上改造的菌株,培养基中均增添0.3 g/L腺嘌呤。
1.4 基因编辑方法
本研究采用CRISPR/Cas9基因编辑方法[7]对大肠杆菌进行基因改造。该方法由pGRB质粒、pREDCas9质粒和DNA重组片段3个元件共同介导,即pGRB质粒和DNA重组片段以电转化的方式导入到含pREDCas9质粒的感受态细胞中,pGRB质粒表达出特异性gRNA来识别目的基因靶位点,pREDCas9质粒表达出Cas9蛋白对该位点进行切割,DNA重组片段通过同源重组的方式对切割位点进行修复,最终实现基因的准确敲除与整合。具体步骤以整合pbuE基因为例:
使用Primer 5软件设计上同源臂引物yjiT-UP-S和yjiT-UP-A、插入片段引物pbuE-S和pbuE-A、下同源臂引物yjiT-DN-S和yjiT-DN-A。通过PCR扩增获得上同源臂yjiT-UP、插入片段Ptrc-pbuE、下同源臂yjiT-DN,纯化回收后再经PCR重叠获得DNA重组片段yjiT∶∶Ptrc-pbuE。使用Cas-Designer工具设计yjiT假基因位点切割靶序列,获得gRNA-yjiT-S和gRNA-yjiT-A两条单链。将上述单链退火所得双链片段与线性化载体进行同源重组,再化转至DH5α化转感受态中,挑取阳性转化子以获得pGRB-yjiT质粒。将重组片段yjiT∶∶Ptrc-pbuE和pGRB-yjiT质粒同时电转化至含pREDCas9质粒的INO3-2感受态细胞中,挑取阳性转化子并消除pGRB质粒和pREDCas9质粒,获得重组菌株INO4-2。
1.5 发酵方法
1.5.1 摇瓶发酵
发酵温度为37 ℃,发酵周期为24 h,发酵过程中通过补加氨水维持pH值为7.0~7.2,初始葡萄糖耗尽后,补加600 g/L葡萄糖溶液维持发酵进行。具体操作可参考文献[8]。
1.5.2 发酵罐分批补料发酵
发酵温度为37 ℃,pH为7.0,溶氧(dissolved oxygen,DO)为25%~35%,接种量为20%,发酵周期为48 h。具体操作可参考文献[8]。
1.6 检测方法
采用高效液相色谱法方法[Thermo U3000,C18色谱柱,V(乙腈)∶V(0.5%三氟乙酸水溶液)=2∶98]对肌苷浓度进行检测,设置柱温30 ℃,流速1 mL/min,紫外检测波长260,进样量20 μL。
残糖浓度和生物量(OD600)测定参考文献[8]进行。
1.7 数据分析方法
参考文献[8]进行数据分析。
2 结果与分析
2.1 肌苷合成途径的强化
强化合成途径是促进产物积累的常用策略之一。在之前的研究中,本实验室通过诱变筛选获得了一株产鸟苷的解淀粉芽孢杆菌突变株TA208。测序结果表明[9],该菌株的嘌呤操纵子已发生突变,推测有助于解除反馈调控,提升嘌呤从头合成酶系的表达水平。本研究首先敲除了野生型大肠杆菌MG1655的rihA、rihB、rihC、deoD、ppnP基因,以阻断肌苷的降解,构建了底盘菌株INO1-1。为强化肌苷合成途径,本研究将突变株TA208的嘌呤操纵子(purEKBCSQLGMNHD)整合在底盘菌株INO1-1的yghE假基因位点,由Ptrc启动子控制,构建了菌株INO1-2。摇瓶发酵结果如图2所示,INO1-2的肌苷产量达到155.2 mg/L。
PRPP和谷氨酰胺在PRPP转酰胺酶(amidophosphoribosyltransferase,PurF)催化下生成5-磷酸核糖胺和谷氨酸,是肌苷合成途径的限速步骤,而PurF的催化活性受腺苷酸(adenosine monophosphate,AMP)和鸟苷酸(guanosine monophosphate,GMP)的协同反馈抑制[10]。据报道,大肠杆菌自身的PurF和枯草芽孢杆菌来源的PurF均可通过定点突变解除其所受反馈抑制[2,11]。于是,本研究将这两种来源的突变基因purFecoK326Q,P410W和purFbsuD293V,K316Q,S400W分别整合至INO1-2的yeeP假基因位点,由Ptrc启动子控制,构建了菌株INO1-3和INO1-4。摇瓶发酵结果如图2所示,INO1-4肌苷产量为308.3 mg/L,相比INO1-2提升98.6%;INO1-3肌苷产量为189.5 mg/L,相比INO1-2提升22.1%。结果表明,两种PurF突变体的过表达均使肌苷产量得以提升,但突变体PurFbsuD293V,K316Q,S400W的效果更好,可显著增强肌苷合成途径通量,提升肌苷的产量。
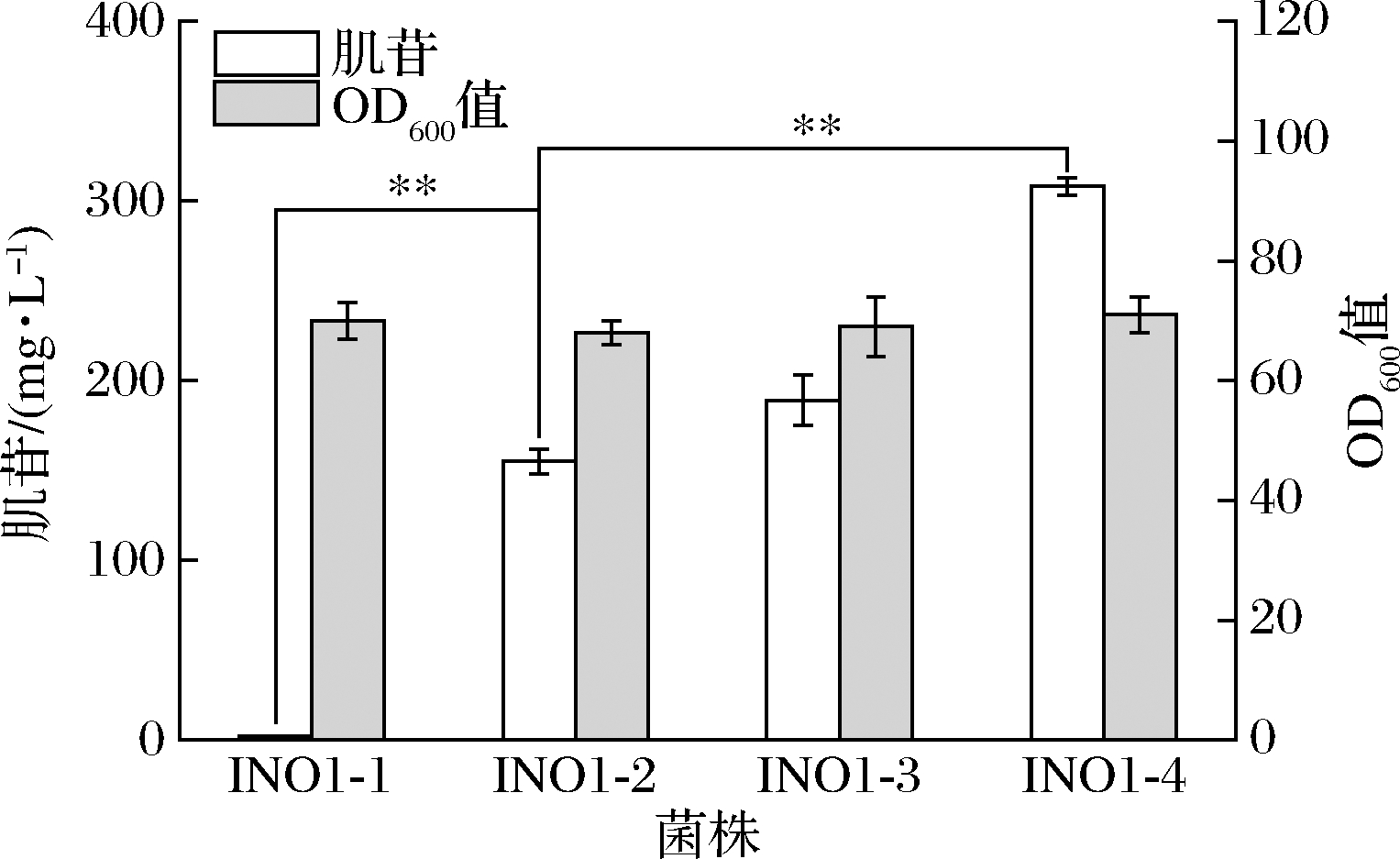
图2 增强肌苷合成途径对肌苷发酵的影响
Fig.2 Effects of enhancing the expression of inosine synthesis pathway on inosine fermentation
注:*表示差异显著(P<0.05),**表示
差异极显著(P<0.01)(下同)
2.2 腺苷合成支路的弱化
IMP到肌苷的分支途径是肌苷合成途径的重要组成部分,然而,IMP还存在另外两条分支途径,分别由腺苷酸合成酶(adenylosuccinate synthetase,PurA)和IMP脱氢酶(IMP dehydrogenase,GuaB)参与催化,向腺苷和鸟苷代谢。研究者对IMP分支代谢通量进行分析,发现流向腺苷支路的碳代谢流远超流向肌苷和鸟苷支路的碳代谢流[3],是限制肌苷合成的重要因素。因此,为提升肌苷支路代谢通量,同时探究阻断腺苷或鸟苷支路对菌体生长和生产的影响,本研究在INO1-4中分别单敲除与双敲除了purA和guaB基因,依次构建了菌株INO2-1、INO2-2和INO2-3。摇瓶发酵结果如图3所示,INO2-1、INO2-2和INO2-3的OD600值均下降至4.0左右,肌苷产量分别下降至75.2、33.7、98.1 mg/L。这表明腺苷和鸟苷支路的阻断使细胞所需能量或营养物质合成受限,进而严重影响到菌体生长与生产。在培养基中添加0.3 g/L腺嘌呤和0.3 g/L鸟嘌呤后,3个菌株的OD600值和肌苷产量均显著提升。其中INO2-1的肌苷产量为1 180.9 mg/L,OD600值为25.1;INO2-2肌苷产量为445.4 mg/L,OD600值为31.2;INO2-3的肌苷产量为778.8 mg/L,OD600值为17.7。这表明阻断腺苷或鸟苷支路均可促进肌苷积累,但腺苷支路的阻断对肌苷产量的提升效果更加明显;同时阻断两条支路代谢会使菌体内代谢严重失衡,即使在添加缺陷物质的条件下仍影响菌体生长。另外,腺苷支路的阻断使AMP从头合成受阻,减弱了PRPP转酰胺酶所受反馈抑制,是肌苷产量提升的另一原因。
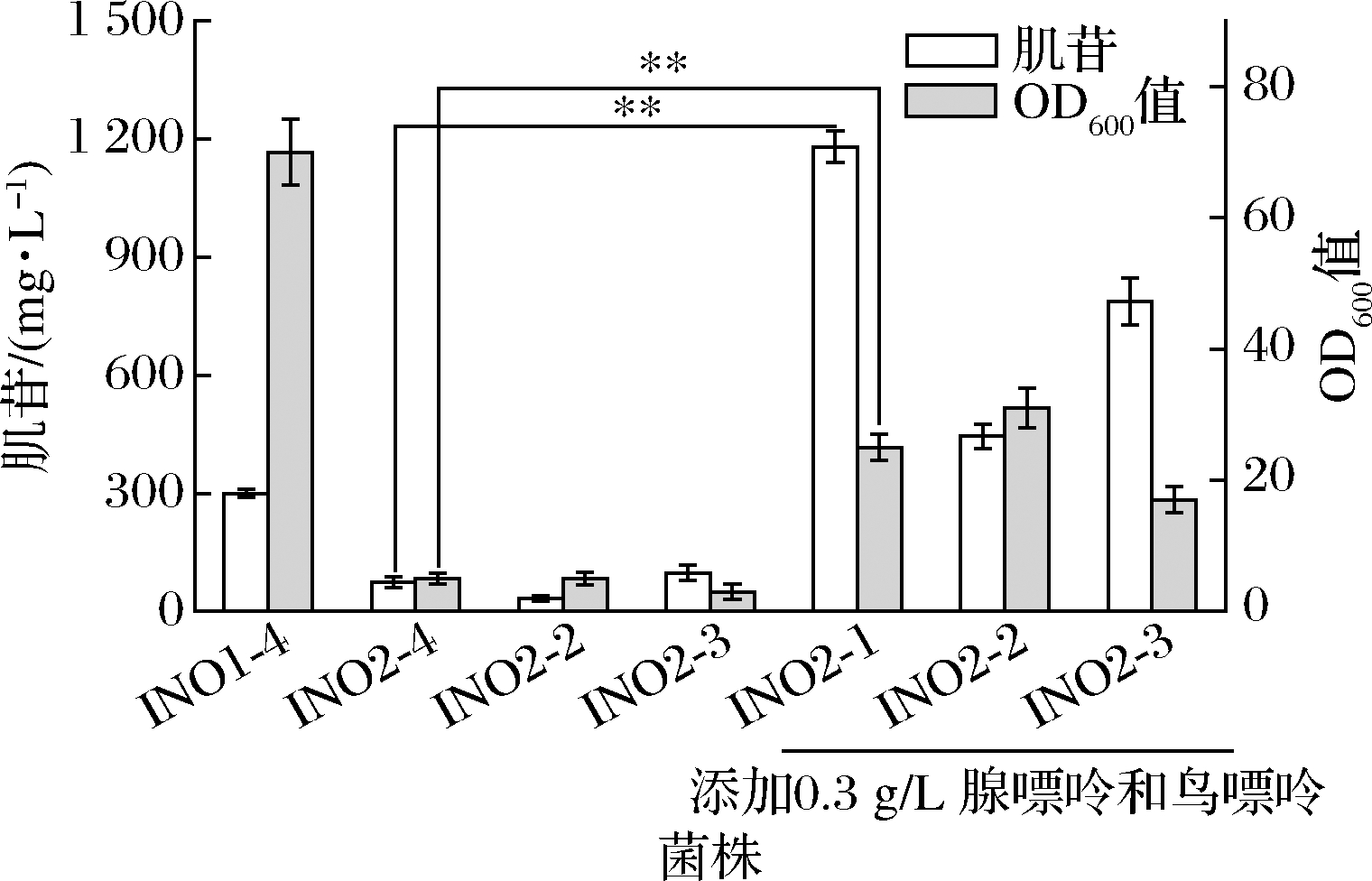
图3 阻断竞争代谢途径对菌体生长和肌苷发酵的影响
Fig.3 Effects of blocking competing metabolic pathways on growth and inosine fermentation
阻断腺苷支路虽可显著提升肌苷产量,但会严重影响菌株生长。为平衡菌株生长和产物合成,适当弱化而非直接阻断腺苷支路是更加理性的选择。研究表明,将枯草芽孢杆菌来源的PurA中第242位的脯氨酸替换为天冬酰胺,可降低53.7%的酶活力[5]。于是,本研究将INO1-4的purA基因替换为purAbsuP242N突变基因,构建了菌株INO2-4。摇瓶发酵结果如图4所示,在无腺嘌呤添加的条件下,INO2-4的肌苷产量为810.2 mg/L,是INO2-1的10.8倍;OD600值为21.4,是INO2-1的5.4倍。在添加腺嘌呤的条件下,INO2-4的肌苷产量达到1 412.5 mg/L,相比INO2-1提升了19.6%;OD600值为30.3,相比INO2-1提升了20.7%。结果表明,突变体PurAbsuP242N的替换既可维持菌株一定的生长水平,又能保证足够的肌苷支路通量,进而使肌苷产量和菌体OD600值均显著提升。另外,添加腺嘌呤可使产量和生长提升至更高的水平。

图4 弱化腺苷合成支路对菌体生长和肌苷发酵的影响
Fig.4 Effects of weakening Adenosine synthesis branch on growth and inosine fermentation
2.3 增强前体物PRPP的供应
增强前体物的供应有利于将代谢流持续导入目标产物合成途径,促进产物的积累。PRPP是嘌呤合成的重要前体物,其合成酶编码基因prs的表达受到嘌呤阻遏物(DNA-binding transcriptional repressor,PurR)的反馈阻遏[12],同时Prs的催化活性受到腺苷二磷酸(adenosine-5′-diphosphate,ADP)和鸟苷二磷酸(guanosine-5′-diphosphate,GDP)的反馈抑制[10]。为解除反馈调控,本研究首先敲除了INO2-4的purR基因,构建了菌株INO3-1。摇瓶发酵结果如图5所示,INO3-1的肌苷产量为1.6 g/L,与INO2-4相比提升了14.3%,OD600值没有明显变化。据报道,在大肠杆菌的Prs中引入D128A位点突变[2]和在解淀粉芽孢杆菌的Prs中引入N120S和L135I位点突变[13],均可解除PRPP合酶所受反馈抑制。因此,本研究在INO3-1中的gapC位点分别整合了prsecoD128A与prsbazN120S,L135I突变基因,由Ptrc启动子控制,构建了菌株INO3-2和INO3-3。摇瓶发酵结果如图5所示,INO3-2的肌苷产量达到3.1 g/L,与INO3-1相比提升了93.8%,而INO3-3的肌苷产量没有提升。这表明突变体PrsecoD128A的过表达有效增强了PRPP的供应,进而提升了肌苷的产量。
2.4 肌苷转运系统的改造
产物在胞内积累过多会给细胞带来代谢负担,也会引起产物介导的反馈抑制,需要及时排到胞外。为促进肌苷外排,本研究在INO3-2的yjiT位点整合了大肠杆菌来源的nepI基因[14]和解淀粉芽孢杆菌来源的pbuE基因[15],由Ptrc启动子控制,构建了菌株INO4-1和INO4-2。摇瓶发酵结果如图6所示,INO4-2的肌苷产量为3.9 g/L,相比INO3-2提升了25.8%;OD600值为35.2,相比INO3-2提升了16.7%;INO4-1的肌苷产量和OD600值均无明显提升。这表明异源转运蛋白的引入有效提升了菌体对肌苷的外排能力,同时缓解细胞内代谢负担,促进了菌体生长。为进一步提升肌苷产量,本研究在INO4-2的ilvG及ylbE位点分别整合了pbuE基因的双拷贝及三拷贝,由Ptrc启动子控制,构建了菌株INO4-3与INO4-4。摇瓶发酵结果显示,INO4-3的肌苷产量为4.5 g/L,相比INO4-2提升了15.4%;INO4-4的肌苷产量没有提升。这表明两个拷贝的pbuE基因已最大化菌体对肌苷的外排能力。另外,nupC[16]、nupG[16]、ydhC[17]和xapB[16]基因也编码肌苷转运蛋白,可将肌苷从胞外摄取到胞内。于是本研究分别敲除了INO4-3的nupC、nupG、ydhC和xapB基因,构建了菌株INO4-5、INO4-6、INO4-7与INO4-8。摇瓶发酵结果如图6所示,INO4-5的肌苷产量为5.0 g/L,与INO4-3相比提升了11.1%,其余菌株的肌苷产量均没有提升。这表明nupC基因所编码的转运蛋白是参与肌苷摄取的主要透性酶,敲除该基因可减弱菌体对肌苷的吸收。
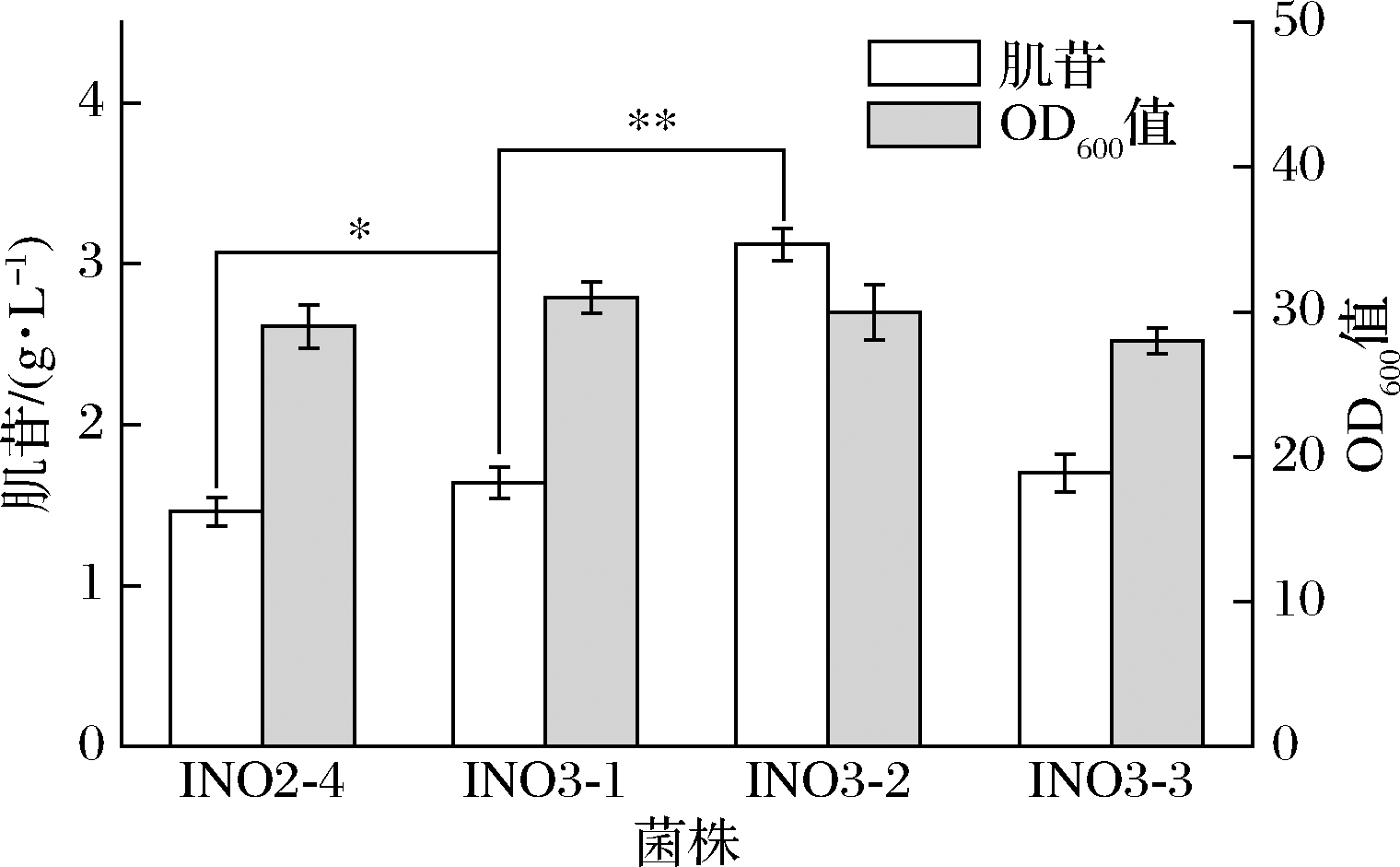
图5 增强PRPP供应对肌苷发酵的影响
Fig.5 Effects of enhancing the supply of PRPP on inosine fermentation
2.5 工程菌发酵罐发酵测试
为测试菌株INO4-5的发酵性能,本研究5 L发酵罐中进行了分批补料发酵。当葡萄糖质量浓度低于2 g/L时,开始流加800 g/L的葡萄糖。发酵结果如图7所示,菌株在发酵初期生长缓慢,20 h后OD600值达到最高并进入稳定期。0~12 h,肌苷合成速率较慢;12~32 h,肌苷保持了较高的合成速率。48 h 时肌苷的最终产量为20.2 g/L,糖酸转化率为0.12 g/g葡萄糖,生产强度为0.42 g/(L·h)。
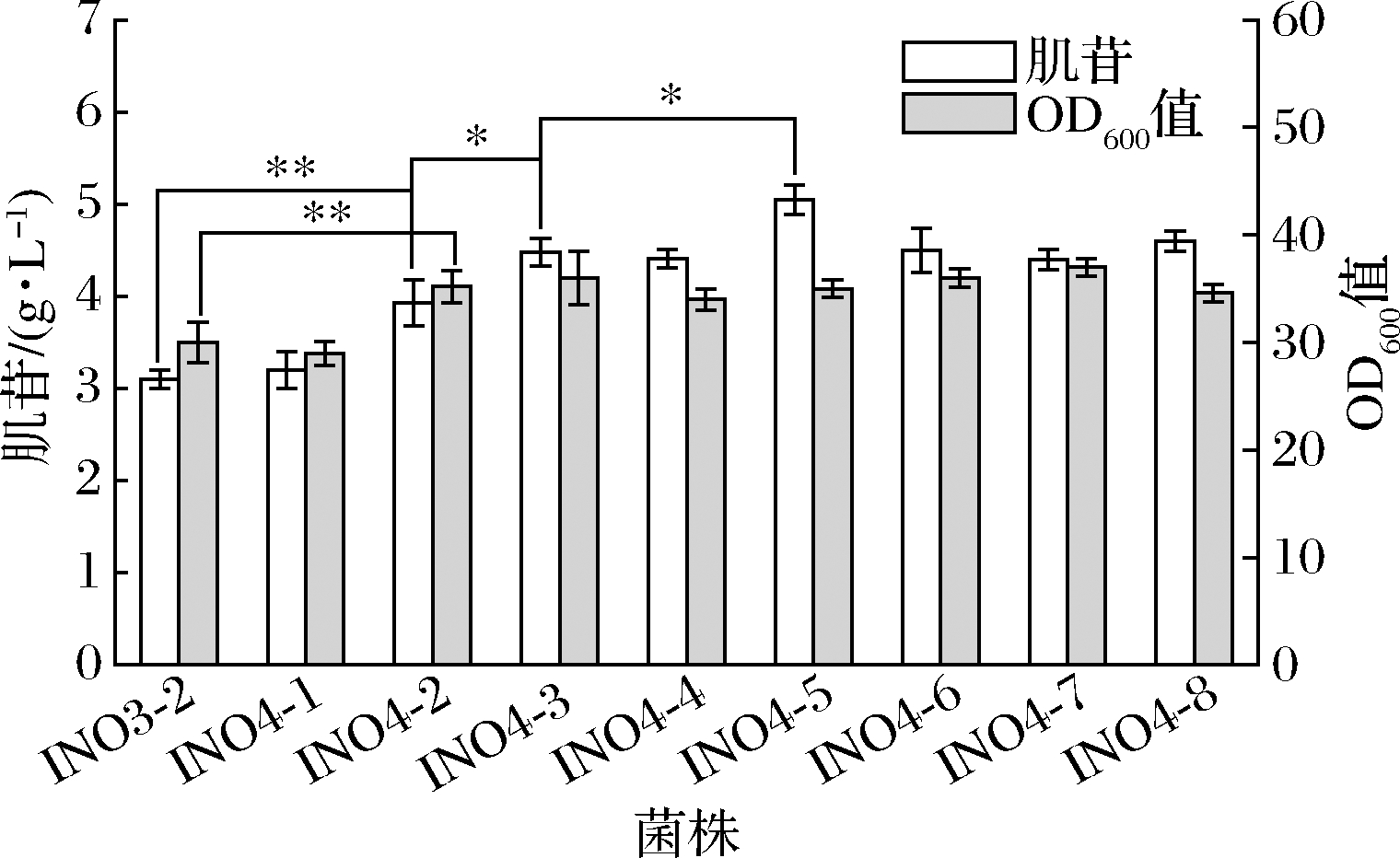
图6 转运系统改造对肌苷发酵的影响
Fig.6 Effects of transport system modification on inosine fermentation
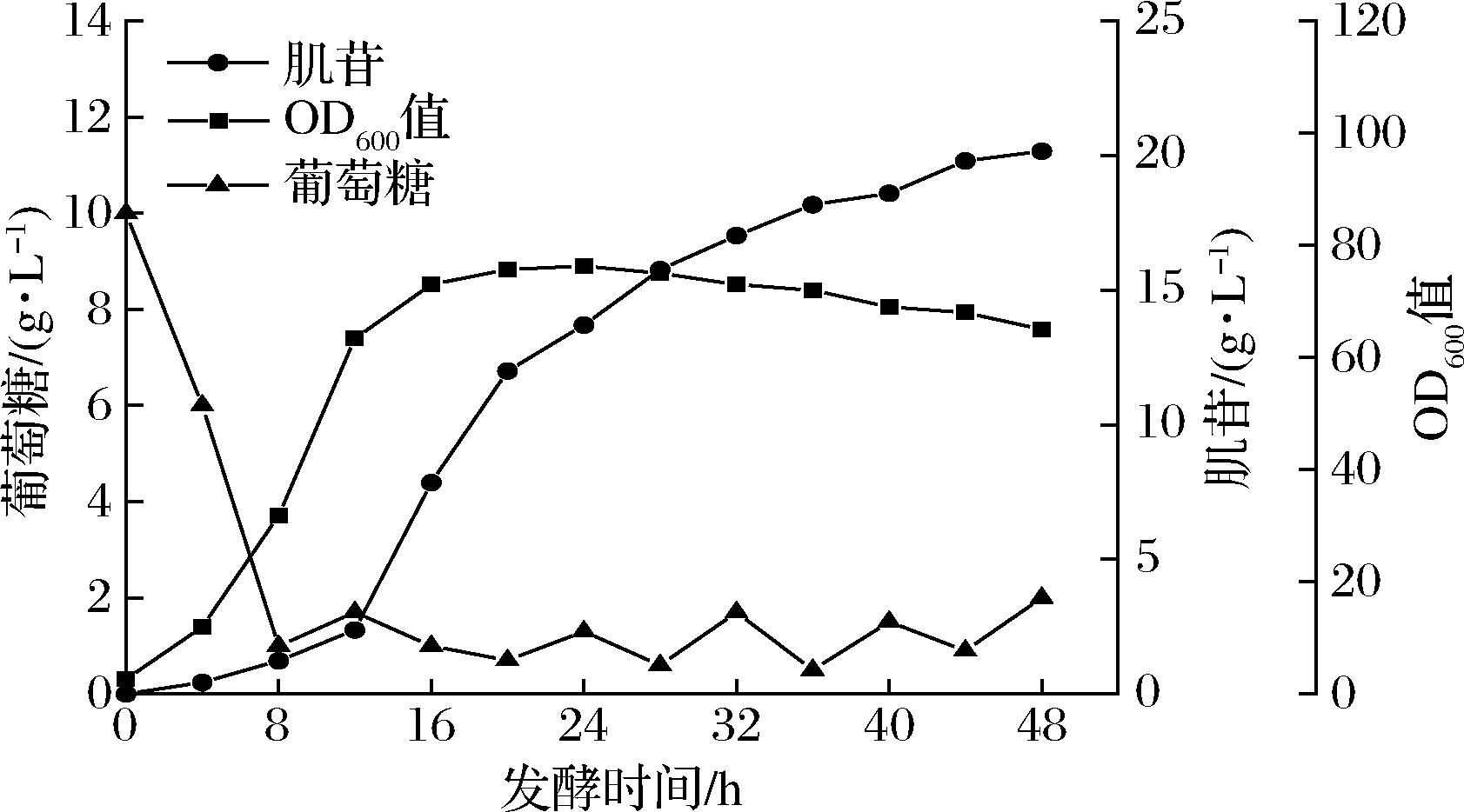
图7 工程菌INO4-5的5 L发酵罐发酵结果
Fig.7 Fermentation results of the engineered strain INO4-5 in a 5 L bioreactor
3 讨论
近年来,随着抗病毒和抗肿瘤药物需求量的增加,嘌呤核苷及其衍生物受到广泛关注。然而,由于代谢途径冗长、代谢调控复杂以及多种前体物需求,肌苷合成十分困难。芽孢杆菌因具有较强的PPP途径和嘌呤合成途径通量,以及较弱的核苷磷酸化酶活力,是肌苷生产菌株选育中常见的底盘菌株。相比芽孢杆菌,大肠杆菌基因操作高效、发酵控制简便、生产周期较短,是发酵法生产肌苷另一个优良的宿主。于是,本研究将Ptrc启动子控制下的芽孢杆菌嘌呤操纵子引入大肠杆菌,不仅绕过了PurR介导的反馈阻遏机制、鸟嘌呤介导的转录衰减机制[18],而且通过借助异源优势,提高了嘌呤从头合成酶系的表达水平。随着各模块的优化组合,肌苷产量持续提高,进一步体现了引入芽孢杆菌嘌呤操纵子的重要作用。
腺苷和鸟苷支路对碳代谢流的分流也是肌苷合成的重要限制因素。本研究对比了阻断这两条支路对肌苷产量的影响,发现腺苷支路的阻断对肌苷产量提升效果更加明显,但会导致菌体营养缺陷。为平衡菌体生长和产物合成,本研究尝试了两种方式来改造腺苷支路。一方面是静态弱化。本研究通过将自身PurA替换为低酶活力的PurAbsuP242N突变体的方式来弱化腺苷支路,相比于直接阻断的方式,菌体生长水平和肌苷产量均大幅提升。为进一步提升菌株发酵性能,本研究尝试拷贝了第二个purAbsuP242N基因。摇瓶发酵结果显示,菌体OD600值达到70,而肌苷产量大幅降低,表明二拷贝purAbsuP242N基因使腺苷支路分流过强,不利于肌苷合成。后期应尝试其他表达强度,或开发新的活性位点突变,使PurA的表达更加均衡,同步提升菌体生长水平和肌苷产量。另一方面是动态调控。本研究选择引入群体感应系统[19],通过响应菌体密度来动态调控purA基因的转录水平。发酵结果显示,在生长初期,菌体OD600值相比对照菌株INO1-4偏低;12 h后,菌体OD600值与对照菌株INO1-4几乎保持一致;24 h摇瓶发酵后,肌苷产量仅为0.3 g/L。推测purA基因转录停止后,胞内已表达的PurA持续催化腺苷支路,导致肌苷支路通量不足。后期应尝试在转录调控的基础上,结合蛋白水平的调节,如引入TEVp[20]、SsrA[21]等元件适时降解PurA,使动态调控更加严谨和高效,真正实现“生长模式”到“生产模式”的自动切换。
PRPP是嘌呤从头合成过程中的重要前体物。本研究通过敲除purR基因、过表达prsecoD128A基因,增强了PRPP的供应,显著提升了肌苷的产量。由于PRPP的前体物5-磷酸核糖经PPP途径生成,为持续提升菌株发酵性能,需对PPP途径进行改造。为此,本研究过表达了PPP途径的关键酶编码基因gnd和zwf,发酵结果显示,肌苷的产量并没有提高,而菌体OD600值略有下降。我们推测PPP途径的强化,使胞内还原力NADPH水平提升,反馈抑制了关键酶6-磷酸葡糖酸脱氢酶和葡萄糖-6-磷酸1-脱氢酶的酶活力,进而限制了PPP碳通量的增强;胞内NADPH/NADP+的比例失衡,可能是影响菌体生长的原因。因此,开发解除反馈抑制的关键酶活性位点突变,同时增强NADPH的代谢以平衡细胞内氧化还原水平,是进一步提升肌苷产量的潜在策略。此外,减弱竞争途径也是提升PPP途径通量的关键考虑。糖酵解途径(embden-meyerhof-parnas,EMP)是PPP的主要竞争途径。WU等[22]通过敲除pgi基因来完全阻断EMP途径,经52 h发酵,组氨酸产量与对照菌株相同,而菌体OD600值大幅下降,糖耗也显著降低。这表明通过减弱EMP途径来增强PPP途径通量的方式,可显著提升以PRPP为前体物的产物的糖酸转化率。但对于EMP途径与PPP途径通量的再分配,应侧重于更加温和可控方式,如通过起始密码子替换或动态调控等方式来减弱EMP途径,才能达到同步提升产量和糖酸转化率的目的。同样地,减弱2-酮-3-脱氧-6-磷酸葡糖酸途径[23]与PPP途径的非氧化分支[24],也有助于降低碳代谢流损失,促使更多碳代谢流流向肌苷合成途径,提升肌苷的产量。
[1] ASAHARA T, MORI Y, ZAKATAEVA N P, et al.Accumulation of gene-targeted Bacillus subtilis mutations that enhance fermentative inosine production[J].Applied Microbiology & Biotechnology, 2010, 87(6):2 195-2 207.
[2] SHIMAOKA M, TAKENAKA Y, KURAHASHI O, et al.Effect of amplification of desensitized purF and prs on inosine accumulation in Escherichia coli[J].Journal of Bioscience & Bioengineering, 2007, 103(3):255-261.
[3] LI H, ZHANG G Q, DENG A H, et al.De novo engineering and metabolic flux analysis of inosine biosynthesis in Bacillus subtilis[J].Biotechnology Letters, 2011, 33(8):1 575-1 580.
[4] PEIFER S, BARDUHN T, ZIMMET S, et al.Metabolic engineering of the purine biosynthetic pathway in Corynebacterium glutamicum results in increased intracellular pool sizes of IMP and hypoxanthine[J].Microbial Cell Factories, 2012, 11:138.
[5] WANG X Y, WANG G L, LI X L, et al.Directed evolution of adenylosuccinate synthetase from Bacillus subtilis and its application in metabolic engineering[J].Journal of Biotechnology, 2016, 231:115-121.
[6] 刘益宁, 秦臻, 李旋, 等.胞苷合成途径改造对大肠杆菌嘧啶核苷发酵的影响[J].食品与发酵工业, 2021, 47(12):10-16.
LIU Y N, QIN Z, LI X, et al.Effect of cytidine synthesis pathway modification on the fermentation of pyrimidine nucleoside by Escherichia coli[J].Food and Fermentation Industries, 2021, 47(12):10-16.
[7] LI Y F, LIN Z Q, HUANG C, et al.Metabolic engineering of Escherichia coli using CRISPR-Cas9 meditated genome editing[J].Metabolic Engineering, 2015, 31:13-21.
[8] 吴鹤云, 张悦, 蒋帅,等.木糖和葡萄糖共发酵生产L-组氨酸[J].食品与发酵工业, 2020, 46(16):115-120.
WU H Y, ZHANG Y, JIANG S, et al.Co-fermentation of xylose and glucose for L-histidine production[J].Food and Fermentation Industries, 2020, 46(16):115-120.
[9] ZHANG G Q, DENG A H, XU Q Y, et al.Complete genome sequence of Bacillus amyloliquefaciens TA208, a strain for industrial production of guanosine and ribavirin[J].Journal of Bacteriology, 2011, 193(12):3 142-3 143.
[10] 王永成, 陈涛, 石婷, 等.嘌呤核苷及其衍生物的代谢工程[J].中国生物工程杂志, 2015, 35(5):87-95.
WANG Y C, CHEN T, SHI T, et al.Progress in biosynthesis of purine nucleosides and their derivatives by metabolic engineering[J].China Biotechnology, 2015, 35(5):87-95.
[11] SHI T, WANG Y C, WANG Z W, et al.Deregulation of purine pathway in Bacillus subtilis and its use in riboflavin biosynthesis[J].Microbial Cell Factories, 2014, 13:101.
[12] HE B, CHOI K Y, ZALKIN H.Regulation of Escherichia coli glnB, prsA, and speA by the purine repressor[J].Journal of Bacteriology, 1993, 175(11):3 598-3 606.
[13] ZAKATAEVA N P, ROMANENKOV D V, SKRIPNIKOVA V S, et al.Wild-type and feedback-resistant phosphoribosyl pyrophosphate synthetases from Bacillus amyloliquefaciens:Purification, characterization, and application to increase purine nucleoside production[J].Applied Microbiology and Biotechnology, 2012, 93(5):2 023-2 033.
[14] GRONSKIY S V, ZAKATAEVA N P, VITUSHKINA M V, et al.The yicM (nepI) gene of Escherichia coli encodes a major facilitator superfamily protein involved in efflux of purine ribonucleosides[J].FEMS Microbiology Letters, 2005, 250(1):39-47.
[15] ZAKATAEVA N P, GRONSKIY S V, SHEREMET A S, et al.A new function for the Bacillus PbuE purine base efflux pump:Efflux of purine nucleosides[J].Research in Microbiology, 2007, 158(8-9):659-665.
[16] NØRHOLM M H, DANDANELL G.Specificity and topology of the Escherichia coli xanthosine permease, a representative of the NHS subfamily of the major facilitator superfamily[J].Journal of Bacteriology, 2001, 183(16):4 900-4 904.
[17] RODIONOVA I A, GAO Y, SASTRY A, et al.Identification of a transcription factor, PunR, that regulates the purine and purine nucleoside transporter punC in E.coli[J].Communications Biology, 2021, 4:991.
[18] MANDAL M, BOESE B, BARRICK J E, et al.Riboswitches control fundamental biochemical pathways in Bacillus subtilis and other bacteria[J].Cell, 2003, 113(5):577-586.
[19] GUPTA A, REIZMAN I M B, REISCH C R, et al.Dynamic regulation of metabolic flux in engineered bacteria using a pathway-independent quorum-sensing circuit[J].Nature Biotechnology, 2017, 35(3):273-279.
[20] GAO C, HOU J S, XU P, et al.Programmable biomolecular switches for rewiring flux in Escherichia coli[J].Nature Communications, 2019, 10:3 751.
[21] BROCKMAN I M, PRATHER K L J.Dynamic knockdown of E.coli central metabolism for redirecting fluxes of primary metabolites[J].Metabolic Engineering, 2015, 28:104-113.
[22] WU H Y, TIAN D G, FAN X G, et al.Highly efficient production of L-histidine from glucose by metabolically engineered Escherichia coli[J].ACS Synthetic Biology, 2020, 9(7):1 813-1 822.
[23] SHIMAOKA M, KAWASAKI H, TAKENAKA Y, et al.Effects of edd and pgi disruptions on inosine accumulation in Escherichia coli[J].Bioscience, Biotechnology, and Biochemistry, 2005, 69(7):1 248-1 255.
[24] KAMADA N, YASUHARA A, TAKANO Y, et al.Effect of transketolase modifications on carbon flow to the purine-nucleotide pathway in Corynebacterium ammoniagenes[J].Applied Microbiology and Biotechnology, 2001, 56(5-6):710-717.

