色氨酸(tryptophan ,Trp)是人体的必需氨基酸之一,参与机体蛋白质的合成和代谢调节,它是烟酸、5-羟色胺、褪黑激素等生物活性物质的前体物,影响蛋白质、糖类和脂肪等营养物质的代谢。色醇(indole-3-ethanol,IET)和色醇类似物是合成几个重要的药用活性分子的常见中间体,包括以色胺为基础的药物,例如曲普坦家族的抗偏头痛药物的某些成员(舒马曲坦、利扎曲坦和佐米曲坦),2α-1选择性肾上腺素受体拮抗剂吲哚,或基于吡喃吲哚结构的化合物,如依托度酸。依托度酸是以IET为主要中间体合成的一种有效的抗炎镇痛化合物[1]。同时,IET也是合成抗肾上腺素能药物吲哚胺的关键中间体[2]。
目前,工业上IET的生产全部是化学合成,方法是连续流合成工艺[3]。以盐酸苯肼(phenyl hydrazine hydrochloride, PHH)和2,3-二氢呋喃(2,3-dihydrofuran, DHF)为原料,采用Fischer吲哚合成路线,在管式反应器中实现了不同温度、不同流速下IET的连续流合成。通过其他方式进行过改进,比如,通过微波进行反应过程的控制,但是反应副产物辛诺啉衍生物和多吲哚等仍然无法解决,这无疑会使成本增加。IET被发现是因为它是酿酒生产的副产物[4],因此,IET的生物发生途径就存在于酿酒酵母中,这便给了IET生物合成的可能。而且酵母中的IET来自Ehrlich途径[5]。几种氨基酸(支链氨基酸和芳香族氨基酸以及蛋氨酸)可以通过Ehrlich途径被同化并转化为高级杂醇。该路径包括3个反应:(1)氨基酸的转氨作用,Aro8p和Aro9p最初分别被表征为芳香族氨基酸氨基转移酶I和Ⅱ,它们在Ehrlich途径中充当广泛底物特异性的氨基酸转氨酶,负责几乎所有相关氨基酸涉及的转氨基作用,并形成对应的α-酮酸;(2)在前一个反应中形成的α-酮酸的脱羧作用,这个不可逆的步骤归因于酵母体内的丙酮酸脱羧酶,这一步骤之后使对应的α-酮酸转变为对应的杂醛;(3)Ehrlich途径的最后一步会产生分支,使产生的杂醛还原成酸或者氧化为醇[6]。
IET的价格高达22 000美元/kg,而且是一种具有广泛应用前景的化合物,作为重要的医药和化工合成的中间体,它的需求量只增不减,拥有广阔的市场前景。但是目前的合成方法存在2个明显的缺点,一是化学合成并不绿色环保,可能对环境和生物造成不可逆的损害;另一个是化学合成过程中的副产物过多,这样会使后期分离得到的产物纯度并不高,而且纯化过程将提高成本。使用酿酒酵母细胞工厂来生产IET不仅绿色环保,而且由于酿酒酵母易于培养、生长迅速等特点[7],可以使生产的成本大大降低,还得益于酿酒酵母基因的可改造性,可以使用其生产的IET直接在细胞内合成更多更有价值的化合物。Trp合成途径本来就存在于酿酒酵母中,可以通过探索相关的基因来增强Trp到IET的生物转化。
本研究对涉及Trp代谢到IET的代谢途径中相关的13个基因(图1)进行了分步骤的过表达,选定Trp到IET的相关基因进行组合探索出了最适合的利于IET生物生产的基因组合。
1 材料与方法
1.1 菌株构建和试剂
本研究所使用的所有质粒的构建和保存均使用大肠杆菌Escherichia coli JM109。Ex Taq®和 PrimeSTAR® Max DNA 聚合酶购买于TaKaRa。用于质粒同源重组的试剂盒MultiF Seamless Assembly Mix购买于武汉爱博泰克生物科技有限公司。限制性核酸内切酶Esp3I (BsmBI)和Eco31I (BsaI)购买于Thermo。T7 DNA连接酶和T4 DNA 连接酶Buffer购买于安诺伦(北京)生物科技有限公司。所有引物(表1)均在苏州金唯智生物科技有限公司合成。除非特殊说明所用化学品,包括各种标准品,均购买于上海阿达玛斯试剂有限公司。
1.2 菌种培养
用于酿酒酵母Saccharomyces cerevisiae菌株BY4741制备感受态细胞的培养基(YPD)包括10 g/L酵母提取物、20 g/L蛋白胨和20 g/L 葡萄糖。为了更好的研究代谢改造的作用,所有使用的人工全合成培养基(SC)均不含有Trp。在缺少URA和Trp的人工全培养基上筛选出含有URA3原养型标记质粒和基因组整合的酿酒酵母菌株;在缺少URA、Trp和His的人工全培养基上筛选出含有URA3和HIS3原养型标记质粒和基因组整合的酿酒酵母菌株,培养基包括6.7 g/L酵母无氨基酸氮源(YNB)、20 g/L葡萄糖、0.72 g/L酵母氨基酸缺失混合物和20 g/L琼脂。带有抗性基因的质粒通过向培养基中添加300 mg/L的潮霉素或者200 mg/L的G418来进行筛选。
生产IET的预培养通过挑取平板菌落在24孔板中进行,每孔添加2 mL SC培养基,30 ℃,220 r/min下过夜发酵。预培养的菌液接种于2 mL的24孔板SC培养基中,30 ℃,220 r/min下发酵36 h。
大肠杆菌JM109被用于质粒的贮存,并在需要时在添加了25 mg/L氯霉素、100 mg/L氨苄西林或者50 mg/L卡那霉素的肉汤培养基(LB)中生长,培养基包括5 g/L酵母抽提物、10 g/L蛋白胨和10 g/L氯化钠。通过在培养基中加入20 g/L的琼脂得到固体培养基。
1.3 质粒构建
本研究所使用的构建和贮存质粒的大肠杆菌均为JM109,按照文献[8]提供的方法制作大肠杆菌感受态细胞并进行转化。本研究所使用的所有引物和质粒列在表1和表2中。质粒构建的方法主要使用Yeast Tool Kit[9],运用Golden Gate原理的模块化的组装方法。对于需要用到的单或多碱基突变使用引物融合聚合酶链式反应(polymerase chain reaction,PCR)或者使用PCR介导的定点突变[10]。
表1 本研究所用的引物
Table 1 Primers in this study

名称序列Seq_GFPGGTATGGATGAACTGTACAAATGACSeq_KanATCCTATGGAACTGCCTCGGSeq-AllTCTTTATAGTCCTGTCGGGTTTCGSeq-All_1CACCTCTGACTTGAGCGTCSeq_KanATCCTATGGAACTGCCTCGGSeq-Con_ECTGTTGTGATATGCCATATATGCACamR-V-All_RCAACAGTACTGCGATGAGTGSeq-Amp_RCGGGGAGTCAGGCAACTATGGGA-ARO8_RATGCCGTCTCAGGTCTCAGGATCCTTTGGAAATACCAAATTCTTCGTATAAAGTGGA-ARO8_FGCATCGTCTCATCGGTCTCATATGACTTTACCTGAATCAAAAGACTTTTCTTGGA-ARO9_FGCATCGTCTCATCGGTCTCATATGACTGCTGGTTCTGCCGGA-ARO9_RATGCCGTCTCAGGTCTCAGGATCCACTTTTATAGTTGTCAAAAAATTCTTTTATGCCMofARO9_FTCCGATGAGACGGAGTCTAACMofARO9_RCTCCGTCTCATCGGAAAAGGGGA-ARO10_FGCATCGTCTCATCGGTCTCATATGGCACCTGTTACAATTGAAAAGTGGA-ARO10_RATGCCGTCTCAGGTCTCAGGATCCTTTTTTATTTCTTTTAAGTGCCGCTGCSeq-ARO10ATATATTCCAGTAAAACACCTGCGGGA-THI3_FGCATCGTCTCATCGGTCTCATATGAATTCTAGCTATACACAGAGATATGCAGGA-THI3_RATGCCGTCTCAGGTCTCAGGATCCGTATCCAACTTGATTTTTTTTTAGAAGTGGTSeq-THI3CTTGGATAGAACAGAGGCCAGGGA-PDC1_FGCATCGTCTCATCGGTCTCATATGTCTGAAATTACTTTGGGTAAATATTTGTGGA-PDC1_RATGCCGTCTCAGGTCTCAGGATCCTTGCTTAGCGTTGGTAGCAGSeq-PDC_RCACCGACGTGTTCAGCGTAGGA-PDC5_FGCATCGTCTCATCGGTCTCATATGTCTGAAATAACCTTAGGTAAATATTTATTTGAAGGA-PDC5_RATGCCGTCTCAGGTCTCAGGATCCTTGTTTAGCGTTAGTAGCGGCPDC5-siteM_FTTGCCTTGCAAAAATTGTTGGATGPDC5-siteM_RAATTTTTGCAAGGCAAATTTCATTTGASeq-PDC5CCCAGTTATCTTGGCTGATGGGA-PDC6_FGCATCGTCTCATCGGTCTCATATGTCTGAAATTACTCTTGGAAAATACTT\
续表1

名称序列GGA-ADH3_RATGCCGTCTCAGGTCTCAGGATCCTTTACTAGTATCGACGACGTATCTACCCADH4-siteM_FACAACTAATAGTCAAATAAGAAAAATGTCTTCADH4-siteM_RCTTATTTGACTATTAGTTGTTGCAAATGTAAGGA-ADH4_FGCATCGTCTCATCGGTCTCATATGCTTGGGATCACGTATGCGGA-ADH4_RATGCCGTCTCAGGTCTCAGGATCCATATTCATAGGCTTTCTTGATAATGGCAADH5-siteM_FAAGACATTGTTGGTGCTATAATAAAGGCADH5-siteM_RAGCACCAACAATGTCTTTTTCTTCCGGA-ADH5_FGCATCGTCTCATCGGTCTCATATGCCTTCGCAAGTCATTCCGGA-ADH5_RATGCCGTCTCAGGTCTCAGGATCCTTTAGAAGTCTCAACAACATATCTACCAACGGA-SFA1_FGCATCGTCTCATCGGTCTCATATGTCCGCCGCTACTGTTGGA-SFA1_RATGCCGTCTCAGGTCTCAGGATCCTTTTATTTCATCAGACTTCAAGACGGTTCURA3_5’_forwardGGGCGGATTACTACCGTTURA3_5’_reverseGTAATGTTATCCATGTGGGCURA3_3’_forwardAGAGCACTTGAATCCACTGCURA3_3’_reverseGATTTGGTTAGATTAGATATGGTTTCHO_5’_forwardCACATCATTTTCGTGGATCCHO_5’_reverseACAGCGATGGAACTTACGGCHO_3’_forwardTATCGTGTTGCATCTGCGGCHO_3’_reverseCTTTGGACTTAAAATGGCGTChr11-5_FGCATCGTCTCATCGGTCTCACAATTAACTCTTCGTATGAGGATTTTCGAChr11-5_RATGCCGTCTCAGGTCTCAAGGGCATGACTGGCATCACTGTACTTCTATGGCACATTTTTCTGTTGAGChr11-3_FGCATCGTCTCATCGGTCTCAGAGTTCGATCGATCGTAGATGCTCATCCACAAGTAAAGCTCGTTGACChr11-3_RATGCCGTCTCAGGTCTCATCGGATGGTTGAAAAGGTTACAGAGGATCChr11-5V_FCGATCCTCATGTTTTTGTTGTTTTCTChr11-5V_RCATGACTGGCATCACTGTACChr11-3V_FTCGATCGATCGTAGATGCTCATChr11-3V_RCCAACCCTGTGCCCTTTCTA
部件质粒pBL617通过引物GGA-ARO8_F和GGA-ARO8_R从酿酒酵母基因组中对应产物,然后通过产物带有的BsmB I酶切位点将其连接至YTK001获得部件质粒。运用相同的方法和原理从酿酒酵母基因组中得到ARO9,ARO10,THI3,PDC1,PDC5,PDC6,ADH1,ADH2,ADH3,ADH4,ADH5,SFA1基因,得到部件质粒pBL618,pBL621,pBL622,pBL623,pBL624,pBL625,pBL626,pBL627,pBL628,pBL629,pBL630,pBL631。
运用Yeast Tool Kit构建了通用的预组装质粒,构建了包含GFP dropout的带有URA3原养型标记整合型的质粒pBL505,带有HIS3原养型标记整合型的质粒pBL602,带有HygRomycin抗性标记的整合型质粒pBL638。
本研究所使用的程序均按照Yeast Tool Kit文献说明进行,构建了对应的可以用于整合的最终质粒,见表2。
表2 本研究使用的质粒
Table 2 Plasmids used in this study

名称特征部件类型或使用的部件来源YTK001 CamR Part Plasmid Entry Vectorentry vectorLEE等[9]pYTK009CamR pTDH32LEE等[9]pBL_617CamR ARO83LEE等[9]pBL_618CamR ARO93LEE等[9]pBL_621CamR ARO103LEE等[9]pBL_622CamR THI33LEE等[9]pBL_623CamR PDC13LEE等[9]pBL_624CamR PDC53LEE等[9]pBL_625CamR PDC63LEE等[9]pBL_626CamR ADH13LEE等[9]pBL_627CamR ADH23LEE等[9]pBL_628CamR ADH33LEE等[9]pBL_629CamR ADH43LEE等[9]pBL_630CamR ADH53LEE等[9]pBL_631CamR SFA13LEE等[9]pYTK047GFP dropout234rLEE等[9]pYTK052tSSA14LEE等[9]pYTK074URA36LEE等[9]pYTK076HIS36LEE等[9]pYTK079HygRomycinR6LEE等[9]pYTK086URA3 3′ Homology7LEE等[9]pYTK088HO 3′ Homology7LEE等[9]pYTK100chrXI 3′ homology7本研究构建pYTK090KanR-ColE18aLEE等[9]pYTK092URA3 5′ Homology8bLEE等[9]pYTK094HO 5′ Homology8bLEE等[9]pYTK101chrXI 5′ homology8b本研究构建pBL505Integration(URA3-loci) KanR-ColE1 URA3 GFP dropoutYTK008,YTK047,YTK073,YTK074,YTK086,YTK090,YTK092本研究构建pBL602Integration(HO-loci) KanR-ColE1 HIS3 HygR dropoutYTK008,YTK047,YTK073,YTK076,YTK088,YTK090,YTK094本研究构建pBL638Integration(Chr11-loci) KanR-ColE1 HYG GFP dropoutYTK008,YTK047,YTK073,YTK079,YTK100,YTK090,YTK101本研究构建pBL804Integration(URA3-loci) KanR URA3 pTDH3-ARO8-tSSA1pBL505,YTK009,pBL617,YTK052本研究构建pBL805Integration(URA3-loci) KanR URA3 pTDH3-ARO9-tSSA1pBL505,YTK009,pBL618,YTK052本研究构建pBL806Integration(HO-loci) KanR HIS3 pTDH3-ARO10-tSSA1pBL505,YTK009,pBL621,YTK052本研究构建pBL807Integration(HO-loci) KanR HIS3 pTDH3-THI3-tSSA1pBL505,YTK009,pBL622,YTK052本研究构建pBL808Integration(HO-loci) KanR HIS3 pTDH3-PDC1-tSSA1pBL505,YTK009,pBL623,YTK052本研究构建pBL809Integration(HO-loci) KanR HIS3 pTDH3-PDC5-tSSA1pBL505,YTK009,pBL624,YTK052本研究构建pBL810Integration(HO-loci) KanR HIS3 pTDH3-PDC6-tSSA1pBL505,YTK009,pBL625,YTK052本研究构建pBL811Integration(Chr11-loci) KanR HygR pTDH3-ADH1-tSSA1pBL638,YTK009,pBL626,YTK052本研究构建pBL812Integration(Chr11-loci) KanR HygR pTDH3-ADH2-tSSA1pBL638,YTK009,pBL627,YTK052本研究构建pBL813Integration(Chr11-loci) KanR HygR pTDH3-ADH3-tSSA1pBL638,YTK009,pBL628,YTK052本研究构建pBL814Integration(Chr11-loci) KanR HygR pTDH3-ADH4-tSSA1pBL638,YTK009,pBL629,YTK052本研究构建pBL815Integration(Chr11-loci) KanR HygR pTDH3-ADH5-tSSA1pBL638,YTK009,pBL630,YTK052本研究构建pBL816Integration(Chr11-loci) KanR HygR pTDH3-SFA1-tSSA1pBL638,YTK009,pBL631,YTK052本研究构建
1.4 菌株构建
本研究所涉及的菌株均列于表3中。酿酒酵母感受态的制作:挑取单菌落过夜培养,次日以OD600=0.2接种于50 mL在250 mL锥形瓶新鲜的YPD培养基中,培养至OD600=1.2~1.5,离心弃上清液,用无菌冰水清洗 2次,0.1 mol/L醋酸锂清洗1次,将菌体重悬在400 μL 0.1 mol/L的醋酸锂中,然后50 μL分装;对分装体系高速离心30 s弃去上清液,向菌体中依次加入240 μL EG 3 350,36 μL 1 mol/L LiAc,1 μg质粒或者片段,50 μL 2.0 mg/mL的Single-stranded carrier DNA,用水补足360 μL体系,充分混匀后在30 ℃水浴30 min,42 ℃水浴25 min,最后离心后用1 mL无菌水重悬,取50~100 μL涂布到对应的缺陷型或者含有抗性的平板上。
表3 本研究使用的菌株
Table 3 Strains used in this study
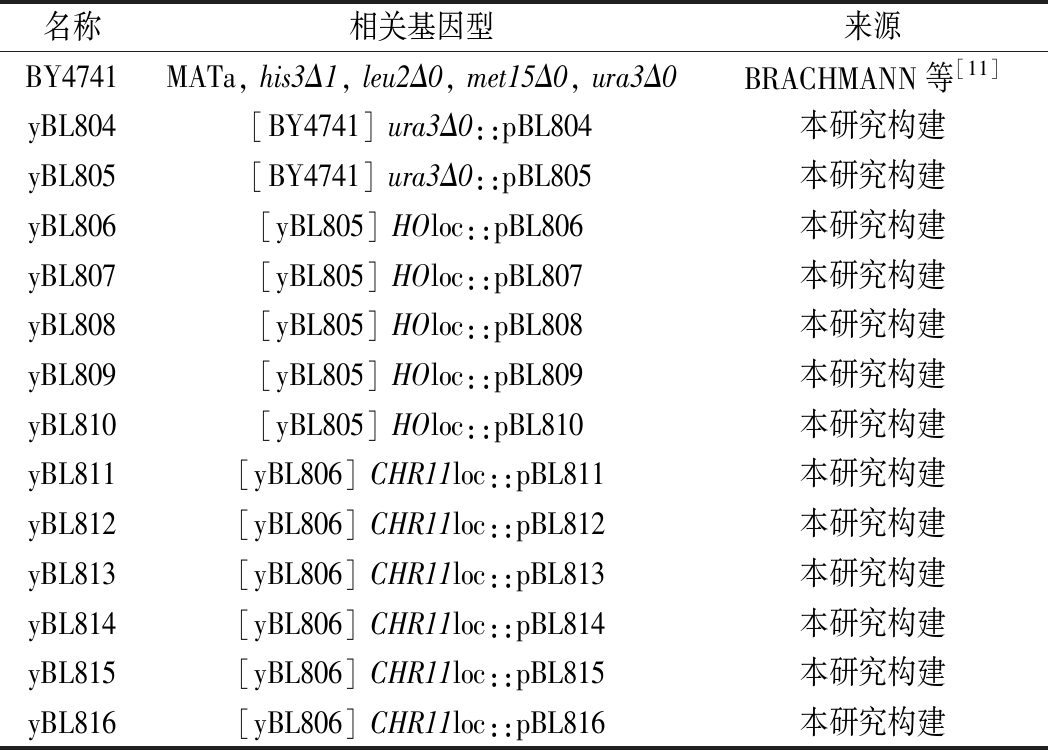
名称相关基因型来源BY4741MATa, his3Δ1, leu2Δ0, met15Δ0, ura3Δ0BRACHMANN等[11]yBL804[BY4741] ura3Δ0::pBL804本研究构建yBL805[BY4741] ura3Δ0::pBL805本研究构建yBL806[yBL805] HOloc::pBL806本研究构建yBL807[yBL805] HOloc::pBL807本研究构建yBL808[yBL805] HOloc::pBL808本研究构建yBL809[yBL805] HOloc::pBL809本研究构建yBL810[yBL805] HOloc::pBL810本研究构建yBL811[yBL806] CHR11loc::pBL811本研究构建yBL812[yBL806] CHR11loc::pBL812本研究构建yBL813[yBL806] CHR11loc::pBL813本研究构建yBL814[yBL806] CHR11loc::pBL814本研究构建yBL815[yBL806] CHR11loc::pBL815本研究构建yBL816[yBL806] CHR11loc::pBL816本研究构建
1.5 菌株的表征
生长速度是从独立的生物副本中生长的好氧间歇培养获得的。用同一培养基和温度的指数生长细胞接种含有2 mL YPD或者SC带档板的24孔板中。
在生产性研究中,所有菌株均以OD600=0.2开始接种发酵。根据耗尽所有葡萄糖所需的时间,细胞统一生长36 h。培养液在13 500×g下离心5 min,上清液在-20 ℃下冷冻。
400 μL的培养上清液与等量的无水乙醇混合,充分混匀,在13 500×g下离心5 min。将上清液用于芳香族化合物的分析。使用高效液相色谱对上清液进行检测,使用岛津Shim-pack GIST C18柱,柱温箱温度30 ℃,流速1 mL/min,进样量10 μL,使用紫外检测器在215 nm和276 nm进行检测,洗脱程序采用梯度法,以20 mmol/L磷酸二氢钾(pH=3.0)含1%(体积分数)乙腈(A)和乙腈(B)为溶剂,程序从0%增加到10%(体积分数)溶剂B (0~6.0 min),然后将其体积分数从10%线性增加到60% (6.0~25 min),然后将体积分数从60%降低到0% (25~26 min),Trp在11 min出峰,对羟基苯乙醇 (p-hydroxy-phenylethanol,pPET) 在13.5 min出峰,苯乙醇(phenylethanol,PET)在19 min出峰,IET在19.75 min出峰。
光密度测量使用紫外可见分光光度计759s(600 nm)。
2 结果与分析
2.1 IET在酿酒酵母中合成途径
IET的合成途径(图1)与Ehrlich途径的过程保持高度一致,首先是带有转氨基作用的双功能2-氨基己二酸转氨酶ARO8(Gene ID:852672)和芳香族氨基酸2-酮戊二酸转氨酶ARO9(Gene ID:856539)[12-14]将Trp转化为吲哚丙酮酸(indole-3-pyruvate ,IPY);之后在脱羧酶的作用下转化为吲哚乙醛(indole-3-acetaldehyde,IAC),能在这个过程中发挥作用的基因有编码吲哚丙酮酸脱羧酶1的PDC1(Gene ID:850733),编码吲哚丙酮酸脱羧酶5的PDC5(Gene ID:850825),编码吲哚丙酮酸脱羧酶6的PDC6(Gene ID:852978)[15],还有苯丙酮酸脱羧酶的ARO10(Gene ID:851987)和支链-2-含氧酸脱羧酶的THI3(Gene ID:851479)[6,16];可催化最后一步IAC到IET的有乙醇脱氢酶1 ADH1(Gene ID:854068)、乙醇脱氢酶2 ADH2(Gene ID:855349)、乙醇脱氢酶3 ADH3(Gene ID:855107)、乙醇脱氢酶4 ADH4(Gene ID:852636)、乙醇脱氢酶5 ADH5(Gene ID:852442)和编码双功能醇脱氢酶的基因SFA1(Gene ID:851386)[17]。其中大多数酶是泛底物酶,在酿酒酵母体内催化多个同一类型的反应,因此有必要探索更合适IET生产的酶。
2.2 酿酒酵母发酵生产IET过程的基本表征
选取酿酒酵母菌株BY4741进行实验,对Trp进入代谢的关键酶基因ARO8和ARO9进行了过表达,获得菌株yBL804、yBL805,进行了不同菌株在YPD富营养培养基中生产IET的实验,并且为了获得更好的检测效果,进行了不同时间的取样。结果如图2-a所示,ARO8和ARO9的过表达有助于菌株的生长,可见菌株的生长在36 h达到顶峰,野生型菌株和实验组菌株生长OD600值分别到达10和25左右,实验组菌株的OD600值比野生型增加约1.5倍。同时检测了IET的产量,如图2-b所示,实验组菌株IET的生产在24 h便趋于稳定,达到0.07 mmol/L,是对照组的2倍。在后续实验中,选定36 h作为之后所有实验的取样时间。过表达菌株的IET产量有明显的提高,但是相对应的菌株生长状况在YPD培养基中得到了加强,但是YPD培养基的成分过于复杂,并不适合检测代谢过程中的其他物质,而且需要后续Trp添加实验进一步确认过表达的效果。
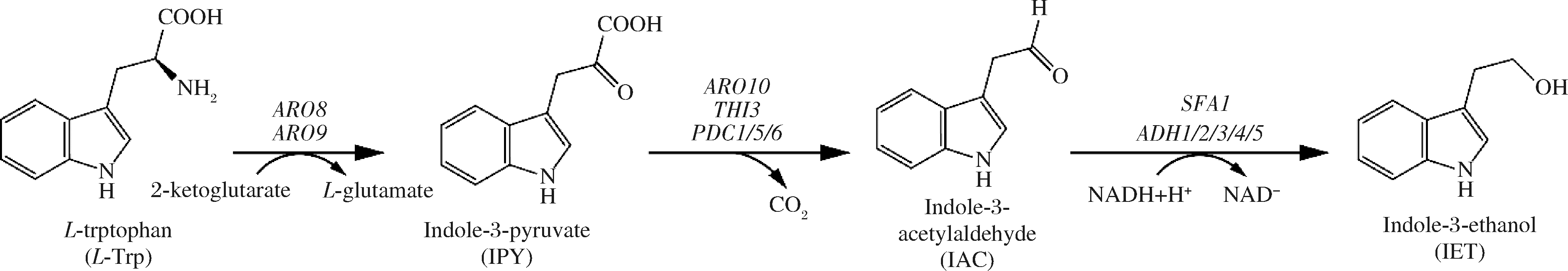
图1 从Trp到IET的代谢途径及相关基因
Fig.1 Metabolic pathway and related genes from tryptophan to tryptophan
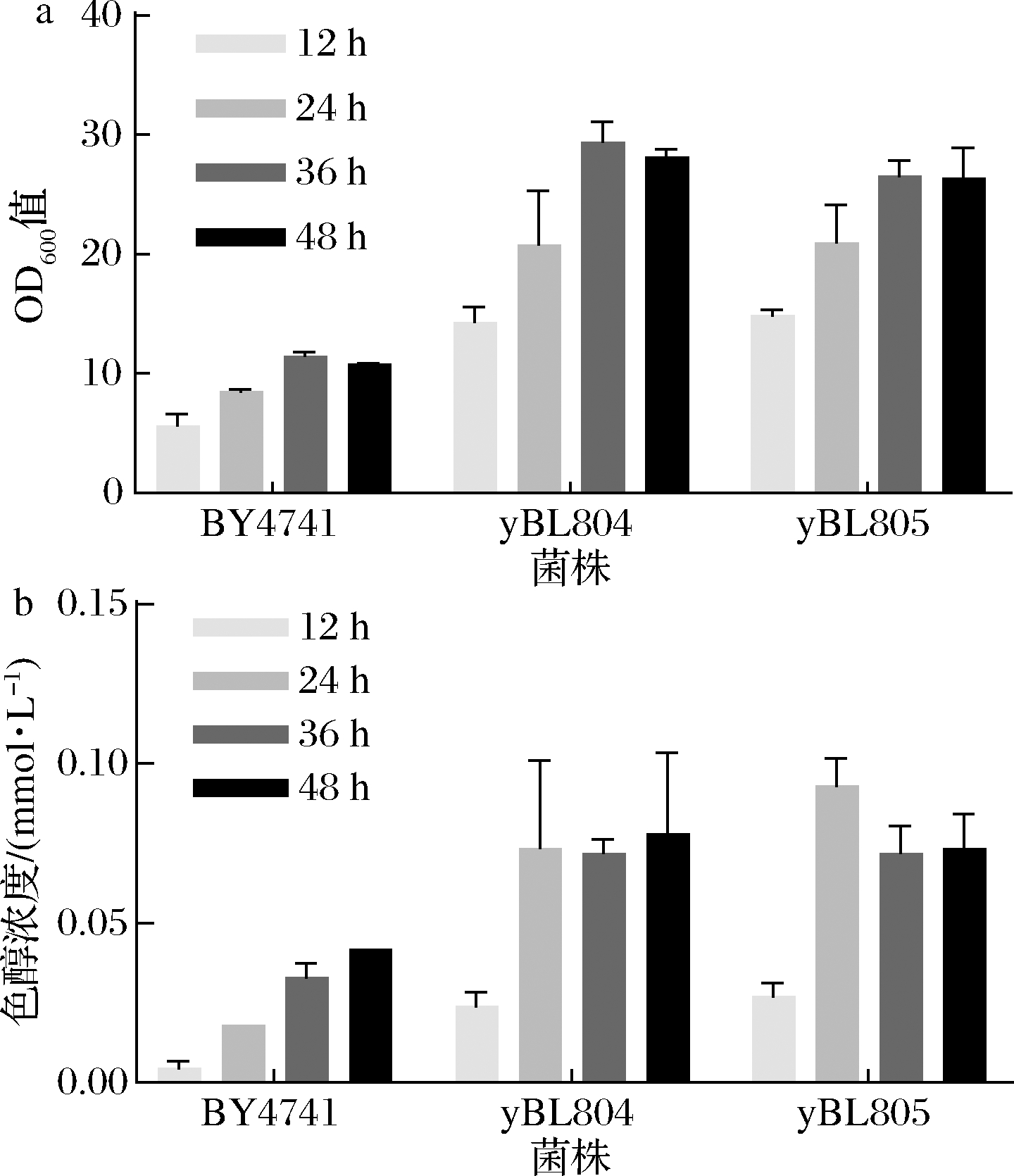
a-菌株不同时间的生长密度;b-对应于不同取样时间的IET产量
图2 BY4741过表达ARO8或ARO9的简单表征
Fig.2 Simple characterization of BY4741 overexpressing ARO8 or ARO9
2.3 添加Trp生产IET
通过向发酵液中添加定量(5 mmol/L)的Trp,检测yBL804、yBL805菌株的IET产量,菌株生长状况如图3-a所示,出于2.2中检测的需要,改用了SC培养基,各菌株的生长不再表现出较大差异,OD600值稳定在10左右。相对应的IET产量如图3-b所示,yBL805 IET产量达到0.58 mmol/L,相比于原始菌株提升47%,作为后续改造的出发菌株。
因为在酿酒酵母分支酸途径中存在苯丙氨酸和酪氨酸的竞争途径,我们同时检测了其中苯丙氨酸和酪氨酸的代谢产物PET和pPET含量,如图3-c所示,PET含量在实验组和对照组都维持在0.08 mmol/L,而pPET含量实验组比对照组下降了36%,综合来看,流向苯丙氨酸和酪氨酸途径的碳流也有一定的减少,说明IET产量的提升也与流向Trp途径的碳流量增加有一定关系。如图3-d所示,对发酵过后Trp的剩余量进行测定,yBL804和yBL805相较于BY4741多消耗Trp 0.23 mmol/L和0.35 mmol/L,这与IET的产量增加呈正相关,说明改造起到了效果。综上,改造通过消耗Trp使更多的碳流量流向Trp分支,并且产量的提升比Trp的消耗要多,表明IET的产生是本身产生的Trp和外源添加Trp的共同作用。
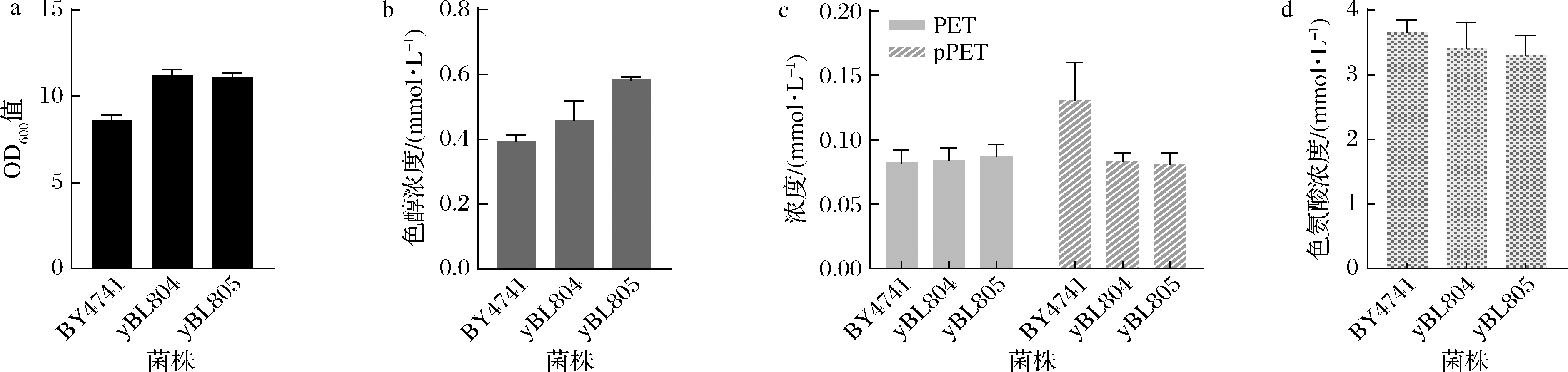
a-36 h菌株的生长密度;b-对应菌株的IET产量;c-菌株副产物PET和pPET的量;d-发酵结束后Trp的剩余量
图3 BY4741过表达ARO8或ARO9
Fig.3 BY4741 overexpressing ARO8 or ARO9
2.4 从IPY到IAC相关基因的过表达
向yBL805菌株转入IPY到IAC的5个相关基因,如图4-a所示,每一个基因都未对菌株生长造成明显的影响。各菌株在IET的生产中表现出了较为显著的差异,如图4-b所示,所有菌株均在IET生产上表现出提升,其中过表达ARO10的菌株yBL806的IET产量相比于yBL805提升51%。如图4-c所示,pPET含量相比于yBL805进一步下降,进一步证实之前的结论。同时如图4-d所示,Trp的消耗与IET的生成也有很好的正相关的关系。yBL806将被用来进行下一步基因操作。
2.5 从IAC到IET相关基因的过表达
向yBL806菌株转入IAC到IET的6个相关基因,如图5-a所示,菌株生长状况基本一致。各菌株在IET的生产中大部分菌株并未表现出较为显著的差异,如图5-b所示,其中产量提升最多的过表达SFA1的菌株yBL816产量提升也只有3%。如图5-c所示,pPET含量出现了进一步下降,但是幅度很小。同时如图5-d所示,Trp的消耗与IET的生成也有很好的对应关系。yBL816检测出最高产量为1.09 mmol/L,但是综合来看生长状况和各相关化合物的量都没有产生明显的变化,说明了此步骤并非整个过程的限速步骤。

a-36 h菌株的生长密度;b-对应菌株的IET产量;c-菌株副产物PET和pPET的量;d-发酵结束后Trp的剩余量
图4 从IPY到IAC相关基因的过表达
Fig.4 Overexpression of genes related to IAC from IPY
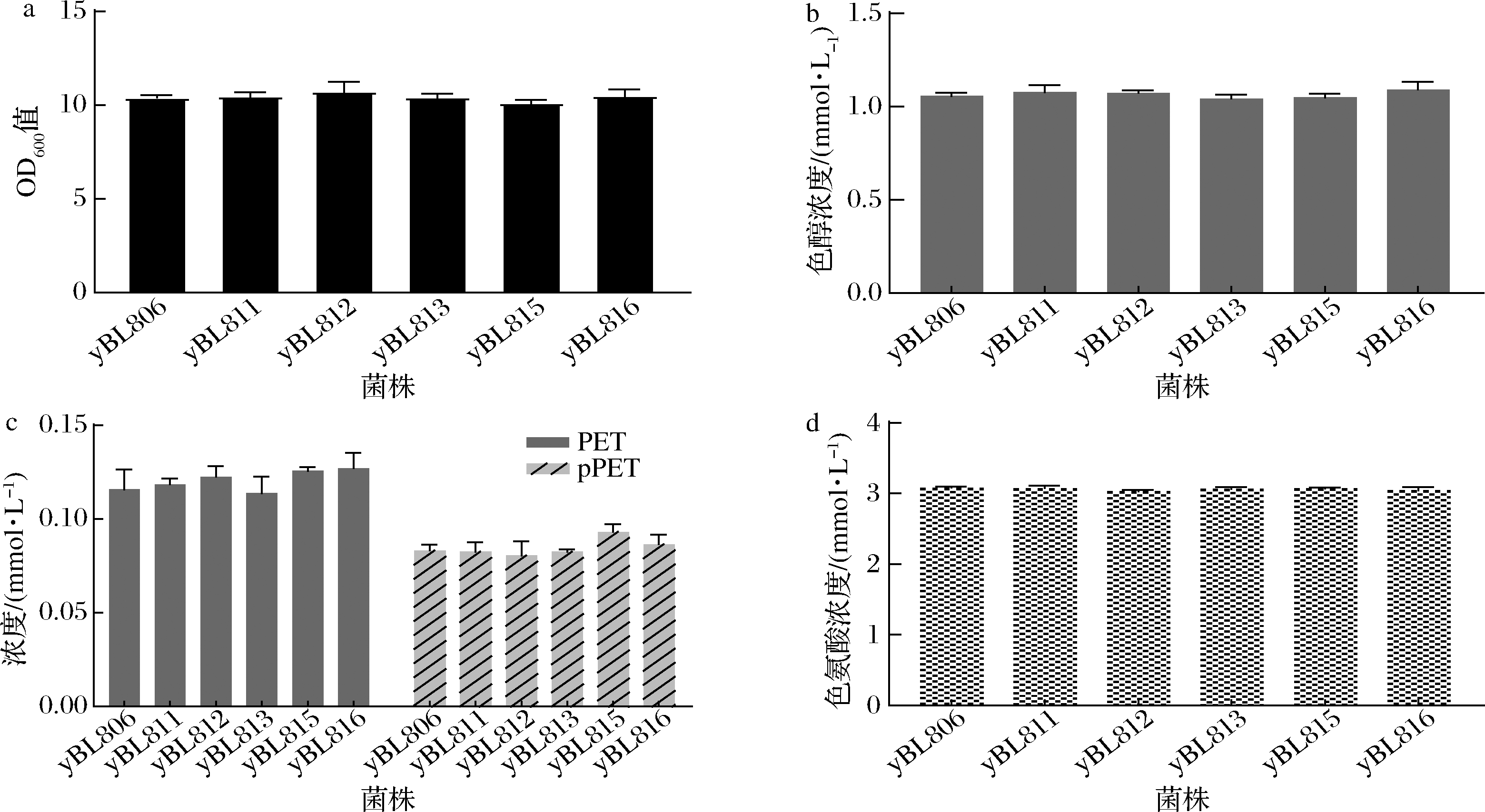
a-36 h菌株的生长密度;b-对应菌株的IET产量;c-菌株副产物PET和pPET的量;d-发酵结束后Trp的剩余量
图5 从IAC到IET相关基因的过表达
Fig.5 Overexpression of genes related to IET from IAC
3 结论与讨论
经过对整个Trp到IET相关的13个基因的分步探究之后,发现从Trp到IPY的2个相关基因中,ARO9基因有更好的效果;从IPY到IAC相关的5个基因中,ARO10的效果最好;从IAC到IET相关的6个基因中,SFA1的效果最好。那么在Trp到IET的生物过程中,ARO9、ARO10和SFA1是最好的生产组合。其中限制Trp到IET的关键步骤为Trp到IAC的2个步骤,从IAC到IET的基因并没有较大作用。可以得出在整个IET的生产过程中,限速步骤便是3个步骤的前2个步骤。本研究开创性的使用IET、PET和pPET来反应碳流在3个芳香族氨基酸分支的流量大小,这是之前的研究未曾涉及的方面。由于色氨酸分支涉及的酶的种类过多,对于酿酒酵母中色氨酸分支的研究很少,关于IET生产的文章更是难以寻见,本研究使生物生产的转化率达到21.8%,可以参考酪氨酸和苯丙氨酸的改造方法进一步提高得率。
在进行最开始的实验表征时,转化菌株表现出过高的生长密度,这是因为培养基为富营养型,而ARO8和ARO9又是很多转氨作用的关键酶[18],所以会表现出更好的生长情况。之后为了排除培养基对检测其他物质的影响,使用了SC培养基,菌株的长势便趋于一致。
不同基因的组合可能有更好的效果,而且对于不同基因使用不同启动子来控制酶的表达[19]也有可能获得更高的IET产量,另外添加更多Trp也可能造成更高的胁迫以提高IET的产率[20],以上皆可继续进行研究。
最后,相比于添加Trp生产IET,通过代谢工程来增加Trp的产量从而实现碳源到IET的从头生产是更为绿色和环保的方式[21],但是所涉及的途径与基因过多,基本上涉及色氨酸分支的改造都不涉及上游Trp通量的改造[22],这可能成为之后的一个研究方向。
[1] GUTMANN B, GOTTSPONER M, ELSNER P, et al.On the fischer indole synthesis of 7-ethyltryptophol-mechanistic and process intensification studies under continuous flow conditions[J].Organic Process Research & Development, 2013, 17(2):294-302.
[2] PIERCE V, SHEPPERSON N B, TODD M H, et al.Investigation into the cardioregulatory properties of the alpha 1-adrenoceptor blocker indoramin[J].British Journal of Pharmacology, 1986, 87(2):433-441.
[3] DUBHASHE Y R, SAWANT V M, GAIKAR V G.Process intensification of continuous flow synthesis of tryptophol[J].Industrial & Engineering Chemistry Research, 2018, 57(8):2 787-2 796.
[4] RIBÉREAU-GAYON P, SAPIS J C.On the presence in wine of tyrosol, tryptophol, phenylethyl alcohol and gamma-butyrolactone, secondary products of alcoholic fermentation[J].mSystems, 1965, 261(8):1 915-1 916.
[5] EHRLICH F.über die bedingungen der fuselölbildung und über ihren zusammenhang mit dem eiweißaufbau der hefe[J].Berichte der deutschen chemischen Gesellschaft, 1907, 40(1):1 027-1 047.
[6] VURALHAN Z, LUTTIK M A, TAI S L, et al.Physiological characterization of the ARO10-dependent, broad-substrate-specificity 2-oxo acid decarboxylase activity of Saccharomyces cerevisiae[J].Applied and Environmental Microbiology, 2005, 71(6):3 276-3 284.
[7] STEWART G G.SACCHAROMYCES | Saccharomyces cerevisiae[M].Oxford:Academic Press, 2014.
[8] CHAN W T, VERMA C S, LANE D P, et al.A comparison and optimization of methods and factors affecting the transformation of Escherichia coli[J].Bioscience Reports, 2013, 33(6):e00086.
[9] LEE M E, DELOACHE W C, CERVANTES B, et al.A highly characterized yeast toolkit for modular, multipart assembly[J].ACS Synthetic Biology, 2015, 4(9):975-986.
[10] CAREY M F, PETERSON C L, SMALE S T.PCR-mediated site-directed mutagenesis[J].Cold Spring Harbor Protocols, 2013(8):738-742.
[11] BRACHMANN C B, DAVIES A, COST G J, et al.Designer deletion strains derived from Saccharomyces cerevisiae S288C:A useful set of strains and plasmids for PCR-mediated gene disruption and other applications[J].Yeast, 1998, 14(2):115-132.
[12] HAZELWOOD L A, DARAN J M, VAN MARIS A J A, et al.The Ehrlich pathway for fusel alcohol production:A century of research on Saccharomyces cerevisiae metabolism[J].Applied and Environmental Microbiology, 2008, 74(8):2 259-2 266.
[13] URRESTARAZU A, VISSERS S, IRAQUI I, et al.Phenylalanine- and tyrosine-auxotrophic mutants of Saccharomyces cerevisiae impaired in transamination[J].Molecular & General Genetics, 1998, 257(2):230-237.
[14] IRAQUI I, VISSERS S, CARTIAUX M, et al.Characterisation of Saccharomyces cerevisiae ARO8 and ARO9 genes encoding aromatic aminotransferases Ⅰ and Ⅱ reveals a new aminotransferase subfamily[J].Molecular & General Genetics, 1998, 257(2):238-248.
[15] TER SCHURE E G, FLIKWEERT M T, VAN DIJKEN J P, et al.Pyruvate decarboxylase catalyzes decarboxylation of branched-chain 2-oxo acids but is not essential for fusel alcohol production by Saccharomyces cerevisiae[J].Applied and Environmental Microbiology, 1998, 64(4):1 303-1 307.
[16] VURALHAN Z, MORAIS M A, TAI S L, et al.Identification and characterization of phenylpyruvate decarboxylase genes in Saccharomyces cerevisiae[J].Applied and Environmental Microbiology, 2003, 69(8):4 534-4 541.
[17] DICKINSON J R, SALGADO L E J, HEWLINS M J E.The catabolism of amino acids to long chain and complex alcohols in Saccharomyces cerevisiae[J].The Journal of Biological Chemistry, 2003, 278(10):8 028-8 034.
[18] KIM B, CHO B R, HAHN J S.Metabolic engineering of Saccharomyces cerevisiae for the production of 2-phenylethanol via Ehrlich pathway[J].Biotechnology and Bioengineering, 2014, 111(1):115-124.
[19] ZHANG J, PETERSEN S D, RADIVOJEVIC T, et al.Combining mechanistic and machine learning models for predictive engineering and optimization of tryptophan metabolism[J].Nature Communications, 2020, 11(1):4880.
[20] CAO M F, GAO M R, SU STEGUI M, et al.Building microbial factories for the production of aromatic amino acid pathway derivatives:From commodity chemicals to plant-sourced natural products[J].Metabolic Engineering, 2020, 58:94-132.
STEGUI M, et al.Building microbial factories for the production of aromatic amino acid pathway derivatives:From commodity chemicals to plant-sourced natural products[J].Metabolic Engineering, 2020, 58:94-132.
[21] FURUYA T, KINO K.Catalytic activity of the two-component flavin-dependent monooxygenase from Pseudomonas aeruginosa toward cinnamic acid derivatives[J].Applied and Environmental Microbiology, 2014, 98(3):1 145-1 154.
[22] WONG G, LIM L R, TAN Y Q, et al.Reconstituting the complete biosynthesis of D-lysergic acid in yeast[J].Nature Communications, 2022, 13(1):712.