豆豉是一种起源于我国几千年前的传统发酵豆制品,豆豉豉香浓郁,口感软硬适中,生产工艺分为前发酵和后发酵两个过程[1],豆豉类型主要分为4种:毛霉型、曲霉型、根霉型和细菌型[2]。利用微生物产生的蛋白酶分解豆类谷物中的蛋白,再经过酵母菌、乳酸菌等后期发酵,使豆豉具有丰富的营养和特殊的香味,从而使这种产品可作为高营养直接食用的食品[3-5]。目前,要实现中国的豆豉工业化生产仍存在许多问题,例如发酵豆豉的方式仍以自然发酵为主,由于微生物种类繁多可变导致产品质量不稳定,自然发酵豆豉中微生物发酵能力差异大、蛋白酶活性低。为解决此类问题,使豆豉进入规范化生产,提高质量,纯种发酵成为提升豆豉行业产品质量的热点。李小永等[6]从临沂细菌型豆豉中筛选出一株适于豆豉生产的高产蛋白酶菌种, 成品豆豉的理化指标和感官评定表明该菌种发酵的豆豉风味好且滋味鲜美;彭昀等[7]从庆阳豆豉中筛选主要作用微生物,获得优势发酵菌株,其蛋白酶和纤维素酶活性最高,分别达到了25.37 U/mL和6.015 U/mL;叶慧兰等[8]从广东阳江豆豉的曲醅中筛选出两株前发酵优势菌,以氨基态氮、还原糖、总酸含量与风味评定为指标,发现以米曲霉与米根霉混合制曲,在36 ℃条件下发酵56 h,生产出的豆豉品质最好。目前工业上豆豉纯种发酵主要集中在霉菌发酵剂[9-11],有关工业细菌发酵剂未见详细报道,筛选蛋白酶活性高的细菌发酵剂是提高工业化豆豉发酵的关键技术之一。
风味酶也是改良调味品质量的途径之一,能够增加鲜味以及脱去苦味。风味酶包含了内切蛋白酶与外切肽酶两种,可以用来去除低水解度产物的苦肽链,使产品鲜香味浓。如诺维信公司的风味酶被广泛应用于食品添加剂行业。风味酶的主要来源之一是各种发酵调味品的微生物菌株,通过基因重组技术对编码风味酶的基因进行高效表达,可以大量生产工业级别的风味酶、蛋白酶,从而用于提高调味品的质量。因此,含有编码风味酶基因的菌株将会拓宽优良发酵剂的研究、应用范围,也是筛选优良发酵剂关注的重点。
本课题组拟从风味良好的传统发酵豆豉中筛选出豆豉发酵剂,并通过全基因组测序及生物信息学分析验证其中存在的与风味相关的功能基因,为后续采用纯种发酵技术制备高品质豆豉,以及风味酶的克隆表达应用提供理论支持。试验首先主要以细菌型的各地豆豉为原料,以透明圈大小以及蛋白酶活力为判定指标,最终确定出高蛋白酶活性菌株,进行分子生物学鉴定以及酶学性质研究。在此试验基础上对该菌株进行全基因组测序分析其风味酶基因及其编码蛋白酶,进一步在基因水平上揭示风味产生的可能原因,旨在筛选出适宜豆豉工业化生产的潜力优良菌种。
1 材料与方法
1.1 材料与试剂
市售风味豆豉以及家庭自制豆豉采自肇庆、临沂、遵义、贺州、六安、南昌、苏州、武汉、北海、长寿、泸州、汉中、永川、潼川、太和、眉山16个地区,置于4 ℃冰箱中冷藏备用。细菌DNA提取试剂盒、通用引物27F、1492R、DNA Maker、Taq MasterMix、ddH2O,生工生物工程(上海)股份有限公司。其他试剂均为分析纯。
1.2 培养基
酪蛋白琼脂培养基(g/L):干酪素10,牛肉膏3,磷酸氢二钠2,氯化钠5,琼脂18,加热溶解,121 ℃高压蒸汽灭菌20 min。
酪素发酵培养基(g/L):干酪素5,葡萄糖1,酵母粉1,K2HPO4 4,KH2PO4 0.5,MgSO4 0.1,121 ℃高压蒸汽灭菌20 min。
LB液体培养基(g/L):胰蛋白胨10,氯化钠10,酵母粉5,121 ℃高压蒸汽灭菌20 min。
1.3 仪器与设备
LDZX-50KBS型立式压力蒸汽灭菌锅,上海申安医疗器械厂;A300型梯度PCR仪,杭州朗基科学仪器有限公司;7200型可见分光光度计,尤尼柯(上海)仪器有限公司;GHP-9050型隔水式恒温培养箱,上海一恒科学仪器有限公司。
1.4 高蛋白酶活菌株的筛选
1.4.1 初筛
将豆豉样品分别称取25 g,碾碎后加入装有225 mL无菌生理盐水中,充分振荡,得到10倍稀释的菌悬液,再依次用无菌生理盐水稀释得到10-2~10-6不同稀释倍数的菌悬液,选取10-2、10-4、10-6 3个梯度,进行3次平行重复涂布,待平板完全干透,将酪蛋白平板倒置于37 ℃培养箱分别培养24 h[12]。
1.4.2 复筛
从初筛所获得的细菌菌株甘油管中吸取100 μL的菌液接入10 mL的LB培养基中,在37 ℃摇床,150 r/min的条件下培养24 h,培养好的菌液按照2%的接种量接种到已灭菌的酪素发酵培养基中,在37 ℃,150 r/min条件下培养48 h,在离心机转速10 000 r/min的条件下离心10 min,取上清液,按照GB/T 23527—2009《蛋白酶制剂 蛋白酶活力的测定 福林法》测定该菌株蛋白酶活力[13]。
1.5 菌株形态观察及鉴定
对高蛋白酶活力菌株进行分离,挑取单菌落制作显微镜涂片。经革兰氏染色后,利用光学显微镜观察细胞的形态特征[14-15]。对已确定的高蛋白酶活力菌株进行基因组DNA抽提。PCR扩增:扩增体系25 μL,包括正向和反向引物各1 μL,模板DNA 2 μL,Taq MasterMix 12.5 μL,ddH2O 8.5 μL。扩增产物需要经过琼脂糖凝胶电泳的检测,获取成功扩增片段再送至生工生物工程(上海)股份有限公司测序。用所得序列在GenBank数据库中做相似性比对分析,进一步于软件MEGA中构建该菌株的系统发育树。
1.6 生长曲线及产酶曲线的测定
将已活化菌液按照2%的接种量接入已灭菌的酪素发酵培养基当中,在37 ℃、150 r/min条件下培养48 h。培养期间每隔4 h取样,测定其600 nm的吸光度(OD600)值,同时将菌液离心取上清液,测定蛋白酶活力。
1.7 蛋白酶酶学性质研究
1.7.1 蛋白酶最适温度测定
根据生长曲线和产酶曲线的绘制,菌株培养适宜发酵时间,设定不同温度条件与1%酪蛋白底物反应,试验设置20、30、40、50、60、70、80 ℃温度梯度,分别测定酶活力,确定反应最适温度。最高酶活力定义为100%[16]。
1.7.2 蛋白酶最适pH测定
在最适发酵时间和温度下,设定不同pH条件与1%酪蛋白底物反应,试验设置4.0、5.0、6.0、7.0、8.0、9.0、10.0 pH梯度,分别测定酶活力,确定反应最适pH。最高酶活力定义为100%[16]。
1.7.3 金属离子、表面活性剂和金属离子螯合剂对酶活力的影响
在最适时间、温度和pH值条件下,将Mn2+、Ca2+、Cu2+、Fe2+、Zn2+金属离子及EDTA和Triton X-100按照2 μmol/mL的浓度分别加入反应体系中,分别测定酶活力,检测金属离子和化学试剂对蛋白酶活性的影响。未添加金属离子、EDTA及Triton X-100的酶活力定义为100%[16]。
1.8 高蛋白酶活力菌株B3的全基因组测序
全基因组测序流程如下:首先收集纯化的DNA基因组,利用Covaris破碎机对纯化的DNA基因组进行破碎片段化,以便构建基因组文库。然后A、B接头连接DNA片段,去除接头自连片段,后经过氢氧化钠变性处理,产生单链DNA片段。将筛选的DNA片段的两端固定在芯片上,两端与引物互补,后经过PCR扩增过程。DNA单链在每次循环过程中加入带有荧光的4种dNTP之一,经过激光扫描识别以及切割基团后,恢复黏性末端,继续聚合,依据每次得到的荧光信号结果,即可得到模板DNA片段的序列。文库检验合格后,进行Illumina Hiseq×10平台测序,测序结果经过GC_depth分布分析和K-mer频率分布分析的基因组评估后,对二代测序后的优化序列采用组装软件SOAPdenovo2进行拼接,组装结果进一步的局部优化后,形成scaffolds开放阅读框。
生信分析过程如下:采用相应软件NR、Swissprot、KEGG、COG、GO、Ex PASy数据库以及PredictProtein(https://www.predictProtein.org/)网站、Clustal Omega、Jalview在线分析工具等分析预测风味酶基因及其编码蛋白酶的氨基酸序列组成、理化性质、高级结构,并构建系统发育树,进行多序列比对。
2 结果与分析
2.1 菌株的筛选
2.1.1 菌株初筛结果
对平板涂布得到的特征菌落进行多次划线分离纯化,共获得51株细菌菌株,对其进行透明圈与菌落直径大小的比值比较,筛选出10株透明圈与菌落直径比值>3.0的菌株(表1),并进行下一步复筛。
2.1.2 菌株复筛结果
经过48 h的发酵培养之后,对上述10株初筛菌株进行蛋白酶活力的测定,结果如图1所示。菌株在前发酵过程中产生大量不同的蛋白酶酶系,快速分解底物中的蛋白质。张芹等[17]以贵州三穗水豆豉为原料,分离出5株优势菌株,其中菌株F复筛蛋白酶活力达到60 U/mL左右。该研究以初筛菌株为基础,进行蛋白酶活力复筛。结果由图1可知,B3菌株在所有初筛菌株中蛋白酶活力最高,达到185.6 U/mL,因此选择该菌株进行后续试验。
表1 豆豉中产高蛋白酶活力菌株的初筛
Table 1 Preliminary screening of high protease activity strains in douchi
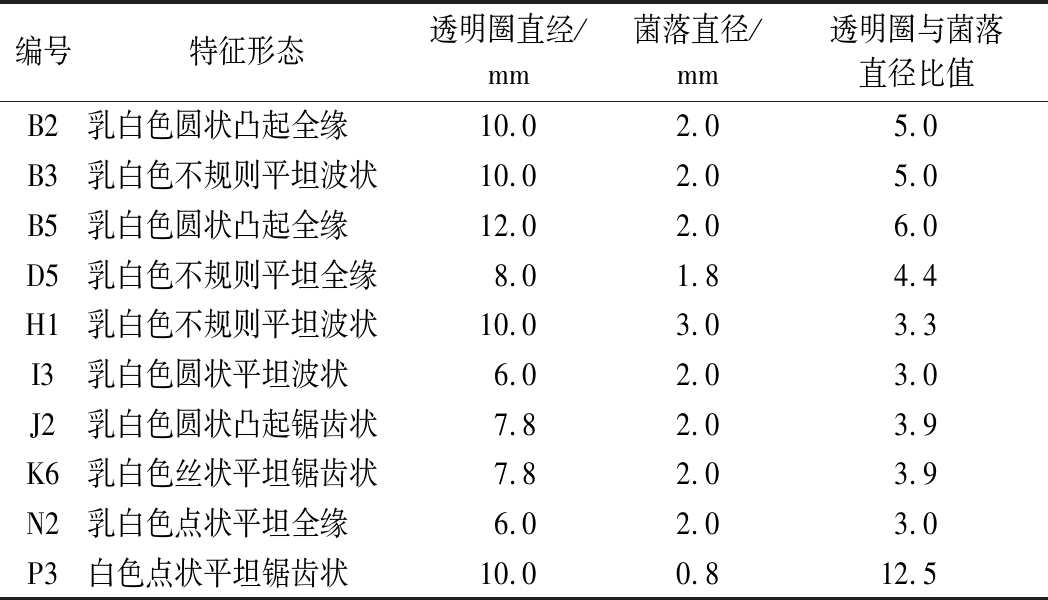
编号特征形态透明圈直经/mm菌落直径/mm透明圈与菌落直径比值B2乳白色圆状凸起全缘10.02.05.0B3乳白色不规则平坦波状10.02.05.0B5乳白色圆状凸起全缘12.02.06.0D5乳白色不规则平坦全缘8.01.84.4H1乳白色不规则平坦波状10.03.03.3I3乳白色圆状平坦波状6.02.03.0J2乳白色圆状凸起锯齿状7.82.03.9K6乳白色丝状平坦锯齿状7.82.03.9N2乳白色点状平坦全缘6.02.03.0P3白色点状平坦锯齿状10.00.812.5

图1 初筛菌株蛋白酶活力对比
Fig.1 Comparison of protease activity of primary strains
2.2 菌种鉴定
2.2.1 菌株菌落形态
复筛优势菌株B3的菌落形态如图2-a所示,平板上生长的菌落呈不规则状,颜色呈乳白色,表面呈凸面,边缘也呈现不规则状,水解透明圈明显,经过革兰氏染色后鉴定为革兰氏阳性菌,如图2-b所示,菌株呈短杆状,成簇或成链排列。

a-单菌落形态图;b-革兰氏染色图
图2 B3菌株形态特征
Fig.2 Morphological characteristics of strain B3
2.2.2 分子生物学鉴定
对B3菌株进行16S rDNA分子鉴定,PCR后系统发育树见图3。B3菌株通过凝胶电泳后在电泳图中出现明显的DNA分子条带,条带处于1 500 bp左右,属于大部分细菌分子质量的范围。利用NCBI网站的Blast功能对送测后获得的菌株序列进行序列比对,同时构建系统发育树了解该菌株的种属关系。建树结果表明该高产蛋白酶活力菌株与Bacillus amyloliquefaciens strain NBRC 15535的相似度高达92%。最终,B3菌株确定为芽孢杆菌属(Genbank 序列号 MZ723401.1)解淀粉芽孢杆菌种。
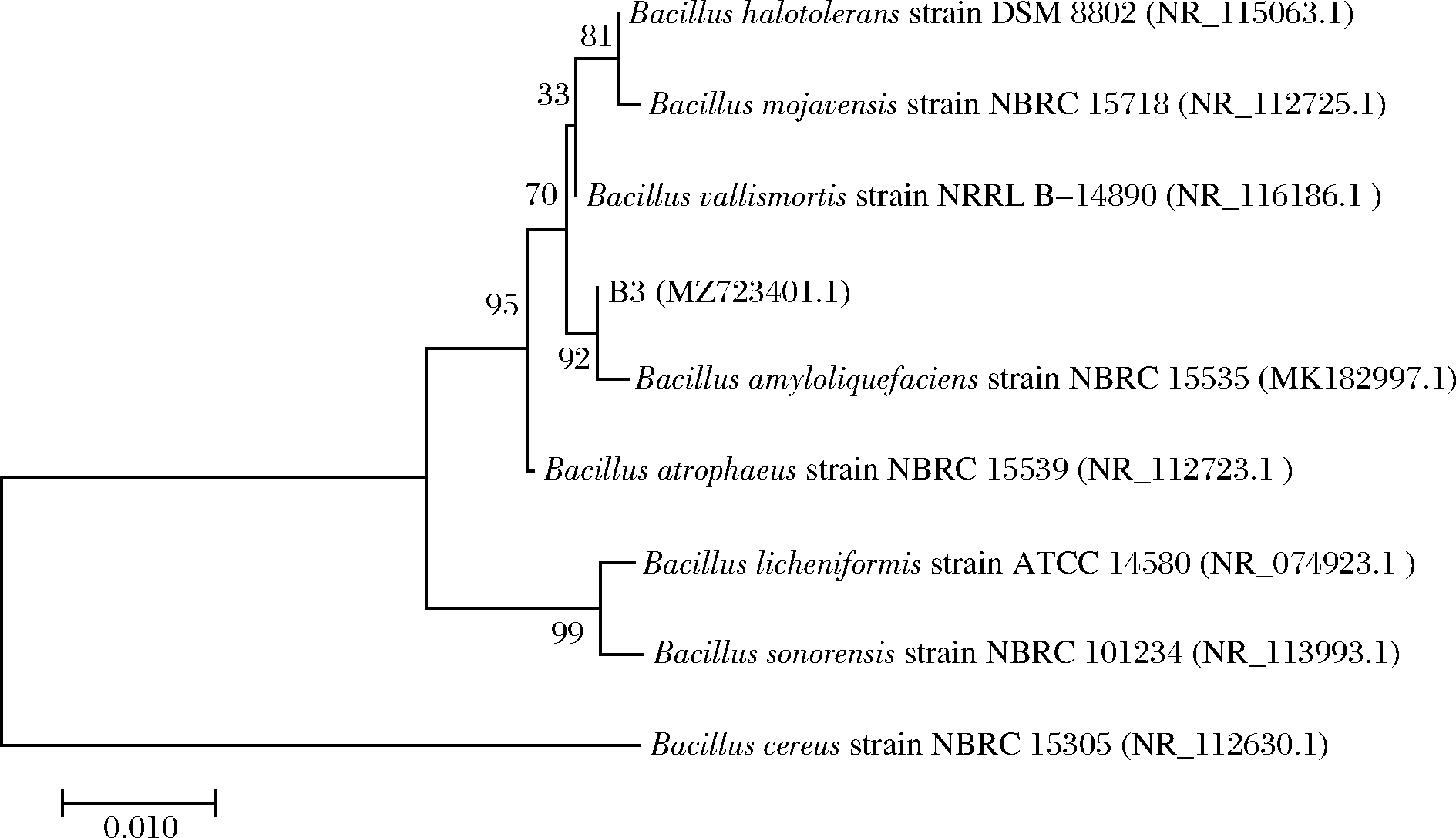
图3 基于菌株 B3 16S rDNA 序列的系统发育树
Fig.3 Phylogenetic tree based on the 16S rDNA sequnences of strain B3
2.3 生长曲线与产酶曲线的绘制
由图4可知,酶活力与生长曲线的变化趋势相似,从0~8 h增长缓慢,从12~36 h逐渐增长,之后酶活趋于平稳,在48 h达到最大值。从培养过程来看,B3菌株在36 h后菌体浓度已达到稳定状态,由于一些产物积累反馈,蛋白酶活也呈现不再增长的状态,所以在48 h的时候,是发酵的最佳时期。
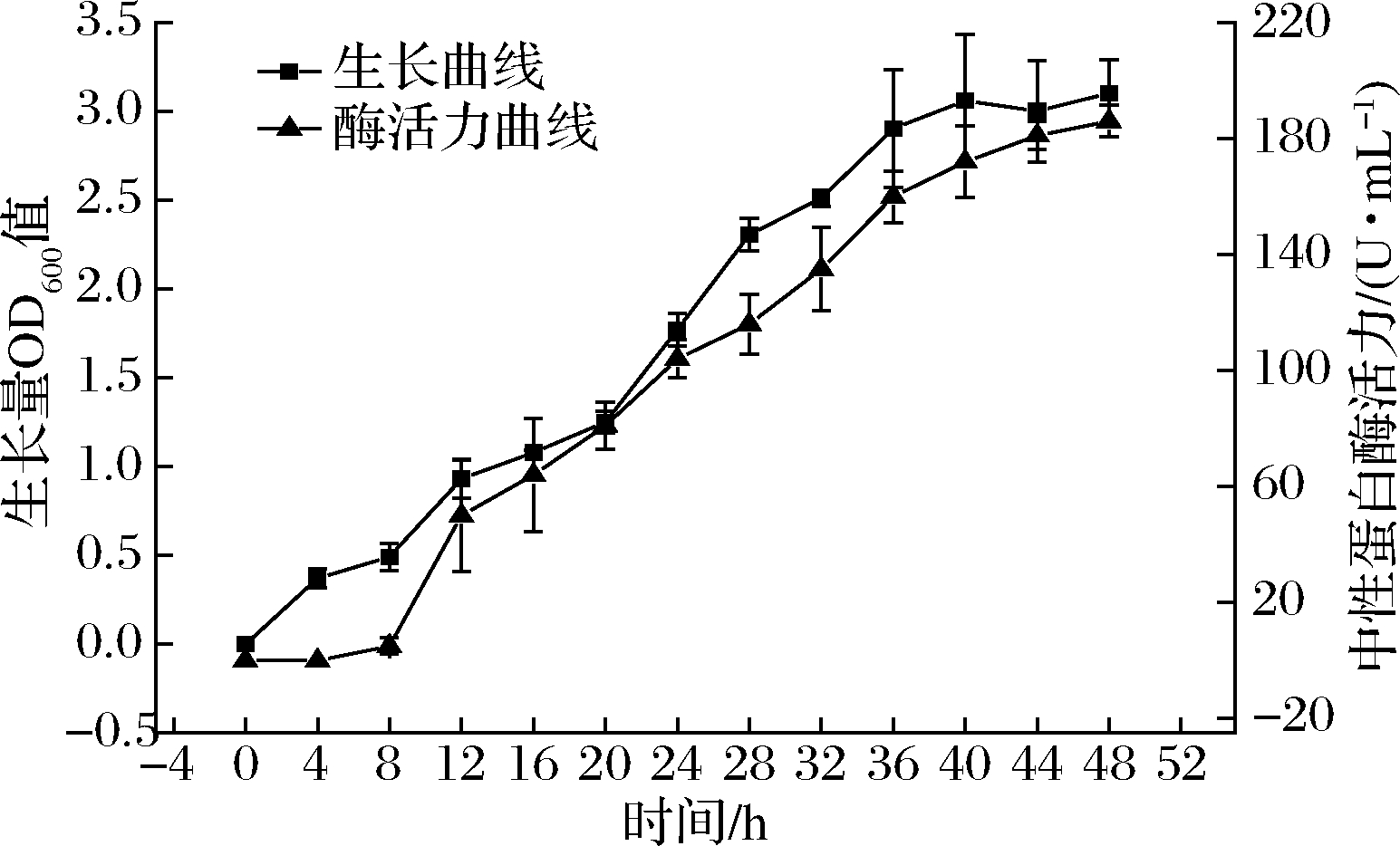
图4 B3菌株生长曲线及产酶曲线
Fig.4 Growth curve and enzyme production curve of strain B3
2.4 酶学性质分析
2.4.1 最适温度
不同温度对蛋白酶的影响结果如图5所示,20~50 ℃时,酶活力随温度的升高逐步上升,温度高于50 ℃后,酶活力迅速降低,50 ℃时达到最高酶活力。高温下,酶活力迅速降低,极可能是破坏了蛋白质结构,而低温下也有20%以上的酶活力存在,表明该菌株具有耐低温的潜在特性。

图5 反应温度对B3蛋白酶活力的影响
Fig.5 The effect of reaction temperature on the activity of B3 protease
2.4.2 最适pH
探究不同pH值对蛋白酶的影响,如图6所示,pH值在4.0~7.0时,酶活力随pH值的升高迅速上升,pH值在7.0~10.0,酶活力逐步降低,在7.0时达到最高酶活力。说明酶最适反应pH为7.0,而pH 7.0以上蛋白酶仍表现出61%以上活力。

图6 pH对B3蛋白酶活力的影响
Fig.6 The effect of pH on the activity of B3 protease
2.4.3 金属离子、表面活性剂和金属离子螯合剂对蛋白酶活性的影响
不同金属离子、EDTA、Triton X-100对蛋白酶的影响结果如图7所示,各种金属离子对酶活力的影响存在差异,仅有Mn2+对酶活力有极其明显的促进作用(P<0.01),相对酶活力可达116%,而Fe2+和EDTA对酶活力有极其明显的抑制作用(P<0.01),相对酶活力分别低至17%和23%。其余的Ca2+、Cu2+、Zn2+及Triton X-100对酶活力也具有抑制作用(P<0.05)。说明蛋白酶活力性受Fe2+和EDTA的影响最大,容易失去活性,其余因素影响下,蛋白酶活力也仍然在60%以上。
2.5 高蛋白酶活力菌株B3全基因组测序结果
通过高通量测序平台对B3基因组的测序发现,该优势菌株基因组全长为3 578 880 bp,获得注释基因3 851个,占基因组全长的89.44%,GC含量为46.16%。通过蛋白编码基因数据库GO、KEGG以及COG注释发现,该菌株参与碳水化合物运输和代谢、氨基酸转运和代谢、膜运输以及能源生产和转化的基因数目较多,这就能够解释该菌株高产蛋白酶能力的原因。
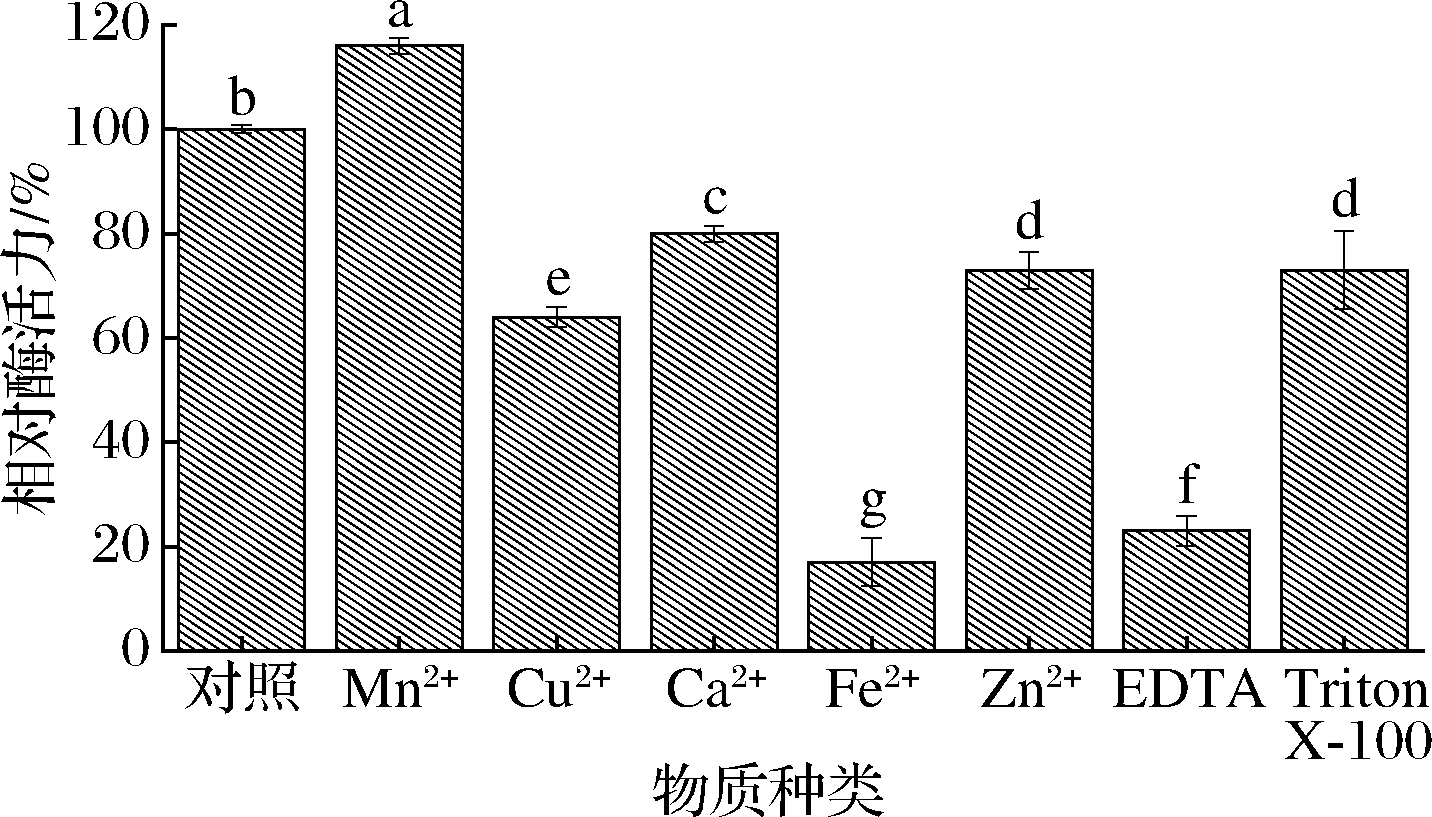
图7 金属离子、表面活性剂、和金属离子螯合剂对B3蛋白酶酶活力的影响
Fig.7 The effects of metal ions, surfactants, and metal ion chelating agents on the enzyme activity of B3 protease
2.5.1 COG功能注释
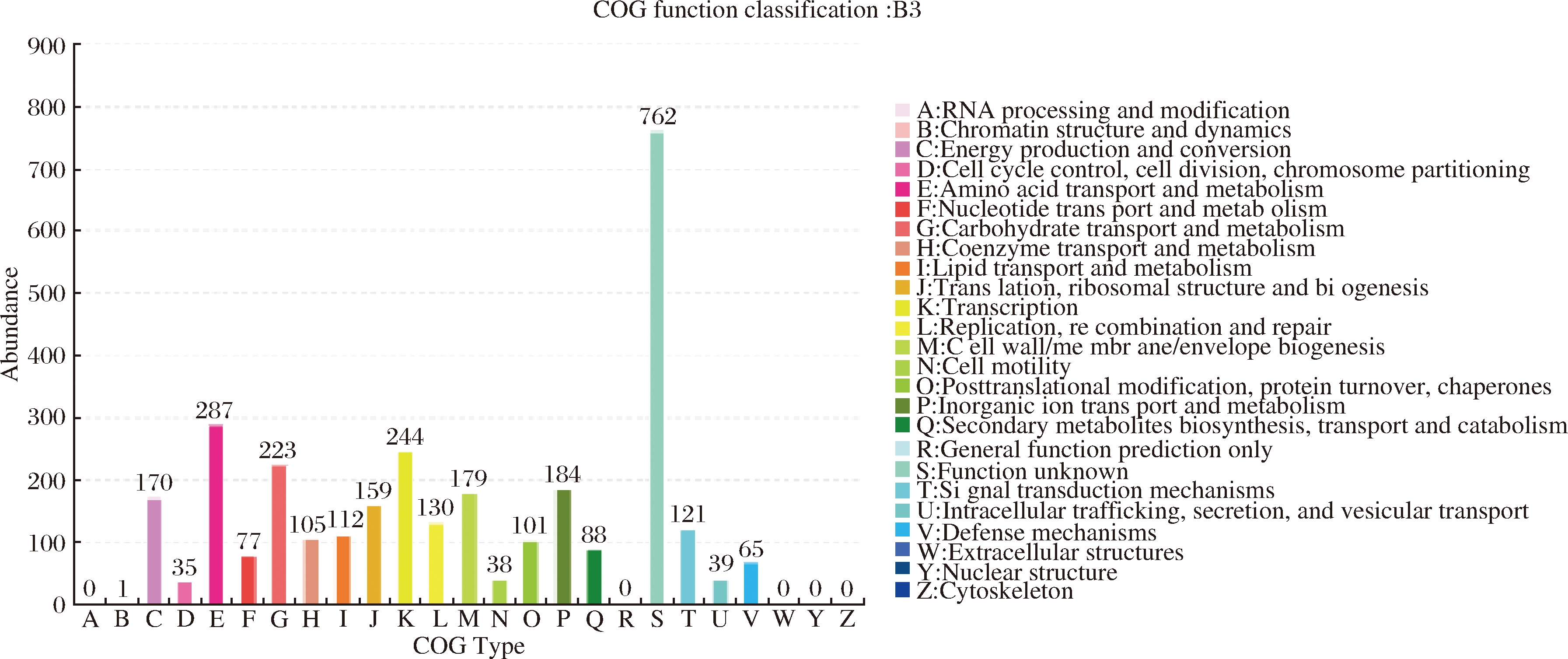
解淀粉芽孢杆菌B3基因组中有3 120个基因成功获得COG功能注释。占比较大的有:氨基酸代谢及转运(amino acid transport and metabolism),287个基因,占总比例的9.2%;碳水化合物转运和代谢(carbohydrate transport and metabolism),223个基因,占总比例的7.1%;转录(transcription),244个基因,占总比例的7.8%。相应结果见附图1(https://kns.cnki.net/kcms/detail/11.1802.TS.20220509.1434.004.html,下同)。
2.5.2 GO功能注释
解淀粉芽孢杆菌B3基因组中有2 666个基因在GO数据库中得到注释,由基因功能可分3大亚类:生物学过程(biological process),14个分支,其中基因数量最多的是转录调控,DNA模板(regulation of transcription, DNA-templated);细胞组分(cellular component),14个分支,其中基因数量最多的是膜的组成部分(integral component of membrane);分子功能(molecular function),14个分支,其中基因数量最多的是结合(binding)和催化活性(catalytic activity)。相应结果见附图2。
2.5.3 KEGG功能注释
对B3基因组进行KEGG注释,共成功获得2 530个基因注释。共分为6大部分代谢途径:细胞过程 (cellular processes,155个基因)、代谢(metabolism,1 759个基因)、人类疾病(human diseases,95个基因)、基因信息处理(genetic informantion processing,189个基因)、有机体系(organismal system,44个基因)、环境信息处理(environmental informantion processing,288个基因)。其中大量的基因参与了代谢中的全局组成部分。相应结果见附图3。
2.6 高蛋白酶活力菌株B3风味酶基因及其编码蛋白酶的预测分析
碱性蛋白酶具有很强的水解能力,并且豆豉中的植物蛋白碱溶性较好,所以碱性蛋白酶有较高的水解效率。碱性蛋白酶多应用于动植物蛋白的加工,以改善蛋白食品的品质和风味,主要是以芽孢杆菌属的碱性蛋白酶为主,通常是丝氨酸蛋白酶[18]。但是在豆豉发酵的过程中,蛋白质被水解,会产生较多的苦味肽,从而刺激味蕾,产生苦味,导致食品口感及品质下降。因此如何在提高豆豉风味的同时脱去苦味,使得豆豉风味更进一步,这个问题显得尤为重要[19]。
氨肽酶是一种外肽酶,这种酶催化肽和蛋白质N端氨基酸残基的水解。在食品工业中使用的氨肽酶也称为风味酶(novo nordisk)。不同来源的微生物氨肽酶结构均不相同且酶学性质也因底物的特异性而不同,绝大多数微生物氨肽酶的最适反应pH一般在碱性范围内(pH 6.0~9.0)。氨肽酶在食品行业中广泛应用于改善食品风味和口感,且在蛋白水解液脱苦中发挥重要作用。在食品中添加氨肽酶可以降解具有苦味的疏水性肽,并且释放其他具有良好风味特征的肽和游离氨基酸[20]。因此氨肽酶经常作为调味品的添加剂,如酱油,调味料混合物等。
促进豆豉发酵成熟,同时增强鲜味和脱除苦味是发酵剂中风味酶所起的主要作用。根据2.4.2中的菌株B3蛋白酶活力测定得知,菌株B3蛋白酶的最适pH值为7.0~9.0,并由此前研究可知,内切蛋白酶——碱性丝氨酸蛋白酶对发酵豆豉中的蛋白质水解有较高效率并可改善其风味,提高鲜味;外切肽酶——氨肽酶可以降解具有苦味的疏水性肽,两种风味酶的最适pH也为7.0~9.0,表明它们均能适应大部分的外界环境,维持稳定状态。本试验通过对菌株B3进行全基因组测序,检索到编码碱性丝氨酸蛋白酶的AprX基因和编码氨肽酶的ampS基因,为该发酵剂产生良好风味从基因层面提供理论验证。除此之外,利用生物信息学方法[21-22]预测分析其编码的碱性丝氨酸蛋白酶和氨肽酶的氨基酸组成、理化性质、高级结构等特性,也为后续风味酶基因的克隆、表达、菌株改良提供理论依据。
2.6.1 风味酶基因AprX和ampS检索及其编码的氨基酸序列保守域预测
(1)编码碱性丝氨酸蛋白酶的AprX基因
由全基因组测序得该基因为一条全长为1 329 bp的序列,经软件分析,该序列是一个完整的阅读框,起始密码子为ATG,终止密码子为TAA,共编码442个氨基酸。将该序列进行Blastx检索,发现该序列与解淀粉芽孢杆菌组(Bacillus amyloliquefaciens group)中所编码的丝氨酸蛋白酶(序列ID:WP_017417817.1)序列相似性为100%,将此氨基酸序列提交到Conserved Domains检测保守结构域,结果表明该序列的保守结构域为含有与胰蛋白酶不同源的Asp/His/Ser催化三联体。
(2)编码氨肽酶的ampS基因
由全基因组测序得到ampS基因核苷酸序列和氨基酸序列。通过NR、Swissprot、KEGG、COG、GO数据库对ampS基因进行基因注释,得到该基因编码的氨肽酶2(aminopeptidase 2)。由全基因组测序得该基因为一条全长为1 233 bp的序列,经软件分析,该序列是一个完整的阅读框,起始密码子为ATG,终止密码子为TAA,共编码410个氨基酸。将该序列进行Blastx检索,发现该序列与解淀粉芽孢杆菌组(Bacillus amyloliquefaciens group)中所编码的氨肽酶(序列ID:WP_033574610.1)序列相似性为99.76%,将此氨基酸序列提交到Conserved Domains检测保守结构域,结果表明该序列的保守结构域与嗜热金属蛋白酶氨肽酶相似,该酶属于肽酶M29家族的氨肽酶具有助催化金属离子,主要是钴,并显示出广泛的底物特异性。
2.6.2 风味酶基因AprX和ampS编码氨基酸序列的理化性质预测
(1)AprX基因编码氨基酸序列的理化性质预测
该基因共有442个氨基酸,带负电荷的残基总数为54,带正电荷的残基总数为48,氨基酸中所占比重最高的是丝氨酸(Ser),比重为8.6%;理论分子质量为48 176.44 u,理论等电点(PI)为5.98,分子式为C2116H3354N584O666S17;在溶液中的不稳定指数为32.45,小于阈值40,稳定性较好。脂溶指数为80.07,且通过Ex PASy的Prot Scale预测,AprX基因编码氨基酸序列疏水性最小值约为-3.000,最大值约为2.756,整个多肽链中亲水性氨基酸比疏水性氨基酸数量多,因此推测该蛋白为亲水性蛋白。
(2)ampS基因编码氨基酸序列的理化性质预测
该基因共有410个氨基酸,带负电荷的残基总数为62,带正电荷的残基总数为44,氨基酸中所占比重最高的是谷氨酸(Glu),比重为10.3%;理论分子质量为45 697.14 u,理论等电点为5.29,分子式为C2 038H3 150N548O634S7;在溶液中的不稳定指数为37.22,小于阈值40,稳定性较好。脂溶指数为77.73,且通过Ex PASy的Prot Scale预测,ampS基因编码氨基酸序列疏水性最小值约为-2.711,最大值约为1.922,整个多肽链中亲水性氨基酸比疏水性氨基酸数量多,因此推测该蛋白为亲水性蛋白。
2.6.3 风味酶基因AprX和ampS编码蛋白酶二级结构预测分析
(1)AprX基因编码蛋白酶二级结构预测分析
运用在线分析工具PredictProtein(https://www.predictProtein.org/)网站对解淀粉芽孢杆菌B3 AprX基因编码的丝氨酸蛋白酶二级结构进行预测,该蛋白酶二级结构α-螺旋比例为25.79%,β-折叠比例为20.36%,其他结构比例为53.85%。相应结果见附图4。
(2)ampS基因编码蛋白酶二级结构预测分析
运用在线分析工具PredictProtein(https://www.predictProtein.org/)网站对解淀粉芽孢杆菌B3 ampS基因编码的氨肽酶二级结构进行预测,该蛋白酶二级结构α-螺旋比例为40.10%,β-折叠比例为23.47%,其他结构比例为36.43%。相应结果见附图5。
二级结构中α-螺旋和β-折叠的比例越高,说明该蛋白质的稳定性越高。由上述的二级结构预测可知,这两种酶的稳定性较强。
2.6.4 风味酶基因AprX和ampS编码蛋白酶三级结构预测分析
(1)AprX基因编码蛋白酶三级结构预测分析
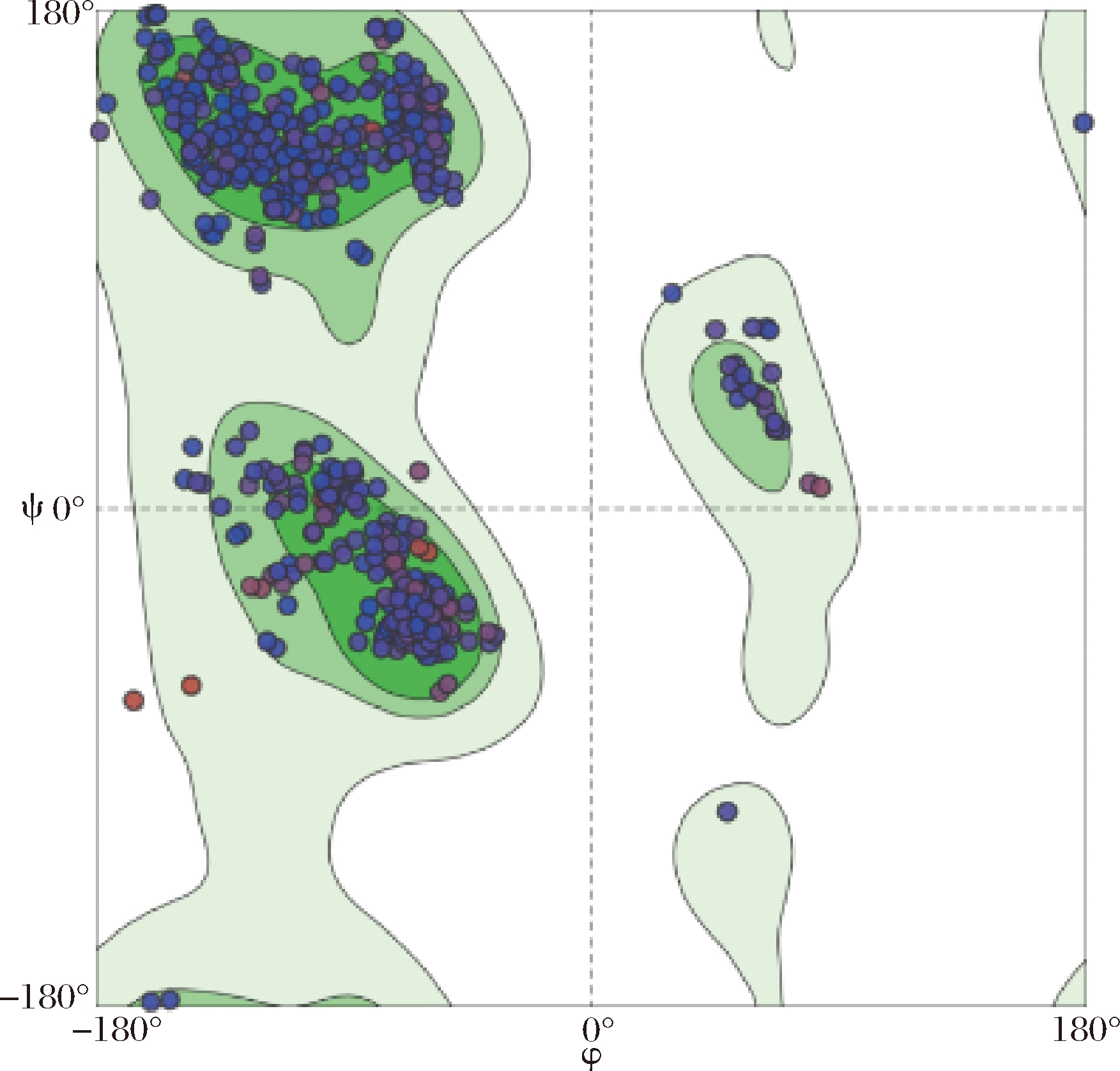
运用Swiss Model预测解淀粉芽孢杆菌AprX基因编码蛋白酶的三级结构模型,选取Subtilisin-like serine protease(SMTL ID:3afg.2)作为模板,两者序列相似度为41.58%,模型显示,该AprX基因编码蛋白酶含有大量α-螺旋、β折叠等二级结构,与二级结构预测结果相符。在同源建模中,两者序列相似度在30%以上,其结果可信度就较大,拉马钱德兰图可以很直观的描述同源建模的合理性,几乎所有位点都位于能量较低的区域,说明该蛋白结构不会发生变化,说明该同源建模所预测的三级结构合理性较高。相应结果见附图6、附图7。
(2)ampS基因编码蛋白酶三级结构预测分析
运用Swiss Model预测解淀粉芽孢杆菌ampS基因编码蛋白酶的三级结构模型,选取Aminopeptidase PepS(SMTL ID:4icr.1.A)作为模板,两者序列相似度为51.3%,模型显示,该ampS基因编码蛋白酶含有大量α-螺旋、β折叠等二级结构,与二级结构预测结果相符。绝大多数位点都分布于能量较低的区域内,说明该三级结构的合理性较高。相应结果见附图8、附图9。
由上述预测的两种酶的三级结构,可以更好的研究这两种酶对豆豉风味的影响,可以预测碱性丝氨酸蛋白酶水解大豆蛋白的结合位点,从而探究氨肽酶对大豆蛋白水解后产生的苦味肽的脱苦作用。
2.6.5 风味酶基因AprX和ampS编码蛋白酶氨基酸序列的系统发育树
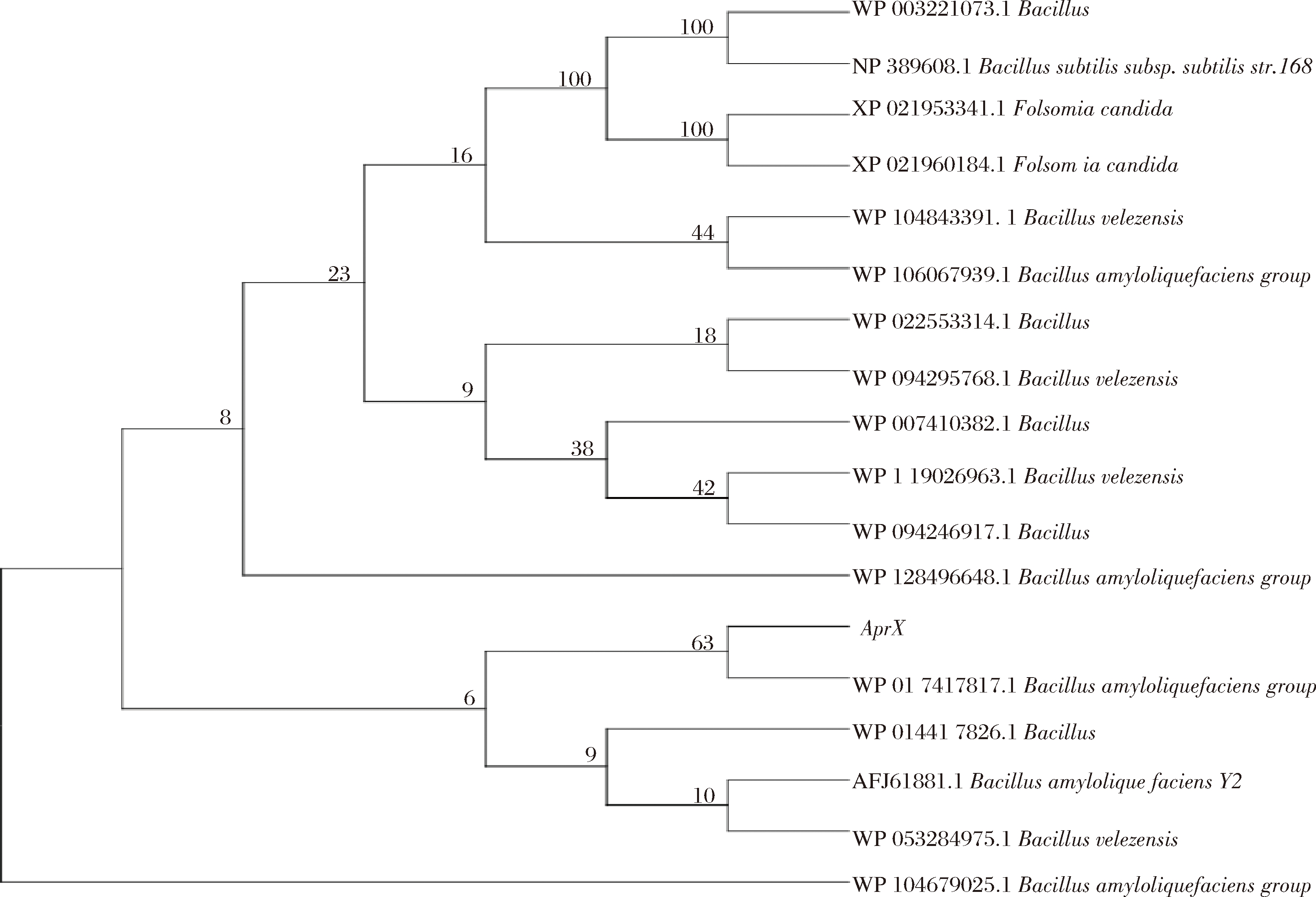
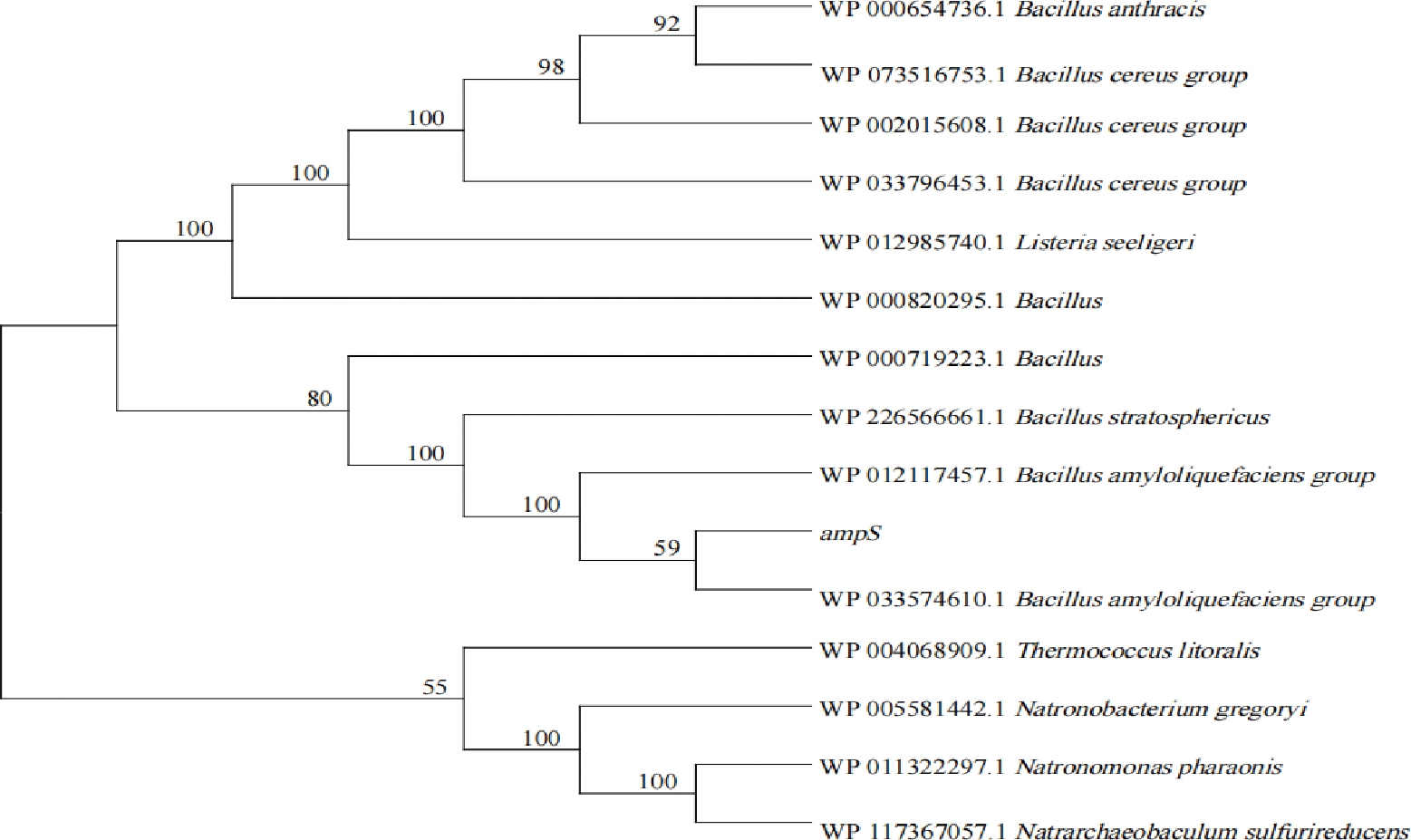
将AprX基因编码丝氨酸蛋白酶的氨基酸序列以及从NCBI的15个Bacillus属、Folsomia candida属来源的丝氨酸蛋白酶氨基酸序列;ampS基因编码氨肽酶的氨基酸序列以及在NCBI的15个Bacillus属、Listeria属、Thermococcus属、Natrarchaeobaculum属来源的氨肽酶氨基酸序列,输入到MEGA软件中,并采用临位相连法构建系统发育树,默认数值,可以较为精确地确定其进化地位。系统发育树结果显示基因AprX和ampS编码氨基酸序列都与Bacillus amyloliquefaciens group亲缘关系较近且不同种属来源的丝氨酸蛋白酶具有一定保守性。相应结果见附图10、附图11。
2.6.6 风味酶基因AprX和ampS编码蛋白酶氨基酸序列的多序列比对
(1)AprX基因编码蛋白酶氨基酸序列的多序列比对
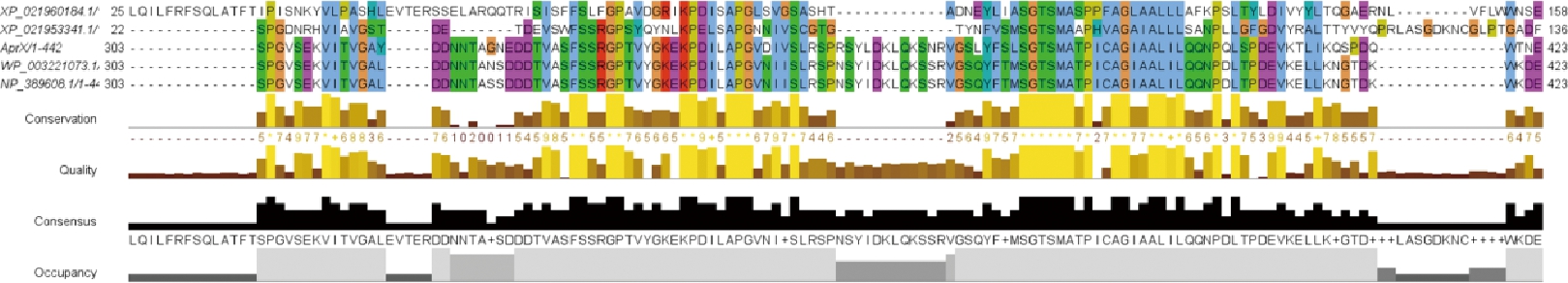
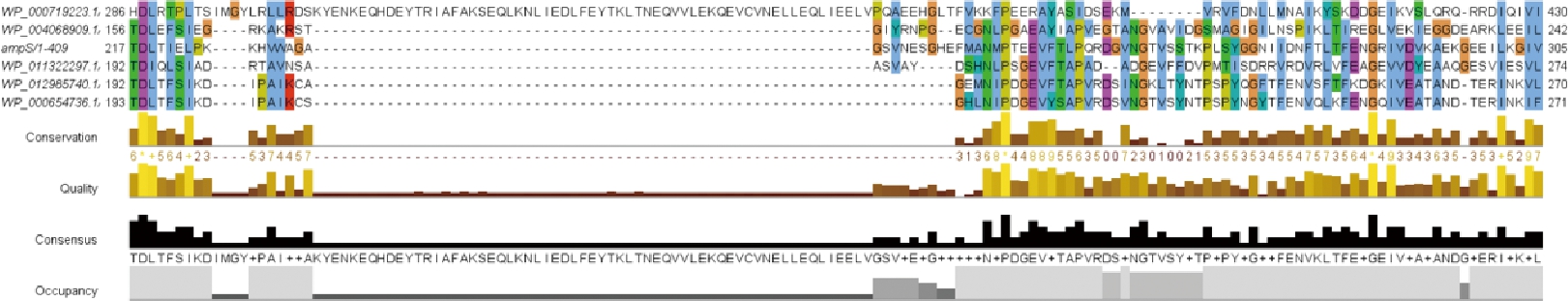
运用在线分析软件Clustal Omega对6种不同源的AprX基因编码蛋白酶氨基酸序列进行多重序列比对,并在Jalview中进行可视化处理,6种AprX基因编码蛋白酶氨基酸序列中芽孢杆菌种间的AprX基因编码蛋白酶氨基酸序列差异很小,保守域大致相同,氨基酸序列为SGTSMATPICAGIAALIL。本研究得到的解淀粉芽孢杆菌B3的AprX基因编码蛋白酶氨基酸序列与Bacillus amyloliquefaciens group相似性最高。相应结果见附图12。
(2)ampS基因编码蛋白酶氨基酸序列的多序列比对
运用相同方法对6种不同源的ampS基因编码蛋白酶氨基酸序列多重序列比对, 6种ampS基因编码蛋白酶氨基酸序列中芽孢杆菌种间的ampS基因编码蛋白酶氨基酸序列差异很小,保守域大致相同,氨基酸序列为EVFTLP。得到的解淀粉芽孢杆菌B3的ampS基因编码蛋白酶氨基酸序列与Bacillus amyloliquefaciens group相似性最高。相应结果见附图13。
3 结论与讨论
本试验从各地豆豉样品中分离纯化得到51株细菌,后通过蛋白酶活力确定了1株B3菌株,并对其进行酶学性质的研究和风味酶基因检索以及其编码的碱性丝氨酸蛋白酶和氨肽酶基酸序列组成、理化性质、高级结构等生信分析。结果表明,B3菌株最高蛋白酶活力可达185.6 U/mL,粗酶液在50 ℃、pH 7.0条件下能够保持最高蛋白酶活性,Mn2+对酶活力有明显的促进作用(P<0.01)。通过形态观察和16S rDNA序列分析,B3属于解淀粉芽孢杆菌种,同时利用全基因组学测序与生物信息学分析表明,解淀粉芽孢杆菌B3中含有风味酶基因AprX和ampS,其中AprX基因全长为1 329 bp,共编码442个氨基酸,理论分子质量为48 176.44 u,理论等电点为5.98,ampS基因全长为1 233 bp,共编码410个氨基酸,理论分子质量为45 697.14 u,理论等电点为5.29。两者均编码位于细胞质中的亲水性蛋白,基因编码氨基酸序列都与Bacillus amyloliquefaciens group相似性最高。以上结果表明B3菌株蛋白酶活力高、酶学性质优良、含有与风味酶密切相关的两种功能基因AprX和ampS,是一株具有发酵潜力的豆豉发酵剂,也是表达风味酶的基因供体。
[1] 李德坤,李欣遥.关于豆豉生产工艺技术的探讨[J].中国调味品,2016,41(8):104-106.
LI D K, LI X Y.Discussion on production technology of fermented soybean[J].China Condiment, 2016, 41(8):104-106.
[2] LIU L B, CHEN X Q, HAO L L, et al.Traditional fermented soybean products:Processing, flavor formation, nutritional and biological activities[J].Critical Reviews in Food Science and Nutrition, 2020,62(7):1 971-1 989.
[3] CAO Z H, GREEN-JOHNSON J M, BUCKLEY N D, et al.Bioactivity of soy-based fermented foods:A review[J].Biotechnology Advances, 2019, 37(1):223-238.
[4] 贾璠,郭霞,何晨,等.传统发酵豆制品营养功能成分研究进展[J].中国酿造,2019,38(4):1-6.
JIA F, GUO X, HE C, et al.Research progress in nutritional and functional ingredients of the traditional fermented soybean products[J].China Brewing, 2019, 38(4):1-6.
[5] JAYACHANDRAN M, XU B J.An insight into the health benefits of fermented soy products[J].Food Chemistry, 2019, 271:362-371.
[6] 李小永,陈伟,程芳,等.一株细菌型豆豉发酵菌种的筛选及鉴定[J].食品工业科技,2011,32(11):212-215;219.
LI X Y, CHEN W, CHENG F, et al.Screening and identification of a strain for lobster sauce fermentation[J].Science and Technology of Food Industry, 2011, 32(11):212-215;219.
[7] 彭昀,杨富民,康红霞,等.庆阳豆豉优势菌株的分离鉴定[J].食品与发酵工业,2013,39(4):58-63.
PENG Y, YANG F M, KANG H X, et al.Isolation and identification of predominant strains in Qingyang douchi[J].Food and Fermentation Industries, 2013, 39(4):58-63.
[8] 叶慧兰,曲直,孙乔润,等.阳江豆豉菌种的筛选及前发酵工艺条件优化[J].食品科学,2012,33(19):264-268.
YE H L, QU Z, SUN Q R, et al.Screening of optimal fermentation starter and optimization of primary fermentation for Yangjiang lobster sauce production[J].Food Science, 2012, 33(19):264-268.
[9] TAN Y, ZHANG R F, CHEN G J, et al.Effect of different starter cultures on the control of biogenic amines and quality change of Douchi by rapid fermentation[J].LWT, 2019, 109:395-405.
[10] HE B, LI H R, HU Z H, et al.Difference in microbial community and taste compounds between Mucor-type and Aspergillus-type Douchi during koji-making[J].Food Research International, 2019, 121:136-143.
[11] 刘敏,张仁凤,陈光静,等.霉菌纯种制曲豆豉在传统后发酵中的成分变化及其与生物胺相关性的研究[J].食品与发酵工业,2019,45(5):51-60.
LIU M, ZHANG R F, CHEN G J, et al.Changes in relevant components of mold starter-making Douchi during traditional post-fermentation and their correlations with biogenic amines[J].Food and Fermentation Industries, 2019, 45(5):51-60.
[12] 魏丕伟,王凌云,罗惠波,等.大曲中高产酸性蛋白酶霉菌的分离鉴定[J].食品与发酵科技,2014,50(4):1-4;21.
WEI P W, WANG L Y, LUO H B, et al.Isolation and identification of acid-protease producing fungi from daqu[J].Food and Fermentation Technology, 2014, 50(4):1-4;21.
[13] 杨林,王筱兰,杨慧林,等.1株传统曲霉型豆豉中高活力蛋白酶产生菌的分离及其鉴定[J].江西师范大学学报(自然科学版),2015,39(5):497-501.
YANG L, WANG X L, YANG H L, et al.A strain of high activity of protease producing bacteria isolation and identification in the traditional type Aspergillus tempeh[J].Journal of Jiangxi Normal University (Natural Science Edition), 2015, 39(5):497-501.
[14] 周德庆. 微生物学教程[M].第3版.北京:高等教育出版社,2011:336-338.
ZHOU D Q.Microbiology Course[M].3rd Edition.Beijing:Higher Education Press, 2011:336-338.
[15] 布坎南, 吉本斯.伯杰细菌鉴定手册[M].8版.北京:科学出版社, 1984.
BUCHANAN, GIBBONS. Berger′s Handbook of Bacterial Identification[M].8th Edition.Beijing:Science Press, 1984.
[16] 权淑静,马焕,王一雯,等.青藏高原土壤低温产蛋白酶菌株的筛选及酶学性质研究[J].中国酿造,2018,37(5):66-70.
QUAN S J, MA H, WANG Y W, et al.Screening and enzymatic characterizations of a low temperature protease-producing strain from Qinghai-Tibet Plateau soil[J].China Brewing, 2018, 37(5):66-70.
[17] 张芹,王修俊,商景天,等.贵州三穗特色豆豉发酵优势菌种的筛选和鉴定[J].食品与机械,2018,34(7):43-47.
ZHANG Q, WANG X J, SHANG J T, et al.Screening and identification of dominant strains of fermented characteristic lobster sauce in Guizhou Sansui[J].Food and Machinery, 2018, 34(7):43-47.
[18] SOLANKI P, PUTATUNDA C, KUMAR A, et al.Microbial proteases:Ubiquitous enzymes with innumerable uses[J].3 Biotech, 2021, 11(10):428.
[19] YAN Z F, YUAN S, QIN Q, et al.Enhancement of rice protein hydrolysate quality using a novel dual enzyme system[J].LWT, 2022, 158:113110.
[20] SONG P, CHENG L, TIAN K M, et al.A novel aminopeptidase with potential debittering properties in casein and soybean protein hydrolysates[J].Food Science and Biotechnology, 2020, 29(11):1 491-1 499.
[21] 刘逸寒,王洪彬,刘夫锋,等.微生物碱性蛋白酶的研究与开发[J].天津科技大学学报,2017,32(2):1-6;12.
LIU Y H, WANG H B, LIU F F, et al.The research and development of microbial alkaline protease[J].Journal of Tianjin University of Science and Technology, 2017, 32(2):1-6;12.
[22] 林晓彤. 亮氨酸氨肽酶的基因挖掘和在黑曲霉中的高效重组表达及其酶学性质研究[D].广州:华南理工大学,2020.
LIN X T.Screening and expression analysis of leucine aminopeptidases genes in Aspergillus niger[D].Guangzhou:South China University of Technology, 2020.