乳酸菌是目前使用最广泛的益生菌,它可以合成多种有益物质,如细菌素、胞外多糖等[1]。植物乳杆菌(Lactobacillus plantarum)作为一种乳酸菌,主要存在于发酵食品及动物肠道中,具有调节代谢、维持菌群平衡、提高人体免疫力等功能,是极具研究价值的一种益生菌。研究表明,L.plantarum不仅被广泛应用于食品生产领域,而且它在饲料生产和临床医学领域也有着不可替代的作用[2-4]。乳酸菌合成的细菌素是其自身的一种次级代谢产物,根据DRIDER等提出的细菌素遗传和生化特点分类方案,该细菌素属于Ⅱb类,与Plantaricin EF、Plantaricin JK相同,可抑制单增李斯特菌或2种肽合成新的肽[5-6]。乳酸菌细菌素无毒副作用,可被蛋白水解酶分解,能够维持肠道菌群的稳定,具备作为抗生素使用的价值[7]。
近年来,随着L.plantarum基因组测序研究的快速发展,截至2022年3月,在NCBI数据库中已经可以查询到671株L.plantarum的全基因组序列[8]。通过全基因组序列分析可以得到微生物的基因信息及其安全性,因此全基因组分析越来越受到重视,并被推荐成为菌株鉴定和安全性评估过程的一部分[9],已有学者进行了相关的研究,例如:TENEAN等[10]通过对L.plantarum UTNGt2进行全基因组测序分析,表明其可产生多种细菌素,具有较强的抑菌能力和良好的安全性,是一种非常有潜力的菌株,可作为细菌素的潜在生产菌株;BARBOSA等[11]通过全基因组测序评估了产细菌素的L.plantarum R23菌株的安全性,并分析了其生产的细菌素的作用方式和在不同环境中的稳定性,确定了其潜在的应用价值。
本研究首先对L.plantarum WUH3进行了全基因组测序分析,以了解其细菌素的合成能力,以及所合成的细菌素的种类和性质。然后,一系列的试验被设计用于验证基因分析得到的结果,并确定该菌株的应用价值。最后,我们对该菌株的安全性进行了分析,以确定其是否会对人体造成危害。该研究有助于促进乳酸菌细菌素的开发和应用。
1 材料与方法
1.1 材料
1.1.1 菌株与培养基
植物乳杆菌WUH3(L.plantarum WUH3),从中国传统腌制黄瓜中分离得到,保藏于上海海洋大学食品学院实验室。
主要指示菌:枯草芽胞杆菌(Bacillus subtilis strain B39),分离自镇沅松子地绿色食品有限公司的胀袋泡菜样品,其他指示菌菌株见表1。
表1 指示菌菌株
Table 1 Indicator strain
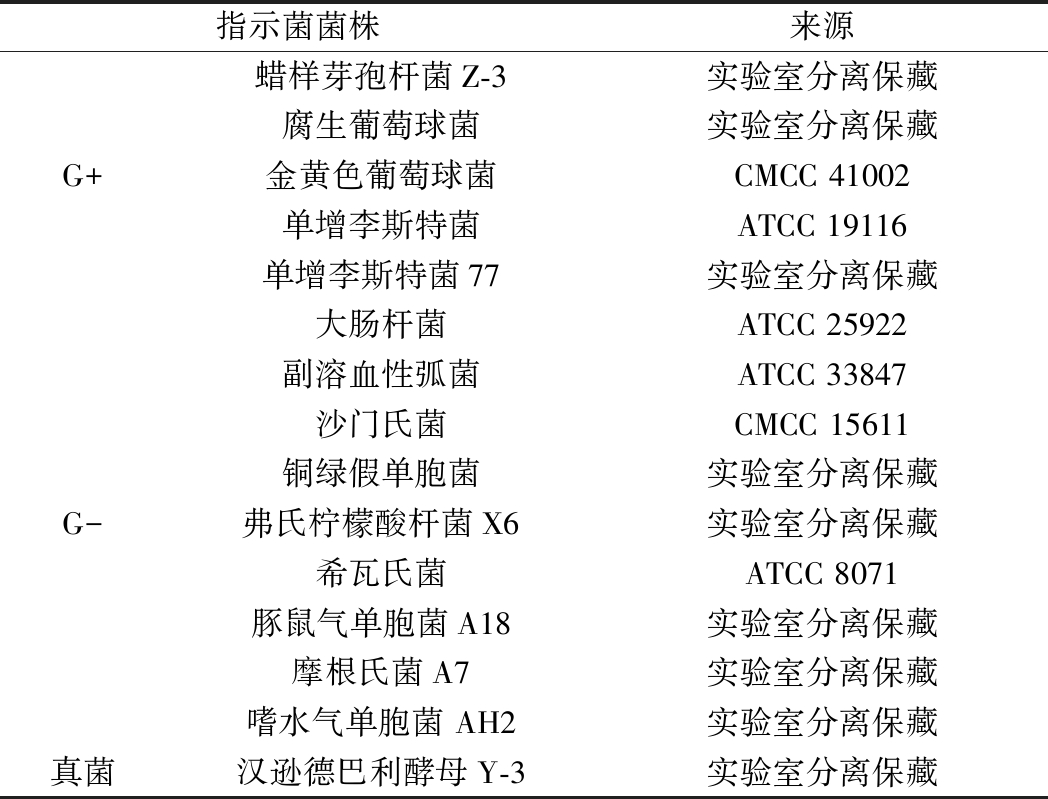
指示菌菌株来源蜡样芽孢杆菌Z-3实验室分离保藏腐生葡萄球菌实验室分离保藏G+金黄色葡萄球菌CMCC 41002单增李斯特菌ATCC 19116单增李斯特菌77实验室分离保藏大肠杆菌ATCC 25922副溶血性弧菌ATCC 33847沙门氏菌CMCC 15611铜绿假单胞菌实验室分离保藏G-弗氏柠檬酸杆菌X6实验室分离保藏希瓦氏菌ATCC 8071豚鼠气单胞菌A18实验室分离保藏摩根氏菌A7实验室分离保藏嗜水气单胞菌 AH2实验室分离保藏真菌汉逊德巴利酵母Y-3实验室分离保藏
培养基MRS培养基(乳酸菌),广东环凯微生物科技有限公司;LB培养基(其他细菌)、YPD培养基(真菌),国药集团化学试剂有限公司。
1.1.2 试剂和仪器
蛋白酶,国药集团化学试剂有限公司;BCA蛋白浓度试剂盒、透析袋,索莱宝生物科技有限公司;其他试剂,生工生物工程(上海)股份有限公司。SW-CJ-1F型洁净工作台,上海博迅实业有限公司;LDZX-50KBS型立式压力蒸汽灭菌器,上海申安医疗器械厂;H2050R冷冻离心机,湖南湘仪离心机仪器有限公司;DKY-II恒温调速回转式摇床,上海社科自动化设备有限公司;FE28 pH计,梅特勒-托利多仪器有限公司。
1.2 实验方法
1.2.1 菌株的活化与培养
L.plantarum WUH3在37 ℃、150 r/min培养24 h,多次划线纯化后,挑取单菌落至MRS培养基中培养。取菌液离心后,收集1.5~2.0 g菌体至1.5 mL无菌冻存管中,用于DNA提取。
1.2.2 基因组DNA提取与测序
采用MP磁珠法,对提取后的DNA进行质量鉴定,保证DNA质量≥10 μg,质量浓度≥50 ng/μL,OD260/OD280≥1.8,OD260/OD230≥1.0,无杂质污染和基因组严重降解。样本使用干冰进行保存,并寄送到检测机构(上海美吉生物有限公司)进行全基因组测序。
1.2.3 基因组装
对使用Illumina Hiseq×10得到的原始数据进行过滤得到有效数据;使用SOAP denovo 2.04对基因组进行组装,并利用GapCloser (Version 1.12)对初步组装结果进行gap区域的填补与优化,从而得到最终的组装结果[12]。
1.2.4 基因预测与功能注释
利用生物信息学软件和数据库对组装后的基因组进行分析,所用软件和数据库的信息见表2。
表2 软件和数据库列表
Table 2 Software and database list
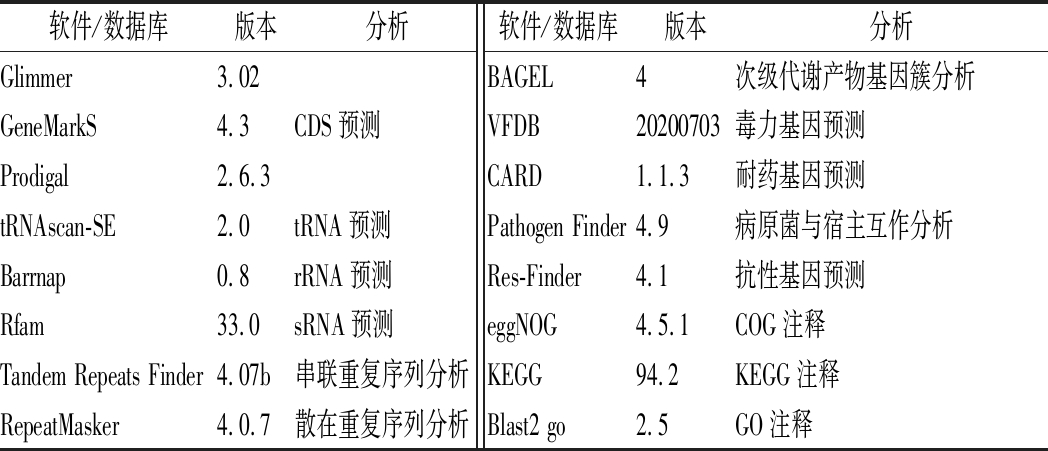
软件/数据库版本分析软件/数据库版本分析Glimmer3.02BAGEL4次级代谢产物基因簇分析GeneMarkS4.3CDS预测VFDB20200703毒力基因预测Prodigal2.6.3CARD1.1.3耐药基因预测tRNAscan-SE2.0tRNA预测Pathogen Finder4.9病原菌与宿主互作分析Barrnap0.8rRNA预测Res-Finder4.1抗性基因预测Rfam33.0sRNA预测eggNOG4.5.1COG注释Tandem Repeats Finder4.07b串联重复序列分析KEGG94.2KEGG注释RepeatMasker4.0.7散在重复序列分析Blast2 go2.5GO注释
1.2.5 细菌素性质分析
1.2.5.1 耐受性分析
细菌素提取:使用乙酸乙酯对除菌发酵上清液离心(8 000 r/min,4 ℃,15 min),以得到细菌素粗提取物。2种溶液首先按体积比1∶1混合,然后用真空旋转蒸发仪除去乙酸乙酯,最后用5 mmol/L PBS(pH=6)重悬,获得L.plantarum WUH3细菌素粗提物溶液[13]。
酶耐受性检测[14]:首先,分别向细菌素粗提物溶液中加入质量浓度为5 mg/mL的蛋白酶(碱性蛋白酶、胃蛋白酶、胰蛋白酶)、脂肪酶、淀粉酶溶液,调节pH至各酶的最适pH;然后,37 ℃恒温水浴4 h,再100 ℃加热使酶失活;最后,调节溶液pH至6,用指示菌检测细菌素的活性。
温度耐受性检测[15]:将细菌素粗提物溶液分别在-20、0、20、40、60、80、100、121 ℃下处理30 min后,用指示菌检测细菌素的活性。
pH耐受性检测[16]:使用缓冲液调节细菌素粗提物溶液pH至2.0、3.0、4.0、5.0、6.0、7.0、8.0、9.0、10.0、11.0,用指示菌检测细菌素的活性。所用缓冲液包括glycine-HCl(pH 2),citrate-phosphate(pH 3~6),sodium-phosphate(pH 7),Tris-HCl(pH 8和pH 9),glycine-NaOH(pH 10和pH 11)[17]。
上述实验均以未经处理的细菌素粗提取物作为对照,所有的试验均重复3次。
1.2.5.2 分子质量测定
采用Tricine-SDS-PAGE测定细菌素粗提物的分子质量[18]。首先将电泳凝胶分为两半,一半用考马斯亮蓝进行染色,并与蛋白Marker对比,观察肽带的位置;另一半用无菌去离子水清洗干净,将其铺在接有枯草芽孢杆菌(1×106 CFU/mL)的LB固体培养基上,37 ℃培养12 h后,将其与染色的另一半凝胶作对比,通过原位抑菌结果来确定细菌素分子质量。
1.2.5.3 吸附性分析
为了研究细菌素的吸附特征,首先,将L.plantarum WUH3在37 ℃培养24 h,沸水浴30 min杀死菌体,用1.0 mol/L NaOH将pH调节至6.5,室温下搅拌30 min,使细菌素最大程度地吸附到菌体上。然后,离心(8 000 r/min,30 min,4 ℃)收获菌体,使用100 mL 5 mmol/L PBS缓冲液(pH 6.5)对收获的菌体进行洗涤,将洗涤后的菌体重新悬浮在40 mL 100 mmol/L NaCl溶液中(pH 2.0),在4 ℃下搅拌12 h,使细菌素解离。最后,离心(10 000 r/min,30 min,4 ℃)回收细菌素粗提取物,用0.22 μm微孔滤膜过滤,透析48 h,得到最终产品。用指示菌测试细菌素活性[18],实验重复3次。
1.2.5.4 抑菌谱的测定
以多种食品腐败菌和病原菌为指示菌,用双层琼脂扩散法测定细菌素粗提物的抑菌效果。所用指示菌浓度为1×106 CFU/mL,抑菌板在4 ℃扩散2 h后,测量抑菌圈直径(mm),实验重复3次。
1.2.5.5 微观结构分析
通过二倍梯度稀释法测定细菌素的最小抑菌浓度(minimum inhibitory concentration, MIC)[19]。首先,收集枯草芽孢杆菌对数期的悬浊液(OD=0.4~0.6),通过添加提纯的细菌素获得浓度为1×MIC和3×MIC的处理液,静置3 h,使用无菌PBS为对照。然后在4 ℃下,用2.5%戊二醛固定菌体形态,接着用无菌PBS缓冲液洗涤菌体,并用乙醇(30%、50%、70%、80%、90%、100%,体积分数)进行梯度脱水,每个梯度静置10 min。最后,离心(8 000 r/min,5 min)除去乙醇获得处理后的菌体,然后用500 μL无水乙醇对得到的菌体进行分散,并吸取10~20 μL溶液均匀涂布到洁净的盖玻片上,干燥并镀金后使用S-4800 SEM(日立,日本)成像。依据TANG等[20]描述的方法观察并记录菌体结构表面的变化。
2 结果与分析
2.1 基因组概况
采用SOAP denovo软件对L.plantarum WUH3的基因组进行组装,组装结果如图1所示。基因组预测共得到3 378个编码基因,总长度2 840 538 bp,占基因组的83.52%,详细的基因组预测结果见表3。

图1 L.plantarum WUH3基因组圈图
Fig.1 Genome circle of L.plantarum WUH3
注:最外面一圈为基因组大小的标识;第2圈和第3圈为正链、负链上的CDS,不同的颜色表示编码蛋白序列(CDS)不同的COG的功能分类;第4圈为rRNA和tRNA;第5圈为GC含量;最内一圈为GC-Skew值
表3 L.plantarum WUH3基因组统计
Table 3 Genome summary of L.plantarum WUH3
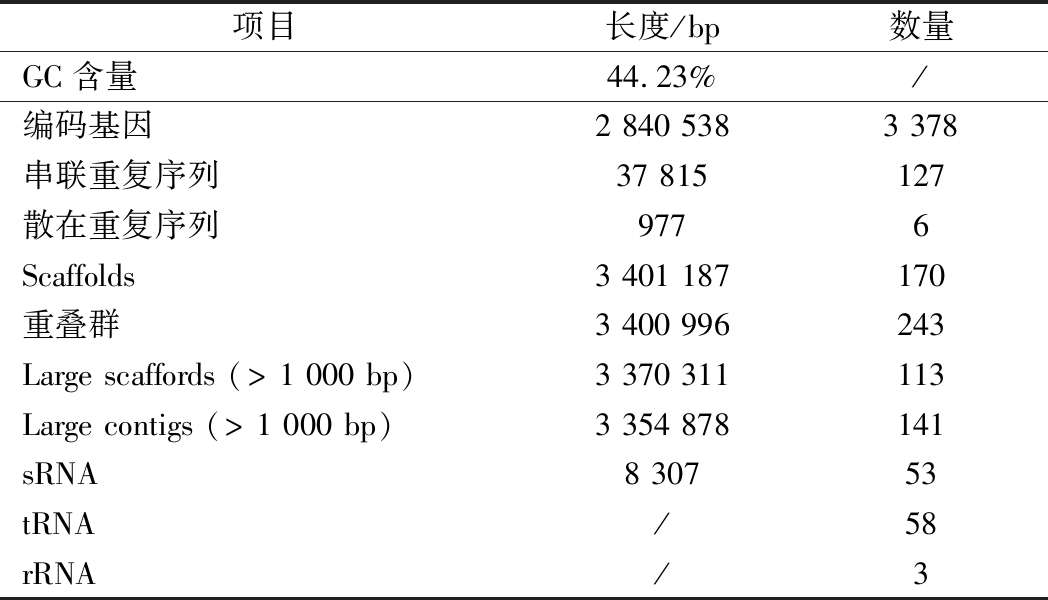
项目长度/bp数量GC含量44.23%/编码基因2 840 5383 378串联重复序列37 815127散在重复序列9776Scaffolds3 401 187 170重叠群3 400 996 243Large scaffords (> 1 000 bp)3 370 311 113Large contigs (> 1 000 bp)3 354 878 141sRNA8 307 53tRNA/58rRNA/3
2.2 基因功能注释
细菌素是一种具有抗菌活性的多肽,属于次级代谢产物。它的合成途径与ABC转运蛋白、氨基酸运输与代谢、次生代谢物的生物合成等密切相关[21]。
分别使用GO、KEGG和COG数据库对L.plantarum WUH3基因组进行注释[22]。如图2所示,根据GO数据库的注释结果可知,该菌株具有合成多种转运蛋白的能力,包括跨膜转运蛋白和ABC转运蛋白等;根据KEGG数据库的注释结果可知,该菌株拥有184条代谢通路,其跨膜运输通路主要集中在ABC转运蛋白的合成,其翻译通路主要集中在核糖体(51个)和氨酰tRNA的合成(28个),其新陈代谢通路主要集中在次级代谢物(228个)和氨基酸的合成(100个);根据COG数据库的注释结果可知,该菌株拥有氨基酸运输与代谢相关基因(208个)和翻译、核糖体结构相关基因(147个)。上述结果表明,L.plantarum WUH3可能具有细菌素的合生与分泌能力,具备作为产细菌素菌株的潜在能力。

a-COG功能分类;b-GO功能分类图;c-KEGG功能分类图
图2 L. plantarum WUH3基因注释图
Fig.2 Gene annotation map of L. plantarum WUH3
2.3 次级代谢产物合成基因簇分析
利用BAGEL4数据库对基因测序结果进行基因簇分析,以探明其合成细菌素的种类及相关基因的位置。结果显示L.plantarum WUH3有3个热点区域,推测其可合成Plantaricin_EF、Pediocin和Enterocin_X_chain_beta共3种细菌素(表4)。
表4 L.plantarum WUH3细菌素合成基因分析
Table 4 Bacteriocin synthesis gene analysis of L.plantarum WUH3

AOIStartEndClassFasta headerScaffold13.52.AOI_0168 86481 20897.2;Enterocin_X_chain_betaScaffold13Scaffold44.64.AOI_01013 863171.2;Plantaricin_EFScaffold44Scaffold55.82.AOI_0107 653159.2;PediocinScaffold55
2.3.1 Plantaricin_EF基因簇
Plantaricin_EF基因簇的具体信息如图3所示。其共有15个ORF,启动子在orf00003之前,orf00003基因片段具有细菌素合成相关组氨酸激酶的基因,Plantaricin_EF基因在Scaffold44的0~13 863碱基之间,该基因簇合成的细菌素属于含有双甘氨酸先导肽的细菌素Ⅱ类的乳球菌素家族,匹配度100%。
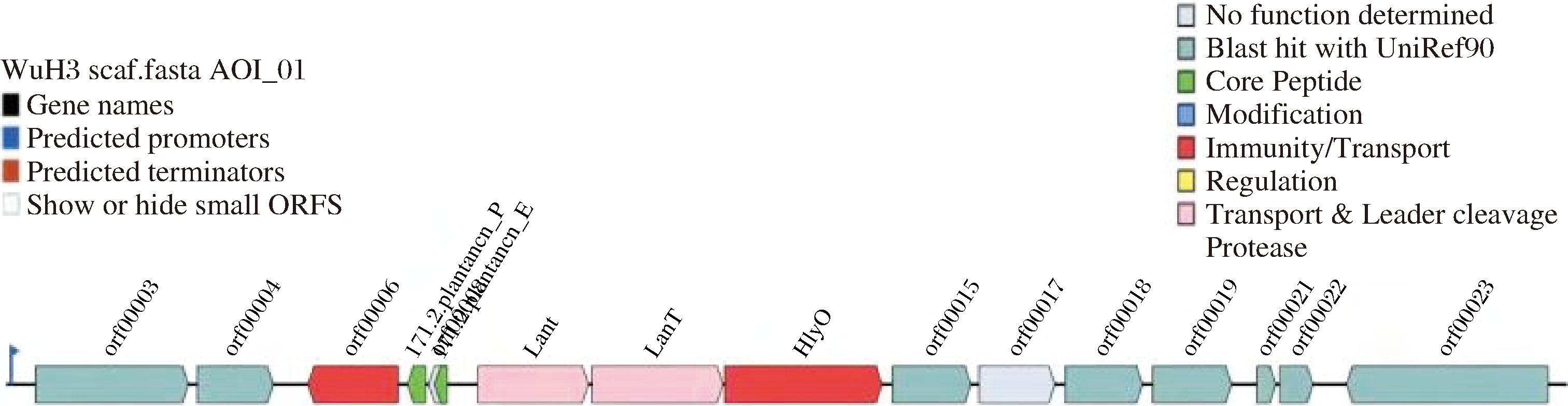
图3 L.plantarum WUH3的Plantaricin_EF基因簇
Fig.3 Plantaricin_EF gene cluster of L.plantarum WUH3
2.3.2 Pediocin基因簇
Pediocin基因簇的具体信息如图4所示。Pediocin基因簇共有9个ORF,启动子在orf00003之前,其合成的细菌素属于片球菌素,匹配度75.248%。

图4 L.plantarum WUH3的Pediocin基因簇
Fig.4 Pediocin gene cluster of L.plantarum WUH3
2.3.3 Enterocin_X_chain_beta基因簇
Enterocin_X_chain_beta基因簇的具体信息如图5所示。Enterocin_X_chain_beta基因簇共有16个ORF,启动子有4个,分别在orf00017~orf00019、orf00022-Enterocin_X_chain_beta、orf00025-orf00028、Plantaricin_K之后,核心肽为Enterocin_X_chain_beta和Plantaricin_K,其合成的细菌素属于乳球菌素,匹配度53.191%。

图5 L.plantarum WUH3的 Enterocin_X_chain_beta 基因簇
Fig.5 Enterocin_X_chain_beta gene cluster of L.plantarum WUH3
2.4 细菌素性质分析
2.4.1 耐受性
以枯草芽孢杆菌为指示菌研究细菌素的耐受性。由表5可知,该细菌素对脂肪酶、淀粉酶及大多数蛋白酶具有耐受性,但可被胰蛋白酶和中性蛋白酶分解,证明该细菌素为蛋白质。由图6-a可知,随着温度的升高,细菌素抑菌活性变化不大,甚至在121 ℃时仍具有抑菌活性,这是由于细菌素具有高度疏水区域、稳定的交联结构,因而往往具有良好的耐温性[23]。由图6-b可知,该细菌素具有良好的酸碱稳定性,能够在pH 2~11内保持抑菌活性。以上结果表明,该细菌素可被人体降解,且具有良好的热稳定性和pH稳定性,因而具有巨大的潜在应用价值。
表5 细菌素酶敏感性测试结果
Table 5 Enzyme sensitive test results of bacteriocin

酶最适pH剩余活性/%胰蛋白酶7.4-胃蛋白酶2.280.64±0.78木瓜蛋白酶9.090.32±0.51中性蛋白酶7.0-过氧化氢酶7.096.60±0.44脂肪酶8.099.60±0.25α-淀粉酶7.0100.00±0
注:“-”表示无抑菌活性

a-温度;b-pH
图6 稳定性测试结果
Fig.6 Stability test results
注:不同小写字母表示差异显著(P<0.05)
2.4.2 分子质量
使用Tricine-SDS-PAGE测定细菌素的分子质量,通过将染色的凝胶与枯草芽孢杆菌重叠来显示细菌素的抗菌活性。如图7所示,在染色的凝胶中有一条4.1~6.5 kDa的单一条带,且原位抑菌实验显示仅该条带对枯草芽孢杆菌具有抑菌活性。据此判断,该细菌素的分子质量在4.1~6.5 kDa。
2.4.3 吸附性
对细菌素进行吸附性研究,研究结果表明离心后的细菌素粗提物中检测不到抑菌活性,而未经处理的上清液检测到了全部的抑菌活性(图8),说明细菌素对宿主细胞不具有吸附效应,后续不能采用pH吸附-解析法进行该细菌素的分离纯化。
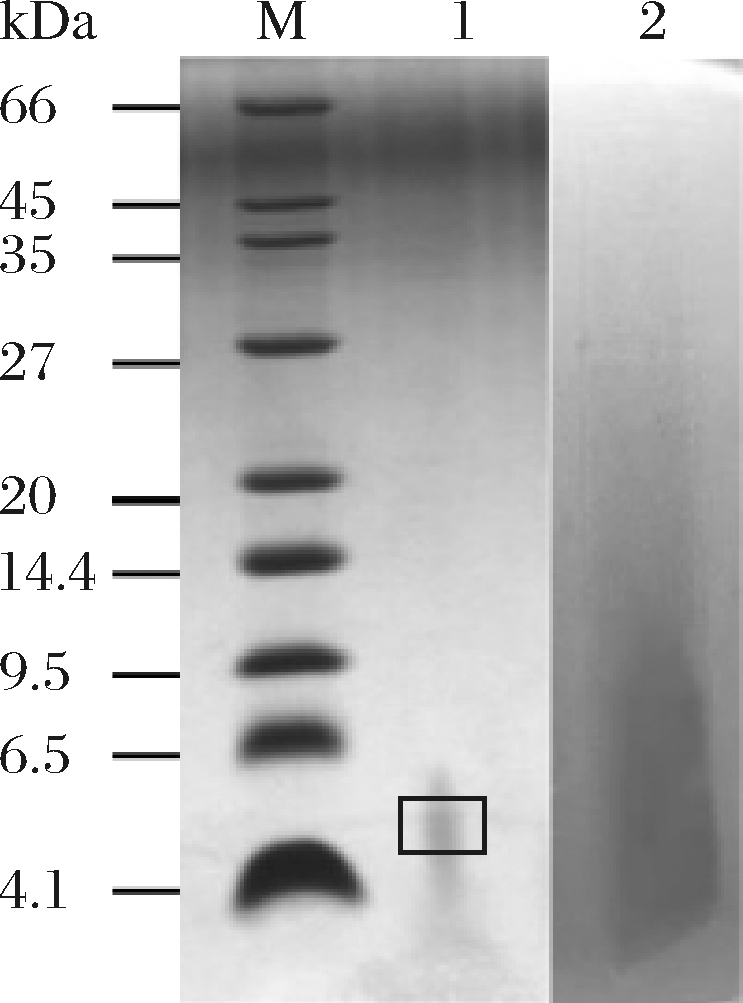
M-蛋白分子质量标准;1-经染色的样品蛋白分子条带;2-在B. subtilis strain B39平板上的抑菌活性条带
图7 细菌素的Tricine-SDS-PAGE电泳
Fig.7 Tricine-SDS-PAGE electrophoresis results of bacteriocin
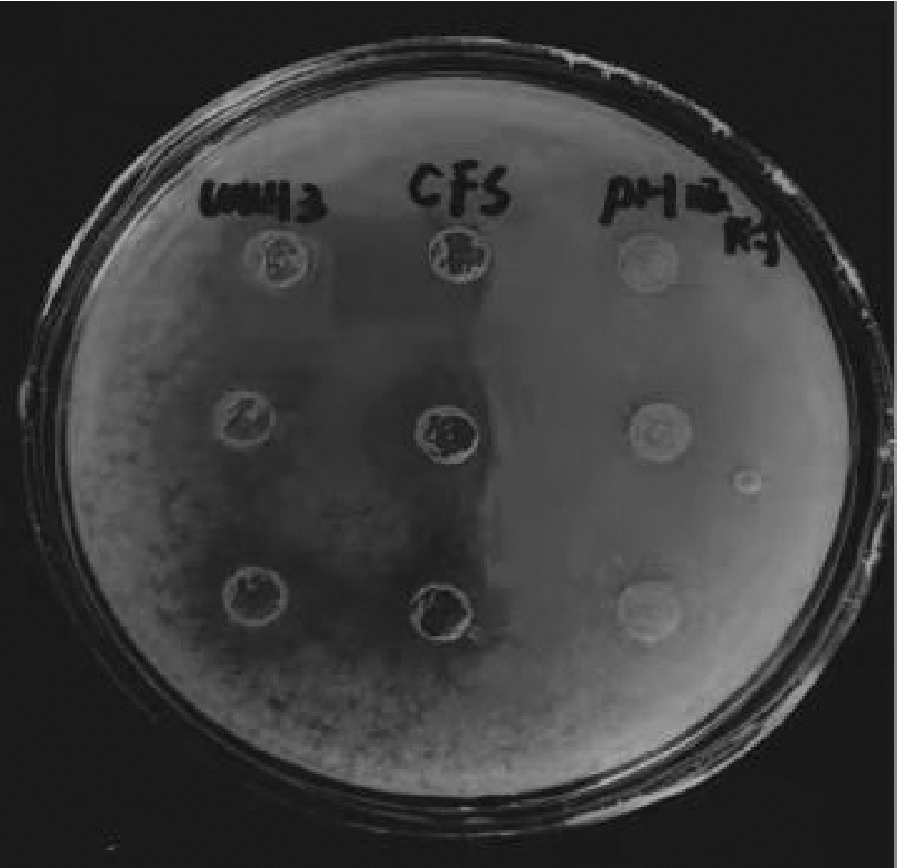
WUH3-菌株发酵液;CFS-无细胞上清液;pH吸附-吸附性实验组细菌素粗提物
图8 细菌素在细胞表面的吸附性研究
Fig.8 Study on the adsorption of bacteriocin on cell surface
2.4.4 抑菌谱
采用双层琼脂扩散法测定细菌素对多种常见食品腐败菌和病原体的抑制效果,如表6所示。该细菌素对多种常见腐败菌均有抑制效果,这表明其具有广谱抑菌性。且该细菌素对于多种食品腐败和水产病原菌均有良好的抑制效果,这是多数已报道的细菌素所不具备的,因此,该细菌素可作为生物防腐剂,在食品工业具有很大的应用潜力。
2.4.5 微观结构
使用二倍稀释法测得该细菌素的MIC为16 μg/mL。由图9可知,未经处理的枯草芽孢杆菌具有光滑、饱满、完整的杆状形态;经细菌素处理后,其形态发生明显变化,在1×MIC处理时,细胞表面出现皱纹,细胞膜向内凹陷。3×MIC处理时,细胞裂解,产生孔洞,内容物流出。上述结果表明,该细菌素可直接作用于枯草芽孢杆菌的细胞膜,引起其细胞膜破裂,从而实现杀菌。
表6 L. plantarum WUH3所产细菌素的抑菌谱
Table 6 Antibacterial spectrum of bacteriocin produced by L. plantarum WUH3

指示菌菌株腐败特性培养条件抑菌圈平均直径/mm蜡样芽孢杆菌Z-3引起食物中毒(呕吐和腹泻)16.00±1.00腐生葡萄球菌引起伤口发炎及食品腐败变质12.67±1.15G+金黄色葡萄球菌产生肠毒素,引起食物中毒LB、12 h、37 ℃22.33±1.53单增李斯特菌败血症、脑膜炎、4 ℃仍可生长,冷藏35.67±0.58单增李斯特菌77食品的主要病原菌之一42.00±1.00大肠杆菌引起胃肠道感染、出血29.33±1.15副溶血性弧菌引起海产性疾病的常见病原体30.00±1.00G-沙门氏菌蛋制品污染,引发发热、恶心、呕吐、腹泻、绞痛LB、12 h、37 ℃20.33±0.58铜绿假单胞菌易引起伤口感染,伴随头痛、呕吐等23.33±2.08弗氏柠檬酸杆菌X6水体污染的重要细菌学指标之一18.33±0.58希瓦氏菌腐败鱼、虾等海产品22.67±2.08豚鼠气单胞菌A18引起人类和动物的腹泻26.33±1.53摩根氏菌A7可引起呼吸道、泌尿道和伤口感染及败血症等LB、12 h、30 ℃28.67±1.15嗜水气单胞菌AH2常见水产病原菌,该菌的毒力因子主要是肠毒素28.00±0.00真菌汉逊德巴利酵母Y-3产生自由脂肪酸、产生油臭、奶臭味、酸化等24.67±1.53
注:G+表示革兰氏阳性菌;G-表示革兰氏阴性菌
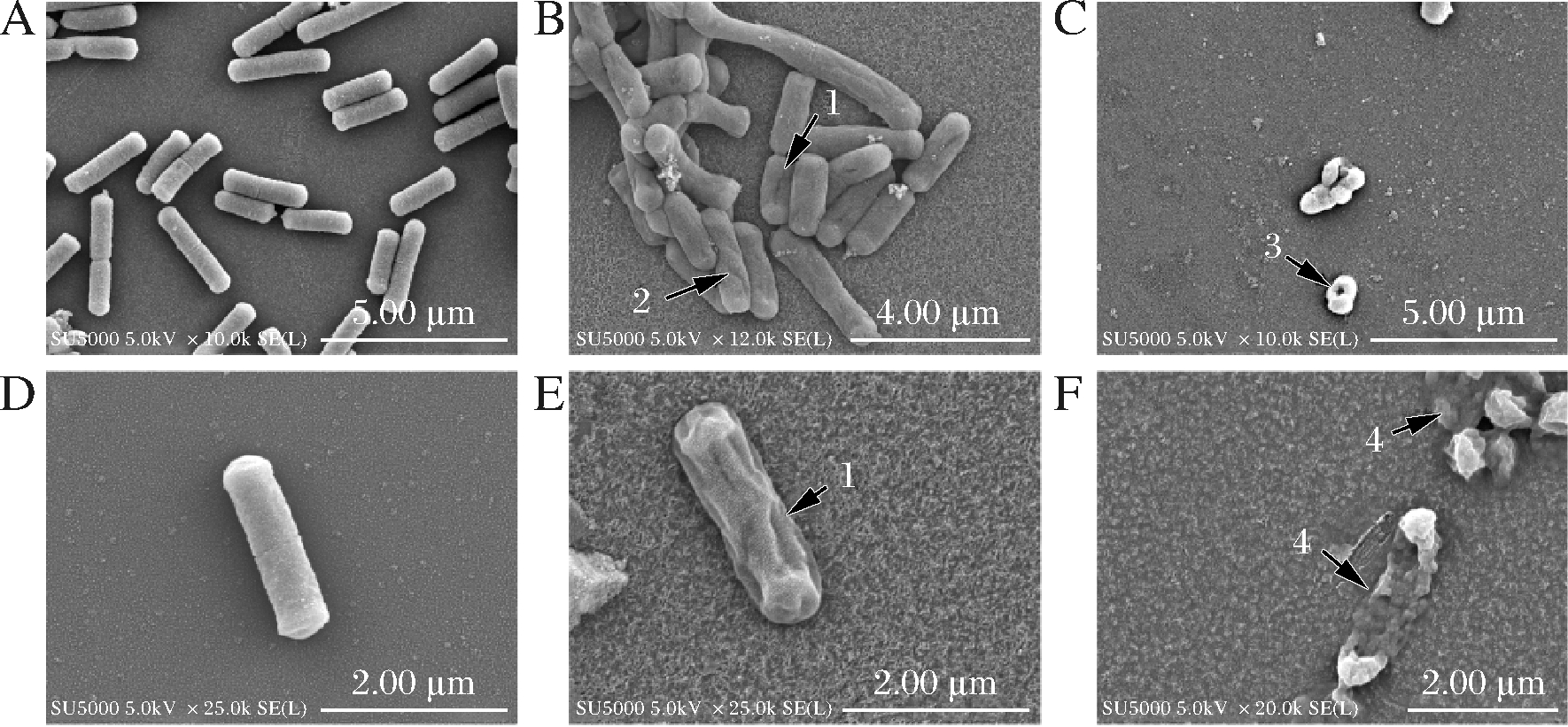
A,D-对照;B,E-1×MIC;C,F-3×MIC
图9 枯草芽孢杆菌的细胞形态
Fig.9 The cell morphology of B.subtilis strain
2.5 菌株安全性分析
使用VFDB数据库对毒力基因进行检测,得到毒力基因数目较多的是铁吸收系统、抗吞噬作用和黏附作用。此外,L.plantarum WUH3还含有一部分帮助细菌逃离宿主免疫系统的毒力因子,如防止吞噬、激发炎症、抑制补体介导的吞噬作用等;同时还含有对细胞有抑制作用的溶细胞素和α溶血素。
使用CARD数据库对耐药基因进行检测,得到160个抗生素耐药基因,共编码了30大类抗生素。其中大环内酯类抗生素的耐药基因最多,其次是四环素类抗生素耐药基因。结合COG注释分析结果,可推测该菌株可能对四环素类抗生素产生抗性。
使用PHI数据库对致病基因进行检测,共注释得到649个与病原菌宿主互作相关的基因,其中,使菌株致病性增加的基因数量较少,可降低菌株致病性的基因占比较大,并且其含有的大多数蛋白保证了L.plantarum WUH3不具有致病性。此外,使用Pathogen Finder网络服务器预测该菌株的致病性,结果显示该菌株不包含任何致病因子,成为人类病原体的的可能性很低(0.213)。因此,L.plantarum WUH3菌株不会对人体产生危害,可被应用于食品防腐领域。
3 结论
本试验对从中国传统腌制黄瓜中分离得到的乳酸菌——L.plantarum WUH3(GenBank数据库登录号:JAKUCW 000000000)进行了研究和分析。首先,从基因层面证明了L.plantarum WUH3具有细菌素的合成分泌能力,并对可能的编码细菌素的基因簇进行了检测和分析。然后,进行了试验验证,结果表明该菌株具有细菌素的合成分泌能力,其能够合成蛋白质类细菌素,分子质量为4.1~6.5 kDa,可被多种胃肠道蛋白酶分解。进一步的分析表明,该细菌素对多种食源性致病菌均有抑制作用,具备广谱抑菌性。对菌株进行安全性分析表明该菌株对人体无害。综上所述,L.plantarum WUH3菌株具有良好的抑菌性能和安全性,且其细菌素具有良好的热稳定性和pH稳定性,因而有望应用于巴氏杀菌食品和热加工食品中,其在食品防腐领域有着广阔的应用前景。本研究对天然防腐剂的开发和应用提供了一定的参考,有助于L.plantarum WUH3的开发和应用。
[1] SU D, LIANG L, LI Z, et al.Genome analysis of two Lactobacillus plantarum strains, LLY-606 and pc-26, for evaluating their potential as probiotics[J].Journal of Microbiology and Biotechnology, 2019.DOI:10.1101/841163.
[2] TREJO-GONZ LEZ L, GUTIÉRREZ-CARRILLO A E, RODR
LEZ L, GUTIÉRREZ-CARRILLO A E, RODR GUEZ-HERN
GUEZ-HERN NDEZ A I, et al.Bacteriocins produced by LAB isolated from cheeses within the period 2009-2021:A review[J].Probiotics and Antimicrobial Proteins, 2022,14(2):238-251.
NDEZ A I, et al.Bacteriocins produced by LAB isolated from cheeses within the period 2009-2021:A review[J].Probiotics and Antimicrobial Proteins, 2022,14(2):238-251.
[3] HUANG Y Y, LIU D M, JIA X Z, et al.Whole genome sequencing of Lactobacillus plantarum DMDL 9010 and its effect on growth phenotype under nitrite stress[J].LWT, 2021, 149:111778.
[4] SIEZEN R J, TZENEVA V A, CASTIONI A, et al.Phenotypic and genomic diversity of Lactobacillus plantarum strains isolated from various environmental niches[J].Environmental Microbiology, 2010, 12(3):758-773.
[5] 李萍, 龙春昊, 赵轩, 等.Ⅱ类细菌素的生物合成及其在食品领域的应用[J].中国食品学报, 2021, 21(10):269-286.
LI P, LONG C H, ZHAO X, et al.Biosynthesis of class Ⅱ bacteriocin and its application in food preservation[J].Journal of Chinese Institute of Food Science and Technology, 2021, 21(10):269-286.
[6] PAL G, SRIVASTAVA S.Inhibitory effect of plantaricin peptides (Pln E/F and J/K) against Escherichia coli[J].World Journal of Microbiology and Biotechnology, 2014, 30(11):2 829-2 837.
[7] HANNY E L L, MUSTOPA A Z, BUDIARTI S, et al.Efficacy, toxicity study and antioxidant properties of plantaricin E and F recombinants against enteropathogenic Escherichia coli K1.1 (EPEC K1.1)[J].Molecular Biology Reports, 2019, 46(6):6 501-6 512.
[8] SUN Z H, HARRIS H M B, MCCANN A, et al.Expanding the biotechnology potential of lactobacilli through comparative genomics of 213 strains and associated genera[J].Nature Communications, 2015, 6(1):1-13.
[9] CHOKESAJJAWATEE N, SANTIYANONT P, CHANTARASAKHA K, et al.Safety assessment of a nham starter culture Lactobacillus plantarum BCC9546 via whole-genome analysis[J].Scientific Reports, 2020, 10(1):10241.
[10] TENEA G N, ORTEGA C.Genome characterization of Lactiplantibacillus plantarum strain UTNGt2 originated from theobroma grandiflorum (white cacao) of ecuadorian amazon:Antimicrobial peptides from safety to potential applications[J].Antibiotics(Basel,Switzerland), 2021, 10(4):383.
[11] BARBOSA J, ALBANO H, SILVA B, et al.Characterization of a Lactiplantibacillus plantarum R23 isolated from arugula by whole-genome sequencing and its bacteriocin production ability[J].International Journal of Environmental Research and Public Health, 2021, 18(11):5 515.
[12] LUO R B, LIU B H, XIE Y L, et al.Erratum:SOAPdenovo2:An empirically improved memory-efficient short-read de novo assembler[J].GigaScience, 2012, 1(1):18.
[13] ZHAO S M, HAN J Z, BIE X M, et al.Purification and characterization of plantaricin JLA-9:A novel bacteriocin against Bacillus spp.produced by Lactobacillus plantarum JLA-9 from Suan-tsai, a traditional Chinese fermented cabbage[J].Journal of Agricultural and Food Chemistry, 2016,64(3):2 754-2 764.
[14] TICHACZEK P S, NISSEN-MEYER J, NES I F, et al.Characterization of the bacteriocins curvacin a from Lactobacillus curvatus LTH1174 and sakacin P from L.sake LTH673[J].Systematic and Applied Microbiology, 1992, 15(3):460-468.
[15] 张瑜. LS-8乳酸菌细菌素的分离纯化[D].杨凌:西北农林科技大学, 2013.
ZHANG Y.The purification and partial characteriaztion of bacteriocin produced by Lactobacillus rhamnosus LS-8[D].Yangling:Northwest A&F University, 2013.
[16] YI L H, QI T, HONG Y, et al.Screening of bacteriocin-producing lactic acid bacteria in Chinese homemade pickle and dry-cured meat, and bacteriocin identification by genome sequencing[J].LWT, 2020, 125:109177.
[17] XU C, FU Y Y, LIU F, et al.Purification and antimicrobial mechanism of a novel bacteriocin produced by Lactobacillus rhamnosus 1.0320[J].LWT, 2021, 137:110338.
[18] YI L H, DANG J, ZHANG L H, et al.Purification, characterization and bactericidal mechanism of a broad spectrum bacteriocin with antimicrobial activity against multidrug-resistant strains produced by Lactobacillus coryniformis XN8[J].Food Control, 2016, 67:53-62.
[19] WANG Y, QIN Y X, XIE Q, et al.Purification and characterization of plantaricin LPL-1, a novel class Ⅱa bacteriocin produced by Lactobacillus plantarum LPL-1 isolated from fermented fish[J].Frontiers in Microbiology, 2018,9:2276.
[20] TANG W T, ZHANG H, WANG L, et al.New cationic antimicrobial peptide screened from boiled-dried anchovies by immobilized bacterial membrane liposome chromatography[J].Journal of Agricultural and Food Chemistry, 2014, 62(7):1 564-1 571.
[21] JIA F F, ZHANG L J, PANG X H, et al.Complete genome sequence of bacteriocin-producing Lactobacillus plantarum KLDS1.0391, a probiotic strain with gastrointestinal tract resistance and adhesion to the intestinal epithelial cells[J].Genomics,.2017, 109(5-6):432-437.
[22] STERGIOU O S, TEGOPOULOS K, KIOUSI D E, et al.Whole-genome sequencing, phylogenetic and genomic analysis of Lactiplantibacillus pentosus L33, a potential probiotic strain isolated from fermented sausages[J].Frontiers in Microbiology, 2021, 12:746659.
[23] YANG R, JOHNSON M C, RAY B.Novel method to extract large amounts of bacteriocins from lactic acid bacteria[J].Applied and Environmental Microbiology, 1992, 58(10):3 355-3 359.