五加科人参属植物人参(Panax ginseng)、三七(Panax notoginseng)和西洋参(Panax quinquefoliusm)是我国常用的珍贵中药材。受环境影响,三者的植物形态和化学成分具有一定的相似性,但药效存在很大差异,单纯依靠传统形态学和化学分析法鉴定植物品种不够精确,尤其是无法对参片及参粉等参类产品进行鉴定。
人参、三七和西洋参为近源物种,三者在基因上差异较小,这导致快速鉴定方法的开发较为困难。随着分子生物学的发展,DNA分子标记技术的应用使得药用植物的鉴定更为准确和便捷。第三代分子标记物单核苷酸多态性(single nucleotide polymorphism,SNP)是指在基因组水平上由单个核苷酸的变异所引起的DNA序列多态性,因其分布广,密度高,遗传稳定性强,易于自动化等优点被广泛应用于藏菖蒲、枇杷、化橘红和紫苏等药用植物的鉴定中[1-4]。
SNP的检测方法有单链构象多态性、温度梯度凝胶电泳、变性高效液相色谱、衍生酶切扩增多态性、TaqMan探针技术、基因芯片和等位基因特异性PCR(allele-specific PCR, AS-PCR)等。其中AS-PCR应用最广,具有成本低,操作简单,便捷,快速等优点,但是其不足在于特异性不强。本研究通过对SNP位点进行锁核酸(locked nucleic acid,LNA)修饰和引入错配碱基2种方式使3′末端实现封闭,增强引物特异性。建立人参、三七和西洋参的多重PCR检测体系,并考察其在市售参类产品中的检测情况。
1 材料与方法
1.1 材料与试剂
供试人参、西洋参及三七样品由国家参茸检验检测中心和吉林省益盛汉参生物科技有限公司提供,储存于-80 ℃冰箱保持样品新鲜(表1)。常见9种市售参类产品:生晒参片、红参切片、西洋参切片、三七粉、人参丸、高丽参茶、人参蜜片、红参药片、西洋参胶囊(图1)。20份粗加工参类产品:生晒参片(5份)、红参切片(5份)、西洋参切片(5份)、三七粉(5份)购自当地及线上销售处。
表1 样品名称及产地表
Table 1 Name and producing area of samples
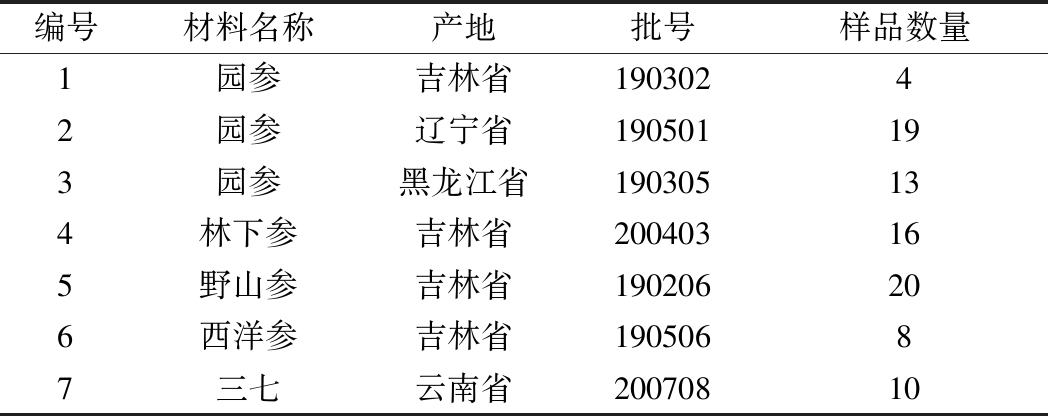
编号材料名称产地批号样品数量1园参吉林省19030242园参辽宁省190501193园参黑龙江省190305134林下参吉林省200403165野山参吉林省190206206西洋参吉林省19050687三七云南省20070810

1-生晒参片;2-人参丸;3-高丽参茶;4-人参蜜片;5-红参切片;6-红参药片;7-西洋参切片;8-西洋参胶囊;9-三七粉
图1 市售样品
Fig.1 Commercially available samples
植物DNA提取试剂盒,北京BioTeke公司;rTaq DNA聚合酶,TaKaRa TaqTM Version 2.0;PrimeSTAR HS、DL2000,大连TaKaRa公司;6×DNA Loading Buffer,天根生化科技有限公司。
1.2 仪器与设备
H-2050R低温高速离心机,长沙湘仪离心机仪器有限公司;ETC811型基因扩增仪,苏州东胜兴业科学仪器有限公司;凝胶成像分析仪,德国耶拿公司;DNA/蛋白质分析仪,Quawell。
1.3 实验方法
1.3.1 特异性引物设计
本研究利用DNAMAN对比了NCBI中现有的人参属植物基因序列,发现人参的18S rRNA序列(以KC593823.1为参照)在496、498和500位碱基处有3个SNP位点,人参为G、T、C,三七为C、A、G,西洋参为C、G、G。三七的rcbL序列(以GQ436707.1为参照)第212位碱基为C,而人参和西洋参为T。西洋参的psbE-petL序列(以JN700483.1为参照)第461位碱基为C,而人参和三七为T。以此为基础利用Primer3设计特异性引物,使正向引物3′端位于SNP位点上并用LNA进行修饰,在3′末端第二或第三碱基处引入错配以确保3′末端实现完全封闭,引物序列见表2。
表2 人参、三七、西洋参特异性鉴别引物
Table 2 Specific identification primers for Panax ginseng, Panax notoginseng, and Panax quinquefolium
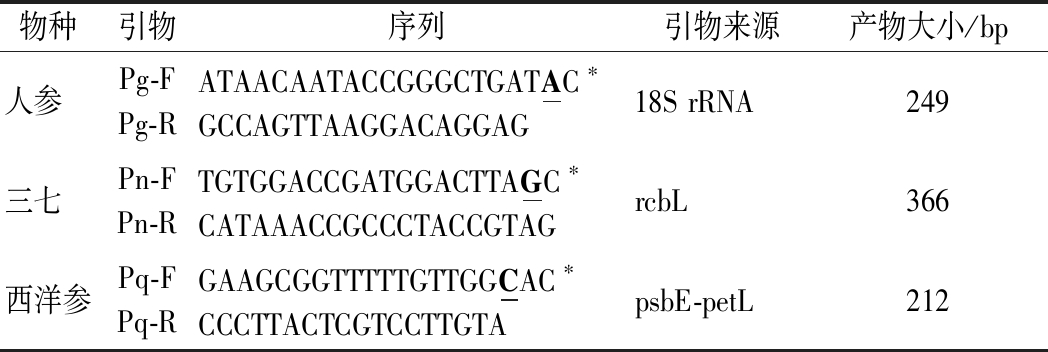
物种引物序列引物来源产物大小/bp人参 Pg-FPg-RATAACAATACCGGGCTGATAC∗GCCAGTTAAGGACAGGAG18S rRNA249三七 Pn-FPn-RTGTGGACCGATGGACTTAGC∗CATAAACCGCCCTACCGTAGrcbL366西洋参Pq-FPq-RGAAGCGGTTTTTGTTGGCAC∗CCCTTACTCGTCCTTGTApsbE-petL212
注:带下划线的粗体字母表示引入的错配,*代表LNA修饰位置
1.3.2 DNA提取
本研究应用改良的CTAB法提取人参、三七和西洋参的DNA[5],用微量核酸蛋白质检测仪检测DNA的纯度及浓度。将质量浓度调整到10 ng/μL用于PCR扩增。
1.3.3 PCR扩增及电泳
20 μL PCR反应体系为:4 μL 10 ng/μL模板DNA,10 μL rTaq DNA聚合酶,人参上下游引物(10 μmol/L)各0.15 μL,三七上下游引物(10 μmol/L)各1.25 μL,西洋参上下游引物(10 μmol/L)各0.3 μL,2.6 μL去离子水,反应在基因扩增仪上进行。反应程序为:94 ℃预变性5 min;95 ℃变性30 s;59 ℃退火30 s;72 ℃延伸20 s,35次循环;72 ℃延伸10 min,4 ℃保存。PCR产物加入Loading buffer后经EB预染的20 g/L(质量浓度)的琼脂糖凝胶进行电泳。
1.3.4 荧光定量PCR
20 μL PCR反应体系为:4 μL 10 ng/μL模板DNA,10 μL PrimeSTAR HS,人参上下游引物(10 μmol/L)各0.15 μL,三七上下游引物(10 μmol/L)各1.25 μL,西洋参上下游引物(10 μmol/L)各0.3 μL,2.6 μL去离子水。反应程序为:95 ℃预变性30 s;95 ℃变性5 s;59 ℃退火20 s;72 ℃延伸20 s,35次循环;每个循环在72 ℃收集荧光信号,每个反应设3个复孔。
1.3.5 方法学评价
1.3.5.1 多重PCR反应条件评价
分析影响多重PCR效果的关键因素,包括退火温度、循环数、DNA含量和引物含量。
1.3.5.2 多重PCR反应体系灵敏性评价
使用ddH2O将人参、三七和西洋参的DNA浓度梯度稀释为10~10-4 ng/μL。按照1.3.3中多重PCR反应体系,保持其中两种模板浓度不变,使剩余一种模板浓度梯度递减进行PCR扩增。
1.3.6 混合样品检测
将人参,三七,西洋参标准品按照一定比例均匀混合,总共分成7组,Ⅰ组是3个样品混合,人参、三七、西洋参质量比为1∶1∶1,6∶3∶1和6∶1∶3。Ⅱ~Ⅶ组为两两混合,分别是人参中混入三七,人参中混入西洋参;西洋参中混入三七,西洋参中混入人参,三七中混入人参,三七中混入西洋参,其质量比分别为1∶1,4∶1和9∶1。混合好的样品用改良的CTAB法提取DNA,PCR扩增后用20 g/L(质量浓度)的琼脂糖凝胶进行电泳。
1.3.7 市售样品检测
用改良的CTAB和试剂盒法提取市售9份人参制品的基因组,将浓度稀释至10 ng/μL,按照1.3.3中多重PCR反应体系进行扩增后用20 g/L(质量浓度)的琼脂糖凝胶进行电泳。
2 结果与分析
2.1 多重PCR反应条件优化
2.1.1 退火温度与循环数
用特异性引物对人参、三七、西洋参进行扩增,在55~60 ℃时分别能扩增出249、366、212 bp的特异性条带,以此为基础对影响多重PCR特异性和灵敏性的因素进行摸索。结果表明退火温度在58~60 ℃时人参、三七、西洋参均能产生特异性条带,但在61 ℃之后西洋参特异性条带亮度明显降低。根据电泳条带的亮度确定多重PCR的退火温度为59 ℃(图2-A)。对循环数的考察,25~40个循环时均能对人参、三七、西洋参进行鉴别,能明显的看出35个循环时条带亮度最高,最清晰,且无非特异性扩增(图2-B)。
2.1.2 DNA含量与最适引物量
20 μL体系中模板含量分别为20、30、40、50 ng,结果人参、三七和西洋参均能扩增出特异性条带,多次重复试验发现模板量在40 ng时条带最亮,确定DNA含量为40 ng(图2-C)。多重PCR中,引物用量及引物间相互作用对PCR结果有重要影响,确保PCR总体系中DNA含量为40 ng,模拟混合掺假调整引物比例致使人参、三七和西洋参的扩增效率一致。在调整引物量的过程中发现人参和西洋参的特异性引物扩增效率明显高于三七。为了确保电泳结果的清晰和准确,最终确定引物配比为人参引物量∶三七引物量∶西洋参引物量体积比为 0.15 μL∶1.25 μL∶0.3 μL(图2-D)。
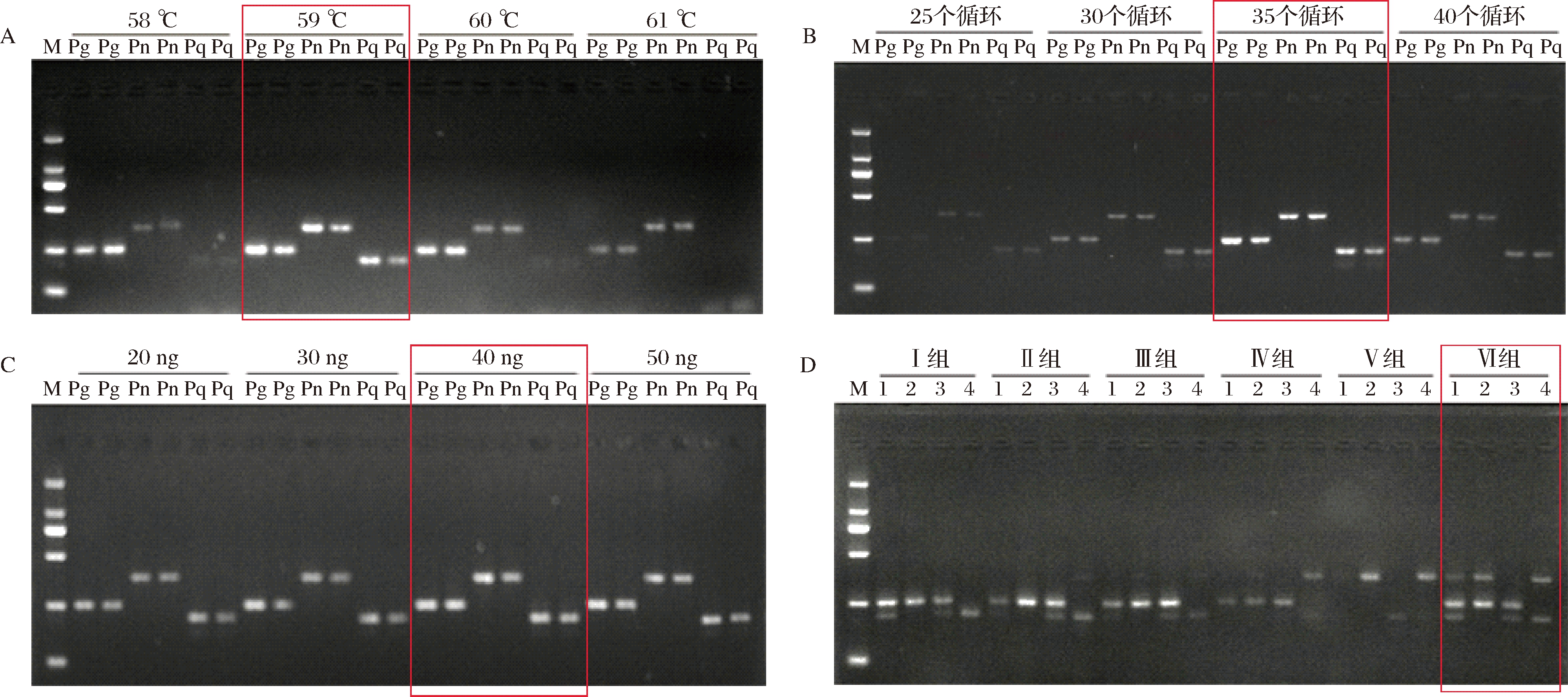
M-DL 2000 DNA Marker;Pg-人参;Pn-三七;Pq-西洋参
A-不同退火温度;B-不同循环数;C-不同模板量;D-最适引物量摸索
图2 人参、三七、西洋参不同PCR反应条件的电泳图
Fig.2 Electrophoresis diagram of Panax ginseng, Panax notoginseng, and Panax quinquefolium under different PCR reaction conditions
注:D中1~4号样品DNA总含量为40 ng;1号样品人参:三七:西洋参DNA含量为1∶1∶1;2号样品人参:三七DNA含量为1∶1;3号样品人参:
西洋参DNA含量为1∶1;4号样品三七:西洋参DNA含量为1∶1;Ⅰ~Ⅵ组人参:三七:西洋参引物量体积比分别为1∶1∶1、0.75∶1∶0.75、
0.5∶1∶0.5、0.25∶1∶0.25、0.1∶1∶0.25、0.15∶1.25∶0.3
2.2 多重PCR反应体系灵敏性评价
如图3-A~C所示,调节人参、三七、西洋参的模板浓度,使其中任意一模板浓度梯度递减,其余两种模板浓度保持不变,按照最佳多重PCR反应条件进行扩增,其中人参最低检测限可达到1 ng/μL,三七和西洋参可达到5 ng/μL。本研究所建立多重PCR检测体系结果稳定,当任意一种DNA减少至5 ng/μL时,仍可准确检测,且无交叉无污染。

M-DL2000 DNA Marker;1-10 ng/μL;2-5 ng/μL;3-1 ng/μL;4-10-1 ng/μL;5-10-2 ng/μL;6-10-3 ng/μL;7-10-4 ng/μL;N-空白对照
A-人参;B-三七;C-西洋参
图3 多重PCR检测体系灵敏度检测
Fig.3 Sensitivity detection of multiplex PCR detection system
2.3 荧光定量PCR
应用荧光定量PCR检测多重PCR检测体系发现其能对3种样品进行准确快速的区分,其出峰时间与片段大小密切相关(图4)。

图4 人参、三七和西洋参多重荧光PCR检测
Fig.4 Multiplex real-time PCR detection of Panax ginseng, Panax notoginseng, and Panax quinquefolium
2.4 混合样品检测
人工模拟混合样品检测,图5中Ⅰ组为人参、三七、西洋参三种样品按照不同比例混合,其余6组为2种样品混合,提取基因组DNA后进行多重PCR检测。各泳道仅出现特异性条带,混合样品检出限为10%。但经过多次重复试验发现,条带亮度并不总是随着掺假比例的减少而增高,如图5中Ⅲ组为人参中混入西洋参,人参和西洋参质量比为4∶1时反而比1∶1和9∶1亮,这可能与2种样品粉末本身质量和基因组提取效率相关。
2.5 市售样品检测
应用改良的CTAB和试剂盒法提取市售产品的基因组,多重PCR检测结果表明生晒参片、红参片、西洋参片和三七粉等粗加工产品的检出率明显高于人参蜜片和人参茶等深加工保健品。选取市场上不同来源的粗加工产品进行多重PCR检测,结果显示在16份试售产品中有13份与其成分相符,检验阳性率达到80%(图6)。

M-DL 2000 DNA Marker
图5 混合样品多重PCR检测
Fig.5 Multiplex PCR detection of mixed samples
注:Ⅰ组1~3号样品中人参:三七:西洋参DNA含量体积比分别为1∶1∶1、
6∶3∶1、6∶1∶3;Ⅱ组1~3号样品中人参∶三七DNA含量体积比分别为1∶1、
4∶1、9∶1;Ⅲ组1~3号样品中人参∶西洋参DNA含量体积比分别为1∶1、4∶1、
9∶1;Ⅳ组1~3号样品中西洋参∶三七DNA含量体积比分别为1∶1、4∶1、9∶1;Ⅴ组1~3号样品中西洋参∶人参DNA含量体积比分别为1∶1、4∶1、9∶1;Ⅵ组1~3号样品中三七∶人参DNA含量体积比分别为1∶1、4∶1、9∶1;Ⅶ组1~3号样品中三七∶西洋参DNA含量体积比分别为1∶1、4∶1、9∶1
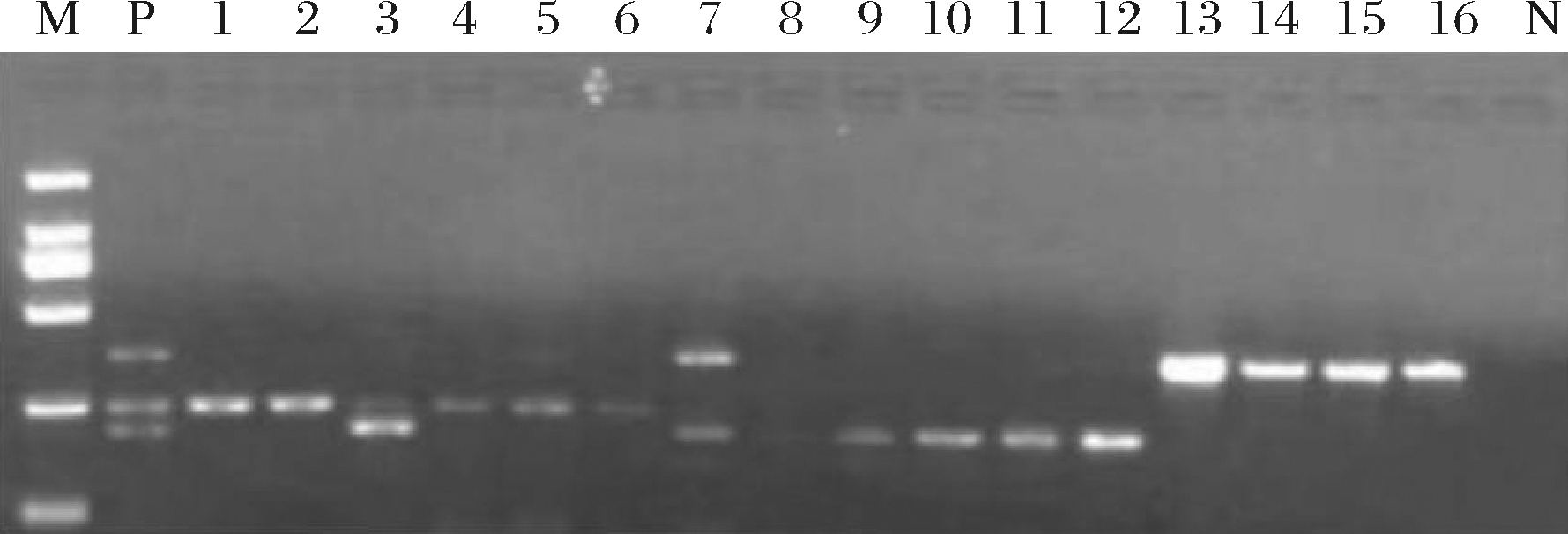
M-DL2000 DNA Marker;P-阳性对照;1~4-生晒参片;5~8-红参切片;9~12-西洋参切片;13~16-三七粉;N-空白对照
图6 市售粗加工样品多重PCR检测电泳图
Fig.6 Electrophoresis diagram of multiplex PCR detection of commercial rough processed samples
3 结论与讨论
随着现代医学的发展,人民生活水平和自我保健意识的提高,人参的价格和使用需求逐步增加,目前市场上的常见的人参产品有人参粉、人参片、人参颗粒和人参胶囊等。由于人参的经济价值和药用价值较高,且人参属植物的形态较为相似,导致一些商人在人参产品中掺入西洋参或三七等其他人参属植物。因此人参及参类产品的鉴别成为目前人参研究的重点。研究者已开发了多种人参的分子标记,其中包括限制性内切酶片段长度多态性(restriction fragment length polymorphism, RFLP)、扩增片段长度多态性(amplified fragment length polymorphism, AFLP)、随机扩增多态性DNA标记(random amplified polymorphic DNA, RAPD)、简单序列重复区间(inter-simple sequence repeat, ISSR)、和简单重复序列(simple sequence repeat, SSR)[6-10]。然而,上述方法均有不足之处,不适用于大批量样本的检测,难以自动化,例如RFLP和AFLP操作较为繁琐,很难比较分析,RAPD和ISSR很容易受到PCR反应条件微小变化的影响,SSR多态性通常需要用到聚丙烯酰胺凝胶电泳和银染等。
近年来,随着下一代测序技术的进步,表达序列标签的获得和高通量技术的应用使得SNP的发现和应用成为可能。与其他DNA分子相比SNP具有检测假阳性率低,分布广,密度高和具有代表性等优点被认为是简单有效的物品种鉴定方法。在人参方面的研究者中常用AS-PCR的方法检测SNP位点[11-14],通过引物3′端碱基与模板严格配对来实现SNP位点的检测,在实验过程中PCR反应体系,循环数和退火温度等均对PCR的扩增产生明显的影响,常常会出现非特异性扩增,且重复性不好。目前多采用引入错配的方式提高引物的特异性[15-16]。在实验的过程中发现仅凭借错配碱基无法确保实现引物3′末端的完全封闭,因此在SNP位点处引入LNA修饰,这使得引物3′末端完全匹配与错配的序列Tm值间存在着显著差异,可以将SNP更好地区分出来[17]。
9份市售样品的检测结果表明4份与所含原料相符,其中粗加工产品多重PCR检测阳性率明显高于深加工保健品。对市场上16份人参粗加工产品进行多重PCR检测,阳性率较高,达到80%以上。这可能存在掺假的问题,也可能由于市售样品中药材本身含量较低,加工过程中高温处理导致DNA严重降解或者产品本身添加辅料(如蜂蜜、淀粉、蔗糖和葡萄糖等)影响DNA提取效率。
本研究利用了基于SNP位点的多重PCR技术建立了人参、三七和西洋参的快速检测体系,通过LNA修饰和引入错配碱基的方式使3′末端实现完全封闭,达到增强引物特异性的目的,该方法灵敏度高,重复性好,基于常规的琼脂糖凝胶电泳即可对样品的成分进行检测,并可用于市售参类产品具体成分的鉴定。
[1] 王丽丽,林余霖,陈晓辰,等.基于SNP位点鉴定藏菖蒲及其近缘种[J].中国现代中药,2014,16(11):895-900.
WANG L L,LIN Y L, CHEN X C, et al.Molecular authentication of the oriental medicine Acorus calamus based on SNP markers[J].Modern Chinese Medicine, 2014,16 (11):895-900.
[2] 赵俊生,杨晓燕,曾祥有,等.利用SNP分子标记分析化橘红种质资源[J].分子植物育种,2016,14(5):1 203-1 211.
ZHAO J S, YANG X Y, ZENG X Y, et al.Analysis on germplasm resources of exocarpium citri grandis using SNP molecular markers[J].Molecular Plant Breeding, 2016,14 (5):1 203-1 211.
[3] LI X Y, XU H X, FENG J J, et al.Mining of genic SNPs and diversity evaluation of landraces in loquat[J].Scientia Horticulturae,2015,195:82-88.
[4] 沈奇,张栋,孙伟,等.药用植物DNA标记辅助育种(Ⅱ)丰产紫苏新品种SNP辅助鉴定及育种研究[J].中国中药杂志,2017,42(9):1 668-1 672.
SHEN Q, ZHANG D, SUN W, et al.Medicinal plant DNA marker assisted breeding(Ⅱ) the assistant identification of SNPs assisted identification and breeding research of high yield Perilla frutescens new variety[J].China Journal of Chinese Materia Medica, 2017,42 (9):1 668-1 672.
[5] JIND C, KOO D, HUR Y K, et al.Variation of RAPD patterns between male and female genomicDNAs in dioecious Rumex acetosa L.[J].Korean J.Plant.Res,2003,16(1):55-56.
[6] NGAN F, SHAW P, BUT P, et al.Molecular authentication of Panax species[J].Phytochemistry,1999,50(5):787-791.
[7] SHAW P C, BUT P P.Authentication of panax species and their adulterants by random-primed polymerase chain reaction[J].Planta Medica,1995,61(5):466-469.
[8] 刘丽,肖炳燚,聂平,等.采用RAPD技术对人参属的亲缘关系和鉴别的分析[J].药物分析杂志,2016,36(2):255-260.
LIU L, XIAO B Y, NIE P, et al.Analysis on genetic relations among 3 species and identification of Panax by RAPD marker[J].Chinese Journal of Pharmaceutical Analysis, 2016,36 (2):255-260.
[9] VASYUTKINA E A, ADRIANOVA I Y, REUNOVA G D, et al.A comparative analysis of genetic variability and differentiation in Panax vietnamensis ha et Grushv.and P.ginseng C.A.Meyer using ISSR markers[J].Russian Journal of Genetics,2018,54(2):262-265.
[10] 陈子易,吕旭楠,程舟,等.微卫星标记在人参和西洋参鉴别中的应用[J].复旦学报(自然科学版),2011,50(2):185-191.
CHEN Z Y, LYU X N, CHENG Z, et al.Application of microsatellite markers to differentiate Panax ginseng C.A.Meyer from Panax quinquefolius L[J].Journal of Fudan University (Natural Science), 2011,50 (2):185-191.
[11] 崔光红,唐晓晶,黄璐琦.利用多重等位基因特异PCR鉴别人参、西洋参[J].中国中药杂志,2006, 31(23):1 940-1 943.
CUI G H, TANG X J, HUANG L Q.Application of multiplex allele-specific PCR for authentication of Panax ginseng and P.quinquefolius[J].China Journal of Chinese Materia Medica, 2006, 31(23):1 940-1 943.
[12] 蒋超,罗宇琴,袁媛,等.多重位点特异性PCR鉴别人参、三七、西洋参掺杂[J].中国中药杂志,2017,42(7):1 319-1 323.
JIANG C, LUO Y Q, YUAN Y, et al.Identification of Panax ginseng, P.notoginseng and P.quinquefolius admixture by multiplex allele-specific polymerase chain reaction[J].China Journal of Chinese Materia Medica, 2017,42 (7):1 319-1 323.
[13] WANG H T, LI G S, KWON W S, et al.Development of EST intron-targeting SNP markers for Panax ginseng and their application to cultivar authentication[J].International Journal of Molecular Sciences,2016,17(6):884.
[14] YANG D U, KIM M K, MOHANAN P, et al.Development of a single-nucleotide-polymorphism marker for specific authentication of Korean ginseng (Panax ginseng Meyer) new cultivar “G-1”[J].Journal of Ginseng Research,2017,41(1):31-35.
[15] WANG H T, SUN H, KWON W S, et al.Molecular identification of the Korean ginseng cultivar "Chunpoong" using the mitochondrial nad7 intron 4 region[J].Mitochondrial DNA,2009,20(2-3):41-45.
[16] 王景,张甜甜,李萌, 等.利用SNP分子标记对人参品种大马牙的特异鉴定研究[J].中药材,2015,38(11):2 298-2 300.
WANG J, ZHANG T T,LI M, et al.Specific identification of ginseng variety horse teeth using SNP molecular markers[J].Journal of Chinese Medicinal Materials, 2015,38 (11):2 298-2 300.
[17] JOHNSON M P, HAUPT L M,GRIFFITHS L R.Locked nucleic acid (LNA) single nucleotide polymorphism (SNP) genotype analysis and validation using real-time PCR[J].Nucleic Acids Research,2004,32(6):e55.