作为谷氨酸的重要衍生物,5-氨基乙酰丙酸在生物体内有重要的作用,如参与血红素等四氢吡咯化合物的合成[1-2]。5-氨基乙酰丙酸具有促进植物生长、抑制昆虫繁殖等生物活性,故被广泛应用于农业和畜牧业等领域[3-4]。此外,5-氨基乙酰丙酸还作为光敏剂被应用于癌症的光动力学诊断和治疗[5-6]。
5-氨基乙酰丙酸的规模生产主要采用化学法合成,但由于合成步骤复杂、得率低等因素限制了其大规模生产和应用[7]。自从在微生物中发现其合成途径以来,生物法合成5-氨基乙酰丙酸被广泛关注。早期人们利用小球藻、类球红细菌等微生物的自身途径合成5-氨基乙酰丙酸,而后开展利用代谢工程等手段以大肠杆菌、谷氨酸棒杆菌等为底盘细胞构建合成菌株[1-2,8]。目前生物法合成5-氨基乙酰丙酸主要包括基于C4途径的前体物添加法和基于C5途径的发酵法[1,9-10]。前者以甘氨酸和琥珀酰辅酶A或琥珀酸为前体,经5-氨基乙酰丙酸合成酶催化合成5-氨基乙酰丙酸[10]。该方法使操作程序复杂化,且甘氨酸对细胞存在抑制作用,限制了其规模化生产[11]。故人们将注意力转向发酵法合成5-氨基乙酰丙酸。在C5途径中,葡萄糖经糖酵解途径和三羧酸循环生成α-酮戊二酸,然后经4步反应生成5-氨基乙酰丙酸(图1)[2,12-14]。与C4途径相比,利用C5途径合成5-氨基乙酰无需额外添加前体物,更易于操作。

Glc-葡萄糖;PEP-磷酸烯醇式丙酮酸;Pyr-丙酮酸;OAA-草酰乙酸;AcCoA-乙酰辅酶A;CIT-柠檬酸;ICI-异柠檬酸;α-KG-α-酮戊二酸;SUCC-琥珀酸辅酶A;Glu-谷氨酸;GlutRNA-谷氨酸酰tRNA;GSA-谷氨酸-1-半醛;5-ALA-5-氨基乙酰丙酸;PBG-胆色素原;Heme-血红素
图1 5-氨基乙酰丙酸C5合成途径及本研究主要策略
Fig.1 C5 pathway for synthesis of 5-aminolevulinic acid and the strategies used in this study
RAMZI等[15]首次在谷氨酸棒杆菌中构建了5-氨基乙酰丙酸合成途径实现其直接发酵合成。YU等[16]发现血红素的过量生成影响了5-氨基乙酰丙酸合成,并通过控制溶氧等方式抑制了血红素的合成。ZHANG等[17]通过敲除谷氨酸高产菌株S914中的谷氨酸分泌蛋白、精氨酸和脯氨酸分泌蛋白编码基因NCgl1221、lysE和putP以减少L-谷氨酸的消耗,从而提高5-氨基乙酰丙酸合成。KO等[18]利用α-酮戊二酸脱氢酶抑制蛋白OdhIT14A/T15A弱化谷氨酸棒杆菌α-酮戊二酸脱氢酶活性以提高α-酮戊二酸供应。ZHANG等[19]以大肠杆菌为宿主对C5途径进行系统代谢工程改造,通过表达谷氨酰-tRNA还原酶编码基因hemA和谷氨酸-1-半醛氨基转移酶编码基因hemL、增强磷酸吡哆醛供应、弱化5-氨基乙酰丙酸降解等策略获得工程菌株,在3 L发酵罐中实验发现5-氨基乙酰丙酸产量达到5.25 g/L。由此推测α-酮戊二酸供应和5-氨基乙酰丙酸的降解可能是影响5-氨基乙酰丙酸合成的关键要素。
有前期研究中,我们通过增强前体物合成、增加辅酶供应等策略强化了5-氨基乙酰丙酸的合成。在此基础上,本研究进一步考察限制5-氨基乙酰丙酸合成的因素,主要策略包括强化α-酮戊二酸合成、弱化α-酮戊二酸降解、动态调控血红素的生成、增强5-氨基乙酰丙酸输出等,为提升5-氨基乙酰丙酸的合成提供参考。
1 材料与方法
1.1 试剂与仪器
KH2PO4等无机盐,国药集团试剂有限公司;基因组DNA纯化试剂盒、PrimeSTAR® HS DNA Polymerase、QuickCut限制酶、RNAiso Plus、Titanium一步法RT-PCR试剂盒等,宝日医生物技术(北京)有限公司;ClonExpress® Ⅱ One-Step Cloning Kit,南京诺唯赞生物科技有限公司。
Ultimate 3000型高效液相色谱仪,美国ThermoFisher Scientific公司;PD-10脱盐滤柱,英国GE Healthcare公司。
1.2 菌株与质粒
本研究所用的菌株和质粒如表1所示。大肠杆菌Escherichia coli DH5α用于重组质粒的构建,E.coli W3110用于rhtA基因的扩增。
表1 本研究所用菌株和质粒
Table 1 Strains and plasmids used in this study

名称特性来源E.coli DH5αF-, Δ(lacZYA-argF)U169 recA1endA1 hsdR17实验室保存E.coli W3110野生型实验室保存AL6C.glutamicum Δcg1377cg1945∶∶Ptuf-pckAPppc∶∶Ptuf PgltA∶∶PtufaceA∶∶Ptuf-pntAB/pXMJ19ΔlacI-hemAM-hemL实验室保存AL7AL6Picd∶∶Ptuf本研究菌株AL8AL7PodhA∶∶PdapB本研究AL9AL8PhemB∶∶PCP_2836本研究AL10AL9携带pEC-XK99E-rhtA本研究AL11AL9携带pEC-XK99E-thrE本研究AL12AL9cg2973∶∶Ptuf-rhtA本研究pEC-XK99EKanar,Ptrc-MCS实验室保存pk18 mobsacBKanar,自杀质粒实验室保存pK18mobsacBPicd∶∶Ptufpk18 mob携带Picd上下游同源臂及Ptuf实验室保存质粒pK18mobsacBPodhA∶∶PdapBpk18 mob携带PodhA上下游同源臂及PdapB本研究pK18mobsacBPhemB∶∶PCP_2836pk18 mob携带PhemB上下游同源臂及PCP_2836本研究pK18mobsacBcg2973∶∶Ptuf-rhtApk18 mob携带cg2973上下游同源臂、Ptuf及rhtA本研究pEC-XK99E-rhtApEC-XK99E携带rhtA本研究pEC-XK99E-thrEpEC-XK99E携带thrE本研究
1.3 引物
本研究所用引物及其序列如表2所示。
1.4 培养基
LB培养基(g/L):蛋白胨10,酵母粉5,NaCl 10,pH 7.0~7.5。
BHIS培养基(g/L):BHI 37,山梨醇91,pH 7.0~7.5。
种子培养基:LB培养基中加入终质量浓度为0.5 g/L的葡萄糖,pH 7.0~7.5。
改良CGXⅡ培养基:CGXⅡ培养基中加入终质量浓度为3 g/L的酵母粉,pH 7.0~7.5。
表2 本研究所用引物
Table 2 Primers used in this study

引物名称序列(5′-3′)odhA-1TGCCTGCAGGTCGACTCTAGAAAACTCCTTTCCCGCCTCTAC∗odhA-2CTGCTAGTCCTGCTCCAGTTGTGACGGTCGGCCCTTTTAGCodhA-3GCTAAAAGGGCCGACCGTCACAACTGGAGCAGGACTAGCAGodhA-4CCTAAGCCCCAGTTGTAGCATTATGCTCCTTCATTTTCGTGGGodhA-5CCCACGAAAATGAAGGAGCATAATGCTACAACTGGGGCTTAGGodhA-6TATGACCATGATTACGAATTCAAGTTCATCTTCTCGGTTTCCTTGhemB-1TGCCTGCAGGTCGACTCTAGAGCTCTTGCGGAACACGTGGhemB-2TACTATTTTCACTGCAGTTGAGCGhemB-3CGCTCAACTGCAGTGAAAATAGTAAAACATGCTTGTCGACGCCGhemB-4TGGGTCTCCTTTGGGCChemB-5GGCCCAAAGGAGACCCAATGAGCACTTCTTCTGATTACTCCChemB-6TATGACCATGATTACGAATTCATCATTCCGGAAGGGCTCAChemB-7GCACTTCTTCTGATTACTCCCACGChemB-8GCAACGAACACCAGCATCTAcg2973-1TGCCTGCAGGTCGACTCTAGAAAATCTGGGAACTCCTCTGCGcg2973-2TTCTATCCACAGGTTTGGAGGATTcg2973-3AATCCTCCAAACCTGTGGATAGAAAGCGTGTCAGTAGGCGCGTAGGGTAAGTGcg2973-4TGTATGTCCTCCTGGACcg2973-5GTCCAGGAGGACATACAATGCCTGGTTCATTACGTAAAATGcg2973-6TTAATTAATGTCTAATTCTTTTATTTTGCTCTcg2973-7AGAGCAAAATAAAAGAATTAGACATTAATTAATGTGTCTGTTGCCGGAAAGGcg2973-8TATGACCATGATTACGAATTCCGCGTTCAGCCATGAGCAcg2973-9CCTATTAGCACACTCCAGCTACCAcg2973-10TGCGAAACTGAGCTATGAGAAAAArhtA-1AGGAAACAGACCATGGAATTCATGCCTGGTTCATTACGTAAAATGrhtA-2GCCAAGCTTGCATGCCTGCAGTTAATTAATGTCTAATTCTTTTATTTTGCTCthrE-1AGGAAACAGACCATGGAATTCATGTTGAGTTTTGCGACCCTTCthrE-2GCCAAGCTTGCATGCCTGCAGTTACCTTTTATTACCGAATCTCTGATTAG16S-FAGAACCACCGCCTGC TCACC16S-RCCGTCGTCGTAGTTGTACTCCTTG
注:*表示同源序列
1.5 质粒及菌株的构建
以大肠杆菌E.coli W3110基因组DNA为模板,利用引物rhtA-1/rhtA-2扩增rhtA基因,产物经电泳并纯化后利用ClonExpress® Ⅱ One-Step Cloning Kit连接至经线性化的pEC-XK99E质粒(经限制性内切酶Sac I酶切、电泳、回收并纯化),转化至E.coli DH5α[20-21]。挑取阳性菌落并提取重组质粒,分别利用EcoR I及EcoR I/Hind Ⅲ进行单、双酶切验证,以pEC-XK99E质粒为对照,将验证正确的质粒命名为pEC-XK99E-rhtA。将pEC-XK-99E-rhtA电转化至AL9,挑取阳性菌落进行菌落聚合酶链式反应(polymerase chain reaction,PCR),将验证正确的重组菌株命名为AL10。采用同样方法构建pEC-XK-99E-thrE和AL11。
采用同源重组的方法进行基因的基因组整合或启动子替换。以整合Ptuf-rhtA为例,利用引物cg2973-1/cg2973-2、cg2973-7/cg2973-8、cg2973-3/cg2973-4及cg2973-5/cg2973-6扩增cg2973上下游同源臂、Ptuf及rhtA,PCR产物经电泳并纯化后利用ClonExpress® Ⅱ One-Step Cloning Kit连接至经线性化的自杀质粒pK18mobsacB(经限制性内切酶Xba I酶切、电泳、回收并纯化),转化至E.coli DH5α经验证后获得pK18mobsacBcg2973∶∶Ptuf-rhtA。将pK18mobsacBcg2973∶∶Ptuf-rhtA电转化至AL9[20-21],挑取阳性菌落利用引物cg2973-9/cg2973-10进行菌落PCR(以AL9为对照),将验证正确的重组菌株命名为AL12。
1.6 实时定量PCR
收集发酵液,用RNAiso Plus提取总RNA并利用Titanium一步法RT-PCR试剂盒进行实时定量PCR检测,数据分析采用2-ΔΔCt法[22]。
1.7 α-酮戊二酸脱氢酶活性测定
收集发酵液经10 000 r/min(4 ℃)离心10 min后收集细胞,用N-三(羟甲基)甲基-2-氨基乙磺钠溶液(100 mmol/L,pH 7.0)洗涤3次后重悬。上述细胞重悬超声破碎20 min后经12 000 r/min(4 ℃)离心30 min,上清液经脱盐滤柱过滤。取100 μL滤液至含100 mmol/L N-三(羟甲基)甲基-2-氨基乙磺钠、5 mmol/L MgCl2、3 mmol/L半胱氨酸、3 mmol/L焦磷酸硫胺、0.2 mmol/L乙酰辅酶A及1 mmol/L NAD+的1 mL体系中,定时测定OD340值[23]。
1.8 发酵实验
将菌株于BHIS液体培养基活化后,以15%(体积分数)接种量接种至种子培养基,于32 ℃、220 r/min振荡培养8~10 h,至OD600=10~12。将种子培养物以15%(体积分数)接种量接种至含40 mL改良CGXⅡ培养基中,于32 ℃、220 r/min振荡培养48 h,发酵期间补加800 g/L葡萄糖,补加氨水使pH维持在7.0~7.5。上述培养中均含15 mg/L氯霉素。利用AL10和AL11发酵时,还需添加20 mg/L卡那霉素,发酵至8 h时添加终浓度为0.1 mmol/L的异丙基-β-D-硫代半乳糖苷(isopropylvbeta-D-thiogalactopyranoside,IPTG)。
1.9 检测与分析方法
菌体生物量以OD600值计,吸取1 mL发酵液适当稀释后利用分光光度计测定。用分光光度法测定5-氨基乙酰丙酸浓度,吸取1 mL发酵液于温室条件下经10 000 r/min离心10 min后取上清液,再次离心后适当稀释备用。配制1 mL反应体系,其中含上述上清液、0.25 mL乙酰丙酮以及0.5 mL的乙酸钠缓冲液(1 mol/L,pH 4.6),于沸水中水浴15 min后冷却至室温。取1 mL上述反应液与等体积二甲氨基苯甲醛混合,30 min后测定554 nm处吸光度[24]。取1 mL发酵液的上清液与等体积二甲氨基苯甲醛混合,30 min后测定555 nm处吸光度以测定胆色素原浓度[25]。用荧光法测定血红素浓度,取1 mL发酵液的上清液与等体积草酸溶液(2 mol/L)混合,测定662 nm处吸光度,激发光波长为400 nm[26]。
2 结果与分析
2.1 过表达icd基因对5-氨基乙酰丙酸合成的影响
在三羧酸循环中,关键酶异柠檬酸脱氢酶(由icd基因编码)催化异柠檬酸合成α-酮戊二酸,同时生成NADPH,二者均为5-氨基乙酰丙酸合成所需[27]。在出发菌株AL6中,已表达柠檬酸合酶编码基因gltA,故推测在此基础上表达icd基因可进一步将代谢流引向α-酮戊二酸,同时提高NADPH的供应,从而有利于5-氨基乙酰丙酸的合成。为考察表达icd基因对菌株合成5-氨基乙酰丙酸的影响,将icd基因的启动子替换为强启动子Ptuf。将pK18-Picd∶∶Ptuf转化至AL6,经两轮重组、筛选后挑取单菌落进行菌落PCR验证。结果如图2所示,从AL6和重组菌中获得碱基数约为1 200、1 600 bp的片段,分别与Picd替换前后碱基数一致(分别为1 179、1 621 bp)。表明启动子替换成功,将其命名为AL7。
摇瓶发酵实验考察AL7的特性,结果如图3-a所示。AL7较出发菌株AL6的生物量略有提高,其5-氨基乙酰丙酸的产量达到1.36 g/L,较AL6提高18.3%。该结果表明过表达icd基因可增强三羧酸循环代谢流从而为5-氨基乙酰丙酸的合成提供更多的α-酮戊二酸。
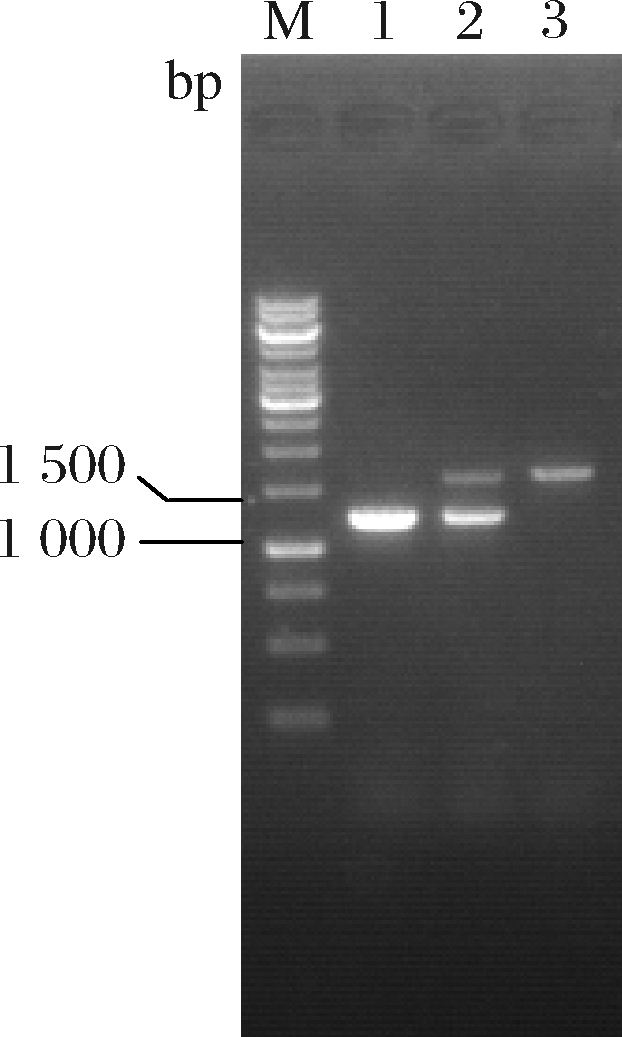
M-Marker;1-AL6菌落PCR;2-第1次重组菌株菌落PCR;3-AL7菌落PCR
图2 AL7菌落PCR鉴定图谱
Fig.2 Map for PCR identification of AL7

a-菌株5-氨基乙酰丙酸产量及生物量;b-菌株α-酮戊二酸脱氢酶活性
图3 菌株AL6、AL7和AL8发酵特性
Fig.3 Characteristics of AL6, AL7, and AL8
2.2 弱化α-酮戊二酸脱氢酶活性对5-氨基乙酰丙酸合成的影响
过表达icd基因后AL7的生物量较AL6高,表明增加的α-酮戊二酸除用于5-氨基乙酰丙酸合成外还用于生长。α-酮戊二酸可分别由谷氨酸脱氢酶和α-酮戊二酸脱氢酶催化生成L-谷氨酸和琥珀酰CoA,其中L-谷氨酸是合成5-氨基乙酰丙酸的前体物。研究发现,谷氨酸脱氢酶对α-酮戊二酸的米氏常数(Km=5.7 mmol/L)远高于α-酮戊二酸脱氢酶(Km=0.02~0.13 mmol/L),故α-酮戊二酸优先用于琥珀酰CoA的合成[27]。因此,调节α-酮戊二酸节点代谢流的分配对5-氨基乙酰丙酸的合成至关重要。有报道表明,通过敲除α-酮戊二酸脱氢酶E1亚基编码基因odhA失活α-酮戊二酸脱氢酶可以显著抑制菌体生长[11]。近年来,随着对谷氨酸棒杆菌系统生物学研究的不断深入,已发现多种不同转录强度的启动子。CHENG等[28]以绿色荧光蛋白编码基因gfp为报告基因,测定了来源于谷氨酸棒状杆菌的多个启动子的转录强度,发现4-羟基-2,3,4,5-四氢二吡啶甲基酸还原酶基因dapB的启动子为弱启动子。
为减少α-酮戊二酸转化为琥珀酰CoA的代谢流,拟将odhA基因启动子PodhA替换为PdapB以期弱化其表达从而降低α-酮戊二酸脱氢酶活性。采用重叠PCR扩增PodhA同源臂片段,并连接至自杀质粒pK18mobsacB,获得重组质粒pK18-PodhA∶∶PdapB。将pK18-PodhA∶∶PdapB转化至AL7,经两轮重组、筛选后挑取单菌落进行菌落PCR验证。结果如图4所示,从AL7和重组菌中获得碱基数约为1 400、1 600 bp的片段,分别与PodhA替换前后碱基数一致(分别为1 400、1 583 bp)。表明启动子替换成功,将其命名为AL8。

M-Marker;1-AL7菌落PCR;2-第1次重组菌株菌落PCR;3-AL8菌落PCR
图4 AL8菌落PCR鉴定图谱
Fig.4 Map for PCR identification of AL8
摇瓶发酵实验结果表明,与AL7相比,AL8生物量降低13.2%,但其5-氨基乙酰丙酸的产量提高41.2%,达到1.92 g/L(图3-a)。酶活性实验结果表明,AL8的α-酮戊二酸脱氢酶活性较AL7降低33.3%(图3-b)。上述结果说明弱启动子PdapB有效降低了odhA的转录致使α-酮戊二酸脱氢酶降低,从而使得一部分代谢流由生长转移至5-氨基乙酰丙酸的合成。
2.3 弱化5-氨基乙酰丙酸脱水酶编码基因hemB减少副产物生成
5-氨基乙酰丙酸可经5-氨基乙酰丙酸脱水酶催化生成胆色素原,后者经数步反应生成血红素。在AL8发酵过程中,发现其发酵液颜色逐渐变红,暗示有血红素生成。经检测,发酵液中胆色素原和血红素质量浓度分别达到12.35、5.31 mg/L。由于血红素是菌体生长必需物质,故不能通过敲除hemB等基因阻断胆色素原和血红素的合成。ZHANG等[17]通过将hemB自身核糖体结合位点序列替换为翻译强度较弱的核糖体结合位点以弱化5-氨基乙酰丙酸向胆色素原的代谢,使得5-氨基乙酰丙酸产量提高18.5%。YU等[16]在5-氨基乙酰丙酸脱水酶C端加入SsrA标签以动态降解该酶,使得胆色素原浓度降低约40%。在前期研究中发现来源于谷氨酸棒杆菌的乙醛脱氢酶启动子(PCP_2836)属于生长依赖型启动子,其转录强度在对数中后期显著下降[29]。故将hemB基因的启动子替换为PCP_2836以期动态调控hemB转录,提高5-氨基乙酰丙酸产量并降低胆色素原和血红素的积累。
采用重叠PCR扩增PCP_2836同源臂同源片段,并连接至自杀质粒pK18mobsacB,获得重组质粒pK18-PhemB∶∶PCP_2836。将pK18-PhemB∶∶PCP_2836转化至AL8,经两轮重组、筛选后挑取单菌落进行菌落PCR验证。结果如图5所示,从AL8和重组菌中获得碱基数约为1 200、1 500 bp的片段,分别与PhemB替换前后碱基数一致(分别为1 168、1 427 bp)。表明启动子替换成功,将其命名为AL9。
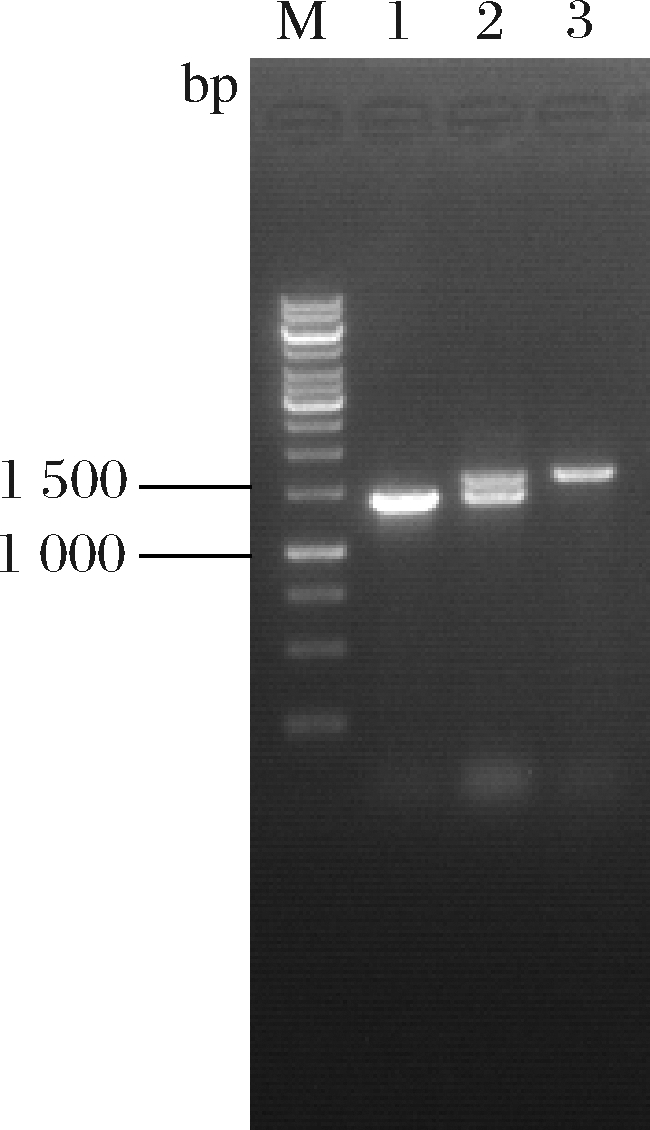
M-Marker;1-AL8菌落PCR;2-第1次重组菌株菌落PCR;3-AL9菌落PCR
图5 AL9菌落PCR鉴定图谱
Fig.5 Map for PCR identification of AL9
与AL8相比,AL9生物量未见显著变化,其5-氨基乙酰丙酸的产量提高12.0%,达到2.15 g/L(图6-a)。发酵液中胆色素原和血红素质量浓度分别达到1.69、0.24 mg/L,较AL8降低86.3%和95.5%(图6-b)。实时定量PCR结果表明,hemB的转录水平在发酵16 h后逐渐降低(图6-c)。上述结果表明,利用PCP_2836实现了对hemB基因转录的动态调控,从而弱化了5-氨基乙酰丙酸向血红素的代谢流,进而提高其产量。
2.4 增强输出对5-氨基乙酰丙酸合成的影响
研究表明来源于大肠杆菌的苏氨酸输出蛋白(由rhtA编码)能够识别5-氨基乙酰丙酸,并将其输出至胞外[8]。谷氨酸棒杆菌亦含有苏氨酸输出蛋白(由thrE编码)[30]。为考察苏氨酸输出蛋白对5-氨基乙酰丙酸合成的影响,扩增rhtA和thrE基因并分别连接至表达质粒pEC-XK99E,获得pEC-XK99E-rhtA和pEC-XK99E-thrE。将二者分别转化至大肠杆菌E.coli DH5α,经筛选后挑取单菌落并提取质粒进行酶切验证。如图7-a所示,经单酶切的pEC-XK99E-rhtA碱基数约为8 000 bp,与pEC-XK99E和rhtA碱基数之和一致(分别为7 018、888 bp);该质粒经双酶切分别获得碱基数约为7 000、900 bp的片段,表明pEC-XK99E-rhtA构建成功。同理证明pEC-XK99E-thrE构建成功(图7-b)。分别将质粒pEC-XK99E-rhtA和pEC-XK99E-thrE转化至AL9,获得菌株AL10和AL11。摇瓶实验结果表明,AL10和AL11的5-氨基乙酰丙酸产量分别达到2.51、2.32 g/L,较AL9提高16.7%和7.9%(图8),说明过表达苏氨酸输出蛋白有利于5-氨基乙酰丙酸的输出,且rhtA效果优于thrE。
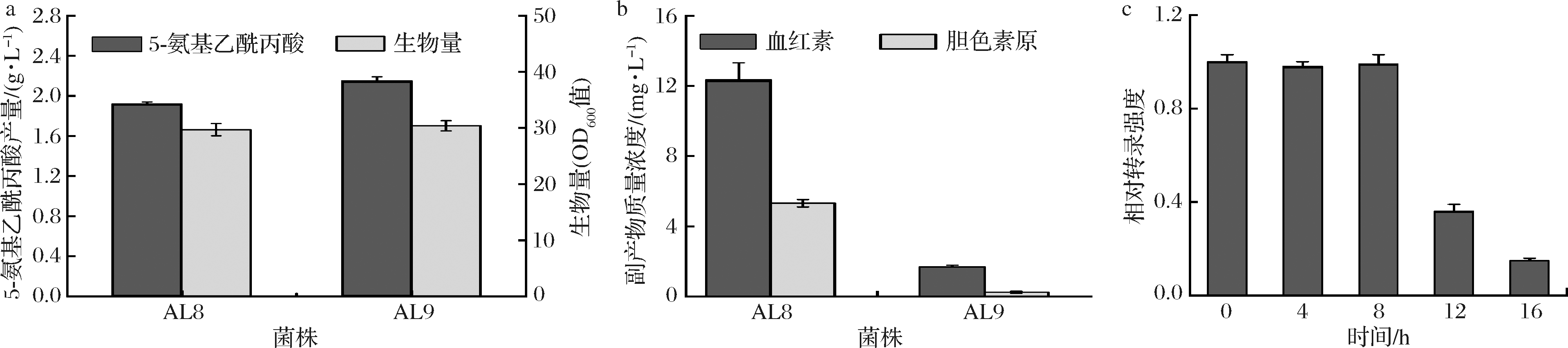
a-菌株5-氨基乙酰丙酸产量及生物量;b-副产物质量浓度;c-AL9中hemB相对转录强度
图6 菌株AL8和AL9发酵特性
Fig.6 Characteristics of AL8 and AL9
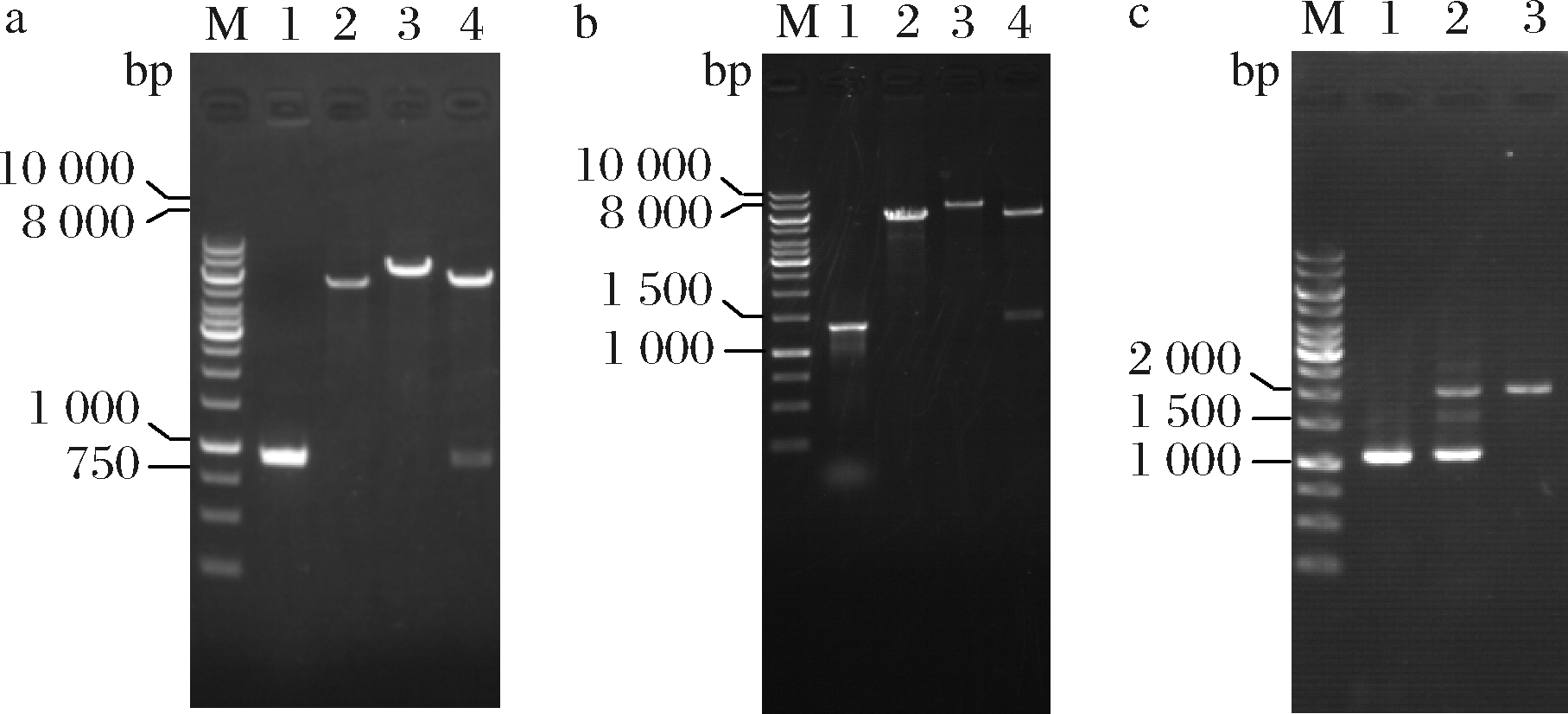
a-AL10和pEC-XK99E-rhtA鉴定图谱(M-Marker;1-AL10菌落PCR;2-pEC-XK99E经EcoR Ⅰ单酶切;3-pEC-XK99E-rhtA经EcoR Ⅰ
单酶切;4-pEC-XK99E-rhtA经EcoR Ⅰ和Hind Ⅲ双酶切) b-AL11和pEC-XK99E-thrE鉴定图谱(M-Marker;1-AL11菌落PCR;2-pEC-XK99E经EcoR Ⅰ单酶切;3-pEC-XK99E-thrE经EcoR Ⅰ单酶切;4-pEC-XK99E-thrE经EcoR Ⅰ和Hind Ⅲ双酶切) c-AL12鉴定图谱(M-Marker;1-AL9菌落PCR;2-第1次重组菌株菌落PCR;3-AL12菌落PCR) 图7 AL10、AL11和A12鉴定图谱
Fig.7 Map for PCR identification of AL10, AL11, and AL12
由图8可知,AL10和AL11的生物量分别较AL9降低12.2%和15.9%,推测可能是质粒增加了菌株的代谢负担。由于过表达rhtA基因的效果优于thrE,故采用基因组整合的方式过表达该rhtA基因,以期减小菌体代谢负担。利用重叠PCR扩增cg2973同源臂片段及强启动子Ptuf,并连接至自杀质粒pK18mobsacB,获得重组质粒pK18-cg2973∶∶Ptuf-rhtA。将pK18-cg2973∶∶Ptuf-rhtA转化至AL9,经两轮重组、筛选后挑取单菌落进行菌落PCR验证。结果如图7-c 所示,从AL9和重组菌中分别获得碱基数约为1 100、2 000 bp的片段,与Ptuf-rhtA整合前后碱基数一致(分别为1 169、2 067 bp)。表明Ptuf-rhtA整合成功,将其命名为AL12。AL12的发酵特性如图8所示,其生物量明显高于AL10,与AL9无显著差异;其5-氨基乙酰丙酸产量达到2.69 g/L,分别比AL9、AL10和AL11高21.5%、7.2%和15.9%。综上所述,采用基因组整合的方式对菌株代谢压力较小,优于质粒过表达的方式。

图8 AL10、AL11和A12的发酵特性
Fig.8 Characteristics of AL10, AL11, and AL12
3 结论
本研究首先通过表达icd基因强化α-酮戊二酸的合成并利用弱启动子下调odhA基因转录进而弱化α-酮戊二酸脱氢酶活性,为5-氨基乙酰丙酸的合成提供了前体物。然后利用生长依赖型启动子动态调控hemB转录,降低了5-氨基乙酰丙酸向血红素的代谢流。在此基础上证明谷氨酸棒杆菌苏氨酸输出蛋白ThrE具有输出5-氨基乙酰丙酸的功能,大肠杆菌苏氨酸输出蛋白RhtA效果更佳;采用基因组整合过表达rhtA的方式效果更好。获得的菌株经摇瓶发酵,其5-氨基乙酰丙酸产量达到2.69 g/L,与已报道的谷氨酸棒杆菌C5途径合成5-氨基乙酰丙酸产量接近。本研究证明调控α-酮戊二酸脱氢酶和5-氨基乙酰丙酸脱水酶是高效合成5-氨基乙酰丙酸的关键,故在后续研究中,拟进一步探索对其精准调控的策略(如利用感受胞内代谢物的启动子、群体感应系统等)。此外,NADPH供应也是合成5-氨基乙酰丙酸的瓶颈,后续拟利用辅因子工程等策略提高其供应,以期进一步提高其合成效率。
[1] KANG Z, DING W W, GONG X, et al.Recent advances in production of 5-aminolevulinic acid using biological strategies[J].World Journal of Microbiology and Biotechnology, 2017, 33(11):200.
[2] 陈久洲, 王钰, 蒲伟, 等.5-氨基乙酰丙酸生物合成技术的发展及展望[J].合成生物学, 2021, 2(6):1 000-1 016.
CHEN J Z, WANG Y, PU W, et al.Advances and perspective on bioproduction of 5-aminolevulinic acid[J].Synthetic Biology Journal, 2021, 2(6):1 000-1 016.
[3] TAN S Y, CAO J, XIA X L, et al.Advances in 5-aminolevulinic acid priming to enhance plant tolerance to abiotic stress[J].International Journal of Molecular Sciences, 2022, 23(2):702.
[4] HENDAWY A O, KHATTAB M S, SUGIMURA S, et al.Effects of 5-aminolevulinic acid as a supplement on animal performance, iron status, and immune response in farm animals:A review[J].Animals, 2020, 10(8):1 352.
[5] BROEKX S, WEYNS F, DE VLEESCHOUWER S.5-Aminolevulinic acid for recurrent malignant gliomas:A systematic review[J].Clinical Neurology and Neurosurgery, 2020, 195:105913.
[6] WAINWRIGHT J V, ENDO T, COOPER J B, et al.The role of 5-aminolevulinic acid in spinal tumor surgery:A review[J].Journal of Neuro-Oncology, 2019, 141(3):575-584.
[7] KAMIYAMA H, HOTTA Y, TANAKA T, et al.Production of 5-aminolevulinic acid by a mutant strain of a photosynthetic bacterium[J].Seibutsu-Kogaku Kaishi, 2002, 78(2):48-55.
[8] KANG Z, WANG Y, GU P F, et al.Engineering Escherichia coli for efficient production of 5-aminolevulinic acid from glucose[J].Metabolic Engineering, 2011, 13(5):492-498.
[9] CUI Z Y, ZHU Z W, ZHANG J H, et al.Efficient 5-aminolevulinic acid production through reconstructing the metabolic pathway in SDH-deficient Yarrowia lipolytica[J].Biochemical Engineering Journal, 2021, 174:108125.
[10] 王丽君, 闫思翰, 杨套伟, 等.代谢改造重组谷氨酸棒杆菌C4途径高效合成5-氨基乙酰丙酸[J].生物工程学报, 2021, 37(12):4 314-4 328.
WANG L J, YAN S H, YANG T W, et al.Engineering the C4 pathway of Corynebacterium glutamicum for efficient production of 5-aminolevulinic acid[J].Chinese Journal of Biotechnology, 2021, 37(12):4 314-4 328.
[11] NOH M H, LIM H G, PARK S, et al.Precise flux redistribution to glyoxylate cycle for 5-aminolevulinic acid production in Escherichia coli[J].Metabolic Engineering, 2017, 43:1-8.
[12] SHIH I T, YI Y C, NG I S.Plasmid-free system and modular design for efficient 5-aminolevulinic acid production by engineered Escherichia coli[J].Applied Biochemistry and Biotechnology, 2021, 193(9):2 858-2 871.
[13] CHEN J Z, WANG Y, GUO X, et al.Efficient bioproduction of 5-aminolevulinic acid, a promising biostimulant and nutrient, from renewable bioresources by engineered Corynebacterium glutamicum[J].Biotechnology for Biofuels, 2020, 13:41.
[14] 李智祥, 赵磊, 梁云龙, 等.生物法合成5-氨基乙酰丙酸的研究进展[J].发酵科技通讯, 2017, 46(3):178-182.
LI Z X, ZHAO L, LIANG Y L, et al.Advance on biosynthesis of 5-aminolevulinic acid[J].Bulletin of Fermentation Science and Technology, 2017, 46(3):178-182.
[15] RAMZI A B, HYEON J E, KIM S W, et al.5-Aminolevulinic acid production in engineered Corynebacterium glutamicum via C5 biosynthesis pathway[J].Enzyme and Microbial Technology, 2015, 81:1-7.
[16] YU X L, JIN H Y, LIU W J, et al.Engineering Corynebacterium glutamicum to produce 5-aminolevulinic acid from glucose[J].Microbial Cell Factories, 2015, 14:183.
[17] ZHANG B, YE B C.Pathway engineering in Corynebacterium glutamicum S9114 for 5-aminolevulinic acid production[J].3 Biotech, 2018, 8(5):247.
[18] KO Y J, YOU S K, KIM M, et al.Enhanced production of 5-aminolevulinic acid via flux redistribution of TCA cycle toward L-glutamate in Corynebacterium glutamicum[J].Biotechnology and Bioprocess Engineering, 2019, 24(6):915-923.
[19] ZHANG J L, WENG H J, ZHOU Z X, et al.Engineering of multiple modular pathways for high-yield production of 5-aminolevulinic acid in Escherichia coli[J].Bioresource Technology, 2019, 274:353-360.
[20] EGGELING L, BOTT M.Handbook of Corynebacterium glutamicum[M].Boca Raton:CRC Press, 2005.
[21] SAMBROOK J, MACCALLUM P, RUSSELL D.Molecular Cloning:A Laboratory Manual[M].New York:Cold Spring Harbor Laboratory Press, 2001.
[22] LIVAK K J, SCHMITTGEN T D.Analysis of relative gene expression data using realtime quantitative PCR and the 2(-Delta Delta C(T)) method[J].Methods:A Companion to Methods in Enzymology, 2013, 25(4):402-408.
[23] SHIIO I, UJIGAWA-TAKEDA K.Presence and regulation of α-ketoglutarate dehydrogenase complex in a glutamate producing bacterium, Brevibacterium flavum[J].Agricultural and Biological Chemistry, 1980, 44(8):1 897-1 904.
[24] 程雪莲, 王倩, 祁庆生.pH及溶氧对重组大肠杆菌发酵生产5-氨基乙酰丙酸的影响[J].中国酿造, 2016, 35(11):145-148.
CHENG X L, WANG Q, QI Q S.Effect of pH and dissolved oxygen on the fermentation production of 5-aminolevulinic acid by recombinant Escherichia coli[J].China Brewing, 2016, 35(11):145-148.
[25] MAUZERALL D, GRANICK S.The occurrence and determination of δ-aminolevulinic acid and porphobilinogen in urine[J].Journal of Biological Chemistry, 1956, 219(1):435-446.
[26] SINCLAIR P R, GORMAN N, JACOBS J M.Measurement of heme concentration[J].Current Protocols in Toxicology, 2001.DOI:10.1002/0471140856.tx0803s00.
[27] KRAWCZYK S, RAASCH K, SCHULTZ C, et al.The FHA domain of OdhI interacts with the carboxyterminal 2-oxoglutarate dehydrogenase domain of OdhA in Corynebacterium glutamicum[J].FEBS Letters, 2010, 584(8):1 463-1 468.
[28] CHENG F Y, YU H M, STEPHANOPOULOS G.Engineering Corynebacterium glutamicum for high-titer biosynthesis of hyaluronic acid[J].Metabolic Engineering, 2019, 55:276-289.
[29] MA Y C, CUI Y, DU L U, et al.Identification and application of a growth-regulated promoter for improving L-valine production in Corynebacterium glutamicum[J].Microbial Cell Factories, 2018, 17(1):185.
[30] SIMIC P, SAHM H, EGGELING L.L-threonine export:Use of peptides to identify a new translocator from Corynebacterium glutamicum[J].Journal of Bacteriology, 2001, 183(18):5 317-5 324.