牛乳是一种营养丰富、老少皆宜的食物,除直接饮用外,现也将一些植物原料如茶、果汁加入牛奶中,使产品形式和口味多样、营养更全面。植物原料中大都含有多酚,其具有保健作用,但也易与牛乳中蛋白形成复合物,进而影响多酚的生物活性、牛乳蛋白的性质和产品品质。很多学者都对蛋白与多酚的结合进行了研究,如两者间的结合类型、结合位点、结合机理等[1-4]。多酚与蛋白的交联主要靠非共价键,如氢键、疏水相互作用、范德华力、静电相互作用,在一定条件下也发生共价交联[5-6]。乳制品的杀菌仍以热杀菌为主,热杀菌对产品品质有负面影响。超高压是一种冷杀菌技术,其具有良好杀菌效果且几乎能够完好保留食物中小分子营养和风味物质,因此在包括乳制品的食品中应用越来越广[7-8]。超高压应用于牛乳时会改变乳蛋白的结构,进而影响乳制品的品质。一些学者报道了超高压处理对牛乳蛋白结构的影响,RUSSO等[9]表明压力处理时β-乳球蛋白的结构较稳定,仅300 MPa以上的压力会使蛋白逐渐展开;CONSIDINE等[10]发现超高压条件下β-乳球蛋白的变性或聚集主要是蛋白中二硫键重排引起的,小于100 MPa为天然构象阶段,150~350 MPa为二硫键发生重排的单体及二聚体,高于500 MPa主要为多聚体。超高压用于植物乳饮料处理后是否影响到多酚与蛋白的结合目前报道很少,CHEN等[11]报道了大豆蛋白与茶多酚在400 MPa高压下主要靠疏水作用和氢键交联,高压下蛋白对茶多酚的生物活性有保护作用。
光谱技术、波谱技术、量热技术、原子力显微镜等都被用于研究蛋白质结构,但测得的只是静态结构;生物体内蛋白质一直处于动态变化,现分子动力学模拟已成为研究蛋白结构的一种有效手段,成为继实验和理论手段后从分子水平了解和认识蛋白质世界的第3种手段,是一种具有足够小的时间尺度和空间尺度的模拟技术[12]。牛乳蛋白与许多小分子的结合都用分子动力学进行了模拟,如柑橘黄酮[13]、姜黄素[14]、芹菜素[15]、茶多酚[4]等。而超高压下的分子模拟较少,一些学者对胰岛素[16]和脂肪酶[12]在高压下的结构变化进行了模拟。
β-乳球蛋白是乳清中含量最为丰富的一种蛋白质,由于其良好的功能和营养特性在食品工业中有广泛的应用,其每个单体含有162个氨基酸,分子质量为180 kDa,是由8条反向平行的β-折叠构成的桶状结构,其外侧含有α-螺旋结构,对疏水及两性配体均具有良好的亲和力[17],而表没食子儿茶素没食子酸酯(epigallocatechin gallate,EGCG)是茶多酚中最主要的成分之一。因此本研究以β-乳球蛋白和EGCG为研究对象,用分子对接和分子模拟技术探讨蛋白经0.1、200、500、800 MPa的压力处理后,其与小分子结合能和结合机理的变化,以期为进一步提高茶乳饮料的质量和超高压杀菌技术在乳饮料中的应用奠定一定的理论基础。
1 实验方法
1.1 β-乳球蛋白与茶多酚的分子对接
β-乳球蛋白数据从RCSB网站下载(ID号:3npo),用PyMOL除去结晶水。EGCG的结构用ChemDraw Professional 17.1和 Chem3D 17.1准备,并用自带的MM2力场进行结构优化,通过AutoDock Tools (1.5.6版本)分别对蛋白和小分子进行处理,其中小分子加氢、计算Gasteiger电荷,蛋白加氢、计算Gasteiger电荷、合并非极性氢,生成β-乳球蛋白和EGCG的pdbqt文件。采用盲对接,小分子设置为全柔性,盒子大小设定为40 Å×40 Å×40 Å,中心坐标为默认值,设置间隔为1 Å。对接过程中采用AutoDock Vina搜索前20个最优的构象,对接参数energy_range设置为5,exhaustiveness设置为100。
1.2 不同压力条件下EGCG与乳球蛋白结合的分子动力学模拟
以1.1中的最优构象作为分子动力学模拟的初始结构,剥离出小分子的pdb结构,并在ATB网站生成小分子的top文件。分子动力学模拟采用Gromacs(2019.6)软件,首先将小分子合并到蛋白中,修改复合物的top文件;模拟时选用GROMOS54a7力场,将蛋白放入立方体水盒子中,水模型采用SPC模型,使蛋白离盒子边缘最短距离为1 nm并添加Na+使体系达到电中性,并使用最速下降法对体系进行能量最小化[13]。然后在NVT和NPT系综下分别进行400 ps的平衡,平衡后温度为300 K,压力达到预先设定的压力值。最后进行150 ns的分子动力学模拟,使用LINCS算法约束所有键,使用PME计算静电作用,范德华相互作用使用截断半径为1.4 nm进行计算。选用Parrinello-Rahman压浴方式,Isotropic为控压方式,使用V-rescale的控温方式。模拟步长为2 fs,每10 ps贮存1次数据,每个处理平行模拟2次。
模拟结束后,先去除周期性边界条件,然后使用gmx的rms命令分析各压力下小分子与蛋白的均方根误差(root mean squared error,RMSD);并用gmx的相应命令分析各压力下蛋白残基的波动[均方根涨落(root mean square fluctuation,RMSF]、蛋白二级结构、可及表面积的变化;并用能量分析命令分析蛋白的体积变化。RMSD分析全部150 ns的数据,而其余指标分析结构稳定后的数据,即100~150 ns。分析时每100 ps选取1个数据点,作图用Origin 8.0软件。
另外,将初始纯蛋白(3npo)也按上述方法在不同压力下进行150 ns的分子模拟,然后分别提取130、135、140、145、150 ns的蛋白结构,并按1.1的方法分别和小分子EGCG进行对接,以观察高压处理后小分子在蛋白的对接位点是否发生了变化。
1.3 MMPBSA计算小分子与蛋白的结合自由能
提取模拟过程中100~150 ns的轨迹(xtc文件),借助于g_mmpbsa工具,利用MMPBSA(molecular mechanics Poisson-Boltzmann surface area)方法计算小分子与蛋白之间的结合自由能[15]。
1.4 小分子与蛋白结合的二维平面图和表面结构图
提取各压力条件下复合物100~150 ns的平均pdb结构,用LigPlot+(V 2.2)软件分析小分子与蛋白在结合位点的疏水和氢键作用;并在PyMOL中绘制蛋白的表面结构图及与小分子结合情况。
2 结果与分析
2.1 茶多酚在蛋白的结合位点
通过分子对接,发现EGCG在β-乳球蛋白前20个能量较高的结合位点主要分布在2个区域,如图1所示,其中主要是结合在疏水腔上部的1号区域,另外还有位于蛋白表面的2号区域,在1号区域的平均对接能大于2号区域。关于多酚在β-乳球蛋白结合位点的报道较多,其中有研究认为在中性pH时小分子主要结合在疏水腔,而在酸性条件下则结合在蛋白表面[4,19],AL-SHABIBN等[20]也发现芦丁既可以结合在疏水腔也可以结合在蛋白表面,其中疏水腔的结合力更大,这与本研究结论一致。另有学者发现小分子吸附在乳球蛋白的表面,未结合在疏水腔[1,21],这可能与小分子结构、对接的蛋白初始结构、模拟条件等差异有关。
蛋白在不同压力条件下进行150 ns模拟后再进行分子对接,对接位点发生了明显变化,500 MPa及以下的压力处理后小分子仍结合到1、2号区域,其中常压下(0.1 MPa)的前20个位点主要仍是位于1号区域;而200和500 MPa处理后则在2号区域为最优区域,其结合能高于1号区域;800 MPa处理后,小分子的结合位点增多,除了1、2号区域外还增加了其他结合位点,如3、4和5号区域,没有明显的规律性,这种现象可能与蛋白的结构变化有关,800 MPa下蛋白结构发生变化后使可结合小分子的位点增加,或1、2号区域发生了不利于小分子结合的构象变化。
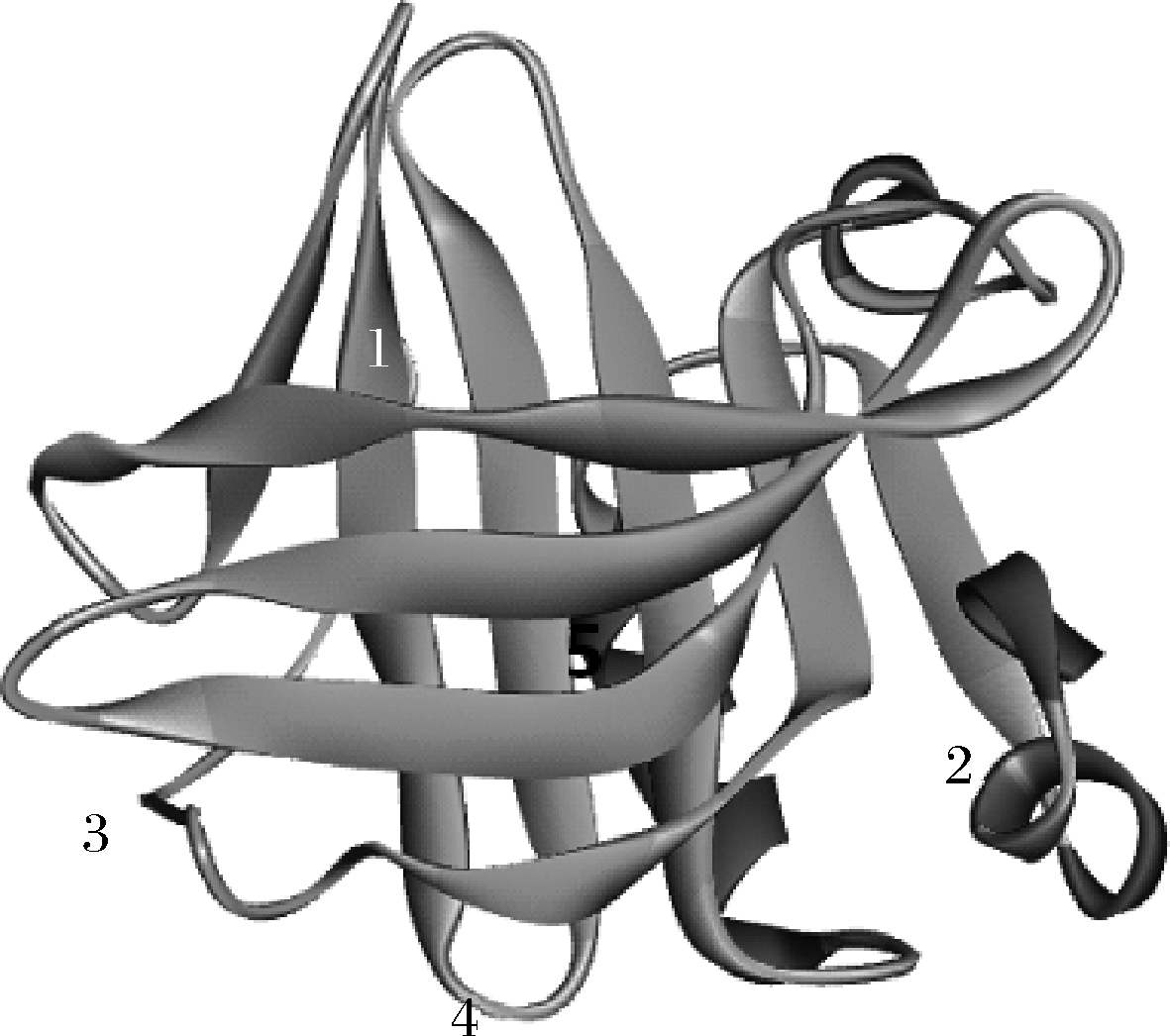
图1 EGCG在β-乳球蛋白的主要结合位点
Fig.1 Main binding sites of EGCG to β-lactoglobulin
2.2 EGCG与β-乳球蛋白结合的分子动力学
2.2.1 分子动力学模拟过程中RMSD的变化
RMSD是衡量特定时间蛋白结构与原始构象的平均偏差,是评价研究体系是否稳定的重要指标。由图2-a可知,500 MPa下小分子的波动较大,100 ns后才达到平衡,而其他压力下基本在30 ns左右就达到了平衡,从平衡后(均取100 ns后的数据)的平均值来对比,常压下为0.192 nm,与200 MPa下相当(0.194 nm),500 MPa(0.266 nm)时最高,800 MPa时为0.213 nm。就蛋白的RMSD而言(图2-b),均是40 ns左右就达到了平衡,且整体波动很小,说明β-乳球蛋白与EGCG的结合过程对蛋白结构影响很小,两者的结合总体平稳。同样,取100 ns后的平均值来衡量,常压下为0.249 nm,200 MPa下蛋白的RMSD低于常压(0.238 nm),随着压力的继续增加,蛋白的RMSD也增加,且高于常压,其中500和800 MPa下分别为0.272和0.259 nm。说明200 MPa的压力处理能使蛋白结构更稳定,而500和800 MPa的压力处理能使β-乳球蛋白的稳定性略为降低。SAHIHI等[13]报道柑橘黄酮与β-乳球蛋白间的结合很平稳,得到的RMSD值也较低;GHOLAMI等[21]研究柚皮苷和β-乳球蛋白的结合时发现柚皮苷的波动比蛋白小;ABDULATIF等[20]发现芦丁与β-乳球蛋白结合时,小分子与蛋白的波动相当,本研究中小分子的波动略低于蛋白,与上面的报道基本一致。KURPIEWSKA等[16]报道胰岛素在200或500 MPa处理时残基位移会减小;HATA等[22]也报道高压下蛋白的结构波动会减小,而本研究只发现200 MPa时蛋白波动小于常压。

a-小分子;b-蛋白
图2 不同压力处理模拟过程中小分子和蛋白的RMSD对比
Fig.2 RMSD of small molecule and protein during different pressure simulations
2.2.2 分子动力学模拟过程中RMSF的变化
RMSF主要反映蛋白中各氨基酸残基在不同压力处理时的波动情况,波动越大,说明该残基在压力处理中柔性越大,平均柔性越高蛋白结构也就越不稳定。从图3可知,不同压力处理体系中RMSF值相当,没有明显差异,说明高压总体上对残基波动影响不大。就小分子和蛋白结合口袋附近而言,残基波动较大的有86号位的Ala,87号位的Leu和88号位的Asn。RMSF大小与模拟常压下柑橘黄酮与β-乳球蛋白结合时得到的数据基本一致[13],但本研究中个别残基的波动更大,可能是受高压的影响。

图3 分子模拟过程中不同压力处理蛋白RMSF对比
Fig.3 RMSF of different pressure treatment during molecular dynamics simulation
2.2.3 分子动力学模拟过程中蛋白体积的变化
如图4所示,随压力的增加,蛋白体积显著减少,且体积在150 ns的模拟过程中保持恒定。根据Le Chatelier原理,高压下蛋白朝体积减小的方向进行,这可能是压力下,水分子进入疏水腔使疏水腔变形,导致蛋白的二级和三级结构发生变化进而体积减小[22]。KURPIEWSKA等[16]也报道了胰岛素在100 MPa下体积减少约1.4%。
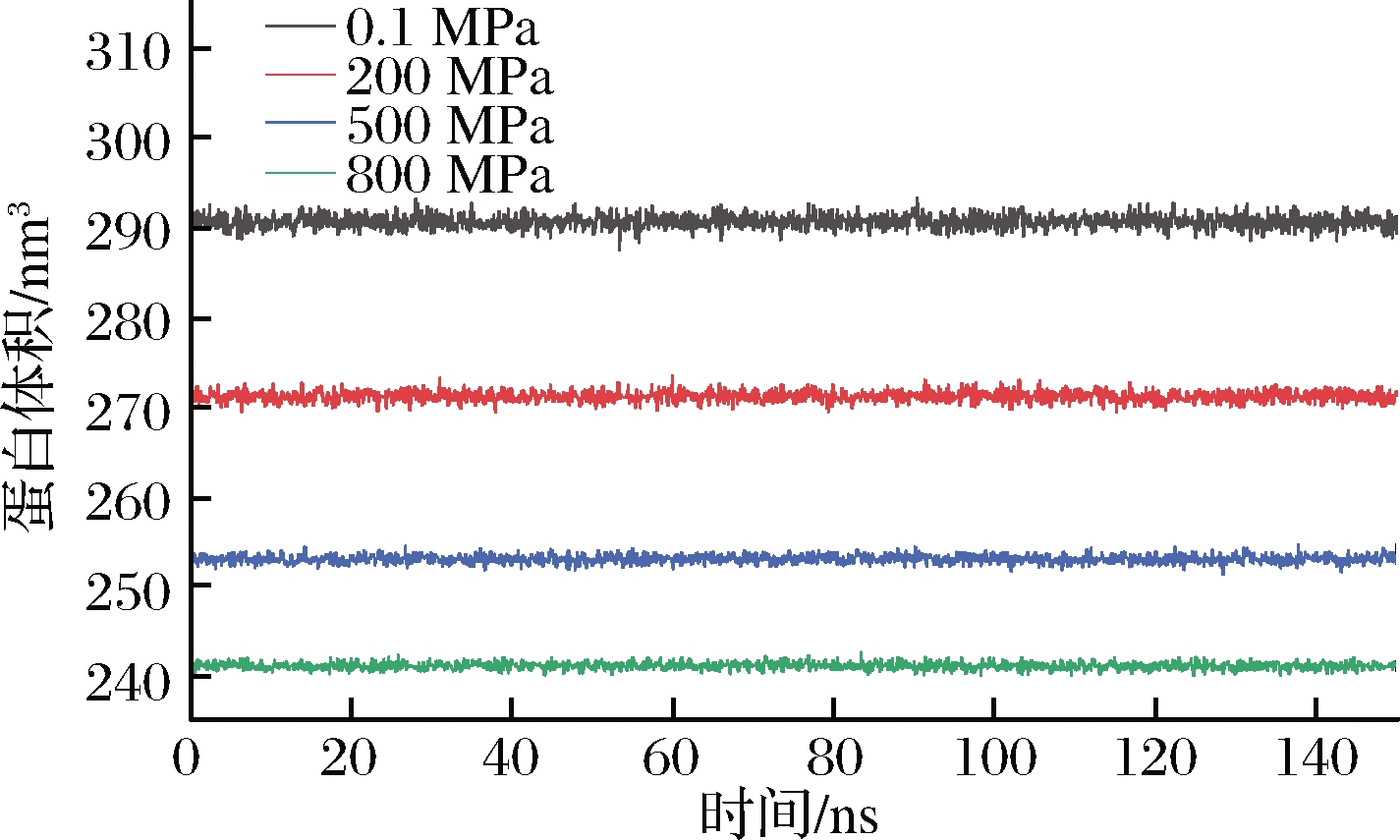
图4 不同压力处理模拟过程中蛋白体积变化
Fig.4 Volume comparison of different pressure treatment during molecular dynamics simulation
2.2.4 分子动力学模拟过程中蛋白溶剂可及表面积的变化
由表1可知,500 MPa以上的高压处理可以明显减少蛋白的溶剂可及表面积,其主要是由疏水表面积的减少所致,而亲水表面积变化不大。这可能是由于高压能导致蛋白总体积减少,从而使溶剂可及表面也减少,更多的疏水表面在压力作用下被掩埋。但各压力下亲水表面积均大于疏水表面积,因此压力作用不会改变蛋白的水溶性。疏水表面积的减少可能是高压条件下水分子渗透进入蛋白分子内部使更多的疏水区域暴露在极性水溶液中[23]。其他一些研究者也报道了高压下蛋白疏水性的变化,但结果并不一致,有的研究者认为随压力的增加蛋白的疏水作用减弱[11,22],而另一些学者报道高压下蛋白的疏水性增加[24-25]。这些研究均是采用实验手段研究表面疏水性,而本研究采用分子模拟研究疏水表面积的变化。
表1 不同压力处理对蛋白溶剂可及表面积的影响 单位:nm2
Table 1 Effect of different pressure on the solvent accessible surface area of lactoglobulin

处理压力总可及表面积疏水表面积亲水表面积0.1 MPa90.31±1.8036.95±1.1053.36±1.38200 MPa90.55±1.5636.60±1.0353.94±1.22500 MPa87.85±1.6034.79±0.9153.06±1.23800 MPa85.49±1.4833.48±0.8852.01±1.29
2.2.5 分子动力学模拟过程中蛋白二级结构的变化
如表2所示,200 MPa时蛋白的二级结构变化不大,主要是弯曲减少而转角增加;而500和800 MPa条件下,蛋白的二级结构发生了较大的变化,α-螺旋明显减少,β-折叠也有所降低,而无规则卷曲显著增加,500和800 MPa两者间差异并不明显。因此,压力较低时(200 MPa)对β-乳球蛋白的二级结构影响不大,而压力较高时(500 MPa以上)蛋白二级结构则变化较大。邱春江[24]研究高压处理肌原纤维蛋白时也发现在高压下蛋白的α-螺旋下降,α-螺旋对压力比较敏感,可能是高压下蛋白的氢键受到一定程度破坏,而氢键对α-螺旋的稳定至关重要[26],本研究也发现500 MPa及以上压力处理对α螺旋的破坏更大。白雨鑫[25]的研究表明500 MPa的压力对β-乳球蛋白的二级结构影响很小,CHEN等[11]研究发现400 MPa的压力处理能使大豆蛋白的α-螺旋减少而β-折叠增加,陈刚[12]报道脂肪酶在400 MPa处理时二级结构变化不大,而500 MPa以上时α-螺旋部分转化为无规则卷曲或β-折叠,这些差异可能是由蛋白不同或测定方法不同引起,上述报道大都采用实验手段测得而本研究是分子模拟。
表2 不同压力处理对蛋白二级结构的影响
Table 2 Effect of different pressure on the secondary structure of lactoglobulin
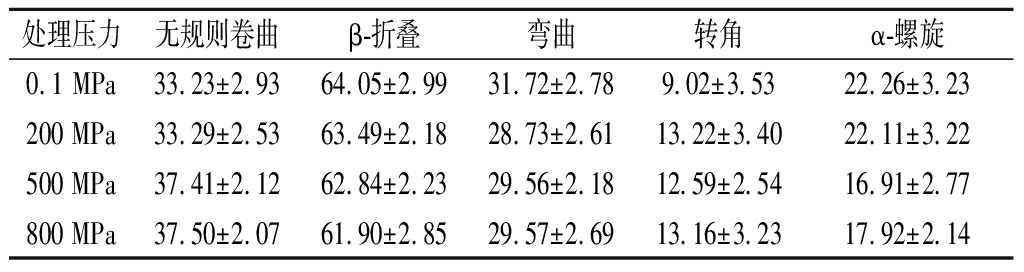
处理压力无规则卷曲β-折叠弯曲转角α-螺旋0.1 MPa33.23±2.9364.05±2.9931.72±2.789.02±3.5322.26±3.23200 MPa33.29±2.5363.49±2.1828.73±2.6113.22±3.4022.11±3.22500 MPa37.41±2.1262.84±2.2329.56±2.1812.59±2.5416.91±2.77800 MPa37.50±2.0761.90±2.8529.57±2.6913.16±3.2317.92±2.14
2.3 MMPBSA分析小分子与蛋白间的结合自由能
由表3可知,小分子和蛋白间的结合主要靠范德华力,静电作用力和非极性溶剂化自由能也有一定的贡献,而极性溶剂化自由能为正值,对蛋白的稳定有负面作用。随着压力的不断增加,静电相互作用力有所增加,但范德华力相比常压模拟体系均降低,另外极性溶剂化所消耗的能量也增加,而非极性溶剂化能基本不变。在以上几种作用力的综合作用下,总结合自由能随压力增加逐渐降低,因此在超高压作用下,小分子与蛋白的结合不如常压下稳定。除了高压对小分子结合的直接影响以外,这也可能与分子模拟只选择了对接能最高的1号结合位点有关,在2.1的分析中可知,高压处理后的蛋白更易结合在2号或其他位点,因此可以推断,β-乳球蛋白高压处理后在1号结合位点可能发生了不利于小分子结合的蛋白结构变化,具体通过后面的机制分析来阐明。
表3 不同压力对蛋白与EGCG结合的MMPBSA自由能的影响 单位:kJ/mol
Table 3 Effect of different pressure on the MMPBSA binding affinity between protein and EGCG

能量处理压力/MPa 0.1 200 500 800范德华力-220.583±11.664-199.349±17.722-163.066±12.591-175.805±14.404静电作用力-13.440±5.807-34.824±11.205-27.767±14.666-40.334±15.305极性溶剂化能97.808±11.017131.710±22.062101.359±26.155145.232±23.533非极性溶剂化能-23.664±1.003-22.229±1.333-18.005±1.120-20.291±0.776总吸附能-159.879±14.306-124.691±20.544-107.479±16.174-91.199±18.644
AL-SHABIBN等[20]研究芦丁和β-乳球蛋白的结合时,用MMGBSA计算出两者在蛋白表面的结合能为-68.011 2 kcal/mol,大于本研究的结果,这可能与小分子结构不同有关,也可能与计算方法不同有关,他们使用的是MMGBSA。ZHAN等[27]研究β-乳球蛋白与辣椒素结合时用MMPBSA计算得到的结合能是-142.744 kJ/mol,也是范德华作用为主要结合力,与本研究结果一致。陈刚[12]发现200 MPa处理时脂肪酶与乙酸乙酯的结合能增加,而400和600 MPa时结合能下降,其中600 MPa时下降更多,与本研究的结果稍有不一致。
从各个残基对能量的贡献来看,4个压力下贡献值均超过1 kJ/mol的有8个残基,分别是Leu31、Leu39、Val41、Leu58、Ile71、Val92、Met107、Glu108,大部分为疏水氨基酸,说明蛋白与小分子的结合疏水作用也是一种主要结合力。特别值得注意的是,压力增加时,Leu87、Met107和Ala118等残基的贡献显著减弱,具体的机制有待于进一步研究。
2.4 蛋白与小分子可能的结合机制分析
由图5-a可以看出0.1 MPa时小分子与蛋白的7个氨基酸存在疏水作用,分别是Leu39、Val41、Lys69、Ile71、Ala86、Met107、Glu108,小分子所处氨基酸环境与CHENG等[28]报道的矢车菊素-3-O-葡萄糖苷和KANAKIS等[17]报道的EGCG结合在β-乳球蛋白后所处的氨基酸环境基本一致。小分子与蛋白间形成6个氢键,分别与Asn88形成2个氢键、与Asn90间1个氢键、与Asn109间2个氢键,另外与Ser116间形成1个氢键。200 MPa时小分子所处的疏水环境和形成的氢键均和0.1 MPa没有差异,500 MPa时所处的疏水环境和0.1 MPa相同,但形成的氢键少了1个,其中与Asn109间只形成了1个氢键(200和500 MPa条件下的二维平面图未显示)。而800 MPa时所处的疏水环境和形成的氢键均发生了比较明显的变化,如图5-b所示,虽然也是和周围的7个氨基酸存在疏水相互作用,但氨基酸与前面3个压力不同,其中多了Leu87和Asn90,而少了Lys69和Ala86;形成的氢键只有4个,分别与Lys69、Asn88、Asn109和Ser116形成氢键。
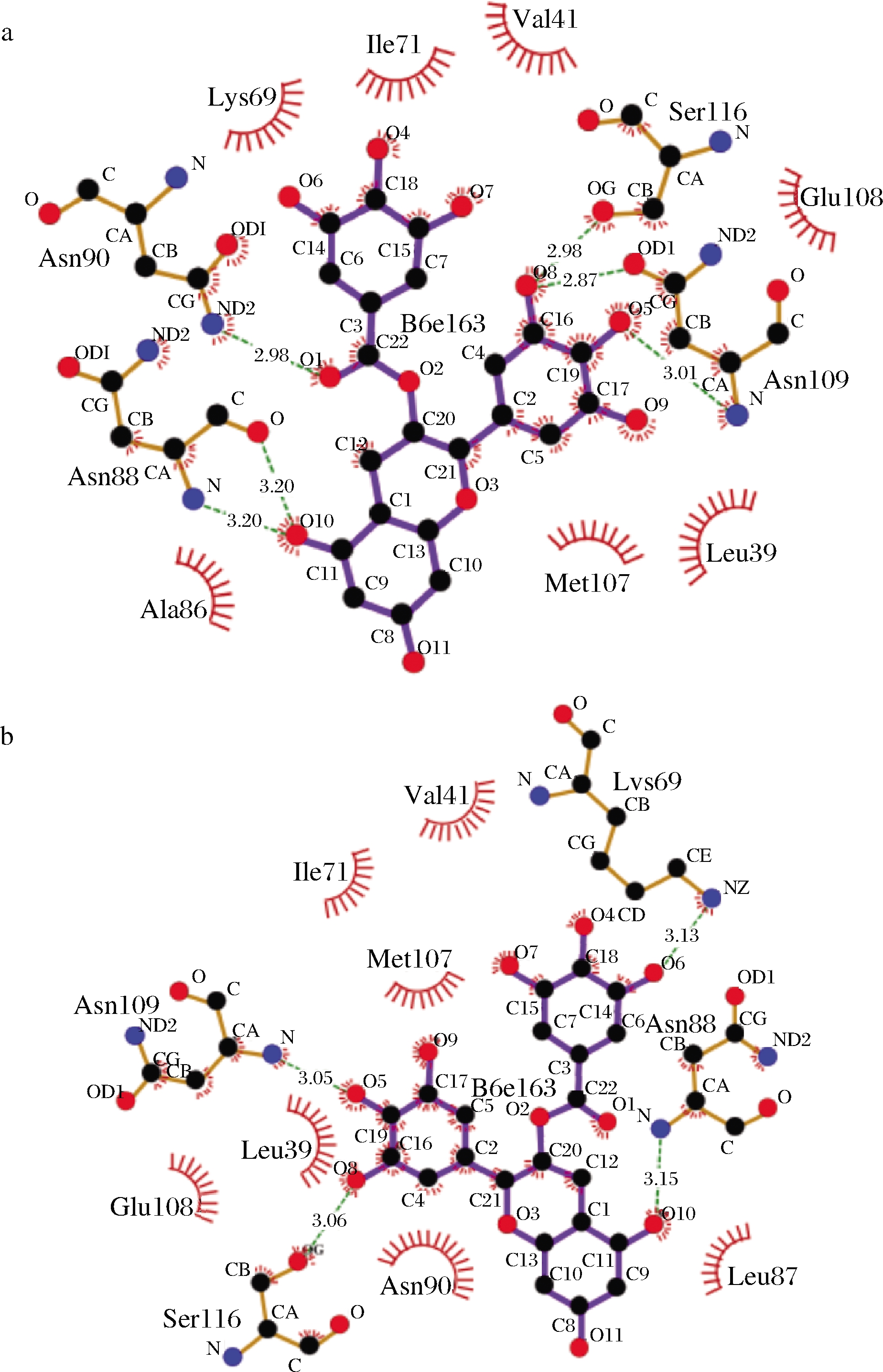
![]() 非配体键;
非配体键;![]() 配体键;
配体键;![]() 氢键及长度;
氢键及长度; ![]() 参与疏水作用的非配体残基;
参与疏水作用的非配体残基;![]() 参与疏水作用的相应原子 a-0.1 MPa;b-800 MPa
参与疏水作用的相应原子 a-0.1 MPa;b-800 MPa
图5 0.1 和800 MPa下小分子与蛋白相互作用的二维平面图
Fig.5 The 2D plot for interaction between EGCG and β-lactoglobulin under 0.1 and 800 MPa
注:B6e163在二维平面图中代表小分子
由表2可知,随着处理压力的增加,小分子与蛋白间的静电作用力也增加,可能的原因是除了氢键以外,蛋白与小分子间的其他弱静电相互作用也很重要[29]。结合2.3的结果可推断,压力处理整体上对小分子与蛋白间的疏水作用影响不大,但800 MPa时小分子所处的疏水环境有微小改变;另外,随着压力的增加(500 MPa及以上),小分子与蛋白间的氢键会有一定程度的破坏,这可能是高压下小分子与蛋白间结合能降低的重要原因之一。
贾晶晶[30]研究EGCG与β-乳球蛋白结合时发现,结合力以疏水作用为主,同时也有范德华力和氢键。CHENG等[28]报道矢车菊素-3-O-葡萄糖苷与β-乳球蛋白交联时发现主要结合力为氢键和疏水作用,且形成的其中2个氢键是与Asn109和Ser116,与本研究基本一致。本研究中随压力增加,小分子与蛋白间结合能逐渐降低应该是氢键、范德华力和静电相互作用力变化引起的综合效果,其中范德华力和氢键均减弱,静电作用有所增加;疏水作用虽然对两者的结合也很重要,但500 MPa及以下的压力没有影响到疏水结合力和小分子的疏水环境。
为进一步了解高压对蛋白表面结构及与小分子结合的影响,用PyMOL软件绘制了蛋白分子表面结构图,图6-a和图6-b分别为0.1和800 MPa下100~150 ns模拟过程中蛋白的平均表面结构及多酚与蛋白的结合情况,可以看出,蛋白在800 MPa时表面结构发生了显著变化,特别是上部临近疏水腔的口袋处,结合位点处蛋白结构的变化肯定会引起小分子结合姿势与结合能的差异,表3的数据也证实了不同压力下小分子与蛋白的结合能差异较大,说明压力越大,小分子与蛋白的结合位点处发生了越不利于两者结合的变化;前面的分子对接也表明压力处理后小分子的最佳结合位点并不在1号位;另外,0.1 MPa时小分子结构更加伸展(图6-a),能与蛋白更多残基发生作用,从而结合力也更高;这也与前面的分析是一致的,0.1 MPa时小分子与蛋白间形成的氢键更多。

a-0.1 MPa;b-800 MPa
图6 0.1和800 MPa下蛋白分子表面结构及与多酚结合情况
Fig.6 Surface structure and polyphenol binding of protein at 0.1 and 800 MPa
3 结论
不同压力下茶多酚在β-乳球蛋白上的结合位点不一样,其中常压下主要结合在疏水腔的1号位点,蛋白在200和500 MPa处理后主要结合在蛋白表面的2号位点,而800 MPa处理后除了1号和2号外,还增加了其他结合位点,这可能与高压下蛋白体积的收缩和蛋白变性引起的蛋白结构变化有关。以1号结合位点为目标进行分子动力学模拟,发现在150 ns的模拟过程中,200 MPa的压力处理能使小分子和蛋白的波动都较小,而500和800 MPa下小分子和蛋白的波动都增加,但蛋白整体上在结合过程中结构稳定。500 MPa及以上的高压下蛋白的α-螺旋和β-折叠会受到一定程度的破坏、蛋白疏水表面积减少,蛋白的表面结构也会在高压下发生较大的改变,特别是小分子与蛋白的结合口袋处,这些差异造成小分子与蛋白的结合自由能随压力的增加而降低,其中主要是范德华力和氢键的减少所致,而静电作用力虽然高压下有所增强,但影响有限。本研究为更好地了解蛋白与多酚在高压下的结合,进而促进高压在植物蛋白饮料杀菌的应用奠定了一定的理论基础。
[1] LI T,HU P,DAI T T,et al.Comparing the binding interaction between,β-lactoglobulin and flavonoids with different structure by multi-spectroscopy analysis and molecular docking[J].Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy,2018,201(18):197-206.
[2] ZHANG G W,WANG L,PAN J H.Probing the binding of the flavonoid diosmetin to human serum albumin by multispectroscopic techniques[J].Journal of Agricultural and Food Chemistry,2012,60(10):2 721-2 729.
[3] GHOLAMI S,BORDBAR A K.Exploring binding properties of naringenin with bovine β-lactoglobulin:A fluorescence,molecular docking and molecular dynamics simulation study[J].Biophysical Chemistry An International Journal Devoted to the Physical Chemistry of Biological Phenomena,2014,187-188:33-42.
[4] 刘婵.体外消化环境下多酚与蛋白、酶间的竞争相互作用及其对多酚和蛋白功能特性的影响研究[D].无锡:江南大学,2017.
LIU C.Competitive interaction between polyphenols and proteins,enzymes in vitro digestion and its effect on functional properties of polyphenols and proteins[D].Wuxi:Jiangnan University,2017.
[5] JAKOBEK L.Interactions of polyphenols with carbohydrates,lipids and proteins[J].Food Chemistry,2015,175(15):556-567.
[6] SUI X N,SUN H B,QI B K,et al.Functional and conformational changes to soy proteins accompanying anthocyanins:Focus on covalent and non-covalent interactions[J].Food Chemistry,2018,245(15):871-878.
[7] STRATAKOS A C,INGUGLIA E S,LINTON M,et al.Effect of high pressure processing on the safety,shelf life and quality of raw milk[J].Innovative Food Science & Emerging Technologies,2019,52:325-333.
![]() E.Can high hydrostatic pressure processing be the best way to preserve human milk?[J].Trends in Food Science & Technology,2020,101:133-138.
E.Can high hydrostatic pressure processing be the best way to preserve human milk?[J].Trends in Food Science & Technology,2020,101:133-138.
[9] RUSSO D,ORTORE M G,SPINOZZI F,et al.The impact of high hydrostatic pressure on structure and dynamics of β-lactoglobulin[J].Biochimica et Biophysica Acta(BBA)-General Subjects,2013,1 830(10):4 974-4 980.
[10] CONSIDINE T,PATEL H A,SINGH H,et al.Influence of binding conjugated linoleic acid and myristic acid on the heat-and high-pressure-induced unfolding and aggregation of β-lactoglobulin B[J].Food Chemistry,2007,102(4):1 270-1 280.
[11] CHEN G,WANG S T,FENG B,et al.Interaction between soybean protein and tea polyphenols under high pressure[J].Food Chemistry,2019,277:632-638.
[12] 陈刚.高压引发脂肪酶分子构象变化与催化行为关系的研究[D].无锡:江南大学,2017.
CHEN G.Study on the correlation between the conformational change and catalytic behavior of lipase induced by high pressure[D].Wuxi:Jiangnan University,2017.
[13] SAHIHI M,GHAYEB Y.An investigation of molecular dynamics simulation and molecular docking:Interaction of citrus flavonoids and bovine β-lactoglobulin in focus[J].Computers in Biology and Medicine,2014,51:44-50.
[14] MOHAMMADI F,SAHIHI M,BORDBAR A K.Multispectroscopic and molecular modeling studies on the interaction of two curcuminoids with β-lactoglobulin[J].Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy,2015,140(1):274-282.
[15] ZHU J X,LI K J,WU H,et al.Multi-spectroscopic,conformational,and computational atomic-level insights into the interaction of β-lactoglobulin with apigenin at different pH levels[J].Food Hydrocolloids,2020,105:105810.
[16] KURPIEWSKA K,MI ACZEWSKA A,K
ACZEWSKA A,K ![]() K.Insulin conformational changes under high pressure in structural studies and molecular dynamics simulations[J].Journal of Molecular Structure,2020,1 202:127251.
K.Insulin conformational changes under high pressure in structural studies and molecular dynamics simulations[J].Journal of Molecular Structure,2020,1 202:127251.
[17] KANAKIS C D,HASNI I,BOURASSA P,et al.Milk β-lactoglobulin complexes with tea polyphenols[J].Food Chemistry,2011,127(3):1 046-1 055.
[18] KUMARI R,KUMAR R,CONSORTIUM O S D D,et al.A_mmpbsa:A GROMACS tool for high-throughput MM-PBSA calculations[J].Journal of Chemical Information and Modeling,2014,54(7):1 951-1 962.
[19] 但倩誉.β-乳球蛋白与川陈皮素相互作用机制研究[D].武汉:华中农业大学,2019.
DAN Q Y.Research of interaction mechanism between beta-lactoglobulin and nobiletin[D].Wuhan:Huazhong Agricultural University,2019.
[20] AL-SHABIBN A,KHAN J M,MALIK A,et al.Molecular insight into binding behavior of polyphenol (rutin) with beta lactoglobulin:Spectroscopic and molecular docking and MD simulation studies[J].Journal of Molecular Liquids,2018,269(1):511-520.
[21] GHOLAMI S,BORDBAR A K,AKVAN N.Spectroscopic and dynamic properties of arachidonoyl serotonin-β-lactoglobulin complex:A molecular modeling and chemometric study[J].Journal of Photochemistry and Photobiology B,2016,162(9):519-528.
[22] HATA H,NISHIYAMA M,KITAO A.Molecular dynamics simulation of proteins under high pressure:Structure,function and thermo dynamics[J].Biochimica et Biophysica Acta (BBA)-General Subjects,2019,1 864(2):129395.
[23] KOLAKOWSKI P,DUMAY E,CHEFTEL J C.Effects of high pressure and low temperature on β-lactoglobulin unfolding and aggregation[J].Food Hydrocolloids,2001,15(3):215-232.
[24] 邱春江.超高压对鲢鱼中关键酶与结构蛋白质构影响的研究[D].无锡:江南大学,2014.
QIU C J.Effect of ultra-high pressure technology on key enzymes and structural proteins in silver carp processing[D].Wuxi:Jiangnan University,2014.
[25] 白雨鑫.四种非热加工对牛乳β-乳球蛋白结构和免疫特性的影响[D].南昌:南昌大学,2013.
BAI Y X.Effects of four non-thermal processing on conformational changes and immunological property of bovine β-lactoglobulin[D].Nanchang:Nanchang University,2013.
[26] MOZHAEV V V,LANGE R,KUDRYASHOVA E V,et al.Application of high hydrostatic pressure for increasing activity and stability of enzymes[J].Biotechnology & Bioengineering,1996,52(2):320-331.
[27] ZHAN F C,DING S,XIE W Y,et al.Towards understanding the interaction of β-lactoglobulin with capsaicin:Multi-spectroscopic,thermodynamic,molecular docking and molecular dynamics simulation approaches[J].Food Hydrocolloids,2020,105:105 767.
[28] CHENG J,LIU J H,PRASANNA G,et al.Spectrofluorimetric and molecular docking studies on the interaction of cyanidin-3-O-glucoside with whey protein,β-lactoglobulin[J].International Journal of Biological Macromolecules,2017,105(1):965-972.
[29] ZHANG J X,ZHANG L L,XU Y C,et al.Deciphering the binding behavior of flavonoids to the cyclin dependent kinase 6/cyclin D complex[J].Plos One,2018,13(5):e0196651.
[30] 贾晶晶.多酚类物质与牛乳β-乳球蛋白及β-半乳糖苷酶相互作用的研究[D].济南:山东师范大学,2018.
JIA J J.Study on interaction mechanism of β-lactoglobulin and β-galactosidase with phenolic compounds[D].Jinan:Shandong Normal University,2018.