蚕豆酱,也称豆瓣酱,通常以豆瓣、谷物、面粉为主要原料,接种米曲霉制曲后与盐水混合经天然晒制发酵而成,是我国传统的发酵调味品[1-2]。蚕豆酱不仅具有易于消化、口感细腻、营养丰富和风味独特等特点,还含有黄酮、花青素、多酚、矿物质及活性肽等多种生物活性物质[2-3],具有抗氧化、抗炎、抗肥胖、预防脂肪肝、清除放射性物质等多种生理功能[4]。蚕豆酱的天然晒制过程中涉及多种微生物,这些微生物与植物基原料降解及风味形成具有重要关系。解析微生物的多样性及其功能是筛选优良微生物菌种和实现精准人工调控的首要任务。近年来,高通量测序技术被广泛用于探究传统酿造食品的微生物群落组成。相关研究[5-7]开展了商业高盐发酵蚕豆酱微生物的多样性研究,结果表明蚕豆酱主要原核微生物包括葡萄球菌属(Staphylococcus)、芽孢杆菌属(Bacillus)、魏斯氏菌属(Weissella)、乳杆菌属(Lactobacillus)、不动杆菌属(Acinetobacter)、片球菌属(Pediococcus)、四联球菌属(Tetragenococcus)等7个属。
食盐作为蚕豆酱发酵的重要配料,形成高渗环境,可抑制有害微生物的生长繁殖,延长保质期,也会抑制有益微生物的生长代谢,对蚕豆酱发酵过程中的微生物群落结构演替有重要影响,从而影响蚕豆酱的抗氧化能力和品质特征[5, 8-12]。深入了解盐浓度对蚕豆酱发酵过程中的微生物群落结构的影响机制,对生产过程中食盐的添加量、工艺优化具有指导意义。本研究采用高通量测序技术对不同盐浓度蚕豆酱的原核微生物多样性进行了分析,测定了理化指标,包括理化因子变化和原核微生物群落演替。本研究不仅加深了我们对食盐调控蚕豆酱发酵的认识,也为今后低盐蚕豆酱的生产提供了指导。
1 材料与方法
1.1 材料与试剂
材料:蚕豆、面粉、食盐,本地超市;米曲霉(Aspergillus oryzae,沪酿 3.042),沂源康源生物科技有限公司。
试剂:甲醛、氢氧化钠、硝酸银(分析纯),国药集团化学试剂有限公司;PowerSoil® DNA提取试剂盒,美国Mobio公司;细菌16S rDNA V3~V4区扩增引物(338F:5′-ACTCCTACGGGAGGCAGCA-3′和806R:5′-GGACTACHVGGGTWTCTAAT-3′),广州基迪奥生物科技有限公司。
1.2 仪器与设备
PB-16 pH计,德国Sartorius公司;AB-40电子分析天平,瑞士Mettler-Toledo公司;MXX1400-30可调温电炉,上海微行机械设备有限公司;SHP-160D生化培养箱,江苏盛蓝仪器制造有限公司;Miseq PE250 Illumina测序仪,美国Illumina公司。
1.3 实验方法
1.3.1 蚕豆酱制备
根据YANG等[10]提供的工艺制备蚕豆酱,具体方法如下:蚕豆置于清水中浸泡8~10 h,沥干水分后于115 ℃蒸煮20 min,冷却后,将煮熟的蚕豆与面粉以质量比1∶3混合,接种1×107 CFU/g原料的米曲霉3.042孢子悬浮液。将接种后的混合物置于35 ℃和90%湿度的生化培养箱中制曲48 h,获得成曲。将成曲分别与不同质量浓度[12 g/100mL(L)、15 g/100mL(M)、18 g/100mL(H)]的盐水以质量比1∶1混合,置于5 L的玻璃发酵罐。用水液封后置于35 ℃培养箱恒温发酵35 d。制备了2批不同盐溶度的蚕豆酱样本。为保证取样的一致性,在发酵罐同一部位取3个样本(30 g/样本),取发酵7、21、35 d的样本,分别对应蚕豆酱的发酵前期、中期和后期[5],-20 ℃贮藏。
1.3.2 理化因子测定
使用pH计直接测定酱醅样本的pH值;参考GB 5009.235—2016《食品中氨基酸态氮的测定》测定氨基酸态氮的含量;参考GB 12456—2021《食品中总酸的测定》测定总酸的含量;参考GB 5009.39—2003《酱油卫生标准的分析方法》测定食盐的含量(以氯化钠计);参照GB 5009.33—2016《食品中亚硝酸盐与硝酸盐的测定》测定亚硝酸盐的含量;参考GB 5009.7—2016《食品中还原糖的测定》测定还原糖的含量(以葡萄糖计)。
1.3.3 DNA提取与Illumina Miseq高通量测序
根据PowerSoil® DNA提取试剂盒说明书的操作流程提取酱醅样本的DNA。为降低取样的误差,将同一发酵时间的3个样本的DNA进行合并。使用0.8%琼脂糖凝胶电泳及超微量分光光度计评估DNA的完整性、浓度及污染程度。将合格的DNA委托广州基迪奥生物科技有限公司完成16S rDNA扩增子V3~V4区建库及Illumina Miseq双端测序。
1.3.4 高通量测序数据分析
将测序数据根据条形码进行划分,随后使用FLASH软件[13]进行拼接,TRIMMOMATIC软件[14]过滤低质量的序列,UCHIME软件[15]去除嵌合体,得到高质量的优质序列。然后使用USEARCH软件[16]在相似性97%的水平上进行可操作分类单元(operational taxonomic unit,OTU)聚类,得到OTU的代表序列。代表序列经由Silva 138数据库[17]比对后进行物种分类注释,比较分析蚕豆酱样本中微生物群落结构及组成。基于注释结果计算各蚕豆酱样本的α-多样性指数及各注释水平的相对丰度。使用R软件对原核微生物群落差异性进行的主坐标分析(principal coordinates analysis,PCoA)、相似性分析(analysis of similarities,ANOSIM)和非加权组平均法(unweighted pair-group method with arithmetic means,UPGMA)聚类分析,理化因子与原核微生物群落间的相互关系进行冗余分析(redundancy analysis,RDA),理化因子与主要属水平微生物的关系进行相关性分析,不同发酵时间和不同盐浓度的原核微生物群落分别进行线性判别分析、效应大小分析(linear discriminant analysis effect size,LEfSe)(http://huttenhower.sph.harvard.edu/galaxy/)。
2 结果与分析
2.1 不同盐浓度发酵蚕豆酱的理化因子
总酸、氨基酸态氮、盐度、pH、亚硝酸盐和还原糖是蚕豆酱发酵过程中的重要理化因子。如图1所示,3种不同盐浓度蚕豆酱样本的pH值在发酵7~21 d内均显著下降(P<0.05),且在随后的发酵过程中保持稳定。样本L的pH值最低,其pH值仅为4.51,而样本H的pH值最高,为5.14。总酸含量的变化与pH值的变化呈相反的趋势,样本L的总酸含量最高,可达13.67 g/kg 鲜重(fresh weight,FW),而样本H的总酸含量最低,只有12.23 g/kg FW。蚕豆酱中总酸含量的增加会给产品带来强烈的酸味[18]。而低盐发酵蚕豆酱中总酸含量最高的原因可能是由于低盐对产酸微生物(乳酸菌)的抑制作用减弱,从而导致这类微生物大量生长[9]。
氨基酸态氮含量的变化与总酸含量的变化趋势一致,在7~21 d均显著增加(P<0.05),样本L的氨基酸态氮含量从6.57 g/kg FW增加至8.77 g/kg FW,而样本H的氨基酸态氮含量从5.21 g/kg FW增加至7.03 g/kg FW。发酵35 d,样本L、M和H中氨基酸态氮含量分别稳定在9.17、8.17、7.41 g/kg FW,其氨基酸态氮含量符合GB/T 24399—2009《黄豆酱》的规定。氨基酸是由芽孢杆菌属和其他微生物分泌的蛋白水解酶水解蛋白产生[5],低盐发酵蚕豆酱中氨基酸态氮含量最高的原因是由于较低盐度下渗透压低,对微生物生长繁殖及其分泌蛋白水解酶的活性抑制要小一些。
在整个发酵过程中,样本L、M和H中食盐含量分别稳定在4.84、6.80、8.86 g/kg FW。蚕豆酱样本的亚硝酸盐含量均在发酵7 d时最高,其在样本L、M和H中的含量分别为7.86、6.35、5.16 mg/kg FW。发酵35 d,蚕豆酱样本中亚硝酸盐含量均低于国家卫生标准GB 2762—2017《食品中污染物限量》规定的安全水平(20 mg/kg)。此外,蚕豆酱样本的还原糖含量在发酵7~21 d内均显著下降(P<0.05),且在随后的发酵过程中保持稳定。发酵35 d,样本L、M和H中还原糖含量分别稳定在32.64、26.83、23.27 g/kg FW。

a-pH;b-总糖;c-氨基酸态氮;d-盐;e-亚硝酸盐;f-还原糖
图1 不同盐浓度蚕豆酱发酵过程中理化因子的变化
Fig.1 The changes of physicochemical factors during the fermentation of broad bean paste with different salt concentrations 注:大写字母代表组件差异显著,小写字母代表组内差异显著(P<0.05)
2.2 不同盐浓度发酵蚕豆酱的原核微生物群落多样性
18个高通量测序样本共产生986 620条优质序列,平均每个样本51 328~59 121条。如图2所示,本研究所有样本的稀释曲线都达到平台期,说明本次测序深度足够覆盖蚕豆酱样本中绝大多数微生物种群信息。

图2 不同盐浓度蚕豆酱发酵过程中原核微生物群落的 稀疏曲线
Fig.2 Rarefaction curves of the prokaryotic microbial community during the fermentation of broad bean paste with different salt concentrations
根据表1可知,3种不同盐浓度蚕豆酱样本的Shannon指数和Simpson指数均随发酵时间的延长而逐渐降低。发酵35 d,样本L具有最高的Shannon指数和Simpson指数,分别为2.31和0.47,而样本H的Shannon指数和Simpson指数最低,分别仅为2.12和0.28,表明低盐发酵蚕豆酱具有更丰富的物种多样性。该结果与YANG等[10]的研究结果相一致,其发现高盐会降低发酵豆瓣酱的物种多样性。此外,3种不同盐浓度蚕豆酱样本的Pielou指数的变化与Shannon指数和Simpson指数的变化相一致。样本H的Pielou指数最低,表明减盐可提高蚕豆酱中原核微生物群落的物种均匀度。
表1 不同盐浓度蚕豆酱过程中原核微生物群落序列 数量和α多样性参数
Table 1 Sequence numbers and α-diversity indices of prokaryotic microbial communities during the fermentation of broad bean paste with different salt concentrations
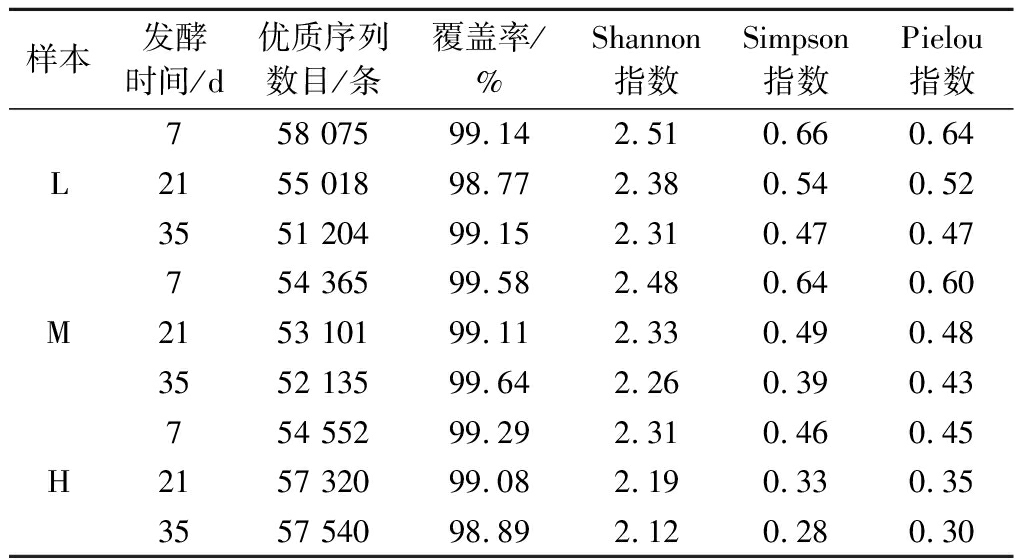
样本发酵时间/d优质序列数目/条覆盖率/%Shannon指数Simpson指数Pielou指数758 07599.142.510.660.64L2155 01898.772.380.540.523551 20499.152.310.470.47754 36599.582.480.640.60M2153 10199.112.330.490.483552 13599.642.260.390.43754 55299.292.310.460.45H2157 32099.082.190.330.353557 54098.892.120.280.30
注:表中数值为2个样本平均值
PCoA用于比较各蚕豆酱样本中原核微生物群落的差异。如图3所示,PCoA1和PCoA2两轴的物种累计解释率可达77.25%。此外,我们还发现组内样本的重复性较好,且具有时间一致性,发酵时间也会影响原核微生物群落组成。因此,我们进一步使用ANOSIM评估发酵时间和盐浓度对蚕豆酱发酵过程中原核微生物群落的影响,揭示不同盐浓度蚕豆酱样本发酵过程中原核微生物群落差异原因。由表2可知,不同发酵时间的蚕豆酱原核微生物群落之间存在显著差异(R=0.382,P=0.002),但盐浓度对蚕豆酱原核微生物群落差异的影响更大(R=0.554,P=0.001),表明发酵时间和盐浓度共同调节蚕豆酱发酵过程中原核微生物群落多样性变化。
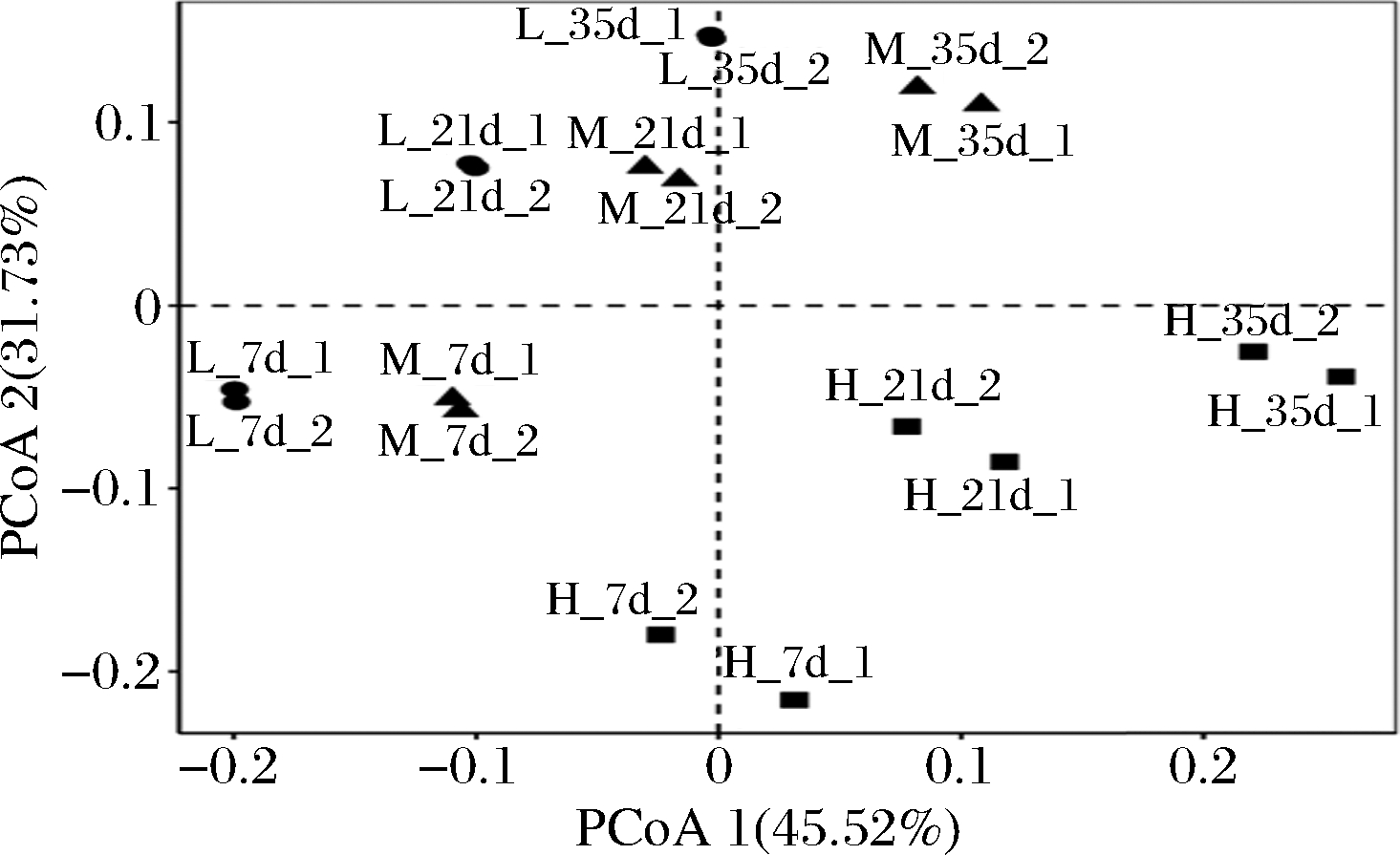
图3 不同盐浓度蚕豆酱发酵过程中原核微生物 群落组成的主坐标分析
Fig.3 PCoA of prokaryotic microbial community composition during the fermentation of broad bean paste with different salt concentrations
表2 不同盐浓度蚕豆酱发酵过程中原核微生物群落 差异的相似性分析统计数据
Table 2 ANOSIM statistics testing differences of prokaryotic microbial community groupings during the fermentation of broad bean paste with different salt concentrations
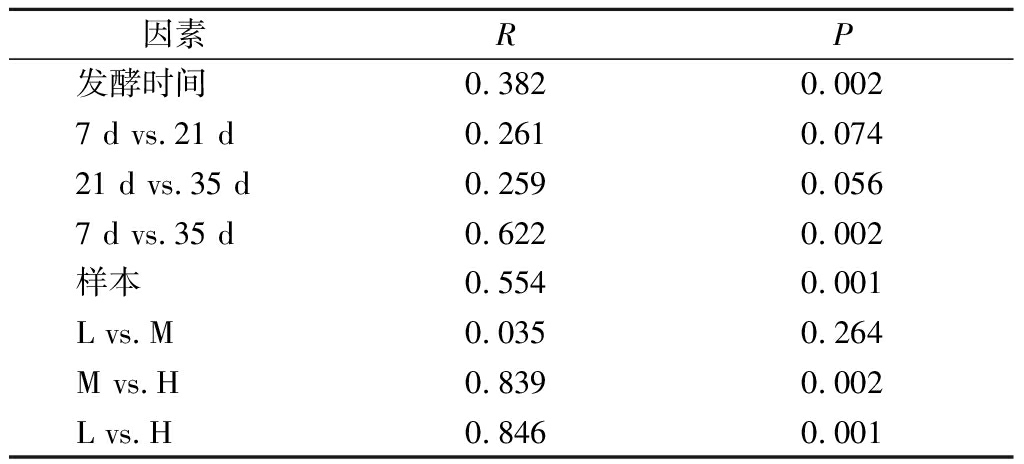
因素RP发酵时间0.3820.0027 d vs.21 d0.2610.07421 d vs.35 d0.2590.0567 d vs.35 d0.6220.002样本0.5540.001L vs.M0.0350.264M vs.H0.8390.002L vs.H0.8460.001
2.3 不同盐浓度发酵蚕豆酱的原核微生物群落组成
如图4所示,经分类学注释,厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)是蚕豆酱发酵的主要优势门(平均相对丰度≥1%),两者平均相对丰度分别为89.32%和7.68%,其他菌门的平均相对丰度之和仅为3%。样本L中厚壁菌门的相对丰度随发酵时间的延长而升高,从81.63%升高至93.31%,而样本M和H中厚壁菌门的相对丰度则呈先增后降的变化趋势。发酵35 d时,样本H中放线菌门(Actinobacteria)和盐厌氧菌门(Halanaerobiaeota)的平均相对丰度分别可达4.16%和1.64%,分别是样本L的4.8倍和5.1倍,表明高盐的酿造环境会促进耐盐微生物生长繁殖。

图4 不同盐浓度蚕豆酱发酵过程中原核微生物群落在 门水平的相对丰度
Fig.4 The relative abundance of prokaryotic microbial communities at phylum level during the fermentation of broad bean paste with different salt concentrations
由图5可知,通过UPGMA聚类可将18个样本划分为2个簇,样本H单独聚为1簇(Ⅰ),其他组样本聚为另一簇(Ⅱ);第Ⅱ簇又可以分为2个亚簇,样本L和M中发酵7 d的样本聚在第ⅰ亚簇,剩余样本聚在第ⅱ亚簇,该结果与图3和表2的结果相一致,说明高盐会显著改变蚕豆酱原核微生物的组成。在属水平上,葡萄球菌属、魏斯氏菌属、乳杆菌属、肠杆菌属(Enterobacter)、芽孢杆菌属、不动杆菌属、肠球菌属(Enterococcus)、欧文氏菌属(Erwinia)、盐厌氧菌属(Halanaerobium)、盐乳杆菌属(Halolactibacillus)、盐单胞菌属(Halomonas)、考克氏菌属(Kocuria)、片球菌属和四联球菌属是蚕豆酱发酵的主要优势属(至少1个样本中相对丰度≥ 1%)。Staphylococcus、Weissella和Bacillus在所有样本中均为绝对优势属,这与YANG等[10]研究结果相一致。葡萄球菌属,如肉葡萄球菌,是豆类发酵食品的主要贡献者之一,其主要参与酸性化合物和氨基酸的生成[19]。芽孢杆菌属能在蚕豆酱发酵过程中分泌蛋白水解酶,有利于促进氨基酸和蚕豆酱独特风味的形成[20]。魏斯氏菌属具有耐盐等特性,且可以代谢碳水化合物,为其自身生长繁殖提供能量[21]。发酵35 d,样本H中盐单胞菌属、片球菌属、盐乳杆菌属、考克氏菌属和四联球菌属的平均相对丰度显著高于其他2组的样本,其平均相对丰度分别为4.14%、5.41%、4.97%、4.16%和2.97%,表明高盐有利于这些微生物在发酵后期成为优势属。盐单胞菌属、片球菌属和考克氏菌属具有耐盐的特性,能分解葡萄糖,产生多种风味物质[22]。四联球菌属广泛存在于含盐的腌制类发酵食品中,可参与氨基酸的合成及生成醛、醇、酮和酯等挥发性风味物质,从而提升发酵食品的风味和口感[23]。肠杆菌属在发酵7 d的样品中为优势属,在21 d与35 d的样品中的丰度降低,也随盐浓度的升高而降低。肠杆菌属经常可以从各种植物表面分离出来,在发酵初期的样品中处于优势是合理的。随着发酵的进行,由于酸和盐形成的环境可以抑制肠杆菌属的生长,使其含量降低。

图5 不同盐浓度蚕豆酱发酵过程中原核微生物群落在 属水平的相对丰度及聚类分析
Fig.5 Cluster analysis and the relative abundance of prokaryotic microbial communities at genus level during the fermentation of broad bean paste with different salt concentrations
2.4 不同盐浓度发酵蚕豆酱的标志性微生物分析
为了探究不同发酵时间和不同盐浓度对发酵蚕豆酱中特定原核微生物种类的改变,使用LEfSe来鉴定这些特征微生物对组间差异的影响程度。由图6可知,发酵7 d样本显示出更高相对丰度的变形菌门、Verrucomicrobiota和乳杆菌科(Lactobacillaceae);发酵21 d样本只有明串珠菌科(Leuconostocaceae)的相对丰度更高;发酵35 d样本显示出潜在的耐盐微生物增加,如盐单胞菌科(Halomonadaceae)、微球菌科(Micrococcaceae)、盐厌氧菌科(Halanaerobiaceae)和芽孢杆菌科(Bacillaceae)。
为进一步明确盐浓度对蚕豆酱原核微生物群落分类特征的影响。由图7可知,样本L显示出来自乳杆菌目(Lactobacillales)的明串珠菌科具有更高的相对丰度;样本M只有棒状菌目(Corynebacteriales)的相对丰度更高;样本H的标志性微生物最多,分别是葡萄球菌科(Staphylococcaceae)和盐单胞菌科。
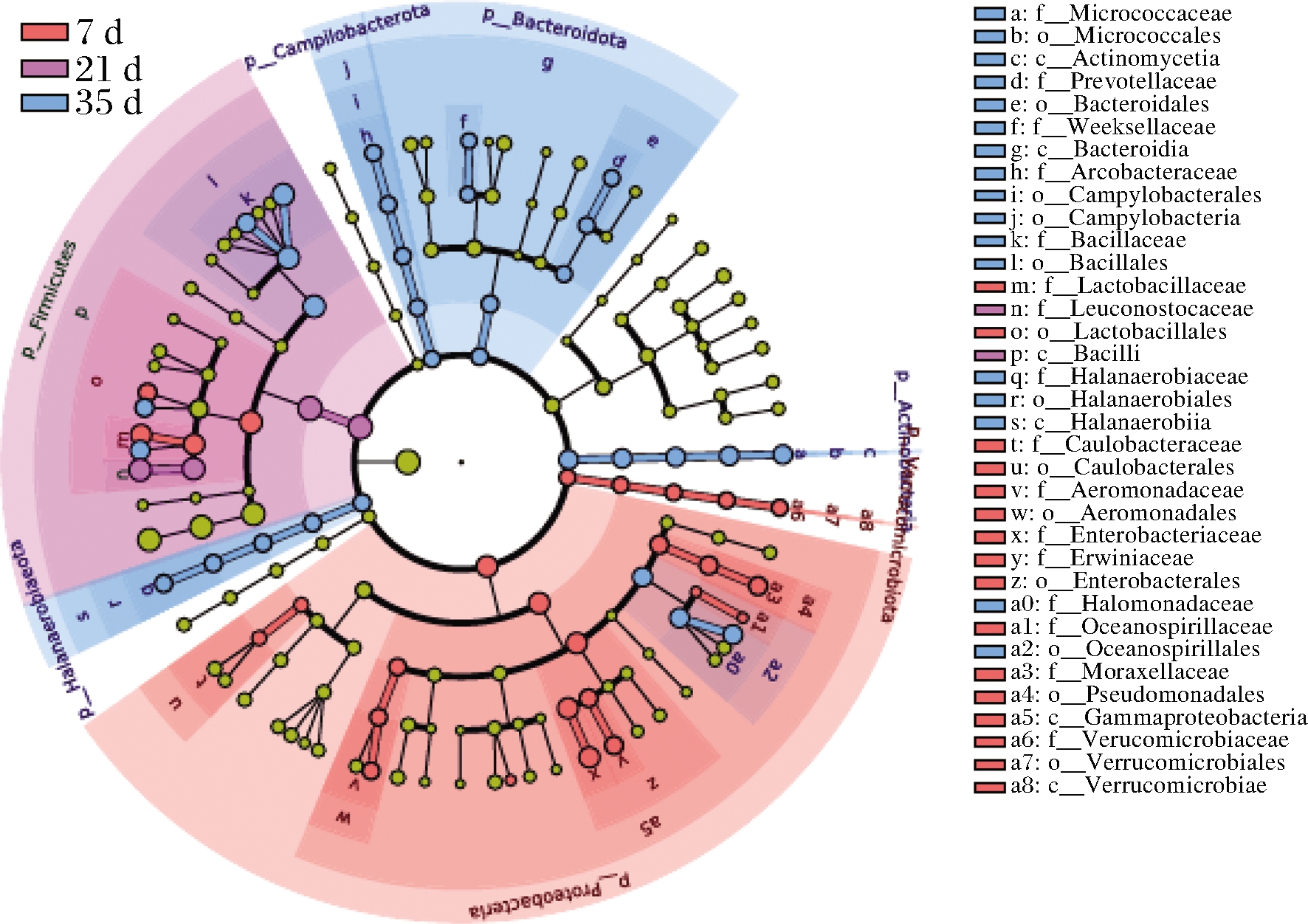
图6 不同盐浓度蚕豆酱发酵过程中不同发酵时期生物 标志性微生物的分析
Fig.6 Analysis of biomarker microorganism in different stage during the fermentation of broad bean paste with different salt concentrations

图7 不同盐浓度蚕豆酱发酵过程中不同盐浓度 生物标志性微生物的分析
Fig.7 Analysis of biomarker microorganism in different salt concentrations during the fermentation of broad bean paste with different salt concentrations
2.5 不同盐浓度发酵蚕豆酱的理化因子与原核微生物群落的关系
冗余分析用于揭示响应变量与解释变量之间的关系。如图8所示,RDA1和RDA2两排序轴解释种群与理化因子的累计变化率分别为46.73%和14.35%,两排序轴和为61.08%,说明冗余分析前2轴即可较好地反映出理化因子与蚕豆酱原核微生物群落的内在关联。pH和亚硝酸盐与发酵7 d的原核微生物群落呈正相关,而还原糖、氨基酸态氮和总酸与发酵35 d的原核微生物群落呈正相关。此外,利用条件限制分析揭示每个理化因子对蚕豆酱原核微生物群落结构的影响程度,发现盐(21.87%)和pH(18.63%)是对群落影响最大的2个理化因子,进一步表明盐和pH是蚕豆酱发酵过程中原核微生物演替的主要驱动力。

图8 不同盐浓度蚕豆酱发酵过程中理化因子与原核 微生物菌群之间的冗余分析关系图
Fig.8 RDA between physicochemical factors and prokaryotic microbial communities during the fermentation of broad bean paste with different salt concentrations 注:箭头表示理化因子与微生物群落结构之间关联性的方向与大小: 箭头的长度代表相关性的大小,中心点样本菌群点之间的连线与箭头 的夹角,锐角表示菌群与相应的理化因子呈正相关,钝角表示负相关
基于相关性分析探究与理化因子相关的主要优势属微生物,由图9可知,芽孢杆菌属、盐单胞菌属、片球菌属、嗜盐杆菌属(Halarcobacter)、盐厌氧菌属、考克氏菌属、盐乳杆菌属和四联球菌属作为发酵35 d的优势微生物,与盐浓度呈显著正相关(P<0.05)。不动杆菌属、乳杆菌属、肠球菌属、肠杆菌属、欧文氏菌属和魏斯氏菌属与盐浓度呈显著负相关(P<0.05)。此外,葡萄球菌属与总酸、氨基酸态氮和还原糖呈显著正相关(P<0.05),而芽孢杆菌属与这些理化因子的关系与葡萄球菌属相反。

图9 不同盐浓度蚕豆酱发酵过程中理化因子与属水平 原核微生物之间的相关性图
Fig.9 Correlation analysis between physicochemical factors and prokaryotic microbial communities in genes level during the fermentation of broad bean paste with different salt concentrations
3 结论
蚕豆酱是我国主要的豆类调味品之一,其含盐量可达9%~12%(质量分数),但关于不同盐浓度对蚕豆酱发酵过程中微生物群落的演替研究鲜有报道。本研究采用高通量测序和理化分析技术,揭示盐浓度对蚕豆酱发酵过程中原核微生物群落多样性和理化因子的影响。理化指标方面,添加12 g/100mL盐水的蚕豆酱pH值最低,发酵后积累的总酸、氨基酸态氮和还原糖较多。盐影响蚕豆酱发酵过程中原核微生物的多样性和组成,但对发酵过程中葡萄球菌属的生长没有影响。在不同盐浓度下,葡萄球菌属始终是发酵过程中的优势菌属。添加18 g/100mL盐水的豆瓣酱,发酵后葡萄球菌属的相对丰度最高。芽孢杆菌属、盐单胞菌属、片球菌属、盐厌氧菌属、盐乳杆菌属、考克氏菌属和四联球菌属受盐浓度的影响显著,且这些微生物与盐浓度呈显著正相关(P<0.05)。此外,冗余分析结果表明盐和pH是蚕豆酱发酵过程中原核微生物演替的主要驱动力。这些结果有助于了解加盐对蚕豆酱发酵生态的影响,为蚕豆酱的质量控制提供依据。
[1] 范子豪, 朱林江, 刘思远, 等.蚕豆酱传统发酵微生物群落中的菌株相互作用分析[J].食品与发酵工业, 2017, 43(4):28-33.
FAN Z H, ZHU L J, LIU S Y, et al.Interaction analysis of the strains isolated from traditional fermented broad bean paste[J].Food and Fermentation Industries, 2017, 43(4):28-33.
[2] 蒯辉, 朱林江, 刘春凤, 等.蚕豆酱传统酿造过程中细菌菌群的动态变化[J].食品与生物技术学报, 2017, 36(3):271-276.
KUAI H, ZHU L J, LIU C F, et al.Monitoring the change in bacterial community during traditional fermentation of broad bean sauce[J].Journal of Food Science and Biotechnology, 2017, 36(3):271-276.
[3] SHUKLA S, PARK J, KIM D H, et al.Total phenolic content, antioxidant, tyrosinase and α-glucosidase inhibitory activities of water soluble extracts of noble starter culture doenjang, a Korean fermented soybean sauce variety[J].Food Control, 2016, 59:854-861.
[4] KIM M J, KWAK H S, KIM S S.Effects of salinity on bacterial communities, Maillard reactions, isoflavone composition, antioxidation and antiproliferation in Korean fermented soybean paste (doenjang)[J].Food Chemistry, 2018, 245:402-409.
[5] JIA Y, NIU C T, LU Z M, et al.A bottom-up approach to develop a synthetic microbial community model:Application for efficient reduced-salt broad bean paste fermentation[J].Applied and Environmental Microbiology, 2020, 86(12).DOI:10.1128/aem.00306-20.
[6] LIU P, XIANG Q, SUN W J, et al.Correlation between microbial communities and key flavors during post-fermentation of Pixian broad bean paste[J].Food Research International, 2020, 137:109513.
[7] 鲍奕达, 张丽杰, 郑鹏飞, 等.郫县豆瓣发酵过程的微生物多样性及溯源分析[J].微生物学报, 2020, 60(11):2 555-2 571.
BAO Y D, ZHANG L J, ZHENG P F, et al.Diversity and source tracking of microbial community in Pixian broad bean paste[J].Acta Microbiologica Sinica, 2020, 60(11):2 555-2 571.
[8] NIU C T, FAN Z H, ZHENG F Y, et al.Isolation and identification of gas-producing spoilage microbes in fermented broad bean paste[J].Food Control, 2018, 84:8-16.
[9] XIONG T, LI J B, LIANG F, et al.Effects of salt concentration on Chinese sauerkraut fermentation[J].LWT, 2016, 69:169-174.
[10] YANG Y, NIU C T, SHAN W X, et al.Physicochemical, flavor and microbial dynamic changes during low-salt doubanjiang (broad bean paste) fermentation[J].Food Chemistry, 2021, 351:128454.
[11] TANG P Y, HU K, LIU C L, et al.Effect of partial sodium chloride replacement by other salts on the quality of western-style ham[J].Meat Research, 2019, 33(11):36-42.
[12] VIDAL V A S, SANTANA J B, PAGLARINI C S, et al.Adding lysine and yeast extract improves sensory properties of low sodium salted meat[J].Meat Science, 2020, 159:107911.
[13] ![]() T, SALZBERG S L.FLASH:Fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics, 2011, 27(21):2 957-2 963.
T, SALZBERG S L.FLASH:Fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics, 2011, 27(21):2 957-2 963.
[14] BOLGER A M, LOHSE M, USADEL B.Trimmomatic:A flexible trimmer for Illumina sequence data[J].Bioinformatics, 2014, 30(15):2 114-2 120.
[15] EDGAR R C, HAAS B J, CLEMENTE J C, et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics, 2011, 27(16):2 194-2 200.
[16] EDGAR R C.UPARSE:Highly accurate OTU sequences from microbial amplicon reads[J].Nature Methods, 2013, 10(10):996-998.
[17] QUAST C, PRUESSE E, YILMAZ P, et al.The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J].Nucleic Acids Research, 2013, 41:590-596.
[18] LEE S H, JUNG J Y, JEON C O.Effects of temperature on microbial succession and metabolite change during saeu-jeot fermentation[J].Food Microbiology, 2014, 38:16-25.
[19] YAO S J, ZHOU R Q, JIN Y, et al.Co-culture with Tetragenococcus halophilus changed the response of Zygosaccharomyces rouxii to salt stress[J].Process Biochemistry, 2020, 95:279-287.
[20] 吴博华, 蒋雪薇, 张金玉, 等.促酱醪发酵芽孢杆菌的筛选及应用[J].食品科学, 2021, 42(6):134-141.
WU B H, JIANG X W, ZHANG J Y, et al.Screening and application of Bacillus spp.promoting moromi fermentation[J].Food Science, 2021, 42(6):134-141.
[21] 胡传旺. 酱油发酵过程微生物群落解析及功能研究[D].无锡:江南大学, 2017.
HU C W.Dynamics of microbial community during soy sauce fermentation and functional analysis strains isolated from moromi[D].Wuxi:Jiangnan University, 2017.
[22] 吴晓红, 高生平, 蒋彩云, 等.榨菜发酵过程中原核微生物群落结构及其理化因子的动态演替[J].食品与发酵工业, 2021, 47(1):27-34.
WU X H, GAO S P, JIANG C Y, et al.Dynamic succession of prokaryotic microbial communities and physicochemical properties during the fermentation of Zhacai[J].Food and Fermentation Industries, 2021, 47(1):27-34.
[23] 王博, 周朝晖, 李铁桥, 等.嗜盐四联球菌及其在发酵食品中的应用[J].食品与发酵工业, 2017, 43(8):267-272.
WANG B, ZHOU Z H, LI T Q, et al.Tetragenococcus halophilus and its application in fermented foods[J].Food and Fermentation Industries, 2017, 43(8):267-272.