白酒是由多种微生物相互作用共同代谢生产的[1]。白酒酿造过程中的微生物主要可分为细菌、酵母和霉菌3大类,白酒发酵过程中这些微生物相互影响一起发挥重要作用。乳酸菌是白酒发酵过程中的优势菌种,其种类和数量的变化对白酒酿造过程至关重要[2-3]。乳酸菌既能代谢产生酸类、醛类等物质直接影响白酒风味,也能通过产生淀粉酶、酯化酶等影响白酒的出酒率和香味[4-5]。与其他菌种的相互作用方面,乳酸菌产生的乳酸、乙酸等有机酸和细菌素等拮抗物质可以影响其他微生物的生长[6-8],而其也能够通过乳糖酶将乳糖分解为葡萄糖和半乳糖,为酵母生长提供碳源[9]。乳酸菌作为白酒酿造过程中的优势菌群在白酒酿造过程中发挥着重要的作用,研究白酒发酵过程中乳酸菌分布特征及消长变化对于指导白酒生产有重要意义。
传统的检测方法是使用不同的选择性培养基对微生物进行培养分离, 但却不能及时反映一般条件难以分离培养的微生物的种类和数量变化。现在多种分子生物学技术手段被应用于食品中乳酸菌的定量[10]。实时荧光定量 PCR(quantitative real-time PCR,q-PCR)技术通过设计特异性引物有效地检验样本中的目标微生物,该方法灵敏、快速,广泛应用于食品微生物的检验中[11-13]。GUO等[14]和包秋华等[15]利用q-PCR检测研究了发酵乳中嗜酸乳杆菌,牛犇等[16]将传统方法q-PCR方法进行比较,发现q-PCR法可快速、高效地检测实际食品基质中的微生物。为了快速有效地分析和检测复杂微生态环境中的乳酸菌, q-PCR技术以其较高的可重复性、灵敏性以及准确性在微生物定量以及乳酸菌定量研究领域得到广泛应用[17-18]。
Lactobacillus jinshani是从中国白酒酿造系统中分离出的酿造微生物。YU等[19]首次从镇江香醋固态发酵过程中分离到1株革兰氏阳性、具有异型乳酸发酵特性的兼性厌氧菌株HSLZ-75T并命名为金山乳杆菌L.jinshani。DU等[20]根据L.jinshani分布频率高、相对丰度高的特点,将L.jinshani作为内标。用种特异性引物定量测定了内标物的绝对丰度后,用内标物归一化方法研究了发酵后的细菌群落及其动态。通过对乳酸菌的定量微生物群分析,确定乳酸菌是一种关键的风味物质产生菌,与8种风味物质相关。邢敏钰等[21]通过高通量测序发现L.jinshani是白酒酿造中后期优势微生物,并且通过相关性分析发现该菌与其他微生物呈负相关,其含量与产出原酒质量正相关,在发酵过程中扮演着重要角色。本研究根据L.inshani的全基因组序列设计特异性引物,建立了L.jinshani的q-PCR检测方法。该方法可快速有效的对L.jinshani进行鉴定,为研究其在白酒酿造系统中的演替及所发挥的功能奠定了坚实的基础。
1 材料与方法
1.1 材料
1.1.1 样品
本研究所用菌株均由本实验室从酒醅中分离获得,冷冻干燥后保藏于-80 ℃冰箱,包括L.jinshani在内的13种微生物分属5个属。实验所用酒醅均由湖北白云边酒业股份有限公司提供。
1.1.2 试剂和仪器
EB-溴乙锭,国药集团化学试剂有限公司;PCR引物(27F、1492R),上海生工生物工程技术服务有限公司;Taq DNA聚合酶、DL 2 000 DNA marker,大连宝生物公司。
电热恒温培养箱,上海新苗医疗器械制造有限公司;BIO-RAD T100 PCR仪,联想生物技术有限公司;超低温冰箱,青岛海尔股份有限公司;DYY-6C电泳仪,北京市六一仪器厂;自动凝胶成像分析系统,美国Syngene公司。
1.2 实验方法
1.2.1 基因组DNA提取
微生物纯培养物基因组DNA提取:收集处于稳定期的菌体,取1 mL于1.5 mL EP管中,离心收集菌体,按照DNA抽屉试剂盒步骤提取DNA。
酒曲及酒醅样本基因组DNA 提取:
(1)将50 g的样品与100 mL的抽提缓冲液放入烧杯中充分混合之后放入全温振荡机,4 ℃,180 r/min,振荡30 min,置入离心设备,中速离心5 min,滤除上层液体,留下底部沉淀。
(2)将沉淀放于烧杯内然后加入500 mL抽提缓冲液,混匀后,放于液氮中冷冻然后解冻融化,得到含目标的微生物体系。
(3)将目标微生物和10 μL 100 mg/mL溶菌酶和20 μL 200 mg/mL溶壁酶放入1.5 mL无菌EP管中混匀,然后放入全温振荡机37 ℃、180 r/min孵育1 h。
(4)加入60 μL 20%的十二烷基硫酸钠,30 μL 5%的十六烷基三甲基溴化铵(hexadecyl trimethyl ammonium bromide,CTAB)/NaCl和100 μL 5 mol/L NaCl,混匀后放入水浴锅中65 ℃水浴1 h;接着置入离心设备,恒温4 ℃中高速完成离心操作。
(5)静置分层后,取上层液体置入小于3 mL的离心管中,加入等量的苯类、酚类混合型物质溶液后进行离心操作,向原玻璃容器中加入0.1体积的,浓度为3 mol/L且pH值为5~7的乙酸钠溶液和2倍体积的无水乙醇溶液,搅拌后置于冷冻设备1 h。
(6)将装入溶液的离心管放入差速离心机内,高转速离心10 min,取出沉淀物置于干净的试管中,用体积分数为90%的医用酒精清洗沉淀,重复2次后恒温干燥,最后加入30 μL ddH2O溶解化合物的DNA,置于冷冻设备保存。
1.2.2 引物设计
通过Nr数据库对L.jinshani的全基因序列(GenBank 登录号为CP034726.1)进行系统的比对分析,选择了L.jinshani特有的单拷贝基因为目的基因,使用NCBI primer-blast设计特异性引物。
1.2.3 PCR 体系和条件
PCR反应体系(50 μL):2×Power Taq MaserMix 25 μL,DNA模板1 μL,引物各1 μL,三蒸水 22 μL。
PCR扩增16S rDNA,采用乳杆菌16S rDNA的通用引物27F(5′-AGAGTTTGATCCTGGCTCAG-3′)与1492R(5′-TACGGTTACCTTGTTACGACTT-3′)
PCR反应条件:94 ℃预变性5 min;94 ℃变性50 s,54 ℃退火50 s,72 ℃延伸90 s,30个循环;72 ℃延伸10 min。
q-PCR反应体系(20 μL):AceQ Universal SYBR Green q-PCR Master Mix 10 μL,上、下游引物(20 μmol/L)各0.2 μL,模板1 μL,补充ddH2O 至20 μL。
q-PCR的反应条件[22]:98 ℃ 1 min;98 ℃ 10 s,55 ℃ 30 s,72 ℃ 25 s,40个循环。溶解曲线均设定为从65 ℃升温至95 ℃,每增加0.5 ℃ 检测1次荧光信号强度,每次检测持续5 s。每次检测均含有3个技术平行。
1.2.4 有效性和特异性
使用含有L.jinshani的质粒、纯种L.jinshani基因以及含有L.jinshan.的酒醅基因组作为阳性对照,选择白酒酿造系统中常见12种非目标微生物的基因组作为阴性对照,通过PCR验证引物的有效性和特异性。根据阳性对照组扩增产物的琼脂糖凝胶电泳结果判断引物的有效性;将PCR 扩增产物送至苏州金唯智生物公司测序,根据测序结果判断引物扩增产物的特异性;根据阴性对照组扩增产物的琼脂糖凝胶电泳结果判断对于其他微生物的特异性。
1.2.5 载体的构建
利用设计的特异性引物,以酒醅样本基因组 DNA 为模板进行目标片段的扩增,取5 μL PCR 产物在1.5%含有染色液EB的琼脂糖凝胶上电泳检测扩增结果。目标片段经胶回收后与pMD19-T载体连接,利用质粒抽提试剂盒抽提质粒,使用特异性引物扩增测序验证是否转化成功。将已完全形成的质粒作为标样,采用蛋白核酸检测定量仪器来验证所测质粒的浓度大小,最后根据检测标准符合度,进行冷藏保存。
1.2.6 标准曲线的构建
构建好的质粒标准品以10倍稀释梯度进行稀释,共制备-1~-7共7个稀释梯度。以原始浓度和经过稀释的质粒标准品作为模板进行q-PCR,测定对应的Cq值,构建质粒拷贝数与Cq值之间的线性关系,绘制标准曲线。扩增效率E计算如公式(1)所示:
(1)
式中:E,扩增效率;a,标准曲线的斜率。
1.2.7 重复性检验
以10倍梯度稀释1.2.5中质粒样本,以稀释104 倍~107 倍的样本为q-PCR检测模板测定对应的Cq值,每个稀释度3次重复,计算Cq值的变异系数,对q-PCR检测方法的重复性进行评价。
1.2.8 q-PCR及高通量测序检测酒曲及酒醅中的L.jinshani
以酿造过程中酒曲及不同轮次的酒醅基因组DNA作为模板进行q-PCR检测,根据标准曲线计算出核酸浓度,样品中L.jinshani的含量计算如公式(2)所示。每次阳性对照选择L.jinshani纯种DNA,阴性对照选择无菌水,保证荧光定量PCR检测结果的准确性。
(2)
式中:K,拷贝数,copies/mL;ρ,标准品质量浓度,g/mL;MW为相对分子质量,Da。
同时将酒曲及酒醅基因组DNA送至武汉华大医学检验所有限公司,对扩增子片段进行高通量测序,分析结果中可操作分类单元(operational taxonomic units,OTUs),得到L.jinshani在样品细菌菌落中的百分比。
2 结果与分析
2.1 引物的设计
为构建q-PCR快速定性及定量检测方法,对L.jinshani的全基因序列(GenBank 登录号为CP034726.1)进行系统的比对分析,选择了L.jinshani特有的单拷贝基因为目的基因,如图1所示,使用NCBI primer-blast设计的特异性引物为:上游引物F:5′-TGCCACAAGGCTATCACCTC-3′, 长度为20 bp,(G+C)mol%为57.75%;下游引物R:5′-AACTCTTGGCTTCGCTGACG-3′, 长度为20 bp,(G+C)mol%为60.95%。扩增的PCR产物长度为226 bp,上下游引物不存在成环、发夹等结构,将此特异性引物进行blast比对结果如图2所示,对目标片段具有特异性,可用于后续q-PCR检测方法的构建。
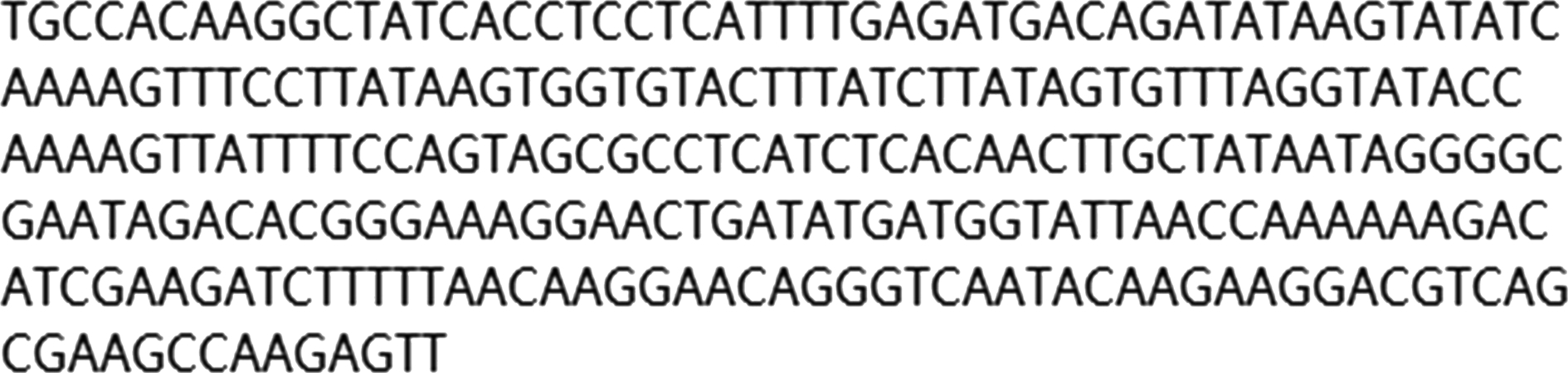
图1 L.jinshani扩增目标基因片段
Fig.1 L.jinshani amplification of target fragment gene
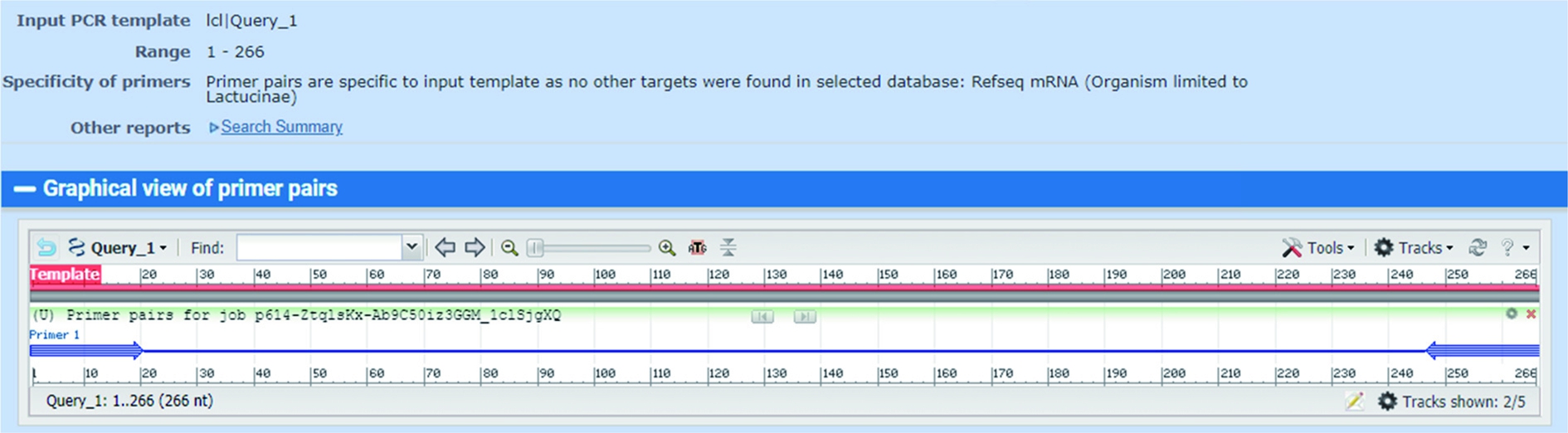
图2 特异性引物blast比对结果
Fig.2 Blast comparison of specific primers
2.2 PCR检测引物特异性
使用2.1中设计的L.jinshani特异性引物对15种不同的样品进行PCR扩增,使用琼脂糖凝胶电泳检验扩增引物,结果如图3所示,泳道1到泳道12在266 bp处未出现特征条带,结果显示为阴性,表明该引物对其他12 种微生物无交叉反应;泳道13、泳道14和泳道15在266 bp处出现特征条带,结果为阳性,均为已知含有L.jinshani目标片段的样品相符。结果表明使用该引物扩增仅能特异性扩增出含有L.jinshani基因片段的样品,说明该引物特异性良好,能够对样品中的L.jinshani进行特异性检测。

M-2 000 bp DNA marker;1-Lactobacillus buchneri;2-Lactobacillus pontis;
3-Lactobacillus casei;4-Lactobacillus farraginis;5-Lactobacillus plantarum;
6-Lactobacillus parafarraginis;7-Lactobacillus panis;8-Bacillus oleronius;
9-Bacillus circμLans;10-Acetobacter pasteurianus;11-Pediococcus pentosaceus;
12-Saccharomyces cerevisiae;13-含有L.jinshan的质粒;
14-L.jinshan.;15-含有L.jinshan.的酒醅;16-ddH2O
图3 PCR验证引物特异性
Fig.3 Validating primer specificity by PCR
2.3 q-PCR检测引物特异性
用2.2中的12种非L.jinshani的菌种DNA样品及L.jinshani的菌株DNA样品分别作为模板,利用特异性引物分别进行q-PCR。结果如图4所示,只有L.jinshani菌株组出现了扩增曲线,其他12种菌种均未出现扩增曲线,对其他12 种微生物无交叉反应,结果表明该引物在q-PCR方法上也具有良好的种间特异性,可用于区分L.jinshani与其他微生物。
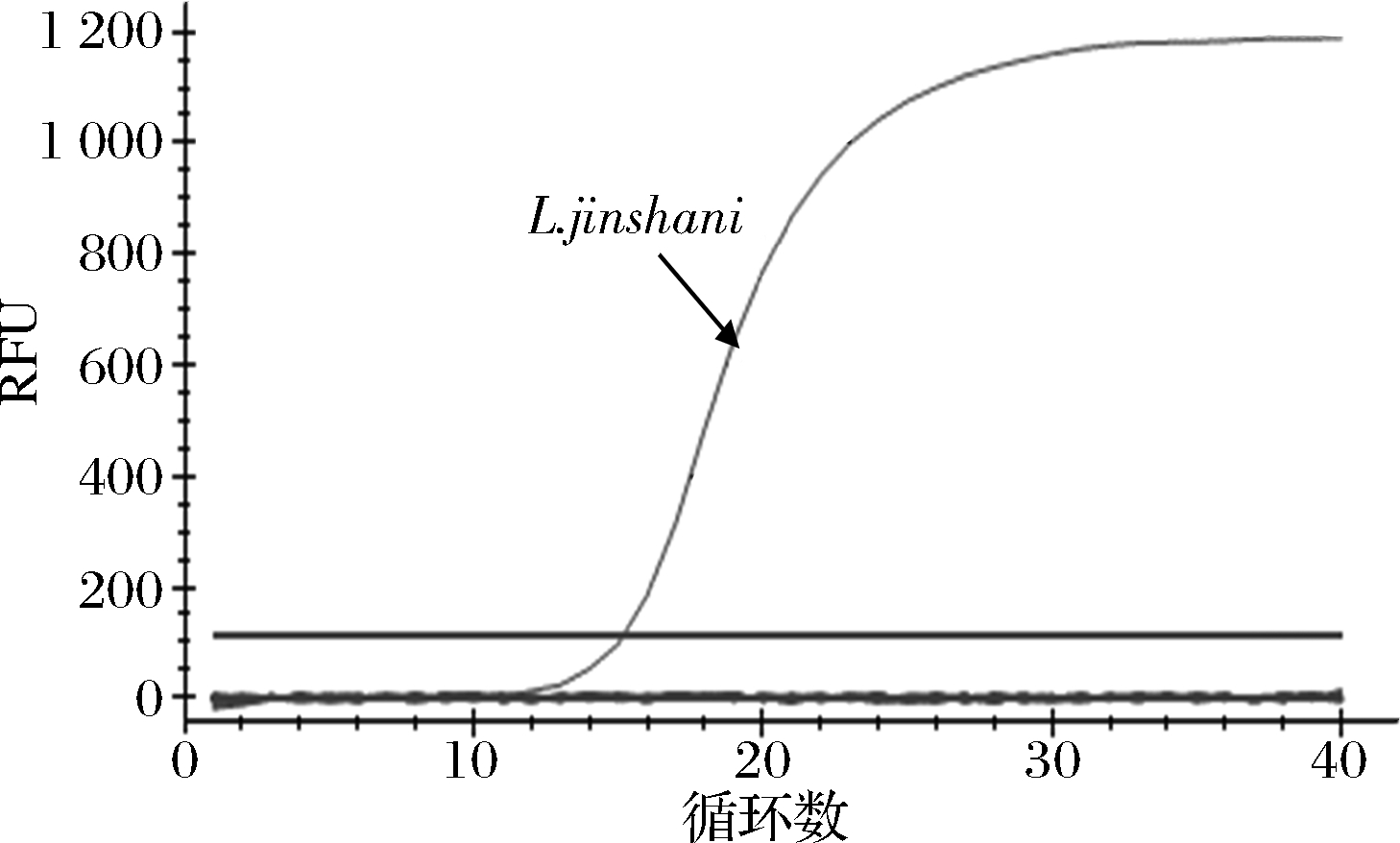
图4 L.jinshani与对照菌株的q-PCR的检测结果
Fig.4 q-PCR detection results of L.jinshani and control strains
2.4 标准曲线
回收的标准质粒质量浓度为135.26 ng/μL,将构建好的质粒标准品以10 倍稀释梯度进行稀释,共制备-1~-7共7个稀释梯度。各浓度梯度质粒样本的稀释曲线均为平稳光滑的S 型,如图5所示,最低检出量为3.08×103 copies/μL;如图6所示,以不同浓度标准品拷贝数的对数作为横坐标,以q-PCR反应过程中到达荧光阈值的初始循环数(Cq)为纵坐标,绘制标准曲线为y=3.354x+10.613,标准曲线的斜率为-3.354,线性R2=0.99,通过斜率计算扩增效率为98.7%,满足标准曲线的要求,可应用于实验和实际生产。
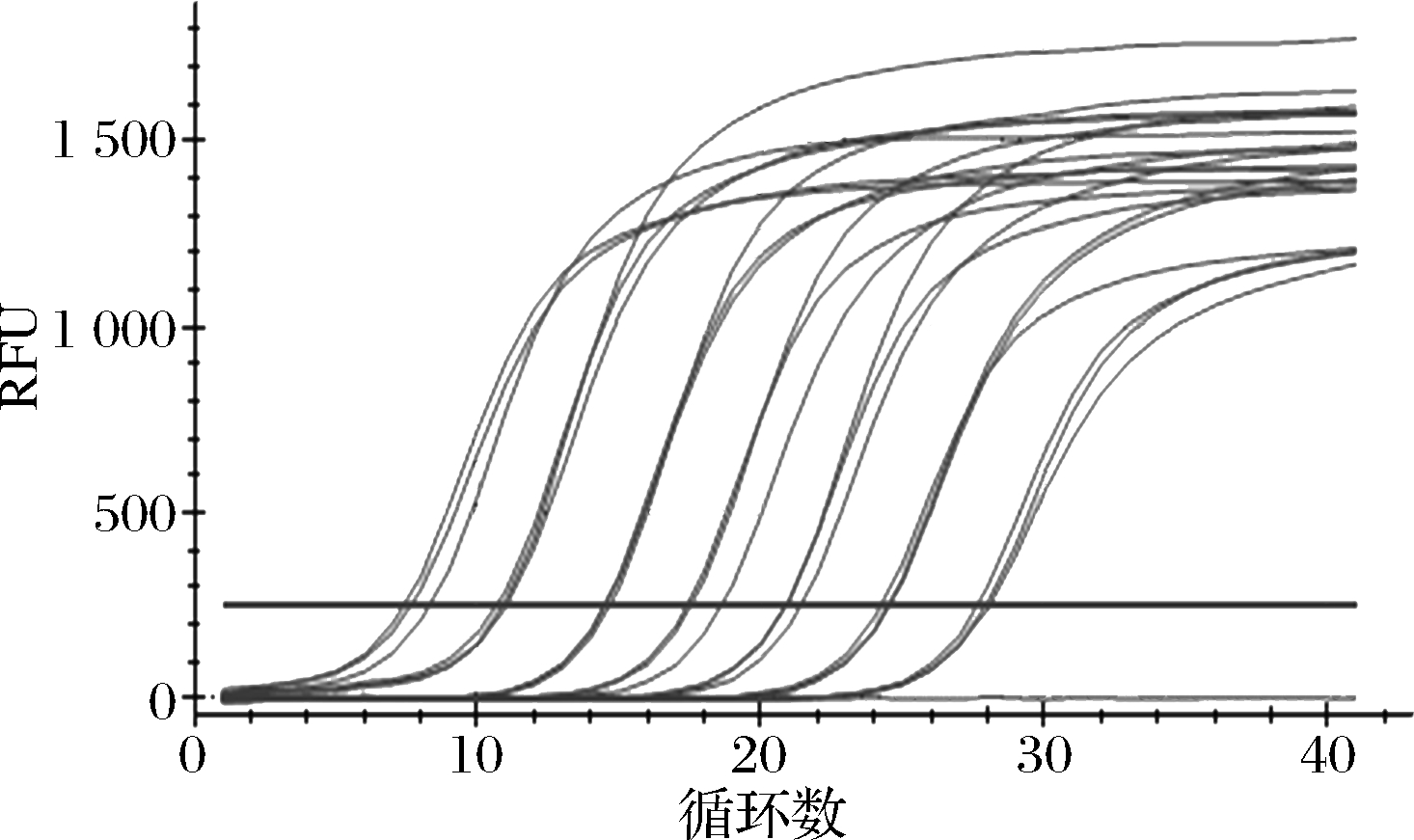
图5 重组质粒梯度的10倍梯度稀释扩增曲线
Fig.5 The amplification curve of serial 10-fold dilutions of recombinant plasmid
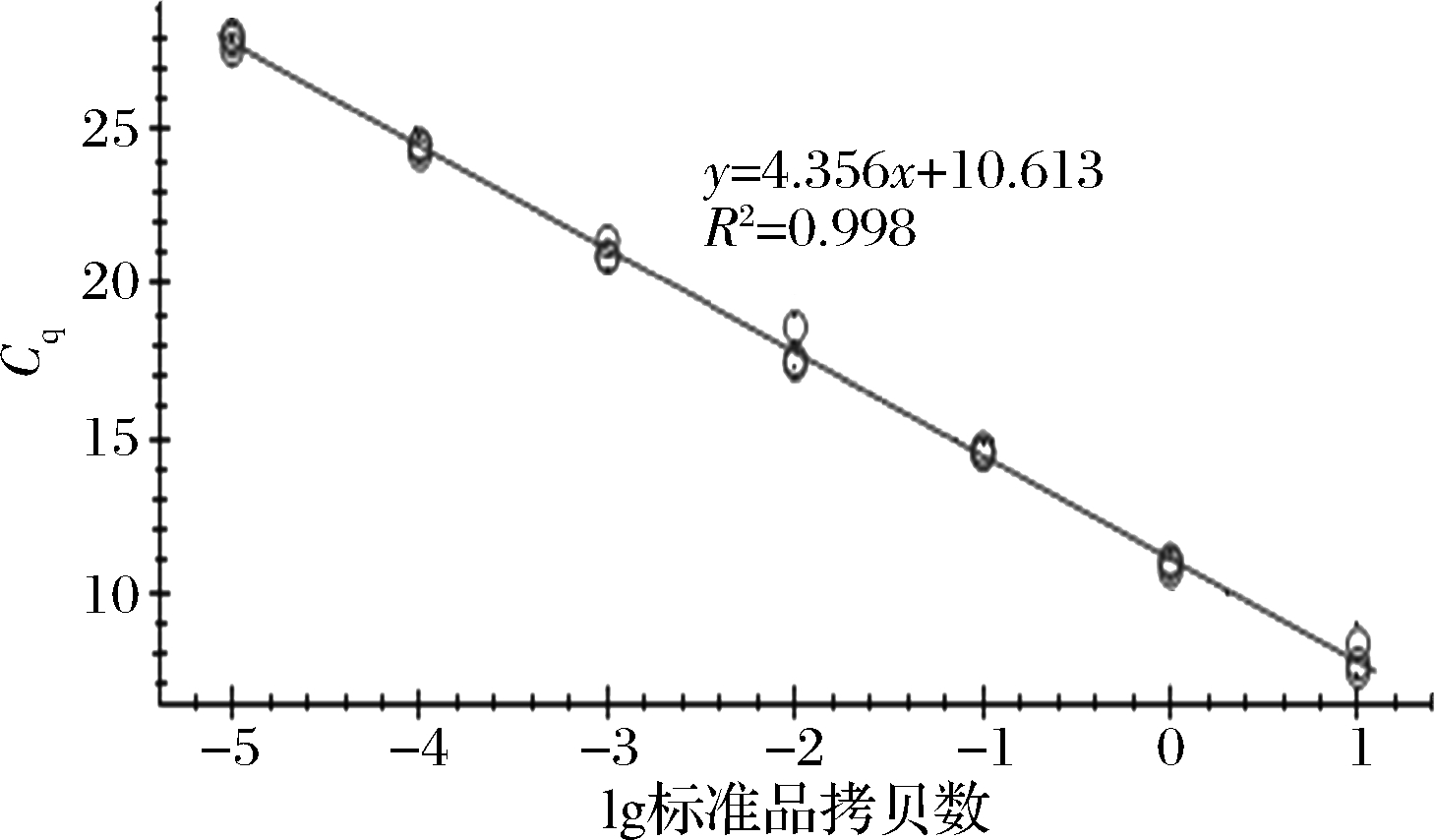
图6 L.jinshani 目的基因拷贝数与Cq值之间的
标准曲线
Fig.6 The standard curve between L.jinshani target fragment gene copies number and Cq value
2.5 重复性检验
以2.4中的质粒为扩增模板将构建好的质粒标准品以10倍稀释梯度进行稀释,共制备-3~-7共5个稀释梯度。研究特异性引物qPCR定量方法的可重复性。结果如表1所示,Cq值的变异系数(coefficient of variation,CV)在0.61%~3.06%,在每个稀释梯度样本的检测结果中均有良好的重复性,证明基于特异性引物构建的q-PCR定量方法具有重复性,适用于定量含有不同浓度L.jinshani的样本。
表1 q-PCR定量检测方法的重复性
Table 1 The reproducibility of q-PCR
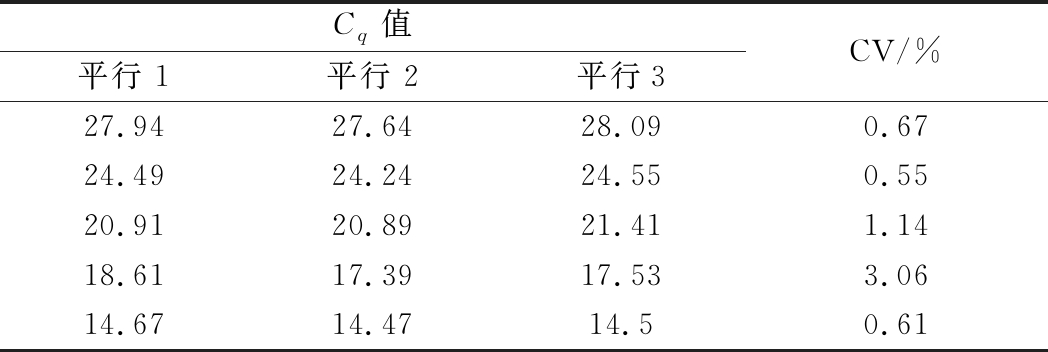
Cq值平行 1平行 2平行3CV/%27.9427.6428.090.6724.4924.2424.550.5520.9120.8921.411.1418.6117.3917.533.0614.6714.4714.50.61
2.6 酒曲及酒醅中L.jinshani含量的检测
如表2所示,通过L.jinshani特异性引物q-PCR的方法定量检测湖北酒厂中酒曲及不同轮次酒醅发现。酒曲中L.jinshani含量较低无法检测到其含量,第1轮次中L.jinshani含量较低无法检测到其含量,在第2轮次酒醅中L.jinshani含量达到(8.9±0.11)lgcopies/g,在第4轮次酒醅中L.jinshani含量达到(9.38±0.11) lgcopies/g,第5轮酒醅中L.jinshani含量达到(9.43±0.11)lgcopies/g。通过高通量检测分析发现,酒曲中未检测出L.jinshani,1轮酒醅中L.jinshani含量为9.05%,2轮酒醅中L.jinshani含量为81.51%,4轮酒醅中L.jinshani含量为99.71%,5轮酒醅中L.jinshani含量为99.47%。q-PCR检测与高通通量检测得到不同轮次酒培的L.jinshani含量相对应。从结果可以看出,L.jinshani在白酒发酵后期数量迅速增长,成为后期产优质原酒的酒醅中的优势菌种。
表2 不同样品中L.jinshani含量
Table 2 The content of L.jinshani in different samples
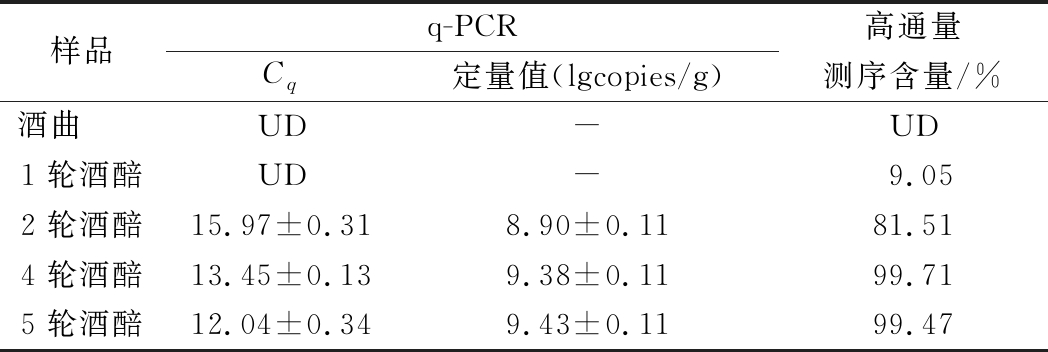
样品q-PCR高通量Cq定量值(lgcopies/g)测序含量/%酒曲 UD-UD1轮酒醅UD - 9.052轮酒醅15.97±0.31 8.90±0.11 81.514轮酒醅13.45±0.139.38±0.1199.715轮酒醅12.04±0.349.43±0.1199.47
注:UD 表示未检出;-表示无
3 结论与讨论
L.jinshani被发现广泛存在于全国各产区白酒酿造体系中[22]。根据本研究结果,酒曲中并未发现L.jinshani,其来源可能为酿酒过程中的其他途径。白云边第1轮酒醅中L.jinshani出现的比率很小,推测与酒醅酸度有关。已有报道L.jinshani在酒醅后期占优势[22],可能与该菌具有较好的耐酸性有关。发酵后期,随着白酒酿造的进行,乳酸菌成为后期的绝对优势微生物,L.jinshani数量得到大量增长,但关于L.jinshani的具体代谢功能与价值还未得到充分研究,其在白酒发酵过程中所起到的作用还有待发掘。目前对L.jinshani的定性定量检测方法的研究报道较少,因此本研究所建立的能够快速检测酒醅中L.jinshani的方法对研究其在白酒酿造过程中的具体功能与价值具有重要意义。
本研究通过设计L.jinshani的特异性引物F:5′-TGCCACAAGGCTATCACCTC-3′;R:5′-AACTCTTGGCTTCGCTGACG-3′建立了q-PCR的方法对L.jinshani进行定量检测。结果表明该方法灵敏度高,特异性好,能够方便快捷地定量检测对样品中L.jinshani。可有效应用于白酒酿造过程中的L.jinshani的定量与定性检测,为进一步揭示白酒功能菌株L.jinshani在酿酒过程中的功能提供了有效方法。
[1] 邱并生. 混菌发酵对白酒液态发酵效率和风味物质的影响[J].微生物学通报, 2014, 41(7):1 477-1 478.
QIU B S, THE EBOMC.Effect of mixed culture on fermentation efficiency and flavor compounds in Chinese liquor[J].Microbiology, 2014, 41(7):1 477-1 478.
[2] 栗连会. 泸型酒酒醅中乳酸菌和乳酸降解菌的多样性和代谢特性[D].无锡:江南大学, 2016
LI L H.Diversity and metabolic characteristics of lactic acid bacteria and lactate-degrading bacteria in fermented grains of Luzhou-flavor liquor[D].Wuxi:Jiangnan University, 2016.
[3] 吴莉莉, 王海燕, 徐岩, 等.酱香型与清香型白酒发酵过程中乳酸菌菌群的差异性分析[J].微生物学通报, 2013, 40(12):2 182-2 188.
WU L L, WANG H Y, XU Y, et al.Differences of lactic acid bacteria community between soy sauce aroma style and light aroma style liquor fermentation[J].Microbiology, 2013, 40(12):2 182-2 188.
[4] WU Z F, ZHUANG B W, WENG P F, et al.Fermentation quality characteristics and flavor formation changes during the process of pickled wax gourd in eastern Zhejiang[J].International Journal of Food Properties, 2016, 19(2):409-419.
[5] 庾昌文, 薛栋升, 周敏, 等.清香型小曲白酒机械化酿造过程中细菌多样性及酿造性能分析[J].湖北农业科学, 2016, 55(11):2 860-2 863;2 881.
YU C W, XUE D S, ZHOU M, et al.Analysis of diversity and brewing characteristics of bacterium in mechanization process of fen-flavor xiaoqu liquor[J].Hubei Agricultural Sciences, 2016, 55(11):2 860-2 863;2 881.
[6] LE LAY C, COTON E, LE BLAY G, et al.Identification and quantification of antifungal compounds produced by lactic acid bacteria and propionibacteria[J].International journal of food microbiology, 2016, 239:79-85.
[7] GEREZ C L, TORRES M J, DE VALDEZ G F, et al.Control of spoilage fungi by lactic acid bacteria[J].Biological Control, 2013, 64(3):231-237.
[8] DALIÉ D K D, DESCHAMPS A M, RICHARD-FORGET F.Lactic acid bacteria-potential for control of mould growth and mycotoxins:A review[J].Food Control, 2010, 21(4):370-380.
[9] SUDUN, WULIJIDELIGEN, ARAKAWA K, et al.Interaction between lactic acid bacteria and yeasts in airag, an alcoholic fermented milk[J].Animal Science Journal, 2013, 84(1):66-74.
[10] 刘艳容, 舒永红, 杨佳玮, 等.食品中乳酸菌定量检测方法研究进展[J].食品工业科技, 2020, 41(12):358-365.
LIU Y R, SHU Y H, YANG J W, et al.Research progress on enumeration methods of lactic acid bacteria in food[J].Science and Technology of Food Industry, 2020, 41(12):358-365.
[11] 刘津, 刘二龙, 谢力, 等.数字聚合酶链式反应技术在食品安全检测领域的研究应用进展[J].食品科学, 2016, 37(17):275-280.
LIU J, LIU E L, XIE L, et al.Progress in research and application of digital polymerase chain reaction(dPCR) in food safety detection[J].Food Science, 2016, 37(17):275-280.
[12] 张娜娜, 刘洋, 俞漪, 等.乳酸菌饮料中嗜酸乳杆菌的实时荧光定量PCR检测方法[J].食品科学, 2019, 40(8):27-32.
ZHANG N N, LIU Y, YU Y, et al.Real-time fluorescent quantitative PCR method for detection of Lactobacillus acidophilus in probiotic beverages [J].Food Science,2019,40(8):27-32.
[13] 刘艳, 王鸣秋, 李诗瑶, 等.基于实时荧光定量PCR和数字PCR的肉制品中牛源性成分检测[J].现代食品科技,2019, 35(7):254-260.
LIU Y, WANG M Q, LI S Y, et al.Detection of bovine components in meat products based on real-time fluorescence quantitative PCR and digital PCR[J].Modern Food Science and Technology, 2019, 35(7):254-260.
[14] GUO Z H, FANG H, XIA Z S, et al.Detection of lactobacillus acidophilus in fermented material by real-time fluorescent quantitative PCR[J].Animal Husbandry and Feed Science, 2016, 8(1):54-57.
[15] 包秋华, 李梅花, 徐洁, 等.1种检测发酵乳中嗜酸乳杆菌的定量定性方法[J].食品研究与开发, 2012, 33(11):153-155.
BAO Q H, LI M H, XU J, et al.Study on the methods of qualitative and quantitative detection L.acidophilus in the fermented milk[J].Food Research and Development, 2012, 33(11):153-155.
[16] 牛犇, 洪斌, 穆丽丽, 等.荧光定量PCR在预测微生物学中的应用[J].食品工业科技, 2019, 40(5):275-282.
NIU B, HONG B, MU L L, et al.Application of fluorescent quantitative PCR in predictive microbiology[J].Science and Technology of Food Industry, 2019, 40(5):275-282.
[17] 程平言, 路虎, 胡峰, 等.实时荧光定量PCR技术在白酒酿造过程中微生物数量检测的应用展望[J].酿酒科技, 2020(5):74-77.
CHENG P Y, LU H, HU F, et al.Application of real-time fluorescent quantitative PCR for monitoring microbes in the production of baijiu[J].Liquor-Making Science & Technology, 2020(5):74-77.
[18] 杨弘华, 宋燕燕, 林晓波, 等.实时荧光定量PCR技术应用研究[J].山东化工, 2016, 45(22):46-47.
YANG H H, SONG Y Y, LIN X B, et al.Summary of research and development of real time fluorescent PCR technology[J].Shandong Chemical Industry, 2016, 45(22):46-47.
[19] YU Y J, LI X, ZHANG J H, et al.Lactobacillus jinshani sp.nov., isolated from solid-state vinegar culture of Zhenjiang aromatic vinegar[J].Antonie Van Leeuwenhoek:Journal of Microbiology, 2020, 113(1):43-54.
[20] DU R B, WU Q, XU Y.Chinese liquor fermentation:Identification of key flavor-producing Lactobacillus spp.by quantitative profiling with indigenous internal standards[J].Applied and Environmental Microbiology, 2020, 86(12).DOI:10.1128/AEM.00 456-20.
[21] 邢敏钰, 杜海, 徐岩.芝麻香型白酒发酵过程中乳酸菌多样性及其演替规律[J].微生物学通报, 2018, 45(1):19-28.
XING M Y, DU H, XU Y.Diversity and succession of lactic acid bacteria during sesame-flavor liquor fermentation[J].Microbiology, 2018, 45(1):19-28.
[22] 杜如冰, 吴群, 徐岩.基于三步荧光定量PCR技术揭示不同产区白酒酿造系统中Lactobacillus sp.的分布特征[J].微生物学通报, 2020, 47(1):1-12.
DU R B, WU Q, XU Y.Distribution of Lactobacillus sp.in Chinese liquor fermentation system from different producing location by three-step fluorescent quantitative PCR[J].Microbiology China, 2020, 47(1):1-12.