酸奶作为乳制品中重要的成员,以其独特的风味、极高的营养价值倍受人们青睐。酸奶不但口感美味,而且具有促进消化吸收、维护肠道健康的功能。随着酸奶生产规模的不断扩大、加工生产产业链的不正规化、低质量奶源等原因,导致酸奶中存在一些有害细菌,使消费者食用后产生不良反应,如腹痛、腹泻甚至中毒。GB 19302—2010《食品安全国家标准 发酵乳》中对大肠菌群、金黄色葡萄球菌、沙门氏菌、酵母和霉菌进行了限量规定。基于现有的研究,我们发现存在于酸奶中并可能带来一定危害的细菌主要有黄色葡萄球菌、布鲁氏杆菌、大肠杆菌、沙门氏菌、志贺氏菌、蜡样芽孢杆菌、单增李斯特氏菌等[1]。部分细菌在国标中并没有明确的规定,对酸奶食用安全性造成的影响不得而知,因此酸奶的安全问题值得引起乳制品相关行业的高度重视。
常见的酸奶分为自然发酵和商品化酸奶两种。自然发酵的酸奶与市售的商品化酸奶在加工方面存在着一定的差异。研究发现南疆地区传统发酵酸奶中厚壁菌门和变形菌门为优势菌群,其中乳杆菌属、链球菌属、醋酸杆菌属、乳球菌属、肠球菌属、芽孢杆菌属的含量较多[2]。自然发酵的酸奶通常是牧民(如青藏高原地区)采用传统而古老的发酵方法制作的,能够产生大量优良的乳酸菌,具有降低胆固醇、抗氧化等保健功能,但自然发酵的酸奶中存在较多发酵菌种外的细菌。市售酸奶为商品化酸奶,通常采用工厂化加工进行大批量生产,上市前对酸奶进行质量检测。一般而言,商业化酸奶的发酵时间相对较短,乳酸菌活菌数较多,后酸化作用较弱,但稳定性较高。
目前有很多检测乳制品中细菌的方法,例如平板培养法、核酸探针法、PCR技术[3]等,这些技术具有简单、易于操作等特点,但当不同菌落混杂在一起时,很难同时进行定性定量的检测,且存在一些缺点如周期长、易受人为因素干扰等[4]。MiSeq系统是一种高端测序系统,可以同时进行合成与测序操作。随着Illumina Miseq高通量测序技术的不断完善、发展,现已成功应用在多个方面,如朱潇等[5]利用该技术分析牦牛发酵乳制品中细菌菌群多样性;卢圣鄂等[6]将该技术用于分析土壤真菌群落结构及多样性。本研究采用宏基因组测序法,对酸奶样品进行细菌采集测序,检测出酸奶中可能存在的细菌,并进行分类分析,从原料乳、加工生产及包装运输3个方面分析可能来源。
1 材料与方法
1.1 材料
酸奶样品于2021年3月2日购于上海泥城大润发超市冷藏酸奶专区(0~4 ℃)。如表1所示,一共为16份样品,包括国内8种知名品牌的酸奶A、B、C、D、E、F、G、H,以及A品牌不同系列的酸奶样品8份(A1~A8),其中A与A5为平行对照样品。全程保证冷链保藏,运送至实验室于0~4 ℃保存备用。
表1 酸奶样品
Table 1 Yogurt samples

编号样品名称发酵菌种保质期/dA风味发酵乳嗜热链球菌,嗜酸乳杆菌,保加利亚乳杆菌,乳双歧杆菌,干酪乳杆菌21B风味发酵乳嗜热链球菌,乳双歧杆菌,乳酸乳球菌乳酸亚种,乳酸乳球菌乳脂亚种,嗜酸乳杆菌,保加利亚乳杆菌21C风味发酵乳嗜热链球菌,乳双歧杆菌,长双歧杆菌,嗜酸乳杆菌,乳酸乳球菌乳脂亚种,乳酸乳球菌乳酸亚种,保加利亚乳杆菌21D风味发酵乳嗜热链球菌,乳双歧杆菌,嗜酸乳杆菌,保加利亚乳杆菌21E风味发酵乳嗜热链球菌,乳双歧杆菌,乳酸乳球菌乳酸亚种,保加利亚乳杆菌21F风味发酵乳嗜热链球菌,保加利亚乳杆菌21G风味发酵乳嗜热链球菌,乳酸乳球菌乳脂亚种,乳酸乳球菌乳酸亚种,保加利亚乳杆菌,乳双歧杆菌,嗜酸乳杆菌,副干酪乳杆菌21H原味裸酸奶嗜热链球菌,保加利亚乳杆菌21A1咕哝咕哝浓缩风味酸牛奶嗜热链球菌,保加利亚乳杆菌21A2黄桃果粒风味发酵乳(复原乳)嗜热链球菌,嗜酸乳杆菌,保加利亚乳杆菌,乳双歧杆菌,干酪乳杆菌21A3大颗果粒风味发酵乳(黄桃)嗜热链球菌,保加利亚乳杆菌21A4健能风味发酵乳乳双歧杆菌,干酪乳杆菌,保加利亚乳杆菌,嗜热链球菌21A5风味发酵乳嗜热链球菌,嗜酸乳杆菌,乳双歧杆菌,保加利亚乳杆菌,干酪乳杆菌21A6赏味风味发酵乳嗜热链球菌,保加利亚乳杆菌,婴儿双歧杆菌21A7如实发酵乳嗜热链球菌,保加利亚乳杆菌,乳酸乳酸菌双乙酰亚种21A8畅优风味发酵乳乳双歧杆菌,嗜酸乳杆菌,保加利亚乳杆菌,嗜热链球菌,植物乳杆菌21
1.2 仪器与设备
FastPre-24 MP型组织均质器,美国MP Biomedicals公司;#DP328型粪便基因组DNA试剂盒,天根生化有限科技公司;SLFPTAD多功能酶标仪,美国伯腾Biotek仪器有限公司;PowerPac HC电泳仪,上海伯乐生命医学产品有限公司;5331PCR仪,美国Biometra公司;22331Eppendorf AG离心机,德国Hamburg公司;DW-40L508医用低温保存箱、HYC-290医用冷藏箱,青岛Haier特种电器有限公司;Illumina MiSeq PE300测序仪,上海美吉生物医药科技有限公司。
1.3 实验方法
1.3.1 样品前处理
用FastPre-24 MP组织均质器将样品进行均质混匀,用移液枪吸取200 μL样品于EP离心管中备用。
1.3.2 样品基因组DNA提取
样品基因组DNA提取采用粪便基因组DNA试剂盒,具体步骤参考试剂盒说明书。
1.3.3 基因组DNA浓度及纯度的检测
通过酶标仪对样品的DNA浓度以及纯度进行测定分析。基因组DNA的完整性通过1%琼脂糖凝胶电泳进行检验。
1.3.4 细菌的16S rRNA V3~V4区PCR扩增
以总DNA为模板利用细菌16S rRNA引物338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)[7]实现V3~V4可变区的PCR扩增。扩增程序为:94 ℃预变性 3 min,35个循环(94 ℃变性1 min,55 ℃退火45 s,72 ℃延伸1 min)。扩增体系为50 μL,25 μL Premix Tap,2 μL dNTPs,4 μL引物(5 mol/L),1 μL DNA模板,18 μL灭菌蒸馏水。
1.3.5 Illumina Miseq 测序
将PCR扩增子产物在MiSeqPE300平台上进行双端测序。美吉平台MiSeq测序得到的是双端序列数据,首先用QIIME(v1.9.1)软件进行数据处理获得合格的reads,根据PE reads之间的overlap关系,将成对的reads拼接成1条序列,根据序列首尾两端的barcode和引物序列区分样品得到有效序列,并校正序列方向,即为优化数据。
1.4 数据分析
样品数据采用稀释曲线分析、可操作分类单元(operational taxonomic units,OTU)分析以及β多样性分析,运用Excel绘制OTU水平上样本菌群分布群落Bar图,运用生物信息学软件 Geneious Prime(v2021.1.1)进行细菌序列的比对分析。
2 结果与分析
2.1 样品16S rRNA V3~V4区扩增电泳结果
酸奶样品总DNA经酶标仪检测后,浓度均在10~15 ng/μL,且OD260/OD280均在1.8~2.0,表明DNA纯度较高。图1为样品16S rRNA V3~V4区PCR扩增产物电泳分析结果,选用100 bp ladder为Marker。16个样品在目标产物区域均出现明显的条带,而阴性对照无条带,说明酸奶样品中存在细菌。此外16个样品的电泳条带均匀单一,表明PCR扩增成功,可以进行后续的测序。
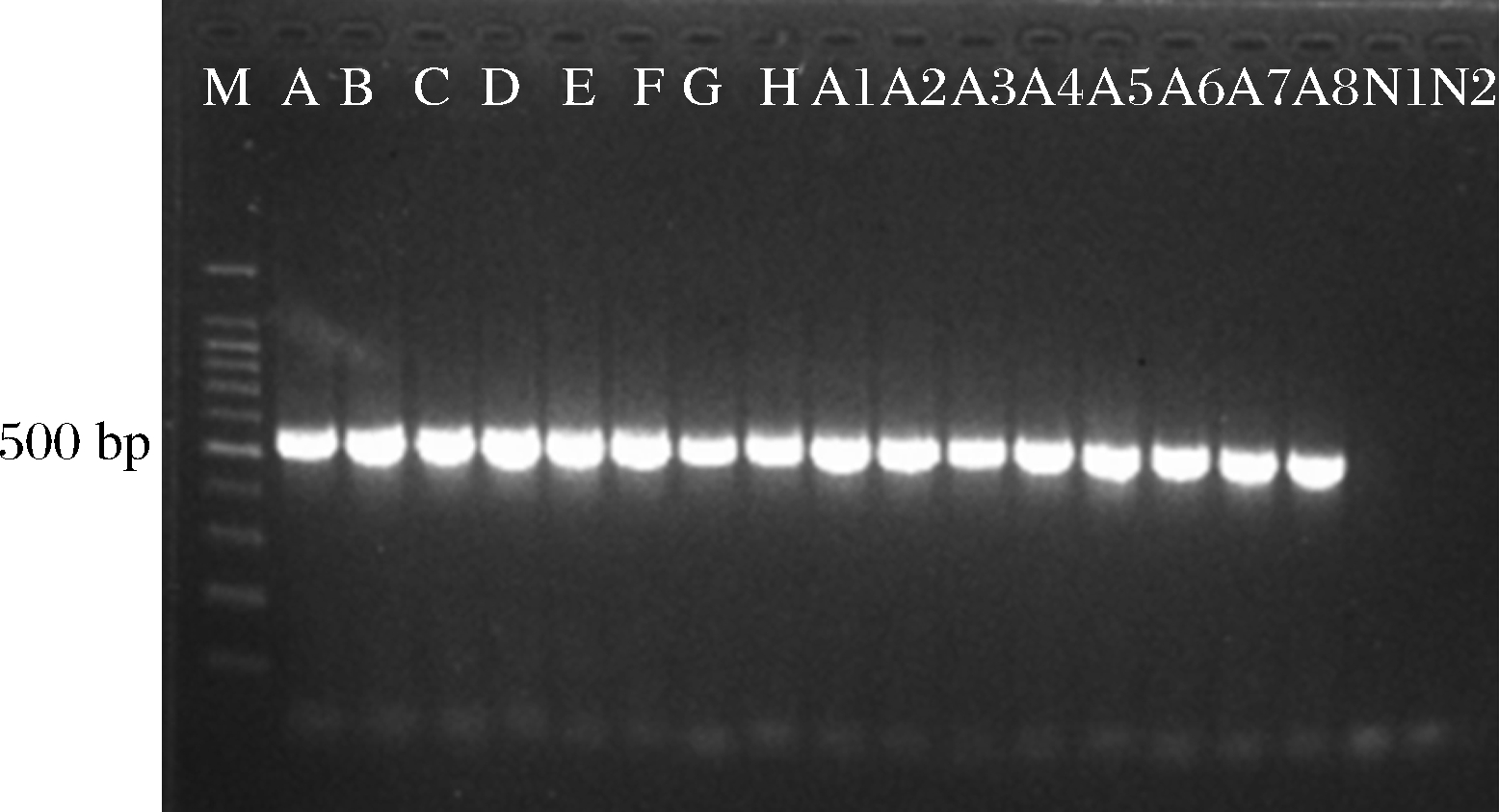
M-marker
图1 16S rRNA V3~V4区PCR产物琼脂糖电泳
Fig.1 The agarose gel electrophoresis of PCR products from V3~V4 region of 16S rRNA
注:N1、N2分别为提取DNA时采用的阴性对照和PCR扩增时采用的阴性对照
2.2 稀释曲线
图2为样品OTU稀释曲线,反映群落丰富度的指数有Sobs、Chao、Ace、Jack、Bootstrap等,其中Sobs描述丰富度实际观测值,更能直观体现本研究样品的丰富度,因此采用的多样性指数为Sobs。由图2可知,随着测序深度增加,曲线趋于平坦,更多的测序量并不会产生更多的物种。表明测序深度合理,可以进行后续分析。
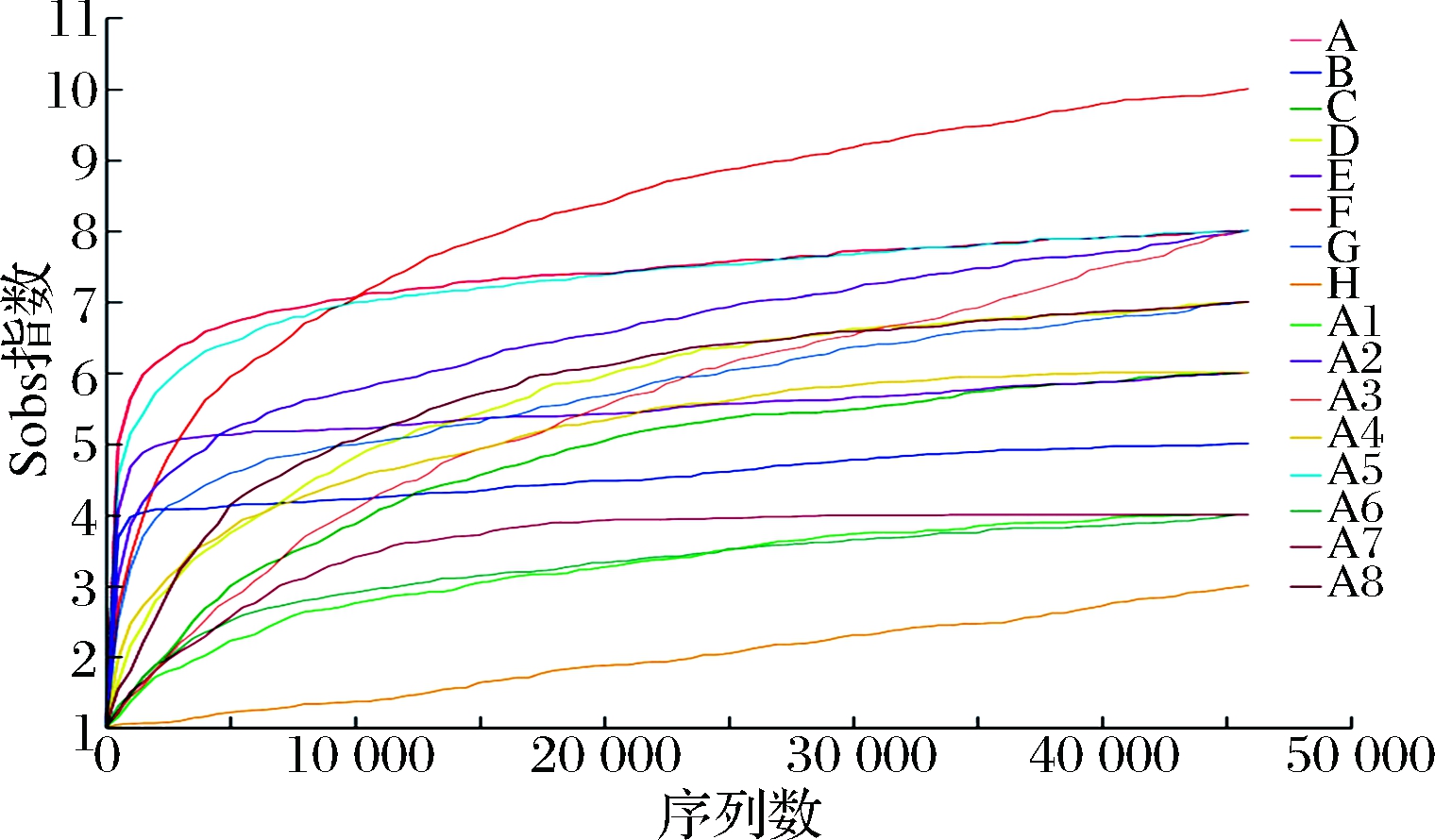
图2 稀释曲线
Fig.2 Rarefraction curve
2.3 OTU分类
按照序列间相似度97%为阈值对非重复序列进行OTU聚类,去除嵌合体,测序结果显示,酸奶样品中的细菌一共包含16个OTU序列。根据分类学分析各分类水平(域、界、门、纲、目、科、属、种、OTU)上的物种组成情况,如表2所示。OTU水平上样本菌群分布群落条形图如图3所示。
2.3.1 发酵菌种
本研究采集的样品共鉴定出9种发酵菌种,包括OTU1(Streptococcus thermophiles)、OTU2(Bifidobacterium animalis subsp.Lactis)、OTU3(uncultured Streptococcus)、OTU4(Lactococcus lactis)、OTU5(Lactobacillus acidophilus)、OTU6(Lactobacillus delbrueckii subsp.Bulgaricus)、OTU9(Bifidobacterium longum subsp.Infantis)、OTU13(Lactococcus piscium)和OTU14(Lactiplantibacillus plantarum)。
根据调查显示,市面上采用的发酵菌种大多为乳酸菌,乳酸菌包括嗜热链球菌属、双歧杆菌属,乳酸球杆菌属、乳酸链球菌属、保加利亚乳杆菌属、嗜酸杆菌等,不同型号的酸奶根据需求对发酵菌种进行适当的调整。在所有样品中均检测到了嗜热链球菌,且在各样品中最为丰富。
2.3.2 污染菌
此外,鉴定出4种污染细菌,即OTU7(Anoxybacillus flavithermus)、OTU10(Acinetobacter baumannii)、OTU11(Enterobacter sp.)和OTU15(Arthrobacter sp.)。
其中OTU10和OTU11可能为条件致病菌,由图3-b可知,OTU10主要存在于D和F样品中,而OTU11存在于F和A3样品中。鲍氏不动杆菌(Acinetobacter baumannii)是一种革兰氏阴性细菌,可引起免疫缺陷宿主的机会性感染,感染后具有很高的死亡率。大肠杆菌为肠杆菌属中主要的细菌,大肠杆菌的分布广泛,在各种动物和食物中较容易被检出,大多不会引发疾病,但一些特殊血清的大肠杆菌具有一定的致病性。
OTU7和OTU15可能造成酸奶的腐败变质。OTU15少量存在于A1与A3中,OTU7在样品A、D和A3中丰度较高。节杆菌属是最常见的分离细菌,常见于土壤和被工业化学品和放射性材料污染的环境中[8]。OTU7是常见的腐败微生物之一,研究表明产芽孢嗜热细菌属的地杆菌和无氧杆菌是乳品工业中常见的污染物[9]。此外,OTU12的序列没有检索到可信度更高的物种信息,可能为不可培养的污染菌,在A8中丰度较高,在G、A2和A4中少量存在。
除了这些细菌之外,OTU8与OTU16不是细菌序列,而是属于植物界中的李属,从图3-b可知,这两种均出现在A2样品中,该样品为黄桃果粒风味发酵乳,推断它们是由酸奶中的黄桃果酱等辅料带入的。
2.4 主成分分析
图4为主成分分析(principal component analysis,PCA),通过比较不同样本中细菌群落的组成来反映酸奶样本间的差异,样本物种组成越相似,其在PCA图中的距离越近。A与A5为平行酸奶样品,从图4可知,两者的测序结果基本一致;而不同品牌的酸奶样品之间差异较大,尤其是B样品。B样品中含有大量的OTU2,故与其他样品产生了较大的差异。
表2 OTU序列物种分类
Table 2 The classification of species represented by OTU

类别界门纲目科属Blast hitBlast identity/%中文名称来源参考文献OTU1BacteriaFirmicutesBacilliLactobacillalesStreptococcaceaStreptococcusStreptococcus thermophiles100嗜热链球菌乳制品、发酵食品[10]OTU2BacteriaActinobacteria/BifidobacterialesBifidobacteriaceaeBifidobacteriumBifidobacterium animalis subsp.Lactis100双歧杆菌乳制品[11]OTU3BacteriaFirmicutesBacilliLactobacillalesStreptococcaceaStreptococcusuncultured Streptococcus100嗜热链球菌发酵乳制品[12]OTU4BacteriaFirmicutesBacilliLactobacillalesStreptococcaceaeLactococcusLactococcus lactis100乳球乳酸菌乳制品、发酵食品[13]OTU5BacteriaFirmicutesBacilliLactobacillalesLactobacillaceaeLactobacillusLactobacillus acidophilus100嗜酸乳杆菌乳制品[14]OTU6BacteriaFirmicutesBacilliLactobacillalesLactobacillaceaeLactobacillusLactobacillus delbrueckii subsp.Bulgari-cus100德氏乳杆菌保加利亚亚种乳制品[15]OTU7BacteriaFirmicutesBacilliBacillalesBacillaceaeAnoxybacillusAnoxybacillus flavithermus100/温泉[16]OTU8ViridiplantaeSpermatophytaMagnoliopsidaRosalesRosaceae AmygdaloideaePrunus/100李属黄桃果酱/OTU9BacteriaActinobacteria/BifidobacterialesBifidobacteriaceaeBifidobacteriumBifidobacterium longum subsp.Infantis100长双歧杆菌乳制品[17]OTU10BacteriaProteobacteriaGammaproteobacteriaPseudomonadalesMoraxellaceaeAcinetobacterAcinetobacter baumannii100鲍氏不动杆菌土壤、水体[18]OTU11BacteriaProteobacteriaGammaproteobacteriaEnterobacteralesEnterobacteriaceaeEscherichiaEnterobacter sp.100肠杆菌属人和动物的肠道、粪便[19]OTU12Bacteria/////uncultured bacterium96.94不可培养的污染菌环境/OTU13BacteriaFirmicutesBacilliLactobacillalesStreptococcaceaeLactococcusLactococcus piscium100Piscium乳球菌乳制品[20]OTU14BacteriaFirmicutesBacilliLactobacillalesLactobacillaceaeLactiplantibacil-lusLactiplantibacillus plantarum100植物乳杆菌乳制品[21]OTU15BacteriaActinobacteria/MicrococcalesMicrococcaceaeArthrobacterArthrobacter sp.100节杆菌属水体、土壤[22]OTU16ViridiplantaeSpermatophytaMagnoliopsidaRosalesRosaceae AmygdaloideaePrunusPrunus dulcis100扁桃/甜李子黄桃果酱/
注:“/”表示未查到

图3 基于属水平15个OTU的菌种组成分析
Fig.3 Bacterial composition analysis based on 15 OTU at the genus level
注:由于OTU1相较于其他16种OTU来说数量过大,无法显著
呈现出其他物种的丰富度。因此图3将OTU1去掉,为充分呈现出样品
之间的物种丰富度差异
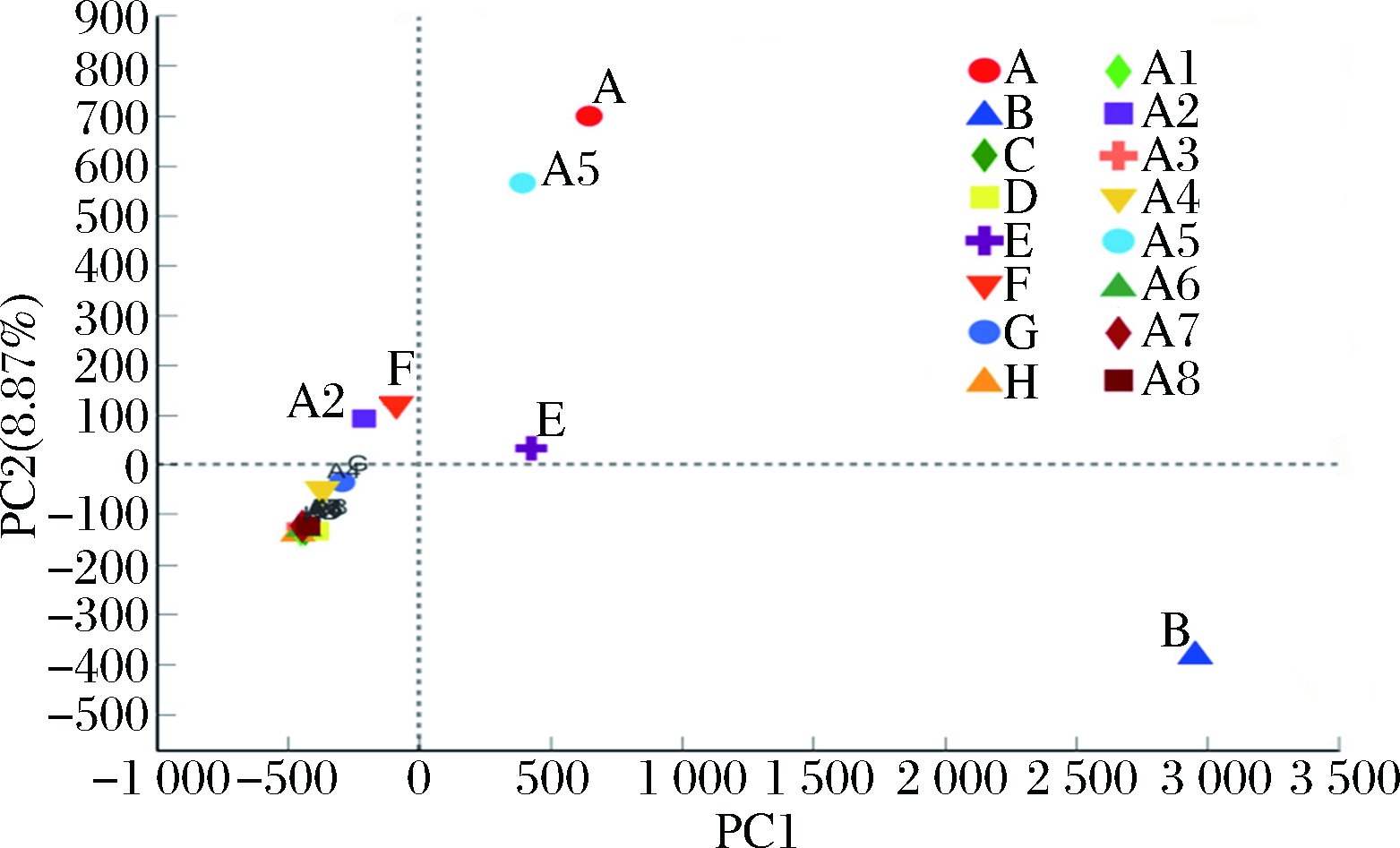
图4 不同酸奶样品细菌菌群的主成分分析
Fig.4 Principal component analysis of bacterial community of different yogurt samples
2.4.1 不同品牌酸奶
样品A、B、C、D、E、F、G和H分别来自8个不同品牌,这8种样品均为以生牛乳为原料,不添加其他成分的原味风味发酵乳。结果表明,B、C、E、G和H品牌酸奶中没有背景细菌(即酸奶中除了人为添加的发酵菌种以外的细菌统称为背景细菌),可初步判断为安全性较高的酸奶。而在A、D和F样品中,A存在较多的OTU7;D相较于A样品,除了含有OTU7之外,还存在较多OTU10;F主要存在OTU10。
由结果可知,这8个品牌的酸奶品质并不相同,存在品牌之间的差异。所产生的差异可能是由于发酵剂、生产工艺等因素不同所致。
2.4.2 同种品牌不同型号酸奶
此外,对A品牌旗下的不同系列的酸奶样品也进行了研究。其中A2样品为复原乳酸奶,且A2与A3样品中均添加了黄桃果酱,其余均为原味发酵乳。结果表明,A6与A7中背景细菌数量相对较少,A2、A4次之;A2中出现OTU8与OTU16两种非细菌序列的污染物,可能是取样时黄桃果酱偶然带入所致;A3样品中主要含有OTU11。A1、A5与A8均含有较多的OTU7。
结果表明同一品牌下的酸奶之间亦存在差异性。导致差异性的原因主要有3点:原料乳不同,分为生牛乳和复原乳两种;发酵菌种不同;添加的辅料不同,添加果粒会对酸奶的菌群造成影响。
3 讨论
3.1 商品化酸奶中发酵菌种的筛查
经比对分析,部分样品中的发酵菌与表1中标签所示完全一致,而部分样品不完全一致。对此,分析有3点原因:其一,该商品化酸奶中实际添加的发酵菌种与标签不符。根据GB 7718—2011《食品安全国家标准 预包装食品标签通则》,标签内容需确保真实,因此标签所示发酵菌种均按要求添加;其二,样品中均按标签添加了发酵菌种,但由于某些乳酸菌属菌株之间的16S rRNA V3~V4区序列相似度较高,因此被归类于同一OTU而没有被区分开。经生物信息学软件Geneious Prime进行序列比对后,发现乳杆菌属的各个菌种之间16S rRNA V3~V4区相似度较高,且该区域片段长度有限(仅400~500 bp左右),无法把某些菌种完全区分,因此某些乳酸菌种可能被归类于乳杆菌属;其三,某些发酵菌种添加量比较少,在后续加工和贮运环节中菌株失活甚至DNA降解,故未完全被检测出。
3.2 酸奶中背景细菌的溯源及防控
本研究是检测酸奶样品中细菌的DNA,因此不能区分菌株是否为存活的状态,只能说明背景细菌曾经在酸奶中存在过。背景细菌的存在,可能导致一些潜在的风险,因此从以下3个方面对其进行溯源分析。
(1)原料乳的质量:影响原料乳质量的因素包括奶牛乳房炎、奶牛饮食、养殖环境、挤奶过程及原奶的运输过程。我国导致奶牛乳房炎最主要细菌是金黄色葡萄球菌和链球菌,发病率高达80%[23]。原料乳中曾检测到不动杆菌属,表明OTU10可能来自于患有乳房炎乳奶牛的原料乳中。此外,FERNANDO等[24]从奶牛粪便存储池和地表水中分离回收到鲍氏不动杆菌,曹慧慧等[25]在奶牛养殖场采集原料乳的环节检测到不动杆菌属、节细菌属、棒状杆菌属等。这些环节均可导致OTU10、OTU11与OTU15混入。
(2)酸奶生产加工过程:酸奶的加工过程包括配料、预热、均质、杀菌、接种、发酵、搅拌、后熟以及包装和运输,其中复原乳酸奶还包括奶粉生产加工过程。污染细菌可能来源于生产器械、加工人员、果酱辅料等。COLERI CIHAN等[26]相关研究表明,OTU7有形成生物膜的潜力使其残存于加工器械上。
(3)包装运输:包装材料的质量、包装环境也会对酸奶质量产生影响,对此可采用紫外灭菌等预处理保证其无菌状态。酸奶大多采用冷链运输,若在运输过程中温度控制不当,可能会造成酸奶腐败变质。
3.3 与国家标准对比
在本研究中发现的7种背景细菌只有肠杆菌属在国标中有明确的规定,虽然本实验只对酸奶样品中的细菌进行定性分析,但在F和A3样品中检出肠杆菌,说明商品化的酸奶样品仍然存在潜在的安全风险。同时,酸奶国家标准中关于细菌部分是基于培养方法进行检测的,大量不可培养的细菌无法有效检出,因此测序结果中发现的部分难以培养细菌也可能存在潜在的隐患。随着现代高通量测序技术的不断发展,酸奶中微生物尤其是细菌的检测和筛查有待进一步的提高和完善。
4 结论
综上所述,本文通过宏基因组测序法分析8个品牌16种低温酸奶样品中的细菌多样性,发现不同品牌和同一品牌的不同种类酸奶之间均存在显著性差异。除了发酵剂中的乳酸菌之外,在商品化酸奶样品中还发现了部分环境污染细菌,表明商品化酸奶在原料乳收集、加工生产和包装运输等环节仍然存在受环境细菌污染的潜在风险。本研究成果为商品化酸奶细菌群落研究和提高酸奶安全性提供一定的参考价值。
[1] 姚宇秀, 陈媛, 郭佩佩, 等.Illumina MiSeq高通量测序技术研究原料乳及环境中细菌多样性和致病菌的分布情况[J].中国食品学报, 2019, 19(1):249-254.
YAO Y X, CHEN Y, GUO P P, et al.Bacterial diversity and pathogenic bacteria distribution of raw milk and its environment by the Illumina MiSeq high-throughput sequencing technologies[J].Journal of Chinese Institute of Food Science and Technology, 2019, 19(1):249-254.
[2] 玛依乐·艾海提, 西热娜依·阿布力克木, 努尔古丽·热合曼.应用高通量测序法检测南疆传统酸奶中微生物多样性[J].食品科学, 2018, 39(20):126-131.
MAYILE A H T, XIRENAYI A B L K M, NUERGULI R H M.Diversity of culturable microorganisms in traditional fermented milk from south Xinjiang as analyzed by high-throughput pyrosequencing[J].Food Science, 2018, 39(20):126-131.
[3] 陈礼福. 食品微生物检测技术应用现状及展望[J].轻工科技, 2020, 36(12):68-69.
CHEN L F.Application status and prospect of food microbiological detection technology[J].Light Industry Science and Technology, 2020, 36(12):68-69.
[4] 石吉勇, 吴胜斌, 邹小波, 等.基于高光谱技术的酸奶中常见致病菌的快速鉴别及计数[J].光谱学与光谱分析, 2019, 39(4):1 186-1 191.
SHI J Y, WU S B, ZOU X B, et al.Rapid identification and enumeration of common pathogens in yogurt using hyperspectral imaging[J].Spectroscopy and Spectral Analysis, 2019, 39(4):1 186-1 191.
[5] 朱潇, 梁琪, 王湘竹, 等.基于Illumina MiSeq高通量技术比较甘肃藏区传统牦牛发酵乳制品细菌菌群多样性[J].中国食品学报, 2021, 21(4):336-344.
ZHU X, LIANG Q, WANG X Z, et al.Comparison of bacterial diversity in traditional yak fermented dairy products in Tibetan areas of Gansu based on Illumina MiSeq high-throughput technology[J].Journal of Chinese Institute of Food Science and Technology, 2021, 21(4):336-344.
[6] 卢圣鄂, 肖波, 任风鸣, 等.基于Illumina Miseq分析黄精根腐病根际土壤真菌群落结构及多样性[J].世界科学技术-中医药现代化, 2021, 23(1):13-19.
LU S E, XIAO B, REN F M, et al.Fungal community structure and diversity of rhizosphere soil of Polygonatum sibiricum with root-rot analyzed by Illumina MiSeq high-throughput sequencing technology[J].World Science and Technology-Modernization of Traditional Chinese Medicine and Materia Materia, 2021, 23(1):13-19.
[7] 刘怡萱, 许国琪, 曹鹏熙, 等.基于16S rRNA高通量测序的西藏农、牧区牦牛酸奶菌群多样性分析[J].食品科学, 2020, 41(18):92-97.
LIU Y X, XU G Q, CAO P X, et al.Microbial diversity of yak yogurt in agricultural and pastoral areas of Tibet analyzed based on high-throughput 16S rRNA gene sequencing[J].Food Science, 2020, 41(18):92-97.
[8] DSOUZA M, TAYLOR M W, TURNER S J, et al.Genomic and phenotypic insights into the ecology of Arthrobacter from Antarctic soils[J].BMC Genomics, 2015, 16(1):36.
[9] SADIQ F A, FLINT S, YUAN L, et al.Propensity for biofilm formation by aerobic mesophilic and thermophilic spore forming bacteria isolated from Chinese milk powders[J].International Journal of Food Microbiology, 2017, 262:89-98.
[10] YULIA N, WIBOWO A, KOSASIH E D.Karakteristik minuman probiotik sari ubi kayu dari kultur bakteri Lactobacillus acidophilus Dan Streptococcus thermophilus[J].Jurnal Kefarmasian Indonesia, 2020:87-94.
[11] YULIANI K, KERTIA N, LESTARI L.Efek konsumsi yoghurt probiotik terhadap Kadar low density lipoprotein pada penyandang diabetes mellitus tipe 2[J].Jurnal Gizi Indonesia (The Indonesian Journal of Nutrition), 2020, 8(1):69.
[12] LIU W J, SUN Z H, ZHANG Y B, et al.A survey of the bacterial composition of kurut from Tibet using a culture-independent approach[J].Journal of Dairy Science, 2012, 95(3):1 064-1 072.
[13] JIAO W S, WANG S, GUAN J Q, et al.Milk fermented with Lactococcus lactis KLDS4.0325 alleviates folate status in deficient mice[J].Food & Function, 2020, 11(5):4 571-4 581.
[14] MUKHTAR H, YAQUB S, HAQ I U.Production of probiotic Mozzarella cheese by incorporating locally isolated Lactobacillus acidophilus[J].Annals of Microbiology, 2020, 70(1):56-69.
[15] LI W X, YANG L, NAN W L, et al.Whole-genome sequencing and genomic-based acid tolerance mechanisms of Lactobacillus delbrueckii subsp.bulgaricus LJJ[J].Applied Microbiology and Biotechnology, 2020, 104(17):7 631-7 642.
[16] KHALIL A, SIVAKUMAR N, ARSLAN M, et al.Novel Anoxybacillus flavithermus AK1:A thermophile isolated from a hot spring in Saudi Arabia[J].Arabian Journal for Science and Engineering, 2018, 43(1):73-81.
[17] 刘国荣, 苏航, 宋振芹, 等.一株长双歧杆菌及其在制备活性双歧杆菌发酵饮料中的应用:中国, CN104630099A[P].2015-05-20.
LIU G R, SU H, SONG Z Q, et al.A strain of Bifidobacterium longum and its application in preparing active Bifidobacterium fermented beverage:China, CN104630099A[P].2015-05-20.
[18] LIU C G, YANG X, LAI Y, et al.Imazamox microbial degradation by common clinical bacteria:Acinetobacter baumannii IB5 isolated from black soil in China shows high potency[J].Journal of Integrative Agriculture, 2016, 15(8):1 798-1 807.
[19] UMAR MUSTAPHA M, HALIMOON N, WAN JOHARI W L, et al.Enhanced carbofuran degradation using immobilized and free cells of Enterobacter sp. isolated from soil[J].Molecules, 2020, 25(12):2 771-2 785.
[20] YANG J, CAO Y, CAI Y, et al.Natural populations of lactic acid bacteria isolated from vegetable residues and silage fermentation[J].Journal of Dairy Science, 2010, 93(7):3 136-3 145.
[21] DE SIMONE N, CAPOZZI V, DE CHIARA M L V, et al.Screening of lactic acid bacteria for the bio-control of Botrytis cinerea and the potential of Lactiplantibacillus plantarum for eco-friendly preservation of fresh-cut kiwifruit[J].Microorganisms, 2021, 9(4):773.
[22] GALLO I F L, FURLAN J P R, SANCHEZ D G, et al.Heavy metal resistance genes and plasmid-mediated quinolone resistance genes in Arthrobacter sp. isolated from Brazilian soils[J].Antonie Van Leeuwenhoek, 2019, 112(10):1 553-1 558.
[23] 赵孟孟, 江吉山.奶牛乳房炎的发病机制与预防措施[J].中国乳业, 2021(2):55-56.
ZHAO M M, JIANG J S.Pathogenesis and preventive measures of mastitis in dairy cows[J].China Dairy, 2021(2):55-56.
[24] FERNANDO D M, KHAN I U H, PATIDAR R, et al.Isolation and characterization of Acinetobacter baumannii recovered from Campylobacter selective medium[J].Frontiers in Microbiology, 2016, 7:1 871-1 880.
[25] 曹慧慧, 董李学, 王磊, 等.奶牛养殖场榨乳环节不同取样点细菌菌群的16S rRNA分子生物学分析[J].黑龙江畜牧兽医, 2021(3):70-74;159.
CAO H H, DONG L X, WANG L, et al.Molecular biological analysis of bacterial flora 16S rRNA at different sampling sites during milking process in dairy farms[J].Heilongjiang Animal Science and Veterinary Medicine, 2021(3):70-74;159.
[26] COLERI CIHAN A, KARACA B, OZEL B P, et al.Determination of the biofilm production capacities and characteristics of members belonging to Bacillaceae family[J].World Journal of Microbiology and Biotechnology, 2017, 33(6):118-131.