芳香族化合物被广泛用作生产高附加值产品的原材料,如聚合物、食品添加剂和药品,或者用作生物燃料,如2-苯基乙醇[1]。芳香族氨基酸是典型的芳香族化学物质,通过微生物莽草酸途径产生。在该途径中,从共同的前体磷酸烯醇式丙酮酸和四磷酸赤藓糖经分支酸产生L-苯丙氨酸、L-色氨酸和L-酪氨酸[2]。由于在药物和工业中的广泛应用,酪氨酸已引起了广泛的关注,并被用于生产食品添加剂[3],苄基异喹啉生物碱[4]、阿片类药物[5]和柚皮苷[6]。除此之外,L-酪氨酸还可用于帕金森氏病药物左旋多巴[7]和芳香族聚合物黑色素[8]的合成。
迄今为止,科研工作者已对多种微生物进行了酪氨酸合成的改造,谷氨酸棒杆菌[9]、球形双节杆菌[10]、乳铁短杆菌[11]和大肠杆菌[12]。其中,大肠杆菌相关的酪氨酸合成研究较为详细。尽管在过去40年中对莽草酸途径进行了大量研究,但是酪氨酸的高效合成仍然具有很大的挑战[13-14]。在大肠杆菌合成酪氨酸的研究中,工作主要集中于影响芳香族氨基酸生物合成的转录调节子tyrR,3-脱氧-7-磷酸庚酮酸合酶(AroGfbr,D146N),预苯酸脱氢酶(TyrAfbr,M53I/A354V),莽草酸脱氢酶(YidB),莽草酸激酶I(AroK)和莽草酸激酶II(AroL)[14-15]。有学者首先将aroGfbr和tyrAfbr基因克隆到低拷贝质粒,并转化入敲除tyrR的大肠杆菌K12中,在发酵过程中可产生0.346 g/L的L-酪氨酸,且aroGfbr,tyrAfbr,ydiB和aroK的组合过表达可以产生0.27 g/g DCW[15-16]。JUMINAGA等[17]通过将整个生物合成途径集中在2个质粒上,并改变质粒拷贝,强启动子替换,优化基因密码子和在基因前添加操纵子来解除瓶颈,酪氨酸产量达到2.17 g/L。KIM等[18]优化了一种质粒系统和DO-stat补料方法,通过在tyrP敲除菌中共表达aroGfbr,aroL和tyrC,合成了43.14 g/L L-酪氨酸。地衣芽胞杆菌是一种安全的革兰氏阳性菌,已被用作生产多糖生物絮凝剂[19]、聚-γ-谷氨酸[2,20]、抗菌肽[21]和碱性蛋白酶[22]。先前研究已实现了地衣芽胞杆菌代谢合成酪氨酸,并找出了酪氨酸合成的主要瓶颈问题[23]。
本研究对酪氨酸合成进行了发酵优化,并进一步探讨了地衣芽胞杆菌莽草酸途径的影响因素,通过以下4个方面对地衣芽胞杆菌合成酪氨酸进行了阐述:(1)碳源、温度以及溶氧;(2)vgb基因过表达;(3)莽草酸途径节点基因不同组合方式;(4)莽草酸补加。该研究为进一步实现地衣芽胞杆菌高效合成酪氨酸提供理论依据和技术支持。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
本研究中所使用的菌株、质粒如表1所示。
表1 本研究所用的菌株及质粒
Table 1 Strains and plasmids used in this study

菌株和质粒相关特征来源菌株JM109Escherichia coli,wild type,used for gene cloning本实验室CICIM B6902Bacillus licheniformis,wild type本实验室CICIM B6902::vgbBacillus licheniformis,△pflB::vgb本实验室质粒ApMA5 carrying the tyrAfbrgene,Ampr,Kanr本研究ACpMA5 carrying the tyrAfbrand aroC genes,Ampr,Kanr本研究ADpMA5 carrying the tyrAfbrand aroD genes,Ampr,Kanr本研究AKpMA5 carrying the tyrAfbrand aroK genes,Ampr,Kanr本研究AGpMA5 carrying the tyrAfbrand aroGfbrgenes,Ampr,Kanr本研究HAHGpMA5 carrying the PHpaIItyrAfbrand PHpaIIaroGfbrgenes,Ampr,Kanr本研究HAPGpMA5 carrying the PHpaIItyrAfbrand PP43aroGfbrgenes,Ampr,Kanr本研究HAPGCpMA5 carrying the PHpaIItyrAfbr,PP43aroGfbrand aroC genes,Ampr,Kanr本研究HAPGDpMA5 carrying the PHpaIItyrAfbr,PP43aroGfbrand aroD genes,Ampr,Kanr本研究HAPGKpMA5 carrying the PHpaIItyrAfbr,PP43aroGfbrand aroK genes,Ampr,Kanr本研究HGPApMA5 carrying the PHpaIIaroGfbrand PP43tyrAfbrgenes,Ampr,Kanr本研究HGPACpMA5 carrying the PHpaIIaroGfbr,PP43tyrAfbrand aroC genes,Ampr,Kanr本研究HGPADpMA5 carrying the PHpaIIaroGfbr,PP43tyrAfbrand aroD genes,Ampr,Kanr本研究HGPAKpMA5 carrying the PHpaIIaroGfbr,PP43tyrAfbrand aroK genes,Ampr,Kanr本研究HGPALpMA5 carrying the PHpaIIaroGfbr,PP43tyrAfbrand aroL genes,Ampr,Kanr本研究
1.1.2 主要仪器与试剂
L-酪氨酸标品,上海源叶生物科技有限公司;胰蛋白胨、酵母提取物,Oxoid公司;限制性核酸内切酶、T4 DNA连接酶,美国Thermo Fisher公司;2×Taq PCR Master Mix,杭州宝赛公司;质粒DNA提取试剂盒、DNA纯化试剂盒、DNA凝胶回收试剂盒、RNA提取试剂盒,康宁生命科学有限公司(上海);卡那霉素、氨苄青霉素、四环素,Sigma公司;其他试剂均为国产或进口分析纯。
1.1.3 培养基与培养条件
LB培养基:蛋白胨10 g,酵母粉5 g,NaCl 10 g,蒸馏水1 L,固体LB培养基需加入20 g/L的琼脂粉。LX发酵培养基:葡萄糖30 g,蛋白胨FP321 20 g,酵母粉FM408 10 g,玉米浆干粉5 g,K2HPO4·3H2O 9.12 g,KH2PO4 1.36 g,CaCl2 0.50 g,MgSO4·7H2O 0.50 g,(NH4)2HPO4 10 g,蒸馏水1 L。菌株构建和发酵过程中,培养基中添加的氨苄青霉素、卡那霉素、四环素终质量浓度分别为100、30、20 mg/L。
1.2 实验方法
1.2.1 细胞生长、乳糖消耗和莽草酸含量检测
为了检测生物量的增长,不同时间点收集1 mL发酵液在12 000×g下离心5 min,去除上清液,细胞沉淀用9 g/L NaCl溶液洗涤并重悬于等体积的水中。分光光度计测定600 nm下吸光值,确保稀释样品OD600读数为在0.3~0.8。日立高效液相示差检测器测定乳糖浓度[16]和紫外检测器210 nm下测定莽草酸浓度[24],液相色谱柱为Shodex SUGAR SH1011色谱柱(300 mm×8 mm),上清液使用终质量浓度50 g/L三氯乙酸4 ℃处理于3 h并12 000×g下离心20 min。将500 μL上清液加入液相瓶中,流动相为10 mmol/L H2SO4,流速为0.8 mL/min,持续25 min。
1.2.2 RT-PCR引物的设计和扩增效率检测
RT-PCR引物由Beacon Designer 7.0 软件设计(http://www.premierbiosoft.com/molecular_beacons/index.html),引物序列如表2所示。
表2 本研究荧光定量PCR所用引物
Table 2 Primers for qRT-PCR in this study

qRT-PCR引物引物序列(5′ to 3′)aroA-F/RTGAGGCAGCAGGCGAATGAAT/TCTGACTGGATCGTAGCGGTTGAaroB-F/RGCTCTTGTCATCCTTGAACCTTCC/GCTTGGCACCGCAACCTTaroC-F/RCCAACCACGATTTCGGGAAGACGC/CAGGCATCACCGCGATCTTAGGGAaroD-F/RGTGCGGCGAGGGCGATATTTAC/GCTGACAAGTCTGCTTCTGCTTCCaroE-F/RACGGAACGGATGCTGTCAATGT/GCACGACTTCACTGTCAGGAACTaroF-F/RGGAATGCCGATTGTCGTCAGAGG/GCAACAACCGCTTCAGCCACTAaroK-F/RCGCTTTCCAACAGGAGGACATCAG/CCATCCGCTGCTTCCAACTCTCaroH-F/RCATTACGGAGCGGACGAAGCA/GCGTGAATATCGGAAGTGGCAGAAtyrA-F/RCGTGTCAGAAGAGCAGGCAAGA/GAAGCGGCAAGTTCATCAAGCAThisC-F/RCGAAGGAGCGGAAGTCAGGGAAAT/TCAGGTTGCGGCTGGATTCATACGrpsE-F/RTGGTCGTCGTTTCCGCTTCG/TCGCTTCTGGTACTTCTTGTGCTT
RT-PCR产物的特异性通过高分辨率凝胶电泳分析,反应得到单一产物所需的长度。引物扩增效率值按公式(1)计算[25]:
(1)
式中:E,引物扩增效率值;k,标准曲线斜率。不同基因引物扩增效率值:aroA,1.95;aroB,1.91;aroC,1.96;aroD,1.94;aroE,1.96;aroF,1.96;aroK,2.00;aroH,1.98;tyrA,1.99;hisC,1.91;rpsE,2.01。
1.2.3 莽草酸途径转录水平分析
重组菌细胞4 ℃,12 000×g下离心1 min。去除上清液,细胞使用冰水洗涤3次,然后使用RNAprep纯细胞/细菌试剂盒(天根,中国)提取总RNA并使用QUAWELL Q5000(上海)检测RNA浓度。根据操作手册,用gDNA Eraser(TaKaRa,日本)处理后总RNA作为模板。根据操作手册,使用PrimeScriptTMRT试剂盒(TaKaRa,日本)在42 ℃下合成cDNA。将cDNA稀释至200 ng/μL并进行下一步的RT-PCR。使用SYBR® Premix Ex TaqTM II(TaKaRa,日本)将上述cDNA与合适的引物混合用于定量RT-PCR并使用CFX96检测系统(Bio-Rad,美国)进行实时定量检测。PCR条件:95 ℃,30 s;95 ℃,5 s和60 ℃,30 s,40个循环。rpsE作为内参基因[23]并使用PFAFFL[25]提出的模型进行数据分析。
1.2.4 L-酪氨酸分析
将发酵液样品以1∶1的体积比用2 mol/L HCl处理,以确保L-酪氨酸完全溶解,然后4 ℃下12 h后将处理液以12 000×g离心20 min,将总体积为500 μL的上清液加入到液相瓶中。上清液通过HPLC(Agilent 1260系列;Agilent Technologies,USA)分析,使用邻苯二甲醛3-巯基丙酸衍生化,并通过二极管阵列检测器使用ODS HYPERSIL色谱柱(4.6 mm×250 mm,3.5 μm)检测。流动相由溶剂A和溶剂B组成。溶剂A是5 g/L乙酸钠溶液,用2%乙酸调整pH值为7.2。溶剂B(1 L)由5 g乙酸钠,400 mL甲醇和400 mL乙腈,200 mL超纯水组成,用5%的乙酸将其调整至pH 7.2。检测方法:0 min,92%溶剂A+8%溶剂B,流速1 mL/min;27.5 min,40%溶剂A+60%溶剂B,流速1 mL/min;31.5 min,100%溶剂B,流速1.5 mL/min;32 min,100%溶剂B,流速1.5 mL/min;34 min,100%溶剂B,流速1 mL/min;35.5 min,92%溶剂A + 8%溶剂B,流速1 mL/min。总运行时间为40 min。用于检测L-酪氨酸的检测器波长为338 nm,带宽为4 nm。
2 结果与分析
2.1 酪氨酸合成重组质粒构建
大肠杆菌来源的aroGfbr与tyrAfbr按照地衣芽胞杆菌密码子偏好性进行密码子优化并送往公司合成(Synbio Technologies,中国)。aroGfbr与tyrAfbr优化后基因核酸序列提交至NCBI,序列号分别为MK002699和MK002700。aroC、aroD和aroK通过PCR扩增于地衣芽胞杆菌基因组,片段大小分别为762、837和528 bp。将重组质粒A以及片段aroC、aroD、aroK以及aroGfbr分别使用BamH I与NheI双酶切,按照1.2.2的方法构建了重组质粒AC、AD、AK和AG。与此同时,将不同强度启动子连接至重组质粒AG,构建了重组质粒HAHG与HAPG。进一步将aroC、aroD与aroK分别连接至HAPG,构建了重组质粒HAPGC、HAPGD和HAPGK。将重组质粒HGHA中tyrAfbr的启动子HpaII替换为启动子P43以及在此基础上分别串联表达基因aroC、aroD、aroK以及aroL,成功构建了重组质粒HGPA、HGPAC、HGPAD、HGPAK、HGPAL。所有插入基因片段均经公司测序正确,说明HGPA等12种酪氨酸合成重组质粒构建成功(如表1所示)。
2.2 碳源、温度、溶氧以及接种量对酪氨酸合成的影响
为了更好地评价构建菌株,首先对发酵碳源以及发酵条件进行优化。根据文献报道,地衣芽胞杆菌发酵过程中在使用不同碳源情况下发酵结果差异性较大,在葡萄糖作为碳源时地衣芽胞杆菌生长比较旺盛,而利用乳糖更有利于合成储存物质[26]。乳糖的添加可将地衣芽胞杆菌合成杆菌肽效价从960 U/mL提升至1 143 U/mL,提高了19%[27]。如图1-a所示,在葡萄糖、蔗糖、乳糖以及木糖做为碳源时,发酵48 h时地衣芽胞杆菌合成酪氨酸单位菌体产量分别为20.10、22.98、31.05和5.15 mg/L。在使用乳糖作为碳源时,酪氨酸单位菌体产量较葡萄糖提高54%,说明乳糖作为碳源时更利于地衣芽胞杆菌合成酪氨酸。同时,地衣芽胞杆菌利用乳糖为碳源不会合成2,3-丁二醇,减少了碳源的损失。在后续重组菌评价过程中,选择了乳糖作为地衣芽胞杆菌发酵碳源。在乳糖作为碳源条件下,对温度、转速以及接种量进行了初步优化(图1-b,1-c,1-d),确认最适的发酵条件为37 ℃、转速200 r/min和3%接种量。在此条件下发酵120 h,重组菌HGPA最终合成酪氨酸单位菌体产量可以达到71 mg/L(图2-a)。
2.3 vgb基因过表达对酪氨酸合成的影响
透明颤菌血红蛋白(Vitreoscilla hemoglobin,VHb)是一种由基因vgb编码的同型二聚氧结合蛋白,可通过促进氧气输送来增强细菌在低氧环境下的呼吸和代谢;该蛋白具有自己的核糖体结合位点,已成功应用到各种异源细菌和真核生物中,以提高细胞密度、蛋白质合成和氧化代谢[28]。vgb基因过表达对左旋多巴合成具有促进作用,而左旋多巴的前体为酪氨酸[29]。因此vgb基因的过表达有可能可以促进酪氨酸的合成。将HGPA电转入vgb基因整合菌株后经验证发现,vgb基因的过表达可以将酪氨酸单位菌体产量从71 mg/L提升至100 mg/L,提升了40.8%(图2-a)。该结果说明提升地衣芽胞杆菌的摄氧能力有助于酪氨酸的合成。

a-碳源;b-发酵温度;c-发酵转速;d-发酵接种量
图1 地衣芽胞杆菌发酵合成酪氨酸的碳源以及发酵条件优化
Fig.1 Optimization of carbon source and fermentation conditions in synthesizing L-tyrosine by B.licheniformis
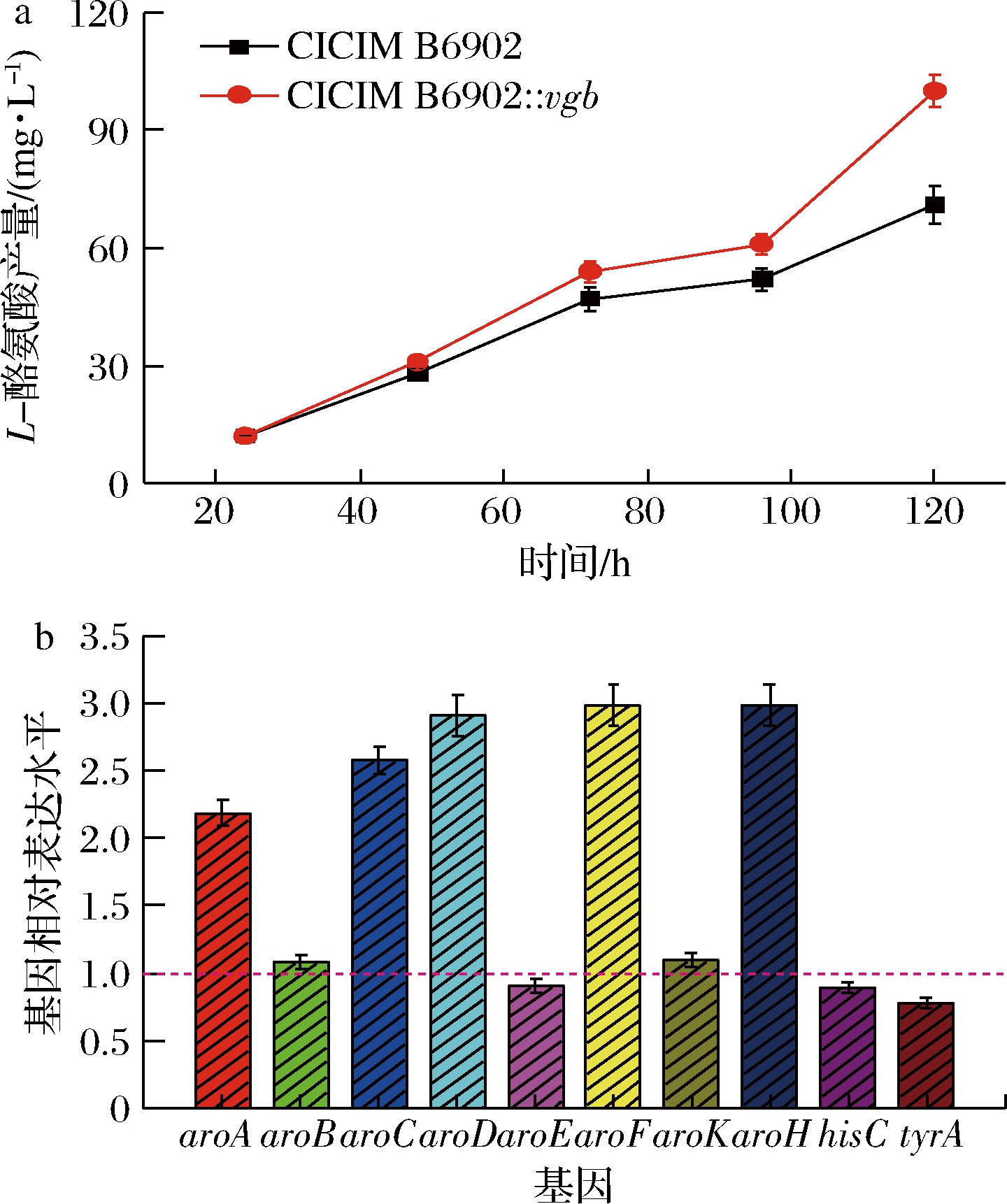
a-酪氨酸合成;b-莽草酸途径基因转录水平
图2 地衣芽胞杆菌过表达vgb基因对酪氨酸合成
以及莽草酸途径基因转录水平的影响
Fig.2 Effect of overexpression of vgb gene on L-tyrosine
synthesis and gene transcription level of shikimate
pathway in B.licheniformis
注:b虚线指示对照菌CICIM B6902莽草酸途径基因转录水平
为了挖掘酪氨酸提升的因素,对原始菌与vgb整合菌进行了莽草酸途径转录分析(图2-b)。vgb重组菌中aroA,aroC,aroD,aroF以及aroH较原始菌分别提升了2.19,2.57,2.91,2.98和2.89倍。值得注意的是,vgb重组菌中aroK的表达水平较原始菌并没有明显的变化。先前文献报道,地衣芽胞杆菌中aroC,aroD和aroK基因是酪氨酸合成的限速步骤并且在消耗葡萄糖的条件下过表达aroK可以提高酪氨酸的合成量[16]。vgb的表达以及莽草酸途径基因的转录水平分析从另一角度证明了增强地衣芽胞杆菌摄氧能力可以提升胞内aroC与aroD的表达水平,进而增强地衣芽胞杆菌合成酪氨酸的能力。同时,该结果表明aroC与aroD的表达与胞内溶氧水平相关联。但高溶氧条件并不是酪氨酸合成的最适条件(图1-c),说明高溶氧条件可能会对其他酪氨酸合成关键代谢步骤起到抑制作用。
2.4 莽草酸途径节点基因不同组合方式对酪氨酸合成的影响
将aroC,aroD,aroK以及aroGfbr与tyrAfbr共表达分别可以合成866,754,686和831 mg/L酪氨酸,比单独表达tyrAfbr分别提升了68.7,53.4,85.8和93.8%(图3-a)。由于过表达aroGfbr对酪氨酸提升幅度最大,研究通过改变aroGfbr启动子来优化其表达强度(图3-b)。在AG质粒基础上分别在aroGfbr基因钱端插入HpaII与P43两种不同强度启动子。在转录强度相对弱的启动子HpaII下表达有利于酪氨酸的合成,产量达到1 089 mg/L。值得注意的是,在强启动子P43下表达aroGfbr后,酪氨酸合成量急剧降低至97 mg/L。该情况可能是由于aroGfbr表达强度过高,导致后续代谢途径堵塞并引起反馈抑制。为验证该推测,在HAPG的基础上分别过表达了aroC、aroD和aroK(图3-c)。在表达aroD与aroK的情况下,酪氨酸分别有不同程度的回升。HAPGD与HAPGK的酪氨酸产量分别回升至1 062和319 mg/L,HAPGC依旧没有酪氨酸合成。该结果证明了aroD和aroK为地衣芽胞杆菌合成酪氨酸的重要节点,aroGfbr表达强度过高会引起该途径节点的堵塞。为了进一步提升酪氨酸合成量,在HGPA基础上分别过表达了aroC、aroD、aroK以及大肠杆菌来源的aroL。其中aroC以及aroL并没有起到促进酪氨酸合成的作用。HGPAD与HGPAK的酪氨酸合成能力类似,酪氨酸产量均可以达到1 200 mg/L(图3-d)。
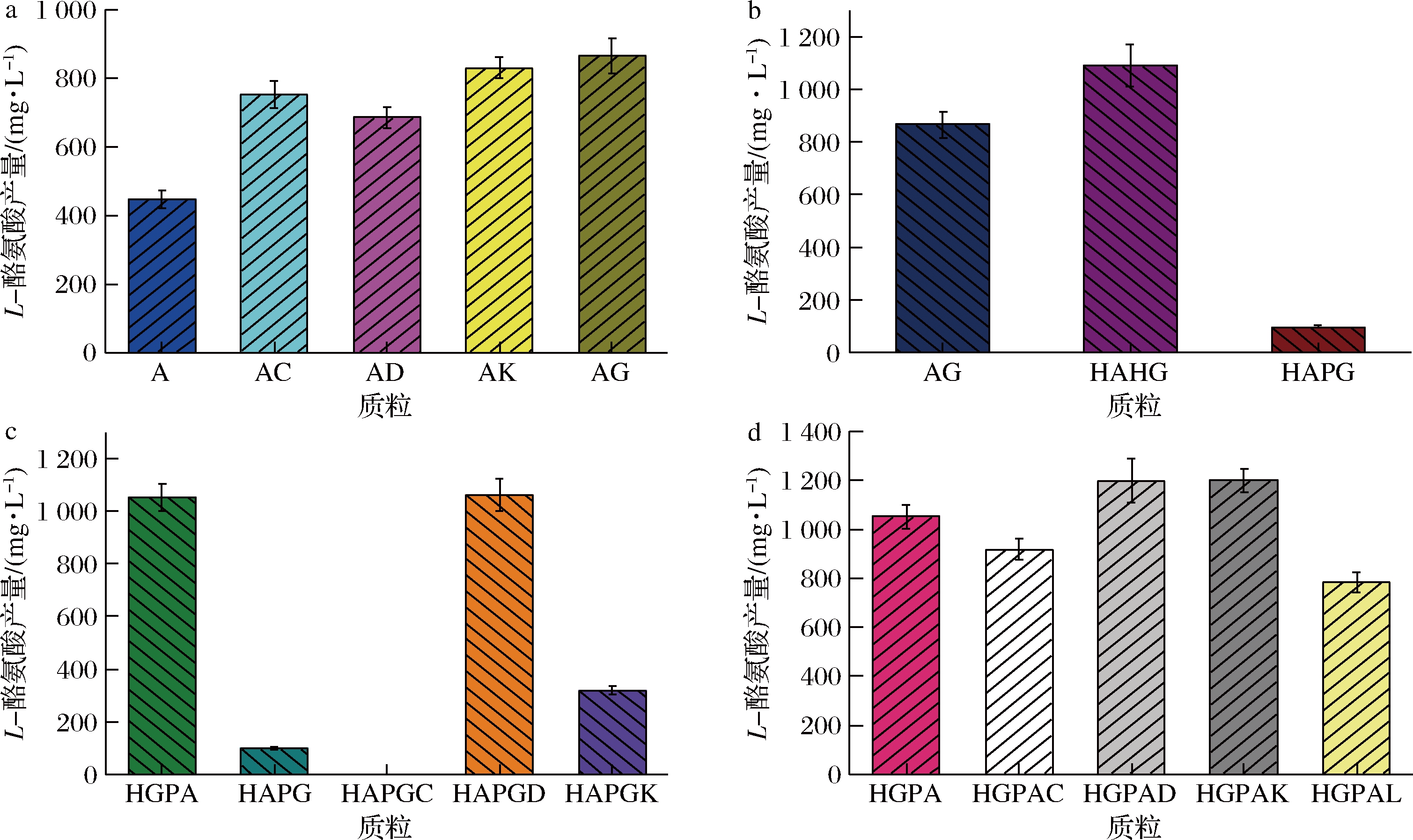
a-aroC、aroD、aroK以及aroGfbr分别与tyrAfbr串联表达;b-aroGfbr的不同表达强度;c-在aroGfbr
强表达下,aroC、aroD与aroK过表达;d-在tyrAfbr强表达下,aroC、aroD、aroK以及aroL过表达
图3 地衣芽胞杆菌中莽草酸途径关键节点基因不同组合方式对酪氨酸合成的影响
Fig.3 Effects of different combinations of key node genes in shikimate pathway on L-tyrosine synthesis by B.licheniformis
2.5 莽草酸补加对酪氨酸合成的影响
虽然HGPAD与HGPAK的酪氨酸合成比HGPA提升了14.1%,但是要远低于AD与AK相对A的提升幅度(53.4%和85.8%)。据此,进一步分析了在不同莽草酸浓度情况下HGPA与HGPAK的酪氨酸合成状况(图4)。在发酵结束时,补加3 g/L莽草酸对HGPA与HGPAK的酪氨酸合成能力并没有明显的提升。当莽草酸质量浓度提升至5 g/L时,HGPA与HGPAK的酪氨酸合成分别可以达到1 311和1 490 mg/L。随着莽草酸质量浓度提升至10 g/L,酪氨酸浓度不再提升,说明添加5 g/L莽草酸时,莽草酸途径代谢流已经处于饱和状态。由此表明,aroD和aroK的过表达未能彻底解除节点代谢流的限制。地衣芽胞杆菌中存在调控该节点的机制并有待挖掘。
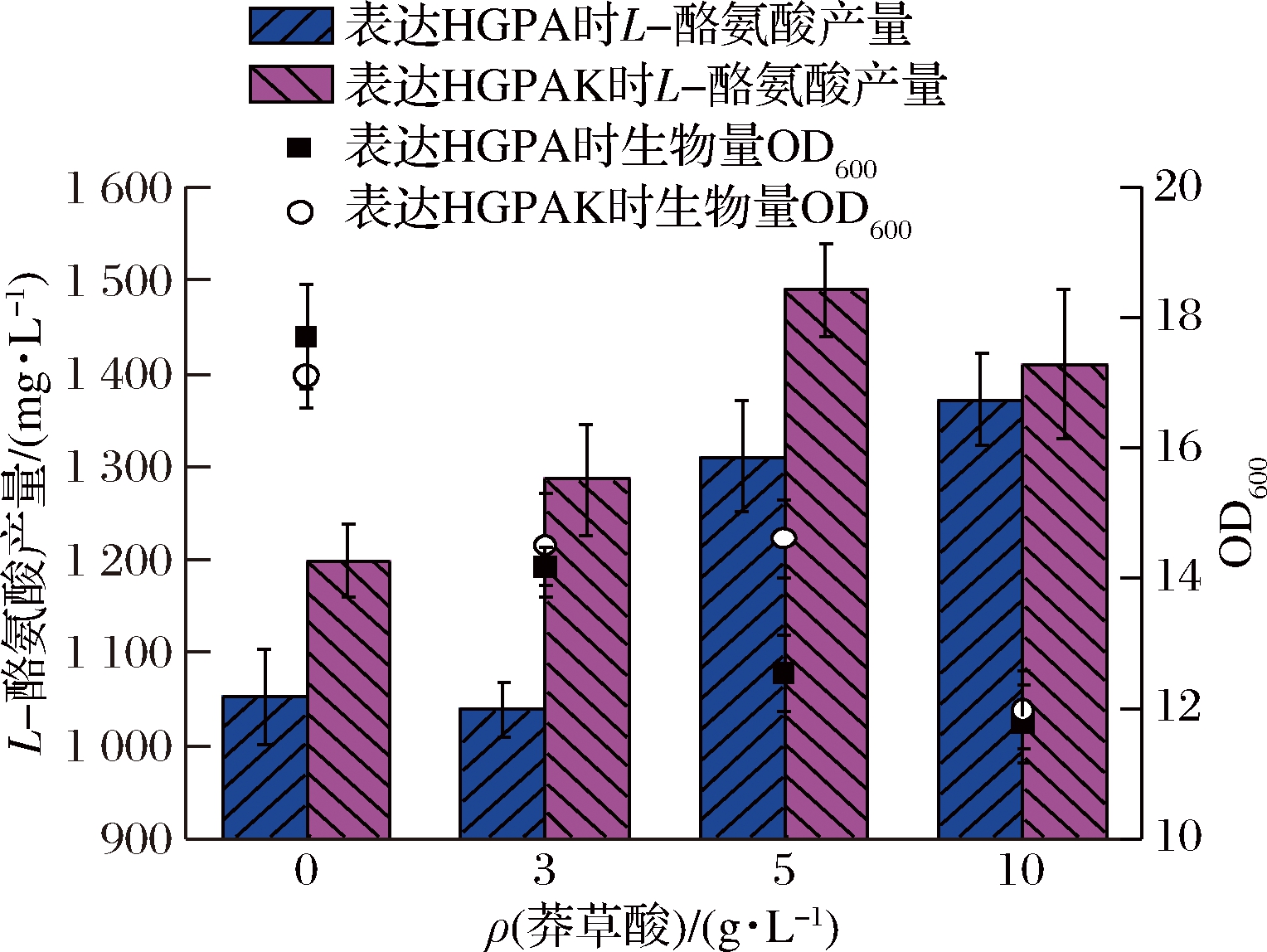
图4 添加不同浓度莽草酸对酪氨酸合成的影响
Fig.4 Effect of different concentrations of shikimic acid on the
synthesis of L-tyrosine by B.licheniformis
3 结论与讨论
本研究以地衣芽胞杆菌为研究对象,通过基因过表达,初步实现L-酪氨酸在地衣芽胞杆菌中的代谢合成。通过检测碳源、温度、转速和接种量对L-酪氨酸产量的影响对发酵条件进行优化,在添加乳糖,温度37 ℃,转速200 r/min和3%接种量的条件下,重组菌HGPA最终合成酪氨酸单位菌体产量可以达到71 mg/L,较优化前提升77%。增强地衣芽胞杆菌摄氧能力可以提升胞内aroC与aroD的表达水平,进而增强地衣芽胞杆菌合成酪氨酸的能力,酪氨酸单位菌体产量从71 mg/L提升至100 mg/L,提升了40.8%。在地衣芽胞杆菌中过表达aroGfbr,tyrAfbr,aroD或aroK,产物质量浓度可以达到1 200 mg/L。进一步通过体外莽草酸补给,L-酪氨酸合成量可以达到1 500 mg/L。地衣芽胞杆菌合成酪氨酸过程中,基因aroD和aroK是莽草酸途径的关键瓶颈,基因的过表达并不能彻底解除节点代谢流的限制,该节点的其他调控机制有待进一步挖掘。
[1] NODA S,KONDO A.Recent advances in microbial production of aromatic chemicals and derivatives[J].Trends in Biotechnology,2017,35(8):785-796.
[2] LIU S P,ZHANG L,MAO J,et al.Metabolic engineering of Escherichia coli for the production of phenylpyruvate derivatives[J].Metabolic Engineering,2015,32:55-65.
[3] ÇAL K P,ÖZDAMAR T H.Mass flux balance-based model and metabolic pathway engineering analysis for serine alkaline protease synthesis by Bacillus licheniformis[J].Enzyme and Microbial Technology,1999,24(10):621-635.
K P,ÖZDAMAR T H.Mass flux balance-based model and metabolic pathway engineering analysis for serine alkaline protease synthesis by Bacillus licheniformis[J].Enzyme and Microbial Technology,1999,24(10):621-635.
[4] CH VEZ-BÉJAR M I,B
VEZ-BÉJAR M I,B EZ-VIVEROS J L,MART
EZ-VIVEROS J L,MART NEZ A,et al.Biotechnological production of L-tyrosine and derived compounds[J].Process Biochemistry,2012,47(7):1 017-1 026.
NEZ A,et al.Biotechnological production of L-tyrosine and derived compounds[J].Process Biochemistry,2012,47(7):1 017-1 026.
[5] CHEN Z,LIU P Z,LI Z P,et al.Identification of key genes involved in polysaccharide bioflocculant synthesis in Bacillus licheniformis[J].Biotechnology and Bioengineering,2017,114(3):645-655.
[6] DEFEYTER R C,PITTARD J.Purification and properties of shikimate kinase II from Escherichia coli K-12[J].Journal of Bacteriology,1986,165(1):331-333.
[7] DICKSON J M J,LEE W J,SHEPHERD P R,et al.Enzyme activity effects of N-terminal His-tag attached to catalytic sub-unit of phosphoinositide-3-kinase[J].Bioscience Reports,2013,33(6):DOI:10.1042/bsr20130075.
[8] ELY B,PITTARD J.Aromatic amino acid biosynthesis:Regulation of shikimate kinase in Escherichia coli K-12[J].Journal of Bacteriology,1979,138(3):933-943.
[9] ESCALANTE A,CALDER N R,VALDIVIA A,et al.Metabolic engineering for the production of shikimic acid in an evolved Escherichia coli strain lacking the phosphoenolpyruvate:Carbohydrate phosphotransferase system[J].Microbial Cell Factories,2010,9:21.
N R,VALDIVIA A,et al.Metabolic engineering for the production of shikimic acid in an evolved Escherichia coli strain lacking the phosphoenolpyruvate:Carbohydrate phosphotransferase system[J].Microbial Cell Factories,2010,9:21.
[10] GALANIE S,THODEY K,TRENCHARD I J,et al.Complete biosynthesis of opioids in yeast[J].Science,2015,349(6252):1 095-1 100.
[11] GOSSET G.Production of aromatic compounds in bacteria[J].Current Opinion in Biotechnology,2009,20(6):651-658.
[12] HOFFMANN K,WOLLHERR A,LARSEN M,et al.Facilitation of direct conditional knockout of essential genes in Bacillus licheniformis DSM13 by comparative genetic analysis and manipulation of genetic competence[J].Applied and Environmental Microbiology,2010,76(15):5 046-5 057.
[13] HU C Y,JIANG P H,XU J F,et al.Mutation analysis of the feedback inhibition site of phenylalanine-sensitive 3-deoxy-D-arabino-heptulosonate 7-phosphate synthase of Escherichia coli[J].Journal of Basic Microbiology,2003,43(5):399-406.
[14] IKEDA M,KATSUMATA R.Metabolic engineering to produce tyrosine or phenylalanine in a tryptophan-producing Corynebacterium glutamicum strain[J].Applied and Environmental Microbiology,1992,58(3):781-785.
[15] ITO H,SAKURAI S,TANAKA T,et al.Genetic breeding of L-tyrosine producer from Brevibacterium lactofermentum[J].Agricultural and Biological Chemistry,1990,54(3):699-705.
[16] LÜTKE-EVERSLOH T,STEPHANOPOULOS G.L-Tyrosine production by deregulated strains of Escherichia coli[J].Applied Microbiology and Biotechnology,2007,75(1):103-110.
[17] JUMINAGA D,BAIDOO E E K,REDDING-JOHANSON A M,et al.Modular engineering of L-tyrosine production in Escherichia coli[J].Applied and Environmental Microbiology,2012,78(1):89-98.
[18] KIM B,BINKLEY R,KIM H U,et al.Metabolic engineering of Escherichia coli for the enhanced production of L-tyrosine[J].Biotechnology and Bioengineering,2018,115(10):2 554-2 564.
[19] KO Y H,GROSS R A.Effects of glucose and glycerol on γ-poly(glutamic acid) formation by Bacillus licheniformis ATCC 9945a[J].Biotechnology and Bioengineering,1998,57(4):430-437.
[20] LI Y R,LI S J,THODEY K,et al.Complete biosynthesis of noscapine and halogenated alkaloids in yeast[J].Proceedings of the National Academy of Sciences of the United States of America,2018,115(17):E3922-E3931.
[21] LIU Z Y,YU W L,NOMURA C T,et al.Increased flux through the TCA cycle enhances bacitracin production by Bacillus licheniformis DW2[J].Applied Microbiology and Biotechnology,2018,102(16):6 935-6 946.
[22] LOBNER-OLESEN A,MARINUS M G.Identification of the gene(aroK) encoding shikimic acid kinase I of Escherichia coli[J].Journal of Bacteriology,1992,174(2):525-529.
[23] XU Y B,LI Y R,ZHANG L,et al.Unraveling the specific regulation of the shikimate pathway for tyrosine accumulation in Bacillus licheniformis[J].Journal of Industrial Microbiology and Biotechnology,2019,46(8):1 047-1 059.
[24] LYU X M,NG K R,LEE J L,et al.Enhancement of naringenin biosynthesis from tyrosine by metabolic engineering of saccharomyces cerevisiae[J].Journal of Agricultural and Food Chemistry,2017,65(31):6 638-6 646.
[25] PFAFFL M W.A new mathematical model for relative quantification in real-time RT-PCR[J].Nucleic Acids Research,2001,29(9):e45.
[26] DA SILVA T L,REIS A,KENT C A,et al.Stress-induced physiological responses to starvation periods as well as glucose and lactose pulses in Bacillus licheniformis CCMI 1034 continuous aerobic fermentation processes as measured by multi-parameter flow cytometry[J].Biochemical Engineering Journal,2005,24(1):31-41.
[27] 刘道奇, 陈守文,李俊辉,等.混合碳源对地衣芽胞杆菌发酵合成杆菌肽的影响[J].食品与发酵工业,2017,43(9):52-57.
LIU D Q,CHEN S W,LI J H,et al.Effects of the mixed carbon resource addition on bacitracin biosynthesis by Bacillus licheniformis[J].Food and Fermentation Industries,2017,43(9):52-57.
[28] ERENLER S O,GENCER S,GECKIL H,et al.Cloning and expression of the Vitreoscilla hemoglobin gene in Enterobacter aerogenes:Effect on cell growth and oxygen uptake[J].Applied Biochemistry and Microbiology,2004,40(3):241-248.
[29] KURT A G,AYTAN E,OZER U,et al.Production of L-DOPA and dopamine in recombinant bacteria bearing the Vitreoscilla hemoglobin gene[J].Biotechnology Journal,2009,4(7):1 077-1 088.