岩藻聚糖硫酸酯主要来源于褐藻、棘皮动物和单壳软体动物[1],是一种富含硫酸基的杂多糖[2],主要由岩藻糖、半乳糖、木糖和葡萄糖等单糖组成[3]。它具有良好的抗病毒、抗氧化、抗凝血、抗肿瘤、抗炎[4-8]等生物活性。然而,岩藻聚糖硫酸酯因其分子质量大、结构复杂、黏度大等特点,影响了其生物利用率和溶解性[9]。目前,通过降解岩藻聚糖硫酸酯制备低分子质量的岩藻寡糖已成为研究热点。
岩藻聚糖硫酸酯的降解方法有很多种,主要分为化学法、物理法和生物酶法[10]。其中的生物酶降解法反应条件较温和,不会造成活性基团的丢失且降解产物分子质量易控制,是一种具有独特优势的降解方法。然而产岩藻聚糖硫酸酯酶的菌株大多来源于海洋,岩藻聚糖硫酸酯酶不易获取,且酶活力普遍较低[11]。本文将从岩藻聚糖硫酸酯酶的分类及来源、性质及酶解产物的结构与活性等方面对岩藻聚糖硫酸酯酶进行综述,以期为岩藻聚糖硫酸酯酶的应用提供理论参考。
1 岩藻聚糖硫酸酯酶的分类及来源
1.1 岩藻聚糖硫酸酯酶的分类
催化岩藻聚糖硫酸酯残基之间糖苷键断裂的酶被称为岩藻聚糖硫酸酯酶。这些酶属于糖苷酶(酶代码EC 3.2.1)。岩藻聚糖硫酸酯降解酶主要有3种分别为α-L-岩藻糖苷酶(Fucosidase,EC 3.2.1.51)、岩藻多糖酶(Fucoidanase,EC 3.2.1.44)和硫酸酯酶(Sulfatase,EC 3.1.6)。它们能将多糖内部或者边缘的糖苷键裂解而达到降解岩藻多糖的目的,α-L-岩藻糖苷酶和岩藻多糖酶为构型保持酶,通过双置换催化机制来催化反应。而硫酸酯酶的催化机制为酯交换机制,在亲核攻击中使硫酸基团从初始底物中释放出来。根据作用机理可以将它们分为内切酶和外切酶,其中α-L-岩藻糖苷酶属于外切酶,其作用方式为从岩藻聚糖硫酸酯的非还原端水解岩藻糖苷键,多使岩藻糖和半乳糖之间的α-1→2-糖苷键断裂来完成水解作用,酶解产物为单糖[12]。岩藻多糖酶和硫酸酯酶属于内切酶,岩藻多糖酶能从中间或末端切开糖链,多作用于α-1→4-糖苷键,而硫酸酯酶主要作用于岩藻聚糖硫酸酯的α-1→3-和α-1→4-连接的岩藻糖残基,使其发生区域选择性脱硫导致多糖的裂解[13]。VANVLIET等[14]研究发现普朗克菌-疣微菌-衣原体(PVC)门的各种海洋细菌具有大量与硫酸化多糖降解相关的硫酸酯酶基因,从其分离出的菌株F1,F2是富含硫酸酯酶的厌氧菌。DESCAMPS等[15]从海洋微生物中出分离出α-L-岩藻糖苷酶,经过一段时间的培养发现该酶有一定的岩藻多糖降解能力。
1.2 岩藻聚糖硫酸酯酶的来源
1.2.1 来源于软体动物
一部分岩藻聚糖硫酸酯酶来源于海洋棘皮动物与单壳软体动物,如海参、海胆、鲍鱼等,多从它们的内脏中分离。THANASSI等[16]从鲍鱼肝胰腺中纯化出一种α-L-岩藻糖苷酶,这种酶能降解岩藻聚糖硫酸酯,虽然这种酶的活性较低,但酶解后的岩藻聚糖寡糖不会出现硫酸基团脱离的现象。TANAKA等[17]从单壳贝类中分离出一种酶,该酶为α-L-岩藻糖苷酶,可降解岩藻聚糖硫酸酯。从海洋软体动物中可纯化出岩藻聚糖硫酸酯降解酶,其原因可能是这些软体动物长期食用含有岩藻聚糖硫酸酯的藻类。但从软体动物中得到的岩藻聚糖硫酸酯酶往往活性较低,无法实现产业化应用。
1.2.2 微生物来源
与海洋动物相比,海洋性微生物是岩藻聚糖硫酸酯酶最主要的来源。SICHERT等[18]从海带中筛选获得了一株降解褐藻多糖硫酸酯的菌株RC2-3,它是一种革兰氏阴性细菌,仅在海水培养基上生长,其最大降解率为54%,用16S RNA基因部分序列的系统分析表明RC2-3菌株属于黄杆菌科(Flavobacteriaceae)。KIM等[19]从朝鲜海域的裙带菜孢子叶分离纯化出一株以岩藻聚糖硫酸酯为唯一碳源的海洋菌株,经过16S rDNA序列分析发现该菌株为鞘氨醇单胞菌,用全细胞酶制剂实验发现显色底物并未对硝基苯基进行水解,表明该酶可能是具有内切作用的岩藻多糖酶,而不是α-L-岩藻糖苷酶。KIM等[20]从海洋细菌鞘氨醇单胞菌PF-1中纯化出一种内切酶岩藻多糖酶,该酶可将从裙带菜孢子叶中分离出的硫酸化半乳岩藻聚糖降解为较小分子质量的半乳岩藻寡糖(1 000~4 000 Da),该酶的活性比粗酶高约112倍。OHSHIRO等[21]从褐藻冈村枝管藻中分离出一株以褐藻聚糖硫酸酯作为唯一碳源的细菌,经鉴定该细菌属于黄杆菌属(Flavobacterium)F-31,该细菌能产生一种使岩藻聚糖硫酸酯降解和脱硫的胞内酶:硫酸酯酶。IVANOVA等[22]从太平洋千岛群岛克拉特尔纳亚湾采集的褐藻岩藻菌体中分离出11株革兰氏阴性海洋细菌。通过16S rDNA分析发现该细菌属于假交替单胞菌(Issachenkonii),该细菌中含有多种酶,其中的岩藻多糖酶可降解海藻多糖。
除此之外,一些真菌也具有降解岩藻聚糖硫酸酯的能力。张袁等[23]从我国厦门海域采集的样品中筛选和复筛得到28株岩藻多糖酶产生菌,其中温特曲霉菌(Aspergillus wentii)的产酶能力最强。该团队还发现Fusarium sp.LD8和Dendryphiella arenaria TM94同样含有岩藻聚糖硫酸酯酶[24-25],可降解岩藻聚糖硫酸酯。RODR GUZE-JASSO等[26]通过2种真菌(PSH 和 Mucor sp.3P)的固态发酵产生能降解岩藻聚糖硫酸酯的岩藻多糖酶,其中Mucor sp.3P菌株的产酶活性较高。
GUZE-JASSO等[26]通过2种真菌(PSH 和 Mucor sp.3P)的固态发酵产生能降解岩藻聚糖硫酸酯的岩藻多糖酶,其中Mucor sp.3P菌株的产酶活性较高。
综上所述,大部分的岩藻聚糖硫酸酯降解酶属于黄杆菌科,还有一部分属于变形菌属及一些单细胞细菌、真菌。岩藻聚糖硫酸酯酶的来源稀少,该酶在市场上价格昂贵,寻找能产生高效岩藻聚糖硫酸酯酶的微生物对岩藻聚糖硫酸酯酶的应用与发展至关重要。
表1 岩藻聚糖硫酸酯酶的来源
Table 1 Resources of fucoidan degrading enzyme

来源产酶软体动物及微生物酶类型特征参考文献鲍鱼鲍鱼肝胰腺Fucosidase胞外[16]软体动物Lambis sp.Fucoidanase胞内[27]贝类单壳贝类Fucosidase胞外[17]昆布FlavobacteriaceaRC2Fucoidanase胞内[12]海带FlavobacteriaceaRC2-3Fucoidanase胞内[18]海洋细菌FlavobacteriaceaCZ1127Fucoidanase胞内[28]裙带菜孢子Sphingomonas paucimobilisFucoidanase胞内[19]海洋细菌Sphingomonas paucimobilisPF-1Fucoidanase胞内[20]冈村枝管藻FlavobacteriumSulfatase胞内[21]软体动物滨螺Fucoidanase胞内[29]褐藻IssachenkoniiFucoidanase胞内[22]真菌Aspergillus wentiiFucoidanase胞内[23]真菌Fusarium sp.LD8Fucoidanase胞内[24,30]真菌Dendryphiella arenaria TM94Fucoidanase胞内[25]真菌PSH and Mucor sp.3PFucoidanase胞内[26]海洋细菌PVCSulfatase胞内[14]海洋细菌Fucobacter裂解酶胞外[31]
2 碳源、氮源、NaCl、金属离子等对菌株产酶活性的影响
在产酶菌株培养过程中,碳源、氮源、NaCl和金属离子等因素会影响菌株的产酶能力从而影响酶的活性。SILCHENKO等[27]研究发现,Zn2+,Cu2+对岩藻聚糖硫酸酯酶活力有抑制作用,Ca2+对酶活力有促进作用。
霍利华[32]选择5种不同氮源发酵产酶,研究发现以硫酸铵为唯一氮源的菌株产生的岩藻聚糖硫酸酯酶活力为100%,高于蛋白胨89.5%。以不同浓度的岩藻聚糖硫酸酯为唯一碳源,研究发现溶液浓度越高,酶活性越强。同时,NaCl的浓度为2.5%左右时岩藻聚糖硫酸酯酶活性最强,其原因可能是海洋产酶菌的生长发酵的盐浓度在2.5%左右。且Mg2+对酶活力促进作用明显,Hg2+对酶活力抑制作用明显,Cu2+、Ca2+对酶活力抑制作用不明显。王莹[12]从昆布中筛选出能产生岩藻聚糖硫酸酯酶的菌株RC2,发现岩藻聚糖硫酸酯为该菌株的最佳碳源,最佳氮源为蛋白胨和牛肉膏,且氮源浓度为0.4%时酶活性最强;NaCl的浓度对岩藻聚糖硫酸酯酶活性有很大的影响,反应体系中无NaCl时酶活性几乎为零;金属离子同样对酶活性有一定的影响,Zn2+,K+,Ca2+对酶活力有促进作用,Cu2+对酶活力抑制作用明显。张袁等[23]从厦门海域的海水,海沙,藻类中分离得到降解岩藻聚糖硫酸酯的真菌菌株温特曲酶菌,该酶的最佳氮源为(NH4)2SO4。
目前研究中,通常采用岩藻聚糖硫酸酯作为唯一碳源来筛选含有岩藻聚糖硫酸酯降解酶的微生物,其浓度会对酶活力产生影响;其最佳氮源一般为蛋白胨、硫酸铵;培养条件中的NaCl对岩藻聚糖硫酸酯酶的活性影响较大。Cu2+一般对酶活力抑制作用明显,其他金属离子如Zn2+,K+,Ca2+等对不同岩藻聚糖硫酸酯酶活力的影响有所不同,可能是菌株的生长环境及生长特性不同所导致的。
3 岩藻聚糖硫酸酯酶的酶学性质
3.1 岩藻聚糖硫酸酯酶活性的测定方法
岩藻聚糖硫酸酯酶酶活性的测定方法并未统一,目前多用还原糖含量的测定间接测定酶的活性。DNS法和Nelson法是测定岩藻聚糖硫酸酯酶活性的常用方法。它们都是根据还原糖含量的变化来反映酶的活性,其中DNS法为常量测定方法,Nelson法为微量测定方法。DNS法更为简单方便,而Nelson法稳定性更佳。DNS法的原理为还原糖与DNS的氧化还原反应,生成棕红色的C7H6 N2O5,用比色法或吸光光度法测定糖的含量,最后用糖含量间接反映酶的活性,这种方法操作简单,测定岩藻聚糖硫酸酯酶的活性稳定性较好。SHUANG等[30]通过固态发酵法酵解菌株Fusarium sp.(LD8),该菌株能产生岩藻聚糖硫酸酯酶,并用DNS法测定酶的活性。将由1 mL底物溶液和0.1 mL酶溶液组成的混合物在50 ℃培养10 min,使用灭活的酶溶液作为空白,做标准曲线,以还原糖浓度表示酶的活性。王春霞[33]用DNS法测定岩藻聚糖硫酸酯酶的活性,并绘制DNS曲线,根据还原糖的量来表示酶的活性。
Nelson法的基本原理是还原糖的羰基,其本身易被氧化,且蔗糖酶可将蔗糖水解为葡萄糖和果糖。它是一种用铜试剂和砷酸盐试剂测定葡萄糖(或还原当量)的光度方法,显色的光密度(510 nm)与摄取的葡萄糖成正比,并能在长时间内保持稳定,从而确定蔗糖酶的活力。SILCHENKO等[27]从海洋软体动物 Lambis sp中纯化出岩藻聚糖硫酸酯酶,并用尼尔森法测定其活性。王莹等[34]从海水中筛选出l株高产菌株MD3,用Nelson法以每小时产生1 μmol/L的岩藻糖所需的酶量来表示岩藻聚糖硫酸酯酶的活性。
除此之外还有一些其他测定方法。张翠玉等[35]发现了测定岩藻聚糖硫酸酯酶活性的PHBH法,该方法为将PHBH溶液加入稀释后的酶溶液中,迅速冷却后测定吸光度。最后根据单位时间内产糖量计算酶的活性。该方法灵敏度及精密度优于DNS法,且操作简单,此方法是酶活性的测定的微量方法。DECAMPS等[15]将岩藻聚糖硫酸酯在20 mmol/L Tris-HCl缓冲液(pH 7.5)中与酶在室温下培养,将样品与含有蔗糖的缓冲溶液混合,用碳水化合物-聚丙烯酰胺凝胶电泳(carbohydrate-polyacrylamide gel electrophoresis,C-PAGE)法进行分析,通过在流动凝胶中出现的阴离子寡糖来检测岩藻聚糖硫酸酯酶的活性。
3.2 岩藻聚糖硫酸酯酶的最适pH值与温度
不同来源的岩藻聚糖硫酸酯酶的酶学特性不同,其最适pH及温度亦存在不同。
一部分岩藻聚糖硫酸酯酶为酸性酶。THANASSI等[16]从鲍鱼肝胰腺中纯化出的α-L-岩藻糖苷酶在pH 值为6.1时活性最强,其最佳温度约为38 ℃。KUSAYKIN等[29]从海洋软体动物滨螺中纯化岩藻多糖酶,岩藻聚糖硫酸酯酶的最适pH值为5.4,最适温度在35 ℃左右。姜琪等[36]用岩藻聚糖硫酸酯酶降解马尾藻岩藻聚糖的最适pH值为6,最适反应温度为50 ℃。
另一部分岩藻聚糖硫酸酯酶为中性或碱性酶。王春霞[33]从海参中筛选分离出一种能降解岩藻聚糖硫酸酯的海洋细菌,将其命名为HDj3。该岩藻聚糖硫酸酯酶的最适pH为7.5,最适温度为25 ℃。常耀光[28]用黄杆菌科细菌CZ1127筛选出的岩藻聚糖硫酸酯酶对海参岩藻聚糖硫酸酯进行降解处理,该酶的最适pH值为7,最适温度为28 ℃。这与王春霞的结论相似。SILCHENKO等[37]研究发现来自海洋细菌福尔摩沙藻类KMM 3553T的墨角藻聚糖酶在pH值6.5~9.1的范围内显示最大活性,最适温度为45 ℃。
表2 岩藻聚糖硫酸酯酶的最适pH与温度
Table 2 Optimal pH and temperature of fucoidan
degrading enzyme
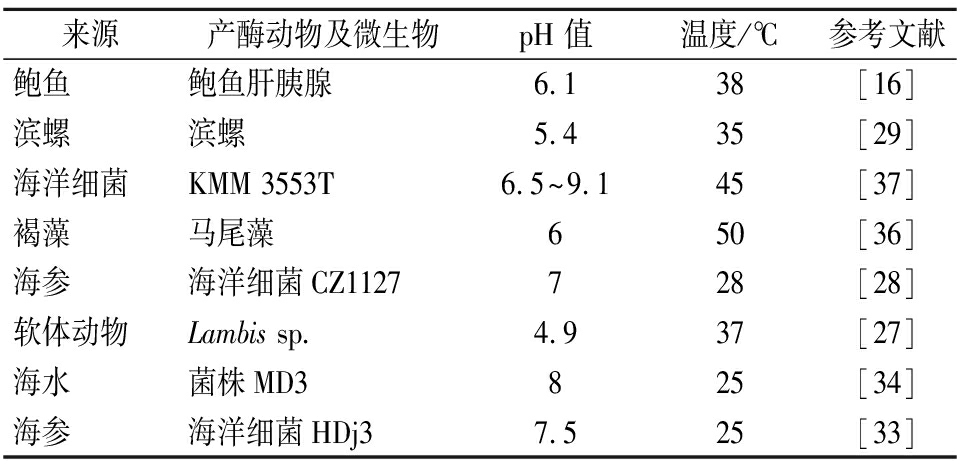
来源产酶动物及微生物pH 值温度/℃参考文献鲍鱼鲍鱼肝胰腺6.138[16]滨螺滨螺5.435[29]海洋细菌KMM 3553T6.5~9.145[37]褐藻马尾藻650[36]海参海洋细菌CZ1127728[28]软体动物Lambis sp.4.937[27]海水菌株MD3825[34]海参海洋细菌HDj37.525[33]
4 岩藻聚糖硫酸酯酶的基因鉴定及外源基因的表达
岩藻聚糖硫酸酯酶是目前岩藻聚糖降解研究的热点。目前已有多种岩藻聚糖硫酸酯酶的基因序列被测定。截止到2020年7月,CAZy数据库包含来自海洋细菌的岩藻聚糖硫酸酯酶的大约17个氨基酸序列的信息,其中4个存在于海洋细菌Wenyingzhuangia fucanilytica CZ1127T中。近几年,随着基因组学的不断完善,新的能降解岩藻聚糖硫酸酯的酶被发现并被测序。TAKAYAMA等[38]从海洋细菌Alteromonas sp.发现2种岩藻多糖酶,经过基因测序将其称为Fda1和Fda2。SAKAI等[31]从海洋细菌Fucobacter中发现胞外硫酸化岩藻聚糖甘露聚糖裂解酶FdlA和FdlB,作用于来自厚叶海带岩藻聚糖硫酸化甘露聚糖。通过序列分析,发现这种裂解酶活性显然由2个独立的编码区编码,有56%的氨基酸序列同一性。
为了解决海洋细菌产酶活力低,携带毒性因子等问题,人们将岩藻聚糖硫酸酯降解酶在大肠杆菌菌株中进行外源基因的表达,从而为岩藻聚糖硫酸酯酶的工业化生产奠定基础。COLIN等[39]从海洋细菌Mariniflexile岩藻聚糖酶SW5T中克隆了编码岩藻多糖酶的基因,并在大肠杆菌中产生了其蛋白,获得了重组酶FcnA。该酶被证明是内α-L-1→4-岩藻聚糖酶,它是糖苷水解酶家族(GH107)的第一种酶。Fda1和Fda2和重组酶FcnA同属GH107家族,Fda1和Fda2与FcnA的氨基酸序列相同性分别为24%和23%。SILCHENKO等[40]对来自海洋细菌 Formosa algae(KMM 3553T)的2种岩藻多糖酶FFA1和FFA2进行了鉴定,并在大肠杆菌中产生了该蛋白,序列分析显示岩藻聚糖酶FFA1属于GH107 家族。ZUEVA等[41]克隆了编码2种岩藻聚糖酶fwf1和fwf2的基因,并在大肠杆菌细胞中产生了FWf1和FWf2蛋白,对其生化特性进行了研究,发现FWf1和FWf2的氨基酸序列分别与Wenyingzhuangia fucanilytica CZ1127T有41%和51%的同一性,具有已建立的三维结构。
5 酶降解产物的结构
岩藻聚糖硫酸酯的结构显示出复杂性,通过岩藻聚糖硫酸酯酶降解能保持天然多糖的结构特征,岩藻聚糖硫酸酯酶的结构更易被人们了解。岩藻聚糖硫酸酯的结构为α-(1→2),α-(1→3)或α-(1→4)连接构成主链和较多的分支结构,主链通常被大量的硫酸盐、乙酸盐和各种单糖残基取代,产生极其复杂的多相分子混合物。硫酸基团主要结合在C4位上,其他单糖在聚合物糖链上随机连接。
岩藻多糖的化学组成和糖基连接方式多样,其结构复杂,而岩藻多糖降解酶可作用于特定的糖苷键,与红外光谱、核磁共振、气相质谱联用等方法联合使用是推断岩藻多糖结构的有效手段。不同来源的岩藻聚糖硫酸酯及酶解产物的结构一般不同。YU等[42]用酶解法制备了低分子质量梅花参岩藻聚糖硫酸酯,并用核磁共振进行了分析,发现该海参岩藻聚糖硫酸酯的基本结构为重复的四岩藻糖单元,且保留了岩藻聚糖硫酸酯的结构特征,结构为:[→3-α-L-Fucp-1→3-α-L-Fucp2,4(OSO3-)-1 →3-α-L-Fucp-1→3-α-L-Fucp2(OSO3-)-1→]n。CHEN等[43]和LI等[44]经降解得到的海参岩藻聚糖硫酸酯的基本结构也为四糖单元,这与YU等[42]得到的结论一致。SAKAI等[45]用从海洋细菌中获得的岩藻多糖酶降解褐藻岩藻聚糖硫酸酯,并通过核磁共振分析确定其结构,它的结构为:[-3L-Fucpα1-3L-Fucp(4-O-sulfate)α1-3L-Fucp(4-O-sulfate)α1-3(D-GlcpUAα1-2)L-Fucpα1]n-3L-Fucpα1-3L-Fucp(4-O-sulfate)α1-3L-Fucp(4-O-sulfate)α1-3L-Fucp(n=0,1,2,or 3)。COLIN等[39]用新型岩藻多糖酶FcnA降解鹿角藻岩藻聚糖硫酸酯,得到的低分子质量岩藻聚糖硫酸酯的基本结构为重复的二糖单元:[3)-α-L-Fucp-(2OSO3-)-1→4-α-L-Fucp-(2,3OSO3-)-(1→]n。SILCHENKO等[40]鉴定、克隆了海洋细菌F.algae中编码岩藻聚糖硫酸酯酶ffa1的基因,并在大肠杆菌中产生了蛋白(FFA1),重组岩藻聚糖酶FFA1用于降解马尾藻岩藻聚糖硫酸酯。通过核磁共振谱确定了聚合度为4~10的5种硫酸化寡糖的结构,基本结构为重复的三糖单元:[→3-α-L-Fucp(2SO3-)-1→4-α-L-Fucp(2,3SO3-)-1→3-α-L-Fucp(2,4SO3-)-1]n,具有(α-L-Fucp-1→2-α-L-Fucp-1→)结构的侧链结合在C4位上。CHEVOLOT等[46]研究发现泡叶藻岩藻聚糖硫酸酯寡糖的基本结构为重复的二糖单元:[→3)-α-L-Fuc(2SO3-)-(1→4)-α-L-Fuc(2,3 diSO3-)-(1]n。
综上所述,来源于棘皮动物的岩藻聚糖硫酸酯通常由重复的四糖单元组成,而来自于褐藻的岩藻聚糖硫酸酯多由重复的二或三糖单元组成。由于岩藻聚糖硫酸酯的结构十分复杂,很难通过常规的分析手段来确定其精细的化学结构。但使用岩藻聚糖硫酸酯酶降解后,通过降解得到的低分子质量岩藻聚糖硫酸酯的结构解析将有助于推测岩藻聚糖硫酸酯的结构。
表3 岩藻聚糖硫酸酯及酶降解产物的结构
Table 3 Structures offucoidan degrading enzyme and its degradation products

来源结构糖单元(n)参考文献海参[→3-α-L-Fucp-1→3-α-L-Fucp2,4(OSO3-)-1 →3-α-L-Fucp-1→3-α-L-Fucp2(OSO3-)-1→]n4[42]海参[→3-α-L-Fucp-1→3-α-L-Fucp2(OSO3-)-1→3-α-L-Fucp2(OSO3-)-1→3-α-L-Fucp2,4(OSO3-)-1→]n4[43]海参[→3Fuc(2S,4S)α1→3Fuc(2S)α1→3Fuc(2S)α1→3Fucα1→]n4[44]褐藻[-3L-Fucpα1-3L-Fucp(4-O-sulfate)α1-3L-Fucp(4-O-sulfate)α1-3(D-GlcpUAα1-2)L-Fucpα1]n3[45]马尾藻[→3-α-L-Fucp(2SO3-)-1→4-α-L-Fucp(2,3SO3-)-1→3-α-L-Fucp(2,4SO3-)-1]n3[40]泡叶藻[→3)-α-L-Fuc(2SO3-)-(1→4)-α-L-Fuc(2,3 diSO3-)-(1]n2[46]鹿角藻[3)-α-L-Fucp-(2OSO3-)-1→4-α-L-Fucp-(2,3OSO3-)-(1→]n2[39]
6 酶解产物的活性
研究发现,经酶解后得到的低分子质量岩藻聚糖硫酸酯较未处理的岩藻聚糖硫酸酯具有更强的生物活性,如抗氧化活性、抗肿瘤活性、抗黑色素生成能力及增强免疫活性。HIFNEY等[47]用发酵真菌中获得的岩藻聚糖硫酸酯酶对其进行处理,产生具有较低分子质量结构松散的岩藻聚糖硫酸酯寡糖,研究发现酶处理后的寡糖具有更优异的抗氧化能力。RASIN等[48]用重组岩藻聚糖酶FFA1水解岩藻聚糖硫酸酯,其较高分子质量部分用阴离子交换色谱法分级,得到3种不同分子质量(63~138 kDa)的低分子质量寡糖片段F1,F2,F3。研究发现经过酶处理后的F3组分对DLD-1细胞的集落形成表现出最有效的抗肿瘤和抗辐射作用。CHEN等[49]用从黄杆菌RC2-3纯化的酶降解岩藻聚糖硫酸酯,发现经酶处理后的分子质量在5~10 kDa之间的岩藻聚糖组分在B16细胞中表现出最佳的酪氨酸酶抑制活性、抗氧化活性和优异的抗黑素生成能力,可用作美白功能化妆品的开发。KUZNETSOVA等[50]比较了褐藻岩藻聚糖硫酸酯及其酶转化产物和低分子质量产物对人外周血多形核白细胞先天免疫细胞功能活性的影响,研究发现酶法生产的低分子质量产物比相应的天然高分子质量岩藻聚糖具有更好的体外先天免疫细胞功能活性。
7 展望
海洋大型藻类是低成本,安全的可再生资源,用于许多生物技术应用,如生物活性化合物和生物能源生产。大多数藻类中含有岩藻聚糖硫酸酯,而岩藻聚糖硫酸酯的高分子质量、不规则结构和黏度是提供用于可溶性和浓缩药物用途的均匀制剂的障碍。通过酶法降解处理,可得到分子质量较窄且均一性好的低分子质量岩藻聚糖硫酸酯。因此,如何获得高效降解岩藻聚糖硫酸酯的酶成为低分子质量岩藻聚糖硫酸酯生产的关键。
今后,人们对岩藻聚糖硫酸酯酶的研究还将集中在:找寻能产生高效岩藻聚糖硫酸酯酶的微生物,岩藻聚糖硫酸酯酶的作用方式与机理,岩藻聚糖硫酸酯酶的序列分析与重组技术研究等。并在此基础上,进一步深入研究酶法得到的岩藻聚糖硫酸酯寡糖的结构及生物活性作用等。随着基因测序的快速发展,多种岩藻聚糖硫酸酯酶已被鉴定,这为岩藻聚糖硫酸酯酶的工业化生产提供了可能,岩藻聚糖硫酸酯酶将具有广阔的应用前景。
[1] WANG S H,HUANG C Y,CHEN C Y,et al.Isolation and purification of brown algae fucoidan from Sargassum siliquosum and the analysis of anti-lipogenesis activity[J].Biochemical Engineering Journal,2021,165:107798.
[2] WANG Y,XING M C,CAO Q,et al.Biological activities of fucoidan and the factors mediating its therapeutic effects:A review of recent studies[J].Marine Drugs,2019,17(3):183.
[3] YIN J Y,YANG X Q,XIA B,et al.The fucoidan from sea cucumber Apostichopus japonicus attenuates lipopolysaccharide-challenged liver injury in C57BL/6 J mice[J].Journal of Functional Foods,2019,61:103493.
[4] PRADHAN B,PATRA S,NAYAK R,et al.Multifunctional role of fucoidan,sulfated polysaccharides in human health and disease:A journey under the sea in pursuit of potent therapeutic agents[J].International Journal of Biological Macromolecules,2020,164:4 263-4 278.
[5] GAO W J,GUO Y Y,WANG L Z,et al.Ameliorative and protective effects of fucoidan and sodium alginate against lead-induced oxidative stress in Sprague Dawley rats[J].International Journal of Biological Macromolecules,2020,158:662-669.
[6] MANSOUR M B,BALTI R,YACOUBI L,et al.Primary structure and anticoagulant activity of fucoidan from the sea cucumber Holothuria polii[J].International Journal of Biological Macromolecules,2019,121:1 145-1 153.
[7] CHUNG C H,LU K Y,LEE W C,et al.Fucoidan-based,tumor-activated nanoplatform for overcoming hypoxia and enhancing photodynamic therapy and antitumor immunity[J].Biomaterials,2020,257:120227.
[8] SANJEEWA K K A,JAYAWARDENA T U,KIM S Y,et al.Fucoidan isolated from invasive Sargassum horneri inhibit LPS-induced inflammation via blocking NF-κB and MAPK pathways[J].Algal Research,2019,41:101561.
[9] ZHAO X,GUO F,HU J,et al.Antithrombotic activity of oral administered low molecular weight fucoidan from Laminaria japonica[J].Thrombosis Research,2016,144:46-52.
[10] FL REZ-FERN
REZ-FERN NDEZ N,TORRES M D,GONZ
NDEZ N,TORRES M D,GONZ LEZ-MU
LEZ-MU OZ M J,et al.Potential of intensification techniques for the extraction and depolymerization of fucoidan[J].Algal Research,2018,30:128-148.
OZ M J,et al.Potential of intensification techniques for the extraction and depolymerization of fucoidan[J].Algal Research,2018,30:128-148.
[11] SILCHENKO A S,RASIN A B,KUSAYKIN M I,et al.Modification of native fucoidan from Fucus evanescens by recombinant fucoidanase from marine bacteria Formosa algae[J].Carbohydrate Polymers,2018,193:189-195.
[12] 王莹.岩藻聚糖硫酸酯酶产生菌的筛选,酶学性质研究及酶解产物抗氧化活性预测系统的建立[D].青岛:中国海洋大学,2013.
WANG Y.Screening of fucoindanase-producing bacteria,fucoindanase characterization,and establishment of the hydrolyzate antioxidant activity predication system[D].Qingdao:Ocean University of China.2013.
[13] BELIK A A,TABAKMAKHER K M,SILCHENKO A S,et al.Sulfated steroids of Halichondriidae family sponges-Natural inhibitors of polysaccharide-degrading enzymes of bacterium Formosa algae,inhabiting brown alga Fucus evanescens[J].Carbohydrate Research,2019,484:107776.
[14] VAN VLIET D M,PALAKAWONG NA AYUDTHAYA S,DIOP S,et al.Anaerobic degradation of sulfated polysaccharides by two novel Kiritimatiellales strains isolated from black sea sediment[J].Frontiers in Microbiology,2019,10:253.
[15] DESCAMPS V,COLIN S,LAHAYE M,et al.Isolation and culture of a marine bacterium degrading the sulfated fucans from marine brown algae[J].Marine Biotechnology,2006,8(1):27-39.
[16] THANASSI N M,NAKADA H I.Enzymic degradation of fucoidan by enzymes from the hepatopancreas of abalone,Haliotus species[J].Archives of Biochemistry and Biophysics,1967,118(1):172-177.
[17] TANAKA K,SORAI S.Hydrolysis of fucoidan by abalone liver alpha-L-fucosidase[J].FEBS Letters,1970,9(1):45-48.
[18] SICHERT A,CORZETT C H,SCHECHTER M S,et al.Verrucomicrobia use hundreds of enzymes to digest the algal polysaccharide fucoidan[J].Nature Microbiology,2020,5(8):1 026-1 039.
[19] KIM W J,KIM S M,LEE Y H,et al.Isolation and characterization of marine bacterial strain degrading fucoidan from Korean Undaria pinnatifida Sporophylls[J].Journal of Microbiology and Biotechnology,2008,18(4):616-623.
[20] KIM W J,PARK J W,PARK J K,et al.Purification and characterization of a fucoidanase(FNase S) from a marine bacterium Sphingomonas paucimobilis PF-1[J].Marine Drugs,2015,13(7):4 398-4 417.
[21] OHSHIRO T,OHMOTO Y,ONO Y,et al.Isolation and characterization of a novel fucoidan-degrading microorganism[J].Bioscience,Biotechnology,and Biochemistry,2010,74(8):1 729-1 732.
[22] IVANOVA E P,SAWABE T,ALEXEEVA Y V,et al. Pseudoalteromonas issachenkonii sp.nov.,a bacterium that degrades the thallus of the brown alga Fucus evanescens[J].International Journal of Systematic and Evolutionary Microbiology,2002,52(1):229-234.
[23] 张袁, 吴茜茜,王杏文,等.岩藻多糖酶产生菌的筛选,鉴定及发酵条件初探[J].包装与食品机械,2010,28(3):32-35;45.
ZAHNG Y,WU Q Q,WANG X W,et al.Screeniing,identification and fermentation of fucoidanase producing strains[J].Packaging and Food Machinery,2010,28(3):32-35;45.
[24] 吴克, 刘斌,吴茜茜,等.海洋真菌 Dendryphiella arenaria TM94 产岩藻多糖酶发酵及酶学性质[J].生物学杂志,2003,20(2):14-16.
WU K,LIU B,WU Q Q,et al.Fermentation of fucoidanase production by marine fungus Dedry arenaria and enzyme properties[J].Journal of Biology,2003,20(2):14-16.
[25] 吴茜茜, 吴克,张洁,等.海洋真菌 Fusarium sp.LD8 岩藻多糖酶的液态发酵条件研究[J].菌物学报,2006,25(1):77-82.
WU Q Q,WU K,ZHANG J,et al.Liguid state fermentation of fucoidanase from marine fungi Fusarium sp.LD8[J].Mycosystema,2006,25(1):77-82.
[26] RODR GUEZ-JASSO R M,MUSSATTO S
GUEZ-JASSO R M,MUSSATTO S ![]() L,et al.Fungal fucoidanase production by solid-state fermentation in a rotating drum bioreactor using algal biomass as substrate[J].Food and Bioproducts Processing,2013,91(4):587-594.
L,et al.Fungal fucoidanase production by solid-state fermentation in a rotating drum bioreactor using algal biomass as substrate[J].Food and Bioproducts Processing,2013,91(4):587-594.
[27] SILCHENKO A S,KUSAYKIN M I,ZAKHARENKO A M,et al.Endo-1,4-fucoidanase from Vietnamese marine mollusk Lambis sp.which producing sulphated fucooligosaccharides[J].Journal of Molecular Catalysis B:Enzymatic,2014,102:154-160.
[28] 常耀光. 海参岩藻聚糖硫酸酯及其酶解产物的制备,结构与活性研究[D].青岛:中国海洋大学,2010.
CHANG Y G.Study on the producing,sturcuture and activity of sea cucumber fucoidan and its enzymatic degradation products[D].Qingdao:Ocean University of China,2010.
[29] KUSAYKIN M I,BURTSEVA Y V,SVETASHEVA T G,et al.Distribution of O-glycosylhydrolases in marine invertebrates.Enzymes of the marine mollusk Littorina kurila that catalyze fucoidan transformation[J].Biochemistry(Moscow),2003,68(3):317-324.
[30] WU Q Q,MA S,XIAO H R,et al.Purification and the secondary structure of fucoidanase from Fusarium sp.LD8[J].Evidence-Based Complementary and Alternative Medicine,2011,2011:196190.
[31] SAKAI T,KIMURA H,KOJIMA K,et al.Marine bacterial sulfated fucoglucuronomannan(SFGM) lyase digests brown algal SFGM into trisaccharides[J].Marine Biotechnology,2003,5(1):70-78.
[32] 霍立华.岩藻聚糖硫酸酯酶产生菌筛选和酶学性质的研究以及岩藻寡糖的应用研究[D].青岛:中国海洋大学,2003.
HUO L H.Screening of fucoidanase sulfate producing bacteria and study on its enzyme characters and the application of fucoidan oligosaccharides[D].Qingdao:Ocean University of China,2003.
[33] 王春霞. 海参岩藻聚糖硫酸酯酶产生菌的筛选鉴定,发酵条件优化及酶学性质研究[D].青岛:中国海洋大学,2011.
WANG C X.Screening and identifying of a fucoidan-degrading marine bacterial strain, optimization of enzyme production condition, and characterization of the fucoidanase[D].Qingdao:Ocean University of China,2011.
[34] 王莹, 李八方,赵雪.产岩藻聚糖硫酸酯酶微生物的筛选及产酶条件优化[J].中国食品学报,2013,13(5):100-105.
WANG Y,LI B F,ZHAO X.Screening of fucoidanase-producing microorganisms and optimization of condition for fucoidanase production[J].Journal of Chinese Institute of Food Science and Technology,2013,13(5):100-105.
[35] 张翠玉, 薛长湖,于龙,等.基于 pHBH 法的岩藻聚糖硫酸酯酶酶活测定方法[J].中国食品学报,2013,13(7):200-206.
ZHANG C Y,XUE C H,YU L,et al.Fucoidanase activity determination method on basis of pHBH method[J].Journal of Chinese Institute of Food Science and Technology,2013,13(7):200-206.
[36] 姜琪, 谌素华,郑琼仪,等.可控酶降解马尾藻岩藻聚糖硫酸酯的研究[J].农产品加工(上),2015(7):5-7;11.
JING Q,CHEN S H,ZHENG Q Y,et al.Enzyme-catalyzed controllable degradation of Sargassum fucoidan[J].Farm Products Processing,2015(7):5-7;11.
[37] SILCHENKO A S,KUSAYKIN M I,KURILENKO V V,et al.Hydrolysis of fucoidan by fucoidanase isolated from the marine bacterium,Formosa algae[J].Marine Drugs,2013,11(7):2 413-2 430.
[38] TAKAYAMA M,KOYAMA N,SAKAI T,et al.Enzymes capable of degrading a sulfated-fucose-containing polysaccharide and their encoding genes:US6489155[P].2002-12-03.
[39] COLIN S,DENIAUD E,JAM M,et al.Cloning and biochemical characterization of the fucanase FcnA:Definition of a novel glycoside hydrolase family specific for sulfated fucans[J].Glycobiology,2006,16(11):1 021-1 032.
[40] SILCHENKO A S,RASIN A B,KUSAYKIN M I,et al.Structure,enzymatic transformation,anticancer activity of fucoidan and sulphated fucooligosaccharides from Sargassum horneri[J].Carbohydrate Polymers,2017,175:654-660.
[41] ZUEVA A O,SILCHENKO A S,RASIN A B,et al.Expression and biochemical characterization of two recombinant fucoidanases from the marine bacterium Wenyingzhuangia fucanilytica CZ1127T[J].International Journal of Biological Macromolecules,2020,164:3 025-3 037.
[42] YU L,GE L,XUE C H,et al.Structural study of fucoidan from sea cucumber Acaudina molpadioides:A fucoidan containing novel tetrafucose repeating unit[J].Food Chemistry,2014,142:197-200.
[43] CHEN S G,HU Y Q,YE X Q,et al.Sequence determination and anticoagulant and antithrombotic activities of a novel sulfated fucan isolated from the sea cucumber Isostichopus badionotus[J].Biochimica et Biophysica Acta(BBA)-General Subjects,2012,1820(7):989-1 000.
[44] LI C,NIU Q F,LI S J,et al.Fucoidan from sea cucumber Holothuria polii:Structural elucidation and stimulation of hematopoietic activity[J].International Journal of Biological Macromolecules,2020,154:1 123-1 131.
[45] SAKAI T,ISHIZUKA K,SHIMANAKA K,et al.Structures of oligosaccharides derived from Cladosiphon okamuranus fucoidan by digestion with marine bacterial enzymes[J].Marine Biotechnology,2003,5(6):536-544.
[46] CHEVOLOT L,MULLOY B,RATISKOL J,et al.A disaccharide repeat unit is the major structure in fucoidans from two species of brown algae[J].Carbohydrate Research,2001,330(4):529-535.
[47] HIFNEY A F,FAWZY M A,ABDEL-GAWAD K M,et al.Upgrading the antioxidant properties of fucoidan and alginate from Cystoseira trinodis by fungal fermentation or enzymatic pretreatment of the seaweed biomass[J].Food Chemistry,2018,269:387-395.
[48] RASIN A B,SILCHENKO A S,KUSAYKIN M I,et al.Enzymatic transformation and anti-tumor activity of Sargassum horneri fucoidan[J].Carbohydrate Polymers,2020,246:116635.
[49] CHEN Q R,KOU L Y,WANG F W,et al.Size-dependent whitening activity of enzyme-degraded fucoidan from Laminaria japonica[J].Carbohydrate Polymers,2019,225:115211.
[50] KUZNETSOVA T A,SMOLINA T P,BESEDNOVA N N,et al.Effect of sulfated polysaccharides from brown alga Fucus evanescens and their enzymatic transformation product on functional activity of innate immunity Cells[J].Antibiot Khimioter,2016,61(7-8):10-14.