清香型小曲白酒作为我国主要白酒种类之一,有着清香纯正、醇甜柔和、余味爽净的独特风味[1-2],备受人们喜爱。其以富含淀粉质的粮食为原料,以富含多种微生物的绿衣观音土曲为糖化发酵剂酿造而成。绿衣观音土曲以观音土、米粉、米糠为主要原料,接入曲种,通过加水拌料,制成坯,入箱进行微生物培育,开箱后转入培养室进行一至七烧共7轮培养,然后烘干制成[3]。霉菌作为重要的小曲白酒酿造微生物,在白酒发酵过程中发挥着重要作用,主要表现在能产生多种活性酶[4-5],如糖化酶、纤维素酶、蛋白酶、酯化酶、果胶酶等,对降解酿造原料和推动呈香呈味物质的形成具有重要贡献[6-8]。因此解析白酒发酵过程中霉菌种群结构,有助于对其中功能菌株的发酵特性进行研究[9-10]。
目前研究者对酱香型[11-12]、浓香型白酒[13-14]中的霉菌菌群研究较多。向玉萍等[11]从茅台酱香型大曲中分离鉴定出55株10个属的霉菌,并对不同种类大曲间霉菌种属差异性进行了分析;刘冰冰等[13]从浓香型白酒的酒醅中分离出15株霉菌,并对其产酶特性进行了研究。而对清香型小曲白酒中的霉菌研究多数集中于易于分离的几种,如根霉[15],对于难分离、难鉴定的霉菌鲜有报道,导致清香型小曲白酒中可以明确应用于酿造生产中的功能霉菌较少[16-17]。且同一种属的不同菌株间的生理生化特性存在一定差异,王旭亮等[18]从清香、浓香、酱香大曲中分离得到8株糖化酶活力较高的根霉,其与酵母模拟白酒酒精发酵结束后,各菌株培养基中的酒精含量和出酒率均不同。
近几年高通量测序技术被广泛应用于酿造微生物的检测中,该技术可以对样品中微生物信息进行解析,适合于微生物区系、群落结构、分布情况及生长变化规律研究,但一般只可鉴定到属,且无法获得菌株。经典分离培养鉴定技术可鉴定到种,并分离得到活菌株,有利于对获得的菌株进行进一步研究,适合于微生物生理生化特性和生产应用研究。
本研究使用经典分离培养鉴定法,通过优化培养基,从清香型小曲白酒酒曲和酒醅中分离出20株疑似霉菌,借助分子生物学技术进行了种属鉴定,并优化了适合霉菌分离、生长的培养基。对部分难以确定种属的霉菌,通过改变PCR扩增体系及扩增条件,成功进行了鉴定,并还原了不同发酵阶段的主要霉菌种类。同时,对霉菌产水解酶组成及酶活力进行了解析。为进一步提升清香型小曲原酒酿造品质提供了霉菌资源。
1 材料与方法
1.1 实验材料
1.1.1 实验样品及培养基
绿衣观音土曲、糖化醅和发酵醅为2019年10~12月由劲牌有限公司提供。
PDA培养基(g/L):马铃薯200,葡萄糖20,琼脂20,pH自然。
察氏培养基(g/L):NaNO3 3,K2HPO4 1,MgSO4·7H2O 0.5,KCl 0.5,FeSO4 0.01,蔗糖30,琼脂20,pH自然或7.0~7.2。
孟加拉红培养基(g/L):蛋白胨5,葡萄糖10,KH2PO4 1,MgSO4·7H2O 0.5,琼脂20,孟加拉红0.033。
改良PDA培养基[19](g/L):PDA培养基/L,牛肉膏2,酵母粉2,KH2PO4 2.5,吐温-20 1.5 mL。
YPD培养基(g/L):酵母膏10,蛋白胨20,葡萄糖20,琼脂20。
以上培养基在121 ℃灭菌20 min,冷却至50~60 ℃,加入质量分数为0.01%青霉素,摇匀倒平板。
1.1.2 实验材料
土壤基因组DNA提取试剂盒,天根生化科技(北京)有限公司。
1.1.3 主要仪器与设备
104C ECLIPSE E200显微镜,日本尼康有限公司;SF-CF-2A超净工作台,郑州南北仪器设备有限公司;WD-9413B凝胶成像系统,北京六一电泳仪器厂;2720 PCR仪,赛默飞世尔科技公司;HH-S恒温水浴锅,上海索谱仪器有限公司;Neofuge 18R高速冷冻离心机,上海Heal Force公司;721型分光光度计,尤尼柯上海仪器有限公司。
1.2 实验方法
1.2.1 取样方法
酒曲:每份曲样200 g左右,存放于4 ℃冰箱中。
糖化醅、酒醅:毛铺三分厂酿造三车间,跟踪同一批次1个糖化池、4个发酵槽车,共计取3批次。收集糖化0、6、12、18 h和终点的糖化醅,每种糖化醅收集1份,每份250 g;收集发酵0、1、2、3、4、5、7、9、11、14 d的酒醅,每天每个发酵槽车收集1份,每份250 g,样品存放于4 ℃。糖化池取样位置如图1所示,4.2 m×2.0 m的中格为取样格,每个取样格内有5个点,先取中心点作为糖化0 h的样品,余下4点为6、12、18 h和终点的样品。每个取样点收集中层的样品(约0.3 m深)。4个取样格即为4个平行。

图1 糖化池取样位置(俯视图)
Fig.1 Sampling location of the saccharification tank(top view)
发酵槽车取样位置如图2所示,实心点即为取样的实际位置。每个取样点收集中层的样品(约0.5~0.6 m深)。4个发酵槽车即为4个平行。
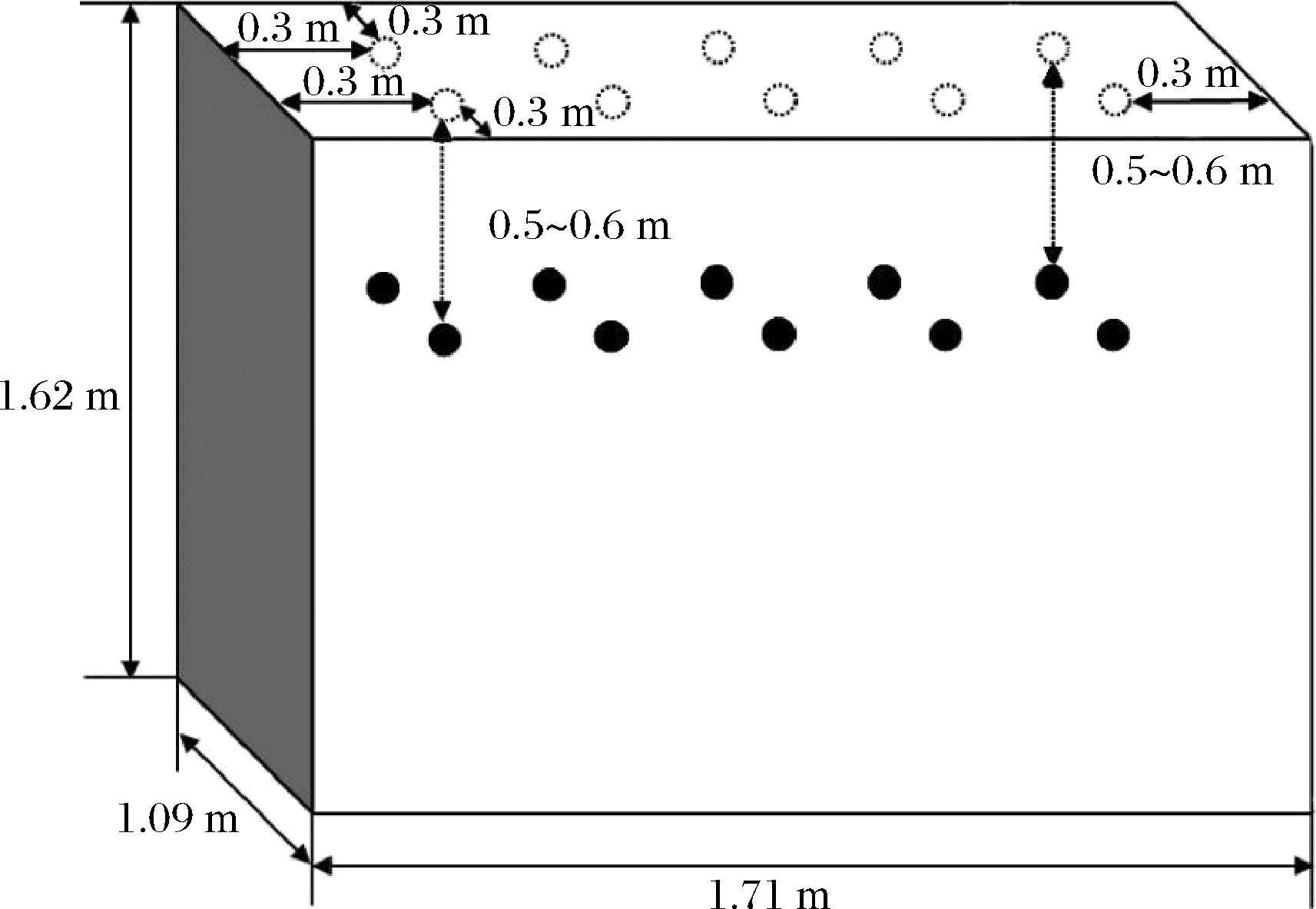
图2 发酵槽车取样位置(侧视图)
Fig.2 Sampling location of the fermentation tank car(side view)
1.2.2 真菌分离
准确称取样品5 g加入95 mL无菌水中,150 r/min振荡20~30 min,配制成菌悬液。分别取1 mL混合液至盛有9 mL灭菌水的试管中,依次制作成10-1、10-2、10-3、10-4梯度菌液,取200 μL的不同稀释液涂布于上述5种培养基中,放置于30 ℃培养,2~5 d后形态观察。
1.2.3 菌落形态观察
将在PDA上活化菌株,然后分别接种到孟加拉红、YPD、PDA培养基上,将平板倒置在30 ℃温箱中培养,观察第5天的菌落形态特征,主要记录其菌落形态、生长速度、气生菌丝疏密和产色素情况等,记录并拍照。
1.2.4 显微形态观察
取无菌盖玻片,以倾斜约45°方式插入PDA培养基中,每皿插入3~4片,30 ℃温箱中倒置培养5 d,取盖玻片在显微镜下观察包括菌丝颜色及形态、孢子形态在内的显微形态特征,记录并拍照[20]。
1.2.5 分子生物学鉴定
真菌模板的制备:使用土壤中微生物基因提取试剂盒提取法提取模板DNA。
PCR反应体系为30 μL:PremixTaq(EX Taq Version 2.0)15 μL,DNA模板2 μL,引物1 μL,采用真菌通用引物ITS1(5′-TCCGTAGGTGAACCTGCGG-3′)和ITS4(5′-TCCTCCGCTTATTGATATGC-3′),dd H2O 12 μL。
PCR扩增程序为:95 ℃ 5 min;95 ℃ 45 s,52 ℃ 45 s,72 ℃ 110 s,循环35次;72 ℃ 10 min,4 ℃保温。
琼脂糖凝胶电泳检测PCR产物,送往武汉华大基因科技股份有限公司完成测序。测序结果与美国国家生物信息技术中心(National Center of Biotechnology Information,NCBI)数据库中已知序列进行BLAST比对。采用基因序列分析软件DNAMAN和构建系统发育树软件MEGA 6,与GenBank数据库中相关属种基因序列进行系统发育分析。
1.2.6 酶活力测定方法
酸性蛋白酶测定参照国标GB 1886.174—2016《食品安全国家标准 食品添加剂 食品工业用酶制剂》的方法测定蛋白酶活力;糖化酶测定方法采用3,5-二硝基水杨酸(3,5-dinitrosalicylic acid,DNS)法[21];纤维素酶测定方法采用DNS法[22];果胶酶测定方法采用DNS法[23]。
2 结果与分析
2.1 霉菌的初筛结果分析
选用5种不同培养基对清香型小曲白酒酒曲和酒醅中的霉菌进行分离筛选,共计筛选得到46株菌落形态不同的疑似霉菌,5种培养基中观察到疑似霉菌的种类,统计结果如表1所示。
表1 不同培养基中筛选得到的疑似霉菌菌种数
Table 1 The number of suspected mold species screened
from different media

培养基疑似菌种数PDA17YPD9孟加拉红6改良PDA5察氏9总计46
对5种培养基中的疑似霉菌菌落观察发现,PDA、YPD和孟加拉红3种培养基中的菌落颜色较鲜艳,各菌落间形态差异较大,相同生长时间内菌落直径较大。而改良PDA培养基中加入盐类和吐温-20,其菌株生长速度较PDA培养基中慢,且菌丝蔓延的情况得到了很好的抑制,但菌株形态种类明显减少,菌丝变短且多聚成小絮状。察氏培养基通过加入高含量的盐,以抑制杂菌生长,但明显也影响霉菌的生长,培养3 d仍无明显菌落长出,至5 d有部分菌落长出,且菌落颜色较浅,菌丝短而稀疏。
故选择PDA、YPD和孟加拉红培养基,对46株疑似霉菌进行复筛。
2.2 霉菌复筛结果分析
根据同一种菌株在不同培养基中菌落形态不同的特点,将46株菌株分别点接到PDA、YPD和孟加拉红培养基上,平板倒置在30 ℃下培养观察5 d,根据3种培养基上的菌落形态、颜色和菌落大小等因素将46株进一步归属为20个种类,编号为M1~M20,结果如图3所示。

图3 二十种疑似霉菌菌株菌落形态
Fig.3 Colonial morphology of 20 suspected mold strains
注:从左至右依次为孟加拉红、PDA、YPD
初筛时20种菌株,在PDA中生长的有9种,在YPD中生长的有9种,在孟加拉红中生长的有3种,在改良PDA中生长的有2种,在察氏培养基中生长的有4种。仅在1种培养基中生长的菌株情况为:PDA上有3种,YPD上有4种,察氏培养基上有2种,结果如表2所示。
表2 不同培养基中筛选得到的疑似霉菌菌种数
Table 2 Comparison of the number of suspected mold
species obtained in different media

培养基初筛菌种数复筛菌种数仅在该培养基上生长菌种数PDA1793YPD994孟加拉红630改良PDA520察氏942
通过复筛可知PDA、YPD有利于霉菌的筛选,其中YPD中霉菌生长菌落较大,颜色鲜艳,复筛菌种数多,仅在该培养基上生长的菌种多,为较好的霉菌筛选培养基。察氏培养基虽然生长较慢,但复筛菌种数为4种,且有2种菌株仅在该培养基上生长,是很好的霉菌初筛培养基。故PDA、YPD和察氏培养基可作为霉菌良好的筛选培养基。
2.3 复筛菌株的显微形态观察结果分析
复筛得到的20株疑似霉菌,使用插片法观察菌株的显微形态,在PDA培养基中培养5 d后观察到的显微形态,结果如图4所示。

图4 二十株菌株的显微形态图
Fig.4 Microscopic morphologies of 20 strains
根据显微形态观察发现菌种M1和M15疑似根霉,M2疑似毛霉,M3和M5疑似毛白地霉,M4疑似酵母菌,M7、M9、M10、M16、M19疑似青霉,M8、M11~M14、M17、M18疑似曲霉,M20疑似为枝孢霉,M6为待定菌株。为科学确定菌株种属类别,对以上20株菌种进行分子生物学鉴定。
2.4 分子生物学鉴定结果分析
对复筛获得的20株疑似霉菌,提取菌株DNA为模板,并进行PCR扩增,经琼脂糖凝胶电泳检测PCR产物,除菌株M7和M16外,均有条带。将PCR产物验证成功的菌株,送往华大基因有限公司进行测序。结果显示,绝大部分峰图较理想,波峰与波谷清晰,峰与峰之间的距离均匀,底部基本未见杂峰。而M1、M4、M12号菌株底部套峰贯穿整个峰图,降低了BLAST比对结果的可靠性。
霉菌M7和M16 PCR扩增产物检测无条带,可能内转录间隔区(internal transcribed spacer,ITS)引物在该PCR反应体系及反应程序下,无法与其DNA模板结合进行扩增,可改变扩增引物;M1、M4、M12号菌株测序结果出现套峰,说明测序反应产物不纯,有不止一种测序反应产物存在,可提高退火温度,提高反应的特异性。
为详细鉴定微生物种属,了解微生物种群结构,对未成功鉴定的菌株M1、M4、M7、M12、M16进行扩增引物和退火温度的实验探索。选用18S rRNA引物[18S-P1(5′-CCAGGCTTTACACTTTATGC-3′)和18S-P2(5′-GCGATTAAGTTGGGTAACGC-3′)]和ITS引物;选用退火温度为56 ℃和52 ℃。通过设计3个实验方案,方案1:引物18S-P和退火温度56 ℃;方案2:引物18S-P和退火温度52 ℃;方案3:引物ITS和退火温度56 ℃。电泳结果如图5所示,选取方案1中的M1、M4、M12、M16菌株和方案2中的M7菌株的PCR扩增产物,送往华大基因有限公司进行测序,均测序成功。
将测序结果通过BioEdit软件进行剪切,并输入NCBI Gen Bank数据库进行同源性检索,获得近似序"列及分子生物学鉴定结果,综合分析形态学和分子生物学鉴定结果,如表3所示。
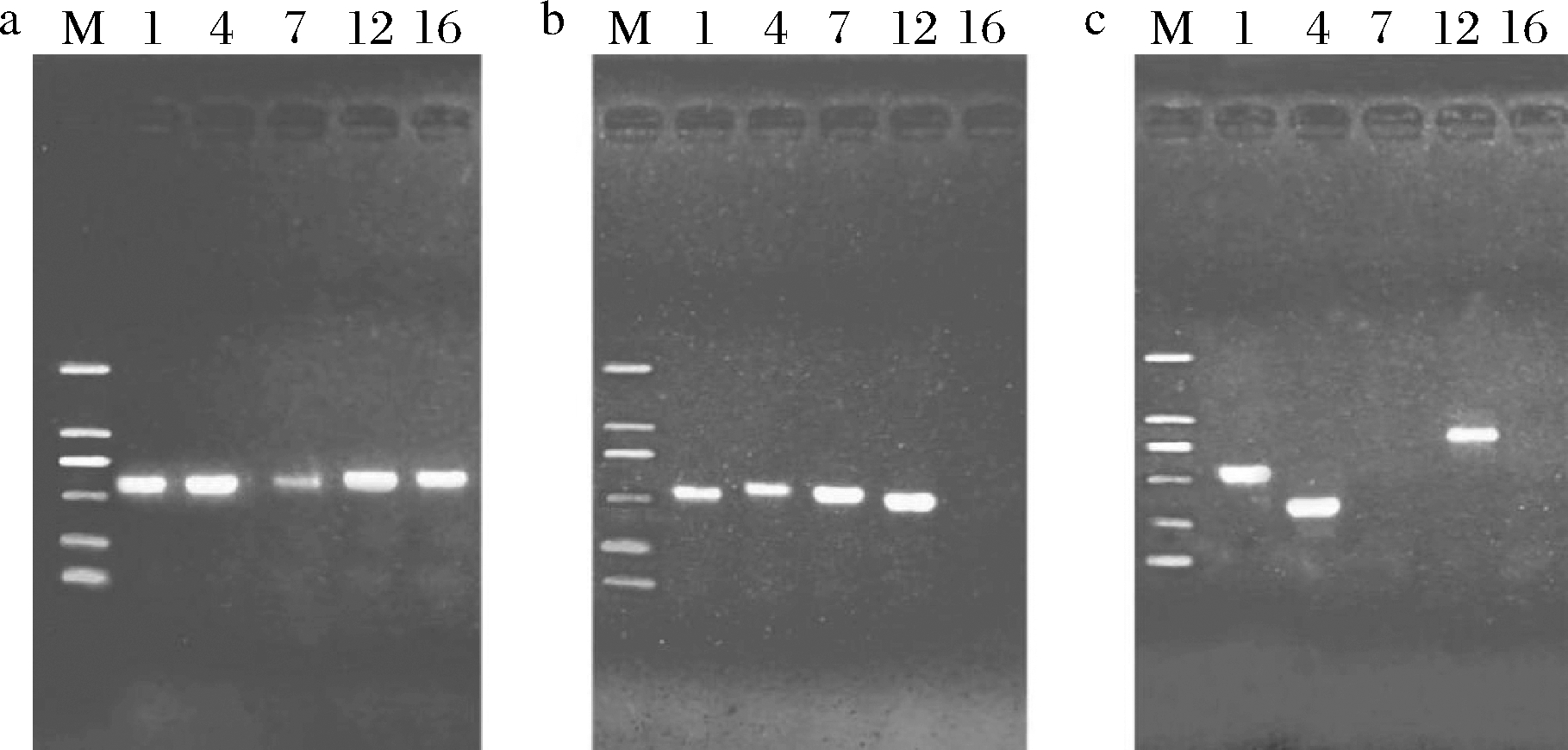
M-Marker; 1-M1菌株;4-M4菌株;7-M7菌株;
12-M12菌株;16-M16菌株
a-方案1;b-方案2;c-方案3
图5 菌株PCR产物验证电泳图
Fig.5 Electrophoregram of PCR product verification of strains
表3 酒曲和酒醅中分离霉菌鉴定结果
Table 3 Identification results of molds isolated from Xiaoqu and fermented grains
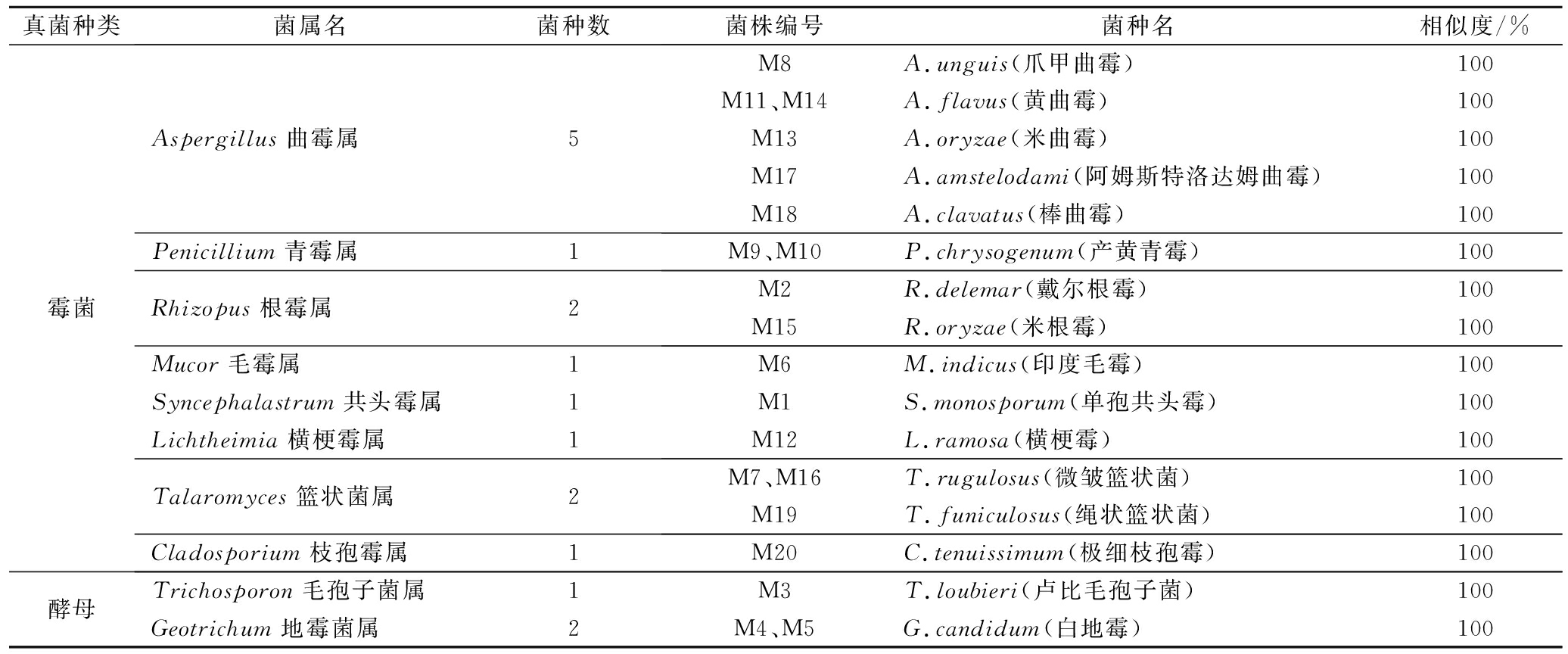
真菌种类菌属名菌种数菌株编号菌种名相似度/%霉菌Aspergillus曲霉属5M8A.unguis(爪甲曲霉)100M11、M14A.flavus(黄曲霉)100M13A.oryzae(米曲霉)100M17A.amstelodami(阿姆斯特洛达姆曲霉)100M18A.clavatus(棒曲霉)100Penicillium青霉属1M9、M10P.chrysogenum(产黄青霉)100Rhizopus根霉属2M2R.delemar(戴尔根霉)100M15R.oryzae(米根霉)100Mucor毛霉属1M6M.indicus(印度毛霉)100Syncephalastrum共头霉属1M1S.monosporum(单孢共头霉)100Lichtheimia横梗霉属1M12L.ramosa(横梗霉)100Talaromyces篮状菌属2M7、M16T.rugulosus(微皱篮状菌)100M19T.funiculosus(绳状篮状菌)100Cladosporium枝孢霉属1M20C.tenuissimum(极细枝孢霉)100酵母Trichosporon毛孢子菌属1M3T.loubieri(卢比毛孢子菌)100Geotrichum地霉菌属2M4、M5G.candidum(白地霉)100
通过形态学和分子生物学鉴定,20株菌中有3株为酵母,17株为霉菌,其中霉菌属于14个种8个属,分别是曲霉属、青霉属、根霉属、毛霉属、共头霉属、横梗霉属、篮状菌属、枝孢霉属。其中M3~M5菌株鉴定结果为酵母,但其菌落形态类似霉菌,菌落直径较大,有菌丝蔓延现象,显微形态中M3和M5菌株有菌丝结构,故在霉菌形态鉴定过程中,需注意毛孢子菌、白地霉等的鉴别。M6菌株显微形态较为特殊,但结果鉴定为印度毛霉菌。
对分离的20株菌,采用系统发育树软件MEGA6,与GenBank数据库中相关菌株基因序列进行系统发育分析,结果见图6。
从系统发育进化树看,青霉属与曲霉属聚在一起,根霉属、毛霉属、共头霉和横梗霉属聚在一起,这对菌株产酶性能的研究有较大指导意义。篮状菌属是青霉的有性型,M19篮状菌属与青霉属、曲霉属聚在一起,符合真菌分类的规则。
2.5 菌种来源
统计霉菌的样品来源,还原不同发酵阶段的主要霉菌种类(主要霉菌的数量为1×105~1×107 CFU/g),在酒曲中包括M9产黄青霉、M12横梗霉、M14黄曲霉、M15米根霉、M16微皱篮状菌、M18棒曲霉;在糖化过程中包括霉菌M2戴尔根霉、M6印度毛霉菌、M7微皱篮状菌、M8爪甲曲霉、M10产黄青霉、M12横梗霉、M15米根霉、M16微皱篮状菌;在发酵初期(0~3 d)存在M1单孢共头霉、M11黄曲霉、M13米曲霉、M14黄曲霉;在发酵前期(4~5 d)M14黄曲霉、M17阿姆斯特洛达姆曲霉、M18棒曲霉;发酵6 d后未筛选出霉菌。因本实验是通过培养组学技术分离得到各个阶段的霉菌种类,该结果可以表示为在该阶段较为优势霉菌种类或该阶段易于培养的霉菌种类。糖化阶段的霉菌主要来自于酒曲中,由于开放式的发酵环境,M6印度毛霉菌、M7微皱篮状菌、M8爪甲曲霉可能来自于环境中,而部分霉菌未在糖化阶段筛选出来,可能还未形成较优势霉菌种类,如:M14黄曲霉和M18棒曲霉;在发酵过程中的优势菌种主要为曲霉属,多个曲霉属菌种在不同的发酵阶段生长成较优势霉菌菌种,而M1单孢共头霉、M17阿姆斯特洛达姆曲霉可能由酒糟和环境中带入。发酵6~14 d未筛选出霉菌,说明在发酵第6天后,霉菌存在较少甚至没有,可能是由于随着发酵深入,酒醅中酸类物质的积累,如乙酸、乳酸等,抑制了霉菌的生长[24]。
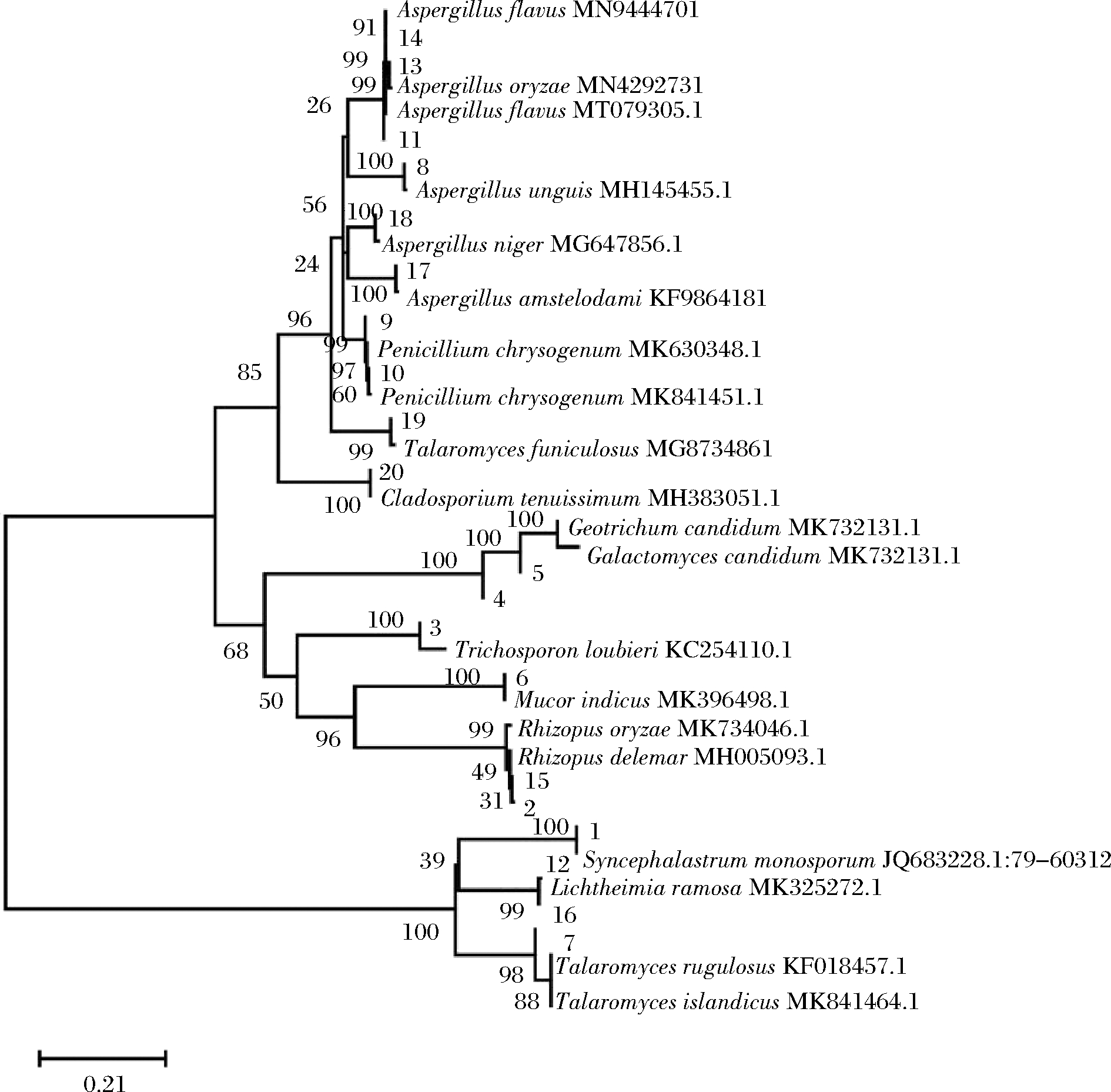
图6 系统发育进化树
Fig.6 Phylogenetic tree based on 18S rRNA and ITS sequences
2.6 霉菌酶活力测定
以分离得到的17株霉菌为实验材料,制备成麸皮种,测定其蛋白酶、糖化酶、纤维素酶、果胶酶等酶活力大小,结果如表4所示。
测定霉菌的水解酶活力,有助于了解分析原料的分解利用情况。在糖化酶上,夏艳秋等[25]在黄酒中筛选出的黄曲霉高产糖化酶活力在16 196~18 352 U/(g·h),本研究中M11号黄曲霉糖化酶活力高达18 156.45 U/(g·h),其余根霉属和曲霉属的酶活力普遍较高,但爪甲曲霉和阿姆斯特洛达姆曲霉除外,极细枝孢霉的糖化酶活力也较高[>10 000 U/(g·h)]。在清香型小曲白酒酿造过程中通常以米根霉作为糖化发酵剂,而实验发现黄曲霉及米曲霉表现出较高的糖化酶活力。霉菌菌株也是清香型小曲白酒酿造过程中蛋白酶的主要来源。印丽等[26]检测茅台镇酱香型白酒核心产区大曲中分离霉菌的酸性蛋白酶活力在12.76~69.75 U/g,本研究中单孢共头霉、横梗霉和微皱蓝状菌的酸性蛋白酶活力较高(>80 U/g),其中横梗霉在酒曲中数量较多,菌丝蔓延生长较快,有利于快速分解原料中的蛋白质等物质。在纤维素酶上,检测的霉菌均能产纤维素酶,但酶活力均较低,多数霉菌产纤维素酶活力0.6~11.09 U/g。在产果胶酶上,仅部分霉菌产果胶酶,且酶活力均不高,仅戴尔根霉、印度毛霉菌的果胶酶酶活力较高(>36 U/g)。
表4 霉菌麸皮种的水解酶系
Table 4 The hydrolytic enzymes in bran seed of mold
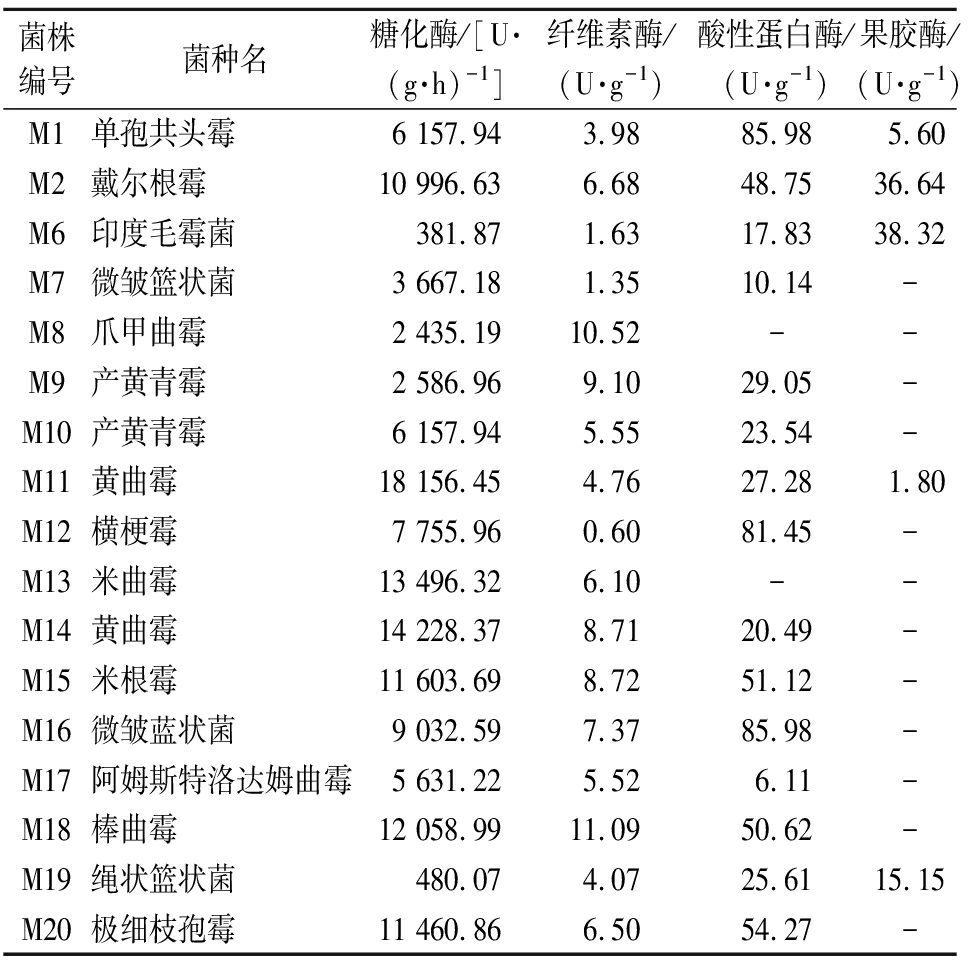
菌株编号菌种名糖化酶/[U·(g·h)-1]纤维素酶/(U·g-1)酸性蛋白酶/(U·g-1)果胶酶/(U·g-1)M1单孢共头霉6 157.943.9885.985.60M2戴尔根霉10 996.636.6848.7536.64M6印度毛霉菌381.871.6317.8338.32M7微皱篮状菌3 667.181.3510.14-M8爪甲曲霉2 435.1910.52--M9产黄青霉2 586.969.1029.05-M10产黄青霉6 157.945.5523.54-M11黄曲霉18 156.454.7627.281.80M12横梗霉7 755.960.6081.45-M13米曲霉13 496.326.10--M14黄曲霉14 228.378.7120.49-M15米根霉11 603.698.7251.12-M16微皱蓝状菌9 032.597.3785.98-M17阿姆斯特洛达姆曲霉5 631.225.526.11-M18棒曲霉12 058.9911.0950.62-M19绳状篮状菌480.074.0725.6115.15M20极细枝孢霉11 460.866.5054.27-
注:-未检出
3 结论
为了解清香型小曲白酒酿造的主要霉菌种类及其主要特性和作用,本研究采用PDA、YPD、孟加拉红、改良PDA和察氏培养基对酒曲和酒醅中的霉菌进行分离筛选,共计筛选得到46种菌落形态不同的疑似霉菌。进一步选择PDA、YPD和孟加拉红3种培养基对46株菌株进行复筛,根据在3种培养基上的菌落形态、颜色、菌落大小等因素将46株归类为20株疑似霉菌,结合形态学及分子生物学分析,20株疑似霉菌为3株酵母,17株霉菌,其中霉菌归为14个种8个属,分别是曲霉属、青霉属、根霉属、毛霉属、共头霉属、横梗霉属、篮状菌属、枝孢霉属。还原不同发酵阶段中的主要霉菌种类,糖化阶段的霉菌主要来自于酒曲中,部分霉菌可能来自于环境中;在发酵过程中的优势菌种主要为曲霉属,多个曲霉属菌种在不同的发酵阶段生长成较优势霉菌菌种,少量霉菌可能由酒糟和环境中带入。另外,对霉菌产水解酶组成及酶活力进行了解析,17株霉菌均能产生糖化酶和纤维素酶,仅部分霉菌可产生蛋白酶和果胶酶,且多数霉菌糖化酶和蛋白酶活力较高,纤维素酶和果胶酶的酶活力整体偏低。说明清香型小曲白酒生产中的霉菌类群产酶特性多样,酶活力差异较大。本研究为后续提升清香型小曲白酒的发酵质量,提供了霉菌资源。
[1] TANG Q X,HE G Q,HUANG J,et al.Characterizing relationship of microbial diversity and metabolite in Sichuan Xiaoqu[J].Frontiers in Microbiology,2019,10:696.
[2] SU M Y,TZENG W S,SHYU Y T.An analysis of feasibility of bioethanol production from Taiwan sorghum liquor waste[J].Bioresource Technology,2010,101(17):6 669-6 675.
[3] 唐洁,陈申习,张磊,等.绿衣观音土曲培养过程中微生物及酶系的动态变化[J].中国酿造,2020,39(5):97-104.
TANG J,CHEN S X,ZHANG L,et al.Dynamic changes of microorganisms and enzymes in the cultivation of Green-covering Guanyin Tuqu[J].China Brewing,2020,39(5):97-104.
[4] JIN G Y,ZHU Y,XU Y.Mystery behind Chinese liquor fermentation[J].Trends in Food Science & Technology,2017,63:18-28.
[5] ZHENG X W,HAN B Z.Baijiu(白酒),Chinese liquor:History,classification and manufacture[J].Journal of Ethnic Foods,2016,3(1):19-25.
[6] XIAO C,YANG Y,LU Z M,et al.Daqu microbiota exhibits species-specific and periodic succession features in Chinese baijiu fermentation process[J].Food Microbiology,2021,98:103766.
[7] LI X R,MA E B,YAN L Z,et al.Bacterial and fungal diversity in the starter production process of Fen liquor,a traditional Chinese liquor[J].Journal of Microbiology,2013,51(4):430-438.
[8] 李兵,张超,王玉霞,等.白酒大曲功能微生物与酶系研究进展[J].中国酿造,2019,38(6):7-12.
LI B,ZHANG C,WANG Y X,et al.Research progress on functional microbes and enzymes in Daqu of Baijiu[J].China Brewing,2019,38(6):7-12.
[9] WU H C,ZHANG S Y,MA Y Y,et al.Comparison of microbial communities in the fermentation starter used to brew Xiaoqu liquor[J].Journal of the Institute of Brewing,2017,123(1):113-120.
[10] 雷振河.采用高通量测序技术分析清香型白酒酿造微生物[J].食品与发酵工业,2015,41(9):164-167.
LEI Z H.Primary analysis on the fermented microorganism of Fen-flavor liquor by high-throughput sequencing[J].Food and Fermentation Industries,2015,41(9):164-167.
[11] 向玉萍,邱树毅,曹文涛,等.酱香型白酒核心产区大曲中霉菌的分离及鉴定[J].食品与发酵科技,2021,57(2):56-65.
XIANG Y P,QIU S Y,CAO W T,et al.Isolation and identification of molds in Daqu of the core producing area of Maotai-flavor liquor[J].Food and Fermentation Sciences & Technology,2021,57(2):56-65.
[12] 朱治宇.茅台镇酱香型白酒酿造区域霉菌多样性特征研究[D].贵阳:贵州大学,2020.
ZHU Z Y.Study on the characteristics of mold diversity in Maotai town sauce-flavor liquor brewing area[D].Guiyang:Guizhou University,2020.
[13] 刘冰冰,刘正宇,宋豪锋,等.宋河浓香型白酒酒醅纯培养霉菌群落的组成及功能酶筛选[J].现代食品科技,2019,35(12):197-207;156.
LIU B B,LIU Z Y,SONG H F,et al.Composition and functional enzymes of the pure-culture mold community from fermented grains of Songhe Nongxiang liquor[J].Modern Food Science and Technology,2019,35(12):197-207;156.
[14] 张超,田鹏,徐秋硕,等.浓香型白酒生态洞酿糟醅中微生物的变化研究[J].酿酒科技,2019(9):65-67;70.
ZHANG C,TIAN P,XU Q S,et al.Microbial change in fermented grains of Nongxiang Baijiu in cave fermentation[J].Liquor-Making Science & Technology,2019(9):65-67;70.
[15] FAN G S,SUN B G,XU D,et al.Isolation and identification of high-yield ethyl acetate-producing yeast from Gujinggong Daqu and its fermentation characteristics[J].Journal of the American Society of Brewing Chemists,2018,76(2):117-124.
[16] 李锐利,方尚玲,陈茂彬,等.绿衣观音土曲中霉菌糖化酶活力的研究[J].酿酒,2010,37(1):50-52.
LI R L,FANG S L,CHEN M B,et al.Research of glucoamylase activity of molds in green-covering Guanyin Taqu[J].Liquor Making,2010,37(1):50-52.
[17] 陈申习,唐洁,张龙,等.清香型小曲白酒机械化生产中微生物动态变化研究[J].中国酿造,2018,37(6):68-72.
CHEN S X,TANG J,ZHANG L,et al.Dynamical variation of microbe in mechanization process of Fen-flavor Xiaoqu Baijiu[J].China Brewing,2018,37(6):68-72.
[18] 王旭亮,王异静,王德良,等.白酒发酵高糖化性能霉菌的筛选及鉴定[J].酿酒科技,2012(9):22-28.
WANG X L,WANG Y J,WANG D L,et al.Screening and identification of mold strains with high saccharifying properties for improving ethanol fermentation efficiency in liquor production[J].Liquor-Making Science & Technology,2012(9):22-28.
[19] 颜松. PDA培养基的改良在霉菌计数中的应用研究[J].农产品加工(学刊),2013(13):18-20.
YAN S.Application and research of the improved PDA medium on the enumeration of moulds[J].Academic Periodical of Farm Products Processing,2013(13):18-20.
[20] 朱丽萍,陈福生,杨强,等.ISSR标记技术辅助形态学的红曲菌株分类鉴定[J].中国酿造,2020,39(8):175-181.
ZHU L P,CHEN F S,YANG Q,et al.Classification and identification of Monascus strains by ISSR marker-assisted morphology[J].China Brewing,2020,39(8):175-181.
[21] 郝萍萍,吴发萍,张文学,等.三种米曲霉糖化酶活及其红薯酒质量指标分析[J].中国酿造,2012,31(5):35-37.
HAO P P,WU F P,ZHANG W X,et al.Determination of saccharifying enzyme activity of three Aspergillus oryzae and quality analysis of sweet potato Shochu[J].China Brewing,2012,31(5):35-37.
[22] 何颂捷,左勇,张鑫,等.降解白酒酒糟中纤维素的细菌的分离鉴定[J].食品与发酵工业,2019,45(24):64-70.
HE S J,ZUO Y,ZHANG X,et al.Isolation and identification of bacteria degrading cellulose in distillers’ grains[J].Food and Fermentation Industries,2019,45(24):64-70.
[23] 徐鹏,王大红,陈亚欣,等.一株大麻脱胶菌株的分离鉴定及其产果胶酶发酵培养基的优化[J].中国麻业科学,2019,41(3):122-129.
XU P,WANG D H,CHEN Y X,et al.Isolation and identification of a hemp degumming strain and the optimization of fermentation medium for pectinase production[J].Plant Fiber Sciences in China,2019,41(3):122-129.
[24] 关凯乐,韩培杰,周森,等.低温大曲酵母菌分离和计数培养方法优化[J].菌物学报,2019,38(7):1 191-1 201.
GUAN K L,HAN P J,ZHOU S,et al.Development of mold-inhibiting methods for selective isolation and counting of yeasts from low-temperature liquor starter “Daqu”[J].Mycosystema,2019,38(7):1 191-1 201.
[25] 夏艳秋,朱强,汪志君.高产糖化酶黄曲霉菌的选育及初步应用[J].微生物学通报,2009,36(10):1 542-1 546.
XIA Y Q,ZHU Q,WANG Z J.Breeding of high-glucoamylase activity Aspergilius flavus and application[J].Microbiology 2009,36(10):1 542-1 546.
[26] 印丽,邱树毅,曹文涛,等.酱香型白酒核心产区大曲的酶系分析[J].现代食品科技,2021,37(3):89-96.
YIN L,QIU S Y,CAO W T,et al.Analysis of Daqu enzymes from the core production area of Maotai-flavor liquor[J].Modern Food Science and Technology,2021,37(3):89-96.