红曲色素是由红曲菌(也称红曲霉)(Monascus spp.)合成的一类次级代谢产物[1-2],作为天然食用色素,在我国以及东南亚地区使用至今已有1 000多年历史[3]。近年来,红曲色素以其着色自然、健康无毒以及色调温和等特点,成为国内销量较高的天然食用色素之一,并出口到欧美等发达国家[4]。
目前红曲色素生产菌株的产率仍不能完全满足工业生产需要。因此,提高红曲菌产率仍是本领域中的核心课题[5-6]。前期研究发现,萘醌化合物能够促进红曲菌合成红曲色素[7],如果能够找到红曲菌中响应萘醌化合物的基因,将有可能利用萘醌化合物提高红曲色素产率。然而,目前红曲菌的萘醌响应基因仍未见报道[7]。
随着转录组学与人工智能的发展,已有报道可以借助这2种工具来挖掘目标基因[8-9]。人工智能是一种利用不同计算机算法来进行数据学习,得到描述生物等过程模型的预测方法[10]。近年来发展起来的人工神经网络(artificial neural network,ANN)模型,能够较为精确地描绘微生物中基因与表型之间的内在关系,用于挖掘参与特定生物过程的关键基因[8-10]。
本研究首先利用转录组方法初步筛选了M.ruber M7菌株中参与萘醌响应的基因,然后选择并优化得到ANN模型,借此预测出2个可能性最高的萘醌响应基因,利用基因敲除菌株验证mga2基因是萘醌响应基因,以期利用白花丹醌诱导mga2基因过表达菌株获得较高的红曲色素产率。
1 材料与方法
1.1 材料与试剂
Monascus ruber M7(CCAM 070120,Culture Collection of State Key Laboratory of Agricultural Microbiology)为本实验保存。红曲菌基因敲除菌株ΔlaeA、ΔmrpigB和Δmga2,以及mga2基因过表达菌株M7::PtrpC-mga2为本课题组前期构建并保存。萘醌类化合物1,4-萘醌、甲萘醌、1,2-萘醌、白花丹醌、5-羟基-1,4-萘醌与拉帕醇,上海阿拉丁试剂有限公司。
1.2 仪器与设备
UV-1700型分光光度计,日本Shimadzu公司;LC-20AT高效液相色谱仪,日本岛津公司。
1.3 实验方法
1.3.1 M.ruber M7的生物量测定
将M.ruber M7接种于马铃薯葡萄糖琼脂(potato dextrose agar,PDA)斜面培养基上,在28 ℃条件下培养10 d,用无菌水洗涤斜面菌丝上的孢子,得到5×105 孢子/mL的孢子液。将5 mL孢子液接种到50 mL马铃薯葡萄糖肉汤(potato dextrose broth,PDB)培养基中,在28 ℃,120 r/min的条件下培养。发酵第12天时,离心(5 000 r/min)分离菌丝体和发酵液,冷冻干燥后称量菌丝体干质量。
1.3.2 萘醌化合物诱导发酵实验
将萘醌化合物预先溶解于二甲基亚砜(dimethylsulfoxide,DMSO)。在液态发酵第5天时,将萘醌溶液加入发酵液中,继续培养。
1.3.3 红曲色素的测定
取冷冻干燥的菌丝体0.1 g,研磨成细粉后加入5 mL甲醇溶液(体积分数为80%),于60 ℃条件下提取胞内红曲色素1 h,12 000 r/min离心5 min,弃去沉淀收集上清液。上清液用甲醇溶液(体积分数为80%)稀释到适当倍数,分别测定上清液在380、470、520 nm处的吸光度值,分别计算菌丝体中黄、橙、红色素的色价,总色价为三者之和[11]。色价计算如公式(1)所示:
色价=A×总稀释倍数
(1)
式中:A为特定波长的吸光度值。
1.3.4 红曲菌总RNA的提取和转录组测序
选取受萘醌化合物诱导的M.ruber M7为样本,以未诱导的M.ruber M7为空白对照。采用Trizol法提取M.ruber M7的总RNA,检测RNA的OD260/OD280在1.8~2.2,确保总RNA质量,然后利用Agilent 2100生物分析仪验证RNA样品的完整性。由微浪生物科技有限公司进行cDNA文库的构建及高通量测序,得到转录组数据。
基因mga2的相对转录水平按前期所建立的实时荧光定量PCR(quantitative real-time PCR,qRTPCR)方法测定[7]。
1.3.5 转录组分析与萘醌响应基因预测
首先根据reads长度、paired-end关系、质量值等,采用FPKM法估算基因表达水平,然后对read count进行标准化,再进行假设检验概率计算,最后进行多重假设检验校正。对不同萘醌类化合物处理的红曲菌样品,分析各样品中的转录组数据,当同一个基因的表达量在不同样品间显示出差异时,如果其假阳性率<0.05,且差异倍数>3,则认为该基因在不同样品中具有显著差异表达。
1.3.6 人工智能预测萘醌响应基因
将萘醌响应基因及其对应样本的红曲色素产量信息,分别定义为输入变量。采用Auto-sklearn软件[12],建立线性模型(linear models)、偏最小二乘回归(partial least squares regression model,PLSR)、弹性网络模型(elastic net model)、随机森林模型(random forest model)、ANN模型这5种预测模型[13]。
为了提高ANN的预测精度并降低计算量,通过筛选激活函数和优化器对ANN进行优化,并计算和校验ANN的预测准确率[14]。利用优化后的ANN预测得到萘醌响应基因,如果这些基因的贡献率>95%,则认为这些基因即为萘醌响应基因。
2 结果与分析
2.1 萘醌化合物提高红曲色素产率
选择6种萘醌化合物,以不同浓度(10、30或50 μmol/L)分别添加到培养5 d的M.ruber M7培养物中。1,4-萘醌、甲萘醌、白花丹醌和拉帕醇这4种萘醌能够显著提高红曲色素产率。其中,30 μmol/L的白花丹醌效果最明显,红曲色素产率从1 131 U/mg增加到1 436 U/mg(图1)。HU等[15]也发现萘醌化合物在低浓度下能促进M.ruber M7合成红曲色素。结果证明萘醌化合物能够促进红曲色素合成,也说明红曲菌中可能存在萘醌响应基因。
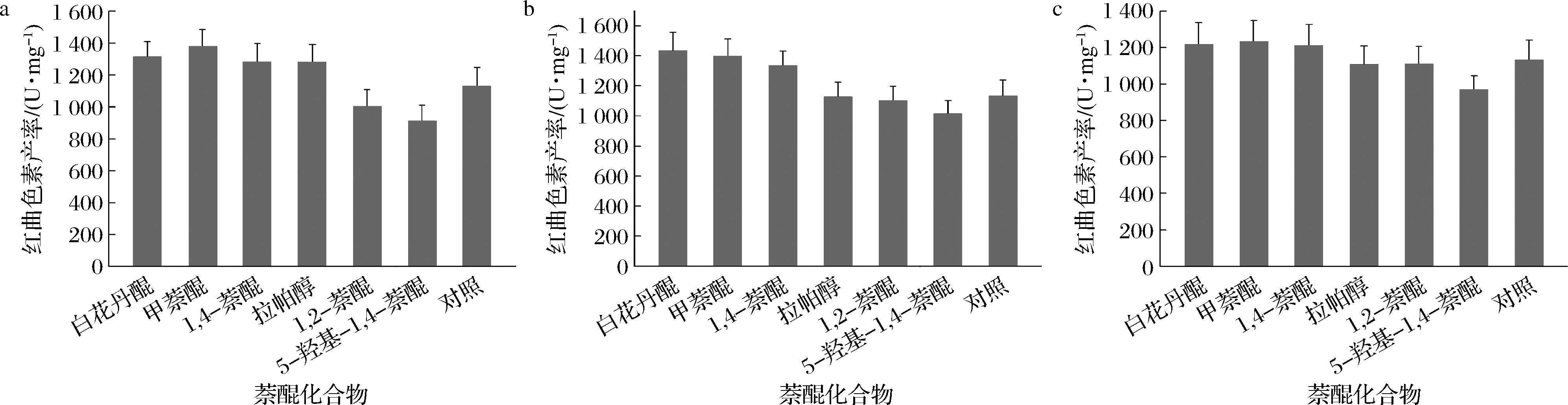
a-10 μmol/L萘醌化合物;b-30 μmol/L萘醌化合物;c-50 μmol/L萘醌化合物
图1 添加外源萘醌化合物提高M.ruber M7的红曲色素产率
Fig.1 Improvement of Monascus pigments yield by exogenous naphthoquinone
2.2 转录组学与人工智能模型联用预测萘醌响应基因
分别测定了1,4-萘醌、甲萘醌、白花丹醌和拉帕醇在不同浓度条件(10、30或50 μmol/L)处理M.ruber M7的转录组数据和红曲色素产率数据。采用PLS-DA对基因转录表达数据进行分析,共得到20个差异基因(表1)。其中,17个基因涉及细胞内的信号传导途径,其余3个基因分别涉及萘醌降解途径[16]、麦角固醇合成途径[17]以及红曲色素合成途径[18]。
表1 转录组学方法预测的萘醌响应基因
Table 1 Naphthoquinone-responsive genes predicted by transcriptomics analysis

编号潜在的萘醌响应基因相关细胞通路基因表达水平样品1LaeA全局调控↑a,b,c,d 2LaeB全局调控↑a,b,c,d3VeA全局调控↑a,b,d4HapB全局调控↓a,b,c,d5G protein-coupled receptorG蛋白信号通路↑a,b,d6G protein α subunit 1(mga1)G蛋白信号通路↑a,b,d7G protein α subunit 2(mga2)G蛋白信号通路↑a,b,c,d8DCP1ARNA降解↓a,b,d9RhlBRNA 降解↓a,b,c,d10Sphinganine kinase肌醇磷酸代谢通路↓b,c,d11Dephospho-CoA kinase肌醇磷酸代谢通路↓a,b,c,d12Phosphatidate phosphatase磷脂酰肌醇信号通路↓b,c,d13Protein kinase磷脂酰肌醇信号通路↓a,b,d,14Protein kinase A MAPK信号途径↑a,c,d,15MAP kinase kinase 1MAPK信号途径↑a,b,c,d16MAP/ERK kinase kinaseMAPK 信号途径↑a,c,d,17Histone deacetylase表观遗传↓a,c,d,18MrPigB红曲色素合成途径↑a,b,c,d19ArtR麦角固醇合成途径调控↓a,b,c20Phenol 2-monooxygenase甲苯降解↑a,b,c,d
注:“↑”代表基因表达水平提高3倍以上;“↓”代表基因表达水平降低1/3以上。样品:a,14 NQ;b,menadione;c,plumbagin;d,lapachol
为了缩小萘醌响应基因的范围,利用红曲色素产率和差异表达基因的数据,分别对Linear、PLSR、Elastic Net、Random forest和ANN这5种人工智能模型进行了训练。平均交叉验证系数(R2)通常被认为是判断模型的预测值和实测值之间相关性的重要指标,而拟合标准差(root mean square error,RMSE)是反映模型预测值与实际测量值之间平均差距的关键参数。比较5种模型,发现ANN的R2最高,而RMSE最低(图2-a和图2-b),说明ANN能够更好地预测M.ruber M7的萘醌响应过程。目前人工智能模型中,ANN也被认为是预测生物体内复杂过程的重要工具[19]。
尽管ANN在这些模型中较优,但该ANN模型的预测精度仅有0.72。为进一步优化该模型(图2-d),采用激活函数来提高预测精度和降低计算量[20]。本研究尝试了3种激活函数,其中LReLu表现最好,使ANN预测精度提高到0.81(图2-d)。但此时ANN仍需要较高的训练步数(>25 000步)。进一步采用优化器Adam降低转录组数据中的噪声以提高拟合水平[21],结果表明训练步数仅需要6 000步就能够将精度提高至0.87(图2-e)。
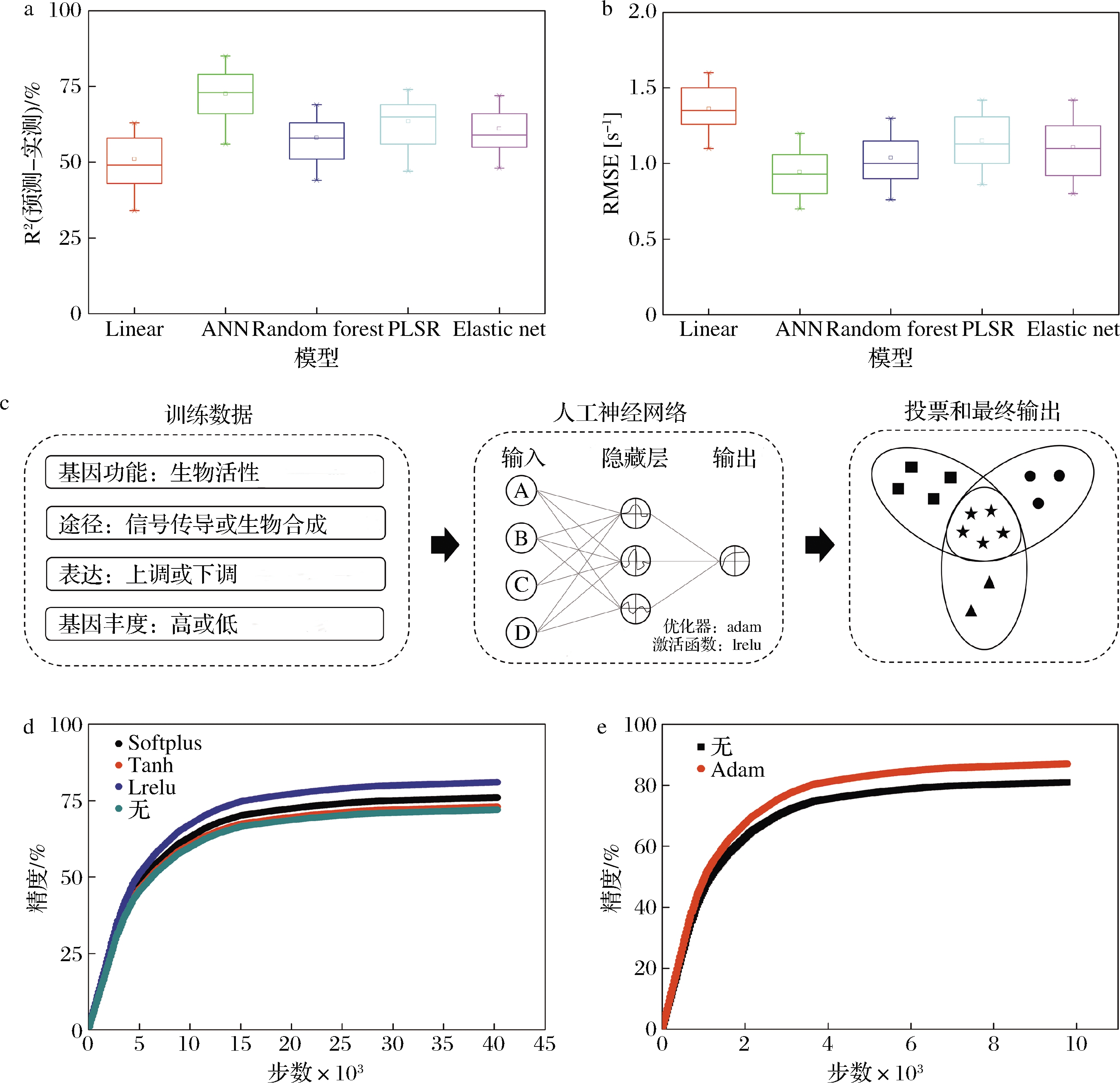
a-不同模型的平均交叉验证系数R2;b-不同模型的拟合标准差RMSE;c-ANN模型计算过程示意图;d-激活函数对ANN模型预测精度的影响;e-优化器对ANN模型预测精度与计算步数的影响
图2 人工智能模型的筛选以及ANN模型的优化
Fig.2 Selection and optimization of ANN model
利用优化后的ANN模型进一步从20个基因中预测出4个萘醌响应基因:laeA、mga2、artR和sk。laeA和mga2在4种萘醌类化合物条件下都被ANN模型预测为萘醌响应基因。而artR和sk这2个基因却仅在一定条件下被作为萘醌响应基因(表2)。基因artR在丝状真菌中调控麦角固醇的生物合成[22]。基因sk是鞘磷脂生物合成途径中的关键基因,可能涉及细胞生长和发育[23]。因此artR和sk也可能参与了M.ruber M7的萘醌响应过程,但需要一定的条件才能有明显表现。因此,最终选择laeA和mga2作为进一步研究对象。
表2 优化ANN模型预测的萘醌响应基因
Table 2 Naphthoquinone-responsive genes revealed by the optimized ANN model
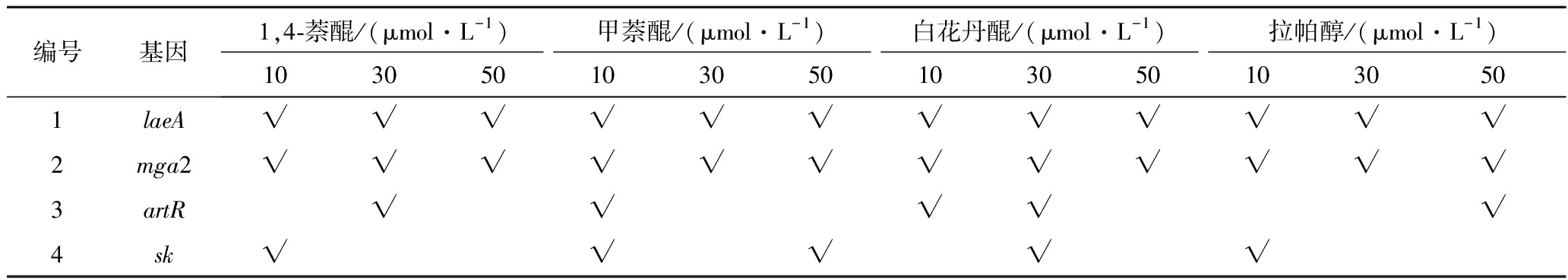
编号基因1,4-萘醌/(μmol·L-1)甲萘醌/(μmol·L-1)白花丹醌/(μmol·L-1)拉帕醇/(μmol·L-1)1030501030501030501030501laeA√√√√√√√√√√√√2mga2√√√√√√√√√√√√3artR√√√√√4sk√√√√√
2.3 萘醌添加发酵实验验证mga2基因是萘醌响应基因
为了验证laeA和mga2这2个基因中哪个是萘醌响应基因,我们利用相应的基因敲除菌株进行萘醌添加发酵实验。萘醌诱导条件下Δmga2菌株的萘醌响应显著减弱,红曲色素产率的诱导效果消失(图3-a~图3-c),说明缺乏mga2基因后红曲菌株无法响应萘醌化合物。ΔlaeA菌株在萘醌化合物诱导条件下,依然保留了一定的诱导效果(图3-d~图3-f),说明laeA不是主要的响应基因。基因mga2是G蛋白中的β亚基,参与G蛋白信号转导途径,因此萘醌化合物可能激活质异三聚体G蛋白,mga2亚基则向下游传导信号,促进红曲色素合成过程[24]。
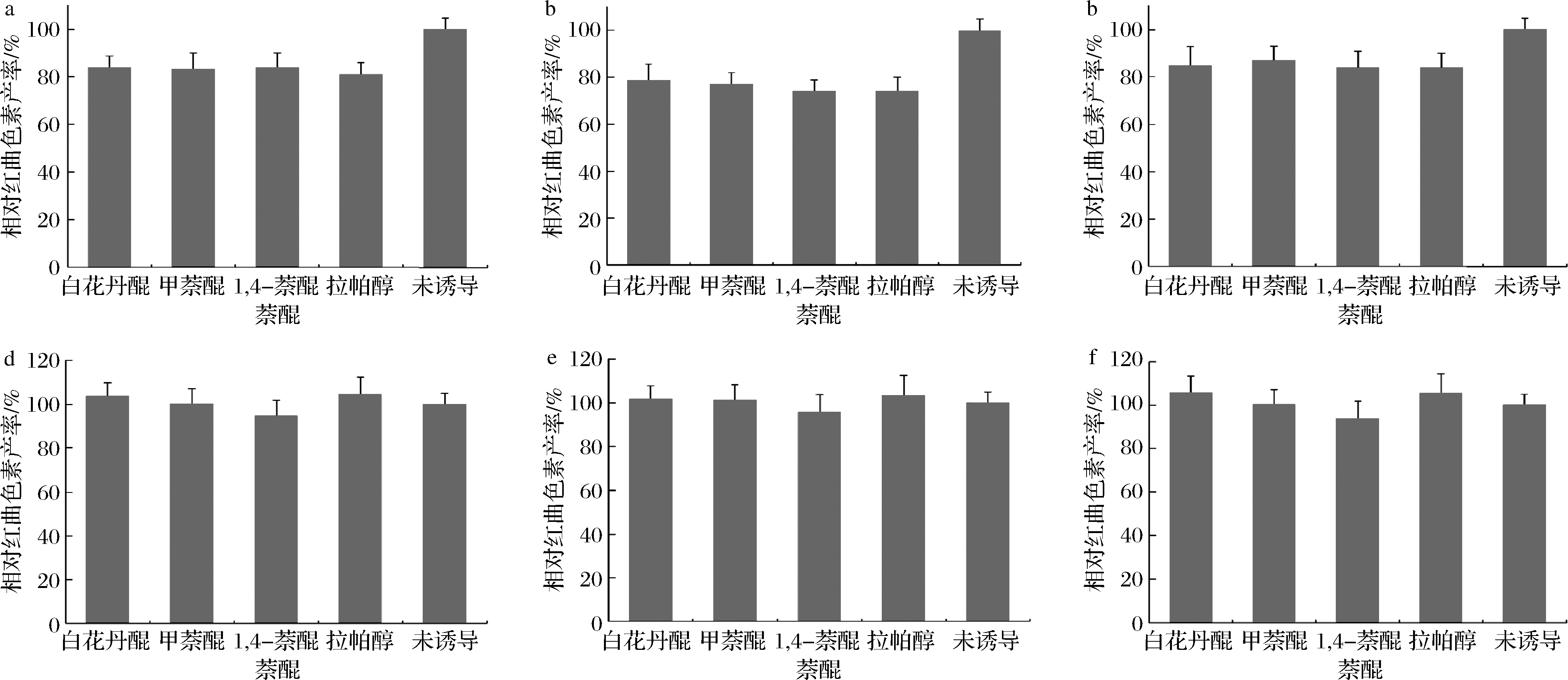
a-10 μmol/L萘醌诱导Δmga2菌株;b-30 μmol/L萘醌诱导Δmga2菌株;c-50 μmol/L萘醌诱导Δmga2菌株;d-10 μmol/L萘醌诱导ΔleaA菌株;e-30 μmol/L萘醌诱导ΔleaA菌株;f-50 μmol/L萘醌诱导ΔleaA菌株
图3 萘醌添加实验验证mga2基因是萘醌响应基因
Fig.3 Identification of mga2 as the naphthoquinone-responsive gene by naphthoquinone feeding assay
2.4 萘醌诱导mga2基因过表达菌株提高红曲色素产率
研究进一步利用mga2的过表达菌株M7::PtrpC-mga2实现萘醌介导的红曲色素产率提高。该过表达菌株为实验室前期构建,其红曲色素产率为1 126 U/mg,与原始菌株M7没有显著差异。首先测定了不同种类萘醌化合物在30 μmol/L浓度下对菌株红曲色素产率的影响,发现白花丹醌条件下红曲色素产率最高,比对照组提高了52%,达到1 719 U/mg(图4-a)。然后测定了不同浓度(0、10、20、30、40 μmol/L)白花丹醌的影响,结果表明,在20 μmol/L浓度下,过表达菌株的红曲色素产率达到1 877 U/mg(图4-b)。研究还测定了白花丹醌的添加时机,发现在菌株培养第3天加入20 μmol/L的白花丹醌,红曲色素产率进一步提高到了2 002 U/mg(图4-c)。同时,我们还测定了野生菌株M7和过表达菌株M7::PtrpC-mga2中mga2基因的转录表水平,发现过表达菌株中该基因的转录水平是野生菌株的4.2倍(图4-d)。综合以上结果说明提高mga2基因的表达量能够增强红曲菌对萘醌的响应能力。最后,研究还测定了红曲色素发酵的过程曲线,可见白花丹醌诱导后红曲色素产率显著增加(图4-e)。
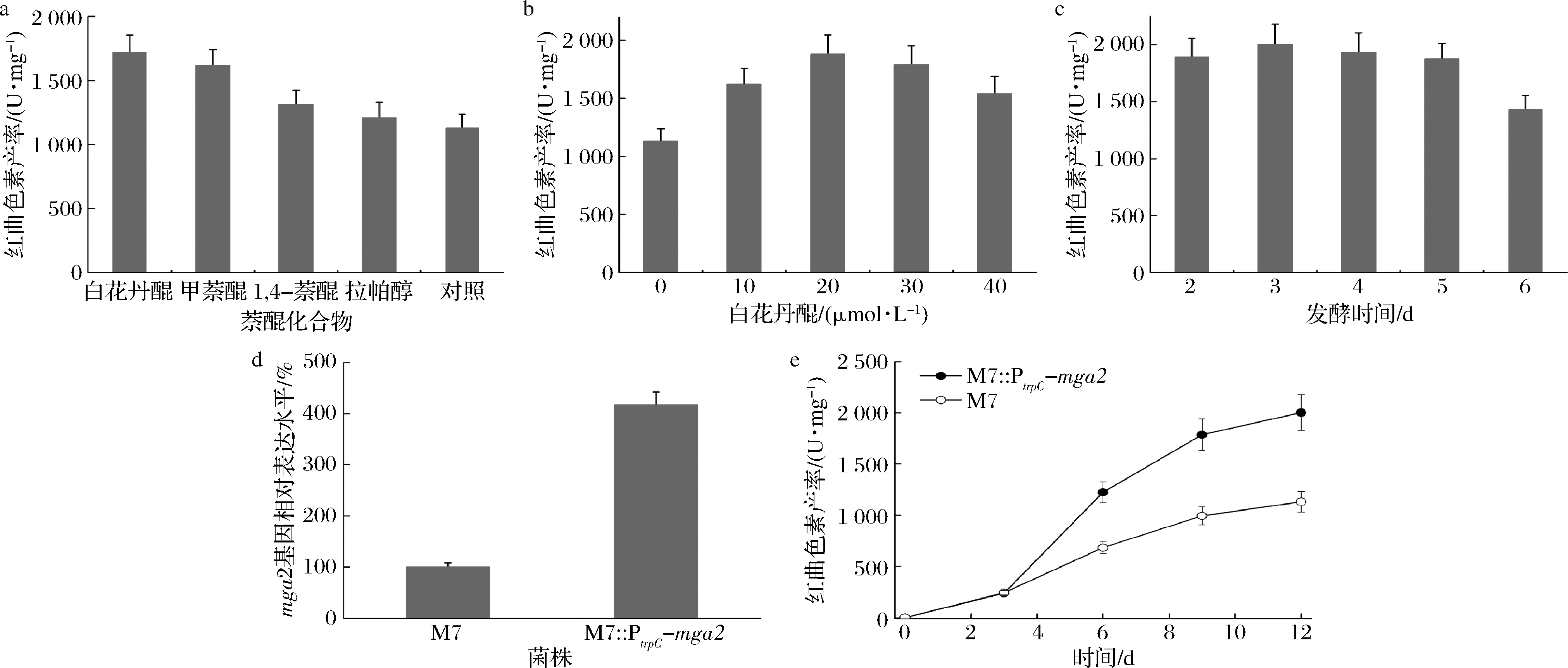
a-不同萘醌对菌株M7::PtrpC-mga2红曲色素产率的影响;b-不同浓度白花丹醌对菌株M7::PtrpC-mga2红曲色素产率的影响;c-不同诱导时机对菌株M7::PtrpC-mga2红曲色素产率的影响;d-原始菌株与M7::PtrpC-mga2菌株中mga2基因的相对表达水平;e-原始菌株与M7::PtrpC-mga2菌株发酵红曲色素的过程曲线
图4 白花丹醌诱导提高M7::PtrpC-mga2菌株的红曲色素产率
Fig.4 Improvement of Monascus pigments yield in strain M7::PtrpC-mga2 by plumbagin induction
3 结论
本研究发现萘醌化合物能够提高红曲色素产率,然后通过转录组学和人工智能模型联用的方法预测到可能的萘醌响应基因,利用基因工程菌株验证后,成功用于提高红曲色素产率。研究发现,G蛋白中的β亚基mga2是红曲菌中的萘醌响应基因,利用该基因的过表达菌株,在白花丹醌诱导下红曲色素产率显著提高。本研究不仅率先发现红曲菌中的萘醌响应基因,并应用于提高红曲色素产率,也为其他真菌中相似研究提供了方法上的借鉴。
[1] CHEN W P,CHEN R F,LIU Q P,et al.Orange,red,yellow:Biosynthesis of azaphilone pigments in Monascus fungi[J].Chemical Science,2017,8(7):4 917-4 925.
[2] CHEN W P,FENG Y L,MOLN R I,et al.Nature and nurture:Confluence of pathway determinism with metabolic and chemical serendipity diversifies Monascus azaphilone pigments[J].Natural Product Reports,2019,36(4):561-572.
R I,et al.Nature and nurture:Confluence of pathway determinism with metabolic and chemical serendipity diversifies Monascus azaphilone pigments[J].Natural Product Reports,2019,36(4):561-572.
[3] YANG Y,LIU B,DU X J,et al.Complete genome sequence and transcriptomics analyses reveal pigment biosynthesis and regulatory mechanisms in an industrial strain,Monascus purpureus YY-1[J].Scientific Reports,2015,5:8331.
[4] 李琦,高健信,陈福生,等.不产桔霉素高产红曲色素的基因工程红曲菌株构建[J].中国酿造,2018,37(6):30-35.
LI Q,GAO J X,CHEN F S,et al.Construction of genetically engineered Monascus strains with no citrinin yield but high yield Monascus pigments[J].China Brewing,2018,37(6):30-35.
[5] LIU Q P,CAI L,SHAO Y C,et al.Inactivation of the global regulator LaeA in Monascus ruber results in a species-dependent response in sporulation and secondary metabolism[J].Fungal Biology,2016,120(3):297-305.
[6] 林琳,王昌禄,李贞景,等.mok E基因过表达对红曲霉Monacolin K产量、菌丝及孢子形态的影响[J].食品科学,2018,39(8):45-49.
LIN L,WANG C L,LI Z J,et al.Effect of mok E overexpression on Monacolin K production and morphology of mycelia and spores in Monascus[J].Food Science,2018,39(8):45-49.
[7] LI M,KANG L J,DING X L,et al.Monasone naphthoquinone biosynthesis and resistance in Monascus fungi[J].mBio,2020.DOI:10.1128/mbio.02 676-19.
[8] 陈晓艳,董朝轶.动态贝叶斯网络结构搜索法辨识生物神经网络连接[J].生命科学研究,2017,21(6):527-533.
CHEN X Y,DONG C Y.Identification of biological neural network connections by dynamical Bayesian network structure searching[J].Life Science Research,2017,21(6):527-533.
[9] 谢媛媛,苏加坤,应旭辉,等.基于人工智能技术的烟气暴露大鼠代谢生物标志物筛选方法研究[J].分析测试学报,2017,36(6):705-710.
XIE Y Y,SU J K,YING X H,et al.Study on screening of cigarette smoke exposure biomarkers for rat’s metabolites on the basis of artificial intelligence technologies[J].Journal of Instrumental Analysis,2017,36(6):705-710.
[10] ![]() K M,SCHMID M,BURCH N,et al.DeepStack:Expert-level artificial intelligence in heads-up no-limit poker[J].Science,2017,356(6 337):508-513.
K M,SCHMID M,BURCH N,et al.DeepStack:Expert-level artificial intelligence in heads-up no-limit poker[J].Science,2017,356(6 337):508-513.
[11] HUANG T,TAN H L,LU F J,et al.Changing oxidoreduction potential to improve water-soluble yellow pigment production with Monascus ruber CGMCC 10910[J].Microbial Cell Factory,2017,16(1):208.
[12] HOWARD N,CHOUIKHI N,ADEEL A,et al.BrainOS:A novel artificial brain-alike automatic machine learning framework[J].Frontiers in Computational Neuroscience,2020,14:16.
[13] 李苍柏,肖克炎,李楠,等.支持向量机、随机森林和人工神经网络机器学习算法在地球化学异常信息提取中的对比研究[J].地球学报,2020,41(2):309-319.
LI C B,XIAO K Y,LI N,et al.A comparative study of support vector machine,random forest and artificial neural network machine learning algorithms in geochemical anomaly information extraction[J].Acta Geoscientica Sinica,2020,41(2):309-319.
[14] 刘开放,席志文,黄林娜,等.布拉酵母高密度发酵培养基及发酵工艺优化[J].食品科学,2019,40(8):56-62.
LIU K F,XI Z W,HUANG L N,et al.Optimization of high cell density fermentation of Saccharomyces boulardii for enhanced biomass production[J].Food Science,2019,40(8):56-62.
[15] HU Y,ZHOU Y X,MAO Z J,et al.NAD+-dependent HDAC inhibitor stimulates Monascus pigment production but inhibit citrinin[J].AMB Express,2017,7(1):166.
[16] CHEN W P,HE Y,ZHOU Y X,et al.Edible filamentous fungi from the species Monascus:Early traditional fermentations,modern molecular biology,and future genomics[J].Comprehensive Reviews in Food Science and Food Safety,2015,14(5):555-567.
[17] LIU J,CHAI X Y,GUO T,et al.Disruption of the ergosterol biosynthetic pathway results in increased membrane permeability,causing overproduction and secretion of extracellular Monascus pigments in submerged fermentation[J].Journal of Agricultural and Food Chemistry,2019,67(49):13 673-13 683.
[18] BIJINU B,SUH J W,PARK S H,et al.Delineating Monascus azaphilone pigment biosynthesis:Oxidoreductive modifications determine the ring cyclization pattern in azaphilone biosynthesis[J].RSC Advances,2014,4(103):59 405-59 408.
[19] 张奇,荣雯雯,刘艳.基因表达谱数据分类算法综述[J].实用预防医学,2018,25(8):1 018-1 021.
ZHANG Q,RONG W W,LIU Y.Review of classification algorithms for gene expression profile data[J].Practical Preventive Medicine,2018,25(8):1 018-1 021.
[20] 屈景怡,叶萌,渠星.基于区域残差和LSTM网络的机场延误预测模型[J].通信学报,2019,40(4):149-159.
QU J Y,YE M,QU X.Airport delay prediction model based on regional residual and LSTM network[J].Journal on Communications,2019,40(4):149-159.
[21] BOCK S,WEIS M.A proof of local convergence for the Adam optimizer[C].2019 International Joint Conference on Neural Networks(IJCNN).Budapest:Institute of Electrical and Electronic Engineers,2019.
[22] HAGIWARA D,MIURA D,SHIMIZU K,et al.A novel Zn2-Cys6 transcription factor AtrR plays a key role in an azole resistance mechanism of Aspergillus fumigatus by co-regulating cyp51A and cdr1B expressions[J].PLoS Pathogens,2017,13(1):e1006096.
[23] JOHNSON E L,HEAVER S L,WATERS J L,et al.Sphingolipids produced by gut bacteria enter host metabolic pathways impacting ceramide levels[J].Nature Communications,2020,11(1):2 471.
[24] LEI M,LIU J,FANG Y,et al.Effects of different G-protein α-subunits on growth,development and secondary metabolism of Monascus ruber M7[J].Frontiers in Microbiology,2019,10:1555.