金枪鱼族是属于硬骨鱼纲鲈形目鲭科的鱼类,包含细鲣属(Allothunnus)、舵鲣属(Auxis)、巴鲣属(Euthynnus)、鲣属(Katsuwonus)、金枪鱼属(Thunnus)等5属15种金枪鱼[1]。鲣鱼(Katsuwonus pelamis)是鲣属中唯一品种,在金枪鱼渔业中有着举足轻重的地位。2018年,鲣鱼捕捞量为316万 t,占金枪鱼捕捞总量的56%以上[2]。
金枪鱼肉质鲜美,具有高蛋白、低脂肪等特点,并且富含丰富的二十碳五烯酸(eicosapentaenoic acid,EPA)与二十二碳六烯酸(docosahexaenoic acid,DHA)等不饱和脂肪酸,是最受消费者欢迎的鲭科鱼类品种之一。然而,金枪鱼较少以鲜活的形式进行出售,通常被加工成生鱼片、罐头、肉松等制品。由于加工破坏了其外形特征,传统的形态特征鉴别法已经不能满足金枪鱼的物种鉴别。在这种情况下,一些不法商贩为牟取暴利,在金枪鱼及其制品的贸易过程中掺假、造假,以次充好的现象时有发生。安丽艳等[3]采用PCR测序技术鉴定金枪鱼罐头中金枪鱼品种,在17个市售样品中,发现58.8%来源于价格低廉的鲣鱼。SOTELO等[4]对6个欧洲国家的545份金枪鱼(新鲜、冷冻和罐头产品)进行调查,发现总体标签错误率为6.79%,新鲜和冷冻产品为6.70%,罐头产品为7.84%。金枪鱼市场鱼目混杂,标示不清,水产品的掺假行为在媒体中频频曝光,引起了消费者的恐慌。因此,为了保护消费者经济利益、维护消费者的合法权益,急需一种灵敏、特异、快速的金枪鱼及其制品的鉴定方法。
基于DNA的检测技术具有灵敏度高、特异性好等优点,已经被广泛运用于水产品检测[5-7]。目前,已经报道的鉴定方法有DNA条形码法[8]、核苷酸测序法[3]、实时荧光定量PCR法[9]、PCR限制性片段长度多态性法[10-11]、多重PCR琼脂糖电泳法[12]。其中,多重PCR琼脂糖电泳法灵敏度较低,近物种鉴定易出现假阳性;DNA条形码法需要测序,鉴定周期长。此外,DNA条形码法还存在依赖参考的数据库建设不够完善、核内DNA条形码和混合物种样品单一条形码获取困难等不确定因素。但至今为止,采用环介导恒温扩增技术(loop-mediated isothermal amplification,LAMP)检测金枪鱼的研究报道比较少见。
LAMP技术是NOTOMI等[13]于2000年提出的一种核酸扩增技术,该技术依赖Bst DNA聚合酶在恒温条件下实现靶基因的自循环链置换合成反应,引物能识别基因的6个或8个独立区域,特异性较PCR大大提高。另外,反应的温度处于恒温条件(60~65 ℃),普通水浴锅就可以满足扩增的需求,节省了检测费用。检测结果可以通过可视化判别,在扩增产物中加入荧光染料SYBR Green I,产物结合染料后发出绿色荧光。目前已有LAMP技术应用于食源性致病菌、转基因成分、过敏源成分[14-15]、动物源成分[19-20]等检测方面的研究。本研究以鲣鱼的线粒体Cyt b基因为靶基因,设计LAMP反应特异性引物,构建LAMP反应体系,建立一种鲣鱼的特异性快速检测技术,为金枪鱼制品的掺假鉴伪提供科学依据。
1 材料与方法
1.1 材料与试剂
1.1.1 样品的采集
22种水产品标准品来自本课题组前期研究,包括Katsuwonus pelamis(MK843764)、Thunnus obesus(MN893173)、T.albocaves(MN893174)、T.alalunga(MN893175)、Scomber niphonius(MN879568)、Scomber spp(MK843721)、Scomber scombrus(MN893176)、Oyprinus carpio(MK843655)、Colla ectenes(MK84666)、Larimichrhy scrocea(MK843674)、Lateolabrax japonicus(MK843686)、Odontobutis potamophila(MK843730)、Patagonotothen ramsayt(MK843753)、Alabtrosstd pecoralls(MK843767)、Pangashus hypophthalms(MK843766)、Micropterus salmoides(MK843768)、Clenopharyngodon idella(MK843773)、Lophius Hitulon(MN850429)、Oncorhymnchus mykiss(MN850431)、Carassius auratus(MK843660)、Mugil cephalus(MN893207)、S.salar(MN850430)。22种样品均进行形态学鉴定和DNA条形码鉴定,且标准品的DNA条形码序列已提交至GenBank,并获得登录号(已备注在对应物种的括号内)。
此外,2019年11月~12月,从市场共采集39份预包装金枪鱼产品(表1),包括19份肉松产品(编号为XWJ1-XWJ19)和20份罐装制品(编号为XWJ20-XWJ39)。所有产品抵达实验室后需进行拍照和标记,并记录好产品标签上的信息。当产品包装内有多个独立小包装时,每个小包装需单独取样,共计取样59份(表1),样品于-18 ℃保存备用。
1.1.2 试剂
DP304细胞/血液/组织基因组DNA提取试剂盒,天根生化科技(北京)有限公司;6×Loading Buffer、dNTP Mix,宝生物工程(大连)有限公司;等温扩增混合液、2×Taq PCR Master Mix、引物合成,上海生工生物工程股份有限公司;Bst DNA聚合酶大片段、MgSO4溶液、10×ThermoPol缓冲液,美国New England Biolabs公司;SYBR Green I染料(10 000×),北京索莱宝科技有限公司。
表1 市售样品的采集与鉴定结果
Table 1 Collection and identification results of municipal samples

产品类型产品编号产地样品编号标签原料LAMPBLAST (100%)肉松XWJ1中国XWJ1-1鲔鱼(-)Thunnus spp.XWJ2XWJ2-1金枪鱼(-)Tetrapturus sppXWJ2-2金枪鱼(-)Tetrapturus sppXWJ3XWJ3-1鲔鱼(-)Thunnus spp.XWJ4XWJ4-1鲔鱼(-)Thunnus spp.XWJ5XWJ5-1金枪鱼(-)Thunnus spp.XWJ6XWJ6-1金枪鱼(-)Thunnus spp.XWJ7XWJ7-1金枪鱼(-)Makaira spp.XWJ8XWJ8-1鲔鱼(-)Thunnus spp.XWJ9XWJ9-1金枪鱼(-)Thunnus spp.XWJ10XWJ10-1金枪鱼(-)Thunnus spp.XWJ11XWJ11-1金枪鱼(-)Thunnus spp.XWJ12XWJ12-1鲔鱼(-)Thunnus spp.XWJ12-2鲔鱼(-)Thunnus sppXWJ13XWJ13-1金枪鱼(-)Thunnus spp.XWJ14XWJ14-1金枪鱼(-)Scomber scombrusXWJ15XWJ15-1金枪鱼(-)重叠峰XWJ15-2金枪鱼(-)Scomber scombrusXWJ15-3金枪鱼(-)Scomber scombrusXWJ16XWJ16-1金枪鱼(-)重叠峰XWJ17XWJ17-1金枪鱼(-)Thunnus spp.XWJ18XWJ18-1金枪鱼(-)Thunnus spp.XWJ19XWJ19-1金枪鱼(-)Thunnus spp.罐头XWJ20XWJ20-1金枪鱼(-)重叠峰XWJ20-2金枪鱼(-)Makaira spp.XWJ21XWJ21-1吞拿鱼(-)重叠峰XWJ21-2吞拿鱼(-)重叠峰XWJ22XWJ22-1金枪鱼(-)重叠峰XWJ22-1金枪鱼(-)Makaira spp.XWJ23泰国XWJ23-1金枪鱼(-)Makaira spp.XWJ23-2金枪鱼(-)Makaira spp.XWJ24西班牙XWJ24-1金枪鱼(-)Makaira spp.XWJ24-2金枪鱼(-)Makaira spp.XWJ25泰国XWJ25-1鲣鱼(+)K. pelamisXWJ25-2鲣鱼(+)K. pelamisXWJ26中国XWJ26-1金枪鱼(-)重叠峰XWJ26-2金枪鱼(-)重叠峰XWJ27韩国XWJ27-1金枪鱼(-)重叠峰XWJ27-2金枪鱼(-)重叠峰XWJ28西班牙XWJ28-1金枪鱼(-)重叠峰XWJ28-2金枪鱼(-)重叠峰XWJ29中国XWJ29-1金枪鱼(-)重叠峰XWJ30泰国XWJ30-1吞拿鱼(-)重叠峰XWJ31泰国XWJ31-1金枪鱼(-)重叠峰XWJ32泰国XWJ32-1金枪鱼(-)重叠峰XWJ32-2金枪鱼(-)重叠峰XWJ33泰国XWJ33-1金枪鱼(-)重叠峰XWJ33-2金枪鱼(-)重叠峰XWJ34中国XWJ34-1金枪鱼(-)重叠峰XWJ34-2金枪鱼(-)重叠峰XWJ35泰国XWJ35-1金枪鱼(-)重叠峰XWJ35-2金枪鱼(-)重叠峰XWJ36中国XWJ36-1鲣鱼(+)K. pelamisXWJ36-2鲣鱼(+)K. pelamisXWJ37XWJ37-1金枪鱼(-)重叠峰XWJ37-2金枪鱼(-)重叠峰XWJ38菲律宾XWJ38-1鲣鱼(+)K. pelamisXWJ38-2鲣鱼(+)K. pelamisXWJ39中国XWJ39-1金枪鱼(-)重叠峰
注:(+)表示阳性结果,(-)表示阴性结果
1.2 仪器与设备
BioPhotometer D30核酸蛋白测定仪,德国艾本德股份有限公司;1-14k台式小型离心机,德国Sigma公司;MS-100恒温混匀仪,杭州奥盛仪器有限公司;Gentier 96全自动医用PCR分析系统,西安天隆科技有限公司。
1.3 实验方法
1.3.1 DNA提取
按照DNA提取试剂盒说明书对市售样品的DNA进行提取,取50 mg样品于1.5 mL离心管中,先加入200 μL Buffer GA,涡旋振荡15 s;再加20 μL蛋白酶K(20 mg/mL)溶液,涡旋混匀后将其置于恒温混匀器中1 500 r/min 56 ℃加热45 min。最后,加入200 μL Buffer GB,充分颠倒混匀后置于恒温混匀器中1 500 r/min 70 ℃加热10 min。将所得混合物转移至吸附柱CB3中,依次加入500 μL Buffer GD和600 μL漂洗液PW,混匀后均以12 000 r/min离心30 s,最后,向吸附膜的中间悬空滴加100 μL洗脱液TE。
DNA的质量检测:采用BioPhotometer D30核酸蛋白测定仪分别测定DNA在230、260和280 nm的吸光值,计算A260/A280和A260/A230值,同时测定DNA浓度。对符合纯度要求的DNA进行琼脂糖凝胶电泳,检测其完整性,纯度和完整性均符合要求的DNA置于-20 ℃冰箱保存备用。
1.3.2 引物的设计
从GeneBank数据库下载鲣鱼(JN086155、KJ617258、EF392591、EF439208、DQ080321)的基因序列信息。并使用BioEdit软件进行序列比对,筛选出种间差异比较大的Cyt b序列作为检测基因,此外,下载非目标物种,长鳍金枪鱼(AB101291)、黄鳍金枪鱼(JN086153)、马苏金枪鱼(KF925362)、大眼金枪鱼(GU256525)、蓝鳍金枪鱼(KF906720)等的Cyt b基因序列,确保鲣鱼和非目标物种之间的错配,以保证引物的特异性。根据LAMP引物设计原则,利用在线PrimerExplorerV4 (http://primerexplorer.jp/elamp4.0.0/index.html)设计LAMP引物,包括外引物(F3、B3)、内引物(FIP、BIP),引物则委托上海生工生物有限公司合成,序列见表2。
表2 本研究所用引物
Table 2 The sequence of primers

目标物种基因引物名称引物序列(5′➝3′)片段大小参考文献FOR16SpcTGCCCGTGCAGAAGCGG327 bp通用引物16S rDNAREV16SpcCAACATCGAGGTCGTAAACCC[21]FOR16S-1GACGAGAAGACCCTATGG242 bpREV16S-2CTGTTATCCCTAGGGTAACTZJ-F3CCATTCCTACATACTTCAAAAC-K. pelamisCyt bZJ-B3CATCCGAGGATTTTGTTCTCT-本研究ZJ-FIPCCGATTCAGGTAAGGATTGCCTTTTCACTTACATTCCGACCAGTC-ZJ-BIPCCTTCATTATCATCGGCCAAGTTTTTCTGCAAGTGGGAAGAAGATG-
注:-表示无数据
1.3.3 LAMP 反应体系建立及条件优化
LAMP反应体系25 μL,含1.4 mmol/L dNTP Mix、1 μL Bst DNA 聚合酶、6 mmol/L MgSO4、2.5 μL 10×ThermoPol缓冲液、1 μL 5×SYBR Green Ⅰ、上游外引物(F3)0.2 μmol/L、下游外引物(B3)0.2 μmol/L、上游内引物(FIP)1.6 μmol/L、下游内引物(BIP)1.6 μmol/L、模板DNA 1 μL,剩下用无菌水补足至25 μL。采用恒温实时荧光法检测,将混合物置于65 ℃恒温反应60 min,最后在80 ℃下10 min结束反应。也可通过向LAMP产物中加入1 μL稀释倍数为100的 SYBR Green I染料肉眼观察扩增情况,在紫外灯下,颜色变绿表明发生了扩增,橙色表明未发生扩增。
对该LAMP反应体系中的Mg2+浓度、外引物、内引物、dNTP Mix、10×ThermoPol缓冲液等因素进行优化,确定最终的最优反应体系。
1.3.4 PCR扩增
采用DNA条形码通用引物(表2)扩增市售DNA样品,采用25 μL的PCR体系,其中2×Taq PCR Master Mix体系预混液12.5 μL,上下游引物(10 μmol/L)各1 μL,模板DNA 1 μL,加无菌水补足至25 μL。PCR扩增条件为:94 ℃预变性3 min;95 ℃ 变性30 s,53 ℃退火20 s,72 ℃延伸30 s,35个循环;72 ℃延伸10 min。反应结束后,以2%琼脂糖凝胶电泳检测PCR产物。
1.3.5 市售样品检测
利用试验建立的LAMP方法进行市售样品的检测,并与DNA条形码检测所得数据进行对比,验证其方法的准确性。
2 结果与分析
2.1 LAMP试验条件优化结果
2.1.1 内外引物浓度比的优化
选取外引物与内引物浓度比梯度依次为1∶4、1∶6、1∶8、和1∶10分别进行LAMP反应,观察阳性反应出现的快慢,从而确认引物浓度比对扩增反应的影响,以此结果确定最佳引物浓度比。由图1可知,试验选用1∶8引物浓度比扩增效果较好。

图1 内外引物浓度比的优化
Fig.1 Optimization of primer concentration ratio inside and outside
2.1.2 Mg2+浓度的优化
在最佳的引物浓度比的反应体系下,选择优化Mg2+浓度,Mg2+是较为重要的试剂之一,能影响Bst DNA聚合酶的活性,反应过程中起到降低反应所需要的活化能的作用。选取Mg2+浓度梯度依次为0、2、4和6 mmol/L分别进行LAMP反应。由图2可知,在检测浓度范围内随Mg2+浓度的升高,促进了反应的进行,当达到6 mmol/L浓度时,最利于反应的进行。

图2 Mg2+浓度的优化
Fig.2 Mg2+ optimization of concentration
2.1.3 dNTP Mix浓度的优化
dNTP是LAMP扩增反应的原料,dNTP Mix浓度分别为1.0、1.2、1.4和1.6 mmol/L进行扩增反应。由图3可知,当dNTP Mix浓度为1.6 mmol/L为最适浓度。

图3 dNTP Mix浓度的优化
Fig.3 dNTP Mix concentration optimization
2.1.4 10×ThermoPol缓冲液浓度的优化
10×ThermoPol缓冲液体系添加量梯度为0.5、1.5、2.5和3.5 μL,其他条件下相同。由图4可以看出,在检测浓度范围内随浓度的升高,促进了反应的进行。当添加量为3.5 μL时,最利于反应的进行。
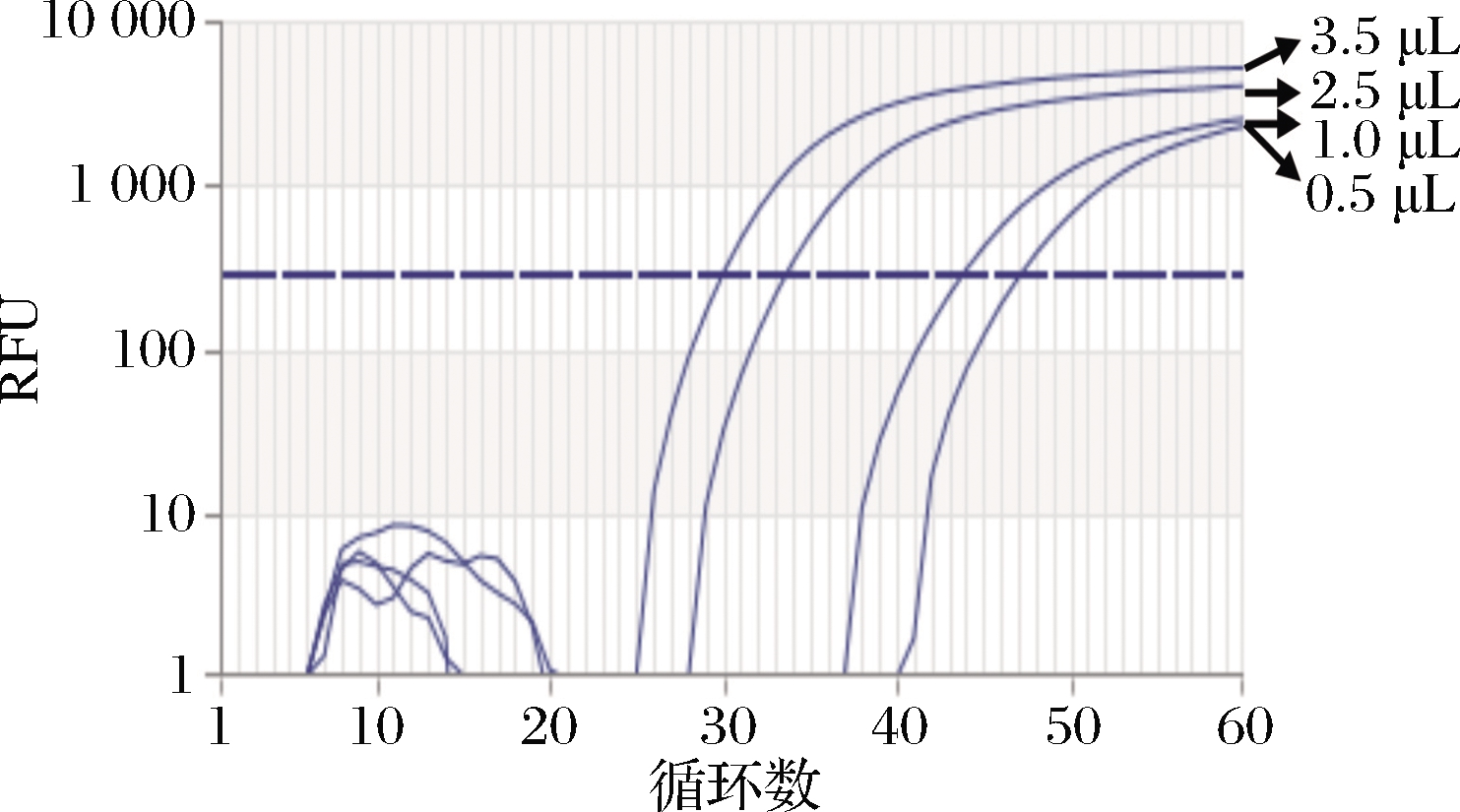
图4 10×ThermoPol缓冲液浓度的优化
Fig.4 10×ThermoPol optimization of buffer concentration
2.2 特异性试验结果
以鲣鱼和其他22个非目标物种的DNA为模板,进行LAMP扩增,验证引物的特异性。扩增产物采用实时荧光法进行检测。如图5所示,空白对照和对照样品均未发生扩增,以鲣鱼DNA为模板的试验组出现扩增,表明该体系具有良好的特异性。

图5 特异性检测
Fig.5 Specific detection
2.3 灵敏度试验分析
将鲣鱼的基因组DNA进行倍比稀释,质量浓度设定为50、5、0.5、0.05 ng/μL。作为反应模板进行扩增,确定方法的灵敏度。如图6所示,该试验所建立的LAMP体系的灵敏度为0.05 ng/μL。

图6 灵敏度检测
Fig.6 Sensitivity detection
2.4 市售样品检测结果
为了评价所建立LAMP方法的适用性和可行性,本研究采集了59份市售样品进行了鉴定(图7、图8)。检测结果通过扩增曲线和可视化2种方式进行判别,观察反应产物在紫外灯下是否有荧光产生。结果显示,肉松制品鉴定结果不含目标成分,罐头制品中6个样本鉴定结果为鲣鱼,其余样本均为非目标物种。由此可得,LAMP鉴定结果与DNA条形码鉴定结果一致。

图7 市售样品鉴定
Fig.7 Sample identification

a-肉松制品;b-罐头制品
图8 市售样品可视化结果
Fig.8 Visual results of marketed samples 注:(-):无菌水;(+):阳性对照
2.5 讨论
在物种属性鉴别领域,DNA条形码技术是一种较为成熟的分子检测技术。其理论依据是使用一段标准DNA片段,通过测序获得样品的线粒体序列,达到物种鉴定的目的。但是对于深加工制品,由于含有多种混合DNA,而检测用引物是通用引物,存在极大的漏检可能性。
LAMP技术目前在鱼类物种鉴定中已广泛使用,而建立高效快速的LAMP体系的基础是特异性引物的设计。常用于鱼类物种鉴定中的目的基因有COⅠ基因、16S rRNA基因、Cyt b基因和12S rRNA基因。通过大量序列进行比对,根据相互间的遗传距离,证实了Cyt b基因作为目的基因的可行性。
传统的LAMP法仍存在一些不足。浊度法检测需要积累大量的扩增子,当低拷贝的模板存在时焦磷酸镁沉淀产生较少,肉眼判断困难;钙黄绿素法引入的Mn2+增加了Bst聚合酶错配的机率,且钙黄绿素的荧光背景降低了检测的灵敏度;可视化LAMP荧光染料SYBR GreenⅠ在水溶液中本身不发光,DNA存在时荧光显著增强。该方法直接通过可视化比色判定检测结果,具有快速、直观、低成本等优点。
3 结论
本研究建立了基于Cyt b基因的鲣鱼LAMP检测方法,建立的方法具有特异性强、快速的优点。在基因组DNA水平为0.05 ng/μL时,即可检出。同时将LAMP扩增技术与荧光染料相结合,即添加指示剂实现可视化检测结果。与DNA条形码鉴定方法相比,该方法不需要复杂的热循环控温仪器,同时也提高了检测效率降低了检测成本,适用于大规模样品的初筛,为监管机构快速检测金枪鱼制品的物种提供技术支持。
[1] COLLETTE B B, REEB C, BLOCK B A.Systematics of the tunas and mackerels (Scombridae)[J].Fish Physiology, 2001, 19:1-33.
[2] FAO Fisheries and Aquaculture Department.The State of World Fisheries and Aquaculture[M].Rome:Food and Agriculture Organization of the United Nations, 2010.
[3] 安丽艳, 孟镇, 仇凯, 等.应用PCR-FINS技术鉴定金枪鱼罐头中金枪鱼种类[J].食品与发酵工业, 2016, 42(6):159-163.
AN L Y, MENG Z, QIU K, et al.Development of a method for the identification of tuna species in canned tuna by FINS[J].Food and Fermentation Industries, 2016, 42(6):159-163
[4] SOTELO C G, VELASCO A, PEREZ-MARTIN R I, et al.Tuna labels matter in Europe:Mislabelling rates in different tuna products[J].PLoS One, 2018, 13(5):e0196641.
[5] ZAHN R J, SILVA A J, HELLBERG R S.Development of a DNA mini-barcoding protocol targeting COI for the identification of elasmobranch species in shark cartilage pills[J].Food Control, 2020, 109:106918.
[6] ARMANI A, GUARDONE L, CASTIGLIEGO L, et al.DNA and Mini-DNA barcoding for the identification of Porgies species (Family Sparidae) of commercial interest on the international market[J].Food Control, 2015, 50:589-596.
[7] CARVALHO D C, PALHARES R M, DRUMMOND M G, et al.Food metagenomics:Next generation sequencing identifies species mixtures and mislabeling within highly processed cod products[J].Food Control, 2017, 80:183-186.
[8] BOTTI S, GIUFFRA E.Oligonucleotide indexing of DNA barcodes:Identification of tuna and other scombrid species in food products[J].BMC Biotechnology, 2010, 10:60.
[9] BOJOLLY D, DOYEN P, LEFUR B, et al.Development of a qPCR method for the identification and quantification of two closely related tuna species, bigeye tuna (Thunnus obesus) and yellowfin Tuna (Thunnus albacares), in canned tuna[J].Journal of Agricultural and Food Chemistry, 2017, 65(4):913-920.
[10] XU K H, FENG J L, MA X T, et al.Identification of tuna species (Thunnini tribe) by PCR-RFLP analysis of mitochondrial DNA fragments[J].Food and Agricultural Immunology, 2016, 27(3):301-313.
[11] LIN W F, HWANG D F.Application of PCR-RFLP analysis on species identification of canned tuna[J].Food Control, 2007, 18(9):1 050-1 057.
[12] GIUSTI A, CASTIGLIEGO L, RUBINO R, et al.A conventional multiplex PCR assay for the detection of toxic gemfish species (Ruvettus pretiosus and Lepidocybium flavobrunneum):A simple method to combat health frauds[J].Journal of Agricultural and Food Chemistry, 2016, 64(4):960-968.
[13] NOTOMI T, OKAYAMA H, MASUBUCHI H, et al.Loop-mediated isothermal amplification of DNA[J].Nucleic Acids Research, 2000, 28(12):e63.
[14] MORI Y, KANDA H, NOTOMI T.Loop-mediated isothermal amplification (LAMP):Recent progress in research and development[J].Journal of Infection and Chemotherapy, 2013, 19(3):404-411.
[15] ZHANG Y Q, SHAN X X, SHI L, et al.Development of a fim Y-based loop-mediated isothermal amplification assay for detection of Salmonella in food[J].Food Research International, 2012, 45(2):1 011-1 015.
[16] FAN H Y, LONG B G, WU X B, et al.Development of a loop-mediated isothermal amplification assay for sensitive and rapid detection of Cronobacter sakazakii[J].Foodborne Pathogens and Disease, 2012, 9(12):1 111-1 118.
[17] NOTOMI T, MORI Y, TOMITA N, et al.Loop-mediated isothermal amplification (LAMP):Principle, features, and future prospects[J].Journal of Microbiology, 2015, 53(1):1-5.
[18] ZAHRADNIK C, MARTZY R, MACH R L, et al.Detection of the food allergen celery via loop-mediated isothermal amplification technique[J].Analytical and Bioanalytical Chemistry, 2014, 406(27):6 827-6 833.
[19] ZAHRADNIK C, MARTZY R, MACH R L, et al.Loop-mediated isothermal amplification (LAMP) for the detection of horse meat in meat and processed meat products[J].Food Analytical Methods, 2015, 8(6):1 576-1 581.
[20] AHMED M U, HASAN Q, HOSSAIN M M, et al.Meat species identification based on the loop mediated isothermal amplification and electrochemical DNA sensor[J].Food Control, 2010, 21(5):599-605.
[21] ARMANI A, TINACCI L, XIONG X, et al.Fish species identification in canned pet food by BLAST and forensically informative nucleotide sequencing (FINS) analysis of short fragments of the mitochondrial 16 s ribosomal RNA gene (16S rRNA)[J].Food Control, 2015, 50:821-830.