山羊(Capra hircas)和绵羊(Ovis aries)为偶蹄目牛科羊亚科的不同属生物,山羊有60条染色体,而绵羊只有54条[1-2]。除了分类学上不同之外,二者在生活习性、繁殖能力等方面也存在差异。例如山羊食性杂,除牧草外还喜食灌木嫩枝、藤蔓等,而绵羊喜食非禾本科草、阔叶草[3]。山羊繁殖能力强,每胎可产羔2~3只,哺乳期7~9个月,年产乳量450~600 kg;而绵羊多为单胎,哺乳期为3~5个月,年产乳量仅为30~350 kg[4-5]。
以上因素导致绵羊养殖范围小、难度高,产乳量仅占羊乳总产量38%。但由于绵羊乳铁蛋白含量是牛乳的3倍,其蛋白质、酪蛋白、共轭亚油酸和维生素C含量均显著高于山羊乳[6-8]。与牛乳和山羊乳相比,绵羊乳在我国乳品加工领域尚处于初级阶段,且绵羊乳功能成分研究较为缺乏。因此本研究通过测定山羊乳与绵羊乳外泌体miRNAs表达谱,并对其进行对比分析,揭示2种羊乳的潜在功能差异。
外泌体是细胞自然产生的胞外囊泡,几乎存在于所有生物液体中。作为一种“纳米载体”,粒径在30~150 nm[9],通过携带脂质、蛋白质、mRNA、非编码RNA和DNA,在各种生物学过程中以及物种间传递遗传信息[10]。乳外泌体在自然界中含量丰富,并表现出许多有利于治疗作用的纳米载体特性[11]。母乳与牛乳外泌体中的miR-146a等分子,具有抑制Toll样受体(Toll like receptors,TLR)依赖的免疫炎症反应激活[12-13];猪乳源外泌体miRNAs可以提高葡聚糖硫酸钠诱导肠炎模型肠道紧密连接蛋白ZO-1的表达[14]。药物负载效果研究中也发现山羊乳外泌体的药物装载能力显著高于牛乳和水牛乳[15]。此外,还有研究发现山羊乳外泌体能够显著降低了登革热病毒的复制和成熟病毒粒子的分泌[16]。以上数据提示羊乳外泌体与其天然负载大分子具有重要的生物学功能。然而,目前关于山羊和绵羊乳外泌体miRNAs表达谱研究十分有限,这在一定程度上限制了研究人员对羊乳功能的进一步认识,也影响了羊乳外泌体的深入研究与应用。
本研究利用超高速离心法分离山羊与绵羊乳外泌体,接着构建了山羊乳及绵羊乳外泌体sRNA文库,利用Illumina平台对其中的miRNAs分子进行测序和生物信息学分析,并分析其功能及差异性。
1 材料与方法
1.1 样品采集和制备
本研究采用前中泌乳期的关中奶山羊和东弗里生绵羊乳液样本。分别将30份山羊乳样品和绵羊乳样品随机分为3组,并充分混合。样品采集后立即冷冻,并置于干冰中运输至实验室,保存于超低温冰箱(-80 ℃)直至后续研究。
1.2 仪器与设备
OptiamTM L-90k Ultracentrifuge超速离心仪,Beckman Coulter公司;NanoPhotometer®分光光度计,IMPLEN公司;Qubit®2.0荧光定量仪,Life Technologies公司;2100型生物分析仪,Agilent公司。
1.3 方法
1.3.1 羊乳外泌体提取
取出样本25 ℃水浴解冻后,在4 ℃、2 000×g离心10 min,将上清液继续在4 ℃、10 000×g离心30 min,随后将样本上清转移至超高速离心管中,在4 ℃、110 000×g离心75 min弃上清液。用 1 mL PBS 重悬沉淀并稀释,用0.22 μm膜过滤后,继续将样本转移至超高速离心管,在4 ℃、110 000×g继续离心75 min收集沉淀,用PBS重悬,并利用透射电镜和纳米颗粒跟踪分析技术(nanoparticle tracking analysis,NTA)对外泌体进行鉴定,其余外泌体置于-80 ℃保存。
1.3.2 总RNA提取
取外泌体样本用TRIzol法提取后在1%(质量分数)琼脂糖凝胶上监测RNA降解和污染,并利用NanoPhotometer®分光光度计和Qubit®2.0 荧光定量仪分别检测RNA纯度和浓度。最后使用Agilent Bioanalyzer 2100系统评估RNA完整性。
1.3.3 sRNA文库构建及测序
每个样品的总RNA量为3 μg。使用NEBNext®Multiplex Small RNA Library Prep Set for Illumina®(NEB, USA)生成测序文库,并将索引代码添加到每个样品的属性序列中。具体是将NEB 3′ SR Adaptor直接特异连接到miRNA、siRNA和piRNA的3′端,SR RT引物杂交到多余的3′ SR 接头,将单链DNA接头转化为双链DNA分子。5′ SR Adaptor连接到miRNA、siRNA和piRNA的5′端。然后利用M-MuLV逆转录酶合成首链cDNA。随后进行PCR扩增。PCR产物在8%(体积分数)聚丙烯酰胺凝胶(100V, 80 min)上纯化。回收140~160 bp(小非编码RNA长度加上3′和5′接头)的DNA片段,溶解于8 μL洗脱缓冲液中,在Agilent Bioanalyzer 2100系统上对文库质量进行评估。之后在cBot聚类生成系统上对样本进行聚类。聚类生成后,文库制备在Illumina HiSeq 2500平台上测序,生成50 bp的单端reads。
1.3.4 测序数据质量控制
使用Fastq软件筛选原始数据,去除山羊乳和绵羊乳外泌体miRNA样品溶液中低质量序列、接头污染序列和PolyA/T/G/C序列,获得纯净序列。同时计算原始数据的Q20、Q30和GC含量,然后从纯净序列中选择一定长度范围进行所有下游分析。
1.3.5 sRNAs定位及序列特征分析
利用Bowtie对照参考基因组数据,分析它们在基因组上的表达和分布。将sRNA与miRBase及Rfam数据库比对,对sRNAs进行鉴定,最终鉴定出的已知miRNAs用于后续分析。去除其中的rRNAs、tRNAs、snRNAs和snoRNAs等非miRNAs序列,同时对比对成功的非miRNAs序列进行数量统计。
1.3.6 已知miRNAs鉴定和新miRNAs预测
以基因组序列为参考,筛选出已知的miRNAs。整合 miREvo和mirdeep2软件,通过探索二级结构、Dicer酶切位点和未注释的sRNAs的最小自由能来预测新的miRNAs。同时对不同长度的已知miRNAs的首位碱基分布及各位点碱基分布情况进行统计。
1.3.7 靶基因预测与生物信息学分析
使用miRanda和RNAhybrid软件分别对山羊乳及绵羊乳外泌体中检测到的所有miRNAs进行靶基因预测并取交集,对预测到的靶基因进行GO富集分析和KEGG通路分析。
2 结果与分析
2.1 山羊乳和绵羊乳外泌体鉴定
如图1所示,在透射电镜下山羊乳和绵羊乳外泌体呈圆形、大小均匀、有典型的“茶托”结构。

a-山羊乳外泌体;b-绵羊乳外泌体
图1 透射电镜下的羊乳外泌体形态(×30 000)
Fig.1 Exosomes morphology under transmission electron microscopy (×30 000)
粒径分析结果如图2所示,山羊乳和绵羊乳外泌体粒径大多分布于50~150 nm,浓度分别为4.37×109和9.42×1010个/mL。以上结果与以往山羊乳外泌体形态报道[11, 17]基本相符。
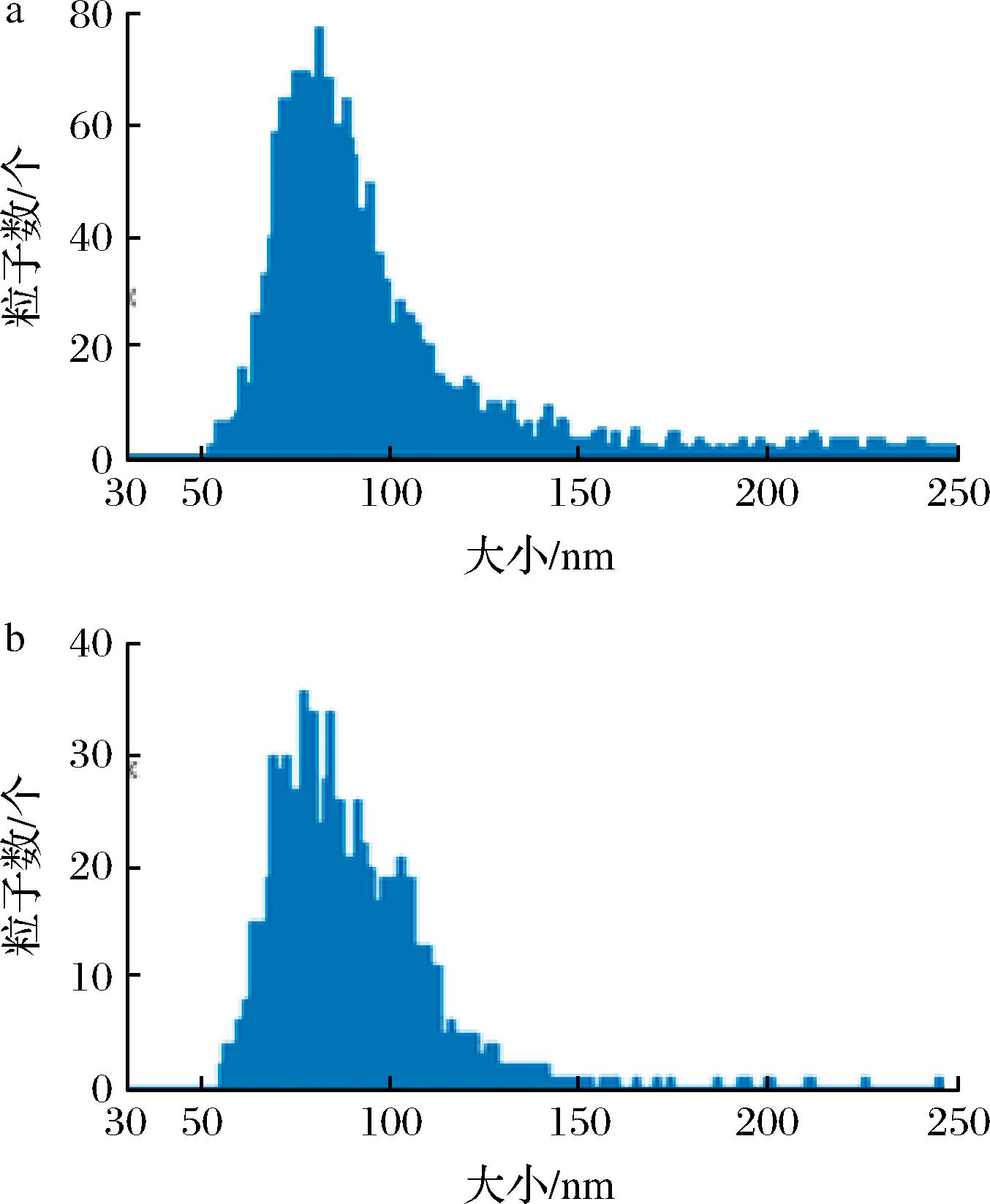
a-山羊乳外泌体;b-绵羊乳外泌体
图2 外泌体粒径分布
Fig.2 Exosomes particle size distribution
2.2 测序数据和质控结果
如表1所示,山羊乳和绵羊乳外泌体测序数据通过质控,分别得到纯净序列8 866 439条(98.37%)和8 210 089条(97.75%)。接下来,对山羊乳和绵羊乳外泌体长度区间为18~35 nt的sRNAs纯净序列进行后续分析。
表1 山羊乳及绵羊乳外泌体中测序数据统计结果
Table 1 Statistical results of sequencing data in goat milk and sheep milk exosomes
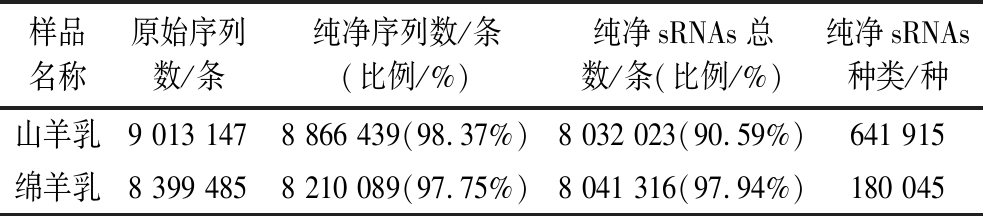
样品名称原始序列数/条纯净序列数/条(比例/%)纯净sRNAs总数/条(比例/%)纯净sRNAs种类/种山羊乳9 013 1478 866 439(98.37%)8 032 023(90.59%)641 915绵羊乳8 399 4858 210 089(97.75%)8 041 316(97.94%)180 045
2.3 羊乳sRNAs序列比对及miRNAs首位碱基偏好性
将长度筛选后的sRNAs定位到参考序列上(山羊:Capra hircus_Ensemble_97和NCBI_GCA_001704415.1_ARS1;绵羊:Ovis aries_Ensemble_97),分析了sRNAs和miRNAs在参考序列上的分布情况,结果如表2。通过比较发现,绵羊乳外泌体与基因组比对率为87.11%,而山羊乳外泌体的比对率仅为27.09%。
表2 山羊乳及绵羊乳外泌体中sRNA序列比对分析结果
Table 2 Comparison results of goat and sheep milk exosomes sRNA sequence and reference database

样品名称比对到的sRNA序列数/条(比例/%)比对到的miRNA前体数/条比对到的miRNA成熟体数/条山羊乳2 175 494 (27.09%) 34 843 34 687绵羊乳7 004 680 (87.11%)1 026 5411 024 143
碱基偏好性有助于了解miRNAs的加工作用机理与测序准确性。由图3可知,不论山羊乳还是绵羊乳,不同长度的已知miRNAs首位碱基均对U具有显著偏好性,其中绵羊乳外泌体miRNAs在25 nt对碱基A有显著偏好。

a-山羊乳外泌体;b-绵羊乳外泌体
图3 乳外泌体已知miRNAs的首位碱基偏好性
Fig.3 First base preference of known miRNAs of milk exosomes
2.4 已知miRNAs及预测的新miRNAs比对和表达情况
在山羊乳和绵羊乳外泌体miRNAs中,分别鉴定出161和80种成熟miRNAs,其中miR-148a、miR-26a和let-7b均在山羊乳和绵羊乳外泌体中表达量较高,miR-151只在山羊乳外泌体中检出(表3)。
表3 部分羊乳外泌体miRNAs表达情况
Table 3 Partial miRNAs expression levels of goat and sheep milk exosomes

山羊乳miRNAs表达量/%绵羊乳miRNAs表达量/%chi-miR-148a-3p77.97oar-miR-148a79.53chi-miR-21-5p4.16oar-miR-26a4.76chi-miR-26a-5p2.31oar-let-7b2.95chi-let-7b-5p1.98oar-let-7a1.82chi-miR-30a-5p1.52oar-miR-200c1.35chi-miR-146b-5p1.42oar-let-7i1.21chi-miR-200a0.90oar-miR-99a1.18chi-miR-223-3p0.85oar-miR-30a-5p1.14chi-miR-378-3p0.67oar-let-7f0.90chi-miR-186-5p0.49oar-miR-210.83chi-miR-151-3p0.48oar-miR-30d0.76chi-miR-30b-5p0.43oar-miR-200a0.73chi-miR-423-5p0.39oar-miR-200b0.61chi-miR-200c0.31oar-let-7g0.47chi-miR-191-5p0.31oar-let-7c0.41
此外,对新miRNAs进行预测。在山羊乳和绵羊乳中分别预测到了5和7种新miRNAs,其中表达量最高的分别是Novel-62和Novel-82,其成熟体序列分别是:Novel-62:UCAGGACCUGUUGUUUCU和Novel-82:UAGAAAGUUUCUUUGGGGUUUU。这些新miRNAs在一定程度上丰富了山羊和绵羊乳miRNA的序列信息。
2.5 靶基因预测及GO功能分析
对山羊乳及绵羊乳外泌体中所有miRNAs靶基因进行GO富集,发现山羊乳及绵羊乳外泌体miRNAs在细胞组成、生物学过程和分子功能层面均具有显著差异。例如:山羊乳外泌体miRNAs中有49.86%的靶基因参与组成细胞骨架(图4-a);而绵羊乳中有43.35% 参与各类细胞器的组成,如内质网系统、高尔基体等(图4-b)。
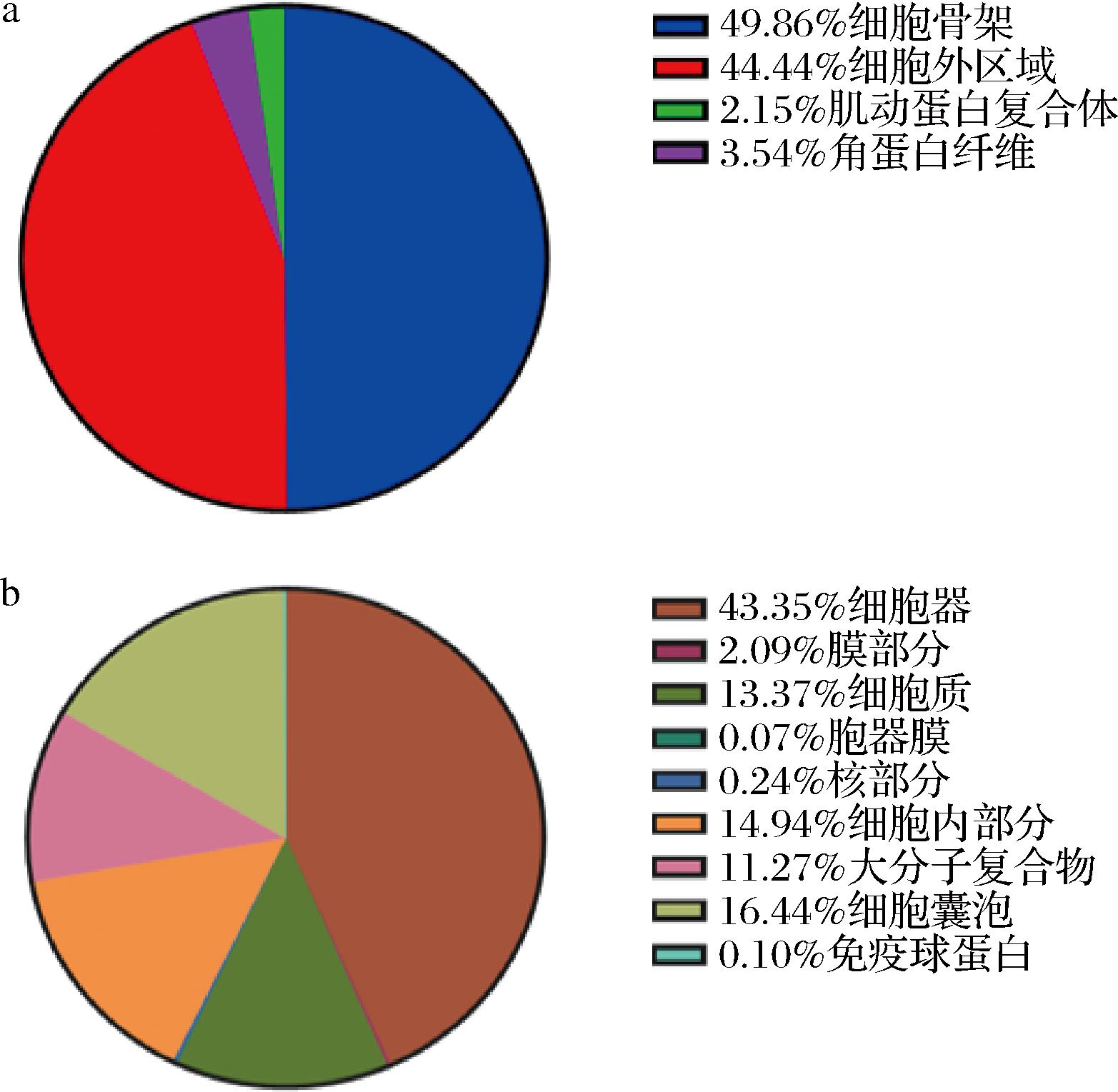
a-山羊乳外泌体;b-绵羊乳外泌体
图4 乳外泌体miRNAs靶基因在细胞组成层面的GO分析结果
Fig.4 GO analysis results of miRNAs target genes in milk exosomes at cell composition level
在生物学过程层面,山羊乳的种类丰富,可参与定位、运输、蛋白质修饰过程等(图5-a);绵羊乳外泌体miRNAs靶基因大多集中在代谢过程,占比82.77%(图5-b)。分子功能层面上,山羊乳外泌体miRNAs靶基因有13 706个,绵羊乳外泌体miRNA靶基因有571个。但可以看出,山羊乳及绵羊乳外泌体miRNAs靶基因蛋白质结合、催化活性和受体结合3种类型中均有富集(图6)。通过分析还发现:绵羊乳外泌体miRNAs靶基因还具有肠菌素结合的功能。研究发现肠菌素作为一种由肠杆菌分泌的细菌素,具有调节菌群数量的作用,还可通过与ATP合成酶互作来促进微生物对铁的吸收及宿主发育[18]。

a-山羊乳外泌体;b-绵羊乳外泌体
图5 乳外泌体miRNAs靶基因在生物学过程层面的GO分析结果
Fig.5 GO analysis results of miRNAs target genes in milk exosomes at the level of biological processes

a-山羊乳外泌体;b-绵羊乳外泌体
图6 乳外泌体miRNAs靶基因在分子功能层面的GO分析结果
Fig.6 GO analysis results of miRNAs target genes in milk exosomes at molecular functional level
2.6 靶基因通路富集分析
对山羊乳和绵羊乳外泌体miRNAs中的靶基因进行KEGG通路富集分析,图7展示了富集显著的20条通路。

a-山羊乳外泌体;b-绵羊乳外泌体
图7 乳外泌体miRNAs靶基因KEGG富集散点图
Fig.7 Milk exosomes miRNAs target gene KEGG enrichment distribution maps
山羊乳外泌体中miRNAs中共10 512个靶基因,被富集到274条通路,其中花生四烯酸代谢、醚脂质代谢、自然杀伤细胞介导的细胞毒性、α-亚麻酸代谢、Ras信号通路、甘油磷脂新陈代谢、轴突引导和移植物抗宿主病这8条通路获得显著富集(P<0.05),最为显著的是花生四烯酸代谢通路。
花生四烯酸是20碳链多不饱和脂肪酸,作为生物细胞膜的组成部分,包括环氧化酶(cyclooxygenase,COX)、脂氧合酶和细胞色素P450等3种途径。其中COX通路是动脉粥样硬化性和缺血性心脏病的主要治疗靶点之一,可能影响该疾病的病理生理特征,包括血小板聚集、血管壁张力和动脉粥样硬化病变中的炎症过程[19],这些将为开发心血管疾病和癌症的新治疗药物提供参考。
绵羊乳外泌体中miRNAs中共有1 067个靶基因被注释到256 条通路中,其中11条获得显著富集(P<0.05),分别是氨基酸的生物合成,碳代谢,果糖和甘露糖代谢,蛋白质消化吸收,癌症中的转录失调,叶酸生物合成,苯丙氨酸,酪氨酸和色氨酸的生物合成,吞噬体,核苷酸切除修复和NF-κB信号通路,最显著富集的通路为氨基酸的生物合成。氨基酸的合成是生物合成代谢的关键,与儿童生长发育、免疫系统完善具有重要作用,这提示绵羊乳外泌体miRNAs在以上方面或许具有独特的功能。
3 讨论
本研究利用超高速离心法对羊乳样本进行外泌体提取和鉴定,随后构建了山羊乳及绵羊乳外泌体的sRNA文库,并利用高通量测序技术研究了山羊乳和绵羊乳外泌体miRNAs的类型和表达量,最后对2种羊乳外泌体miRNAs的特点、靶基因参与的生物学过程进行分析。
在对miRNAs序列的鉴定分析时发现:山羊乳外泌体中鉴定出161种已知miRNAs和5种新型miRNAs序列;而在绵羊乳外泌体中鉴定出80种已知miRNAs和7种新型miRNAs序列。在已知miRNAs序列中,miR-148a、miR-26a和let-7b均在山羊乳和绵羊乳外泌体中表达量较高,其中miR-148a在2种羊乳样本中表达量最高,分别占77.97%和79.53%,而该miRNA也在人乳中高度表达,它可通过靶向TLR4介导的途径抑制NF-κB 和p65的激活,且在小鼠子宫内膜炎模型发现miR-148a的上调具有缓解炎症的作用[20]。miR-151只在山羊乳外泌体中表达,它通过靶向白细胞介素17A(IL-17A) 减弱动脉粥样硬化内皮细胞凋亡,是一种动脉粥样硬化治疗的潜在途径[21]。通过文献进行检索后发现,这些高表达miRNAs在减轻病原微生物感染、炎症、抑制癌细胞增殖迁移,甚至可作为缓解某些疾病的靶点或手段,这也提示上述羊乳外泌体miRNAs可能通过以上过程参与到了人体生理功能的调节过程。
此外,生物信息学分析时发现山羊乳和绵羊乳外泌体miRNAs的靶基因在分子功能性上也存在差异,如山羊乳miRNAs靶基因数量较多,集中在结合和催化活性等方面;绵羊乳外泌体miRNAs部分靶基因具有肠菌素结合的功能,且对宿主发育具有一定促进作用,上述结果提示山羊与绵羊乳外泌体miRNAs的差异化表达,使得不同羊乳外泌体可能具有不同功能,而这在今后研究中应当给予关注。
本研究也存在某些不足之处,例如:(1)本研究采用透射电镜和纳米颗粒跟踪分析技术对外泌体进行鉴定,由于目前对山羊与绵羊乳研究的局限,与特异性抗体的缺乏,难以通过免疫印迹等免疫学方法鉴定外泌体;(2)在miRNAs注释过程中发现,绵羊乳外泌体sRNAs总数为8 041 316,与参考序列比对之后有87.11%的序列可以被准确注释;虽然山羊乳外泌体sRNAs序列与绵羊乳相当,但仅有27.09%的序列被准确注释,本研究先后使用了Capra hircus_Ensemble_97和NCBI_GCA_001704415.1_ARS1两套基因组序列进行比对但结果依然未能改善。造成这一情况的可能原因包括:样本问题,即山羊乳样品杂合度高,在进行扩增时使得其他序列种类增多;二是基因组重复序列的复杂性,即山羊乳外泌体重复序列种类占比22.83%,而绵羊乳仅为3.27%;2个物种的RNA数据库信息尚不完善,也可能是造成注释比例偏低的原因。这方面应当在后续的研究中予以重视。虽然存在上述2点不足,但对于2类样本外泌体miRNAs的序列提取与表达分析仍然具有可比性。
羊乳营养丰富且与母乳成分较为接近,羊乳脂肪颗粒仅为牛奶的1/3,更利于人体吸收。近年来随着羊乳及其制品逐渐被大众接受,羊乳中功能成分的研究成为羊乳深加工与开发利用的关键。本研究构建了山羊乳和绵羊乳外泌体miRNAs表达谱,为不同类型羊乳外泌体的生理功能与各自优势提供基础数据,也为羊乳外泌体的后续研究提供参考。
[1] YANG Z C, LIU J, LIU H L, et al.Isolation and characterization of SSEA3+ stem cells derived from goat skin fibroblasts[J].Cellular Reprogramming, 2013, 15(3):195-205.
[2] WU X L, GAO F, WU Y N, et al.Isolation and biological characteristics of sheep amniotic epithelial cells[J].Cytotechnology, 2019, 71(2):539-551.
[3] ROGOSIC J, PFISTER J A, PROVENZA F D, et al.The effect of polyethylene glycol on intake of Mediterranean shrubs by sheep and goats1[J].Journal of Animal Science, 2008, 86(12):3491-3496.
[4] 王赛赛, 丁建平, 姜勋平.世界奶绵羊品种资源[J].养殖与饲料, 2022, 21(4):31-34.
WANG S S, DING J P, JIANG X P.World milk sheep breed resources[J].Animals Breeding and Feed, 2022, 21(4):31-34.
[5] CHILLIARD Y, DELAVAUD C, BONNET M.Leptin expression in ruminants:Nutritional and physiological regulations in relation with energy metabolism[J].Domestic Animal Endocrinology, 2005, 29(1):3-22.
[6] 李文斐, 张磊, 宋宇轩, 等.绵羊、山羊和牛乳的营养成分比较分析[J].食品工业科技, 2020, 41(24):286-291.
LI W F, ZHANG L, SONG Y X, et al.Comparative analysis of nutrients in sheep, goat and cow milk[J].Science and Technology of Food Industry, 2020, 41(24):286-291.
[7] 张磊, 刘晓瑞, 李文斐, 等.东佛里生绵羊奶的常规营养成分和脂肪酸分析[J].家畜生态学报, 2021, 42(7):24-27.
ZHANG L, LIU X R, LI W F, et al.Analysis of conventional nutrients and fatty acids of east Friensian milk sheep[J].Journal of Domestic Animal Ecology, 2021, 42(7):24-27.
[8] 赵丽丽, 葛武鹏, 宋宇轩, 等.绵羊奶组成和营养特性[J].中国食品学报, 2022, 22(1):413-423.
ZHAO L L, GE W P, SONG Y X, et al.The composition and nutrition characteristics in sheep milk[J].Journal of Chinese Institute of Food Science and Technology, 2022, 22(1):413-423.
[9] JOHNSTONE R M, ADAM M, HAMMOND J R, et al.Vesicle formation during reticulocyte maturation.Association of plasma membrane activities with released vesicles (exosomes)[J].Journal of Biological Chemistry, 1987, 262(19):9412-9420.
[10] JOHNSTONE R M.Revisiting the road to the discovery of exosomes[J].Blood Cells, Molecules, and Diseases, 2005, 34(3):214-219.
[11] MELNIK B C, STREMMEL W, WEISKIRCHEN R, et al.Exosome-derived microRNAs of human milk and their effects on infant health and development[J].Biomolecules, 2021, 11(6):851.
[12] LIAO Y L, DU X G, LI J, et al.Human milk exosomes and their microRNAs survive digestion in vitro and are taken up by human intestinal cells[J].Molecular Nutrition &Food Research, 2017, 61(11):10.1002/mnfr.201700082.
[13] RUBIO M, BUSTAMANTE M, HERNANDEZ-FERRER C, et al.Circulating miRNAs, isomiRs and small RNA clusters in human plasma and breast milk[J].PLoS One, 2018, 13(3):e0193527.
[14] XIE M Y, CHEN T, XI Q Y, et al.Porcine milk exosome miRNAs protect intestinal epithelial cells against deoxynivalenol-induced damage[J].Biochemical Pharmacology, 2020, 175:113898.
[15] AHMED F, TAMMA M, PATHIGADAPA U, et al.Drug loading and functional efficacy of cow, buffalo, and goat milk-derived exosomes:A comparative study[J].Molecular Pharmaceutics, 2022, 19(3):763-774.
[16] YENUGANTI V R, AFROZ S, KHAN R A, et al.Milk exosomes elicit a potent anti-viral activity against dengue virus[J].Journal of Nanobiotechnology, 2022, 20(1):317.
[17] SANTOS-COQUILLAT A, GONZ LEZ M I, CLEMENTE-MORAG
LEZ M I, CLEMENTE-MORAG N A, et al.Goat milk exosomes As natural nanoparticles for detecting inflammatory processes by optical imaging[J].Small, 2022, 18(6):e2105421.
N A, et al.Goat milk exosomes As natural nanoparticles for detecting inflammatory processes by optical imaging[J].Small, 2022, 18(6):e2105421.
[18] QI B, HAN M.Microbial siderophore enterobactin promotes mitochondrial iron uptake and development of the host via interaction with ATP synthase[J].Cell, 2018, 175(2):571-582.e11.
[19] WANG B, WU L J, CHEN J, et al.Metabolism pathways of arachidonic acids:Mechanisms and potential therapeutic targets[J].Signal Transduction and Targeted Therapy, 2021, 6(1):94.
[20] JIANG K F, YANG J, YANG C, et al.miR-148a suppresses inflammation in lipopolysaccharide-induced endometritis[J].Journal of Cellular and Molecular Medicine, 2020, 24(1):405-417.
[21] GUO Z, CAI X C, GUO X, et al.Let-7b ameliorates Crohn’s disease-associated adherent-invasive E coli induced intestinal inflammation via modulating Toll-like receptor 4 expression in intestinal epithelial cells[J].Biochemical Pharmacology, 2018, 156:196-203.