合成生物学是在工程学的指导下,通过生物元器件的标准化设计和构建,对生物体进行有目标的重设计和改造,以解决医药、环境、材料和能源等国计民生的重大问题[1-2]。在生物体重设计过程中,基因的精细表达调控是影响合成生物体构建的关键因素。因此,开发设计有效的生物控制元件是合成生物学研究的重要内容。核糖体结合位点(ribosome binding site, RBS)是指起始密码子ATG上游的一段富含嘌呤的非翻译区[3],又叫SD(Shine-Dalgarno)序列,是控制翻译起始和蛋白质表达的关键区域。随着合成生物学的发展,利用RBS工程对目标基因的表达进行精细调控,优化目标产物合成途径通量,构建具有高效生产能力的细胞工厂,已经逐渐成为合成生物学研究的热点。
谷氨酸棒杆菌(Corynebacterium glutamicum)是一种从土壤中分离的形态为短杆棒状,无芽孢的兼性好氧型的革兰氏阳性细菌,是公认的安全食品级菌株[4]。随着对谷氨酸棒杆菌研究的不断深入,谷氨酸棒状杆菌已经发展成为重要的工业底盘菌,广泛用于L-谷氨酸、L-赖氨酸、L-精氨酸等氨基酸[5],以及有机酸[6]、高级醇[7]和多聚物[8]的工业化生产中,其中L-谷氨酸和L-赖氨酸年产超过200万t[9]。近年来,谷氨酸棒杆菌也被开发用于虾青素等天然产物[10]合成过程中。在构建细胞工厂的过程中,往往需要对代谢途径进行精细和定量调控,以提高目的产物的合成效率,因此关于谷氨酸棒杆菌基因表达调控工具的研究也越来越受到关注。在前期研究中,本实验室通过设计人工随机序列,构建了合成启动子文库,并应用于L-苏氨酸合成途径的表达调控[11]。然而目前,关于谷氨酸棒杆菌RBS元件文库的报道仍然较少。WEI等[12]利用RBS元件文库优化策略,在谷氨酸棒杆菌中重建和优化了甘油利用途径,显著提升了工程菌株的甘油利用能力。WANG等[13]利用基于CRISPR/Cas系统的碱基编辑技术,在谷氨酸棒杆菌中建立了RBS原位调控方法,并先后对木糖代谢途径和番茄红素合成途径的基因表达进行调控,大幅提升了木糖的摄取速率和番茄红素产量。ZHANG等[14]以谷氨酸棒杆菌通用的SD序列构建了RBS文库,优化了莽草酸合成途径aroGBDE基因的表达水平,提升了莽草酸的产量。然而该研究采用低通量平板筛选方法,构建的RBS文库调控范围较窄,调节丰度较低,不利于谷氨酸棒杆菌基因的精细表达调控。因此,本研究设计构建了人工RBS文库,通过偶联流式细胞分选(fluorescence-activated cell sorting,FACS)技术对RBS文库进行高通量筛选,获得了一个调控范围宽,覆盖范围均匀的人工RBS文库。利用人工RBS文库对L-高丝氨酸合成途径的基因进行精细调控,提高了L-高丝氨酸的合成能力,为谷氨酸棒杆菌中基因的表达调控提供了重要研究工具。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒
大肠杆菌(Escherichia coli)DH5α用于质粒构建,由中国科学院天津工业生物技术研究所保藏。C.glutamicum ATCC 13032菌株由中国科学院天津工业生物技术研究所保藏。谷氨酸棒杆菌穿梭质粒pEC-XK99E用于RBS评价和筛选,由本实验室保藏。本研究所用菌株与质粒见表1。
表1 本研究所用质粒与菌株
Table 1 The plasmids and strains used in this study

菌株和质粒相关描述来源pXMJ19-GFP大肠杆菌和谷氨酸棒杆菌的穿梭质粒,GFP,Ptac,CmR实验室保存[11]pEC-XK99E大肠杆菌和谷氨酸棒杆菌的穿梭质粒,Ptrc,KanR实验室保存DH5α用作质粒克隆的宿主菌实验室保存C.glutamicumC.glutamicum ATCC 13032, wild type实验室保存Bacillus subtilisBacillus subtilis 168实验室保存CgH1C.glutamicum ATCC13032,ΔthrB实验室保存pEC-XK99E-ΔLacO-GFPDH5α, pEC-XK99E质粒,LacO缺失本研究amyE-4C.glutamicum ATCC 13032, pEC-XK99E-ΔLacO-RBS4-amyE质粒本研究amyE-10C.glutamicum ATCC 13032, pEC-XK99E-ΔLacO-RBS10-amyE质粒本研究amyE-20C.glutamicum ATCC 13032, pEC-XK99E-ΔLacO-RBS20-amyE质粒本研究amyE-23C.glutamicum ATCC 13032, pEC-XK99E-ΔLacO-RBS23-amyE质粒本研究amyE-37C.glutamicum ATCC 13032, pEC-XK99E-ΔLacO-RBS37-amyE质粒本研究amyE-33C.glutamicum ATCC 13032, pEC-XK99E-ΔLacO-RBS33-amyE质粒本研究1CgH1, pEC-XK99E-ΔLacO-RBS37-LysC-RBS37-Hom质粒本研究2CgH1, pEC-XK99E-ΔLacO-RBS37-LysC-RBS20-Hom质粒本研究3CgH1, pEC-XK99E-ΔLacO-RBS37-LysC-RBS4-Hom质粒本研究4CgH1, pEC-XK99E-ΔLacO-RBS20-LysC-RBS37-Hom质粒本研究5CgH1, pEC-XK99E-ΔLacO-RBS20-LysC-RBS20-Hom质粒本研究6CgH1, pEC-XK99E-ΔLacO-RBS20-LysC-RBS4-Hom质粒本研究7CgH1, pEC-XK99E-ΔLacO-RBS4-LysC-RBS37-Hom质粒本研究8CgH1, pEC-XK99E-ΔLacO-RBS4-LysC-RBS20-Hom质粒本研究9CgH1, pEC-XK99E-ΔLacO-RBS4-LysC-RBS4-Hom质粒本研究
1.1.2 培养基
LB培养基(g/L):酵母粉5.0,胰蛋白胨10.0,NaCl 10.0[固体培养基加2%(质量分数,下同)的琼脂粉];硫酸卡那霉素(kanamycin)终质量浓度为100 μg/mL。主要用于大肠杆菌的培养。
LBHIS培养基(g/L):酵母粉2.5,胰蛋白胨5.0,NaCl 5.0,脑心浸液1.85,山梨醇9.1(固体培养基加2%的琼脂粉);Kanamycin终质量浓度为50 μg/mL。主要用于谷氨酸棒杆菌的培养。
种子培养基(g/L):玉米浆30,尿素5,(NH4)2SO4 5,KH2PO4 1,MgSO4 0.5,葡萄糖25,苏氨酸0.4。
发酵培养基(g/L):玉米浆60,FeSO4-柠檬酸盐溶液0.55 mL/L(FeSO4·7H2O 20 g/L,柠檬酸18.14 g/L),(NH4)2SO4 40,KH2PO4 0.25,MgSO4 0.5,H3PO4(85%) 0.225 mL/L,维生素溶液7 mL/L(生物素300 mg/L,维生素B1 500 mg/L,泛酸钙盐2 g/L,烟酰胺600 mg/L),乙酸钾0.5,葡萄糖70,苏氨酸0.4,CaCO3 20。
1.1.3 试剂
Phusion DNA聚合酶、限制性内切酶Dpn I、限制性内切酶FastDigest Eco31 I、T4 DNA连接酶、蛋白Marker,Thermo Fisher公司;0.1% BSA,NEB公司;质粒提取试剂盒、胶回收试剂盒,天根生物公司;卡那霉素,索莱宝生物科技有限公司;其他试剂如3,5-二硝基水杨酸、酒石酸钾钠、可溶性淀粉、NaOH、苯酚、Na2SO3、葡萄糖、冰醋酸、盐酸等均为国产分析纯。
1.1.4 仪器与设备
Synergy NEO2多功能酶标仪,美国Biotek公司;摇床,美国精骐公司;Scientz-IID超声破碎仪,宁波新芝公司;高速冷冻离心机,德国Eppendoff公司;MoFlo XDP贝克曼分选型流式细胞仪,美国Beckman公司;Agilent 1260高效液相色谱仪,美国Agilent公司。
1.2 实验方法
1.2.1 谷氨酸棒杆菌人工RBS文库的构建
本研究根据文献报道的谷氨酸棒杆菌通用的SD序列AAAGGA,设计了AAAGGA(N)7-8的RBS随机序列,使SD序列距离起始密码子7或8 bp。设计随机引物,该引物包含Bsa I限制性内切酶识别位点、RBS随机序列以及报告基因gfp(green fluorescent protein, GFP)上游引物序列。引物序列具体为CCAGGTCTCAGAGCAAAGGANNNNNNN(N)ATGAGTAAAGGAGAAGAACTTTTCACTGGAG。采用Phusion DNA聚合酶,以gfp基因为模板,通过PCR方法获得含有人工RBS序列的gfp基因片段,利用Golden Gate DNA组装方法,将gfp基因片段整合连接至质粒pEC-XK99E上,转化到E.coli DH5α感受态中,涂布于含100 μg/mL kanamycin的LB平板上,37 ℃静置培养12 h。收集平板上的所有菌落,提取质粒,构建谷氨酸棒杆菌人工RBS序列文库。
1.2.2 RBS文库的高通量筛选
将构建的RBS文库质粒电转到C.glutamicum ATCC 13032感受态细胞中,涂布到含50 μg/mL kanamycin LBHIS的平板上,在30 ℃下培养24 h。用PBS(pH 7.5)冲洗平板上的细胞,取2 mL的菌液,按照1%(体积分数,下同)的接种量转接到LBHIS液体培养基中,30 ℃振荡培养12 h。随后收取2 mL菌液,用PBS洗涤菌体,重复2次,将菌液浓度稀释至OD600=0.1。将制备的样品振荡混匀5 min,使用MoFlo XDP流式细胞仪对样品细胞进行筛选,激发波长488 nm,发射波长530 nm,检测GFP荧光变化强度。为保证细胞阳性率,本研究采用纯化模式对RBS文库细胞进行流式细胞分选。将FACS技术分选出的细胞置于LBHIS的平板上,30 ℃摇床培养24 h。挑取平板上的细胞到含有600 μL LBHIS培养基的96深孔板中,采用多功能酶标仪检测GFP荧光强度变化,激发波长488 nm,发射波长520 nm。相对荧光强度(relative fluorescence unit, RFU)的计算如公式(1)所示:
相对荧光强度![]()
(1)
随后将分离的含有不同强度的GFP荧光的菌株,置于含有5 mL LBHIS培养基的试管中,进一步评价细胞的荧光强度。
1.2.3 人工RBS文库调控强度表征
1.2.3.1 质粒构建
本研究利用α-淀粉酶(1,4-α-D-glucan-glucanohydrolase, α-amyE)进一步测试人工RBS文库的调控强度。从人工RBS文库中挑选不同强度的RBS元件,嵌入到α-淀粉酶基因amyE上游引物上,采用Phusion DNA聚合酶,以枯草芽孢杆菌(Bacillus subtilis)基因组为模板,通过PCR方法获得包含不同RBS元件的amyE基因片段。利用Golden Gate方法,将含有不同RBS序列的amyE基因片段连接至载体pEC-XK99E上。将构建的载体转入C.glutamicum ATCC 13032感受态细胞中,构建含有不同强度的RBS序列的α-淀粉酶菌株。
1.2.3.2 α-淀粉酶的酶活测定
本研究采用3,5-二硝基水杨酸(3,5-dinitro-2-hydroxybenzoic acid, DNS)比色法测定还原糖含量来反映α-淀粉酶的酶活力[15]。挑取上述构建的含有不同强度的RBS序列的α-淀粉酶菌株,接种到含有50 μg/mL kanamycin的5 mL的LBHIS液体培养基中,过夜活化培养。按照1%的接种量转接到LBHIS液体培养基中,30 ℃、200 r/min振荡培养24 h。收集菌液,加入裂解缓冲液(20 mmol/L Na2HPO4,200 mmol/L NaCl,pH 7.5)重悬,调节OD600至10,冰水浴超声破碎处理20 min,随后在4 ℃、6 000 r/min离心10 min,收集上清液。
反应混合液包括:50 μL粗酶液,450 μL 20 mmol/L醋酸钠Buffer B(0.02 mol/L NaAC固体,0.2 mL/L冰醋酸,pH 5.5),500 μL 10 g/L可溶性淀粉(溶于醋酸钠Buffer B,现配现用)。将装有反应混合液的试管放置在60 ℃的水浴锅,准确反应5 min后,加入3滴0.5 mol/L HCl,中断反应,然后加入1.0 mL DNS试剂(0.87 mol/L酒石酸钾钠,0.03 mol/L DNS,0.52 mol/L NaOH,0.06 mol/L苯酚,0.04 mol/L Na2SO3),在90 ℃水浴锅中反应5 min,冰浴后冷却至室温,测定540 nm处的吸光值。α-淀粉酶的酶活单位定义为:在60 ℃条件下,1 min释放1 μmol葡萄糖所需要的酶量。
1.2.4 利用人工RBS文库精细调控L-高丝氨酸生物合成途径
本研究基于人工RBS文库对L-高丝氨酸生物合成途径的关键模块进行精细调控。天冬氨酸激酶(aspartate kinase, LysC)和高丝氨酸脱氢酶(homoserine dehydrogenase, Hom)是L-高丝氨酸生物合成途径的2个关键酶。为移除关键酶LysC和Hom的反馈抑制,本研究分别引入点突变LysCT311I和HomG378E,并从人工RBS文库中挑选3个不同强度的RBS元件,分别调控LysCr和Homr的表达。将RBS元件序列分别嵌合到lysC和hom基因上游引物上,以C.glutamicum基因组为模板,通过PCR方法获得含有不同RBS元件序列的lysC和hom基因DNA片段。通过用Golden Gate方法,将lysC和hom基因片段两两组合,连接到载体pEC-XK99E上,将上述构建的质粒转化至菌株CgH1中,构建L-高丝氨酸合成菌株。
1.2.5 L-高丝氨酸的发酵
挑取上述构建的高丝氨酸合成菌株,于LBHIS液体培养基中,30 ℃过夜活化培养。按照1%的接种量,接种到种子培养基中,30 ℃、200 r/min培养12 h。随后以初始OD600=10,接种到含有20 mL发酵培养基的250 mL摇瓶中,30 ℃、200 r/min进行发酵培养,每24 h取样,使用分光光度计测定菌株的生长情况OD600,使用HPLC测定发酵液中L-高丝氨酸的含量[16]。
2 结果与分析
2.1 谷氨酸棒杆菌人工RBS文库的设计与构建
研究表明,谷氨酸棒杆菌16S rRNA 3′末端的anti-Shine-Dalgarno序列为AAAGGAGG,被认为是通用RBS的保守序列。在谷氨酸棒杆菌中,RBS元件和起始密码子之间的距离大概是5~10 bp[17]。而ZHANG等[14]通过对RBS序列随机筛选,发现有效RBS元件的保守序列AAAGG与起始密码子之间的距离大多数为7~8 bp。基于上述结果,本研究设计了谷氨酸棒杆菌人工RBS随机序列为AAAGGA(N)7-8,其中AAAGGA为RBS元件的保守序列,为保证筛选效率,AAAGGA与起始密码子的距离设定为7~8 bp,如图1-a所示。将设计的人工RBS序列嵌合到报告基因gfp上游引物上,设计随机引物。以gfp基因为模板,通过使用设计的随机引物进行PCR扩增,获得含有随机RBS元件的gfp序列,采用Golden Gate的方法,将gfp片段插入到载体pEC-XK99E上,构建质粒文库(图1-b)。将质粒文库电转至C.glutamicum ATCC 13032感受态细胞中,建立谷氨酸棒杆菌人工RBS文库。通过随机引物设计,人工RBS文库的理论库容量为6.6×104。因此,本研究共挑选了大约105个克隆,保证RBS文库的100%覆盖。

a-人工RBS文库设计策略;b-人工RBS文库的构建;c-高通量筛选方法的建立
图1 人工RBS文库的设计、构建和高通量筛选
Fig.1 Design, construction and high-throughput screening of the artificial RBS library.
2.2 人工RBS文库的高通量筛选
本研究利用两步筛选法对人工RBS文库进行高通量筛选(图1-c)。流式细胞技术是比较先进的单细胞定量分析和分选技术,能够根据细胞形态和荧光信号,快速准确地完成细胞性状分析和分选工作,大大加快了菌株筛选进程。因此,本研究为加快筛选效率,首先利用流式细胞技术对人工RBS文库进行评价和分选。以谷氨酸棒杆菌常用的RBS元件AAAGGAGGTTGTC(CK1)和AAAGGAGGACAACC(CK2)为阳性对照,分别选择RBS文库中细胞荧光强度前1%和前0.04%的细胞(图2-a)。经过多轮筛选,共筛选获得10 000个克隆,置于LBHIS琼脂平板上30 ℃培养48 h。为了验证高通量筛选效率,从平板上挑选3个具有不同荧光强度的菌株,利用流式细胞仪对菌株荧光强度进行评价。结果如图2-b所示,筛选的菌株荧光强度均显著高于对照组CK1,说明基于流式细胞技术的高通量筛选方法具有较好的筛选效率。随后本研究利用96孔板筛选法对RBS文库进行再次筛选和评价。挑取筛选的平板菌落,置于含有LBHIS培养基的96孔板中,30 ℃培养18 h,采用多功能酶标仪对培养细胞的荧光强度进行分析。结果表明,通过两步筛选方法构建的人工RBS文库,调控范围广,调控强度分布均匀,荧光强度从3×103~105 a.u.,具有较好的调节强度多样性(图2-c)。
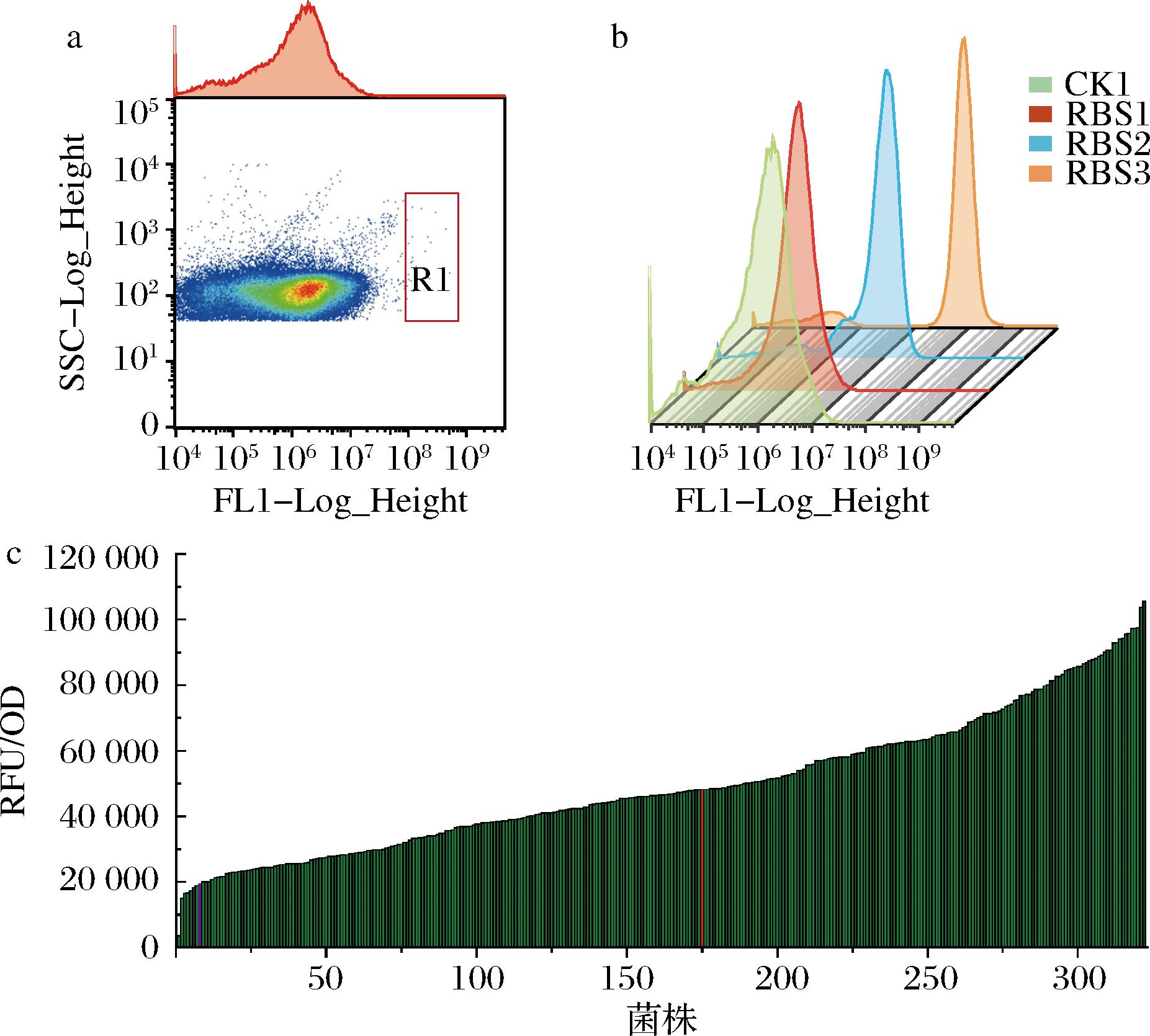
a-利用流式细胞技术高通量筛选人工RBS文库(R1为流式细胞分选框);b-流式细胞分析验证高通量筛选结果的可靠性;c-利用96孔筛选技术对人工RBS文库进行复筛
图2 利用两步筛选法对人工RBS文库进行高通量筛选
Fig.2 High throughput screening of the artificial RBS library using two-step screening method
注:RFU/OD为20 000和50 000处的紫色和红色线条分别代表CK1和CK2的荧光强度。
2.3 人工RBS文库的评估与表征
为评估人工RBS元件的稳定性,本研究根据荧光强度,从构建的人工RBS文库中挑选了37个RBS元件进行放大验证。所选37个RBS序列见表2。将选择的含有不同RBS元件的菌株,接种于含有5 mLLBHIS培养基的试管中,30 ℃、200 r/min培养18 h。随后采用多功能酶标仪测定培养细胞的荧光强度。结果如图3-a所示,选择的RBS元件在放大培养条件下,其荧光强度变化趋势与96孔板的测定结果基本一致,说明构建的人工RBS元件文库具有较高的稳定性。选择的RBS元件调控范围广,最高荧光强度为105 508 a.u.,是对照的8.55倍,最低荧光强度为3 633 a.u.,是对照的0.29倍,调控幅度为29.04倍。根据荧光强度不同,将RBS文库分为3类,高强度RBS(荧光强度>80 000),中强度RBS(荧光强度为80 000~40 000),低强度RBS(荧光强度<40 000)。

a-人工RBS文库稳定性测试;b-α-淀粉酶表达系统的建立;c-利用α-淀粉酶表达系统评估人工RBS文库的通用性
图3 人工RBS文库的评估与表征
Fig.3 Evaluation and characterization of the artificial RBS library
表2 37个RBS序列及相对强度
Table 2 37 RBS sequences and relative intensity

注:红色字体为筛选得到的随机序列。
低强度中强度高强度编号RBS序列(5′-3′)相对强度编号RBS序列(5′-3′)相对强度编号RBS序列(5′-3′)相对强度CK1AAAGGAGGTTGTC-11AAAGGAGTCACTG3.7326AAAGGAGGCTTGTA6.751AAAGGAAGCTCGG0.2912AAAGGAGCAGGCT4.1027AAAGGAGATATTC6.842AAAGGAACCA0.6613AAAGGAGTGGGAT4.5128AAAGGAGAGAATTC7.023AAAGGAGGTCTGTC0.7214AAAGGAGTAACTC4.9729AAAGGAAGGTATTT7.044AAAGGAATTC1.2215AAAGGAGCATTTGC5.0330AAAGGAGACTCATT7.105AAAGGAGTGGCTC1.8716AAAGGAGTTTGTGC5.0931AAAGGAGTGGAAGC7.146AAAGGAATGGGCG1.9017AAAGGAGTAGTTAA5.1432AAAGGAGAAAGTC7.527AAAGGAGATCGCCG2.4918AAAGGAAGGCAGC5.2533AAAGGAGTGAAGC7.628AAAGGAATTCATG2.9819AAAGGAAGGGCGT5.7734AAAGGAGAGGTACT7.649AAAGGAGGCTCGAA3.1520AAAGGAGGCTTT5.7735AAAGGAGAGCCTTT7.7410AAAGGACCCCTTTC3.1621AAAGGAGTATATT5.8436AAAGGAGAGGTTGC7.8822AAAGGAAGGAATC5.9837AAAGGAGTAAAGGT8.5523AAAGGAGGTATAAG6.1224AAAGGAGACAGGAC6.2325AAAGGATGGAGAA6.38
为进一步验证人工RBS文库的通用性,本研究利用α-淀粉酶表达系统对RBS元件文库进行评估(图3-b)。分别从3个不同强度的RBS文库中各挑选2个序列,连接至α-淀粉酶上游,调控α-淀粉酶的表达。将上述构建的不同RBS序列和α-淀粉酶的质粒转入谷氨酸棒杆菌中,于LBHIS培养基中,30 ℃、200 r/min振荡培养24 h,测定α-淀粉酶的酶活性。结果如图3-c所示,含有高强度RBS元件菌株表现出较高的α-淀粉酶活性,含有低强度RBS元件菌株表现出较低的α-淀粉酶活性,与GFP荧光强度具有较高的一致性。其中含有高强度RBS元件37-amyE菌株的α-淀粉酶活性为3.00 U/mg,是对照的4.43倍,含有低强度RBS元件4-amyE菌株的α-淀粉酶活性为0.70 U/mg,是对照的1.04倍。上述结果表明,本研究构建的人工RBS元件文库具有较宽的调控范围,较好的稳定性和通用性,为谷氨酸棒杆菌代谢改造和基因表达调控提供了多样性的系列调控元件。
2.4 利用人工RBS文库调控L-高丝氨酸的生物合成途径
L-高丝氨酸是一种重要的非蛋白质氨基酸[17],在食品、饲料、化工和医药领域具有广泛的应用。L-高丝氨酸是合成苏氨酸等天冬氨酸家族氨基酸的重要前体物质,具有重要的生理功能,也是一种重要的化工原料,用于L-草铵膦[18]和γ-丁内酯[19]的生产。近年来,随着L-高丝氨酸需求量的逐渐增大,利用环境友好的微生物发酵法生产L-高丝氨酸受到广泛关注。在构建微生物细胞工厂的过程中,对代谢途径通量的平衡调控是影响目标产物合成的关键因素。研究表明,天冬氨酸激酶(aspartate kinase, LysC)和高丝氨酸脱氢酶(homoserine dehydrogenase, Hom)是谷氨酸棒杆菌中L-高丝氨酸生物合成途径的2个关键酶。因此,本研究利用人工RBS文库精细调控L-高丝氨酸生物合成途径中关键酶LysCr和Homr的表达,平衡代谢途径通量,提高L-高丝氨酸合成能力。
本研究从构建的人工RBS元件文库选择了3个具有不同强度的RBS序列,即高强度RBS-37、中强度RBS-20、低强度RBS-4。将3个具有不同强度的RBS元件分别置于基因lysC和hom的上游,构建含有不同强度RBS的基因表达元件。将获得的lysC和hom基因表达元件通过Golden Gate方法,连接至pEC-XK99E上。通过排列组合方法,构建了9个含有lysC和hom基因的表达质粒。将构建的表达质粒电转化至谷氨酸棒杆菌CgH1菌株中,构建系列L-高丝氨酸合成菌株(图4-b)。
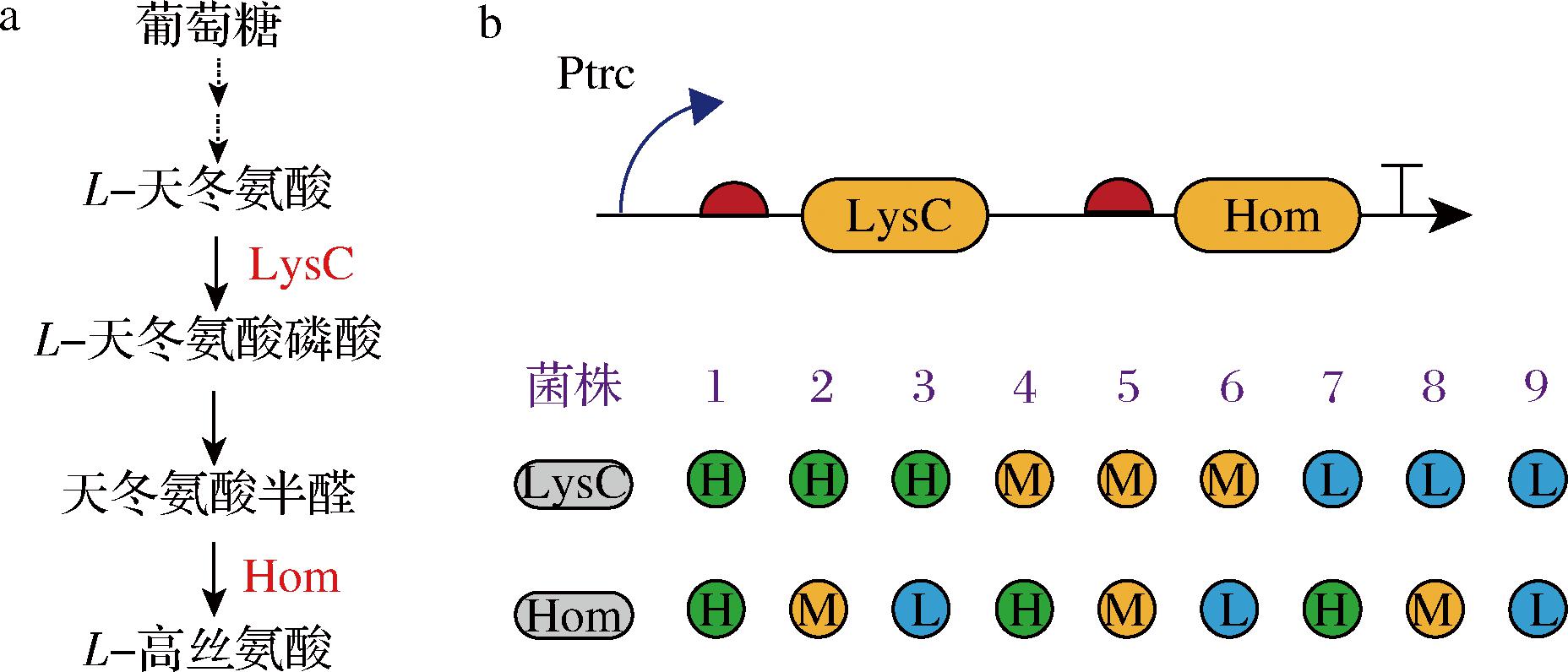
a-L-高丝氨酸生物合成途径;b-利用人工RBS文库精细调控关键酶LysC和Hom表达示意图
图4 利用人工RBS文库精细调控L-高丝氨酸的生物合成途径
Fig.4 Fine- regulation of the key enzymes expression in L-homoserine biosynthesis pathway using artificial RBS library
按照1.2.6节所述方法,以CgH1包含空质粒为对照,对构建的L-高丝氨酸合成菌株进行摇瓶发酵,测定LysCr和Homr组合表达对L-高丝氨酸产量的影响。如图5所示,与对照菌株相比,1号菌株生长略低于对照菌株,而其他菌株生长与对照基本相当。相比之下,各菌株的L-高丝氨酸合成能力存在明显差异,其中4号菌株表现出最高的L-高丝氨酸合成能力,L-高丝氨酸产量到达12.7 g/L,比野生型菌株高丝氨酸含量(3.5 g/L)提高了3.6倍。而低表达LysCr和Homr的菌株,其高丝氨酸含量最低,仅为5.7 g/L。结果表明,精细调控关键酶LysCr和Homr的表达,能有效平衡代谢途径通量,提升L-高丝氨酸的合成能力。而本研究构建的人工RBS文库为谷氨酸棒杆菌中基因的表达调控提供了有力工具。

a-L-高丝氨酸合成菌株的生长情况;b-L-高丝氨酸产量
图5 人工RBS文库精细调控关键酶的表达对L-高丝氨酸产量的影响
Fig.5 Effects of fine-regulation of key enzymes expression using artificial RBS library on L-homoserine production
3 结论
本研究基于谷氨酸棒杆菌RBS序列的基本特征,设计构建了一个随机RBS文库。以GFP为报告基因,偶联FACS技术和96孔板筛选技术,对随机RBS文库进行高通量筛选,成功构建了调控范围广,覆盖范围均匀的人工RBS元件文库,调控范围达到29.04倍。通过α-淀粉酶活力测试和表征,验证了人工RBS文库具有较高的稳定性和通用性。随后本研究将人工RBS文库应用于L-高丝氨酸代谢途径的调控优化。利用具有不同强度的RBS元件对L-高丝氨酸合成途径关键酶LysCr和Homr进行精细表达调控,获得了具有高效L-高丝氨酸合成能力的最佳组合,平衡了代谢途径通量,提高了L-高丝氨酸产量。与之前的研究相比,本研究建立的RBS文库调节宽度大,调控强度范围广,具有较好的普适性,能够“即插即用”,可直接对代谢途径中各个基因进行精细表达调控,为谷氨酸棒杆菌代谢改造和基因精细表达调控提供了有力工具。在下一步研究中,我们将利用不同强度的启动子库与RBS元件文库进行组合,建立具有不同强度的精细调控工具,并应用于其他生物合成代谢途径的精细调控研究。
[1] 严伟, 信丰学, 董维亮, 等.合成生物学及其研究进展[J].生物学杂志, 2020, 37(5):1-9.
YAN W, XIN F X, DONG W L, et al.Synthetic biology and research progress[J].Journal of Biology, 2020, 37(5):1-9.
[2] CHEN Y, BANERJEE D, MUKHOPADHYAY A, et al.Systems and synthetic biology tools for advanced bioproduction hosts[J].Current Opinion in Biotechnology, 2020, 64:101-109.
[3] HANCOCK J M.Ribosome Binding Site (RBS)[M].New York:American Cancer Society, 2014:1-14.
[4] 刘莫识, 刘娇, 孙冠男, 等.谷氨酸棒杆菌人工合成启动子文库的构建及应用[J].生物工程学报, 2022, 38(2):831-842.
LIU M S, LIU J, SUN G N, et al.Construction and application of a synthetic promoter library for Corynebacterium glutamicum[J].Chinese Journal of Biotechnoligy, 2022, 38(2):831-842.
[5] KINOSHITA S, NAKAYAMA K, AKITA S.Taxonomical study of glutamic acid accumulating bacteria, Micrococcus glutamicus nov.sp[J].Bulletin of the Agricultural Chemical Society of Japan, 1958, 22(3):176-185.
[6] MCKINLAY J B, VIEILLE C, ZEIKUS J G.Prospects for a bio-based succinate industry[J].Applied Microbiology and Biotechnology, 2007, 76(4):727-740.
[7] NIIMI S, SUZUKI N, INUI M, et al.Metabolic engineering of 1, 2-propanediol pathways in Corynebacterium glutamicum[J].Applied Microbiology and Biotechnology, 2011, 90(5):1721-1729.
[8] SONG Y Y, MATSUMOTO K, YAMADA M, et al.Engineered Corynebacterium glutamicum as an endotoxin-free platform strain for lactate-based polyester production[J].Applied Microbiology and Biotechnology, 2012, 93(5):1917-1925.
[9] EGGELING L, BOTT M.A giant market and a powerful metabolism:L-lysine provided by Corynebacterium glutamicum[J].Applied Microbiology and Biotechnology, 2015, 99(8):3387-3394.
[10] HENKE N A, WIEBE D, PÉREZ-GARC A F, et al.Coproduction of cell-bound and secreted value-added compounds:Simultaneous production of carotenoids and amino acids by Corynebacterium glutamicum[J].Bioresource Technology, 2018, 247:744-752.
A F, et al.Coproduction of cell-bound and secreted value-added compounds:Simultaneous production of carotenoids and amino acids by Corynebacterium glutamicum[J].Bioresource Technology, 2018, 247:744-752.
[11] WEI L, XU N, WANG Y R, et al.Promoter library-based module combination (PLMC) technology for optimization of threonine biosynthesis in Corynebacterium glutamicum[J].Applied Microbiology and Biotechnology, 2018, 102(9):4117-4130.
[12] WEI L, ZHAO J H, WANG Y R, et al.Engineering of Corynebacterium glutamicum for high-level γ-aminobutyric acid production from glycerol by dynamic metabolic control[J].Metabolic Engineering, 2022, 69:134-146.
[13] WANG Y, CHENG H J, LIU Y, et al.In-situ generation of large numbers of genetic combinations for metabolic reprogramming via CRISPR-guided base editing[J].Nature Communications, 2021, 12(1):678.
[14] ZHANG B, ZHOU N, LIU Y M, et al.Ribosome binding site libraries and pathway modules for shikimic acid synthesis with Corynebacterium glutamicum[J].Microbial Cell Factories, 2015, 14(1):1-14.
[15] 朱海霞, 石瑛, 张庆娜, 等.3, 5-二硝基水杨酸(DNS)比色法测定马铃薯还原糖含量的研究[J].中国马铃薯, 2005, 19(5):266-269.
ZHU H X, SHI Y, ZHANG Q N, et al.Applying 3, 5-dinitrosalicylic acid(DNS) method to analyzing the content of potato reducing sugar[J].Chinese Potato, 2005, 19(5):266-269.
[16] 赵金花, 魏亮, 徐宁, 等.双酶偶联法测定发酵液中L-高丝氨酸的含量[J].食品与发酵工业, 2023, 49(2):239-245.
ZHAO J H, WEI L, XU N, et al.Determination of L-homoserine in fermentation broth by double enzyme coupling method[J].Food and Fermentation Industries, 2023, 49(2):239-245.
[17] PFEIFER-SANCAR K, MENTZ A, RÜCKERT C, et al.Comprehensive analysis of the Corynebacterium glutamicum transcriptome using an improved RNAseq technique[J].BMC Genomics, 2013, 14:888.
[18] JEON J, LEE J Y, LEE C, et al.L-Glufosinate intermediate and L-glufosinate preparation method:US, US2022024955A1[P].2022-01-27.
[19] HONG K K, KIM J H, YOON J H, et al.O-succinyl-L-homoserine-based C4-chemical production:Succinic acid, homoserine lactone, γ-butyrolactone, γ-butyrolactone derivatives, and 1, 4-butanediol[J].Journal of Industrial Microbiology &Biotechnology, 2014, 41(10):1517-1524.