沙门氏菌是最常见的食源性致病菌之一,能够导致肠胃炎和败血症等疾病。据世界卫生组织估计,在全球范围内沙门氏菌每年造成约1.53亿例肠道感染病例以及5.7万人死亡[1]。在中国,沙门氏菌也成为了细菌性食源性疾病的第二大常见原因[2],肠炎沙门氏菌(Salmonella enterica serovar Enteritidis)是其中重要的血清型之一,主要通过禽类及其副产品等食品传播感染[3]。
随着抗生素的广泛甚至不合理使用,细菌耐药性普遍增强给细菌感染的控制带来挑战。据2018年中国细菌耐药性监测(China antimicrobial surveillance network,CHINET)的报道,细菌耐药性整体呈上升趋势,耐药菌的分离比2017年增加了28.7%[4]。国内外研究发现沙门氏菌主要对四环素、氨苄西林、磺胺类与链霉素等抗生素药物表现出较强的耐药性[5]。其中,氨苄西林作为第一种广谱β-内酰胺类抗生素,可通过抑制细菌细胞壁的合成发挥作用[6],在细菌中的耐药状况较为严重[7]。细菌主要通过修饰青霉素结合蛋白、生产β-内酰胺酶、改变膜的通透性以及促进外排泵等方式提高对氨苄西林的耐药性[8],而耐药基因则通过染色体基因突变与水平基因转移等方式获得[9]。水平基因转移既是细菌整体耐药性快速上升的重要原因,也是耐药基因传播的主要手段,耐药基因由质粒、转座子等载体以低于染色体基因突变的成本,赋予细菌耐药的特性[10],而肠炎沙门氏菌中含有许多的耐药质粒。
细菌在获得耐药性后通常会发生生长速率下降及对不利环境的抗性降低,这种现象称为适应性代价[11]。也有研究发现耐药性的获得会诱导细菌对常见的食品相关压力产生交叉抗性,导致细菌在食品链中具有更强的存活能力[12],造成食品安全隐患。对于氨苄西林此类作用于细胞壁的抗生素,细菌可通过一系列信号通路的协调反应改变细菌的代谢和抗性,加之不同种类细菌间的异质性以及抗生素作用机制不同,变化有待进一步探究。因此,本研究将通过质粒导入的方式赋予肠炎沙门氏菌氨苄西林的耐药特征,并检测转化后菌株的耐药性、生长特性、热与酸抗性的变化,以期为沙门氏菌的风险评估以及在食品加工环节中的防治提供参考,从而保障食品安全。
1 材料与方法
1.1 试验材料
实验中所用的5株抗生素敏感肠炎沙门氏菌及含pKD46的大肠埃希氏菌为本实验室保存。野生型肠炎沙门氏菌均分离于食品,1、4号来自鸡肉,2、3、5号来自猪肉。经药敏测试发现对包括氨苄西林、头孢唑啉、四环素、亚胺培南等在内17种抗生素药物敏感。100 μg/mL的LA培养基与培养液由1 000 mL灭菌的LB培养基与培养液中加入100 mg/mL的氨苄西林原液1 mL配制而成。
LB培养基,青岛海博生物有限公司;SOC培养液、MH培养液(Mueller-Hinton Broth),上海生工生物工程有限公司;质粒小量提取试剂盒,上海捷瑞生物工程有限公司;甘油、盐酸(分析纯),国药集团化学试剂有限公司;氨苄西林原液,北京博奥森生物科技有限公司;GN4F革兰氏阴性菌药敏板,美国赛默飞世尔科技有限公司。
Bioscreen C全自动微生物生长曲线分析仪,芬兰Oy Growth Curves Ab公司。
1.2 实验方法
1.2.1 菌株活化
野生沙门氏菌(WT)在LB培养液中于37 ℃,静置培养24 h到达稳定期。含有pKD46质粒的大肠埃希氏菌和质粒转化后的沙门氏菌在含有100 μg/mL氨苄西林的LA培养基中培养,30 ℃静止培养至稳定期。
1.2.2 质粒提取与感受态细胞制备
大肠埃希氏菌活化后,根据上海捷瑞生物工程有限公司质粒小量提取试剂盒说明书步骤提取pKD46质粒。使用电转法进行沙门氏菌转化,采用甘油洗涤法制备感受态细胞。将活化后的沙门氏菌在LB培养液中培养至OD值为0.5。将培养完成的菌液置于冰上冷却30 min后分装至15 mL离心管中,4 ℃下2 500 r/min离心10 min,除去上清液。再用双蒸水重悬菌体,离心去除上清液。随后再用预冷的10%(体积分数)甘油,重悬离心2次以至彻底去除菌液中的离子。最后加入500 μL预冷的10%(体积分数)甘油重悬。
1.2.3 质粒转化
采用电转法转化沙门氏菌,先取3 μL质粒与0.2 cm电击杯置于冰上预冷,再将质粒与感受态细胞混合均匀,置于电击杯中,轻轻敲击杯壁使混合液体进入杯底。将电转仪调至2.5 kV,电击后,迅速加入1 mL的SOC溶液重悬细胞,轻轻吹吸数次后转移至2 mL离心管中。将转化后的细胞悬液在30 ℃下220 r/min振荡复苏2 h。取100 μL复苏菌液于LA平板上涂布,培养观察转化情况。挑取单菌落于LA培养液中进行培养,培养至稳定期与50%(体积分数)甘油混合保存于-80 ℃下,为后续试验所用。
1.2.4 最小抑菌浓度(minimal inhibitory concentration,MIC)值测定
使用革兰氏阴性菌药敏板对转化后的沙门氏菌(pKD46)对多种抗生素的MIC值进行测定。从LA培养基上挑取3~5个生长正常的单菌落,在无菌水中乳化,充分混合并调为0.5麦氏浊度。取10 μL配制好的菌液与10 mL的MH培养液混合,移至药敏板中,每孔30 μL。将药敏板于37 ℃下静置培养24 h,观察孔中是否生长,并参照美国临床和实验室标准化委员会(Clinical and Laboratory Standardization, CLSI)对抗生素耐药性进行判断。
1.2.5 生长特性测定
对野生及转化后沙门氏菌在25、30、35 ℃下的生长特性,包括最大比生长速率(μmax)和延滞期(λ)进行测定。将培养至稳定期的菌液进行梯度稀释,每个浓度取200 μL于100微孔板中在全自动微生物生长曲线分析仪(BioscreenC)测定生长状态,温度设定为25、30、35 ℃,中等强度振动,每10 min检测1次OD600。通过不同稀释梯度的菌液浓度与检测时间(time to detection,Tdet)做线性回归计算最大比生长速率μmax,检测时间即定义为初始的OD值至达到菌液浓度107 CFU/mL时所对应OD值的时间,计算如公式(1)所示:
(1)
式中:slope,斜率。
基于以上公式所求μmax值,根据BARANYI等[13]提出的方法计算延滞期λ,计算如公式(2)所示:
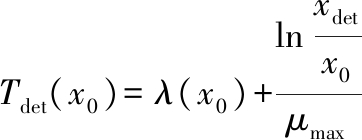
(2)
式中:x0,初始菌量,CFU/mL;xdet,达到检测时间的菌数,CFU/mL;Tdet,OD值达到107 CFU/mL浓度的时间,h;μmax,对数期细菌生长的最大比生长速率,lg CFU/h;λ,生长延滞期,h。
1.2.6 热失活特性测定
采用WANG等[14]的方法对菌在55 ℃/12 min、57.5 ℃/3 min、60 ℃/12 s的处理下观察存活情况。取培养至稳定期的菌液30 μL于PCR管中,按照对应的温度与处理时间在PCR仪中从37 ℃开始升温,处理结束后迅速将PCR管置于冰水中停止热失活,对初始菌液及热失活后菌液进行梯度稀释并进行平板计数。以近似D值作为热失活表征参数,计算如公式(3)所示:
(3)
式中:t,热处理时间,min;N0,初始菌浓度,CFU/mL;Nt,热处理后菌浓度,CFU/mL。
1.2.7 酸失活特性测定
依据沙门氏菌在酸化培养液中处理后细菌对数减少量判断耐酸情况,实验中采用1%(体积分数)盐酸将培养液pH调至3.0。取1 mL培养至稳定期的沙门氏菌至酸化的LB培养液中,4 ℃培养18 h后,3 600 r/min离心5 min,用普通的LB培养液重悬离心2次,沉淀菌体用1 mL的LB重悬后于梯度稀释并在平板上计数,对比耐药质粒导入前后的对数减少值。
1.2.8 统计分析
每组试验重复不少于2次,计算平均值及标准差。单因素方差分析(one-way analysis of variance,ANOVA)和图基(Tukey’s)检验对不同耐药表型沙门氏菌之间的生长延滞期、最大比生长速率、热失活特性及酸抗性的差异进行显著性分析。试验采用95%置信限,P<0.05则差异有统计学意义。所有数据均通过SPSS 25进行分析。
2 结果与分析
2.1 耐药沙门氏菌MIC测定
经耐药质粒转化后共获得5株沙门氏菌耐药菌株。革兰氏阴性菌药敏板对质粒导入后的沙门氏菌进行了16种抗菌药物的MIC测定,表1展示了其中MIC值发生变化的6种抗生素耐药情况。整体而言,经质粒导入氨苄西林耐药基因不易导致广泛的交叉耐药。其中,转化后的沙门氏菌对氨苄西林/舒巴坦钠、头孢唑啉与替卡西林/克拉维酸的MIC值普遍增加;米诺环素与呋喃妥因的MIC值普遍下降,1号与5号菌对氨苄西林/舒巴坦钠的耐药表型和1号、4号与5号对头孢唑啉的耐药表型均从敏感变为耐药。
表1 质粒导入前后肠炎沙门氏菌对不同抗生素的MIC值
Table 1 The MIC of different antibiotics aganist S. Enteritidis before and after plasmid introduction

注:A/S2-氨苄西林/舒巴坦钠;FAZ-头孢唑啉;MIN-米诺环素;TIM2-替卡西林/克拉维酸;SXT-甲氧苄啶/磺胺甲恶唑;NIT-呋喃妥因;LEVO-左氧氟沙星;括号内为药物敏感性为中介时的抗生素浓度。
菌株处理抗生素/(μg/mL)A/S2(8/4^32/16)FAZ(2^8)MIN(4^16)TIM2(16/4^128/4)SXT(2/38^4/76)NIT(32^128)1WT<4/2<14<8/2<2/3864pKD4632/1616264/28/156<322WT<4/228<8/2<2/38<32pKD4616/82816/2<2/38<323WT<4/224<8/2<2/3864pKD4616/84216/2<2/38<324WT<4/248<8/2<2/3864pKD468/432832/2<2/38<325WT<4/224<8/2<2/3864pKD4632/168264/24/76<32
2.2 生长特性分析
质粒转化后的沙门氏菌整体生长特性存在较为显著的变化(图1)。在3种温度条件下,沙门氏菌的平均生长延滞期λ均发生了显著的延长(P<0.05),25 ℃时由3.02 h延至5.59 h;30 ℃时由2.09 h延至4.48 h;35 ℃时由2.27 h延至3.02 h。而对于最大比生长速率μmax的平均值在转化前后则无显著差异。

a-生长延滞期;b-最大比生长速率
图1 野生型与耐药型肠炎沙门氏菌延滞期与最大比生长速率
Fig.1 Lag time and maximum specific growth rate of S.Enteritidis for different phenotypes:WT and pKD46
注:*表示差异显著(P<0.05),**表示差异极显著(P<0.01),下同。
2.3 热抗性分析
质粒转化后的5株耐药沙门氏菌,在3种温度条件下近似D值均呈现下降趋势(图2),即耐药沙门氏菌对热的抗性降低。其中,平均近似D值在55 ℃下由4.76 min降至3.35 min,差异不显著;在57.5 ℃下由1.70 min显著降至0.64 min(P<0.01);在60 ℃下由0.77 min显著降至0.24 min(P<0.01)。
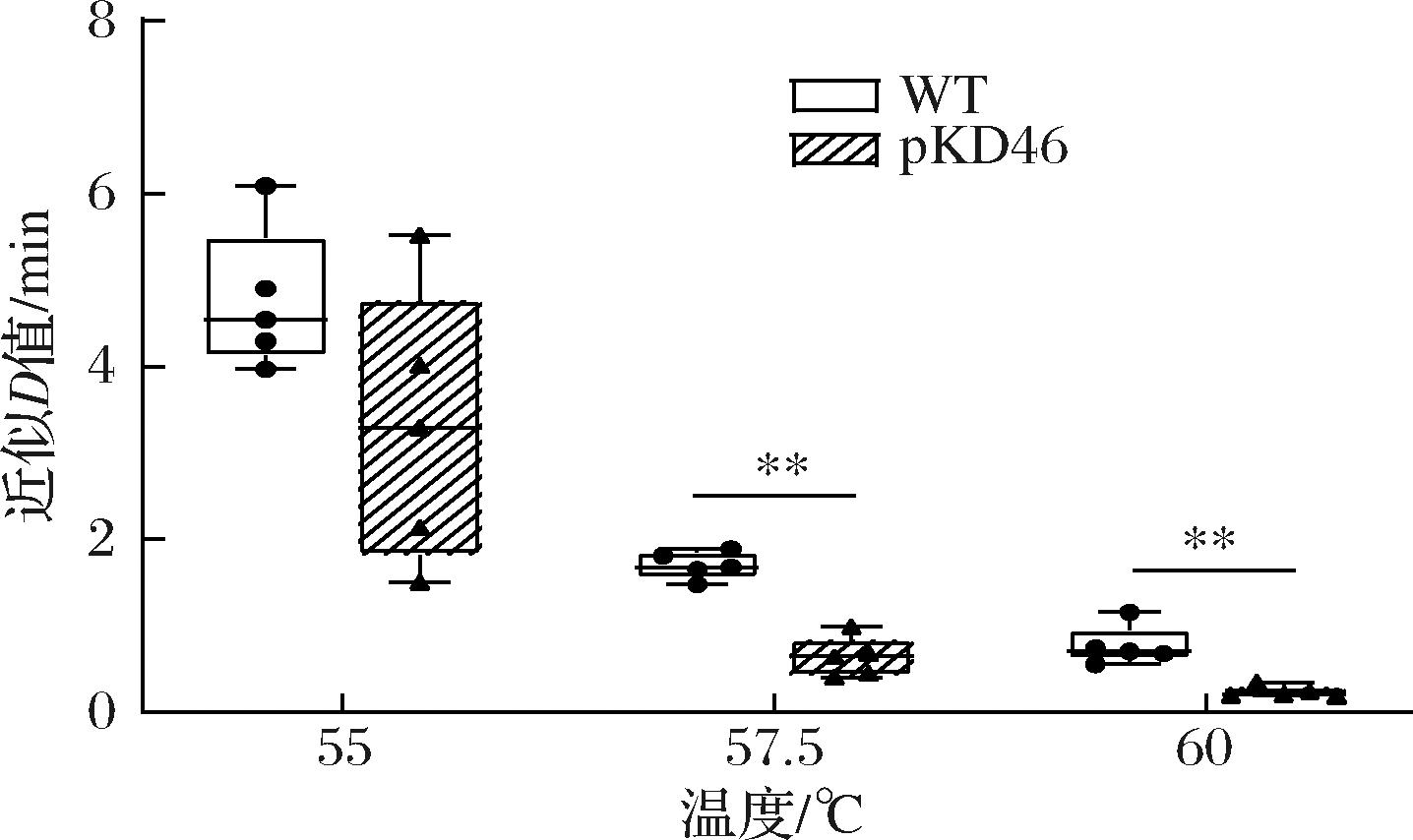
图2 野生型与耐药型肠炎沙门氏菌近似D值
Fig.2 Apparent D-values of S.Enteritidis for different phenotypes:WT and pKD46
2.4 酸抗性分析
野生型与质粒导入的耐药型沙门氏菌整体在pH=3.0条件下细菌减少量如图3所示,耐药菌的平均对数减少值虽然低于野生型,但两者之间的酸抗性无显著性差异。其中,野生型沙门氏菌的对数减少平均值为2.90 lg CFU/mL,耐药型沙门氏菌为1.81 lg CFU/mL。
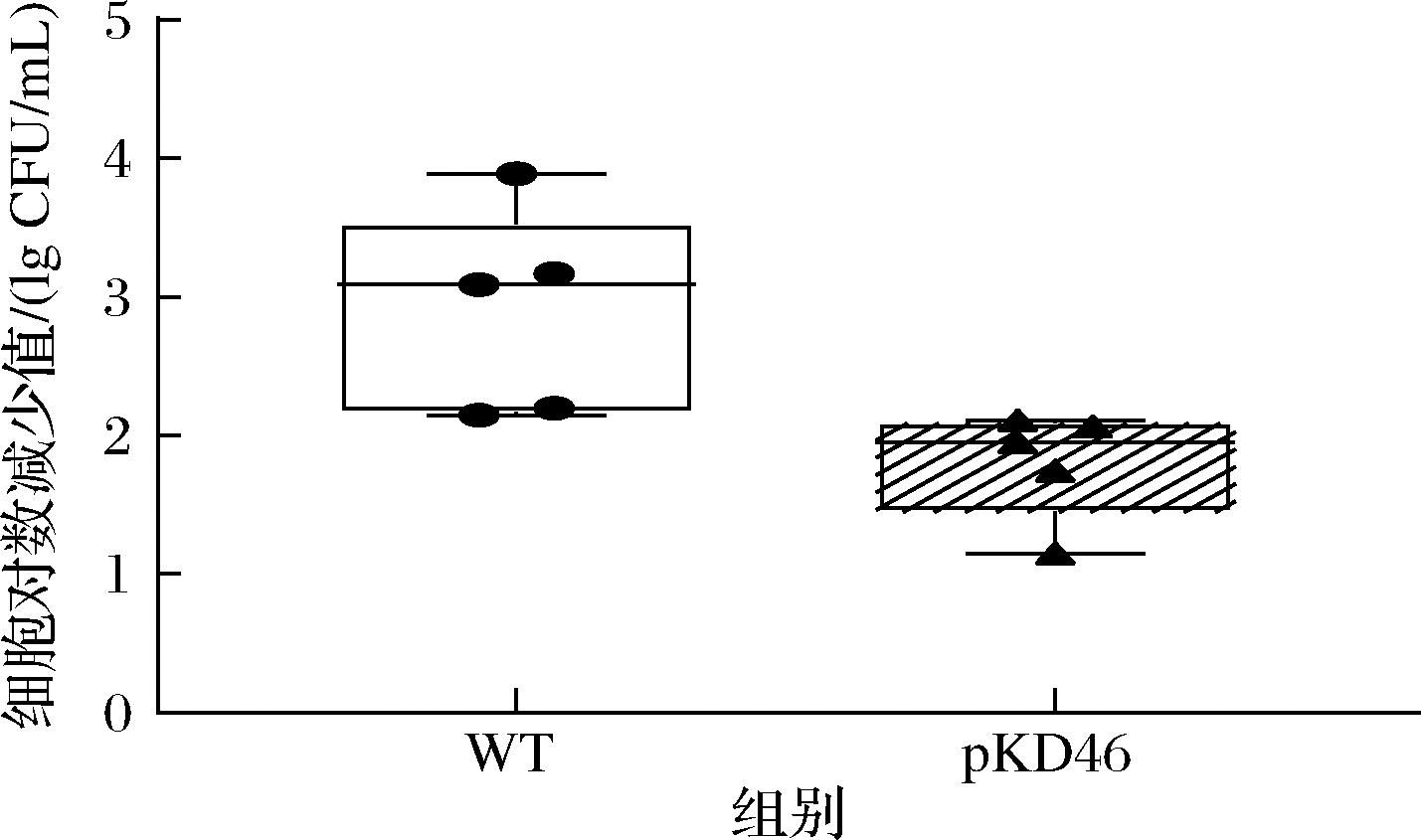
图3 野生型与耐药型肠炎沙门氏菌对数减少值
Fig.3 Logarithmic reduction of S.Enteritidis for different phenotypes:WT and pKD46
3 讨论与结论
肠炎沙门氏菌是一种人畜共患的食源性致病菌,质粒介导的耐药基因传播以及所带来的生长特性与抗性给耐药细菌的风险评估带来新的挑战。本研究将含有氨苄西林耐药基因的外源质粒pKD46导入敏感的肠炎沙门氏菌中,并对转化后沙门氏菌的耐药性、生长特性以及热与酸抗性进行了探究,发现质粒介导氨苄西林耐药的肠炎沙门氏菌对其他不同种类的抗生素交叉抗性较弱,延滞期λ增长而最大比生长速率μmax无显著差异,热抗性降低,酸抗性无显著差异。
CHOI等[15]在探究质粒介导的耐黏菌素大肠埃希氏菌生长特征时得到了与本研究相似的结论,3株耐药大肠埃希氏菌的最大比生长速率μmax与敏感菌无显著性差异。JIANG等[16]同样发现导入含有广谱β-内酰胺酶的pHK01质粒对大肠埃希氏菌的生长曲线无影响,然而当质粒中的一些sRNA过表达时会使延滞期改变。推测质粒本身或许比其携带的耐药基因对细菌生长特性的影响更大。减毒的鼠伤寒沙门氏菌在导入真核表达质粒plRES2-RGFP-4-1BBL后,生长繁殖较亲本变慢[17]。在对万古霉素耐药肠球菌的研究中发现,在自然营养条件下具有较大质粒的菌株比无质粒的敏感菌株衰减更快[18]。而在自然环境中,适应性代价较低的质粒在传播中占据更大优势[19],WANG等[20]比较了自然分离的沙门氏菌生长特性,发现敏感、耐药与多重耐药菌株间的生长并无显著性差异。
目前,关于研究耐药质粒与食品相关压力抗性关联的研究较少。在自然分离的菌株中,质粒介导的耐药基因传播是细菌耐药性增强的主要原因,研究发现金黄色葡萄球菌[21]、单增李斯特菌[22]和大肠埃希氏菌[23]等的敏感与耐药菌株间热抗性不存在显著性差异,然而本次研究中发现耐药沙门氏菌的耐热性是显著降低的。热激蛋白是生物体在应急条件下诱导产生的蛋白质,通常作为分子伴侣协助热损伤的蛋白进行修复与折叠[24]。耐药质粒的导入能在短时间内扰乱宿主原有的基因表达,包括热激蛋白,而自然界分离的耐药菌中耐药质粒则在长久的传播中实现了补偿性进化从而尽可能减小了对宿主基因组的影响,这可能是解释实验现象的原因之一。此外,研究发现携带四环素抗性基因的质粒与沙门氏菌中的热激诱导相关[25],肺炎克雷伯氏菌中预测细菌热抗性蛋白的基因与广谱β-内酰胺酶的基因通过质粒共传播的现象[26],均表明耐药质粒的传播与细菌热抗性之间存在着复杂的联系。而酸抗性的结果更加多样,研究发现大肠埃希氏菌的敏感菌株耐酸能力更强[23],相反,单增李斯特菌[22]与金黄色葡萄球菌[21]的耐药菌株酸抗性更强,而在沙门氏菌中酸抗性则与耐药性无关[27-28]。细菌的酸抗性主要包括3种机制:提高胞内pH、酸激蛋白的修复以及细胞膜修饰[29],其中许多与耐药机制相同,如外排泵、双组分系统调节以及改变膜的通透性等[30],因此不同菌株各自的特性及不同抗生素耐药机制导致研究间出现了较强的异质性。
综上所述,肠炎沙门氏菌在导入含有特定基因的耐药质粒后,耐药谱变化较小,交叉抗性主要体现在作用机制相似的抗生素中;细菌的最大比生长速率μmax不变,延滞期λ增长;耐药菌的耐热性显著降低,对食品生产加工过程中致病菌的防控有利;耐药菌的酸抗性有所增加,但不显著,不足以产生抗性的交叉保护,然而这可能与细菌的种类以及抗生素的耐药机制相关。试验结果一定程度上说明质粒介导的耐药基因传播不会增加食品生产加工过程中致病菌防控的负担,但耐药性的提高仍会增加动物饲养以及临床治疗的难度,且目前相关的研究数量较少,细菌与抗生素种类的多样性同样不容忽视,需要更多深入的研究为耐药性食源性致病菌的防控提供支持与参考。
[1] KIRK M D, PIRES S M, BLACK R E, et al.World health organization estimates of the global and regional disease burden of 22 foodborne bacterial, protozoal, and viral diseases, 2010:A data synthesis[J].PLoS Medicine, 2015, 12(12):e1001921.
[2] SUN L A, ZHANG H X, CHEN J A, et al.Epidemiology of foodborne disease outbreaks caused by nontyphoidal Salmonella in Zhejiang Province, China, 2010—2019[J].Foodborne Pathogens and Disease, 2021, 18(12):880-886.
[3] LI S T, HE Y S, MANN D A, et al.Global spread of Salmonella Enteritidis via centralized sourcing and international trade of poultry breeding stocks[J].Nature Communications, 2021, 12:5109.
[4] 胡付品, 郭燕, 朱德妹, 等.2018年CHINET中国细菌耐药性监测[J].中国感染与化疗杂志, 2020, 20(1):1-10.
HU F P, GUO Y, ZHU D M, et al.CHINET surveillance of bacterial resistance in China:2018 report[J].Chinese Journal of Infection and Chemotherapy, 2020, 20(1):1-10.
[5] NEUERT S, NAIR S, DAY M R, et al.Prediction of phenotypic antimicrobial resistance profiles from whole genome sequences of non-typhoidal Salmonella enterica[J].Frontiers in Microbiology, 2018, 9:592.
[6] LI M C, LIU Q L, TENG Y L, et al.The resistance mechanism of Escherichia coli induced by ampicillin in laboratory[J].Infection and Drug Resistance, 2019, 12:2853-2863.
[7] DUC V M, NAKAMOTO Y, FUJIWARA A, et al.Prevalence of Salmonella in broiler chickens in Kagoshima, Japan in 2009 to 2012 and the relationship between serovars changing and antimicrobial resistance[J].BMC Veterinary Research, 2019, 15(1):1-8.
[8] BECEIRO A, TOM S M, BOU G.Antimicrobial resistance and virulence:A successful or deleterious association in the bacterial world?[J].Clinical Microbiology Reviews, 2013, 26(2):185-230.
S M, BOU G.Antimicrobial resistance and virulence:A successful or deleterious association in the bacterial world?[J].Clinical Microbiology Reviews, 2013, 26(2):185-230.
[9] MOHSEN A, CHERN C W, HENRIETTA V.Intrinsic, adaptive and acquired antimicrobial resistance in Gram-negative bacteria[J].Essays in Biochemistry, 2017, 61(1):49-59.
[10] VOGWILL T, MACLEAN R C.The genetic basis of the fitness costs of antimicrobial resistance:A meta-analysis approach[J].Evolutionary Applications, 2015, 8(3):284-295.
[11] MELNYK A H, WONG A, KASSEN R.The fitness costs of antibiotic resistance mutations[J].Evolutionary Applications, 2015, 8(3):273-283.
[12] LIAO X Y, MA Y N, DALIRI E B M, et al.Interplay of antibiotic resistance and food-associated stress tolerance in foodborne pathogens[J].Trends in Food Science &Technology, 2020, 95:97-106.
[13] BARANYI J, PIN C.Estimating bacterial growth parameters by means of detection times[J].Applied and Environmental Microbiology, 1999, 65(2):732-736.
[14] WANG X, DEVLIEGHERE F, GEERAERD A, et al.Thermal inactivation and sublethal injury kinetics of Salmonella enterica and Listeria monocytogenes in broth versus agar surface[J].International Journal of Food Microbiology, 2017, 243:70-77.
[15] CHOI Y, LEE J Y, LEE H, et al.Comparison of fitness cost and virulence in chromosome- and plasmid-mediated colistin-resistant Escherichia coli[J].Frontiers in Microbiology, 2020, 11:798.
[16] JIANG X L, LIU X, LAW C O K, et al.The CTX-M-14 plasmid pHK01 encodes novel small RNAs and influences host growth and motility[J].FEMS Microbiology Ecology, 2017, 93(7):fix090.
[17] 仉元亭, 叶建新, 陈卫昌, 等.pIRES2-EGFP-4-1BBL质粒的导入对减毒沙门菌生物学行为的影响[J].苏州大学学报(医学版), 2009, 29(2):236-239.
ZHANG Y T, YE J X, CHEN W C, et al.The biological behaviour change of attenuation Salmonella imported pIRES2-EGFP-4-1BBL plasmid[J].Suzhou University Journal of Medical Science, 2009, 29(2):236-239.
[18] YOUNG S, ROHR J R, HARWOOD V J.Vancomycin resistance plasmids affect persistence of Enterococcus faecium in water[J].Water Research, 2019, 166:115069.
[19] CARROLL A C, WONG A.Plasmid persistence:Costs, benefits, and the plasmid paradox[J].Canadian Journal of Microbiology, 2018, 64(5):293-304.
[20] WANG X, XIE Y N, CAI H, et al.Growth and survival characteristics of Salmonella enterica regarding antibiotic resistance phenotypes[J].LWT, 2021, 149:111872.
[21] MA Y N, LAN G A, LI C, et al.Stress tolerance of Staphylococcus aureus with different antibiotic resistance profiles[J].Microbial Pathogenesis, 2019, 133:103549.
[22] KOMORA N, BRUSCHI C, MAGALH ES R, et al.Survival of Listeria monocytogenes with different antibiotic resistance patterns to food-associated stresses[J].International Journal of Food Microbiology, 2017, 245:79-87.
ES R, et al.Survival of Listeria monocytogenes with different antibiotic resistance patterns to food-associated stresses[J].International Journal of Food Microbiology, 2017, 245:79-87.
[23] AKHTAR M, MASERATI A, DIEZ-GONZALEZ F, et al.Does antibiotic resistance influence shiga-toxigenic Escherichia coli O26 and O103 survival to stress environments?[J].Food Control, 2016, 68:330-336.
[24] GUISBERT E, YURA T, RHODIUS V A, et al.Convergence of molecular, modeling, and systems approaches for an understanding of the Escherichia coli heat shock response[J].Microbiology and Molecular Biology Reviews:MMBR, 2008, 72(3):545-554.
[25] HÜTTENER M, PRIETO A, AZNAR S, et al.Tetracycline alters gene expression in Salmonella strains that harbor the Tn10 transposon[J].Environmental Microbiology Reports, 2018, 10(2):202-209.
[26] BOJER M S, HAMMERUM A M, JØRGENSEN S L, et al.Concurrent emergence of multidrug resistance and heat resistance by CTX-M-15-encoding conjugative plasmids in Klebsiella pneumoniae[J].APMIS, 2012, 120(9):699-705.
[27] BACON R T, SOFOS J N, KENDALL P A, et al.Comparative analysis of acid resistance between susceptible and multi-antimicrobial-resistant Salmonella strains cultured under stationary-phase acid tolerance-inducing and noninducing conditions[J].Journal of Food Protection, 2003, 66(5):732-740.
[28] HUGHES M K, YANAMALA S, SAN FRANCISCO M, et al.Reduction of multidrug-resistant and drug-susceptible Salmonella in ground beef and freshly harvested beef briskets after exposure to commonly used industry antimicrobial interventions[J].Journal of Food Protection, 2010, 73(7):1231-1237.
[29] BEGLEY M, HILL C.Stress adaptation in foodborne pathogens[J].Annual Review of Food Science and Technology, 2015, 6:191-210.
[30] MUNITA J M, ARIAS C A.Mechanisms of antibiotic resistance[J].Microbiology Spectrum, 2006, 13(3):177-184.