2-苯乙醇(2-phenylethanol,2-PE)是一种芳香族化合物,广泛用于食品和化妆品行业。2-PE主要通过化学合成生产,但此法会产生有毒副产物。利用微生物转化进行天然2-PE的生产受到了广泛关注[1]。其中,酿酒酵母(Saccharomyces cerevisiae)是最主要的生产菌株。酿酒酵母通过莽草酸途径(从头合成)和艾氏途径合成2-PE[2]。最近研究表明通过强化酿酒酵母莽草酸途径构建的工程菌株JM26产2-PE达643 mg/L[3]。然而,从头途径合成2-PE产量低,要实现工业化生产,仍然面临着代谢工程方面的巨大挑战[4]。当L-苯丙氨酸(L-Phe)是培养基中唯一的氮源时,2-PE主要通过艾氏途径合成。L-Phe由芳香族氨基酸氨基转移酶Aro9催化生成苯丙酮酸,然后通过苯丙酮酸脱羧酶Aro10脱羧为苯乙醛,再由醇脱氢酶(alcohol dehydrogenase,ADH)还原为2-PE[5],2-PE的工业生产主要依赖于该途径。
为了提高酿酒酵母的2-PE产量,可用诱变育种、基因组改组、基因工程和基因编辑等方法[2,6]。这些方法在菌株改良的初始阶段有一定效果,尤其是在产量较低的菌株中。酿酒酵母S288C中过量表达转氨酶ARO8和苯丙酮酸脱羧酶ARO10后,2-PE产量达到2.61 g/L[7]。多个相关基因组装构建的酿酒酵母菌株YS58在5 L发酵罐中的2-PE产量达到6.3 g/L[8]。我们前期对酿酒酵母CWY132菌株进行了研究,通过优化培养条件,2-PE产量达到3.52 g/L[9],发酵罐小试其产量达4.1 g/L[10]。以糖蜜为碳源,进一步利用液-液两相的分批补料发酵及工艺进行转化,2-PE产量最高达到9.03 g/L[11]。
当产量达到一定水平时,2-PE对酵母细胞的毒性便成了限制产量的瓶颈。此外,酿酒酵母在转化2-PE的过程中会产生较多的副产物乙醇,从而与2-PE产生协同抑制效应[12]。通过原位产物回收技术,2-PE的产量从2.28 g/L增加到5.14 g/L[13]。然而,去除回收残留有机溶剂存在困难,限制了其应用[2]。因此,提高菌株对2-PE的耐受性是进一步提高产量的潜在途径。酿酒酵母菌株的耐受性是由多基因控制的复杂综合性状,通过随机诱变或基因工程提高菌株耐受性的策略相对来说效率较低。因此,有必要将传统方法和基因工程方法相结合来改良菌株。
我国酿酒业高度发达,酵母菌株的多样性非常丰富,是重要的资源宝库。例如,在制备茅台酒时,酿酒酵母在高温、高酸和高乙醇浓度等多种应激因素的环境中生长。在制曲过程中,温度达60 ℃,pH值在2.0左右,乙醇体积分数达82%以上[14]。生长在这种特殊环境中的微生物区系具有独特的物理和化学特征,可以从中筛选出耐受性更好的菌株。例如,筛选出的耐热胁迫酿酒酵母菌株可耐受2.5 g/L的2-PE,显著高于不耐热菌株(1 g/L),相应地,2-PE产量达4.5 g/L[15]。
在获得具有不同优良性状的多个酵母菌株后,有必要将这些性状聚合到一个酵母菌株中。原生质体融合显示出高效的潜力,可以在不需要了解具体机制的情况下实现。该方法已用于改善酿酒酵母的乙醇生产性能,融合菌株同时获得双亲的发酵性能和乙醇耐受性[16]。酿酒酵母和巴斯德酵母菌的融合菌株SP2-18的乙醇产量为74.65 g/L,而亲本的乙醇产量分别为33.12和65.44 g/L[17]。酿酒酵母和乙醇假丝酵母的融合菌株R6在生产高品质低醇苹果酒方面具有巨大潜力[18]。这些研究表明,原生质体融合是酿酒酵母菌株改良的有效策略。
CRISPR/Cas9技术已成功应用于酿酒酵母的基因操作,DI-CRISPR(Delta整合的CRISPR)可以实现外源基因的高拷贝整合[19]。研究表明酿酒酵母cdc25(Ras家族鸟嘌呤核苷酸交换因子)的点突变菌株(W1416C)可以在39 ℃高温下有效地进行乙醇发酵,产量为6 g/L[20]。但是cdc25突变对2-PE产量是否有促进作用尚不明确。在本研究中,我们筛选出3株耐受性好、2-PE产量高的酿酒酵母菌株,经原生质体融合后,进一步利用DI-CRISPR对融合菌株进行研究,探索cdc25及艾氏途径关键酶基因对2-PE产量的影响。构建策略见电子增强出版附件1(https://doi.org/10.13995/j.cnki.11-1802/ts.031898)。
1 材料与方法
1.1 菌株、培养基和试剂
1.1.1 菌株
酿酒酵母CWY132、酿酒酵母NGER,诺睿特生物公司;LSC-1(CICC 33068)、LSC-2(CICC 1005)和LSC-3(CICC 32304),中国工业微生物保藏中心;S.C-1分离于浙江绍兴黄酒酿造样品。其他各菌株及来源见电子增强出版附件2(https://doi.org/10.13995/j.cnki.11-1802/ts.031898)。
1.1.2 试剂及其配制方法
NaH2PO4· 2H2O、山梨醇、β-巯基乙醇、EDTA-2 Na、酵母基因组DNA提取试剂盒、蜗牛酶,上海生工生物工程有限公司;崩溃酶,美国Sigma-Aldrich公司;cDNA转录试剂盒、Power SYBR®green PCR Master Mix试剂,美国Thermo Fisher Scientific公司。
0.2 mol/L PB溶液:87.7 mL 0.2 mol/L NaH2PO4· 2H2O和12.3 mL 0.2 mol/L Na2HPO4·2H2O,pH 6.0。
PBS溶液:50 mL 0.2 mol/L PB溶液和450 mL 1 mol/L山梨醇溶液,pH 6.0。
细胞壁裂解预处理液:取0.2 mL β-巯基乙醇和2.233 g EDTA-2Na,溶解于PBS中,定容至100 mL,0.22 μm过滤器过滤。
1.1.3 培养基
YPD培养基用于菌株活化和分离。YPDS培养基(原生质体再生培养基):YPD中添加182.17 g/L山梨醇。
2-PE发酵培养基(g/L):葡萄糖 30,MgSO4 0.5,K3PO4 5,酵母提取物 1.5,L-Phe 5。
1.2 酵母菌株的分离与鉴定
样品进行稀释后涂布在YPD平板上于30 ℃培养48 h,通过菌落形态和显微观察选择酵母菌,分离纯化后进行分子鉴定。酵母基因组DNA用试剂盒提取。ITS(Internal Transcribed Spacer)的全序列用引物对Seq-F(5′-CGCCAGGTTTTCACAGAC-3′)/Seq-R(5′-GAGCGATACATTTCACAGC-3′)进行PCR扩增。将ITS序列与GenBank核酸数据库进行相似性分析,用MEGA7.0软件进行系统发育树分析。
1.3 2-PE生长曲线及耐高温性的测定
用光密度法(OD600)测定各菌株的生长曲线。用梯度稀释滴板实验分析2-PE的耐受性,将酵母细胞浓度调整为107个细胞/mL,取5 μL稀释液滴在含有2-PE的YPD平板上培养。
1.4 原生质体制备、融合、再生
通过离心收集新鲜培养的酵母细胞,在PBS溶液中与4 mL酶液混合,酶液由70 mg/mL蜗牛酶和2 mg/mL崩溃酶组成,30 ℃、60 r/min下酶解2 h。利用双亲灭活的方法进行细胞融合和选择,使灭活效率≥95%。离心收集原生质体,PBS洗涤并悬浮,一亲本的悬浮液置于25 W紫外线灯下照射,涂布于YPDS平板,在30 ℃培养,另一亲本在60 ℃水浴中处理。处理后的双亲原生质体浓度调整为107/mL,各取0.5 mL混合并离心后悬浮在1 mL 40%(质量分数) 聚乙二醇6000,30 ℃避光培养25 min。离心收集原生质体,PBS稀释后涂布于YPDS板在30 ℃培养观察,等待融合子出现。
1.5 融合子鉴定
细胞DNA含量按照公式(1)计算[18]:
(1)
式中:Ki,稀释因子;E,1个紫外吸收单位代表DNA质量浓度等于50 μg/mL。
1.6 使用DI-CRISPR构建艾氏途径和cdc25突变体中的基因过度表达
融合菌株RH2-16为宿主,将艾氏途径中关键酶基因ARO8、ARO10和ADH2分别由组成性启动子磷酸甘油酸激酶驱动表达,整合在酿酒酵母基因组的delta位点。将这3个基因组合,获得7种类型菌株:↑ARO8,↑ARO10,↑ADH2,↑ARO8+↑ARO10,↑ARO8+↑ADH2,↑ARO10+↑ADH2和↑ARO8+↑ARO10+↑ADH2(↑表示超表达)。DI-CRISPR基因编辑参考文献[20]进行。通过LiAc介导的方法转化酵母,利用构建特异性引物(正向引物位于目标基因的开放阅读框内,反向引物位于delta位点)对转化子进行了PCR鉴定。通过PCR引入cdc25基因的点突变,并如上所述构建突变株。引物见电子版增强出版附件3(https://doi.org/10.13995/j.cnki.11-1802/ts.031898)。
1.7 qRT-PCR分析
使用cDNA转录试剂盒进行转录,以1 μg总RNA为模板合成cDNA。以ACT1基因作为内参,进行qRT-PCR,在7500 Real-Time PCR系统使用Power SYBR®green PCR Master Mix试剂进行扩增。引物见电子版增强出版附件3(https://doi.org/10.13995/j.cnki.11-1802/ts.031898)。
1.8 2-PE发酵生产及效价测定
对数生长期的酵母细胞以10%(体积分数)的接种量添加到含有5 g/L L-Phe的50 mL发酵培养基中,30 ℃,200 r/min发酵36 h。发酵液离心后取上清液用气相色谱测定2-PE的浓度。向样品中加入200 μL内标溶液(甲基异丁基甲醇1 g/L水溶液),并混合500 μL乙酸乙酯。取上层有机相测量(岛津GC 2014)。独立进行3次实验,每个处理至少3个重复。
1.9 数据处理
实验重复3次,使用GraphPad作图及数据分析。
2 结果与讨论
2.1 酵母菌分离鉴定
共筛选获得25株酵母菌株(电子版增强出版附件2)。通过菌落形态分析、显微观察和分子鉴定,鉴定出19株酿酒酵母、2株Wickerhamomyces anomalus、2株Pichia pastoris、1株Torulaspora delbrueckii和1株Candida anglica。根据ITS序列分析,工业菌株CWY132与从源于四川的样品中分离的SC001、SC003和SC009之间相似性高,与GenBank中的酿酒酵母JYC2558菌株遗传关系接近。来自CICC的LSC-1和来自甘肃的SC008显示出高度同源性,而从浙江衢州分离的S.C-1与绍兴分离的S.C-2遗传上并不相近,亲缘关系见系统发育树(电子版增强出版附件4,https://doi.org/10.13995/j.cnki.11-1802/ts.031898)。
2.2 分离菌株的性状测定
以工业菌株CWY132为参考,对分离的酵母菌株从2-PE产量、2-PE耐受性及高温耐受性进行评价。结果表明,不同菌株的2-PE产量差异显著。酿酒酵母的2-PE合成能力高于其他酵母菌。有6株酵母的2-PE产量在1~2 g/L,4株低于1 g/L。5株酿酒酵母(LSC-1、SC002、SC003、SC004和SC005)的2-PE产量稳定在3.4 g/L以上,高于工业菌株CWY132(电子版增强出版附件5,https://doi.org/10.13995/j.cnki.11-1802/ts.031898),表明酿酒酵母在2-PE的合成中具有天然优势。上述5株2-PE产量较高的酿酒酵母生长速率显著高于CWY132,其中LSC-1生长速率最高,其2-PE产量也高。2-PE耐受性测定发现在2.80 g/L时,NGER、S.C-1和LSC-1的生长明显优于CWY132。在3.60 g/L时,NGER生长最好(图1-a)。高温耐受性测定发现,在40 ℃时,CWY132的生长受到显著抑制,而其他3株仍能生长。在41 ℃培养48 h后,只有S.C-1生长(图1-b),表明S.C-1能够抵抗高温胁迫。综上,以LSC-1、NGER和S.C-1这3株菌作为原生质体融合的亲本菌株。

a-不同浓度2-PE胁迫;b-高温胁迫
图1 酿酒酵母菌株在2-PE和高温胁迫下的耐受性测定
Fig.1 Tolerance test of S.cerevisiae strains under stress of 2-PE and high temperature
2.3 第一轮原生质体融合获得耐受性增强的融合子
以NGER和S.C-1为亲本进行第一轮原生质体融合,共获得20个融合子,其中RH1-4与NGER生长性能相近,但在2-PE质量浓度为3.60 g/L时,耐受性提高(图2-a)。在40 ℃下生长性能比较发现,RH1-4与S.C-1耐热性相近,表明其从亲本中同时获得了高温和2-PE耐受性(图2-b)。RH1-4的2-PE产量为2.11 g/L,比亲本分别增加了11%和24%(图2-c)。
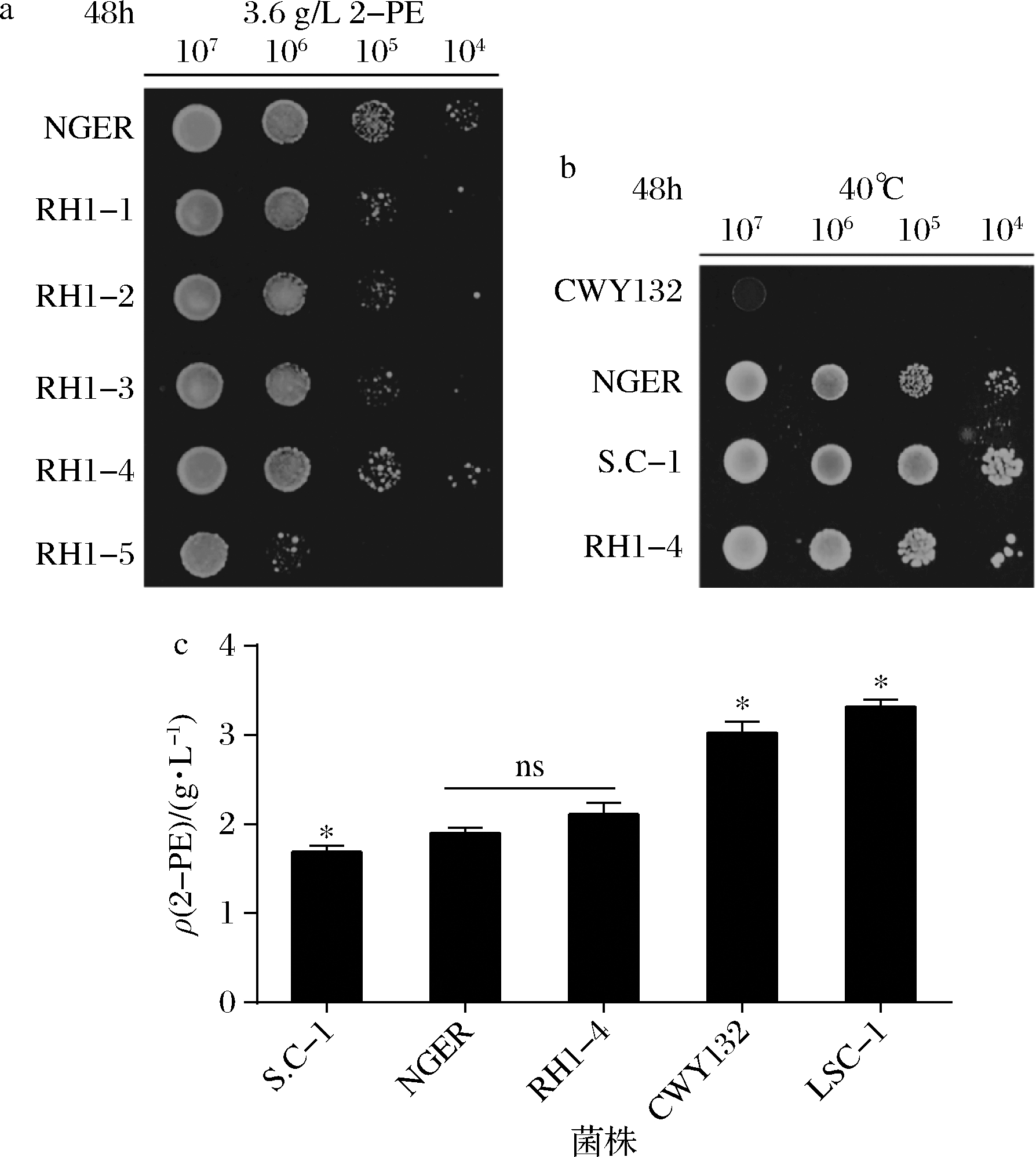
a-在3.6 g/L 2-PE胁迫下生长;b-在40 ℃下生长;c-2-PE产量
图2 第一轮原生质体融合子筛选
Fig.2 Screening of strains with 2-PE tolerance from the first round of protoplast fusants
注:*表示差异显著性(P<0.05)(下同)
2.4 第二轮原生质体融合获得2-PE高产融合子
将RH1-4和LSC-1作为亲本进行第二轮原生质体融合,共获得约100个融合菌株,根据高温耐受性分为A至C级。A:接近LSC-1水平;B:LSC-1和RH1-4之间;C:接近RH1-4的水平(图3-a)。C级融合子中只有几株的2-PE产量接近RH1-4(2.11 g/L),其余都低于RH1-4,接近第一轮亲本SC-1的产量1.67 g/L。B级融合子的耐受性没有达到RH1-4的预期水平,但它们的产量相对较高。其中一株融合子RH2-16的2-PE为4.05 g/L,比亲本LSC-1增加18.8%。另一株RH2-26的产量达3.96 g/L,增加了16.5%(图3-b)。

a-40 ℃胁迫下;b-2-PE产量测定
图3 在原生质体融合的第二轮中筛选2-PE产量 和耐受性增加的融合子
Fig.3 Screening of fusants with increased 2-PE titre and tolerance in the second round of protoplast fusion
2.5 融合子的遗传稳定性和2-PE产量测定
细胞DNA含量测定结果(每107个细胞中):亲本NGER、SC-1和LSC-1的分别为0.71、0.67和1.08 μg;融合子RH1-4和RH2-16的分别为1.05和1.49 μg,符合杂合子的特征。融合子连续传代后检测2-PE和乙醇产量,发现各菌株连续培养10代、20代、30代后2-PE产量基本稳定(电子版增强出版附件6,https://doi.org/10.13995/j.cnki.11-1802/ts.031898),说明融合子遗传稳定性良好。RH2-16的最高产量为4.31 g/L,5代的平均产量为4.04 g/L。亲本和融合子菌株的2-PE产量、耐性等性状的综合比较见电子版增强出版附件7(https://doi.org/10.13995/j.cnki.11-1802/ts.031898)。此外发现,在最适生长温度(30 ℃)下2-PE产量最高,即使是耐热菌株(电子版增强出版附件8),有的高产2-PE菌株也具有较好的乙醇生产能力(电子版增强出版附件6和附件8)。
2.6 cdc25突变株表型测定及基因表达分析
cdc25W1416C和cdc25N1393T的2个点突变分别被引入融合子菌株RH2-16。结果表明,cdc25W1416C在40 ℃、105 CFU/mL时生长较好,而野生型几乎不能生长(图4-a)。cdc25W1416C在3.0 g/L的2-PE平板上生长优于野生型(图4-b),表明该突变株提高了综合耐受性。cdc25N1393T未发现上述表型。
qRT-PCR技术分析了RH2-16、cdc25W1416C、CWY132、NGER和S.C-1菌株的耐热相关基因(hsp12、hsp104)、乙醇脱氢酶基因(adh2)和艾氏途径分支途径酶基因(ald3)的转录水平。结果表明,与RH2-16相比,RH2-16(cdc25W1416C)中hsp104转录水平增加了5倍,S.C-1中hsp12增加了近9倍(图4-c)。cdc25W1416C的2-PE产量增加8%(图4-d)。说明cdc25W1416C点突变可有效提高耐热性,增加2-PE产量。

a-40 ℃胁迫;b-2-PE胁迫;c-基因转录水平差异分析;d-2-PE产量测定
图4 cdc25W1416C突变株的表型鉴定
Fig.4 Phenotype identification of cdc25W1416C mutant strains
2.7 艾氏途径相关基因过表达对2-PE合成的影响
选择芳香族氨基酸氨基转移酶(ARO8)、苯丙酮酸脱羧酶(ARO10)和醇脱氢酶(ADH2)基因,构建了7株过表达单基因或组合的菌株,分别记为↑ADH2,↑ARO8,↑ARO10,↑ARO8+↑ARO10,↑ARO8+↑ADH2,↑ARO10+↑ADH2和↑ARO8+↑ARO10+↑ADH2(图5-a)。测定2-PE产量发现,RH2-16为3.92 g/L,RH2-16 ↑ADH2和RH2-16↑ARO8+↑ARO10分别为3.91和3.88 g/L。其他菌株产量下降,其中RH2-16↑ARO10为3.49 g/L,降低了12%(图5-b)。结果表明,该策略不能有效提高菌株RH2-16的2-PE产量。
3 结论与讨论
微生物合成2-PE等天然代谢物的能力,菌株之间的差异很大。筛选优良的菌株并采取有效的改良措施,是工业化生产开发的途径[21]。我国酵母菌株资源丰富,分离获得综合性状优良的菌株用于2-PE生产的潜能很大,本文研究结果证实了这一点。
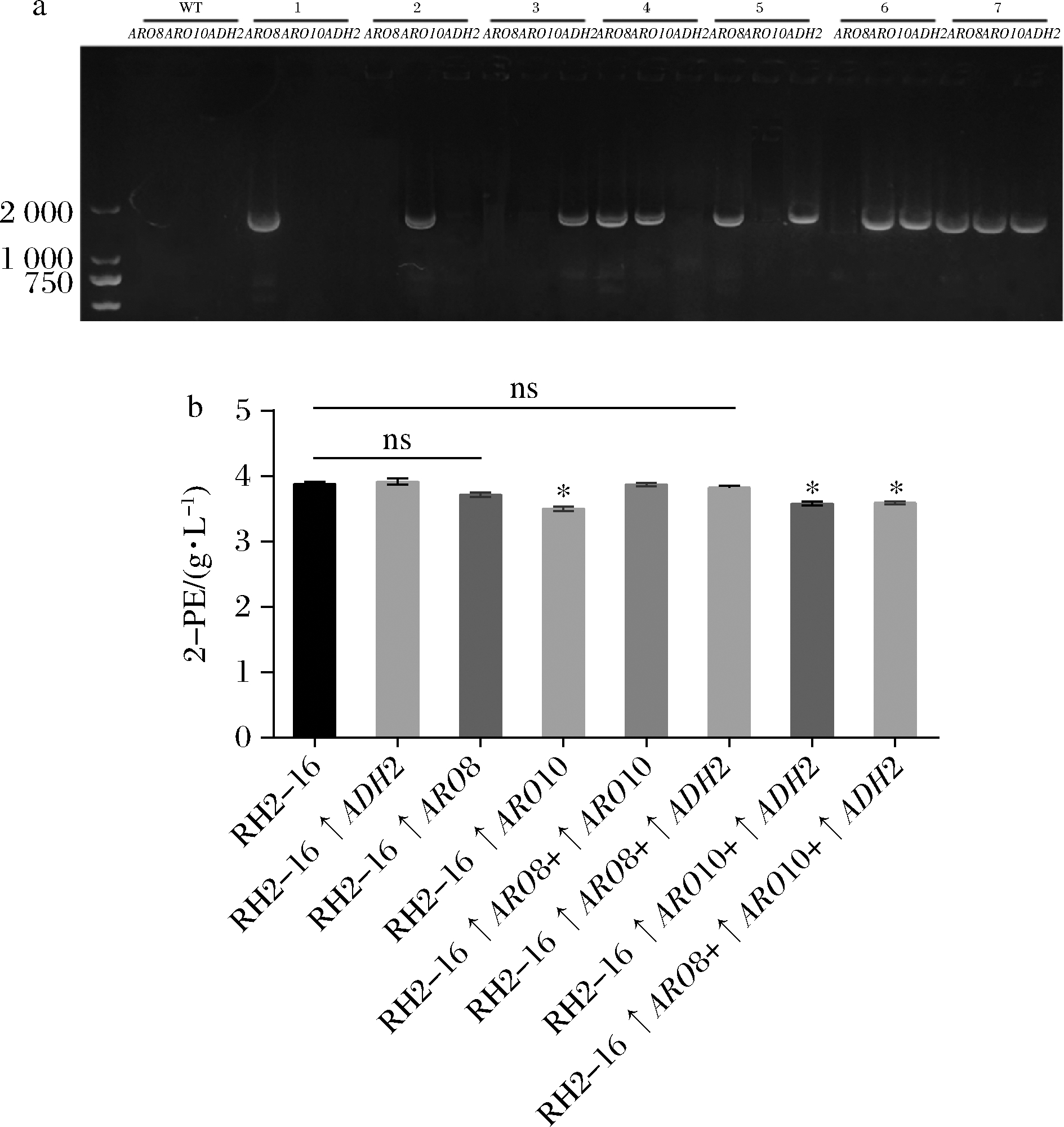
a-特异性引物PCR鉴定;b-2-PE产量测定
图5 过表达艾氏途径基因各菌株的分子鉴定和2-PE产量
Fig.5 Molecular characterization of strains overexpressing Ehrlich pathway genes and determination of 2-PE titre
原生质体融合可以绕过有性繁殖的障碍,通过重组基因组DNA的大片段,获得具有亲本遗传特征的稳定“杂交”细胞。此外,在细胞融合过程中,可以有效地从亲本基因组或突变的随机组合中获得新的正向遗传变异。酿酒酵母菌株LSC-1、NGER和SC-1经两轮原生质体融合后,优良性状已成功整合到融合子菌株中,其2-PE产量显著高于工业菌株CWY132,产量提高主要归因于其耐受性的增强。研究表明,耐热性是其他胁迫耐受性的良好指标,耐热菌株可以帮助减少2-PE的毒性作用[22]。耐高温的菌株对氧化和渗透胁迫也有更强的抵抗力[23]。耐热菌株Ye9-612在30~40 ℃下具有恒定的生长速率,但非耐热菌株Ye9-596(同一亲本的后代)则受到严重影响[15],本文研究结果与这些发现一致。
尽管一些研究表明,在艾氏途径中超表达与2-PE合成相关的基因可以提高产量[2,5-6],但在本研究中,用相同的策略未能提高融合子RH2-16的2-PE产量。可能是RH2-16的2-PE产量相对较高,进一步提高对酵母细胞产生更强的毒性。此结果与之前报道的类似,即4.0 g/L 2-PE可以完全抑制所试酵母细胞的生长[21]。
综上,从自然资源中筛选优良性状的菌株做亲本,通过原生质体融合是一种有效的酵母育种方法。与需要突变文库构建和递归融合的基因组改组相比,该法更简单、高效。对具有良好抗逆性的酵母菌株进行转录组分析,有助于理解多重耐受机制[24]。RH2-16可以作为生产2-PE的工业菌株,也可以作为分析2-PE合成机理或用于育种的优良候选菌株。
[1] ETSCHMANN M W, BLUEMKE W, SELL D, et al.Biotechnological production of 2-phenylethanol[J].Applied Microbiology and Biotechnology, 2002, 59(1):1-8.
[2] QIAN X J, YAN W, ZHANG W M, et al.Current status and perspectives of 2-phenylethanol production through biological processes[J].Critical Reviews in Biotechnology, 2019, 39(2):235-248.
[3] ZHU L H, WANG J H, XU S, et al.Improved aromatic alcohol production by strengthening the shikimate pathway in Saccharomyces cerevisiae[J].Process Biochemistry, 2021, 103:18-30.
[4] HASSING E J, DE GROOT P A, MARQUENIE V R, et al.Connecting central carbon and aromatic amino acid metabolisms to improve de novo 2-phenylethanol production in Saccharomyces cerevisiae[J].Metabolic Engineering, 2019, 56:165-180.
[5] KIM B, CHO B R, HAHN J S.Metabolic engineering of Saccharomyces cerevisiae for the production of 2-phenylethanol via Ehrlich pathway[J].Biotechnology and Bioengineering, 2014, 111(1):115-124.
[6] 严伟, 高豪, 蒋羽佳, 等.2-苯乙醇生物合成的研究进展[J].合成生物学, 2021, 2(6):1 030-1 045.
YAN W, GAO H, JIANG Y J, et al.Research progress in 2-phenylethanol production through biological processes[J].Synthetic Biology Journal, 2021, 2(6):1 030-1 045.
[7] YIN S, ZHOU H, XIAO X, et al.Improving 2-phenylethanol production via Ehrlich pathway using genetic engineered Saccharomyces cerevisiae strains[J].Current Microbiology, 2015, 70(5):762-767.
[8] WANG Z Y, JIANG M Y, GUO X N, et al.Reconstruction of metabolic module with improved promoter strength increases the productivity of 2-phenylethanol in Saccharomyces cerevisiae[J].Microbial Cell Factories, 2018, 17(1):60.
[9] CUI Z F, YANG X, SHEN Q J, et al.Optimisation of biotransformation conditions for production of 2-phenylethanol by a Saccharomyces cerevisiae CWY132 mutant[J].Natural Product Research, 2011, 25(7):754-759.
[10] 汪琨, 沈情佳, 魏秀燕, 等.酿酒酵母CWY132发酵罐小试生产2-苯乙醇[J].食品与发酵工业, 2011, 37(4):102-106.
WANG K, SHEN Q J, WEI X Y, et al.Studies on the biotransformation processes for 2-phenylethanol production by Saccharomyces cerevisiae CWY132 in small fermentor test[J].Food and Fermentation Industries, 2011, 37(4):102-106.
[11] 崔志峰, 沈情佳, 汪琨, 等.酿酒酵母CWY132以糖蜜为碳源生产2-苯乙醇的培养条件[J].食品与发酵工业, 2010, 36(11):10-13.
CUI Z F, SHEN Q J, WANG K, et al.Studies on the fermentation conditions for production of 2-phenylethanol by Saccharomyces cerevisiae CWY132 in a molasses-based medium[J].Food and Fermentation Industries, 2010, 36(11):10-13.
[12] WANG H, DONG Q F, GUAN A, et al.Synergistic inhibition effect of 2-phenylethanol and ethanol on bioproduction of natural 2-phenylethanol by Saccharomyces cerevisiae and process enhancement[J].Journal of Bioscience and Bioengineering, 2011, 112(1):26-31.
[13] SHU C H, CHEN Y J, NIRWANA W O C, et al.Enhanced bioconversion of L-phenylalanine into 2-phenylethanol via an oxygen control strategy andin situproduct recovery[J].Journal of Chemical Technology & Biotechnology, 2018, 93(10):3 035-3 043.
[14] WU Q, XU Y, CHEN L.Diversity of yeast species during fermentative process contributing to Chinese Maotai-flavour liquor making[J].Letters in Applied Microbiology, 2012, 55(4):301-307.
[15] ESHKOL N, SENDOVSKI M, BAHALUL M, et al.Production of 2-phenylethanol from L-phenylalanine by a stress tolerant Saccharomyces cerevisiae strain[J].Journal of Applied Microbiology, 2009, 106(2):534-542.
[16] XIN Y, YANG M, YIN H, et al.Improvement of ethanol tolerance by inactive protoplast fusion in Saccharomyces cerevisiae[J].BioMed Research International, 2020, 2020:1979318.
[17] JETTI K D, GNS R R, GARLAPATI D, et al.Improved ethanol productivity and ethanol tolerance through genome shuffling of Saccharomyces cerevisiae and Pichia stipitis[J].International Microbiology:the Official Journal of the Spanish Society for Microbiology, 2019, 22(2):247-254.
[18] WANG Z L, XU K D, CAI R, et al.Construction of recombinant fusant yeasts for the production of cider with low alcohol and enhanced aroma[J].European Food Research and Technology, 2020, 246(4):745-757.
[19] SHI S B, LIANG Y Y, ZHANG M M,et al.A highly efficient single-step, markerless strategy for multi-copy chromosomal integration of large biochemical pathways in Saccharomyces cerevisiae[J].Metabolic Engineering, 2016, 33:19-27.
[20] SATOMURA A, MIURA N, KURODA K, et al.Reconstruction of thermotolerant yeast by one-point mutation identified through whole-genome analyses of adaptively-evolved strains[J].Scientific Reports, 2016, 6:23157.
[21] ETSCHMANN M M, SELL D, SCHRADER J.Screening of yeasts for the production of the aroma compound 2-phenylethanol in a molasses-based medium[J].Biotechnology Letters, 2003, 25(7):531-536.
[22] WELKER S, RUDOLPH B, FRENZEL E, et al.Hsp12 is an intrinsically unstructured stress protein that folds upon membrane association and modulates membrane function[J].Molecular Cell, 2010, 39(4):507-520.
[23] NIR N, BAHALUL M, FEINGERSCH R, et al.Improvement of natural isolates of Saccharomyces cerevisiae strains for synthesis of a chiral building block using classic genetics[J].Applied Microbiology and Biotechnology, 2008, 78(4):659-667.
[24] INOKUMA K, IWAMOTO R, BAMBA T, et al.Improvement of xylose fermentation ability under heat and acid co-stress in Saccharomyces cerevisiae using genome shuffling technique[J].Frontiers in Bioengineering and Biotechnology, 2017, 5:81.