脂肪酶(EC3.1.1.3)可以催化长链酰基甘油在脂质-水界面分解为二酰基甘油酯、单甘油酯、甘油和游离脂肪酸[1-2]。目前研究发现脂肪酶还可以催化酯合成、酰胺化、醇解、酯交换等一系列反应[3],在食品工业、纸浆工业、化妆品行业以及精细化学品加工中有着广泛的应用。
近年来,真菌脂肪酶得到了广泛关注,已有不少真菌脂肪酶如来源于南极假丝酵母的脂肪酶B(Novozyme 435)和来源于米黑根毛霉的脂肪酶(Novozyme Lipozyme RMIM)等实现了商业化,并被应用于食品酿造行业中[4]。例如,王丹丹等[5]研究了Novozym 435在两相体系中合成白酒中四大酯类物质的能力,结果表明己酸乙酯在有机相合成转化率最高,丁酸乙酯在水相合成转化率最高。丝状真菌黑曲霉因其独特的食品安全特性和优良的蛋白质分泌系统和修饰能力,已成为食品酶生产的重要宿主之一[6]。2007年,黑曲霉CBS513.88基因组被完全解析并公布[7],生物信息学预测发现其基因组中存在大量编码脂肪酶基因的开放阅读框。此后,黑曲霉脂肪酶得到了广泛的关注。2009年,SHU等[8-9]首次克隆得到黑曲霉F044脂肪酶基因并实现在大肠杆菌和毕赤酵母中的异源表达。2020年,XING等[10]克隆黑曲霉胞外脂肪酶基因EXANL1在毕赤酵母中异源表达,研究了其结构与功能之间的关系。研究表明,黑曲霉脂肪酶种类繁多且其三维结构和催化特性也有着显著的差异[11-13],因此,研究不同类型黑曲霉脂肪酶的催化特性对其在食品工业中的应用具有非常重要的指导意义。
脂肪酸乙酯是白酒中重要的风味化合物[14],白酒中脂肪酸乙酯的合成主要依赖于羧酸水解酶家族、脂肪酶、酯酶和角质酶[15]。目前,关于脂肪酶催化酯化反应的研究主要集中在有机相体系中进行[16],其中少数酶在水相条件下可以催化酯的合成[17]。2020年,XU等[18]报道了一种来源于Monascus purpureus的羧酸酯酶在水相体系中催化脂肪酸乙酯的合成。本实验室早期的研究中发现黑曲霉可以显著提高白酒中脂肪酸乙酯的含量,因此对黑曲霉进行了转录组分析,本研究根据黑曲霉基因组注释,从中挑选出一个转录水平较高的脂肪酶基因LipA进行了生物信息学分析,成熟的脂肪酶Lip A由279个氨基酸组成,之后对该酶的酶学性质和酯化特性进行深入研究,为其在食品工业中得到有效应用奠定理论基础。
1 材料与方法
1.1 质粒与菌株
Escherichia coli DH5α、Escherichia coli Rosetta(DE3)、质粒pET-28a(+),均由本实验室保存。
1.2 主要试剂
限制性内切酶,大连宝生物工程有限公司;2×Taq Master Mix DNA聚合酶、同源重组酶、质粒提取试剂盒,Vazyme公司;IPTG、卡那霉素、氯霉素、Bradford蛋白浓度测定试剂盒,Solarbio公司;Ni柱、PD-10脱盐柱,GE公司;10 kDa超滤管,Millipore公司;对硝基苯酚丁酸酯等系列底物,Sigma公司;其他生化试剂均为国产分析纯,天津市江天化工技术有限公司。
1.3 重组菌株的构建及诱导表达
根据NCBI数据库中脂肪酶的氨基酸序列,将黑曲霉脂肪酶成熟蛋白编码序列(去掉信号肽及内含子)进行合成并根据大肠杆菌表达系统进行密码子优化。将合成后的片段以上、下游引物5′-TAAGAAGGAGATATACCATGGCGCCGACCCCGCTGGATG-3′和5′-ACGGAGCTCGAATTCGGATCCTTAATGATGATGATGATGATGCAGC-3′进行PCR扩增后通过同源重组与载体pET-28a(+)连接,重组产物转化至E.coli DH5α中,提取质粒并测序验证成功后,将重组质粒转化至E.coli Rosetta(DE3)中进行诱导表达。将重组菌株在37 ℃、220 r/min条件下培养至OD600=0.6~0.8,加入终浓度为1 mmol/L的IPTG溶液,在25 ℃、220 r/min条件下诱导发酵20 h后,离心收集菌体,使用pH 7.4 PBS洗涤菌体后重悬菌体。使用高压均质机破碎菌体收集上清液即为粗酶液,将粗酶液经Ni柱进行洗脱,洗脱后的重组蛋白使用PD-10和超滤管去除咪唑分子,蛋白质浓度测定使用Bradford试剂盒测定。
1.4 脂肪酶Lip A的酶活力测定及气相检测
1.4.1 脂肪酶活力测定
参照文献[19]的方法进行。500 μL反应体系中包括400 μL的Tris-HCl缓冲液(20 mmol/L、pH 8.0)、50 μL的pNPB(10 mmol/L)和50 μL酶液,37 ℃下反应5 min,500 μL无水乙醇终止反应,410 nm下测量吸光度值。酶活力定义:在一定温度和pH下,1 min内催化生成1 μmol对硝基苯酚所需要的酶量定义为一个活力单位(U)。
1.4.2 气相色谱的检测
气相色谱检测条件:色谱柱为HP-INNOWax(30 m×0.32 mm×0.5 μm),检测器为氢火焰离子化检测器(flame ionization detector, FID)。进样器温度260 ℃,进样量1.0 μL,检测器温度300 ℃。柱温在65 ℃保持5 min,以5 ℃/min上升到150 ℃,保持2 min,共24 min。载气N2,色谱柱流速0.8 mL/min。
1.5 脂肪酶Lip A的生化特性分析
按照1.4.1的方法,对脂肪酶进行生化特性分析,每个反应平行3次,以最高酶活力定义为100%,计算相对酶活力。
(1)最适温度和热稳定性
在不同温度(20~70 ℃)测定重组脂肪酶的酶活力,将酶液分别放置在不同温度(40~70 ℃)保温,间隔2 h取样,以未经处理的酶的酶活力定义为100%,测定残余酶活力。
(2)最适pH和pH稳定性
在不同pH条件下(pH 5.0~11.0)测定重组脂肪酶的酶活力,将酶液分别置于pH 4.0~10.0的不同缓冲液中,于4 ℃放置1 h后,测定残留酶活力。所用缓冲液为:20 mmol/L Na3PO4缓冲液(pH 4.0~6.0)、20 mmol/L磷酸钠缓冲液(pH 6.0~7.0)、20 mmol/L Tris-HCl缓冲液(pH 7.0~9.0)、20 mmol/L甘氨酸-KOH缓冲液(pH 9.0~11.0)。
(3)金属离子和螯合剂对酶活力的影响
在反应体系中加入不同金属离子(Co2+、Cu2+、Fe3+、Mn2+、Ca2+、Mg2+、Zn2+、Li2+)和金属螯合剂至终浓度为1和10 mmol/L,以未处理酶的酶活力为100%,计算相对酶活力。
(4)有机试剂耐受性
将酶液与有机试剂按照9∶1(体积比)混合均匀后,置于4 ℃放置1 h后,对照组使用缓冲液代替有机试剂,以未处理酶的酶活力为100%,计算相对酶活力。
(5)底物特异性及动力学参数测定
分别配制10 mmol/L不同碳链长度的对硝基苯酚酯(p-nitrophenyl, pNP)为底物,测量脂肪酶的酶活力。使用异丙醇配制不同浓度的底物,测定脂肪酶在不同浓度下的酶活力,利用Graphpad Prime 7.0 软件中的Michaelis-Menten方程拟合得到不同底物的动力学参数[20]。
1.6 脂肪酶Lip A酯化乳酸乙酯特性分析
参考文献[21-22]的方法,乳酸和乙醇浓度分别为0.3和1.2 mol/L,溶剂为PBS,反应体系为5 mL,酶10 mg,40 ℃,摇床转速200 r/min,反应时间12 h。同时以高温灭活的脂肪酶Lip A作空白对照离心,取上清液。留作气相分析。
(1)温度对酯化反应的影响
低温下脂肪酶无法发挥应有的催化活性,温度过高又会使脂肪酶变性而无法进行催化反应,因此找到合适的催化温度会大大提高乳酸乙酯合成量,按照上述的反应体系,反应温度在30、40、50、60 ℃操作,计算催化乳酸乙酯生成量。
(2)反应体系中酸醇物质的量比对酯化反应的影响
因为乳酸的成本高于乙醇,同时乳酸的电离系数高,含量过多会使整个体系的pH急剧下降,因此固定乳酸含量,改变乙醇的含量探究酸醇比对酯化反应的影响。按照上述的反应体系,酸醇物质的量比为1∶1、1∶2、1∶3、1∶4,计算催化乳酸乙酯生成量。
1.7 脂肪酶生物信息学分析
使用The Lipase Engineering Database(LED,http://www.led.uni-stuttgart.de/)BLAST查询与脂肪酶Lip A亲缘性较近的蛋白序列,使用MEGA 7.0软件进行系统发育树分析,采用邻域连接法(neighbor-joining, NJ)构建系统发育树。
2 结果与讨论
2.1 黑曲霉脂肪酶Lip A表达菌株的构建及其表达纯化
构建质粒pET-28a(+)-LipA的结果如图1-a所示,重组质粒提取验证如图1-b所示,重组质粒大小为6 127 bp。重组菌经诱导、菌体破碎以及Ni柱纯化后SDS-PAGE结果如图1-c所示,目的蛋白条带在大约27 kDa处出现,蛋白的分子质量大小与理论值一致,采用PD-10去除咪唑小分子,测定纯化后的脂肪酶Lip A测定比酶活力为58.31 U/mg,超滤后的脂肪酶Lip A比酶活力为132.4 U/mg,纯化倍数为2.27倍。

a-表达质粒pET-28a(+)-LipA图谱;b-重组质粒(M-Marker; 1-重组质粒6 127 bp);c-SDS-PAGE分析(M-Protein Marker; 1-重组脂肪酶粗酶液;2-纯化流穿液;3-高盐洗脱液; 4-纯化后样品)
图1 脂肪酶Lip A表达质粒的构建及诱导表达结果分析
Fig.1 Construction and induced expression of lipase Lip A
2.2 黑曲霉脂肪酶Lip A的生化性质解析
2.2.1 温度与pH对脂肪酶Lip A的影响
如图2-a所示,脂肪酶Lip A最适温度为50 ℃,且在40~60 ℃下孵育8 h后残余酶活力均在60%以上(图2-b),随着时间延长,重组酶活力出现显著下降。当孵育温度升高至70 ℃时,酶活力下降较快,8 h后其残余酶活力仅为40%左右(图2-b),此时酶的构象在高温下已经被破坏,导致酶的不可逆变性[23]。

a-最适反应温度;b-温度稳定性;c-最适反应pH;d-pH稳定性
图2 温度和pH对脂肪酶Lip A活性的影响
Fig.2 Effect of temperature and pH on the activity of lipase Lip A
如图2-c所示,脂肪酶Lip A的最适pH为8.0,在pH 6.0~8.0,酶活力保持在70%以上,具有较宽泛的pH适用范围,当pH>10后,酶活力骤降,表明该酶不适应在极端碱性条件下使用。如图2-d所示,在pH 4.0的环境下孵育1 h后,重组酶剩余酶活力依然可以保持在60%左右,在pH 5.0出现降低是由于脂肪酶Lip A的等电点在这一范围,该酶在酸性及弱碱性条件下具有较好的稳定性可能取决于蛋白质的一级结构,环境pH的变化会影响蛋白质中三维结构中离子键的改变,从而导致酶的构象发生变化[24]。
2.2.2 金属离子对脂肪酶Lip A的影响
如表1所示,在1 mmol/L金属离子条件下,Ca2+和Mg2+对脂肪酶Lip A具有促进作用,其他金属离子则有不同程度的抑制作用。在10 mmol/L金属离子条件下,Ca2+对于脂肪酶Lip A的促进作用更加明显,相较于对照提高了55%。而高浓度的Mg2+对脂肪酶Lip A的酶活力却由低浓度时的促进作用转变为抑制作用,其他金属离子对酶活力的抑制作用也进一步增强,其中Cu2+、Fe3+等最为明显,重金属离子作为亲电试剂可能与脂肪酶中的氨基酸残基发生电荷相互作用,改变其构象,从而使脂肪酶变性[25]。
表1 金属离子及螯合剂对脂肪酶Lip A活性的影响 单位:%
Table 1 Effects of metal ions and chelating agent on the activity of lipase Lip A

浓度/(mmol/L)金属离子或螯合剂对照Co2+Fe3+Cu2+Mn2+Zn2+Mg2+Ca2+Li+EDTA1100±2.4576.78±1.2729.18±3.5676.20±0.7985.70±1.7377.30±2.8107.25±3.66122.70±0.3491.67±3.2180.90±4.7210100±1.6166.27±1.2710.32±2.9314.72±1.7378.40±0.933.62±3.6897.37±4.12155.73±2.7990.65±4.6250.59±1.45
2.2.3 有机试剂对脂肪酶Lip A的影响
对照组用PBS替代有机溶剂,实验组中酶液与有机溶剂按照9∶1(体积比)混合。如图3所示,正庚烷、正己烷明显提高了脂肪酶Lip A的催化活性,正庚烷对该酶活性提高50%左右;醇对脂肪酶Lip A的酶活力基本无明显影响,其他有机溶剂则呈现出不同的抑制作用。脂肪酶的结构通常由疏水的催化核心和亲水的表面构成,其催化活性需要一定极性的溶剂来维持,正庚烷和己烷作为极性溶剂使得蛋白构象发生改变,底物更容易进入催化口袋进行催化反应[26],然而大多数有机溶剂则会削弱蛋白质的疏水键,使酶分子内部斥力增加,造成蛋白质的三级结构延展,最终导致脂肪酶丧失催化活性[27]。由此可见,脂肪酶Lip A可以耐受有机相的环境,为其在有机相中应用奠定了基础。

图3 有机试剂对脂肪酶Lip A活性的影响
Fig.3 Effect of organic solvents on the activity of lipase Lip A
2.2.4 脂肪酶Lip A底物特异性分析
由图4可见,脂肪酶Lip A作用于对硝基苯酚乙酸酯(pNP-C2)的水解活性最高,作用于对硝基苯酚月桂酸酯(pNP-C12)和对硝基苯酚棕榈酸酯(pNP-C16)的水解活性分别为作用于pNP-C2水解活性的23.04%和9.71%,可见,脂肪酶Lip A偏好于水解中短链对硝基苯酚酯,对长链底物表现出较低的水解活性。

图4 脂肪酶Lip A的底物特异性
Fig.4 Substrate specificity of lipase Lip A
米氏常数Km代表酶与底物的亲和力,Km越小表明酶与底物的亲和力最强,以pNP-C2为底物时,该酶的Km为(0.228±0.05) mmol/L(表2),表明pNP-C2为脂肪酶Lip A的最佳底物,脂肪酶Lip A对C2~C8的对硝基苯酚酯等具有较高水解酶活性,表明该酶更可能为羧酸酯酶,而不是真正的脂肪酶[28-29]。
表2 脂肪酶Lip A动力学参数
Table 2 Kinetic parameters of lipase Lip A

底物Km/(mmol·L-1)Kcat/(s-1)Kcat/Km/[L·(s·mol)-1]对硝基苯酚乙酸酯(pNP-C2)0.228±0.050.023±0.001102.01±2.31 对硝基苯酚丁酸酯(pNP-C4)0.989±0.0970.086±0.01393.048±14.62对硝基苯酚己酸酯(pNP-C6)0.553±0.0730.118±0.016213.54±30.47对硝基苯酚辛酸酯(pNP-C8)0.817±0.1550.102±0.014125.27±17.18
2.3 脂肪酶Lip A酯化特性分析
脂肪酶Lip A催化脂肪酸乙酯的研究中发现,脂肪酶Lip A在水相条件下可以催化乳酸和乙醇合成乳酸乙酯。温度对其催化乳酸和乙醇合成乳酸乙酯的影响结果如图5-a所示,50 ℃时脂肪酶催化乳酸乙酯合成含量最高,为40.81 mg/L,实验结果表明脂肪酶Lip A在较低温度下无法最大程度发挥催化活性,而较高的温度又会使酶失活,使其蛋白结构发生不可逆性损伤[23]。由图5-b可知,随着乙醇浓度的增加,乳酸乙酯生成量逐渐降低,说明乙醇作为一种短链醇,同时也作为酶的失活剂,高浓度的乙醇会破坏酶的三维构象,并最终导致酶的催化效率降低。

a-最佳反应温度;b-最佳酸醇物质的量比
图5 脂肪酶Lip A催化合成乳酸乙酯的最佳反应 温度和最佳酸醇物质的是比
Fig.5 Optimum reaction temperature and substrate molar ratio for the synthesis of ethyl lactate by lipase Lip A
2.4 脂肪酶Lip A系统发育树的构建
蛋白序列相似性分析是蛋白质研究过程中不可或缺的一步,构建系统进化树可以快速准确地概括不同生物体间的亲缘关系,进而反映进化关系[30]。本研究中利用LED数据库比对脂肪酶Lip A编码的氨基酸序列,发现该酶属于abH23同源家族[29],abH23同源家族中包含大量不同来源的脂肪酶序列,它们被划分为4个组(图6),脂肪酶Lip A被划分到Rhizomucor mihei组,该酶与来源于米曲霉的二酰基甘油酯酶(BAA12912.1)和来源于粗糙脉孢菌的三酰基甘油酯酶(EAA32130.1)同属于一个进化分支,表明它们有共同的祖先,并且可能在某些催化特性方面具有相似的性质。
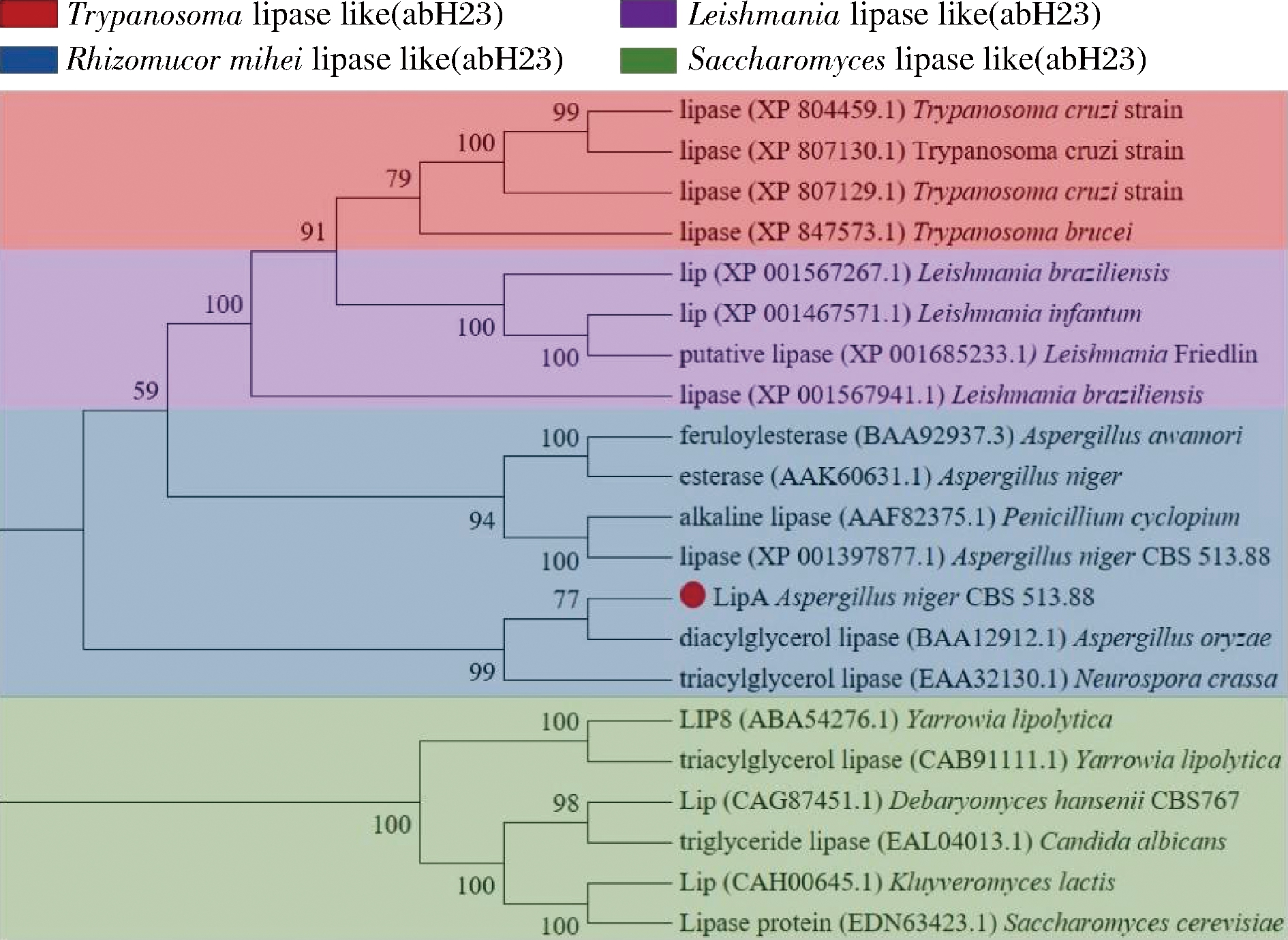
图6 脂肪酶Lip A与abH23同源家族的系统进化树
Fig.6 Phylogenetic tree of lipase Lip A and abH23 homologous family
3 结论
本论文成功克隆表达了来源于黑曲霉CBS513.88中具有潜在脂肪酶催化特性的Lip A蛋白,该酶的蛋白分子质量约为27 kDa。酶学性质分析表明,脂肪酶Lip A的最适温度和pH分别为50 ℃和8.0,且在40~60 ℃和pH 5.0~8.0有较好的稳定性。该酶对有机试剂有较好的耐受性,Ca2+、Mg2+、正庚烷和正己烷对脂肪酶Lip A的水解活性有促进作用,该酶对不同长度碳链底物均表现出活性,动力学参数表明其作用于对硝基苯酚乙酸酯时催化活性最高。序列相似性分析结果表明脂肪酶Lip A为abH23同源家族Rhizomucor mihei脂肪酶中的一员。此外,该酶可以在水相介质催化乳酸乙酯的合成,这一特性使其在食品加工与酿酒工业中具有一定的应用价值。
[1] HILLS G.Industrial use of lipases to produce fatty acid esters[J].European Journal of Lipid Science and Technology, 2003, 105(10):601-607.
[2] STERGIOU P Y, FOUKIS A, FILIPPOU M, et al.Advances in lipase-catalyzed esterification reactions[J].Biotechnology Advances, 2013, 31(8):1 846-1 859.
[3] ANGAJALA G, PAVAN P, SUBASHINI R.Lipases:An overview of its current challenges and prospectives in the revolution of biocatalysis[J].Biocatalysis and Agricultural Biotechnology, 2016, 7:257-270.
[4] FILHO D G,SILVA A G, GUIDINI C Z..Lipases:Sources, immobilization methods, and industrial applications[J].Applied Microbiology and Biotechnology, 2019, 103(18):7 399-7 423.
[5] 王丹丹, 林萌莉, 周晓宏.脂肪酶对白酒四大酯的可逆合成与分解[J].食品科技, 2014, 39(12):74-78;83.
WANG D D, LIN M L, ZHOU X H.Reversible synthesis and hydrolysis of four main esters in Chinese liquor catalyzed by lipase[J].Food Science and Technology, 2014, 39(12):74-78;83.
[6] LI C, ZHOU J W, DU G C, et al.Developing Aspergillus niger as a cell factory for food enzyme production[J].Biotechnology Advances, 2020, 44:107630.
[7] PEL H J, DE WINDE J D, ARCHER D B, et al.Genome sequencing and analysis of the versatile cell factory Aspergillus niger CBS 513.88[J].Nature Biotechnology, 2007, 25(2):221-231.
[8] SHU Z Y, YAN Y J, YANG J K, et al.Aspergillus niger lipase:Gene cloning, over-expression in Escherichia coli and in vitro refolding[J].Biotechnology Letters, 2007, 29(12):1 875-1 879.
[9] SHU Z Y, DUAN M J, YANG J K, et al.Aspergillus niger lipase:Heterologous expression in Pichia pastoris, molecular modeling prediction and the importance of the hinge domains at both sides of the lid domain to interfacial activation[J].Biotechnology Progress, 2009, 25(2):409-416.
[10] XING S Q, ZHU R N, LI C Q, et al.Gene cloning, expression, purification and characterization of a sn-1,3 extracellular lipase from Aspergillus niger GZUF36[J].Journal of Food Science and Technology, 2020, 57(7):2 669-2 680.
[11] FERN NDEZ-LORENTE G, ORTIZ C, SEGURA R L, et al.Purification of different lipases from Aspergillus niger by using a highly selective adsorption on hydrophobic supports[J].Biotechnology and Bioengineering, 2005, 92(6):773-779.
NDEZ-LORENTE G, ORTIZ C, SEGURA R L, et al.Purification of different lipases from Aspergillus niger by using a highly selective adsorption on hydrophobic supports[J].Biotechnology and Bioengineering, 2005, 92(6):773-779.
[12] MHETRAS N C, BASTAWDE K B, GOKHALE D V.Purification and characterization of acidic lipase from Aspergillus niger NCIM 1207[J].Bioresource Technology, 2009, 100(3):1 486-1 490.
[13] HARIDASAN NAMBOODIRI V M, CHATTOPADHYAYA R.Purification and biochemical characterization of a novel thermostable lipase from Aspergillus niger[J].Lipids, 2000, 35(5):495-502.
[14] XU Y Q, ZHAO J R, LIU X, et al.Flavor mystery of Chinese traditional fermented Baijiu:The great contribution of ester compounds[J].Food Chemistry, 2022, 369(1):130920.
[15] LEVISSON M, VAN DER OOST J, KENGEN S W M.Carboxylic ester hydrolases from hyperthermophiles[J].Extremophiles, 2009, 13(4):567-581.
[16] KRUIS A J, BOHNENKAMP A C, PATINIOS C, et al.Microbial production of short and medium chain esters:Enzymes, pathways, and applications[J].Biotechnology Advances, 2019, 37(7):107407.
[17] SUN J C, YU B, CURRAN P, et al.Optimisation of flavour ester biosynthesis in an aqueous system of coconut cream and fusel oil catalysed by lipase[J].Food Chemistry, 2012, 135(4):2 714-2 720.
[18] XU Y Q, WANG X C, LIU X, et al.Discovery and development of a novel short-chain fatty acid ester synthetic biocatalyst under aqueous phase from Monascus purpureus isolated from Baijiu[J].Food Chemistry, 2021, 338:128025.
[19] YAO J, GUI L, YIN S C.A novel esterase from a soil metagenomic library displaying a broad substrate range[J].AMB Express, 2021, 11(1):38.
[20] KARI J, ANDERSEN M, BORCH K, et al.An inverse Michaelis-Menten approach for interfacial enzyme kinetics[J].ACS Catalysis, 2017, 7(7):4 904-4 914.
[21] 赵江, 胡松青.响应曲面法优化非水相酶催化合成乳酸乙酯[J].现代食品科技, 2009, 25(11):1 320-1 322;1 319.
ZHAO J, HU S Q.Optimization of lipase-catalyzed synthesis of ethyl lactate in non-aqueous media by response surface methodology[J].Modern Food Science and Technology, 2009, 25(11):1 320-1 322;1 319.
[22] XU Y Q, HUANG H Q, LU H Y, et al.Characterization of an Aspergillus niger for efficient fatty acid ethyl ester synthesis in aqueous phase and the molecular mechanism[J].Frontiers in Microbiology, 2022, 12:820380.
[23] ZHANG X, LI X Q, XIA L M.Expression of a thermo-alkaline lipase gene from Talaromyces thermophilus in recombinant Pichia pastoris[J].Biochemical Engineering Journal, 2015, 103:263-269.
[24] SCHWEIKER K L, MAKHATADZE G I.Protein stabilization by the rational design of surface charge-charge interactions[J].Methods in Molecular Biology (Clifton, N.J.), 2009, 490:261-283.
[25] SINCHAIKUL S, SOOKKHEO B, PHUTRAKUL S, et al.Optimization of a thermostable lipase from Bacillus stearothermophilus P1:Overexpression, purification, and characterization[J].Protein Expression and Purification, 2001, 22(3):388-398.
[26] KOUTINAS M, YIANGOU C, OS RIO N M, et al.Application of commercial and non-commercial immobilized lipases for biocatalytic production of ethyl lactate in organic solvents[J].Bioresource Technology, 2018, 247:496-503.
RIO N M, et al.Application of commercial and non-commercial immobilized lipases for biocatalytic production of ethyl lactate in organic solvents[J].Bioresource Technology, 2018, 247:496-503.
[27] BORNSCHEUER U T.Microbial carboxyl esterases:Classification, properties and application in biocatalysis[J].FEMS Microbiology Reviews, 2002, 26(1):73-81.
[28] PETROVSKAYA L E, NOVOTOTSKAYA-VLASOVA K A, SPIRINA E V, et al.Expression and characterization of a new esterase with GCSAG motif from a permafrost metagenomic library[J].FEMS Microbiology Ecology, 2016, 92(5):fiw046.
[29] FISCHER M, PLEISS J.The Lipase Engineering Database:A navigation and analysis tool for protein families[J].Nucleic Acids Research, 2003, 31(1):319-321.
[30] ZHANG Y P, CHEN P C, GAO Y, et al.DBP-PSSM:Combination of evolutionary profiles with the XGBoost algorithm to improve the identification of DNA-binding proteins[J].Combinatorial Chemistry & High Throughput Screening, 2022, 25(1):3-12.