东北三省素有“大豆之乡”的美誉,其大豆制品通常分为发酵豆制品和非发酵豆制品。传统发酵大豆食品包括豆酱、豆豉、豆浆、酱油、臭豆腐与腐乳等产品[1]。而东北传统大酱是以大豆为原料,经天然存在的细菌和真菌发酵而成的半固体状调味食品[2]。在厌氧发酵过程中,厌氧菌快速繁殖,微生物群落的动态变化会对大酱发酵产生一定的影响[3],蛋白质在酶的作用下分解为小分子的肽、氨基酸,从而产生独特风味。因此传统发酵大豆制品营养丰富、质地独特、风味醇厚,且在其发酵过程中会发生非酶褐变并产生褐色物质——类黑精,即大豆中富含的蛋白质及其分解产物与还原糖类发生美拉德反应的产物[4-5]。类黑精作为非消化性高分子物质,除具有食物纤维的生理功能外[6],还具有降血压[7]、抗氧化[8]、抗肿瘤[9]等多种作用生物活性。
但传统豆酱发酵工艺为自然发酵,因此其品质因参与发酵的微生物种类而异。基于16S rRNA与26S rRNA基因的非培养技术能够揭示微生物群落结构,为规避传统微生物群落分析的局限性而开发的各种非培养的方法中,变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)分析是用于快速、经济地研究微生物多样性的最广泛应用的分子方法之一。一般在聚丙烯酰胺凝胶电泳时,双链DNA分子迁移距离由其分子大小与电荷决定,因此同样长度的DNA片段在胶中的迁移距离相同,从而不能被区分出来,DGGE技术在一般的聚丙烯酰胺凝胶基础上加入了变性剂梯度,从而能够把同样长度但序列不同的DNA片段区分开来。
因此,本研究以自制的韩式大酱与中式大酱为原料,不进行微生物培养,直接提取样品总DNA,采用聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophoresis, PCR-DGGE)测定分子指纹图谱,比较生物体所携带基因中的保留性结合序列(conserved region)来确认微生物的变化,分析中式与韩式大酱中的优势菌群,对传统酱类由原材料和加工方法引起的发酵微生物群落变化进行补充,为大酱的标准化生产与质量控制提供理论依据。
1 材料与方法
1.1 材料与试剂
琼脂糖,上海源叶生物科技有限公司;PBS、TAE缓冲液,江苏恩莫阿赛生物技术有限公司;DNA提取试剂盒,广州健仑生物科技有限公司;聚丙烯酰胺凝胶试剂盒,上海生工生物工程有限公司;SYBR Green,Invitrogen公司。
1.2 仪器与设备
FA1104分析天平,上海天平仪器有限公司;Z400K冷冻离心机,莱比信(中国)科技发展有限公司;SX-700高压蒸汽灭菌器,日本TOMY公司;Bio-Rad T100型PCR仪、电泳仪、变形梯度电泳仪、GelDoc XR凝胶成像仪,伯乐生命医学产品(上海)有限公司;SW-CJ-IFD超净工作台,上海新苗医疗器械机械制造有限公司。
1.3 实验方法
1.3.1 韩式大酱制作工艺流程
大豆→浸泡→沥水蒸煮→破碎→造型→稻草捆绑→吊挂→自然发酵→酱曲
酱曲→清洗晾晒→破碎→装坛→加盐水(25%,质量分数,下同)→自然发酵→去除酱油→破碎→装坛→撒食盐→白布封口→封盖发酵
1.3.2 中式大酱制作工艺流程
大豆→浸泡→沥水蒸煮→破碎→造型→包装纸包装→自然发酵→酱曲
酱曲→清洗晾晒→破碎→装坛→加盐水(25%)→自然发酵
1.3.3 大酱样品DNA提取
分别称取10 g样品与20 mL PBS(pH 7.0)混匀均质并经4层滤布过滤,将滤液于4 ℃下400×g离心5 min,弃上清液,沉淀用PBS重复洗涤3次后重新加入3 mL PBS,使用基因组DNA提取试剂盒提取其DNA,并在10 g/L琼脂糖凝胶上对DNA的产量与质量进行电泳分析,将提取DNA作为PCR模板以扩增16S rRNA与26S rRNA基因[10]。
1.3.4 PCR扩增
PCR反应体系均为50 μL,体系包括5 μL 10×Buffer(含有Mg2+),4 μL 25 mmol/L dNTP混合物,1 U Taq DNA聚合酶,每种引物50 pmol,DNA模板用量为1 μL。Reconditioning PCR用来消除普通PCR过程中的杂合双链DNA污染。
A:16S rRNA基因V3区PCR扩增条件:引物为341F GC和518R。PCR反应程序为94 ℃预变性4 min后进入30个循环,94 ℃ 1 min;65 ℃ 1 min(注:每循环二次后温度降低1 ℃);72 ℃ 1 min,最后72 ℃延伸10 min。Reconditioning PCR反应程序:94 ℃预变性4 min后进入循环,94 ℃ 1 min:65 ℃ 1 min;72 ℃ 1 min,5至10个循环后72 ℃延伸10 min。
B:26S rRNA基因PCR扩增条件:引物为NL1(5′-GCATATCAATAAGCGGAGGAAAAG-3′)与LS2(5′- ATTCCCAAACAACTCGACTC-3′),并且在NL1的5′端上加入富含GC的DNA片段(5′-CGCCCGCCGC GCGGCGGGCGGGGCGGGGGC-3′)。PCR反应程序为94 ℃预变性4 min进入30个循环,94 ℃ 1 min;50 ℃ 1 min;72 ℃ 1 min,最后72 ℃延伸10 min。Reconditioning PCR反应程序:94 ℃预变性4 min后进入循环,94 ℃ 1 min;55 ℃ 1 min;72 ℃ 1 min,8个循环后72 ℃延伸10 min。
1.3.5 DGGE
PCR产物采用Bio-Rad DCode检测系统进行DGGE分析。将80 g/L聚丙烯酰胺凝胶置于TAE缓冲液中,上样后在100 V下电泳200 min,并在40%~60%的变性梯度下实现了最佳分离,然后用SYBR green染色45 min,使用GelDoc XR凝胶成像仪对凝胶进行拍照[11]。
1.3.6 DGGE条带回收、PCR扩增与测序
参考郑艳等[12]的方法,将DGGE后的凝胶,置于凝胶成像仪上,选取目标条带切割后放入无菌离心管中,加入60 μL的灭菌去离子水清洗后加入60 μL的灭菌去离子水并于4 ℃静置过夜。取储存样品液2 μL作为PCR反应模板,进行PCR扩增并测序。
2 结果与分析
2.1 韩式大酱与中式大酱中的细菌多样性分析
扩增大酱样品中微生物的16S rRNA基因,并通过变性梯度凝胶电泳方法调查细菌的群落分布。韩式大酱PCR-DGGE结果如图1所示,并对出现的每个条带进行碱基序列分析,鉴定结果如表1所示。韩式大酱中芽孢杆菌属(Bacillus)为优势种,其次为不动杆菌属(Acinetobacter)、四联球菌属(Tetragenococcus)和嗜盐单胞菌属(Halomonas)。芽孢杆菌在发酵过程中能分泌多种酶类与抗生素,可协助真菌分解大豆中的蛋白质和淀粉。此外,酱曲在清洗晾晒、破碎装坛后使用盐水浸泡发酵,分离出的四联球菌属和单胞菌属都具有一定的耐盐性,是大豆产品发酵过程中常接种的微生物之一[13],可用于酿造酱油、腐乳、黄豆酱等制品中,改善产品风味。

图1 韩式大酱中16S rRNA扩增产物的DGGE指纹图谱
Fig.1 DGGE fingerprints of 16S rRNA amplification products in Korean soybean paste
表1 韩式大酱中PCR-DGGE指纹图谱上条带的测序结果
Table 1 Sequencing results of PCR-DGGE fingerprints in Korean soybean paste

编号菌株名称NCBI登陆号相似度C1Tetragenococcus halophilus IAM 1673EU689054100%C2Lactobacillus pobuzihiiAB32636798%C3Bacillus amyloliquefaciens KFRI-GS4-1KJ56086999%C4Tetragenococcus halophilus NBRC 12172AP01204699%C5Bacillus polyfermenticus BF1-1GQ86152199%C6Acinetobacter gyllenbergii S-B9DKJ80648499%C7Bacillus amyloliquefaciens ARC365KJ38114699%C8Bacillus methylotrophicus SCKB 1354KJ46979999%C9Uncultured bacterium clone Ll142-1O12FJ67175497%C10Tetragenococcus halophilus NBRC 12172AP012046100%C11Bacillus amyloliquefaciens KFRI-GS4-1KJ56086999%C12Bacillus amyloliquefaciens KFRI-GS4-1KJ56086999%C13Bacillus subtilis Bac3EU55767196%C14Bacillus casamancensis TN3JQ41597699%C15Bacillus safensis HA 552KJ535359100%C16Uncultured Peptostreptococcaceae bacteri-um clone BR16BF04DQ85721994%C17Bacillus subtilis XB157KF44742099%C18Bacillus amyloliquefaciens KFRI-GS4-1KJ56086999%C19Uncultured bacterium clone A1-H10GU08356292%C20Bacillus amyloliquefaciens SCKB 1329KJ46979299%C21Acinetobacter gyllenbergii S-B9DKJ806484100%C22Acinetobacter junii T-B7BKJ80646099%C23Halomonas cibimaris 10-C-3NR_10854097%C24Acinetobacter gyllenbergii S-B9DKJ806484100%C25Nocardiopsis alba DW-10KJ55653499%C26Acinetobacter gyllenbergii S-B9DKJ806484100%
中式大酱PCR-DGGE结果如图2所示,并对出现的每个条带进行碱基序列分析,鉴定结果如表2所示。在中式大酱中,乳酸菌如片球菌属(Pediococcus)、乳杆菌属(Lactobacillus)、明串珠菌属(Leucanostoc)和肠杆菌属(Enterobacter)均为优势种,且与芽孢杆菌属相比分布更多。
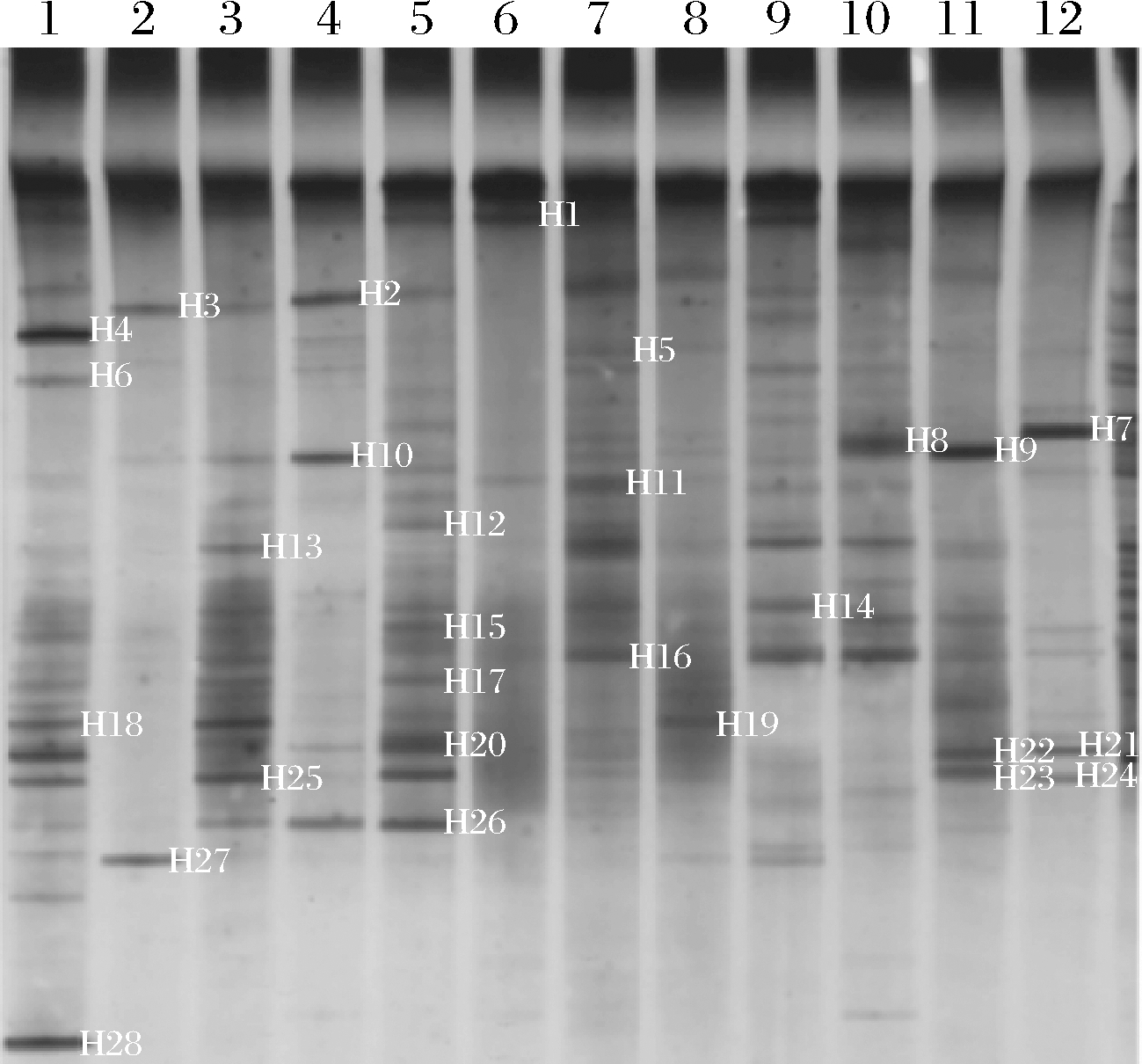
图2 中式大酱中16S rRNA扩增产物的DGGE指纹图谱
Fig.2 DGGE fingerprints of 16S rRNA amplification products in Chinese soybean paste
表2 中式大酱中PCR-DGGE指纹图谱上条带的测序结果
Table 2 Sequencing results of PCR-DGGE fingerprints in Chinese soybean paste

编号菌株名称NCBI登陆号相似度H1Bacillus subtilis P1HQ62549199%H2Bacillus amyloliquefaciens KFRI-GS4-1KJ56086999%H3Bacillus amyloliquefaciens ARC365KJ38114699%H4Clostridium arbusti SL206NR_116458100%H5Lactobacillus curvatus 24-3KJ47738599%H6Acinetobacter gyllenbergii S-B9DKJ806484100%H7Bacillus flexus KJ1-5-910KJ623600100%H8Acinetobacter gyllenbergii S-B9DKJ80648499%H9Pediococcus acidilactici TUB/2013/3-5KJ531397100%H10Halomonas cibimaris 10-C-3NR_10854097%H11Bacillus subtilis LCB56FJ86792599%H12Leuconostoc mesenteroides Ycay2anJQ44651993%H13Uncultured bacterium clone K.2_99KF783333100%H14Tetragenococcus halophilusAB91154388%H15Pediococcus acidilactici TUB/2013/3-5KJ531397100%H16Enterococcus durans D4KJ70257799%H17Bacillus mojavensis SWFU16KJ75633599%H18Pediococcus acidilactici TUB/2013/3-5KJ53139799%H19Lactobacillus pobuzihiiAB32636799%H20Lactobacillus pobuzihiiAB32636799%H21Bacillus amyloliquefaciens DBT57KJ58489999%H22Lactobacillus pobuzihiiAB32636799%H23Enterobacter hormaecheiAB93112799%H24Bacillus amyloliquefaciens SCKB 1329KJ46979299%H25Bacillus subtilis P1HQ62549199%H26Kocuria kristinae 7L1HQ28485290%H27Corynebacterium variabileNR10287499%
乳酸菌是人体肠道系统中最主要的有益菌,通常在发酵食品中分离得到,在成品酱风味形成过程中也起到了重要作用,能产生良好的感官风味和丰富的营养物质[14-16]。在大酱发酵过程中,乳酸菌将酪氨酸、精氨酸、组氨酸分解,对苏氨酸、丝氨酸、苯丙氨酸等进行异型脱羧基作用从而生成多类化合物,这些化合物对豆酱风味形成有重要的贡献。
左丽丽等[17]对传统发酵豆酱中乳酸菌进行分离和鉴定,初步推断出戊糖片球菌是吉林市农家酱发酵过程中的优势乳酸菌群,对传统豆酱的风味起着至关重要的作用。此外,不动杆菌属、四联球菌属和嗜盐单胞菌属也显示为优势种。
2.2 韩式大酱与中式大酱中的真菌多样性分析
扩增大酱样品中微生物的26S rRNA基因,并通过变性梯度凝胶电泳方法调查真菌的群落分布,韩式大酱PCR-DGGE结果如图3所示,并对出现的每个条带进行碱基序列分析,鉴定结果如表3所示。

图3 韩式大酱中26S rRNA扩增产物的DGGE指纹图谱
Fig.3 DGGE fingerprints of 26S rRNA amplification products in Korean soybean paste
表3 韩式大酱中PCR-DGGE指纹图谱上条带的测序结果
Table 3 Sequencing results of PCR-DGGE fingerprints in Korean soybean paste

编号菌株名称NCBI登陆号相似度1Rhizomucor endophyticus CBS 385.95HM62331391%2Penicillium solitum 20-01JN642222100%3Penicillium solitum 20-01JN642222100%4Galactomyces geotrichum LMA-70JQ668740100%5Aspergillus oryzae SEMCC-3.248HM064501100%6Tyrophagus putrescentiae voucher AHBLoo511AY499563100%7Circinella umbellataAF15712899%8Mucor plumbeus UPSC 1492AF54807899%9Mucor plumbeus UPSC 1492AF548078100%10Rhizopus oryzae CS1217GU12637596%11Exophiala halophila CBS 121512JN85601593%12Exophiala halophila CBS 121512JN85601592%13Mucor plumbeus UPSC 1492AF548078100%14Rhizomucor endophyticus CBS 385.95HM62331399%15Rhizomucor endophyticus CBS 385.95HM623313100%16Rhizomucor endophyticus CBS 385.95HM62331399%17Penicillium chrysogenum ZJ-T2HQ88217799%18Mucor circinelloides f. circinelloides WA0000017591HM64168999%19Mucor circinelloides f. circinelloides WA0000017591HM64168999%20Penicillium solitum 20-01JN64222299%21Mucor circinelloides f. circinelloides WA0000017591HM64168999%22Penicillium solitum 20-01JN642222100%23Rhizomucor endophyticus CBS 385.95HM62331399%
韩式大酱的真菌分布呈多样性,根毛霉属(Rhizomucor)、青霉菌属(Penicillium)、毛霉属(Mucor)、外瓶霉属(Exophiala)、曲霉属(Aspergillus)等分布广泛。其中,曲霉属通常能够分泌较复杂酶系,如蛋白酶、谷氨酰胺酶、淀粉酶、果胶酶、纤维素酶等,且具有较强的酶活性,能利用单糖、淀粉、低聚糖、酒精、有机酸等作为碳源,利用氨基酸、尿素、蛋白质等作为氮源,广泛应用于食品发酵[18];毛霉属能产生果胶酶、凝乳酶和脂肪酶等,可用于制曲和酿酒[19]。因此,有必要对大酱的微生物多样性进行研究,了解大酱的微生物群落结构,充分利用优势菌株。
中式大酱PCR-DGGE结果如图4所示,并对出现的每个条带进行碱基序列分析,鉴定结果如表4所示。从表4可以看出,鲁氏接合酵母(Zygosaccharomyces rouxii)分布较多,其次是熊蜂生斯塔莫酵母(Starmerella bombicola)但与韩式大酱相比,其真菌种类较为单一。

图4 中式大酱中26S rRNA扩增产物的DGGE指纹图谱
Fig.4 DGGE fingerprints of 26S rRNA amplification products in Chinese soybean paste
鲁氏接合酵母是豆酱发酵应用相对较多的酵母菌,该菌具有较好的酒精发酵力,且耐盐性极强、抗高渗透压,在高盐浓度下可发酵麦芽糖、葡萄糖产生甘露醇、甘油、乙醇、阿拉伯糖醇、琥珀酸、异戊醇、异丁醇等,此外还可以与乳酸菌代谢产物作用生成酯类物质,产生大酱的特有香味。
近年来,Starmerella属因其优良的发酵特性引起了很多研究兴趣[20],Starmerella bombicola与Starmerella bacillaris是该属的典型种[21-22],TU等[23]分离鉴定出Starmerella davenportii Do18并进行初步的发酵研究和发酵产物的检测,结果发现该菌能产生浓郁的香气和有机酸,降低胆固醇,对高糖、低pH、胆盐等各种抑制剂具有耐受性,并能模拟胃肠环境。
表4 中式大酱中PCR-DGGE指纹图谱上条带的测序结果
Table 4 Sequencing results of PCR-DGGE fingerprints in Chinese soybean paste
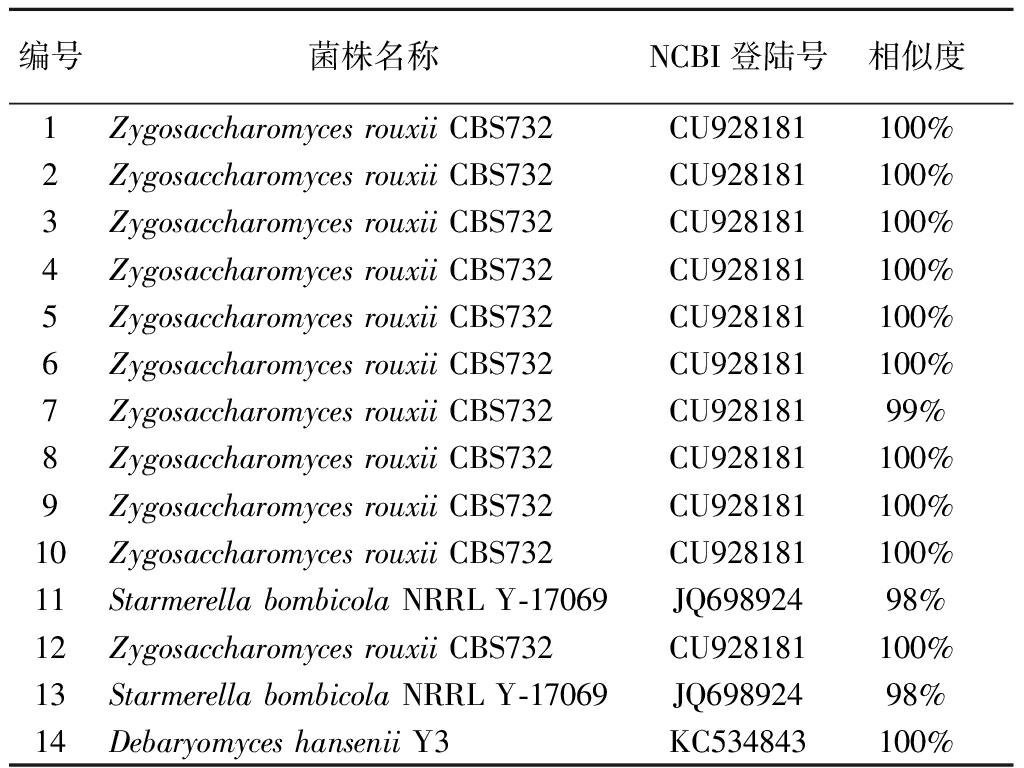
编号菌株名称NCBI登陆号相似度1Zygosaccharomyces rouxii CBS732CU928181100%2Zygosaccharomyces rouxii CBS732CU928181100%3Zygosaccharomyces rouxii CBS732CU928181100%4Zygosaccharomyces rouxii CBS732CU928181100%5Zygosaccharomyces rouxii CBS732CU928181100%6Zygosaccharomyces rouxii CBS732CU928181100%7Zygosaccharomyces rouxii CBS732CU92818199%8Zygosaccharomyces rouxii CBS732CU928181100%9Zygosaccharomyces rouxii CBS732CU928181100%10Zygosaccharomyces rouxii CBS732CU928181100%11Starmerella bombicola NRRL Y-17069JQ69892498%12Zygosaccharomyces rouxii CBS732CU928181100%13Starmerella bombicola NRRL Y-17069JQ69892498%14Debaryomyces hansenii Y3KC534843100%
3 结论
韩式与中式大酱由于制作方法与水分含量的不同,其微生物的分布也不同。制作韩式大酱时,采用稻草捆绑酱曲进行发酵,枯草芽孢杆菌可由稻草分离得到,因此韩式大酱中的芽孢杆菌属较有优势,且制作过程中去除了酱油(酱卤),其水分含量低,适合霉菌生长。此外,分离出的四联球菌属与嗜盐单胞菌属具有一定的耐盐性,是豆制品发酵过程中常接种的微生物之一。
中式大酱的制作过程中未去除酱油,水分含量高,多为乳酸菌,如片球菌属、乳杆菌属、明串珠菌属、肠杆菌属等,这些菌株可将氨基酸进行异型脱羧基从而生成多类化合物,在大酱风味形成过程中起到了重要作用。此外,鲁氏接合酵母与熊蜂生斯塔莫酵母不仅具有一定的耐盐性,发酵过程中产生的醇类物质还可与乳酸菌产生的酸类物质发生酯化反应,从而赋予大酱独特的酱香味。
本研究成功提取了大酱宏基因组DNA,并经PCR扩增靶基因后可较完整地反映相应微生物群落组成,显示出了菌群多样性,从未培养角度证实了微生物在大酱发酵过程中具有重要地位。但PCR-DGGE只能分析在总微生物数量上占有一定优势的微生物,由于其分析的DNA片段较短,信息量较小,所以只能分析到一定范围内。综上所述,DGGE可以作为分析豆酱中微生物组成的方法,对豆酱中微生物种类进行估算,其精确分析则需要结合其他方法。此外,要了解微生物的生态功能,尤其是特殊菌种在大酱生产中的具体应用,仍需要得到它们的纯培养,因此传统分离培养仍然是研究微生物多样性不可缺少的手段。本研究对大酱的微生物区系结构进行了初步探讨,但对与之相对应的功能还有待进一步研究。
[1] 张蒙冉, 李淑英, 高雅鑫, 等.传统发酵豆制品研究进展[J].食品科技, 2021, 46(1):98-104.
ZHANG M R, LI S Y, GAO Y X, et al.Research progress of traditional fermented soybean products[J].Food Science and Technology, 2021, 46(1):98-104.
[2] 安飞宇, 武俊瑞, 尤升波, 等.基于宏转录组学技术对豆酱中活菌群落分析方法的建立[J].食品科学, 2020, 41(4):96-101.
AN F Y, WU J R, YOU S B, et al.Metatranscriptomic analysis of the microbial community in soybean paste, a traditional Chinese fermented condiment[J].Food Science, 2020, 41(4):96-101.
[3] WU J R, TIAN T, LIU Y M, et al.The dynamic changes of chemical components and microbiota durifng the natural fermentation process in Da-Jiang, a Chinese popular traditional fermented condiment[J].Food Research International, 2018, 112:457-467.
[4] 王璋. 食品化学[M].北京:中国轻工业出版社.1991:77-83.
WANG Z.Food Chemistry[M].Beijing:China Light Industry Press, 1991:77-83.
[5] KIM M J, KWAK H S, KIM S S.Effects of salinity on bacterial communities, Maillard reactions, isoflavone composition, antioxidation and antiproliferation in Korean fermented soybean paste (doenjang)[J].Food Chemistry, 2018, 245(15):402-409.
[6] BORRELLI R C, FOGLIANO V.Bread crust melanoidins as potential prebiotic ingredients[J].Molecular Nutrition & Food Research, 2005, 49(7):673-678.
[7] 徐清萍. 食品类黑精的功能、结构及制备研究进展[J].中国酿造, 2008, 27(22):13-15.
XU Q P.Function, structure and preparation of melanoidins[J].China Brewing, 2008, 27(22):13-15.
[8] HABINSHUTI I, CHEN X, YU J, et a1.Antimicrobial, antioxidant and sensory properties of Maillard reaction products (MRPs) derived from sunflower, soybean and corn meal hydrolysates[J].LWT-Food Science and Technology, 2019, 101:694-702.
[9] 韩易, 赵燕, 徐明生, 等.美拉德反应产物类黑精的研究进展[J].食品工业科技, 2019, 40(9):339-345.
HAN Y, ZHAO Y, XU M S, et al.Research progress on the melanoidins of Maillard reaction product[J].Science and Technology of Food Industry, 2019, 40(9):339-345.
[10] LEE S M, LEE S, SINGH D, et al.Comparative evaluation of microbial diversity and metabolite profiles in doenjang, a fermented soybean paste, during the two different industrial manufacturing processes[J].Food Chemistry, 2017, 221:1 578-1 586.
[11] LYU X C, JIANG Y J, LIU J, et al.Evaluation of different PCR primers for denaturing gradient gel electrophoresis (DGGE) analysis of fungal community structure in traditional fermentation starters used for Hong Qu glutinous rice wine[J].International Journal of Food Microbiology, 2017, 255:58-65.
[12] 郑艳, 姚婷.PCR-DGGE分析甘薯酸浆自然发酵过程中细菌多样性[J].食品科学, 2016, 37(7):99-103.
ZHENG Y, YAO T.Analysis of bacterial diversity during natural fermenation of sweet potato sour liquid by PCR-DGGE[J].Food Science, 2016, 37(7):99-103.
[13] 王博, 周朝晖, 李铁桥, 等.嗜盐四联球菌及其在发酵食品中的应用[J].食品与发酵工业, 2017, 43(8):267-272.
WANG B, ZHOU Z H, LI T Q, et al.Tetragenococcus halophilus and its application in fermented foods[J].Food and Fermentation Industries, 2017, 43(8):267-272.
[14] WASSIE M, WASSIE T.solation and identification of lactic acid bacteria from Iranian camel milk[J].International Journal of Advanced Biological and Biomedical Research, 2020, 8(1):67-74.
[15] TESHOME G.Review on lactic acid bacteria function in milk fermentation and preservation[J].African Journal of Food Science, 2015, 9(4):170-175.
[16] STELLA CAPPELLO M, ZAPPAROLI G, LOGRIECO A, et al.Linking wine lactic acid bacteria diversity with wine aroma and flavour[J].International Journal of Food Microbiology, 2017, 243:16-27.
[17] 左丽丽, 高永欣, 王钰涓, 等.传统发酵豆酱中乳酸菌的分离及鉴定[J].中国调味品, 2020, 45(11):117-120.
ZUO L L, GAO Y X, WANG Y J, et al.Separation and identification of lactic acid bacteria in traditionally fermented soybean paste[J].China Condiment, 2020, 45(11):117-120.
[18] 黄永光, 徐岩.酱香白酒酿造环境曲霉的分离及Aspergillus hennebergii酶分泌胁迫条件[J].食品与生物技术学报, 2015, 34(8):814-821.
HUANG Y G, XU Y.Isolation of Aspergillus from Jiangxiang liquor fermentation environment and enzyme secretion stress conditions of Aspergillus hennebergii[J].Journal of Food Science and Biotechnology, 2015, 34(8):814-821.
[19] 沈弘洋, 邓微, 赵云珠, 等.传统大豆酱不同发酵阶段微生物多样性变化[J].食品与发酵工业, 2021, 47(23):118-124.
SHEN H Y, DENG W, ZHAO Y Z, et al.Changes of microbial diversity in different fermentation stage of traditional soybean paste[J].Food and Fermentation Industries, 2021, 47(23):118-124.
[20] DE GRAEVE M, DE MAESENEIRE S L, ROELANTS S L K W, et al.Starmerella bombicola, an industrially relevant, yet fundamentally underexplored yeast[J].FEMS Yeast Research, 2018, 18(7):foy072.
[21] ENGLEZOS V, RANTSIOU K, CRAVERO F, et al.Starmerella bacillaris and Saccharomyces cerevisiae mixed fermentations to reduce ethanol content in wine[J].Applied Microbiology and Biotechnology, 2016, 100(12):5 515-5 526.
[22] ENGLEZOS V.Starmerella bacillaris in winemaking:Opportunities and risks[J].Current Opinion in Food Science, 2017, 17:30-35.
[23] TU C H, HU W X, TANG S J, et al.Isolation and identification of Starmerella davenportii strain Do18 and its application in black tea beverage fermentation[J].Food Science and Human Wellness, 2020, 9(4):355-362.