血红素作为一种含铁卟啉化合物,广泛存在于各种生物体中[1],其对细胞的转录、翻译和蛋白质后修饰等起着一定的调控作用[2-3]。这些生理功能使其在食品、医学诊断等领域具有广泛的应用前景[4]。随着市场需求不断扩大,当前通过动植物提取法获得的血红素已无法满足日益增加的应用需求。此外,动植物提取法存在原料来源有限且产品质量不稳定等缺点。利用合成生物学技术理性设计细胞代谢网络,以廉价生物质为底物发酵生产血红素是近来的研究热点,且该方法具有价格低廉、产物纯度高和环境友好等优势,被寄予厚望。
近年来,随着合成生物学的快速发展,研究人员已成功实现多种生物基化学品的工业化生产,如丙酮酸、聚谷氨酸等[5-7]。在血红素的生物合成方面,研究人员通过优化C4和C5途径来合成血红素取得了一定的成果,但是存在一些不足。KWON等[8]通过在大肠杆菌中构建C4途径以及优化胞内代谢流,实现了血红素的合成,但发现C4途径合成血红素的效率非常低。此外,ZHAO等[9]在大肠杆菌中通过理性优化C5合成途径、调控血红素合成的下游途径、补加合成前体等策略,显著改善了血红素的积累,并发现C5途径合成血红素的效率明显高于C4途径,但获得血红素合成的底物转化率仅为0.000 138 mol/mol(血红素/葡萄糖),远低于理论转化率。因此,如何提升C5途径合成血红素的底物转化率是下一步研究的重点。与大多数天然产物合成途径一样,血红素的生物合成途径非常长且涉及到28个途径基因(从葡萄糖到合成血红素)。目前,几乎所有血红素生物合成研究都是采用多个质粒在一个宿主细胞中表达合成途径基因的策略,这会对菌株的生长产生不利影响。为避免在单一宿主细胞中设计天然产物合成途径造成的胞内代谢流紊乱和细胞代谢压力增高等问题,共培养体系设计策略被提出并用于构建细胞生长与产物合成平衡、无有毒中间代谢产物累积、生产效率显著提高的细胞工厂,取得了令人瞩目的成绩[10-11]。例如,ZHOU等[12]将紫杉醇前体的合成途径分为2个模块,分别构建在酿酒酵母和大肠杆菌中,通过进一步的途径优化实现了紫杉醇前体的高效合成。
本课题组前期研究获得了1株谷氨酸非依赖性高产聚谷氨酸的解淀粉芽孢杆菌(Bacillus amyloliquefaciens) NX-2S154,该菌株具有较高的三羧酸循环代谢通量和谷氨酸合成代谢流。此外,对该菌株的代谢途径进行相应的设计和优化后,实现了其利用葡萄糖等碳源高效合成聚谷氨酸等产品[13-15]。B.amyloliquefaciens NX-2S154菌株的这些特点为理性设计C5途径合成血红素的研究提供了一个难得的底盘菌株。本研究以具有高谷氨酸合成代谢通量的解淀粉芽孢杆菌NX-2S154和具有异源酶高表达能力的大肠杆菌BL21为出发菌株,通过将血红素的合成途径分为前体5-氨基乙酰丙酸(5-aminolevulinic acid, 5-ALA)合成模块和5-ALA合成血红素模块,分别理性设计在解淀粉芽孢杆菌与大肠杆菌中构建血红素生物合成的共培养体系,并对获得的共培养体系进行进一步的代谢途径设计和过程优化实现了血红素的合成,以期提高菌株发酵的底物转化率。
1 材料与方法
1.1 材料
1.1.1 菌株
解淀粉芽孢杆菌NX-2S154以及大肠杆菌Escherichia coli BL21(DE3)为本实验室保藏菌株,大肠杆菌GM2163用于质粒的去甲基化。本研究所用菌株与质粒详见表1,质粒构建所有引物详见表2。
表1 菌株和质粒
Table 1 Strains and plasmids

菌株与质粒特征性来源菌株NX-2S154Bacillus amyloliquefaciens with high yield of γ-PGA本实验室B.amy-0Deletion of genes pgsBCA in strain NX-2S154 chromosome本研究B.amy-1B.amy-0 harboring plasmid pMA5-gltXE.coli -hemAE.coli - hemAE.coli本研究B.amy-2B.amy-0 harboring plasmid pMA5-gltXB.amy-hemAB.amy- hemLE.coli本研究B.amy-3Deletion of gene ldh in strain B.amy-2 chromosome本研究B.amy-4Deletion of gene pta in strain B.amy-2 chromosome本研究B.amy-5Deletion of gene nas in strain B.amy-2 chromosome本研究B.amy-6Deletion of gene prob in strain B.amy-2 chromosome本研究B.amy-7Deletion of genes prob、ldh、pta and nas in strain B.amy-2 chromosome本研究B.amy-8B.amy-7 harboring gene rhtA本研究E.coli-aBL21(DE3) harboring plasmid pRSF Duet1-hemB-hemC-hemD-hemE-hemF-hemG-hemH本研究E.coli-bE.coli-a harboring gene ccmABC本研究质粒pMA5Kanr, Ampr, Expression vector with two replicons, shuttle plasmid, HpaII pro-moter本实验室pRSFDuet1Kanr, Expression vector with two MCS, T7 promoter本实验室pHT01-cas9npHT01 carrying gene cas9n本实验室pDR-sgpgsBCApDR244 carrying gene sgpgsBCA本研究pDR-sgldhpDR244 carrying gene sgldh本研究pDR-sgptapDR244 carrying gene sgpta本研究pDR-sgnaspDR244 carrying gene sgnas本研究pDR-sgprobpDR244 carrying gene sgprob本研究pMA5-gltX E.coli -hemAE.coli- hemLE.colipMA5 carrying gene gltX from E.coli,hemA from E.coli and hemL from E.coli本研究pMA5-gltX E.coli -hemAE.coli- hemLB.amypMA5 carrying gene gltX from E.coli,hemA from E.coli and hemL from B.amy本研究pMA5-gltX E.coli -hemAB.amy- hemLB.amypMA5 carrying gene gltX from E.coli,hemA from B.amy and hemL from B.amy本研究pMA5-gltX B.amy-hemAB.amy- hemLE.colipMA5 carrying gene gltX from B.amy,hemA from B.amy and hemL from E.co-li本研究pMA5-gltX E.coli -hemAB.amy- hemLE.colipMA5 carrying gene gltX from E.coli,hemA from B.amy and hemL from E.coli本研究pMA5-gltX B.amy -hemAB.amy- hemLB.amypMA5 carrying gene gltX from B.amy,hemA from B.amy and hemL from B.amy本研究pMA5-gltX B.amy -hemAE.coli- hemLE.colipMA5 carrying gene gltX from B.amy,hemA from E.coli and hemL from E.coli本研究pMA5-gltX B.amy -hemAE.coli- hemLB.amypMA5 carrying gene gltX from B.amy,hemA from E.coli and hemL from B.amy本研究pRSF Duet1-hemBCDEFGHpRSF Duet1 carrying gene hemB、hemC、hemD、hemE、hemF、hemG and hemH本研究pRSF Duet1-hemBCDEFGH-ccmABCpRSF Duet1 carrying gene hemB、hemC、hemD、hemE、hemF、hemG、hemH and cc-mABC本研究
表2 本研究所使用引物
Table 2 Primers used in the study

引物序列 (5’-3’)sgpgsBCA-FAAAAAGGAGCGATTTAGTCGACTCTCCCGCTGCTCAGCACGAGTTTTAGAGCTAGAAATAGCAsgpgsBCA-RTTTGAATCGGCTGTACGAAAAAAAAAAGCACCGACTCGGTGCCACTTTTTC pgsBCA-UP-FGAAAAAGTGGCACCGAGTCGGTGCTTTTTTTTTTCGTACAGCCGATTCAAApgsBCA-UP-RAAGCGGATTACAAAAGA TCCGTGATTTTTGACATCTCCTTCTpgsBCA-DN-FAGAAGGAGATGTCAAAAATCACGGATCTTTTGTAATCCGCTTpgsBCA-DN-RAGGTGTTTTTTTATTACCCTCGAGAAAACAAATGAAAATCATTAApgsBCA-out-FCTCCTGAACGATCGGA TTATCAGCCAGCTCpgsBCA-out-RAATCTCCGCAAACA CACATCAGTTCCTGCAgltX1-FAAAAGGAGCGATTTACATATGATGAAAATCAAAACTCGCTTCgltX1-RACCGAGTGCTAAAAGGGTCATATGTAAATCGCTCCTTTTTACTGCTGATTTTCGCGTTChemA1-FGAACGCGAAAATCAGCAGTAAAAAGGAGCGATTTACATATGACCCTTTTAGCACTCGGThemA1-RAAGATTTTCAGACTTACTCATATGTAAATCGCTCCTTTCTACTCCAGCCCGAGGCTGTChemL1-FGACAGCCTCGGGCTGGAGTAGAAAGGAGCGATTTACATATGAGTAAGTCTGAAAATCTThemL1-RGAGCTCGACTCTAGAGGATCCTCACAACTTCGCAAACACCCGgltX2-RTCCCACAACAAGAATATGCATATGTAAATCGCTCCTTTTTACTGCTGATTTTCGCGTTChemA2-FGAACGCGAAAATCAGCAGTAAAAAGGAGCGATTTACATATGCATATTCTTGTTGTGGGAhemA2-RTGATTTTTCATAGCTTCTCATATGTAAATCGCTCCTTTTCACTCACTTACAAGCGGACGhemL2-FCGTCCGCTTGTAAGTGAGTGAAAAGGAGCGATTTACATATGAGAAGCTATGAAAAATCAhemL2-RGAGCTCGACTCTAGAGGATCC TCAGCGGCTGATCGCCTGGAAhemA3-RAAGATTTTCAGACTTACTCATATGTAAATCGCTCCTTTTCACTCACTTACAAGCGGACGhemL3-FCGTCCGCTTGTAAGTGAGTGAAAAGGAGCGATTTACATATGAGTAAGTCTGAAAATCTThemA4-RTGATTTTTCATAGCTTCTCATATGTAAATCGCTCCTTTCTACTCCAGCCCGAGGCTGTChemL4-FGACAGCCTCGGGCTGGAGTAGAAAGGAGCGATTTACATATGAGAAGCTATGAAAAATCAgltX5-FAAAAGGAGCGATTTACATATGATGGGAAATGAAGTACGCGTTgltX5-RTCCCACAACAAGAATATGCATATGTAAATCGCTCCTTTTTAAATGCTTTTCAGGCGTCThemA5-FAGACGCCTGAAAAGCATTTAAAAAGGAGCGATTTACATATGCATATTCTTGTTGTGGGAhemA5-RTGATTTTTCATAGCTTCTCATATGTAAATCGCTCCTTTTCACTCACTTACAAGCGGACGhemL5-FCGTCCGCTTGTAAGTGAGTGAAAAGGAGCGATTTACATATGAGAAGCTATGAAAAATCAhemL5-RGAGCTCGACTCTAGAGGATCC TCAGCGGCTGATCGCCTGGAAgltX6-RACCGAGTGCTAAAAGGGTCATATGTAAATCGCTCCTTTTTAAATGCTTTTCAGGCGTCThemA6-FAGACGCCTGAAAAGCATTTAAAAAGGAGCGATTTACATATGCATATTCTTGTTGTGGGAhemL7-RTCTGGCTTCAAGCATACCCACATGTAAATCGCTCCTTTTCACAACTTCGCAAACACCCGccmABC-FCGGGTGTTTGCGAAGTTGTGAAAAGGAGCGATTTACATGTGGGTATGCTTGAAGCCAGAccmABC-RGAGCTCGACTCTAGAGGATCCTCATTTACGGCCTCTTTTCAGhemB-FATAAGGAGATATACCATGGGCATGACAGACTTAATCCAACGChemB-RTCTTAAAACATTGTCTAACAT CTCCTT TTAACGCAGAATCTTCTTCTChemC-FGAGAAGAAGATTCTGCGTTAA AAGGAG ATGTTAGACAATGTTTTAAGAhemC-RGCGGGTGACAAGGATACTCAT CTCCTT TCATGCCGGGGCGTCTCCGTThemD-FAACGGAGACGCCCCGGCATGA AAGGAG ATGAGTATCCTTGTCACCCGChemD-RCTGGTGTGCGTCGGGTTTCAT CTCCTT TTATTGTAATGCCCGTAAAAGhemE-FCTTTTACGGGCATTACAATAA AAGGAG ATGACCGAACTTAAAAACGAThemE-RGCATTATGCGGCCGCAAGCTT TTAGCGGTGATACTGTTCAGAhemF-FTAAGAAGGAGATATACATATG ATGAAACCCGACGCACACCAGhemF-RGAAAAGAATTAATGTTTTCAC CTCCTT TTACACCCAATCCCTGACCTThemG-FAAGGTCAGGGATTGGGTGTAA AAGGAG GTGAAAACATTAATTCTTTTChemG-RACCGGTTTTAGTCTGACGCAT CTCCTT GACAAACCGACGCTGAAATAAhemH-FGACAAACCGACGCTGAAATAAAAGGAGATGCGTCAGACTAAAACCGGThemH-RGGTTTCTTTACCAGACTCGAG TTAGCGATACGCGGCAACAAGrhtA-FCGGGTGTTTGCGAAGTTGTGAAAAGGAGCGATTTACATATGCCTGGTTCATTACGTAAArhtA-RGGTTTCTTTACCAGACTCGAG TTAATTAATGTCTAATTCTTTsgpta-FAAAAAGGAGCGATTTAGTCGACTTTGACCCGAATACATATGAGTTTTAGAGCTAGAAATsgpta-RATGACAACTGGTACATGCTTTCTAAAAAAAGCACCGACTCGGTGCCACpta-UP-FGTGGCACCGAGTCGGTGCTTTTTTTAGAAAGCATGTACCAGTTGTCATpta-UP-RAATTAACGCAAGATTGTAGACGTCTTTTCCTGCTACTTTTTCTTGTACpta-DN-FGTACAAGAAAAAGTAGCAGGAAAAGACGTCTACAATCTTGCGTTAATT
续表2

引物序列 (5’-3’)pta-DN-RAGGTGTTTTTTTATTACCCTCGAGTTTGCAATTGCCGTCTGCTGTATGpta-out-FTCGTCAAAACCGACAGATCCCGTGATAATCTGTTTGACTTTGCCGpta-out-RGGGGATTGCGTTCTCAAGCAAATACTTGCTGAAAGCAGGCATTATsgldh-FAAAAAGGAGCGATTTAGTCGACGCCATTTTCAAAAGCATTGTGTTTTAGAGCTAGAAATsgldh-RATTGTATCAGTCATCCTCTATATCAAAAAAGCACCGACTCGGTGCCACldh-UP-FGTGGCACCGAGTCGGTGCTTTTTTGATATAGAGGATGACTGATACAATldh-UP-RGAGAATATCTTTCAGCACGTTTGCACCTGCTCCGATTAATGCTACTTTldh-DN-FAAAGTAGCATTAATCGGAGCAGGTGCAAACGTGCTGAAAGATATTCTCldh-DN-RAGGTGTTTTTTTATTACCCTCGAGTTTCCTTTACGGAGCCACATTTGTldh-out-FAATAGAACTTTCCGGCATCGGGAAGCGGTTCGGATGACTGGACATldh-out-RTCTTTATCGCCTTTTTTATTGTGGAAAGAAAAGCGGAAGAACCGAsgnas-FAAAAAGGAGCGATTTAGTCGACCGCTATTCAAAAAAAGATCTGTTTTAGAGCTAGAAATsgnas-RCGGCGCGGCTATGAACATGACATCAAAAAAGCACCGACTCGGTGCCACnas-UP-FGTGGCACCGAGTCGGTGCTTTTTTGATTCATAACCCGGCCGTGTATGAnas-UP-RGATTTTGATATAATCATAGGTTAAATACGCCGCCTTTTATTTTCGTGAnas-DN-FTCACGAAAATAAAAGGCGGCGTATTTAACCTATGATTATATCAAAATCnas-DN-RAGGTGTTTTTTTATTACCCTCGAGTCCTTGAAAGGCTTGATCAGTGAAnas-out-FGATGTCATGTTCATAGCCGCGCCGCCCGGCGTTTCCGGAGAGTGGnas-out-RGCGATTGTCCCTTTCGCTTTTTTGATATCGATAGGCCAGCGGCTG
1.1.2 主要试剂
氨苄青霉素、氯霉素、壮观霉素,上海麦克林生化科技有限公司;基因组抽提试剂盒、2×Phanta Max Master Mix、One Step Cloning Kit,南京诺唯赞生物科技股份有限公司;谷氨酸检测试剂盒,南京建成生物工程研究所;酵母粉、蛋白胨,OXOID公司。
1.1.3 培养基
LB(Luria-Bertani)培养基(g/L):酵母粉5,蛋白胨10,NaCl 10。
发酵培养基(g/L):葡萄糖50,酵母粉10,蛋白胨5,(NH4)2SO4 15,Na2HPO4·12H2O 10,KH2PO4 2,FeSO4·7H2O 0.05。
1.2 实验方法
1.2.1 发酵方法
5-ALA摇瓶发酵:发酵种子液培养12 h后以1%(体积分数,下同)的接种量接种于100 mL发酵培养基中,32 ℃,200 r/min摇床培养,发酵周期为54 h,定时取样。
血红素共培养体系摇瓶发酵:分别单独培养解淀粉芽孢杆菌菌株与大肠杆菌的发酵种子液12 h,各以1%的接种量转接于含有100 mL发酵培养基的摇瓶中,于32 ℃,200 r/min的摇床中培养3 h,加入终浓度为0.4 mmol/L的IPTG进行诱导,定时取样测定共培养体系中的血红素和生物量等。
7.5 L发酵罐发酵:发酵罐中装液量为3 L,解淀粉芽孢杆菌工程菌株与大肠杆菌工程菌株种子液分别培养12 h,各以1%的接种量同时转接到发酵罐中,发酵条件为32 ℃,400 r/min,通气量为1 vvm,共培养3 h后加入IPTG进行诱导并定时取样测定相关发酵参数。
1.2.2 转化方法
参考SHA等[13]的解淀粉芽孢杆菌转化方法进行相关质粒转化。
1.2.3 基因敲除与载体构建
利用QIU等[16]报道的CRISPR-cas9n基因组编辑技术对解淀粉芽孢杆菌的基因组DNA进行编辑。以敲除聚谷氨酸合酶基因pgsBCA为例,流程如下:以解淀粉芽孢杆菌基因组为模板,利用引物sgpgsBCA-F/R、pgsBCA-UP-F/R和pgsBCA-DN-F/R分别通过扩增得到crRNA-tracRNA、UPgene和DNgene基因片段。将上述基因片段通过重叠PCR进行连接,获得crRNA-tracRNA-UPgene-DNgene片段,接着,将上述片段组装到线性化的pDR244载体上,获得重组质粒pDR-sgpgsBCA。将pDR-sgpgsBCA以及pHT01-cas9n质粒(先前研究构建[16])同时转入解淀粉芽孢杆菌中。通过平板筛选和菌落PCR获得正确的阳性克隆子。挑取阳性转化子转接到液体LB培养基中,并加入IPTG诱导Cas9蛋白表达,利用PCR技术验证目的基因是否敲除成功。同样,利用上述方法对解淀粉芽孢杆菌的prob、nas、ldh和pta基因进行敲除以构建不同工程菌株。
基因表达质粒构建流程:以大肠杆菌MG1655基因组为模板,利用引物gltX1-F/R、hemA1-F/R和hemL1-F/R分别通过PCR扩增获得gltX、hemA、hemL基因片段,之后通过重叠PCR连接上述基因片段,获得gltX-hemA-hemL多基因片段。将gltX-hemA-hemL基因片段与线性化pMA5载体进行组装,构建成功后转入相应的解淀粉芽孢杆菌中,获得目的基因表达的工程菌株。此外,利用表2所示引物从大肠杆菌基因组上扩增获得hemB、hemC、hemD、hemE、hemF、hemG和hemH基因片段并按照上述质粒构建方法将其组装到pRSF-Duet1中,获得质粒pRSF Duet1-hemBCDEFGH。从大肠杆菌以及解淀粉芽孢杆菌基因组上扩增出不同来源gltX、hemA、hemL基因片段,通过重叠PCR进行连接,获得不同组合的gltX、hemA、hemL多基因片段。将不同组合的gltX-hemA-hemL基因片段与pMA5载体组装,获得不同的gltX、hemA、hemL表达质粒。以大肠杆菌基因组为模板,扩增获得rhtA以及ccmABC基因片段,将上述片段组装到相关的质粒上,获得表达质粒。
1.2.4 5-ALA与血红素检测方法
参考TAN等[17]的化学检测法检测5-ALA;参考ZHAO等[9]的液相检测方法检测血红素。
2 结果与分析
2.1 构建血红素生物合成的共培养体系
前体供应对于微生物细胞工厂合成目标产物至关重要[18]。谷氨酸作为血红素合成的前体,其胞内水平将在很大程度上影响血红素的合成。为了验证B.amyloliquefaciens NX-2S154在血红素生物合成过程中能否提供充足谷氨酸前体。首先,利用CRISPR-cas9n技术敲除B.amyloliquefaciens NX-2S154中编码聚谷氨酸合酶的基因pgsBCA,获得工程菌株B.amy-0(NX-2S154ΔpgsBCA)。接着,将出发菌B.amyloliquefaciens NX-2S154与工程菌株B.amy-0分别在LB平板上划线培养。通过对这2株菌株的菌落形态观察发现,在敲除pgsBCA基因后,菌落形态由饱满湿润变得扁平且十分干燥(图1-a、图1-b)。通过对上述2株菌株进行摇瓶发酵来验证聚谷氨酸合酶基因敲除前后的胞内谷氨酸含量以及菌株生长情况。由图1-c可知,工程菌株B.amy-0的OD600达到7.84,与原始菌株相比提升12.90%。此外,通过测定B.amyloliquefaciens NX-2S154以及B.amy-0菌株的胞内谷氨酸含量,发现B.amy-0菌株的胞内谷氨酸含量达到674.67 μg/g,相对于原始菌株(75.64 μg/g)提升了791.94%。这些结果充分说明B.amy-0菌株具有高效的胞内谷氨酸合成通量,可为血红素合成的共培养体系提供充足谷氨酸前体。

a-NX-2S154菌落形态;b-NX-2S154ΔpgsBCA菌落形态;c-聚谷氨酸合酶基因敲除前后菌株生长情况及胞内谷氨酸浓度
图1 NX-2S154与NX-2S154ΔpgsBCA菌落形态及发酵代谢产物产量
Fig.1 The NX-2S154 and NX-2S154ΔpgsBCA colony morphology and fermentation metabolite yield
以葡萄糖为底物合成血红素涉及到28个的途径酶。如将这些途径基因全部过表达于单个宿主细胞中将会严重影响菌株的胞内代谢流平衡。因此,我们将血红素的合成途径分为前体5-ALA合成模块和5-ALA合成血红素模块,分别构建于解淀粉芽孢杆菌与大肠杆菌中。在解淀粉芽孢杆菌中异源表达大肠杆菌来源的gltX,hemA和hemL基因构建前体5-ALA合成模块,获得工程菌株B.amy-1[NX-2S154ΔpgsBCA(pMA5-gltXE.coli-hemAE.coli-hemAE.coli)]。由图2-a可知,工程菌株B.amy-1在发酵过程中5-ALA的积累量不断增加,达到136.81 mg/L。而对照菌株B.amy-0仅存在微量的本底表达(约1.45 mg/L)。同时,在大肠杆菌中异源表达大肠杆菌来源的hemB、hemC、hemD、hemE、hemF、hemG和hemH基因构建血红素合成模块,获得工程菌株E.coli-a。将工程菌株B.amy-1和E.coli-a进行血红素的共培养发酵,获得7.84 mg/L的血红素积累。此时,共培养体系的5-ALA质量浓度相对较低,仅为76.62 mg/L,较B.amy-1菌株下降了43.99%(图2-b)。

a-B.amy-1工程菌株发酵过程中5-ALA产量;b-共培养体系发酵过程中5-ALA和血红素产量
图2 B.amy-1工程菌株以及共培养体系发酵参数
Fig.2 Fermentation parameters of co-cultivation system and B.amy-1 engineering strain
2.2 代谢工程优化共培养体系改善血红素合成
为了提高上述共培养体系合成血红素的能力,需要采取组合多种代谢工程策略对共培养体系进行进一步的代谢途径优化。首先,通过对不同宿主来源的5-ALA途径基因gltX、hemA、hemL进行基因表达的适配性优化,获得8种含有不同基因组合的工程菌株。这些工程菌株的摇瓶发酵结果表明工程菌株B.amy-2[NX-2S154ΔpgsBCA(pMA5- gltXB.Amy-hemAB.Amy-hemLE.coli)]的5-ALA产量提升最为明显,其5-ALA积累量达到298.72 mg/L,较对照菌株B.amy-1(136.8 mg/L)菌株提升了118.3%(图3)。因此,菌株B.amy-2将作为后续研究的出发菌株。
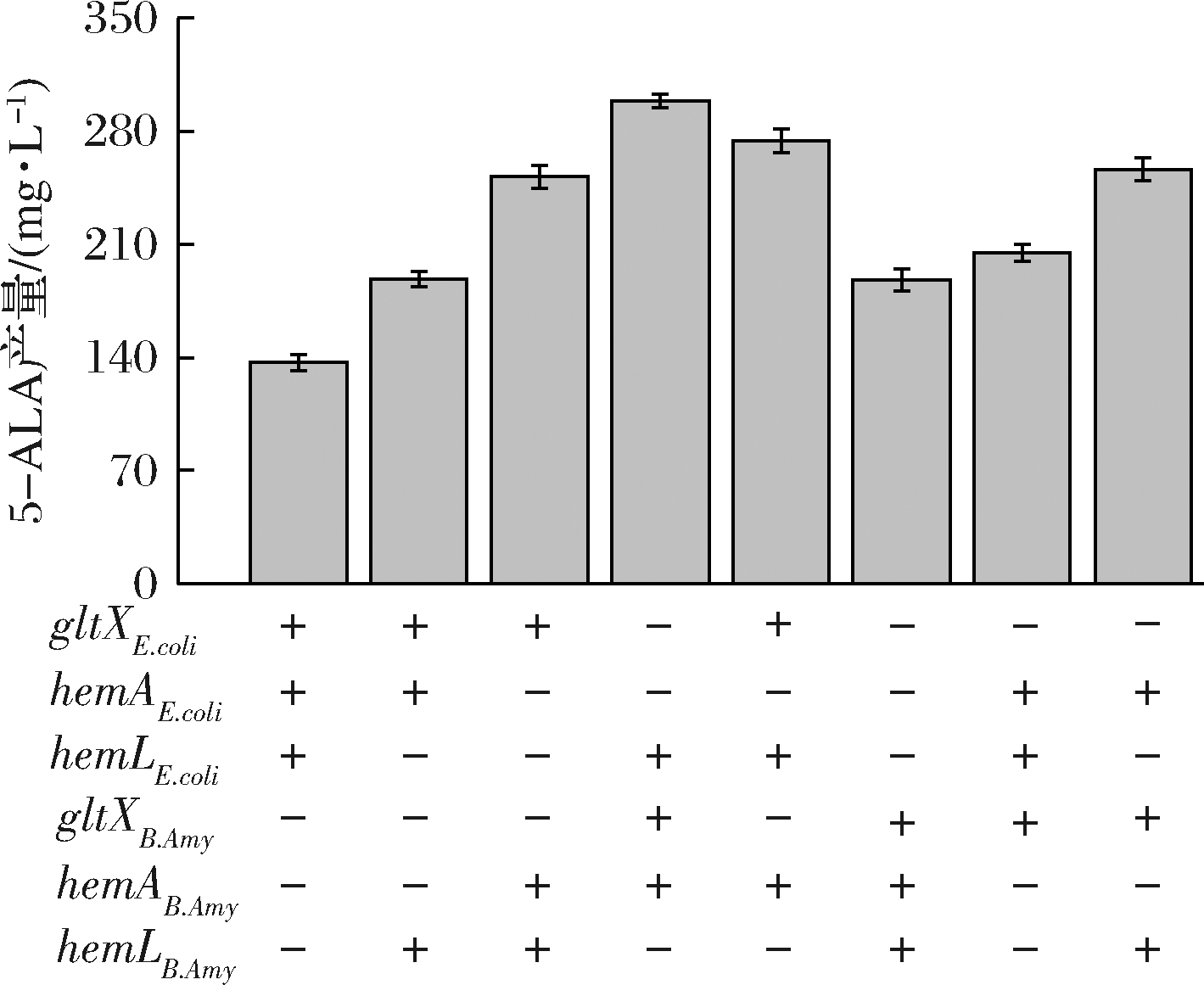
图3 不同来源5-ALA途径基因组合的产量
Fig.3 Titer of 5-ALA pathway gene combinations from different sources
阻断目标产物合成的竞争代谢通路是改善目标产物合成效率的一种高效策略[19]。为了改善共培养体系合成血红素的能力,利用CRISPR-cas9n技术分别敲除B.amy-2菌株的ldh、pta、nas以及prob基因来阻断副产物乳酸、乙酸、N-乙酰氨基谷氨酸以及脯氨酸的生物合成,获得B.amy-3、B.amy-4、B.amy-5和B.amy-6工程菌株。这些菌株的摇瓶发酵的5-ALA积累水平相对于B.amy-2菌株分别提升32.47%、29.40%、42.03%和6.01%(图4-a~图4-d)。
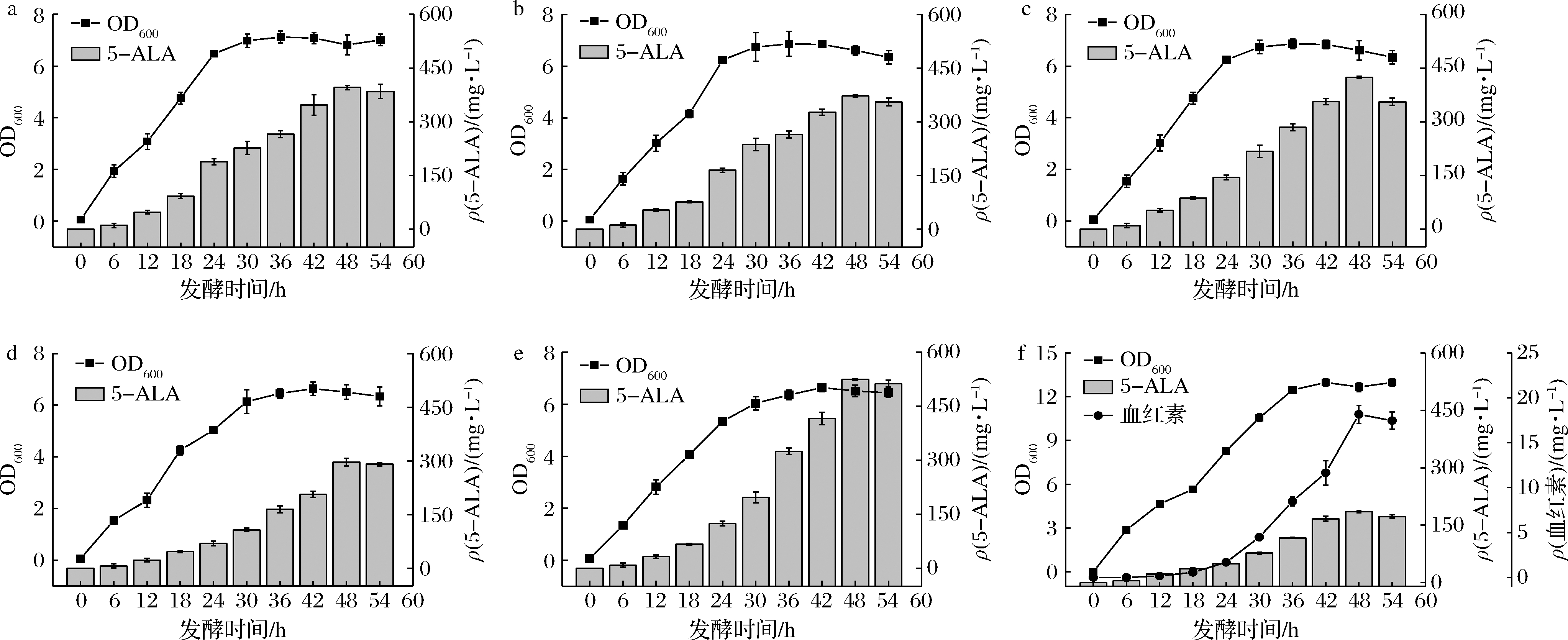
a-敲除ldh基因对菌株生长和5-ALA浓度的影响;b-敲除pta基因对菌株生长和5-ALA浓度的影响;c-敲除nas基因对菌株生长和5-ALA浓度的影响;d-敲除prob基因对菌株生长和5-ALA浓度的影响;e-敲除4个旁路基因对菌株生长和5-ALA浓度的影响;f-敲除4个旁路基因对共培养体系的影响
图4 敲除旁路基因对菌株生长、5-ALA以及共培养体系的影响
Fig.4 Effects of knockout bypass genes on strain growth, 5-ALA, and co-culture systems
其中,敲除解淀粉芽孢杆菌的nas基因使得5-ALA产量提升最明显,达到424.26 mg/L。接着,为了进一步改善解淀粉芽孢杆菌合成5-ALA的能力,在B.amy-2菌株中同时敲除上述4个旁路基因获得了B.amy-7工程菌株,该菌株摇瓶发酵5-ALA的产量达到524.26 mg/L,较B.amy-2菌株(298.72 mg/L)提高了75.50%(图4-e)。这些结果进一步说明阻断5-ALA和血红素生物合成的竞争代谢途径对改善前体5-ALA的合成至关重要。最后,将工程菌株B.amy-7与E.coli-a菌株进行血红素的共培养发酵,发现共培养体系中血红素的产量达到18.16 mg/L,相比于阻断副产物合成途径之前提高了131.62%。此外,工程菌株B.amy-7与E.coli-a的发酵体系中5-ALA的积累量为185.72 mg/L,较B.amy-7单独发酵下降了64.57%(图4-f)。
为加速共培养体系中解淀粉芽孢杆菌的胞内5-ALA向胞外转运进入大肠杆菌的效率,避免5-ALA胞内积累对5-ALA生物合成途径的反馈抑制,在工程菌株B.amy-7中表达rhtA基因来构建5-ALA胞外转运通路,获得工程菌株B.amy-8。通过对工程菌株B.amy-8和E.coli-a进行血红素共培养发酵,发现在强化5-ALA胞外转运后,共培养体系中5-ALA的积累量较B.amy-7与E.coli-a的共培养体系有所降低,并且共培养体系中血红素含量提升了16.91%,达到21.23 mg/L(图5)。
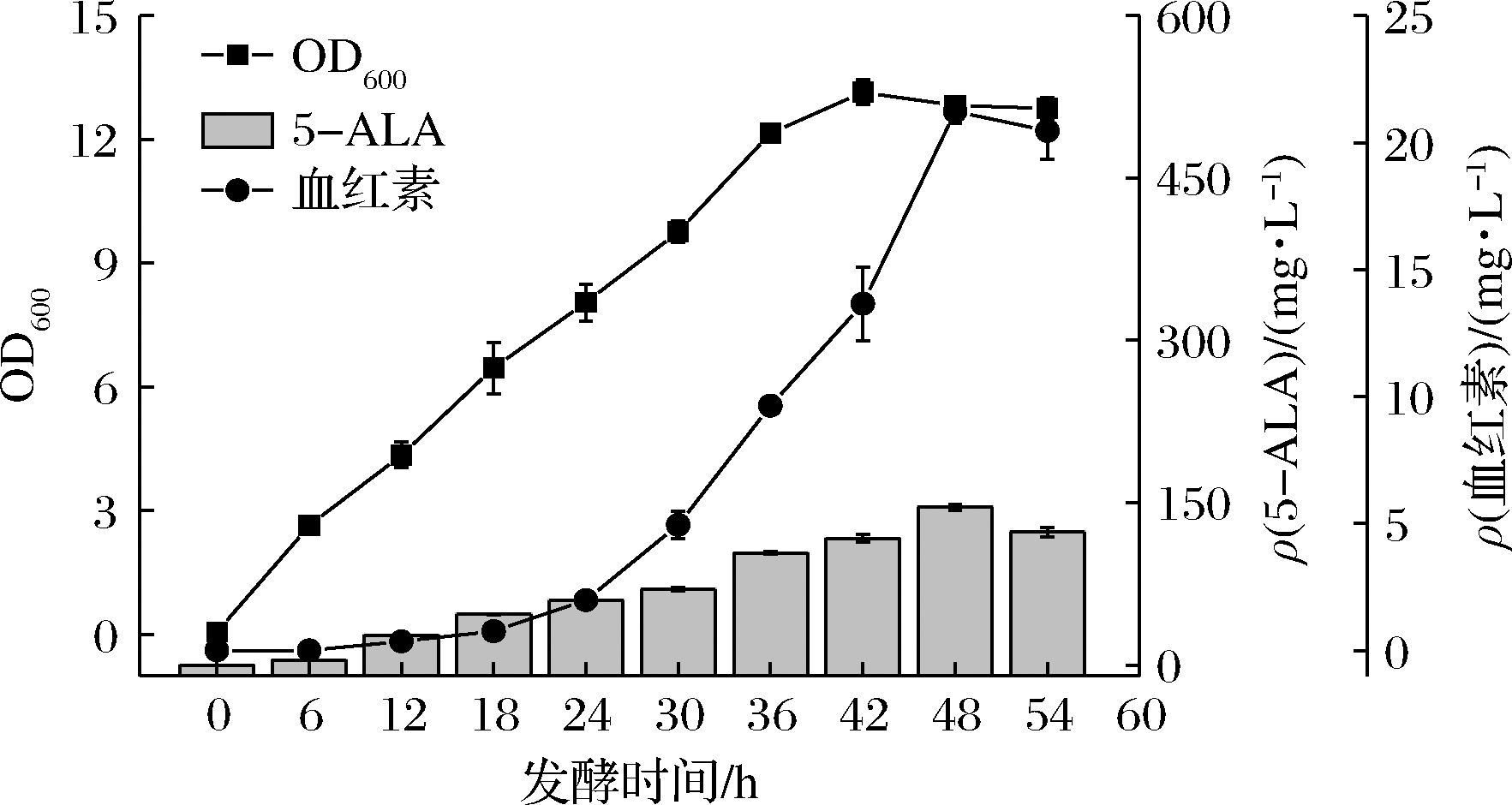
图5 构建5-ALA胞外转运蛋白对共培养体系的影响
Fig.5 Effects of constructing 5-ALA extracellular transporter on co-culture system
2.3 转运途径设计强化血红素的合成
血红素的胞内合成受到严格调控,为减少血红素胞内积累对其生物合成途径关键酶的反馈抑制,在E.coli-a菌株中过表达编码血红素胞外转运蛋白的ccmABC基因构建血红素的胞外转运通路,获得E.coli-b工程菌株。工程菌株B.amy-8与E.coli-b的共培养发酵结果表明构建血红素胞外转运通路使得血红素的积累量提高了34.02%,达到28.36 mg/L,其中胞外血红素浓度达到14.94 mg/L(图6)。
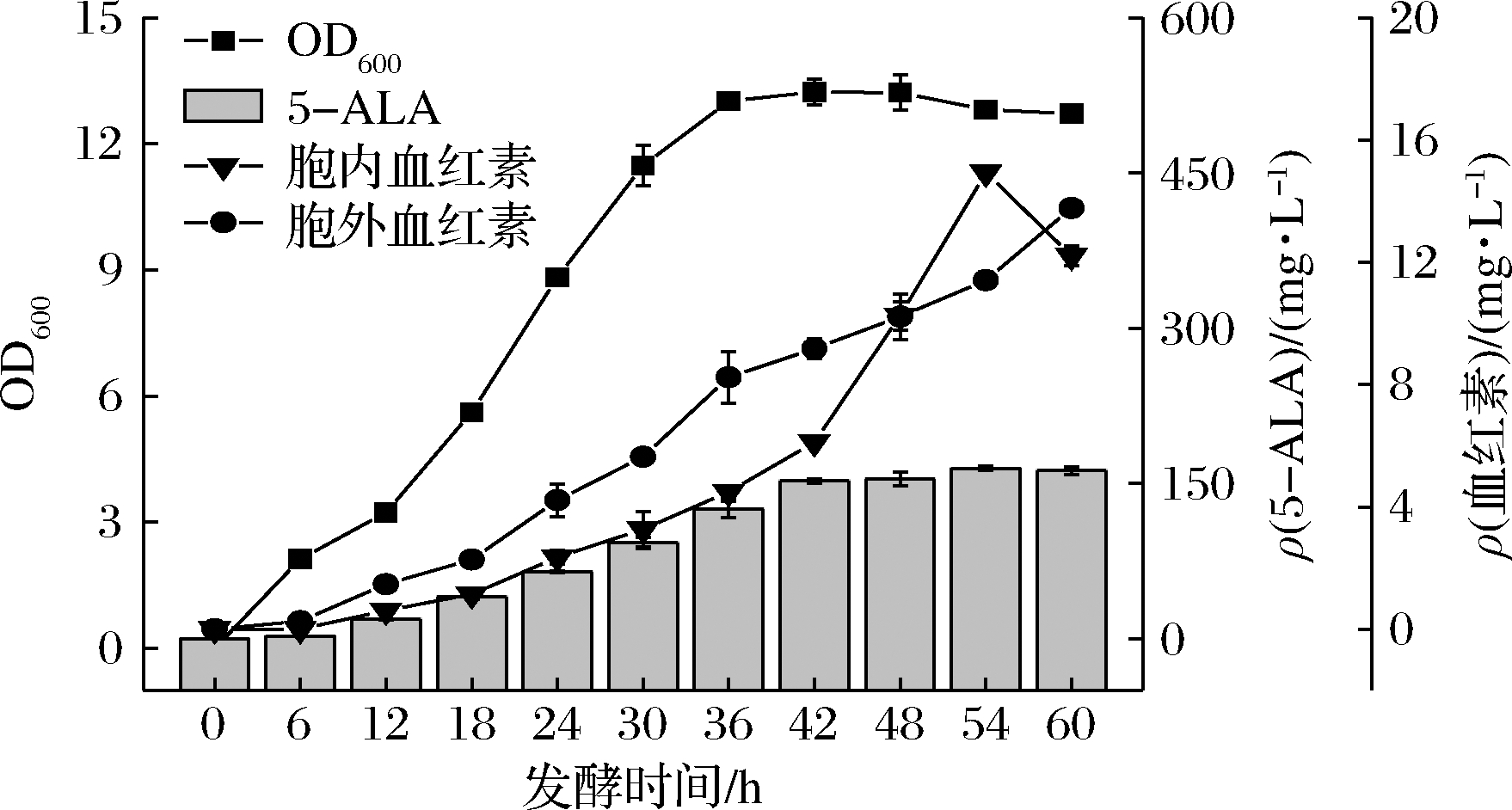
图6 表达血红素胞外转运蛋白对 5-ALA以及胞内、胞外血红素浓度的影响
Fig.6 Effects of expression of heme extracellular transporter on 5-ALA and intracellular and extracellular heme concentration
通过对共培养体系进行多种代谢工程改造后,解淀粉芽孢杆菌与大肠杆菌共培养体系合成血红素的能力得到了不断提升,相对于优化前的共培养体系,血红素的产量提升了2.62倍(表3)。
表3 本研究所获工程菌株和共培养体系合成5-ALA与血红素的产量比较
Table 3 Titer of 5-ALA and heme in the engineering strains and the co-cultivation system obtained in this study

菌株 策略5-ALA/(mg·L-1)血红素/(mg·L-1)B.amy-1gltXE.coli、hemAE.coli and hemAE.coli were expressed in B.amyloliquefaciens NX-2S154ΔpgsBCA136.81-E.coli-ahemB、hemC、hemD、hemE、hemF、hemG and hemH were overexpressed in E.coli BL21(DE3)--B.amy-1+E.coli-aCo-culture fermentation76.627.84B.amy-2gltXB.Amy、hemAB.Amyand hemLE.coli were expressed in B.amyloliquefaciens NX-2S154ΔpgsBCA298.72-B.amy-3ldh was knocked out in B.amy-2395.72-B.amy-4pta was knocked out in B.amy-2372.54-B.amy-5nas was knocked out in B.amy-2424.26-B.amy-6prob gene was knocked out in B.amy-2296.68-B.amy-7ldh、pta、nas and prob were knocked out in B.amy-2524.26-
续表3

菌株 策略5-ALA/(mg·L-1)血红素/(mg·L-1)B.amy-7+E.coli-aCo-culture fermentation185.7218.16B.amy-8rhtA was expressed in B.amy-7--B.amy-8+E.coli-aCo-culture fermentation145.7221.23E.coli-bccmABC was overexpressed in E.coli-a--B.amy-8+E.coli-bCo-culture fermentation164.4528.36
2.4 7.5 L发酵罐分批及分批补料发酵
为了提升上述共培养体系合成血红素的能力,将B.amy-8与E.coli-b的共培养体系在7.5 L发酵罐进行血红素的分批发酵,其5-ALA和血红素产量分别达到168.48 mg/L与36.31 mg/L,与摇瓶发酵相比分别提升了2.45%和28.03%(图7-a)。

a-分批发酵;b-分批补料发酵
图7 共培养体系发酵参数
Fig.7 Parameters of co-culture systems
此外,有文献报道在发酵初期过高的葡萄糖浓度会导致乙酸等副产物的积累从而影响细胞的生长[20]。为了提高发酵过程中菌体密度以及目标产物的合成,分批补料发酵策略被采用。首先将初始葡萄糖质量浓度设为20 g/L,并在发酵12、24和36 h分别流加10 g/L的葡萄糖。由图7-b可知,在补料发酵过程中,共培养体系的菌株生长迅速进入对数生长期,并且在48 h菌株的OD600达到最高。血红素的积累量达到65.38 mg/L,相对于分批发酵提升1.8倍。
3 讨论与结论
本研究以B. amyloliquefaciens NX-2S154和E. coli BL21为出发菌株构建血红素合成的共培养体系,获得了7.84 mg/L的血红素积累。通过对解淀粉芽孢杆菌中合成5-ALA的关键途径基因进行适配性优化,并敲除多个副产物合成途径关键基因实现了碳代谢流高效流向血红素,使得血红素的产量达到18.16 mg/L,相比于优化前提高了131.62%。此外,通过载体工程策略构建5-ALA与血红素的胞外转运通路,获得了28.36 mg/L的血红素。最后,在7.5 L发酵罐进行血红素的分批与分批补料发酵,使得血红素的产量达到65.38 mg/L。
本研究虽然首次采用共培养体系实现血红素的生物合成,但目前血红素的产量及其转化率仍然相对较低。因此,后续可能需要进一步开展以下研究工作来进一步改善血红素合成效率。首先,开发基因表达的动态调控系统来理性调节关键基因的表达水平,提高代谢流流向血红素生物合成的效率[21]。其次,血红素的生物合成受到严格调控,需要研究血红素对关键酶的反馈抑制机制,并通过蛋白质工程手段缓解反馈抑制作用。另外,需要探索血红素共培养体系中解淀粉芽孢杆菌与大肠杆菌之间的信息交流以及物质交换机制,为构建上述菌株共培养体系高效合成血红素及其他产物提供新的研究方向[22]。
[1] BRYANT D A, HUNTER C N, WARREN M J.Biosynthesis of the modified tetrapyrroles:The pigments of life[J].Journal of Biological Chemistry, 2020, 295(20):6 888-6 925.
[2] PERNER J, HATALOVA T, CABELLO D M, et al.Haem-responsive gene transporter enables mobilization of host haem in ticks[J].Open Biology, 2021, 11(9):210048.
[3] ZAMARRE O BEAS J, VIDEIRA M A M, SARAIVA L M.Regulation of bacterial haem biosynthesis[J].Coordination Chemistry Reviews, 2022, 452:214286.
O BEAS J, VIDEIRA M A M, SARAIVA L M.Regulation of bacterial haem biosynthesis[J].Coordination Chemistry Reviews, 2022, 452:214286.
[4] 潘斐, 严一凡, 朱逸凡, 等.四吡咯化合物生物合成研究进展[J].生物工程学报, 2022, 38(4):1 307-1 321.
PAN F, YAN Y F, ZHU Y F, et al.Advances in the biosynthesis of tetrapyrrole compounds[J].Chinese Journal of Biotechnology, 2022, 38(4):1 307-1 321.
[5] QIU Y B, ZHU Y F, SHA Y Y, et al.Development of a robust Bacillus amyloliquefaciens cell factory for efficient poly(γ-glutamic acid) production from jerusalem artichoke[J].ACS Sustainable Chemistry & Engineering, 2020, 8(26):9 763-9 774.
[6] LUO Z S, LIU S, DU G C, et al.Enhanced pyruvate production in Candida glabrata by carrier engineering[J].Biotechnology and Bioengineering, 2018, 115(2):473-482.
[7] 曾娇娇, 余世琴, 周景文.代谢工程改造大肠杆菌增产酪醇[J].食品与发酵工业, 2021, 47(22):8-15.
ZENG J J, YU S Q, ZHOU J W.Metabolic engineering of Escherichia coli for improving tyrosol production[J].Food and Fermentation Industries, 2021, 47(22):8-15.
[8] KWON S J, DE BOER A L, PETRI R, et al.High-level production of porphyrins in metabolically engineered Escherichia coli:Systematic extension of a pathway assembled from overexpressed genes involved in heme biosynthesis[J].Applied and Environmental Microbiology, 2003, 69(8):4 875-4 883.
[9] ZHAO X R, CHOI K R, LEE S Y.Metabolic engineering of Escherichia coli for secretory production of free haem[J].Nature Catalysis, 2018, 1(9):720-728.
[10] LI Z H, WANG X N, ZHANG H R.Balancing the non-linear rosmarinic acid biosynthetic pathway by modular co-culture engineering[J].Metabolic Engineering, 2019, 54:1-11.
[11] 张恕铭, 曾林, 孙向阳, 等.屎肠球菌与植物乳杆菌共培养产γ-氨基丁酸条件优化及关键酶活性研究[J].食品与发酵工业, 2021, 47(9):154-159.
ZHANG S M, ZENG L, SUN X Y, et al.Optimization of γ-aminobutyric acid produced by co-culturing Enterococcus faecium and Lactobacillus plantarum and the activities of key enzyme[J].Food and Fermentation Industries, 2021, 47(9):154-159.
[12] ZHOU K, QIAO K J, EDGAR S, et al.Distributing a metabolic pathway among a microbial consortium enhances production of natural products[J].Nature Biotechnology, 2015, 33(4):377-383.
[13] SHA Y Y, HUANG Y Y, ZHU Y F, et al.Efficient biosynthesis of low-molecular-weight poly-γ-glutamic acid based on stereochemistry regulation in Bacillus amyloliquefaciens[J].ACS Synthetic Biology, 2020, 9(6):1 395-1 405.
[14] SHA Y, ZHANG Y, QIU Y, et al.Efficient biosynthesis of low-molecular-weight poly-γ-glutamic acid by stable overexpression of PgdS hydrolase in Bacillus amyloliquefaciens NB[J].Journal of Agricultural and Food Chemistry, 2019, 67(1):282-290.
[15] QIU Y B, ZHU Y F, ZHANG Y T, et al.Characterization of a regulator pgsR on endogenous plasmid p2Sip and its complementation for poly(γ-glutamic acid) accumulation in Bacillus amyloliquefaciens[J].Journal of Agricultural and Food Chemistry, 2019, 67(13):3 711-3 722.
[16] QIU Y B, ZHANG Y T, ZHU Y F, et al.Improving poly-(γ-glutamic acid) production from a glutamic acid-independent strain from inulin substrate by consolidated bioprocessing[J].Bioprocess and Biosystems Engineering, 2019, 42(10):1 711-1 720.
[17] TAN S I, YOU S C, SHIH I T, et al.Quantification, regulation and production of 5-aminolevulinic acid by green fluorescent protein in recombinant Escherichia coli[J].Journal of Bioscience and Bioengineering, 2020, 129(4):387-394.
[18] MISCEVIC D, MAO J Y, KEFALE T, et al.Strain engineering for high-level 5-aminolevulinic acid production in Escherichia coli[J].Biotechnology and Bioengineering, 2021, 118(1):30-42.
[19] NOH M H, LIM H G, PARK S, et al.Precise flux redistribution to glyoxylate cycle for 5-aminolevulinic acid production in Escherichia coli[J].Metabolic Engineering, 2017, 43:1-8.
[20] DONG Y S, ZHANG H, WANG X Y, et al.Enhancing ectoine production by recombinant Escherichia coli through step-wise fermentation optimization strategy based on kinetic analysis[J].Bioprocess and Biosystems Engineering, 2021, 44(7):1 557-1 566.
[21] CASTRO C C, NOBRE C, DE WEIRELD G, et al.Microbial co-culturing strategies for fructo-oligosaccharide production[J].New Biotechnology, 2019, 51:1-7.
[22] GOERS L, FREEMONT P, POLIZZI K M.Co-culture systems and technologies:Taking synthetic biology to the next level[J].Journal of the Royal Society, Interface, 2014, 11(96):20140065.