蛋白质水解物包括多肽、寡肽和氨基酸,是食品、发酵、化工、医药、饮料、饲料等行业的重要原料。蛋白质的水解程度与水解方式,决定了蛋白水解物的化学组成、生理活性和物化特性等。相比传统酸碱水解制备方法,蛋白水解酶介导的蛋白质酶法水解,具有条件温和、易于控制、产品质量高等优势[1]。但蛋白水解酶具有水解位点选择性和底物分子大小偏好性,单一种类蛋白水解酶对特定底物的水解作用往往具有限制性[2-3]。
MEINLSCHMIDT等[4]以多种商品蛋白酶水解大豆分离蛋白,发现其中碱性蛋白酶(alcalase)水解度最高,可达13%,此时水解产物中仍有相当比例的10~25 kDa大小组分。SCHLEGEL等[5]以9种蛋白酶水解羽扇豆分离蛋白,也发现使用碱性蛋白酶水解度最高,可达9.05%,但水解产物中仍有相当比例的10~20 kDa大小组分。GRUPPI等[6]用2种商品酶Debitrase HYW20TM和ProlyveTM分别水解乳清蛋白、酪蛋白和乳蛋白,水解前后分子质量在10 kDa以上的成分比例由70%~95%降至2%~25%,分子质量在1~10 kDa的成分比例由4%~35%升至35%~55%。ALAHMAD等[7]使用无花果蛋白酶水解鳙鱼蛋白,水解度分别为13.36%、17.09%和20.15%的条件下,分子质量在1~10 kDa的成分比例由水解前的2.67%分别增至8.55%、7.69%和6.86%。ZHANG等[8]用碱性蛋白酶(alcalase)水解紫苏粕蛋白,蛋白电泳结果显示反应体系中主要成分由反应前的52 kDa和32 kDa两种为主转变为水解后的10 kDa以下为主,另有少量37 kDa和30 kDa成分。另外,胃蛋白酶(pepsin)、胰蛋白酶(trypsin)、糜蛋白酶(chymotrypsin)、嗜热菌蛋白酶(thermolysin)、枯草杆菌蛋白酶(subtilisin)、酸性蛋白酶(acidic protease)和蛋白酶K(proteinase K)等均有被应用于寡肽制备的报道[9],从另一方面说明了蛋白水解酶对特定底物水解作用的局限性。
单一蛋白水解酶对底物水解作用的局限性表现为生成特定比例的寡肽产物,此时使用寡肽水解酶进行辅助酶解,理论上可以调节酶解的水解度,同时改变产物化学组成、生理活性和物化特性。因此,寡肽水解酶的获得对于蛋白水解与蛋白质加工具有重要意义和关键价值。为此,本文对来源于黑曲霉的一种未知功能的蛋白水解酶进行分子克隆、表达与功能鉴定,进一步就其蛋白水解偏好性进行分析,为其应用于蛋白质加工和寡肽制备等奠定基础。
1 材料与方法
1.1 菌种与质粒
黑曲霉(Aspergillus niger)CICIM F0215,为本实验室前期分离、鉴定并保藏。大肠杆菌(Escherichia coli)JM109和毕赤酵母(Pichia pastoris)GS115,为本实验室保藏,分别用于本研究的重组质粒构建和酶表达宿主。质粒pPIC9k(Invitrogen),用于本研究的酶编码基因克隆与表达的载体。黑曲霉和大肠杆菌分别采用马铃薯葡萄糖(potato dextrose agar,PDA)培养基和LB(Luria-Bertani)培养基进行培养[10];毕赤酵母及其重组菌的培养基及培养方法等参照毕赤酵母操作手册(Invitrogen)进行。
1.2 主要试剂
限制性内切酶和LA Taq DNA多聚酶,大连宝生物工程有限公司;T4 DNA连接酶、质粒小量提取试剂盒、TRIzol总RNA提取试剂、cDNA合成试剂盒和小量DNA产物纯化回收试剂盒,Thermo Fisher公司;大豆分离蛋白,上海源叶;小麦蛋白、花生分离蛋白、豌豆分离蛋白和芝麻蛋白,江苏锐阳;BODIPY FL Ester,Invitrogen;苯甲基磺酰氟(phenylmethylsulfonyl fluoride,PMSF)、抑肽素(pepstatin)、E-64和EDTA-2 Na,Sigma公司。其他生化试剂为分析纯,国药化学试剂有限公司。
1.3 基因克隆和表达
黑曲霉总RNA提取及cDNA制备,按照试剂盒使用说明进行。基因扩增、连接、转化与鉴定等,按照常规实验方法进行[10]。基因扩增所用引物由生工生物工程(上海)股份有限公司合成,上游引物序列为:5′-GTACTGTCTCTCCATAAACGCGACGG-3′,下游引物为:5′-TGCTCTAGATTACAGAGCGAGAAGTAGGCCGATACC-3′(下划线部分为限制性酶切位点)。
重组酶的发酵制备,按照毕赤酵母操作手册(Invitrogen)所述方法进行,以甲醇诱导发酵并监测发酵液酶活力变化,至酶活力不再上升时终止发酵。离心收集发酵液上清液,制得粗酶液,再经盐析、脱盐、凝胶过滤色谱等进行分离纯化。酶蛋白纯度分析采用SDS-PAGE法,使用5%的浓缩胶和12%的分离胶,蛋白质浓度测定采用BCA(bicinchoninic acid)法[10]。
1.4 酶活力测定与酶学特征分析
1.4.1 蛋白水解酶酶活力检测方法
蛋白水解酶酶活力检测,依照本研究前期建立的均相检测法进行,使用BODIPY NHS Ester标记的大豆分离蛋白作为底物[11]。基本步骤为:荧光底物(10 μg/mL)与适度稀释的酶液分别预热至40 ℃后各取100 μL加入微孔板,40 ℃下反应10 min,用酶标仪在激发波长500 nm、发射波长530 nm条件下检测荧光。以底物和酶液作为对照。所测酶活力用相对荧光强度(relative fluorescence intensity,RFI)表示,计算如公式(1)所示:
RFI=FC-(FA+FB)
(1)
式中:FC,样品荧光值;FA,底物荧光值;FB,酶液荧光值。
1.4.2 温度和pH对酶活力的影响
温度和pH对酶活力的影响,按照实验室常规方法进行[12]。将酶液与底物分别置于25~60 ℃下或pH 3.0~10.0下进行酶活力测定。以酶活力最高者为100%,计算相对酶活力,确定酶的最适作用温度和最适作用pH。
1.4.3 抑制剂对酶的特异性抑制
按1/10或1/100的添加比例,将抑制剂溶液(10 mmol/L PMSF乙醇溶液、100 μmol/L抑肽素二甲亚砜溶液、100 μmol/L E-64去离子水溶液或10 mmol/L EDTA-2 Na去离子水溶液)与适度稀释的酶液混合,其中PMSF、抑肽素、E-64和蛋白水解酶在4 ℃孵育20 min后检测残余酶活力,EDTA-2 Na在4 ℃孵育6 h后检测残余酶活力。以不添加抑制剂的酶活力为Uc(对照),添加抑制剂后检测的酶活力为Us,抑制剂对蛋白酶的抑制率(I)计算如公式(2)所示:
(2)
1.5 酶底物偏好性分析
采用酶法分别对小麦蛋白、花生分离蛋白、豌豆分离蛋白和芝麻蛋白进行水解,制备获得不同水解度的水解物[13]。以此为底物,按照上述酶活力测定分析重组酶对其水解作用,底物质量浓度为10 g/L,反应条件为40 ℃和pH 7.0。
1.6 生物信息学分析方法
黑曲霉基因组信息,参照A.niger CBS 513.88基因组序列(美国国立生物技术信息中心);序列比对分析使用在线分析程序BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行;信号肽分析使用在线分析程序SignalP 6.0(https://services.healthtech.dtu.dk/service.php?SignalP)进行分析;氨基酸序列比对分析使用Clustal Omega(https://www.ebi.ac.uk/Tools/msa/clustalo/)和BioEdit Sequence Alignment Editor软件进行分析,并使用MEGA 11软件以Neighbour-joining法构建遗传发育树。
2 结果与讨论
2.1 黑曲霉蛋白水解酶系中存在一个未知功能的全新蛋白水解酶
黑曲霉具有极为丰富的蛋白水解酶体系,以A.niger CBS 513.88基因组序列为参照序列,进行了黑曲霉蛋白水解酶系的分析,发现其中的XM_001396314开放读框为一疑似天冬氨酸蛋白酶,将此读框的编码基因命名为ypsA,编码产物命名为YpsA。此读框由492个氨基酸残基组成,其N-端含有一个由18个氨基酸残基组成的典型Sec信号肽。其一级结构与氨基酸序列相似性分析结果显示,YpsA与MEROPS数据库中不同来源的天冬氨酸蛋白酶具有较高的一级结构相似性和一定的氨基酸序列相似性(图1、图2),并且与来自于A1A家族的几种酶相对聚集,遗传距离相对接近,与Q12303存在最高相似性(33.05%);而涉及不同家族来源的天冬氨酸蛋白酶的自展值大多较低(<50%),这与其氨基酸序列相似性较低(<22%)吻合。故此,初步分析结果揭示YpsA为A1A家族的新成员。

图1 YpsA在天冬氨酸蛋白水解酶家族中的遗传距离
Fig.1 Genetic distances of YpsA in the aspartic peptidase family
注:线段的长度表示用MEGA 11计算的遗传距离;分支节点上的数字代表的是bootstrap百分比,小于50%的bootstrap值未显示;除YpsA外,各蛋白水解酶以其在UniProt数据库(https://www.uniprot.org/)中的登录号表示,括号中标注为各蛋白水解酶在MEROPS数据 库(https://www.ebi.ac.uk/merops/)中的家族分类号

图2 天冬氨酸蛋白水解酶不同家族成员的氨基酸序列比对与结构分析
Fig.2 Multiple sequence alignment and primary structure analysis of different aspartic peptidases
注:“*”指示保守的氨基酸序列;“:”指示保守的替换;“.”指示半保守的替换;序列中的“-”代表空格;信号肽以阴影进行标注;活性位点以实线方框标出;N-端保守的色氨酸和酪氨酸残基用虚线方框标出
进一步分析YpsA一级结构组成序列特征(图2),发现已报道的5种A1A家族成员均具有该家族典型的4个基序,分别是组成第1个ψ环的Asp-Thr-Gly与Gly-H-H-Gly(H为疏水性氨基酸),以及组成第2个ψ环的Asp-Thr-Gly与H-H-Gly-Asp/Gln/Asn[14-15]。YpsA在序列对应位置也具有全部4个基序,其中第2和第4个基序分别为Gly-Ile-Ala-Gly和Leu-Leu-Gly-Asp。天冬氨酸蛋白酶在多肽链N-端具有高度保守的酪氨酸残基位点和相对保守的色氨酸残基位点[14],通过比对可知,这2个位点在YpsA中分别为Trp86和Tyr218。除此之外,进行比对的6种天冬氨酸蛋白酶的氨基酸序列,还在其他多个位点具有保守的、保守替换的或是半保守替换的氨基酸残基,显示其具有较高的一级结构相似性,可初步确认YpsA的催化活性中心组成为Asp79和Asp274[15]。
2.2 ypsA基因克隆、异源表达及生化特征
基于以上分析,黑曲霉YpsA极有可能是一未知功能的天冬氨酸蛋白酶。为此,进一步通过cDNA克隆技术对其进行克隆与表达。通过提取A.niger CICIM F0215的总RNA并反转录制备cDNA,以此cDNA为模板设计核苷酸引物,对其成熟肽编码序列进行扩增并克隆入表达载体pPIC9K中,获得了重组质粒pPIC-ypsA。经核苷酸序列测定与分析后发现,所克隆的ypsA核苷酸序列与A.niger CBS 513.88中相应序列完全一致。
进一步将线性化后的重组质粒pPIC-ypsA电击转化入毕赤酵母GS115,并通过G418抗性平板筛选获得转化子Pp-YpsA。按照毕赤酵母操作手册中的方法进行摇瓶发酵与酶液制备,经96 h发酵后,发酵液中重组酶的酶活力达到1 034 386 RFU/mL,进一步纯化获得重组YpsA用于后续酶学性质等分析。
在不同温度和不同pH下,分析了重组YpsA的最适作用pH和温度,结果如图3所示。YpsA在40 ℃和pH 7.0的条件下表现出最高酶活力,并且YpsA能够在相对较宽的温度(30~50 ℃)和pH(pH 4.5~9.5)下呈现高活性(相对酶活力>50%)。YpsA的基础酶学性质与来自酿酒酵母的天冬氨酸蛋白酶Yapsin1和YAP3有明显差异,后者的最适作用pH分别为4.5~5.5和4.0~4.5,并且较窄pH范围内(分别为pH 4.3~6.0和pH 3.8~5.3)保持50%以上的相对酶活力[16-17]。YpsA与白色念珠菌(Candida albicans)中10种天冬氨酸蛋白酶的酶学性质也存在较大差异,后者的多数天冬氨酸蛋白酶最适作用pH介于3.0~5.0,仅其中的Sap7最适作用pH为6.5,且在pH 4.8~7.3保持50%以上的相对酶活力[18]。此外,现有研究多认为天冬氨酸蛋白酶是酸性蛋白酶的代表,而本研究鉴定获得的来自于黑曲霉的天冬氨酸蛋白酶YpsA,具有中性最适作用pH并具有较为宽泛的高活性pH范围。可见,中性天冬氨酸蛋白酶在自然界中是存在的,并且可能发挥重要的生理作用。因此,随着研究的深入,天冬氨酸蛋白酶作为酸性蛋白酶代表的认知可以获得进一步完善。此外,中性天冬氨酸蛋白酶这一特征,也有可能使其更便于在特定蛋白质生物加工过程的应用。

a-温度对酶活力的影响;b-pH对酶活力的影响
图3 温度和pH对YpsA活性的影响
Fig.3 Effects of temperature and pH on the activity of YpsA
按照现行蛋白水解酶的分类原则,蛋白水解酶可根据其活性中心位点类别进行区分,除天冬氨酸蛋白酶家族外,还包括丝氨酸蛋白水解酶、半胱氨酸蛋白水解酶、金属蛋白水解酶等家族。不同家族蛋白水解酶的催化活性可被不同种类的抑制剂特异性抑制,因而可使用抑制剂对蛋白水解酶进行家族分类[19]。为此,本研究进一步分析和检测了不同蛋白酶抑制剂对YpsA活性的抑制作用,结果显示,抑肽素对YpsA具有强烈抑制作用,而PMSF、E-64和EDTA对YpsA活性几乎没有抑制作用(图4)。已知抑肽素是一种紧密结合的可逆抑制剂,其结构类似催化反应过程中的四面体中间体,对蛋白水解酶A1和A2家族有特异性抑制作用[20]。依据本实验结果,并结合上述结构特征分析,可以确定黑曲霉来源的YpsA是一种天冬氨酸蛋白酶。
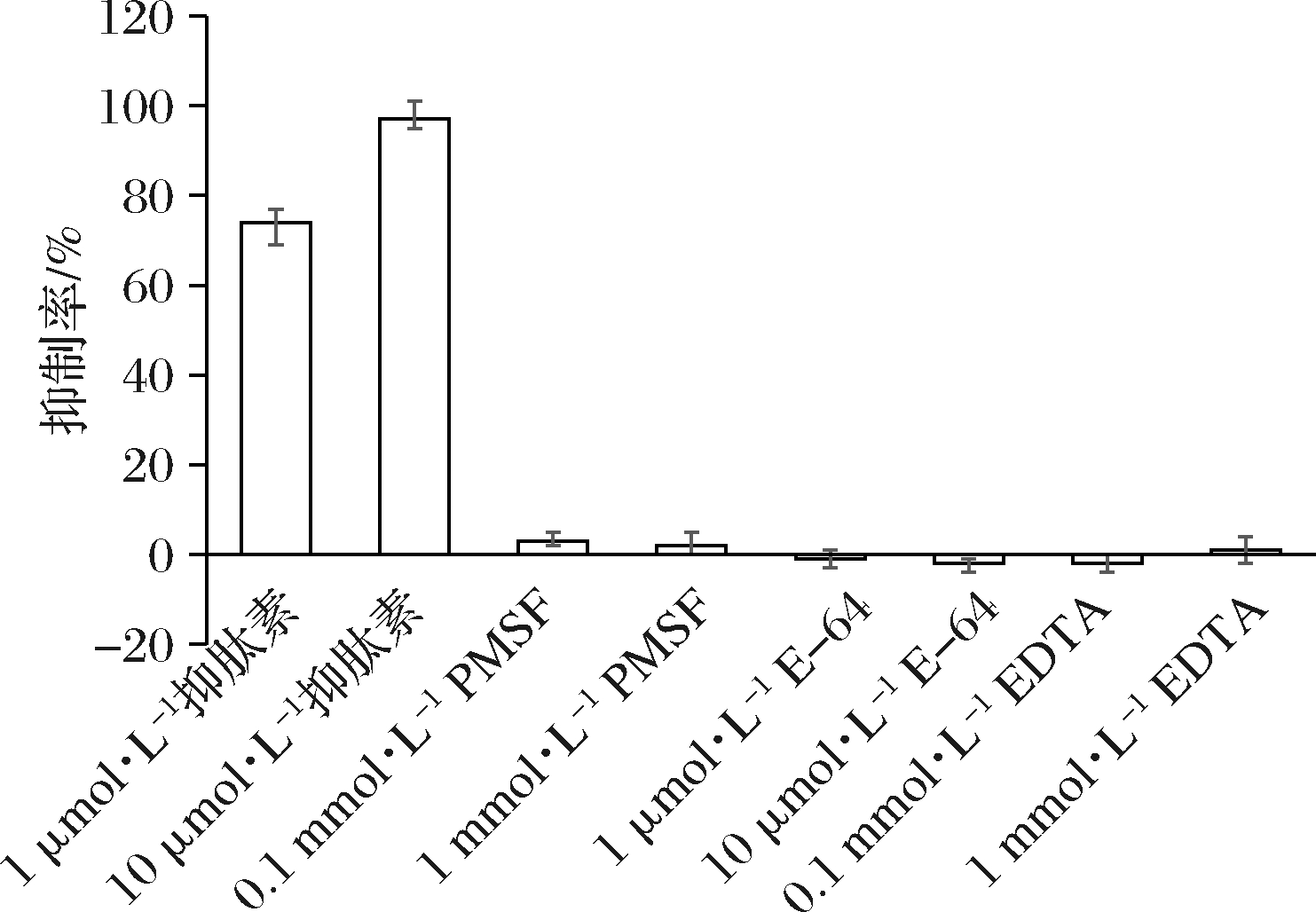
图4 蛋白酶抑制剂对YpsA活性的影响
Fig.4 Effects of different protease inhibitors on the activity of YpsA
2.3 重组酶YpsA水解不同底物特征
已有研究揭示,天冬氨酸蛋白酶具有酸性pH条件下的高活性和稳定性,其被应用于多个重要的工业领域,特别是在食品行业,如在乳制品行业的奶酪加工中用作牛奶凝固剂,在果汁或酿酒工业中用于澄清,在部分食品中用作增味剂等,而特定酶的适合应用场景取决于其催化特征[15]。在上述研究中发现,YpsA的最适作用pH为中性,有效作用pH宽度也与现有报道的大多数天冬氨酸蛋白酶有较大差异。因此,YpsA应该具有独特的蛋白水解特征。为此,分别使用5种植物来源的蛋白、多肽、寡肽作为底物,进一步对YpsA的水解特征进行了分析,结果汇总于图5。可以看出,YpsA对低分子质量的寡肽(0.45~1.45 kDa)具有相对高的活性,而对所测试的大豆蛋白、小麦蛋白、豌豆蛋白、花生蛋白和芝麻蛋白及其多肽制备物(1.45~12.5 kDa)的水解活性皆较低。可见,YpsA对由5~15个氨基酸残基组成的低分子质量寡肽具有水解偏好性,是一种新型天冬氨酸寡肽酶。

图5 YpsA作用于不同底物的活力特征
Fig.5 Relative activities of YpsA on different substrates
3 结论
通过分子克隆、功能鉴定与分析,确认了黑曲霉YpsA为一新型中性天冬氨酸寡肽酶,具有相对更宽泛的最适作用pH范围,在蛋白质生物加工和寡肽制备中可能具有特殊的应用价值。
[1] LI G M, LIU W Y, WANG Y Q, et al.Functions and applications of bioactive peptides from corn gluten meal[J].Advances in Food and Nutrition Research, 2019, 87:1-41.
[2] GURUMALLESH P, ALAGU K, RAMAKRISHNAN B, et al.A systematic reconsideration on proteases[J].International Journal of Biological Macromolecules, 2019, 128:254-267.
[3] BAHARIN A, TING T Y, GOH H H.Post-proline cleaving enzymes (PPCEs):Classification, structure, molecular properties, and applications[J].Plants (Basel, Switzerland), 2022, 11(10):1330.
[4] MEINLSCHMIDT P, SUSSMANN D, SCHWEIGGERT-WEISZ U, et al.Enzymatic treatment of soy protein isolates:Effects on the potential allergenicity, technofunctionality, and sensory properties[J].Food Science & Nutrition, 2015, 4(1):11-23.
[5] SCHLEGEL K, SONTHEIMER K, HICKISCH A, et al.Enzymatic hydrolysis of lupin protein isolates-changes in the molecular weight distribution, technofunctional characteristics, and sensory attributes[J].Food Science & Nutrition, 2019, 7(8):2 747-2 759.
[6] GRUPPI A, DERMIKI M, SPIGNO G, et al.Impact of enzymatic hydrolysis and heat inactivation on the physicochemical properties of milk protein hydrolysates[J].Foods (Basel, Switzerland), 2022, 11(4):516.
[7] ALAHMAD K, XIA W S, JIANG Q X, et al.Effect of the degree of hydrolysis on nutritional, functional, and morphological characteristics of protein hydrolysate produced from bighead carp (Hypophthalmichthys nobilis) using ficin enzyme[J].Foods (Basel, Switzerland), 2022, 11(9):1320.
[8] ZHANG H H, ZHANG Z J, HE D L, et al.Optimization of enzymatic hydrolysis of Perilla meal protein for hydrolysate with high hydrolysis degree and antioxidant activity[J].Molecules (Basel, Switzerland), 2022, 27(3):1079.
[9] L PEZ-GARC
PEZ-GARC A G, DUBLAN-GARC
A G, DUBLAN-GARC A O, ARIZMENDI-COTERO D, et al.Antioxidant and antimicrobial peptides derived from food proteins[J].Molecules (Basel, Switzerland), 2022, 27(4):1343.
A O, ARIZMENDI-COTERO D, et al.Antioxidant and antimicrobial peptides derived from food proteins[J].Molecules (Basel, Switzerland), 2022, 27(4):1343.
[10] 诸葛健, 王正祥.工业微生物实验技术手册[M].北京:中国轻工业出版社, 1994.
ZHU G J, WANG Z X.Industrial Microbiology:A Laboratory Manual[M].Beijing:China Light Industry Press, 1994.
[11] 宋鹏, 程磊, 田康明, 等.工业蛋白水解酶快速检测方法的建立与初步应用[J].食品与发酵工业, 2019, 45(17):216-220.
SONG P, CHENG L, TIAN K M, et al.A novel method for rapid determination of industrial protease and its preliminary application[J].Food and Fermentation Industries, 2019, 45(17):216-220.
[12] 蔡可, 王太康, 王君, 等.黑曲霉内切β-1, 4-半乳聚糖酶AghA的分子克隆与特征解析[J].食品与发酵工业, 2019, 45(1):29-35.
CAI K, WANG T K, WANG J, et al.Molecular cloning and biochemical characterization of endo-beta-1, 4-galactanase agh A from Aspergillus niger[J].Food and Fermentation Industries, 2019, 45(1):29-35.
[13] 宋鹏. 黑曲霉F0215蛋白水解酶系表征及功能解析[D].天津:天津科技大学, 2018.
SONG P.Characterization and functional analysis of proteolytic enzymes from Aspergillus niger F0215[D].Tianjin:Tianjin University of Science and Technology, 2018.
[14] REVUELTA M V, KAN J A L V, KAY J, et al.Extensive expansion of A1 family aspartic proteinases in fungi revealed by evolutionary analyses of 107 complete eukaryotic proteomes[J].Genome Biology and Evolution, 2014, 6(6):1 480-1 494.
[15] MANDUJANO-GONZ LEZ V, VILLA-TANACA L, ANDUCHO-REYES M A, et al.Secreted fungal aspartic proteases:A review[J].Revista Iberoamericana De Micología, 2016, 33(2):76-82.
LEZ V, VILLA-TANACA L, ANDUCHO-REYES M A, et al.Secreted fungal aspartic proteases:A review[J].Revista Iberoamericana De Micología, 2016, 33(2):76-82.
[16] OLSEN V, CAWLEY N X, BRANDT J, et al.Identification and characterization of Saccharomyces cerevisiae yapsin 3, a new member of the yapsin family of aspartic proteases encoded by the YPS3 gene[J].The Biochemical Journal, 1999, 339 (Pt 2):407-411.
[17] AZARYAN A V, WONG M, FRIEDMAN T C, et al.Purification and characterization of a paired basic residue-specific yeast aspartic protease encoded by the yap3 gene.Similarity to the mammalian pro-opiomelanocortin-converting enzyme[J].Journal of Biological Chemistry, 1993, 268(16):11 968-11 975.
[18] AOKI W, KITAHARA N, MIURA N, et al.Comprehensive characterization of secreted aspartic proteases encoded by a virulence gene family in Candida albicans[J].The Journal of Biochemistry, 2011, 150(4):431-438.
[19] RAWLINGS N D.Twenty-five years of nomenclature and classification of proteolytic enzymes[J].Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics, 2020, 1 868(2):140345.
[20] RAWLINGS N D, BARRETT A J, THOMAS P D, et al.The MEROPS database of proteolytic enzymes, their substrates and inhibitors in 2017 and a comparison with peptidases in the PANTHER database[J].Nucleic Acids Research, 2017, 46(D1):D624-D632.