食品组学是基于改善人类营养而开发的一种用于食品科学的综合性、高通量的新技术[1]。“食品组学”一词于2009年被首次提出,被定义为“食品组学是通过应用和整合先进组学技术来研究食品和营养领域,以提高消费者的福祉、健康和信心的科学”,它是基因组学(genomics)、转录组学(transcriptomics)、蛋白组学(proteomics)和代谢组学(metabolomics)等相关组学技术的组合[2-3]。因此,“组学”的根本含义是研究组成细胞的分子扮演的角色和发挥的功能,它们的发展离不开高通量技术的发展[4]。如图1所示,这些组学技术与食品成分、食品质量、食品认证、食品蛋白质和肽的活性、食品过敏原、毒素的鉴定、转基因食品以及人类基因组的破译等相关,并在基因水平上解释食品作用的领域,使人们能够更好地理解食品的功能和加工技术[6]。
食物中含有丰富的营养素,例如蛋白质、脂类、碳水化合物和矿物质、维生素、植物基物质、抗氧化剂等[7],这些营养素在人体内的功能不是单一的,它们经过消化道,一部分可被人体直接吸收,一部分与肠道内的微生物作用产生新的物质被人体利用,还有的会直接排除体外。在研究食品与人体营养、健康等的关系时,这些生物化学变化的多样性和复杂性造就了科学研究不可预测、主观性较强的特点。食品组学出现,可以在复杂反应中找到客观规律,解释和调控食品对机体的有利和有害影响,分析导致这些影响的因素,发掘关键反应通路。食品组学从分子角度研究复杂的体系,避免了传统研究方法使用较多有毒有害试剂、花费较长反应时间、产生大量废液废物和得到较少实验数据等问题,被公认是一种绿色技术[8]。食品组学目前常用于发酵食品研发、转基因食品研发、果蔬保鲜、营养与安全检测、疾病预防与诊断等方面的研究,还可以用于检测一些对人体健康和食品安全有重要影响的外源性化合物[9]。本文主要讨论了食品领域中常用的几种组学技术,分析了几种技术的各自优势,为后续组学技术在食品领域的深入应用提供研究思路。

图1 食品组学涵盖的内容[5]
Fig.1 Scope of foodomics[5]
1 基因组学在食品微生物组成上的应用
基因组学是对DNA结构和功能进行综合分析的一种技术,通过研究基因组的结构、功能、进化、定位和编辑等方式来获取不同食物中微生物的遗传基础[10]。受限于人们对未知微生物探索的局限性,基因组学只能研究微生物的多样性,而不能完全注释微生物的种别[11]。测序技术是基因组学的核心技术,自人类发现DNA以来,测序技术已经发展到了第三代。第一代测序也叫Sanger测序,是由英国生物化学家Frederick Sanger 及其同事于1977年提出的。该方法通常只能测得长度<1 000 bp的DNA核苷酸序列,它在某一固定的核苷酸位点开始,并在特定的碱基处终止,然后在每个碱基后面进行荧光标记,产生以A、T、C、G结束的4组不同长度的一系列核苷酸,并在尿素变性聚丙烯酰胺凝胶电泳上进行检测,从而获得DNA碱基序列的一种方法,其准确度较高,但效率较低[12]。第二代测序也叫下一代测序(next generation sequencing,NGS)或者高通量测序(high-throughput sequencing,HTS),它在DNA复制过程中通过捕捉新添加的碱基所携带的荧光标记,来实现边合成边测序的过程,从而确定DNA的序列,其具有通量高、读长短的特点[13]。NGS 在微生物学中的主要用途是用病原体的基因组定义代替通过形态学、染色特性和代谢标准对病原体进行的常规表征[14]。第三代测序以PacBio公司的SMRT和Oxford Nanopore Technologies纳米孔单分子测序技术为代表。第三代测序技术无需进行PCR扩增,可实现超长的测序长度[15]。
用于微生物多样性测序的方法有两种,一种是扩增子测序,一种是(宏)基因组测序。常见的扩增子测序方法有16S rDNA/rRNA测序[16](主要检测原核微生物物种组成、丰度和多样性)、18S rDNA/rRNA测序、ITS测序[17-18](主要检测真核微生物物种组成、丰度和多样性),而常见的(宏)基因组测序是利用鸟枪法(shotgun methods)将物种DNA打断成小片段后测序再拼接的方式进行[19-20]。扩增子测序由于引用的是细菌、真菌高度保守的基因片段为引物进行扩增测序,准确率可达到84%~95%,因此在微生物种类和丰度的检测上具有独特的优势,已被广泛应用于腌腊肉制品[19,21-22]、乳制品[23-24]、调味料[25-27]、泡菜[28-30]、酒类[31-35]等发酵过程微生物丰度和多样性(表1)。扩增子测序也可以用来检测食品的微生物安全性,例如WANG等[36]基于16S rRNA扩增子测序分析了预制食品(肉类、水产和菜肴)中的微生物,发现变形菌门和厚壁菌门是加工肉制品中的主要细菌门,且肉制品中含有更多的细菌属,其微生物的来源与环境和食物类型密切相关。WONG等[37]使用16S rRNA扩增子测序鉴定到了经微波处理后的咖喱鸡中存在病原微生物——肺炎克雷伯菌,为基因组学在食品安全上的应用提供了思路。
表1 利用扩增子测序分析发酵食品中的天然微生物示例
Table 1 Example of analyzing natural microbes in fermented foods by amplificon sequencing

研究对象鉴定的微生物类型实验方法参考文献中国传统腊肉盐弧菌属、葡萄球菌属、索丝菌属、弧菌属、变色杆菌、不动杆菌属、肉杆菌属、气单胞菌属、假单胞菌属、Cobetia属、巨球菌属、肠杆菌属等16S rRNA扩增子测序[19] 干腌火腿四烯球菌属、中度嗜盐菌属、发光菌属、不动杆菌属、葡萄球菌属、假单胞菌属等16S rRNA扩增子测序[21]酸鱼细菌:乳酸菌属、巨大球菌属、葡萄球菌属;真菌:酵母菌属、念珠菌属、威克汉姆酵母属16S rRNA扩增子测序和ITS扩增子测序[22]蒙古传统乳制品乳球菌属、链球菌属、乳杆菌属、肠球菌属、明串珠菌属16S rRNA扩增子测序[23]奶酪毕赤酵母属、哈萨克斯坦酵母属、半乳糖菌属、结合酵母属、圆孢菌属等18S rRNA扩增子测序[24]酱油魏氏杆菌属、芽孢杆菌属、乳球菌属16S rRNA扩增子测序[25]酱油枯草芽孢杆菌16S rDNA扩增子测序[26]辣椒酱乳杆菌属、魏斯氏菌属、中慢生根瘤菌属16S rRNA扩增子测序[27]泡菜乳酸杆菌属、片球菌属、不动杆菌属、明串珠菌属、魏氏菌属、链球菌属、巨单胞菌属、肠球菌属等16S rRNA扩增子测序和宏基因组测序[28]发酵芥末乳酸杆菌属16S rRNA扩增子测序[29]酸菜乳杆菌属、片球菌属16S rRNA扩增子测序[30]浓香型白酒甲烷八叠球菌属、甲烷杆菌属、水井坊氨基酸杆菌属、乳杆菌属16S rRNA扩增子测序[31]白酒发酵酵泥乙醇生孢产氢菌属、拟杆菌属、嗜蛋白军属、沉积杆菌属16S rRNA扩增子测序[32]酱香型白酒鞘氨醇杆菌属、肠杆菌属、不动杆菌属、魏氏杆菌属、芽孢杆菌属等16S rRNA扩增子测序[33]藏曲细菌:乳杆菌属、明串珠菌属;真菌:根霉菌属、曲霉属16S rDNA扩增子测序和ITS扩增子测序[34]茅台酒糖多胞菌属、芽孢杆菌属、麦氏菌属等16S rRNA扩增子测序[35]
由于扩增子测序扩增的是非全长的基因序列,因而无法注释到微生物的种水平,大多数微生物多样性研究停留在属水平,要进一步研究微生物对食物的影响还必须借助传统的微生物形态进行鉴定,使研究微生物的具体组成存在一定难度。自然界中大多数微生物是不可人工培养的,因此通过传统的微生物形态在种水平进行注释时,不可避免的会忽略掉一些重要信息。另外扩增子测序无法获知微生物具体的基因组成,在判断微生物的抗病性、发酵潜力、代谢能力等性能上具有一定的局限。
基因组学、宏基因组学测序方法很好的弥补了扩增子测序的缺点,不仅可以将微生物注释到种水平,并能对基因进行功能注释,准确分析相应基因参与的代谢通路,预测微生物的具体活动[38]。LI等[39]采用宏基因组学方法对中国南方18个即食食品中细菌群落和抗生素抗性基因的综合特征进行了表征,发现即食食品中最丰富的细菌门是变形菌门、厚壁菌门、蓝细菌门、拟杆菌门和放线菌门,并检测到属于18个抗生素抗性基因的 204个抗生素抗性基因亚型,其中氯霉素、大环内酯-林可酰胺-链霉素、多药耐药、氨基糖苷类、杆菌肽、四环素和β-内酰胺类耐药基因占主导地位。这些发现加深了对抗生素耐药组以及即食食品中抗生素耐药组、微生物群和移动遗传元件之间相关性的全面认识[39]。YANG等[40]采用宏基因组学将香肠中的细菌注释到了种水平,并通过相关性分析发现发酵葡萄球菌(如Staphylococcus carnosus, S.xylosus, S.equorum和S.saprophyticu)与乙酸戊酯、3-甲基丁烷、2-甲基丁烷等的产生密切相关,特别是S.carnosus和S.xylosus与苯醛、1-羟基-2-丁酮、乙醛、酮和酯水平呈正相关。这项结论为功能微生物发酵剂的开发奠定了基础[40]。食品是由众多生物细胞、生物代谢物、生化反应产物混合的大宗复合物,因此要准确预测食品中微生物的多样性和功能受到诸多限制,特别是食物基质中存在的原料如肉、植物的基因会影响宏基因组的测序准确度,影响微生物注释结果。
2 转录组学在食品加工与安全中的应用
转录组学是揭示能够修饰基因表达谱不同影响因素的组学[41]。转录组学的研究基础是生命的中心法则,通过转录调控物质(protein-coding mRNA和non-coding mRNA)构成生命体的多样性[42]。生物体的转录组具有生物活性,它可以随着内在和外在因素的变化而改变。要研究这些活性因子,常采用基因表达微阵列技术(即定量检测转录组)和RNA测序(RNA-seq)技术(即使用高通量测序来捕获所有转录序列)。
基因表达微阵列技术虽然是转录学相关技术中最经济的一种[43],但由于RNA-seq能够提供更完整的信息,非常适合鉴定微生物的全基因组序列,因此RNA-seq技术使用更为广泛。RNA-Seq是指对转录本cDNA的测序,其中丰度来源于每个转录本的计数,因而该技术深受高通量测序技术的制约[44]。基本的分析流程可以分为5个步骤:(1)通过高通量测序获得转录组的基本信息,包括总reads数、转录覆盖水平、质控等;(2)基因表达量的统计;(3)筛选差异表达基因,获得biomarkers,对有效信息进行可视化;(4)差异基因的功能注释及富集分析,描述基因扮演的角色;(5)基因结构与其他分析,例如KEGG信号通路显著性富集分析等。
在食品领域中,转录组学通常用于研究食物中与人体健康相关的代谢物的代谢机制以及食物中具体微生物的代谢情况,与指导食品生产、控制食品腐败、提高食品质量密切相关。虽然RNA-seq和基因表达微阵列技术在生物研究领域有广泛的应用,但是这种技术在食品科学中还处于起步阶段[45]。同基因组学的研究范围一样,转录组学在食品科学领域的应用多局限于食品中的微生物,重点关注食品品质的变化。YANG等[46]为了研究酿酒酵母在苹果酒发酵过程中抑制展青霉素的代谢机制,采用转录组学分析了酿酒酵母的代谢情况,发现酿酒酵母通过增加芳基醇脱氢酶、NADPH脱氢酶3和谷胱甘肽S-转移酶2等抗性酶的活性来应对棒曲霉素的胁迫,从而导致棒曲霉素的有效降解,达到抑菌的效果。QIAN等[47]研究则发现植物乳杆菌 SS-128 的生长特征和代谢是由 AI-2/LuxS QS 系统介导的,同时该系统是参与食品安全的正调节剂,为调控食品安全加工提供了理论基础。YE等[48]研究表明乳球菌因为培养条件的改变,其代谢途径会发生相应的变化,特别是pH值的影响最为显著。这项结论为乳品加工或使用乳球菌为发酵剂的食品的生产提供了理论支持[48]。
不难发现,食品科学领域中,采用转录组学解决的问题关乎食品加工、过程调控与食品安全,为科学解决食品安全问题提出了新的解决方案。也有部分研究直接关注食品原料、加工方式对食品品质的影响,但此类研究多注重引起变化的原因和结果的探究,较少论文从基因层面解释引起这种变化的机制。例如DOU等[49]采用转录组学研究黑骨鸡骨骼肌黑色素生成的分子机制,发现PDZK1基因具有参与色素沉着、黑素生成以及调节黑骨鸡肌肉中的磷脂信号传导过程的双重生理功能,但具体的生成及调节过程有待进一步挖掘。DU等[50]从加工方式方面研究了鲫鱼的肉质问题,研究发现短期微流水系统通过调控三羧酸循环、鸟氨酸循环、嘌呤代谢和氨基酸分解代谢来调节鲫鱼的肉质,但未探索参与代谢途径调节的基因位点。ONO等[51]基于转录组学研究确定了石榴果皮可水解单宁、花青素、类黄酮、萜类和脂肪酸生物合成或调节的候选基因,为提升石榴风味提供的参考,但未具体提出具体调控方。
转录组学在检测食品中有害成分和污染物上也具有独特的优势,例如二恶英、异种雌激素、霉菌素和有害的美拉德反应产物等[52]。通过分析细胞对不同毒素应激做出的反应,来区分细胞表达的各种特征化合物,实现对有害成分的动态检测。例如丙烯酰胺是美拉德反应过程中产生的一种热诱导污染物,即使在高浓度下,也只会引起培养细胞中基因表达的微小变化,利用常规传统的检验方法往往难以捕捉微量变化。然而,将MCF-7乳腺癌细胞暴露于丙烯酰胺的主要活性代谢产物甘氨酰胺后会产生特征性表达指纹,转录水平上表现为一系列保护抗氧化和酶的因子上调,表明美拉德褐变反应增加了加工食品的氧化活性[53]。转录组学中的微阵列技术可以用于转基因食品的分析,用于表征转基因食品的基因表达谱。例如通过谷氨酸-A1,谷氨酸-D1基因改造的小麦表现出更丰富的营养;通过OASA1D基因改造的大米表现出游离色氨酸的积累等,这些可能的表型变化都可以通过转录组学进行分析。由于转录组学可以从基因表达上分析食品的表型效果,可能观察到转基因导致的潜在基因突变等现象,对降低食品安全隐患意义深远[54]。
转录组学能从基因层面解释引起食品变化的原因,发掘潜在的食品安全隐患,调控加工食品质量。由于转录组学需要以具有活性的转录本为研究对象,这就意味着转录组学的研究对象是食品原料、转基因食品或者是参与食品生产、影响食品贮藏的微生物,与食品质量、安全和加工研究密切相关。但是为了充分阐明细胞活动过程中的变化和潜在机制,还需要蛋白表达水平和代谢物水平的联合分析。随着RNA-seq技术的成熟,其检测灵敏度也将会更高、分析范围将会更广,甚至可以研究剪接变体以及检测等位基因特异性差异(多态性),对食品研究来说也将是一种新的分析方式。
3 蛋白组学用于食品中蛋白质与食品品质的 关联分析
蛋白组学以一个生物体或细胞在某一生理状态中的蛋白质为研究对象,从整体水平观察细胞内蛋白质的组成及其变化规律,整体性、概括性地了解蛋白表达、修饰、相互作用等,从而揭示蛋白功能,为生命科学、自然科学的理论研究提供重要的参考[55]。蛋白质在生物体中扮演着重要的角色,蛋白质功能的形成是生物修饰、加工的结果,因此蛋白组学不仅包含对蛋白质本身的研究,还涉及到生物学方面的动态调控[56]。根据不同的检测方法和对象可以将蛋白组学分为非靶向蛋白组学、靶向蛋白组学、修饰蛋白组学、互作蛋白组学等。食品中多将非靶向和靶向蛋白组学用于研究食物生产中蛋白质的代谢情况,以提高食品安全、口感及营养程度。
蛋白组学在食品研究中应用较广,例如可用于食品新鲜度判别:BEGUM[57]首先使用2D-PAGE(two-dimensional polyacrylamide gel electrophoresis,2D-PAGE)方法获得了鸡蛋贮存过程中蛋白质的变化情况,然后利用蛋白组学鉴定了卵清蛋白中的蛋白质并获得了抗菌蛋白,从而成功识别和量化了蛋白降解过程中发生的变化,为识别鸡蛋新鲜度和质量的标志物提供有力途径;食品掺假检测:GALLARDO等[58]综述了蛋白质的氧化修饰如羰基化、硫醇氧化、芳香族羟基化以及美拉德反应可以反映食品加工中的变化,从而成为蛋白氧化的标志物,为鉴别食品提供提供了新的思路;乳品品质调控:剧柠等[55]指出蛋白组学可以对乳品中蛋白质的分子结构、表达模式、生物学功能进行更为详细的表征和注释,能为乳品的开发和利用奠定基础;食品过敏原识别:MONACI等[59]指出基于MS的蛋白组学可以检测完整的食物过敏原信息,追踪与过敏原相关的肽标记物,还能同时分析多种目标,在较高稳定性和可重复性的基础下,为复杂食品基质中的多种过敏原提供了痕量检测;其他与食品蛋白相关的作用机制的研究,例如将蛋白组学用于生鲜肉肉色变化的研究等[60]。
食品风味由于其特有的复杂性一直以来就是食品研究的难点,将蛋白组学用于食品风味形成的研究为食品的开发提供了新的见解。JI等[61]采用鸟枪法宏蛋白质组学在中国发酵鱼中鉴定出2 175种蛋白质,同时还得到了63种存在于链球菌属、芽孢杆菌属、埃希氏菌属中的氨基酸,这些微生物产生的代谢物可能是发酵鱼香气形成的原因。LIU等[62]利用定量蛋白组学分析了俄罗斯鲟鱼片在低温真空加热的异味来源是己醛、庚醛、壬醛和辛醛等物质。JIA等[63]利用无标记蛋白组学得到山羊肉中参与蛋白质氧化、半胱氨酸和蛋氨酸代谢的蛋白质会导致辐射异味形成。但是,由于贮存过程中的代谢过程,辐照异味会逐渐消散[63]。ZHAO等[64]采用TMT策略蛋白组学分析酱油生产过程中,低温高盐条件会刺激米曲霉产生更多独特的芳香风味,如酯类、吡嗪类、呋喃类等。ZHOU等[65]利用无标记MS/MS蛋白质组学发现了原调节蛋白(D0QCY6)和肌钙蛋白 (P63317) 与现代加工火腿味道密切相关。尤丽琴等[66]利用iTRAQ策略蛋白组学发现了宰后滩羊肉成熟过程中代谢酶的调控、结构蛋白的降解和应激蛋白的防护共同对风味前体物质的变化起调控作用。
鉴于蛋白组学的特殊定义,其多用于肉类食品蛋白组成、蛋白互作、蛋白代谢等方面的研究,而在植物蛋白方面的研究鲜有报道。植物蛋白基食品,如植物肉、植物奶和植物冰淇淋等由于其在营养、绿色和健康方面的优势备受消费者青睐[67],将蛋白组学用于植物蛋白基食品的研究具有重大意义。目前蛋白组学方法的研究主要集中在食源性成分的定性分析或是相对比例的定量分析,而在绝对定量分析中的研究应用较少,如何准确测定食品中的蛋白质含量,是当前食品行业的难点之一。
4 代谢组学分析食品组分的动态变化
代谢组学是研究生物产生的或受其刺激或干扰产生的小分子代谢产物(相对分子质量<1 000)以及代谢途径的科学。根据分析目的不同,可以分为4种类型:(1)代谢组学分析:对一定条件下某一生物样品或细胞所有内源性小分子代谢物组分的定性、定量分析;(2)代谢物靶标分析:运用多种技术来量化有限数量的代谢物;(3)代谢物轮廓分析:通过半定量方式检测某些类别化合物的代谢谱[68];(4)代谢物指纹分析:通过比较代谢指纹图谱的差异整体描述代谢物[69]。代谢组学根据分析方式的不同,还可以分为非靶向代谢物分析和靶向代谢物分析,在研究食品组分的动态变化中潜力巨大。
目前,代谢组学已广泛用于食品成分分析:JIANG等[70]基于非靶向代谢组学分析了不同荔枝的多酚代谢差异,共鉴定出8类126种多酚代谢物,揭示了不同荔枝中多酚的组成和差异。同时首次发现每个品种的常见差异代谢物15种,特异性差异代谢物20种。在此基础上推断黄酮类、黄酮醇类、羟基肉桂酰类和儿茶素类是荔枝果肉的主要多酚代谢产物[70];食品营养评估:代谢组学技术虽然还处于起步阶段,但却极大地的扩展了氨基酸营养的研究领域,它可以研究膳食氨基酸的概况和特征、消化、吸收和代谢、安全性和毒性等[71];食品加工效果验证:SPRAUL等[72]将核磁共振结合SGF ProfilingTM用于果汁鉴定、验证和质量控制,发现这种方法除了可以量化大量果汁的特征化合物之外,还可以使用统计模型来估计水果含量和来源,是食品质量控制的新方向[72];食品安全评估:代谢组学也用于食品违禁品的检测,例如化学原料、燃料、硝基呋喃药物等。在转基因食品、食品掺假和食源性检验方面也多有应用,例如利用代谢组学可以在非选择培养基上同时检测大肠杆菌O157∶H7、鼠伤寒沙门氏菌和沙门氏菌等[73];饮食干预:由于饮食成分会影响人体代谢,影响某些疾病的患病率和风险,STELLA等[74]研究不同饮食干预对人体代谢物的影响,研究表明肌酸、肉碱、乙酰肉碱和三甲胺-N-氧化物是高肉类饮食的特征代谢物,说明饮食可以改变人体细菌的组成或新城代谢性能,从而引起代谢物的变化。
种种迹象表明,代谢组学已成为食品研究中的有力工具,在食品质量、溯源、污染、加工以及食品与健康的关系等多方面起到重要的作用。相对于蛋白组学,代谢组学在研究食品风味形成上具有独特的优势,它能从多方面获取风味组成成分,例如脂肪酸、核苷酸、有机酸、氨基酸等,能更加全面的分析食品中物质的组成,为理解食品品质形成、调控食品风味组成、提升食品加工效率带来了积极的作用。WANG等[75]基于代谢组学研究了山羊肉种的风味代谢物,结果表明鲁北白山羊、波尔山羊和济宁灰山羊的脂肪酸、醛、酮、内酯、生物碱、黄酮类化合物、酚类和药物残留物的特征明显不同。YANG等[76]基于代谢组学发现了酱肉成产过程中代表性的代谢物有26种,包括氨基酸、糖类、有机酸、核酸及其衍生物,它们可能是酱肉风味的主要贡献者。SONG等[77]发现泡菜中的游离糖、有机酸、乙醇和氨基酸谱因样品类型而异,正是泡菜风味多样的因素。YAO等[78]基于代谢组学分析发现了16 种与啤酒氧化稳定性相关的潜在代谢物标志物,同时发现啤酒在陈化过程中形成了一些新的化合物。BI等[79]基于代谢组学发现黑胡椒精油包衣降低了金华火腿酸味、甜味、苦味、后味和相对非挥发性味觉代谢物的强度;且强度的降低不依赖于黑胡椒精油的含量。
代谢组学虽然在研究食品组分的变化上具有特有的优势,但受限于现有的仪器设备和材料,并不能完全检测特定时间、特定食品中的全部代谢物,同时局限于检测精度,仍然存在部分关键代谢物不能被有效检测的可能。为了提高分析准确性, 多组学联用成为新的研究热点。代谢组学与基因组学、转录组学和蛋白组学等组学技术联用,不仅可以获得食品品质形成的机制,还可以从分子水平上获得食品质量变化的影响因素,为食品安全控制也带来了新的契机。
5 讨论
基因组学、转录组学、蛋白组学和代谢组学是食品认证、食品质量、食品安全、食品掺假、过敏原和有毒物质检测的有力工具,并参与营养科学的研究,为人类健康提供有力支撑。基因组学发现影响食品安全、质量的潜在因素(生物因素),转录组学表征、定性食品内部的真实代谢活动,蛋白组学获得与食品安全、质量有关的蛋白组分,在此基础上,代谢组学获得与食品安全、质量有关的代谢组分,从而摸清食品生产过程中的所有变化(图2)。虽然单一组学的研究方法已经趋于成熟并且得到了很好的应用,但由于各组学之间存在相互作用,单用一种组学技术并不能全面、客观的揭露食品科学中的动态信息,例如基因组学中微生物注释的精度受食品基质的影响,从而影响转录组学的结果解读;蛋白组学的应用还多局限于动物蛋白,应用于植物基蛋白的准确性有待确认;代谢组学与仪器设备、耗材的发展息息相关等都制约着组学技术在食品中的应用。

图2 食品组学关系图
Fig.2 Diagram of foodomics
食品科学领域是一个动态的研究领域,其发展也与食品组学技术的发展密切相关。而全面、准确的预测食品质量、安全和加工中的具体变化是影响食品标准化和工业化的关键,因此多组学联用将成为未来研究食品的新方向。例如基因组学与蛋白组学联用,可以同时获得影响食品质量和安全的因素及现象;基因组学与代谢组学联用,可以同时获得与微生物相关的代谢活动和其他代谢变化;转录组学与代谢组学联用可以获得代谢物的代谢途径和调控方式等。这些新的研究思路将为研究食品开辟新的领域,促进食品研究的发展。与此同时,随着分析化学、合成生物学等学科的发展,未来分子水平的进步和现有技术的整合将带来新的“组学”和研究方向。
[1] 李媛媛, 买梦奇, 胡小松, 等.食品组学在生物活性化合物营养功能特性研究中的应用[J].食品科学, 2020, 41(3):239-245.
LI Y Y, MAI M Q, HU X S, et al.A literature review of the application of foodomics in studying nutritional and functional properties of bioactive compounds[J].Food Science, 2020, 41(3):239-245.
[2] BORDONI A, CAPOZZI F, FERRANTI P.Preface:FoodOmics.The science for discovering [J].Food Research International, 2014, 63:125.
[3] CIFUENTES A.Foodomics, foodome and modern food analysis [J].TrAC Trends in Analytical Chemistry, 2017, 96:1.
[4] WILLIAMS J K, ANDERSON C M.Omics research ethics considerations [J].Nursing Outlook, 2018, 66(4):386-393.
[5] HERRERO M, SIM C, GARC
C, GARC A-CA
A-CA AS V, et al.Foodomics:MS-based strategies in modern food science and nutrition [J].Mass Spectrometry Reviews, 2012, 31(1):49-69.
AS V, et al.Foodomics:MS-based strategies in modern food science and nutrition [J].Mass Spectrometry Reviews, 2012, 31(1):49-69.
[6] BALKIR P, KEMAHLIOGLU K, YUCEL U.Foodomics:A new approach in food quality and safety [J].Trends in Food Science and Technology, 2021, 108:49-57.
[7] 金强强, 聂秋霞, 田贡洲.探析营养成分检测在食品安全中的重要性[J].食品安全导刊, 2021(20):49;51.
JIN Q Q, NIE Q X, TIAN G Z.Analysis on the importance of nutritional components detection in dood safety[J].China Food Safety Magazine, 2021(20):49;51.
[8] CASTRO-PUYANA M, MENDIOLA J A, IBA EZ E.Strategies for a cleaner new scientific discipline of green foodomics [J].TrAC Trends in Analytical Chemistry, 2013, 52:23-35.
EZ E.Strategies for a cleaner new scientific discipline of green foodomics [J].TrAC Trends in Analytical Chemistry, 2013, 52:23-35.
[9] FASOLI E, RIGHETTI P G.Proteomics of fruits and beverages [J].Current Opinion in Food Science, 2015, 4:76-85.
[10] GERMAN J B, ZIVKOVIC A M, DALLAS D C, et al.Nutrigenomics and personalized diets:What will they mean for food? [J].Annual Review of Food Science and Technology, 2011, 2:97-123.
[11] FRASER C M, EISEN J A, SALZBERG S L.Microbial genome sequencing [J].Nature, 2000, 406(6 797):799-803.
[12] SIKKEMA-RADDATZ B, JOHANSSON L F, DE BOER E N, et al.Targeted next-generation sequencing can replace Sanger sequencing in clinical diagnostics [J].Human Mutation, 2013, 34(7):1 035-1 042.
[13] BOWMAN S K, SIMON M D, DEATON A M, et al.Multiplexed Illumina sequencing libraries from picogram quantities of DNA [J].BMC Genomics, 2013, 14:466.
[14] BEHJATI S, TARPEY P S.What is next generation sequencing? [J].Archives of Disease in Childhood.Education and Practice Edition, 2013, 98(6):236-238.
[15] ROTHBERG J M, HINZ W, REARICK T M, et al.An integrated semiconductor device enabling non-optical genome sequencing [J].Nature, 2011, 475(7 356):348-352.
[16] BURKE C M, DARLING A E.A method for high precision sequencing of near full-length 16S rRNA genes on an Illumina MiSeq [J].PeerJ, 2016, 4:e2492.
[17] CHOI J, PARK J S.Comparative analyses of the V4 and V9 regions of 18S rDNA for the extant eukaryotic community using the Illumina platform [J].Scientific Reports, 2020, 10(1):6519.
[18] GWEON H S, OLIVER A, TAYLOR J, et al.PIPITS:An automated pipeline for analyses of fungal internal transcribed spacer sequences from the Illumina sequencing platform [J].Methods in Ecology and Evolution, 2015, 6(8):973-980.
[19] WANG Y, WANG Z M, HAN Q L, et al.Comprehensive insights into the evolution of microbiological and metabolic characteristics of the fat portion during the processing of traditional Chinese bacon [J].Food Research International,2022,155:110987.
[20] LEHMANN K. Sampling of riverine or marine bacterial communities in remote locations: From field to publication[J]. Methods in Molecular Biology (Clifton, N.J.), 2016, 1452:1-11.
[21] ZHU Y Y, GUO Y, YANG F H, et al.Combined application of high-throughput sequencing and UHPLC-Q/TOF-MS-based metabolomics in the evaluation of microorganisms and metabolites of dry-cured ham of different origins[J].International Journal of Food Microbiology,2021,359:109422.
[22] ZANG J H, XU Y S, XIA W S, et al.Dynamics and diversity of microbial community succession during fermentation of Suan yu, a Chinese traditional fermented fish, determined by high throughput sequencing [J].Food Research International, 2018, 111:565-573.
[23] FAN H, HUO R, ZHAO J N, et al.Microbial diversity analysis of jiaoke from Xilingol, Inner Mongolia [J].Journal of Dairy Science, 2020, 103(7):5 893-5 905.
[24] ZHANG K L, ZHANG Y, LI S, et al. Fungal diversity in Xinjiang traditional cheese and its correlation with moisture content[J]. Indian Journal of Microbiology, 2022, 62(1):47-53.
[25] LIU X Y, QIAN M, SHEN Y X, et al.An high-throughput sequencing approach to the preliminary analysis of bacterial communities associated with changes in amino acid nitrogen, organic acid and reducing sugar contents during soy sauce fermentation [J].Food Chemistry, 2021, 349:129131.
[26] JIANG X W, XU Y T, YE J, et al.Isolation, identification and application on soy sauce fermentation flavor bacteria of CS1.03 [J].Journal of Food Science and Technology, 2019, 56(4):2 016-2 026.
[27] NIU C T, XUE Y, JIA Y, et al. Analysis of bacterial community dynamics in the manufacture process of lajiaojiang (red chili paste)[J]. Lebensmittel-Wissenschaft Und-Technologie, 2019, 122(1):108976.
[28] WANG Z X, SHAO Y Y.Effects of microbial diversity on nitrite concentration in pao cai, a naturally fermented cabbage product from China [J].Food Microbiology, 2018, 72:185-192.
[29] CHAO S H, KUDO Y, TSAI Y C, et al.Lactobacillus futsaii sp.nov., isolated from fu-tsai and suan-tsai, traditional Taiwanese fermented mustard products [J].International Journal of Systematic and Evolutionary Microbiology, 2012, 62(Pt 3):489-494.
[30] LIU Z G, LI J Y, WEI B L, et al.Bacterial community and composition in Jiang-shui and Suan-cai revealed by high-throughput sequencing of 16S rRNA [J].International Journal of Food Microbiology, 2019, 306:108271.
[31] ZHENG Y, HU X L, JIA Z J, et al.Co-occurrence patterns among prokaryotes across an age gradient in pit mud of Chinese strong-flavor liquor [J].Canadian Journal of Microbiology, 2020, 66(9):495-504.
[32] XU J L, SUN L P, XING X, et al. Culturing bacteria from fermentation pit muds of Baijiu with culturomics and amplicon-based metagenomic approaches[J]. Frontiers in Microbiology, 2020, 11:1223.
[33] WANG L, HUANG Y G, HU X X, et al.The impact of environmental factors on the environmental bacterial diversity and composition in the Jiang-flavoured Baijiu production region [J].LWT, 2021, 149:111784.
[34] MA W R, GENG X J, JIA F C, et al. Investigation of microbial composition and functional characterization of Zangqu using high throughput sequencing[J]. Lebensmittel-Wissenschaft Und-Technologie, 2021, 136(8):110342.
[35] GAN S H, YANG F, SAHU S K, et al.Deciphering the composition and functional profile of the microbial communities in Chinese Moutai liquor starters[J].Frontiers in Microbiology, 2019, 10:1540.
[36] WANG P, HU A T, FAN X P, et al.Bacterial communities in prepared foods available at supermarkets in Beijing, China [J].Food Research International, 2019, 120:668-678.
[37] WONG J, VISWESWARA RAO P, SOON-SINCLAIR J M.Identification of Klebsiella pneumoniae in microwavable chicken curries using 16S rRNA analysis [J].International Food Research Journal, 2018, 25(2):620-625.
[38] ZHERNAKOVA A, KURILSHIKOV A, BONDER M J, et al.Population-based metagenomics analysis reveals markers for gut microbiome composition and diversity [J].Science, 2016, 352(6 285):565-569.
[39] LI Y M, CAO W W, LIANG S L, et al.Metagenomic characterization of bacterial community and antibiotic resistance genes in representative ready-to-eat food in Southern China[J].Scientific Reports, 2020, 10(1):15175.
[40] YANG P, ZHONG G X, YANG J C, et al. Metagenomic and metabolomic profiling reveals the correlation between the microbiota and flavor compounds and nutrients in fermented sausages[J]. Food Chemistry, 2022, 375:131645.
[41] LI W V, LI J J.Modeling and analysis of RNA-seq data:A review from a statistical perspective[J].Quantitative Biology, 2018, 6(3):195-209.
[42] HRDLICKOVA R, TOLOUE M, TIAN B.RNA-Seq methods for transcriptome analysis [J].Wiley Interdisciplinary Reviews:RNA, 2017, 8(1):e1364.
[43] VALDÉS A, IB
 EZ C, SIM
EZ C, SIM C, et al.Recent transcriptomics advances and emerging applications in food science [J].TrAC-Trends in Analytical Chemistry, 2013, 52:142-154.
C, et al.Recent transcriptomics advances and emerging applications in food science [J].TrAC-Trends in Analytical Chemistry, 2013, 52:142-154.
[44] LOWE R, SHIRLEY N, BLEACKLEY M, et al.Transcriptomics technologies [J].PLoS Computational Biology, 2017, 13(5):e1005457.
[45] 赵乐, 孟祥晨.转录组测序技术在植物乳杆菌细菌素合成研究中的应用研究进展[J].乳业科学与技术,2021, 44(5):32-37.
ZHAO L,MENG X C.A Review of the application of transcriptome sequencing in research on bacteriocin synthesis by Lactobacillus plantarum[J].Journal of Dairy Science and Technology, 2021, 44(5):32-37.
[46] YANG C, LI M, PENG B Z.Transcriptomic analysis reveals the metabolic mechanism of patulin by Saccharomyces cerevisiae during fermentation [J].LWT, 2021, 149:111808.
[47] QIAN Y L, LI Y, XU T T, et al. Dissecting of the AI-2/LuxS mediated growth characteristics and bacteriostatic ability of Lactiplantibacillus plantarum SS-128 by integration of transcriptomics and metabolomics[J]. Foods (Basel, Switzerland), 2022, 11(5):638.
[48] YE D, DU F, ZOU Y J, et al.Transcriptomics analysis of primordium formation in Pleurotus eryngii[J].Genes, 2021, 12(12):1863.
[49] DOU T F, YAN S X, LIU L X, et al.Integrative analysis of transcriptomics and metabolomics to reveal the melanogenesis pathway of muscle and related meat characters in Wuliangshan black-boned chickens [J].BMC Genomics, 2022, 23(1):173.
[50] DU H Y, XIONG S B, LV H, et al.Comprehensive analysis of transcriptomics and metabolomics to understand the flesh quality regulation of crucian carp (Carassius auratus) treated with short term micro-flowing water system[J].Food Research International, 2021, 147:110519.
[51] ONO N N, BRITTON M T, FASS J N, et al. Exploring the transcriptome landscape of pomegranate fruit peel for natural product biosynthetic gene and SSR marker DiscoveryF[J]. Journal of Integrative Plant Biology, 2011, 53(10):800-813.
[52] LANCOVA K, DIP R, ANTIGNAC J P, et al.Detection of hazardous food contaminants by transcriptomics fingerprinting[J].TrAC Trends in Analytical Chemistry,2011,30(2):181-191.
[53] CLEMENT F C, DIP R, NAEGELI H.Expression profile of human cells in culture exposed to glycidamide, a reactive metabolite of the heat-induced food carcinogen acrylamide[J].Toxicology,2007,240(1-2):111-124.
[54] VALDÉS A, SIM C, IB
C, IB
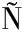 EZ C, et al. Foodomics strategies for the analysis of transgenic foods[J]. Trends in Analytical Chemistry, 2013, 52:2-15.
EZ C, et al. Foodomics strategies for the analysis of transgenic foods[J]. Trends in Analytical Chemistry, 2013, 52:2-15.
[55] 剧柠, 苟萌, 张彤彤.蛋白质组学技术及其在乳及乳制品中的应用研究进展 [J].食品与发酵工业, 2021, 47(3):245-251.
JU N, GOU M, ZHANG T T.Advances in proteomics and its application in milk and dairy products [J].Food and Fermentation Industries,2021,47(3):245-251.
[56] AIZAT W M, HASSAN M.Proteomics in Systems Biology[M]. Cham:Springer,2018:31-49.
[57] BEGUM F.Proteomic measures of albumen degradation as indicators of egg freshness [D].Sydney:The University of Sydney, 2021.
[58] GALLARDO J M, ORTEA I, CARRERA M.Proteomics and its applications for food authentication and food-technology research [J].TrAC-Trends in Analytical Chemistry, 2013, 52:135-141.
[59] MONACI L, DE ANGELIS E, MONTEMURRO N, et al. Comprehensive overview and recent advances in proteomics MS based methods for food allergens analysis[J]. TrAC Trends in Analytical Chemistry, 2018, 106:21-36.
[60] 梁荣蓉, 许宝琛, 张一敏, 等.蛋白质组学在生鲜肉肉色变化机制研究中的应用[J].农业工程学报, 2020, 36(16):283-292.
LIANG R R, XU B C, ZHANG Y M, et al.Application of proteomics in mechanism research of fresh meat color changes[J].Transactions of the Chinese Society of Agricultural Engineering, 2020, 36(16):283-292.
[61] JI C F, ZHANG J B, LIN X P, et al.Metaproteomic analysis of microbiota in the fermented fish, Siniperca chuatsi[J].LWT, 2017, 80:479-484.
[62] LIU F J, SHEN S K, CHEN Y W, et al.Quantitative proteomics reveals the relationship between protein changes and off-flavor in Russian sturgeon (Acipenser gueldenstaedti) fillets treated with low temperature vacuum heating[J].Food Chemistry, 2022, 370:131371.
[63] JIA W, SHI Q Y, ZHANG R, et al.Unraveling proteome changes of irradiated goat meat and its relationship to off-flavor analyzed by high-throughput proteomics analysis[J].Food Chemistry, 2021, 337:127806.
[64] ZHAO G Z, LIU C, LI S, et al.Exploring the flavor formation mechanism under osmotic conditions during soy sauce fermentation in Aspergillus oryzae by proteomic analysis[J].Food &Function, 2020, 11(1):640-648.
[65] ZHOU C Y, WU J Q, TANG C B, et al.Comparing the proteomic profile of proteins and the sensory characteristics in Jinhua ham with different processing procedures[J].Food Control, 2019, 106:106694.
[66] 尤丽琴, 姬琛, 罗瑞明.蛋白质组学揭示滩羊宰后成熟过程中风味前体物质的变化机理[J].食品科学, 2021, 42(19):20-27.
YOU L Q, JI C, LUO R M.Proteomic analysis revealed the mechanism of changes in flavor precursors in Tan sheep meat during post-mortem ageing[J].Food Science, 2021, 42(19):20-27.
[67] 陈小威, 尹文俊, 马传国. 植物蛋白与天然双亲皂苷相互作用及对食品乳液和泡沫的稳态化研究进展[J]. 食品科学, 2022, 43(19):270-276.
CHEN X W, YIN W J, MA C G. Interactions between plant proteins and saponins and their role in the stabilization of food emulsions and foams: A review of recent progress[J]. Food Science, 2022, 43(19):270-276.
[68] VAN DER MEULEN R, SCHEIRLINCK I, VAN SCHOOR A, et al.Population dynamics and metabolite target analysis of lactic acid bacteria during laboratory fermentations of wheat and spelt sourdoughs[J].Applied and Environmental Microbiology, 2007, 73(15):4 741-4 750.
[69] 梁强, 郭晓晖, 周蓓莉, 等.代谢指纹分析及其在食品科学中的应用[J].食品与发酵工业, 2011, 37(10):134-138.
LIANG Q, GUO X H, ZHOU B L, et al.Metabolic fingerprinting analysis and the application in food science[J].Food and Fermentation Industries, 2011, 37(10):134-138.
[70] JIANG N H, ZHU H L, LIU W, et al. Metabolite differences of polyphenols in different litchi cultivars (Litchi chinensis sonn.) based on extensive targeted metabonomics[J]. Molecules (Basel, Switzerland), 2021, 26(4):1181.
[71] HE Q H, YIN Y L, ZHAO F, et al. Metabonomics and its role in amino acid nutrition research[J]. Frontiers in Bioscience (Landmark Edition), 2011, 16(7):2 451-2 460.
[72] SPRAUL M, SCHÜTZ B, RINKE P, et al.NMR-based multi parametric quality control of fruit juices:SGF profiling[J].Nutrients, 2009, 1(2):148-155.
[73] LIU S J, WU Y N, CHAN L.Application of metabonomics approach in food safety research-a review[J].Food Reviews International, 2020, 36(6):547-558.
[74] STELLA C, BECKWITH-HALL B, CLOAREC O, et al.Susceptibility of human metabolic phenotypes to dietary modulation[J].Journal of Proteome Research, 2006, 5(10):2 780-2 788.
[75] WANG W T, SUN B, HU P, et al.Comparison of differential flavor metabolites in meat of lubei white goat, jining gray goat and boer goat[J].Metabolites, 2019, 9(9):176.
[76] YANG Y, YE Y F, PAN D D, et al.Metabonomics profiling of marinated meat in soy sauce during processing[J].Journal of the Science of Food and Agriculture, 2018, 98(4):1 325-1 331.
[77] SONG H S, LEE S H, AHN S W, et al.Effects of the main ingredients of the fermented food, kimchi, on bacterial composition and metabolite profile[J].Food Research International, 2021, 149:110668.
[78] YAO J W, ZONG X Y, CUI C, et al.Metabonomics analysis of nonvolatile small molecules of beers during forced ageing[J].International Journal of Food Science &Technology, 2018, 53(7):1 698-1 704.
[79] BI Y, WANG Y, ZHOU G H, et al. The effect of coating incorporated with black pepper essential oil on the taste quality of Jinhua ham after storage for four months[J]. Journal of Food Science, 2019, 84(11):3 109-3 116.