由核苷酶催化的核苷水解反应普遍存在于生物中,参与核苷的代谢[1]。根据底物和催化机制不同,核苷酶可分为核苷磷酸化酶(nucleoside phosphorylase,EC 2.4.2.-)和核苷水解酶(nucleoside hydrolase,EC 3.2.2.-)[1-2]。前者催化核苷和磷酸生成D-核糖-1-磷酸和含氮碱基(嘌呤或嘧啶),后者催化核苷和水生成D-核糖和含氮碱基[3]。根据最适催化底物不同,核苷水解酶又可分为嘌呤核苷水解酶(EC 3.2.2.1)、次黄嘌呤核苷水解酶(EC 3.2.2.2)和尿嘧啶核苷水解酶(EC 3.2.2.3)。核苷水解酶广泛存在于微生物、植物和非哺乳动物中,业已在戊糖乳杆菌、肠道沙门氏菌、荧光假单胞菌、大肠杆菌、黑曲霉、硕大利什曼原虫、布氏锥虫、蚊、黄羽扇豆和小果咖啡中发现核苷水解酶[4-6]。核苷水解酶是原生动物核苷补救途径中的关键酶,而哺乳动物中未发现此酶,故核苷水解酶已作为药物靶点抑制或杀灭致病原生动物用于治疗相关疾病(如治疗夏格病、疟疾等)[7-9]。在原核及高等真核生物中,核苷水解酶的作用具有多样性,如TODD等[10]发现蜡状芽孢杆菌(Bacillus cereus)和炭疽芽孢杆菌(Bacillus anthracis)芽孢外围附着有核苷水解酶,推测该酶具有降低芽孢对其代谢物敏感性并防止芽孢提前萌发的作用;RIBEIRO等[11]在雌性埃及伊蚊(Aedes aegypti)唾液中发现高活性嘌呤核苷水解酶而在雄性埃及伊蚊中活性极低,推测与其吸食血液有关。
D-核糖、嘌呤和嘧啶参与核酸、维生素、辅酶等多种物质的合成及能量转移等新陈代谢过程,具有重要的生理功能,同时还可用多种药物的合成(如鸟嘌呤是阿昔洛韦、泛昔洛韦等抗疱疹病药物合成的前体物),故在食品、医药等领域具有广泛的应用前景和市场需求[12-16]。目前D-核糖主要采用发酵法合成,嘌呤和嘧啶采用化学法合成,总体而言,后者合成效率不高。以核苷为底物利用核苷水解酶可“一锅法”合成D-核糖及嘌呤或嘧啶,可为其生物合成提供思路。谷氨酸棒杆菌(Corynebacterium glutamicum)是常见工业微生物,已用于氨基酸、有机酸等多种化学品的生物合成[17-19]。经基因组序列和途径分析发现,该菌中含有3个注释的核苷水解酶编码基因(cgl1364、cgl1977和cgl2835),但其功能未见报道。本文首先克隆上述核苷水解酶编码基因并构建大肠杆菌表达系统,然后分离纯化重组酶并对其酶学性质进行研究,以期为其在D-核糖及嘌呤或嘧啶合成中的应用提供参考。
1 材料与方法
1.1 试剂与仪器
葡萄糖等常规生化试剂,国药集团试剂有限公司;D-核糖、腺苷、胞苷、鸟苷、黄苷、肌苷、尿苷、腺嘌呤、胞嘧啶、鸟嘌呤、黄嘌呤、次黄嘌呤、尿嘧啶,上海阿拉丁生化科技股份有限公司;TaKaRa Ex Taq®试剂盒、限制性内切酶等分子生物学试剂,宝日医生物技术(北京)有限公司。
Thermo ULtiMate 3000高效液相色谱仪,美国Thermo Fisher Scientific公司;Mastercycler nexus PCR仪,德国Eppendorf公司。
1.2 菌株与质粒
大肠杆菌Escherich coli BL21(DE3)和谷氨酸棒杆菌Corynebacterium glutamicum ATCC 13032及质粒pET-28a均由本实验室保藏。
1.3 引物
本研究所用引物如表1所示。
表1 本研究所用引物
Table 1 Primers used in this study

引物名称序列(5′-3′)1364-1GCCGGATCCATGACCACCAAGATCATCCTCG (BamH)1364-2CGG AAGCTTCTATCCGATGCGCTTTACTGC (Hind Ⅲ)1977-1CGGCCATGGGTATGAGCAAAAAAGCCATCCTT (Nco Ⅰ)1977-2CGGCTCGAGCTGCTGTGCTGCGACTCG (Xho Ⅰ)2835-1GCCGGATCCATGATTCCTGTTCTCATCGACTGC (BamH Ⅰ)28352CGGAAGCTTTCATTCCACTGCTCTTAGAAGTTCC (Hind Ⅲ)
注:下划线表示酶切位点。
1.4 培养基
LB培养基(g/L):胰化蛋白胨10,酵母提取物5,NaCl 10,pH(7.0~7.5),121 ℃高压蒸汽灭菌20 min。根据需要添加终质量浓度为50 μg/mL的卡那霉素。
基本培养基(g/L):葡萄糖2,NH4Cl 1,Na2HPO4 12.8,KH2PO4 3,MgSO4·7H2O 0.5,FeSO4·7H2O 0.01,pH(7.0~7.4),115 ℃高压蒸汽灭菌20 min[20]。根据需要添加终质量浓度为50 μg/mL的卡那霉素。
1.5 重组蛋白序列比对
于ClustalW网站采用Clustal算法进行序列比对分析,于ENDscript/ESPript网站作图。
1.6 目的基因的PCR扩增
根据NCBI中报道的C.glutamicum ATCC 13032的cgl1364、cgl1977和cgl2835序列,利用软件Primer Premier 5设计扩增引物(序列见表1),由苏州金唯智生物科技有限公司合成。以C.glutamicum ATCC 13032基因组DNA为模板,分别利用相应引物及TaKaRa Ex Taq®试剂盒扩增上述基因。PCR条件为94 ℃ 5 min 1个循环,94 ℃ 30 s、56 ℃ 30 s、72 ℃ 90 s 30个循环,72 ℃ 10 min 1个循环,反应体系为100 μL。产物经琼脂糖凝胶电泳检测并切胶回收。
1.7 表达载体的构建
PCR扩增产物切胶回收后,利用相应的限制性内切酶酶切(表1),经琼脂糖凝胶电泳、切胶、回收后连接至经相同酶切的表达载体pET-28a,然后转化至E.coli BL21(DE3),经活化后涂布于含50 μg/mL卡那霉素的LB固体培养基,于37 ℃倒置培养。挑取转化子单菌落,进行菌落PCR鉴定。转化子经活化后提取质粒,分别进行单、双酶切验证[cgl1977(Xho Ⅰ单酶切、Nco Ⅰ和Xho Ⅰ双酶切),cgl1364和cgl2835(BamH Ⅰ单酶切、BamH Ⅰ和Hind Ⅲ双酶切)]。
1.8 重组蛋白的表达及分离纯化
分别将BL-cgl1364、BL-cgl1977和BL-cgl2835的种子培养物接种至2 L含50 μg/mL卡那霉素的LB液体培养基中,利用终浓度为0.1 mmol/L的IPTG对其诱导表达。收集上述细胞后超声破碎,并利用十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis, SDS-PAGE)检测重组蛋白[21],对照为不含目的基因、未经IPTG诱导的转化有pET28a的E.coli BL21(DE3)。利用Ni-NTA树脂法收集重组蛋白。
1.9 重组核苷水解酶活性检测
采用1 mL反应体系,10 mmol/L底物和适量酶溶于磷酸盐缓冲液(0.2 mol/L,pH 7.0)。将反应体系于37 ℃水浴1~20 min,反应结束后加入100 μL稀盐酸[22]。上述反应体系于10 000 r/min离心10 min后取上清液,测定底物和D-核糖浓度。将底物浓度设定为0~10 mmol/L,采用Lineweaver-Burk双倒数作图法分析重组酶的Km和Vmax值。分别于10~60 ℃(pH 7.0)以及pH 3~11(35 ℃)测定重组酶活性,以考察其最适温度和pH;分别于25、35、45、55 ℃放置1 h后于35 ℃测定重组酶活性以考察其热稳定性;分别将重组酶于25、35、45 ℃放置1~6 h后测定其活性,进一步考察其热稳定性。
1.10 利用静息细胞催化水解尿苷
将IPTG诱导4 h的BL-cgl1364培养物经6 000×g离心10 min后收集菌体细胞,用生理盐水清洗2次后置于-80 ℃冰箱12 h,即为静息细胞。取6 g静息细胞于室温放置10 min后转移至含200 mL反应体系(pH 7.0、10 mmol/L磷酸盐缓冲液含20 mmol/L尿苷)的摇瓶中,于35 ℃、200 r/min反应4 h。将反应液于4 ℃、6 000×g离心10 min后取上清液,测定D-核糖浓度。
1.11 检测与分析方法
采用二喹啉甲酸(bicinchoninic acid,BCA)蛋白定量试剂盒测定重组蛋白浓度[20]。按照参考文献[22]和[23]的方法采用高效液相色谱法测定核苷、嘌呤、嘧啶及D-核糖的浓度。
2 结果与分析
2.1 cgl1364、cgl1977和cgl2835的克隆及表达体系构建
经KEGG和NCBI数据库分析,C.glutamicum ATCC 13032含有3个核苷水解酶,分别为Cgl1364、Cgl1977和Cgl2835,均被注释为肌苷/尿苷水解酶。对Cgl1364、Cgl1977和Cgl2835进行同源性分析,结果如图1所示,三者具有一定的同源性,其中Clg1364与Cgl1997和Cgl2835同源性分别为54%和45%。由此可见,Clg1364与Cgl1997同源性更高。

图1 Cgl1364、Cgl1977和Cgl2835同源性分析
Fig.1 Alignment analysis of Cgl1364, Cgl1977 and Cgl2835
为考察其功能,克隆并表达cgl1364、cgl1977和cgl2835。以C.glutamicum ATCC 13032基因组DNA为模板扩增获得与上述基因的理论碱基数接近(分别为951、939、906 bp)的片段(图2)。

M-marker;a1-cgl1364;b1-cgl1977;c1-cgl2835;a-cgl1364基因的PCR扩增图谱;b-cgl1977基因的PCR扩增图谱;c-cgl2835基因的PCR扩增图谱
图2 cgl1364、cgl1977和cgl2835基因PCR扩增图谱
Fig.2 Maps for cgl1364,cgl1977 and cgl2835 amplifying products
回收cgl1364、cgl1977和cgl2835基因PCR扩增产物,经双酶切后连接至pET-28a,经转化大肠杆菌E.coli BL21(DE3)后提取重组子质粒进行酶切验证,结果如图3所示。

a:M-Marker;1-cgl1364基因PCR扩增片段;2-pET28a经BamH Ⅰ单酶切;3-pET28a经BamH Ⅰ和Hind Ⅲ双酶切;4-pET-1364经BamH Ⅰ单酶切;5-pET-1364经BamH Ⅰ和Hind Ⅲ双酶切;b:M-Marker;1-cgl1977基因PCR扩增片段;2-pET28a经Xho Ⅰ单酶切;3-pET28a经Nco Ⅰ和Xho Ⅰ双酶切;4-pET-1977经Xho Ⅰ单酶切;5-pET-1977经Nco Ⅰ和Xho Ⅰ双酶切;c:M-Marker;1-cgl2835基因PCR扩增片段; 2-pET28a经BamH Ⅰ单酶;3-pET28a经BamH Ⅰ和Hind Ⅲ双酶切;4-pET-2835经BamH Ⅰ单酶切;5-pET-2835经BamH Ⅰ和Hind Ⅲ双酶切;a-重组质粒pET-1364酶切鉴定图谱;b-重组质粒pET-1977酶切鉴定图谱;c-重组质粒pET-2835酶切鉴定图谱
图3 重组质粒酶切鉴定图谱
Fig.3 Maps for identification of recombinant plasmids
质粒经双酶切后分别获得碱基数为5 350和939 bp,5 237和951 bp以及5 350和906 bp的条带,表明重组质粒和菌株构建成功。分别将鉴定正确的重组质粒命名为pET-1364、pET-1977和pET-2835,相应的重组菌株命名为BL-cgl1364、BL-cgl1977和BL-cgl2835。
2.2 重组酶的表达及纯化
经IPTG诱导培养的重组菌株BL-cgl1364、BL-cgl1977和BL-cgl2835破碎后蛋白质表达图谱如图4所示。全菌破碎液及其上清液中均出现一条分子质量为30~40 kDa的条带,与Cgl1364、Cgl1977和Cgl2835理论分子质量一致(分别为37.6、34.9、37.1 kDa,包含6×His标签),说明重组蛋白均能够可溶性表达;BL-cgl1364和BL-cgl2835的菌体破碎物中还存在着一定量的重组蛋白包涵体。

M-Marker;1~3-转化有pET28a的E.coli BL21(DE3)全菌破碎液、破碎物沉淀和上清液;4~6-BL-cgl1364、BL-cgl1977和BL-cgl2835的全菌破碎液、破碎物沉淀和上清液;a-BL-cgl1364菌体破碎物;b-BL-cgl1977菌体破碎物;c-BL-cgl2835菌体破碎物
图4 BL-cgl1364、BL-cgl1977和BL-cgl2835表达产物的SDS-PAGE图谱
Fig.4 SDS-PAGE maps of proteins in BL-cgl1364,BL-cgl1977 and BL-cgl2835
利用Ni-NTA亲和树脂分离纯化菌体破碎物中的重组酶。经透析、浓缩后进行SDS-PAGE分析,结果如图5所示。重组Cgl1364蛋白存在于含20~500 mmol/L咪唑的洗脱物中,但含200 mmol/L咪唑的洗脱物中浓度最高,而含500 mmol/L咪唑的洗脱物中纯度最高;重组Cgl1977蛋白出现于含20~200 mmol/L咪唑的洗脱物中,含80 mmol/L咪唑的洗脱物中浓度最高,而含200 mmol/L咪唑的洗脱物中纯度最高;重组Cgl2835出现于含80~500 mmol/L 咪唑的洗脱物中,其中含200 mmol/L咪唑的洗脱物中浓度最高,而含500 mmol/L咪唑的洗脱物中纯度最高。经测定,重组Cgl1364、Cgl1977和Cgl2835蛋白质量浓度分别为4.1、3.2和1.9 mg/mL。上述结果表明,重组蛋白纯化质量理想,可用于后续酶学实验。

M-Marker;1-菌体破碎物;2-菌体破碎物上清液;3-穿透液;4~9-含20、50、80、100、200、500 mmol/L咪唑的洗脱物;a-重组Cgl1364 SDS-PAGE图谱;b-重组Cgl1977 SDS-PAGE图谱;c-重组Cgl2835 SDS-PAGE图谱
图5 纯化重组酶SDS-PAGE图谱
Fig.5 SDS-PAGE maps of purified recombinant enzymes
2.3 重组酶酶学特性分析
2.3.1 重组酶的活性及底物特异性分析
选择腺苷、胞苷、鸟苷、黄苷、肌苷及尿苷作为底物,分别测定不同浓度条件下重组Cgl1364、Cgl1977和Cgl2835的反应速度,并采用Lineweaver-Burk双倒数法测定其米氏常数Km及最大反应速度Vmax。结果如表2所示。
表2 重组Cgl1364、Cgl1977和Cgl2835的酶动力学参数
Table 2 The kinetic parameters of recombinant enzymes Cgl1364, Cgl1977 and Cgl2835

底物Cgl1364Cgl1977Cgl2835Km/(μmol·L-1)Vmax/[μmol·(min·mg)-1]Km/(μmol·L-1)Vmax/[μmol·(min·mg)-1]Km/(μmol·L-1)Vmax/[μmol·(min·mg)-1]腺苷434.782.59383.792.63221.262.29胞苷198.512.14203.762.18379.252.40鸟苷230.972.22183.652.17195.572.18黄苷222.572.18180.242.11194.662.14肌苷239.842.31220.752.30202.282.23尿苷183.042.10198.142.16516.912.74
3种核酸酶均能够催化上述6种底物,生成D-核糖及相应的嘌呤或嘧啶。重组酶对各底物的亲和性存在差异:Cgl1364对尿苷和胞苷亲和性最强,其次是黄苷、鸟苷和肌苷,对腺苷亲和性最差;Cgl1977对鸟苷和黄苷亲和性最强,其次是尿苷、胞苷和肌苷,对腺苷亲和性最差;Cgl2835对鸟苷和黄苷亲和性最强,其次是肌苷、腺苷和胞苷,对尿苷亲和性最差。
2.3.2 重组酶的最适反应温度
分别于10~60 ℃条件下测定重组Cgl1364、Cgl1977和Cgl2835的相对活性,结果如图6所示。3种重组酶活性均随温度的提高而不断升高,当温度达到35 ℃时活性最高,然后随着温度的继续提升而迅速下降,60 ℃时均完全丧失活性。由图6还可知,Cgl1364和Cgl1977在低于35 ℃条件下,活性随温度提高而升高的幅度较Cgl2835大;当高于35 ℃时,Cgl1977活性随温度提高而降低的幅度较Cgl1364和Cgl2835小。

图6 温度对重组酶活性的影响
Fig.6 Effect of temperature on the activities of recombinant enzymes
2.3.3 重组酶的最适反应pH
在最适温度(35 ℃)条件下测定不同pH(3~11)对重组酶活性的影响,结果如图7所示。随着pH的升高,重组酶的活性逐渐升高,pH 7时活性最高,随后逐渐降低,pH 3和11时均无活性。

图7 pH对重组酶活性的影响
Fig.7 Effect of pH on the activities of recombinant enzymes
2.3.4 重组酶的热稳定性
分别将重组酶于不同温度(25~55 ℃)条件下水浴后测定其热稳定性,结果如图8所示。随着处理温度的逐渐提高,3个重组酶活性均剧烈下降,经55 ℃处理后完全丧失活性。经35和45 ℃处理的重组Cgl1977残留活性高于Cgl1364和Cgl2835,表明其热稳定性优于后者。分别将重组酶于25、35和45 ℃放置不同时间以进一步测定其稳定性,结果如图9所示。重组酶活性随着处理时间的延长均呈现不同程度的下降,45 ℃条件下最为明显;在35 ℃条件下处理2 h后仍具有80%的活性。由图8和图9所知,Cgl1977的热稳定性优于Cgl1364和Cgl2835。

图8 重组酶的热稳定性
Fig.8 Thermal stability of the recombinant enzymes

a-25 ℃下重组酶热稳定性;b-35 ℃下重组酶热稳定性;c-45 ℃下重组酶热稳定性
图9 不同温度下重组酶的热稳定性
Fig.9 Thermal stability of the recombinant enzymes at different temperatures
2.4 利用含重组酶的静息细胞水解尿苷
采用前期建立的静息细胞催化法,选取BL-cgl1364静息细胞为酶源进行催化反应水解其最适底物尿苷,结果如图10所示。产物生成量随催化时间的延长而增加,3.5 h时产物浓度达到最高(17.9 mmol/L),摩尔转化效率为89.5%。
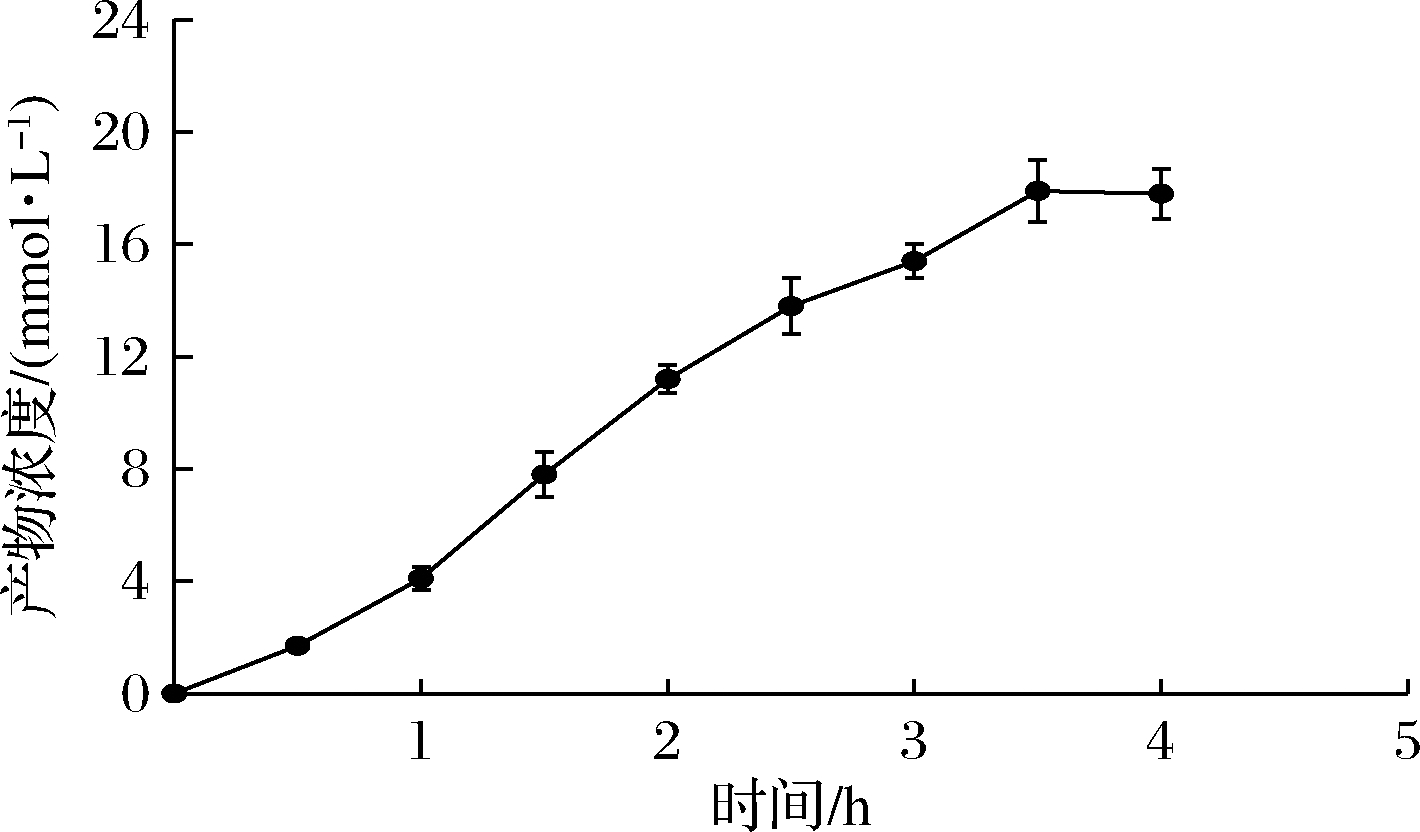
图10 BL-cgl1364静息细胞催化过程曲线
Fig.10 Curve of uridine hydrolysis by BL-cgl1364 resting cells
3 结论
本研究首次表达并纯化了来源于C.glutamicum ATCC 13032的核苷水解酶Cgl1364、Cgl1977和Cgl2835并明确其酶学特性。3种重组核苷水解酶均能够以腺苷、胞苷、鸟苷、黄苷、肌苷及尿苷为底物催化生成D-核糖及相应的嘌呤或嘧啶,但其底物亲和性有所差异;其最适催化温度和pH分别为35 ℃和7.0;Cgl1977的热稳定性优于Cgl1364和Cgl2835。利用BL-cgl1364静息细胞为酶源可催化水解尿苷,底物转化率为89.5%。该研究结果可为D-核糖及碱基的生物合成提供依据。在后续研究中,会进一步考察BL-cgl1977和BL-cgl2835静息细胞对鸟苷、黄苷及腺苷等底物的催化效率;在此基础上,对催化体系进一步放大以评估3种静息细胞的催化性能,并通过条件优化以期获得最佳合成工艺。
[1] WU F F, HU S P, RAN Y, et al.Gene mining and sequence analysis of purine nucleosidase based on RNA-seq[J].Journal of Visualized Experiments:JoVE, 2020(164):e61561.
[2] RENTSEN R L, BRODERSEN D E, ZHANG Y E.Evolution of the bacterial nucleosidase PpnN and its relation to the stringent response[J].Microbial Cell (Graz, Austria), 2019, 6(9):450-453.
[3] KHANDAZHINSKAYA A, FATEEV I, KONSTANTINOVA I, et al.Synthesis of new 5′-norcarbocyclic aza/deaza purine fleximers-noncompetitive inhibitors of E.coli purine nucleoside phosphorylase[J].Frontiers in Chemistry, 2022, 10:867587.
[4] HANSEN M R, DANDANELL G.Purification and characterization of RihC, a xanthosine-inosine-uridine-adenosine-preferring hydrolase from Salmonella enterica serovar typhimurium[J].Biochimica et Biophysica Acta (BBA)-General Subjects, 2005, 1723(1-3):55-62.
[5] MUELLERS S N, NYITRAY M M, REYNAROWYCH N, et al.Structure-guided insight into the specificity and mechanism of a parasitic nucleoside hydrolase[J].Biochemistry, 2022, 61(17):1 853-1 861.
[6] SALES E M, SOUSA G S, BELOUEZZANE C, et al.Expression, purification and spectrophotometric analysis of nucleoside hydrolase from Leishmania chagasi (LcNH)[J].Protein Expression and Purification, 2019, 161:40-48.
[7] VERSÉES W, STEYAERT J.Catalysis by nucleoside hydrolases[J].Current Opinion in Structural Biology, 2003, 13(6):731-738.
[8] CASANOVA L M, RODRIGUES L M, DE AGUIAR P F, et al.An NMR-based chemometric strategy to identify Leishmania donovani nucleoside hydrolase inhibitors from the Brazilian tree Ormosia arborea[J].Journal of Natural Products, 2020, 83(2):243-254.
[9] PARK S M, LEE H A, CHU K B, et al.Production of a polyclonal antibody against inosine-uridine preferring nucleoside hydrolase of Acanthamoeba castellanii and its access to diagnosis of Acanthamoeba keratitis[J].PLoS One, 2020, 15(9):e0239867.
[10] TODD S J, MOIR A J, JOHNSON M J, et al.Genes of Bacillus cereus and Bacillus anthracis encoding proteins of the exosporium[J].Journal of Bacteriology, 2003, 185(11):3 373-3 378.
[11] RIBEIRO J M C, VALENZUELA J G.The salivary purine nucleosidase of the mosquito, Aedes aegypti[J].Insect Biochemistry and Molecular Biology, 2003, 33(1):13-22.
[12] NEUBAUER S.The failing heart:An engine out of fuel[J].The New England Journal of Medicine, 2007, 356(11):1 140-1 151.
[13] FALLER K M, MEDWAY D J, AKSENTIJEVIC D, et al.Ribose supplementation alone or with elevated creatine does not preserve high energy nucleotides or cardiac function in the failing mouse heart[J].PLoS One, 2013, 8(6):e66461.
[14] KRISHNAN V B R, KUMARI MERUGU S, JYOTHSNA GALI S, et al.Efficient multicomponent synthesis of biginelli-dihydro-pyrimidines and evaluation of anti-cancer activity[J].Chemistry Select, 2022, 7(29):e202201630.
[15] NAGHIYEV F, MAMEDOV I, ASKEROV R, et al.Synthesis and biological activity of functionally substituted pyrimidine and pyran derivatives on the basis of isatylidene malononitriles[J].ChemistrySelect, 2022, 7(33):e202202006.
[16] OGUNLADE B, FIDELIS O P, AFOLAYAN O O, et al.Neurotherapeutic and antioxidant response of D-ribose-L-Cysteine nutritional dietary supplements on Alzheimer-type hippocampal neurodegeneration induced by cuprizone in adult male wistar rat model[J].Food and Chemical Toxicology, 2021, 147:111862.
[17] CHUNG S C, PARK J S, YUN J E, et al.Improvement of succinate production by release of end-product inhibition in Corynebacterium glutamicum[J].Metabolic Engineering, 2017, 40:157-164.
[18] TSUGE Y, MATSUZAWA H.Recent progress in production of amino acid-derived chemicals using Corynebacterium glutamicum[J].World Journal of Microbiology &Biotechnology, 2021, 37(3):49.
[19] WANG Y, ZHENG P, XU Z H, et al.Editorial:Engineering Corynebacterium glutamicum chassis for synthetic biology, biomanufacturing, and bioremediation[J].Frontiers in Bioengineering and Biotechnology, 2022, 10:923145.
[20] 张成林, 刘远, 薛宁, 等.苏云金芽孢杆菌重组L-异亮氨酸羟化酶的酶学性质及其在4-羟基异亮氨酸合成中的应用[J].微生物学报, 2014, 54(8):889-896.
ZHANG C L, LIU Y, XUE N, et al.Characterization of recombinant L-isoleucine-4-hydroxylase from Bacillus thuringiensis and its application in 4-hydroxyisoleucine biosynthesis[J].Acta Microbiologica Sinica, 2014, 54(8):889-896.
[21] SAMBROOK J, RUSSELL D W.Molecular cloning:a laboratory manual[M].3rd ed.New York:Cold Spring Harbor Laboratory Press, 2001.
[22] OGAWA J, TAKEDA S, XIE S X, et al.Purification, characterization, and gene cloning of purine nucleosidase from Ochrobactrum anthropi[J].Applied and Environmental Microbiology, 2001, 67(4):1 783-1 787.
[23] ZHENG Q, WANG R, JU X, et al.Enhancement of L-ribulose production from L-ribose through modification of Ochrobactrum sp.CSL1 ribose-5-phosphate isomerase A[J].Applied Biochemistry and Biotechnology, 2022, 194(10):4 852-4 866.