传统发酵食品是以动植物为原料,经过微生物自然发酵或接入微生物制剂后得到的具有丰富营养和独特风味的一类食品[1]。发酵是生产和保存食物的有效方法,它可以通过自然途径延长食物的保存期限,增加营养成分含量,提高食品风味[2]。中国的饮食文化历史悠久,中国传统发酵食品具有几千年的历史,成为了中华饮食文化中独特的一部分。中国传统发酵食品的原料包括谷物、蔬菜、牛奶、肉类、豆类等,通过微生物的发酵作用产生了一系列产品,如白酒、黄酒、陈醋、酱油、腐乳[3]。微生物在食品的发酵中提供动力,研究发酵食品中微生物的多样性及在发酵过程中的菌群相对丰度的动态变化,探究其对发酵食品风味和质量的影响,具有重要意义[4]。目前中国一些传统的发酵食品已开始从自然发酵向接种发酵过渡,从传统发酵食品的自然发酵系统中分离筛选具有特定功能的微生物进行开发利用,此举具有广阔的应用前景[5]。然而,目前绝大多数微生物仍然处在不可培养或难培养状态,即只能使用与培养无关的技术来研究尚未培养微生物的多样性,但这些未培养的微生物可能具有目前已知的微生物尚未研究的代谢功能及代谢途径[6],同时也是巨大的未知微生物资源宝库。探究中国传统发酵食品发酵系统中的未培养微生物,挖掘其功能,有利于抑制发酵过程中有害物质的产生,也有利于开发更多有益的微生物产品应用于工业生产及日常生活[7-8]。
1 自然界中的未培养微生物
德国微生物学家Robert Koch于1881年建立了采用固体平板分离微生物的纯培养技术[6],该技术易于操作,因此成为了微生物菌株分离培养最常用的手段。微生物的纯培养为微生物的分类、生理、遗传等微生物学各个领域的发展提供了前提条件。一个多世纪之前,研究者发现总的微生物数量和可培养微生物数量之间存在差异这一现象。1985年,STALEY等[9]将其称为“平板计数异常(great plate count anomaly)”。近年来,随着测序技术和生物信息学分析的发展,鸟枪宏基因组测序、扩增子测序等高通量测序技术已成为揭示微生物群落中各物种丰度和功能的有力手段[10],但要深入挖掘菌株的代谢特性及其在发酵过程中发挥的作用,获得其纯培养物是必要的[3]。然而目前能够得到纯培养物的微生物仅占微生物总数的一小部分,未培养或难培养的微生物在微生物中仍占据大多数[11-12]。存在未培养微生物的原因主要有以下几点:(1)常规培养无法满足其生长条件。许多微生物的培养严格取决于特定的环境生长条件,依赖于自然生长环境中的各种生长因子(如维生素、氨基酸、核苷酸、无机化合物)的存在,而在常规培养条件下无法满足其自然生长环境的条件[13]。(2)微生物处在活的但不可培养(viable but non-Culturable,VBNC)状态。在自然状态下一些微生物面对恶劣的自然环境如不适宜的高/低温、pH、氧含量等会进入VBNC状态[14],在实验室培养条件下,即使将导致其进入VBNC状态的因素去除,仍然不能使微生物复苏。(3)互生依赖性。微生物在自然生长状态下可能存在相互依赖的关系,即一个微生物物种的代谢产物可能是其他微生物的生长基质,如果两种生物体相互依赖,在实验室培养条件下必须确定满足两种微生物个体的适当条件,才可能激活其在纯培养条件中沉默或表达极低水平的基因[15],但如果人为破坏两者之间的互生关系也会增加难培养微生物的分离难度。(4)寡营养条件无法满足。培养基中基质的浓度会影响特定微生物可培养性[16],大多数微生物在自然条件下处于寡营养状态,当在富营养的培养基上培养时,生长速度快抵抗力强的微生物可快速生长,营养物质被消耗,菌体密度增加,可能会抑制本身生长缓慢或抵抗力差的微生物生长。(5)低丰度和弱竞争力。部分微生物种属在自然界中以低丰度存在,使用扩增子测序等检测微生物多样性的方法无法检出,无法推测这类微生物的生长条件[17]。
2 传统发酵食品发酵系统中的未培养微生物
在中国几千年的饮食文化中,传统发酵食品占有重要地位,传统发酵技术也催生出许多具有中国特色的传统发酵食品。中国地大物博,食物原料众多,传统发酵食品的种类也丰富多彩:豆类制品如酱油、豆豉、大酱、腐乳等;酒精类饮品如白酒、黄酒、米酒等;谷物类制品如馒头、陈醋等;乳制品如酸奶、奶豆腐等以及酸菜、香肠等[18]。在传统发酵食品的发酵过程中,微生物起到关键推动作用。传统发酵食品的发酵系统多为多菌种混合发酵,微生物多样性丰富,经过周围生态环境和发酵过程的不断选择与富集,传统发酵食品的发酵系统形成了特有的微生物群落[19]。当前研究其中微生物群落多样性的方法主要有非培养依赖方法以及培养依赖方法,已有研究者使用不依赖培养的方法在发酵食品中发现了不可培养的微生物,具体统计情况如表1所示。此外,研究者在白酒窖泥、大曲等中国传统发酵食品发酵系统中成功分离得到之前尚未培养的微生物新物种(表2),实现了未培养微生物的可培养化。使用Venny在线绘图网站(https://bioinfogp.cnb.csic.es/tools/venny/index.html)对使用非培养依赖方法和培养依赖方法研究得到的白酒窖泥[20-35]、大曲[36-44]、发酵茶[45-53]发酵系统中的微生物,以及这些发酵系统中存在新物种所在属进行了总结比对,结果如图1所示。

a-窖泥;b-大曲;c-发酵茶
图1 不同方法检测得到的中国传统发酵食品发酵系统中的微生物数量及其比例
Fig.1 Number and proportion of microorganisms in Chinese traditional fermented food fermentation system detected by different methods
表1 使用非培养依赖方法对中国传统发酵食品发酵
系统中的不可培养微生物的测定
Table 1 Determination of non-culturable microorganisms in Chinese traditional fermented food fermentation system using non-culture dependent methods
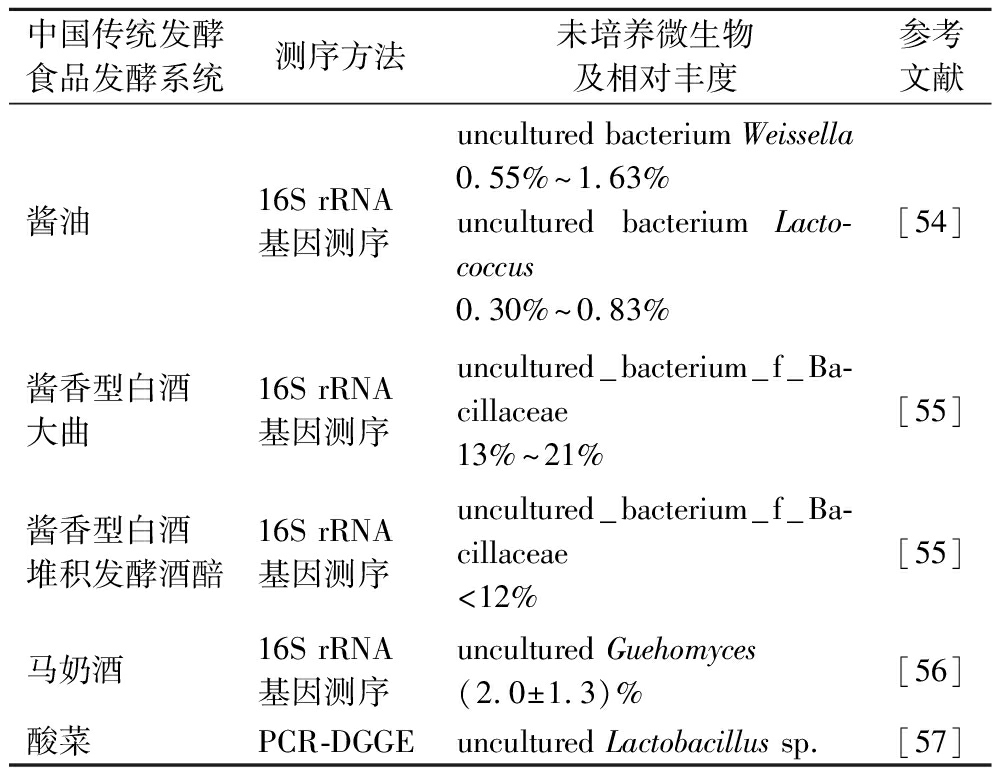
中国传统发酵食品发酵系统测序方法未培养微生物及相对丰度参考文献酱油16S rRNA基因测序uncultured bacterium Weissella0.55%~1.63%uncultured bacterium Lacto-coccus0.30%~0.83%[54]酱香型白酒大曲16S rRNA基因测序uncultured_bacterium_f_Ba-cillaceae13%~21%[55]酱香型白酒堆积发酵酒醅16S rRNA基因测序uncultured_bacterium_f_Ba-cillaceae<12%[55]马奶酒16S rRNA基因测序uncultured Guehomyces(2.0±1.3)%[56]酸菜PCR-DGGEuncultured Lactobacillus sp.[57]
表2 中国传统发酵食品发酵系统中分离到的微生物新物种
Table 2 New microbial species isolated from Chinese traditional fermented food fermentation system

中国传统发酵食品发酵系统新物种参考文献窖泥Sporosarcina beigongshangi, Aminipila luticellarii,Lysobacter zhanggongensis, Bacillus vini,Pseudoxan-thomonas beigongshangi,Clostridium swellfunianum, Clostridium fermenticellae, Blautia liquoris,Novi-syntrophococcus fermenticellae, Caproicibacterium amylolyticum,Caproicibacterium lactatifermentans[20-30]大曲Brevibacterium renqingii, Thermoflavimicrobium daqui, Franconibacter daqui, Scopulibacillus daqui[36-39]发酵茶Aeromicrobium camelliae, Pullulanibacillus camelliae,Paenibacillus yunnanensis,Pueribacillus theae, Bacillus camelliae[45-49]
不依赖培养的方法能够获得较多的微生物物种信息,但通常无法检测到一些低丰度的微生物,因此该方法并不能包含发酵系统中的全部微生物。而传统的培养方法也因为通量低或培养条件的限制存在一定缺陷,2种方法单独使用都具有一定的局限性,因此,要想全面了解发酵系统中的微生物多样性且获得菌株的纯培养物,两种方法结合或可起到相互弥补和相互验证的作用。此外,有些新物种所在的属在2种方法的统计中均未出现,说明其在发酵系统中丰度较低或难于培养,是第一次在发酵食品被成功分离或该属是发酵系统中分离得到的一个新属。发酵食品中未培养微生物的研究能够增加微生物的可培养数量,拓宽人们对微生物的认知领域,发现微生物新的代谢途径等[7-8],有利于微生物产品的进一步开发,使微生物更好地应用于人类的生产生活中。
3 传统发酵食品发酵系统中未培养微生物的研究方法
3.1 培养方法及培养基的优化
通过改善培养条件已成功分离培养出许多不可培养微生物,将其应用于传统发酵食品发酵系统不可培养微生物的分离培养具有很高的可行性。
3.1.1 富集培养
富集培养是一个复杂的混菌培养系统,研究证明富集培养可以使部分微生物的丰度增加,也能使处于休眠状态的微生物复苏,增加未培养微生物可培养的数量和种类[58]。中国传统发酵食品大多数为混菌自然发酵,微生物之间存在着互作关系[59],某些微生物也可能处于休眠或VBNC状态。因此,在纯培养之前进行富集孵育可能会使部分不可培养的微生物得以复苏。LIU等[60]设计了一套富集体系对白酒窖泥中的微生物进行富集,其富集体系DSMZ 827是根据窖泥中的一种优势菌Petrimonas sulfuriphila BN3的培养条件设计的。富集液成分在微生物培养基数据库-布伦施威格州莱布尼茨研究所(DSMZ-german)(https://www.dsmz.de/collection/catalogue)检索获得,该数据库可根据微生物种属推荐相应的培养基。该研究在富集后成功分离培养得到1株Aminobacterium属的厌氧新物种,将其与Methanosarcina barkeri (ACCC00572)在含有氨基酸或蜜二糖的培养基中共培养可促进白酒风味物质挥发性脂肪酸(volatile fatty acids,VFAs)的产生。此外有研究证明,使用速效碳源和氮源含量较少的低营养富集液对寡营养环境中的微生物进行富集培养,会促进生长较慢的寡营养菌(k-strategists)的生长以及复苏处于休眠状态的部分微生物[61]。在发酵系统中,浓香型白酒窖泥长期处于厌氧、微酸性、寡营养的环境中[62],卢萌萌[63]通过使用3种营养成分依次减少的富集液对浓香型白酒窖泥中的微生物进行富集,结果表明速效碳源和氮源浓度最低的寡营养培养液效果最好,富集得到的微生物多样性最高,而富营养培养液富集得到的微生物较为单一。该富集液是根据Clostridium acetobutylicum ATCC 824菌株的生长条件设计的[64]。在海洋微生物研究领域,在寡营养富集培养液中加入使VBNC状态微生物复苏的物质丙酮酸钠[61],已经实现了部分不可培养海洋微生物的纯培养,包括1个疑似新目,1个疑似新科,16个疑似新属和79个疑似新种,后续可将其应用于发酵系统中VBNC状态微生物的复苏。
3.1.2 改良培养基
获得微生物的纯培养物是研究微生物的生物特性及代谢功能的基础,因此,有很多学者针对微生物的生长条件对培养基进行特异性设计,以期实现更多未培养微生物的可培养化。韩颖[65]将窖泥和酒醅加水溶解粉碎离心后得到的液体代替定容的ddH2O设计培养基,更好地模拟原位环境。高凤[66]利用东北酸菜在发酵过程中会产生各种有机酸盐的特点,将实验前期在酸菜发酵过程中检测到的有机酸对应的有机酸盐作为特定碳源和氮源设计培养基,并在培养基中加入硝酸盐和亚硝酸盐,分离酸菜发酵液中起到分解代谢有害有机酸和亚硝酸盐作用的菌株,并分离得到了2株肠杆菌属新种和1株黄杆菌属新种,它们具有同时降解硝酸盐和亚硝酸盐的能力。刘文容[67]通过在常规MRS培养基中加入能使腐败微生物在常规培养基上生长的生长因子——甲瓦龙酸后,第一次在黄酒中分离出导致陈酿黄酒酸败的主要微生物。改良后的培养基更加满足难培养微生物的生长需求,可培养常规培养条件下无法生长的微生物。
3.2 培养组学
单纯不依赖培养的方法得到微生物的纯培养物具有一定难度,阻碍微生物资源的开发利用,培养依赖性方法对于研究微生物功能是必不可少的,而单一的培养条件分离微生物的效率较低,因此,培养组学应运而生。培养组学(culturomics)是LAGIER等[68]于2012年创建的培养微生物的新技术,研究者设计利用多重培养条件对微生物进行分离培养,不同培养条件的设计旨在抑制大多数种群的生长并促进低丰度或难培养微生物的生长,使用基质辅助激光解吸电离-飞行时间质谱(MALDI-TOF-MS)和16S rRNA 基因测序对分离得到的细菌进行种属的鉴定。培养组学因设计的培养条件及筛选条件多样,具有筛选低丰度微生物的潜力,在一定程度上补充了传统培养技术的不足。在传统发酵食品领域,XU等[69]通过培养组学与扩增子测序技术联用的方法对浓香型白酒窖泥中的细菌进行了培养,根据扩增子测序的结果设计适合窖泥中不同种属微生物生存的培养基和条件,培养基的选择主要包括普通商业化培养基、模拟原始环境培养基以及预测培养基,预测的培养基是通过 KOMODO(known media database)培养基推荐系统(http://komodo.modelseed.org)检索获得。通过培养组学对微生物进行高通量分离培养鉴定,共得到215个细菌纯培养物,分属于4个门41个属,其中36个属在以前的研究中未从窖泥中分离出来过,该研究还培养出19株潜在新种,显著增加了从白酒窖泥中可分离培养微生物的数量,同时该研究是第一次将培养组学应用于白酒发酵系统的微生物分离,丰富了窖泥微生物的纯培养库,对研究白酒发酵系统中的未培养微生物具有重要意义。含硫化合物3-(甲硫基)-1-丙醇是白酒中的一种风味物质, DU等[70]开发了基于多模块分工的合成微生物群落,利用培养组学的方法从白酒酒曲和酒醅中分离筛选了每个模块的核心菌株(酵母菌、芽孢杆菌、乳酸菌),以实现3-(甲硫基)-1-丙醇的高效生物合成。设计了选择培养基,分别通过加入抗生素、观察透明水解圈的产生、在培养基中加入显色物质呈现颜色变化对目标菌株进行了筛选。但同时需要注意的是,培养组学需要设置多种培养条件来满足微生物群中不同物种的偏好,且不同样品中的微生物群存在差异,因此目前存在工作量大,所需时间长等缺点。将培养组学与富集培养、高通量测序结合起来,通过选择合适的富集时间、取样后添加新鲜培养基、代表时间点取样[71],并根据高通量测序结果,针对发酵系统中不同相对丰度的微生物种属预测设计不同培养基,优化培养方案,有目的性的分离具有特定功能的菌株,并采用“有经验的”菌落选择(即每个平板挑取有代表性的菌落进行分离纯化)方法,在减小工作量的同时增加可培养微生物的数量。未来,培养组学将在获得未培养微生物的纯培养物中发挥重要作用。
3.3 原位培养
未培养微生物难培养的原因之一是原始环境复杂,人为模拟的环境以及培养条件可能仍然无法满足其对于多种营养及微量元素的需求。原位培养是将样品培养在含有模拟原始环境培养基的多个独立密闭小孔装置中,小孔上下覆盖有半透膜,半透膜可以使原始环境中的多种生长因子透过,进入小孔中为微生物生长提供营养条件。将装置放回原始环境培养,以此实现微生物生长过程中对原始环境营养物质的获取[13]。在发酵系统的应用中,熊盈盈[43]采用原位培养中的分离芯片法(isolation Chip, iChip)研究了酱香型白酒大曲中的细菌多样性,研究者选择了适合细菌生长的孔径为0.03 μm的聚碳酸酯径迹蚀刻滤膜(polycarbonate track etched,PCTE),同时,在原位培养装置小孔中的培养基中加入了酱香型白酒大曲浸出液,使培养基成分更加贴近原始环境,将组装好的装置放在窖池的糟醅中进行原位培养。与传统培养法相比,分离芯片法因为微生物的生长环境为原始生境,分离得到了更多的微生物种属,而多个小孔的设置也提高了培养微生物的通量。将原位培养装置取出后,可设计多种选择性固体培养基对其中的微生物进行分离筛选,增加可培养微生物的数量。
4 传统发酵食品发酵系统中未培养微生物的开发利用
4.1 筛选特定功能微生物进行开发利用
传统发酵食品发酵系统中的微生物资源丰富,且在发酵过程中起到重要作用,可从中筛选具有特定功能的微生物进行开发利用,也可以制备微生物制剂应用于其他生产领域。例如研究者在虾酱中分离到了耐受高盐浓度的嗜盐细菌新物种“盘锦慢生芽孢杆菌”、在浓香型白酒窖泥中发现了能够利用乳酸高效产丁酸的新物种,具体统计情况如表3所示。
表3 中国传统发酵食品发酵系统中具有特定功能的未培养微生物
Table 3 Uncultured microorganisms with specific functions in Chinese traditional fermented food fermentation system

中国传统发酵食品发酵系统菌株功能参考文献麦曲Pontibacter beigongshangensis可产生色素;诱导戊糖片球菌产生抑菌物质,降低黄酒中的生物胺含量 [72-74]虾酱Lentibacillus panjinensis嗜盐,可耐受25%盐浓度[75]窖泥Ruminococcaceae bacterium CPB6利用乳酸高产己酸[76]发酵酸奶Bacillus sp.DU-106产生L-乳酸,具有潜在的益生菌特性[77]发酵腐乳Bacillus sp.ZJ1502能够产生耐过氧化氢的碱性蛋白酶[78]
4.2 人工构建合成微生物群落
探究目前在发酵系统中已成功分离培养的未培养微生物的代谢途径和功能,分析其是否可作为核心菌株以用于人工发酵微生物群的构建。在传统发酵食品中,以白酒为例,WU等[79]指出,白酒发酵的重点是研究形成风味化合物的微生物群以及影响核心微生物群的因素,应确定与产生风味物质相关的关键微生物群,对其进行分离,而未培养微生物可培养化的实现可为构建合成微生物群落提供更多的菌株选择。YU等[80]从传统镇江香醋醋醅中获得了一个乳酸菌新物种,命名为金山醋酸乳杆菌(Acetilactobacillus jinshanensis),它是乳杆菌科(Lactobacillaceae)的新属。孙佳[81]后续分析了Acetilactobacillus jinshanensis的生长特性、功能潜力,并对其在香醋酿造中发挥的功能进行了中试实验。研究结果表明Acetilactobacillus jinshanensis可以利用培养液中的糖类物质产生乳酸、乙酸、柠檬酸,在酿造实验中,Acetilactobacillus jinshanensis在醋酸发酵的初始阶段加入,可以加快酿造体系温度提升和葡萄糖消耗的速度,缩短发酵周期。醋酸发酵终点时,醋醅的理化指标较好,部分有益挥发性化合物含量显著升高,后续将对其在谷物酿醋以及更多发酵食品中的应用进行探索。XU等[72]在北宗黄酒麦曲中分离得到了1株细菌新物种“北工商海洋杆菌”,郝淑月等[73]研究发现 “北工商海洋杆菌”具有诱导戊糖片球菌M28产生细菌素降解黄酒中生物胺的作用;李意涵等[74]进一步对该菌的色素成分进行了研究,结果表明其中含有类胡萝卜素类成分,且该色素在强酸条件和紫外线条件下稳定性强,但在强碱和高温条件下不稳定。上述研究为“北工商海洋杆菌”在黄酒降解生物胺及增色应用方面提供了理论基础。这些新物种的发现增加了可培养微生物的数量,其功能和特性可在构建合成微生物群落中发挥作用。
5 总结与展望
微生物作为中国传统发酵食品发酵过程中的强大动力,其中蕴含着许多待深入挖掘的资源[3]。在自然界中,可培养微生物比例甚微,因此,对未培养微生物的研究十分重要。当前,培养未培养微生物的方法更加多样化,如利用微生物之间的相互作用进行共培养[82]、反向基因组学[83]、绝迹稀释培养[16]等方法已经在不同领域取得了实践的成功。因此,利用原位培养、共培养等技术对发酵系统中的微生物进行分离;将培养组学与绝迹稀释相结合,绝迹稀释可使微生物总体以痕量状态生长,减少微生物对营养物质的竞争,而培养组学设计的多种培养条件也可满足更多微生物的生长条件;探究发酵系统微生物与VBNC的关系,通过添加复苏促进因子等验证并复苏发酵系统中VBNC状态的微生物,或将成为开发中国传统发酵食品发酵系统未培养微生物的重要手段。
中国传统发酵食品发酵系统中的微生物种类多样,功能复杂,含有丰富的未培养微生物。用各种技术手段对未培养微生物进行分离、研究以及开发利用,有助于研究发酵系统中的微生物多样性,了解微生物在发酵过程中与代谢物质的关系;促进中国传统发酵食品的发酵工艺向成熟化、完善化、工业化发展的同时仍然保持独特的中国风味;增加当前可培养微生物数量,为菌种的工业化开发和应用提供更多资源。
[1] 夏傲喃, 李建华, 林祥娜, 等.发酵食品微生物多样性分析方法研究进展[J].食品研究与开发, 2021, 42(4):220-224.
XIA A N, LI J H, LIN X N, et al.Research progress on analysis methods of microbial diversity in fermented foods[J].Food Research and Development, 2021, 42(4):220-224.
[2] KÅRLUND A, G MEZ-GALLEGO C, KORHONEN J, et al.Harnessing microbes for sustainable development:Food fermentation as a tool for improving the nutritional quality of alternative protein sources[J].Nutrients, 2020, 12(4):1020.
MEZ-GALLEGO C, KORHONEN J, et al.Harnessing microbes for sustainable development:Food fermentation as a tool for improving the nutritional quality of alternative protein sources[J].Nutrients, 2020, 12(4):1020.
[3] 任聪, 杜海, 徐岩.中国传统发酵食品微生物组研究进展[J].微生物学报, 2017, 57(6):885-898.
REN C, DU H, XU Y.Advances in microbiome study of traditional Chinese fermented foods[J].Acta Microbiologica Sinica, 2017, 57(6):885-898.
[4] 陈镜如, 边鑫, 杨杨, 等.中国传统发酵食品微生物多样性研究进展[J].中国调味品, 2022, 47(2):205-210.
CHEN J R, BIAN X, YANG Y, et al.Research progress on microbial diversity of traditional fermented food in China[J].China Condiment, 2022, 47(2):205-210.
[5] 张春月, 金佳杨, 邱勇隽, 等.传统与未来的碰撞:食品发酵工程技术与应用进展[J].生物技术进展, 2021, 11(4):418-429.
ZHANG C Y, JIN J Y, QIU Y J, et al.Collision between tradition and future:Technology and application progress in food fermentation engineering[J].Current Biotechnology, 2021, 11(4):418-429.
[6] 朱红惠, 黄力, 李文均, 等.未/难培养微生物:风正扬帆再起航,共辉煌[J].微生物学报, 2021, 61(4):777-780.
ZHU H H, HUANG L, LI W J, et al.Uncultivated microorganisms:It is time to refocus and reflourish to the special issue “Uncultivated Microorganisms”[J].Acta Microbiologica Sinica, 2021, 61(4):777-780.
[7] LU X W, WU Q, ZHANG Y, et al.Genomic and transcriptomic analyses of the Chinese Maotai-flavored liquor yeast MT1 revealed its unique multi-carbon co-utilization[J].BMC Genomics, 2015, 16:1064.
[8] HU X L, DU H, XU Y.Identification and quantification of the caproic acid-producing bacterium Clostridium kluyveri in the fermentation of pit mud used for Chinese strong-aroma type liquor production[J].International Journal of Food Microbiology, 2015, 214:116-122.
[9] STALEY J T, KONOPKA A.Measurement of in situ activities of nonphotosynthetic microorganisms in aquatic and terrestrial habitats[J].Annual Review of Microbiology, 1985, 39:321-346.
[10] HE G Q, LIU T J, SADIQ F A, et al.Insights into the microbial diversity and community dynamics of Chinese traditional fermented foods from using high-throughput sequencing approaches[J].Journal of Zhejiang University-SCIENCE B, 2017, 18(4):289-302.
[11] 江汶钰, 张明星, 李洁, 等.豉香型白酒酒丸、酒饼及酒醪中微生物群落多样性研究[J].中国酿造, 2021, 40(7):33-37.
JIANG W Y, ZHANG M X, LI J, et al.Microbial community diversity in Jiuwan, Jiubing and Jiulao of Chi-flavor Baijiu[J].China Brewing, 2021, 40(7):33-37.
[12] MAO P, HU Y L, LIAO T T, et al.Microbial diversity during fermentation of sweet paste, a Chinese traditional seasoning, using PCR-denaturing gradient gel electrophoresis[J].Journal of Microbiology and Biotechnology, 2017, 27(4):678-684.
[13] LEWIS W H, TAHON G, GEESINK P, et al.Innovations to culturing the uncultured microbial majority[J].Nature Reviews Microbiology, 2021, 19(4):225-240.
[14] 张晓华, 钟浩辉, 陈吉祥.细菌活的非可培养状态研究进展[J].中国海洋大学学报(自然科学版), 2020, 50(9):153-160.
ZHANG X H, ZHONG H H, CHEN J X.Research progress on viable but nonculturable state bacteria[J].Periodical of Ocean University of China, 2020, 50(9):153-160.
[15] ARORA D, GUPTA P, JAGLAN S, et al.Expanding the chemical diversity through microorganisms co-culture:Current status and outlook[J].Biotechnology Advances, 2020, 40:107521.
[16] BARTELME R P, CUSTER J M, DUPONT C L, et al.Influence of substrate concentration on the culturability of heterotrophic soil microbes isolated by high-throughput dilution-to-extinction cultivation[J].mSphere, 2020, 5(1):e00024-e00020.
[17] 熊盈盈, 莫祯妮, 邱树毅, 等.未培养环境微生物培养方法的研究进展[J].微生物学通报, 2021, 48(5):1 765-1 779.
XIONG Y Y, MO Z N, QIU S Y, et al.Research progress on culture methods of uncultured environmental microorganisms[J].Microbiology China, 2021, 48(5):1 765-1 779.
[18] 刘飞翔, 董其惠, 吴蓉, 等.不同国家和地区传统发酵食品及其发酵微生物研究进展[J].食品科学, 2020, 41(21):338-350.
LIU F X, DONG Q H, WU R, et al.Recent advances in traditional fermented foods and starter cultures for their production in different countries and regions[J].Food Science, 2020, 41(21):338-350.
[19] 陈坚, 汪超, 朱琪, 等.中国传统发酵食品研究现状及前沿应用技术展望[J].食品科学技术学报, 2021, 39(2):1-7.
CHEN J, WANG C, ZHU Q, et al.Research status and application prospect of frontier technology of traditional fermented food in China[J].Journal of Food Science and Technology, 2021, 39(2):1-7.
[20] SUN Z B, GUO L Y, YAN Y, et al.Sporosarcina beigongshangi sp.nov, isolated from pit mud of Baijiu[J].Archives of Microbiology, 2022, 204(1):10.
[21] WEI Z X, MA S C, CHEN R, et al.Aminipila luticellarii sp.nov., an anaerobic bacterium isolated from the pit mud of strong aromatic Chinese liquor, and emended description of the genus Aminipila[J].International Journal of Systematic and Evolutionary Microbiology, 2021, 71(10).DOI:10.1099/ijsem.0.004710.
[22] ZHANG X F, WANG H H, SUN X Y, et al.Lysobacter zhanggongensis sp.nov.isolated from a pit mud[J].Current Microbiology, 2017, 74(12):1 389-1 393.
[23] MA K D, CHEN X R, GUO X, et al.Bacillus vini sp.nov.isolated from alcohol fermentation pit mud[J].Archives of Microbiology, 2016, 198(6):559-564.
[24] SUN Z B, DAI F, YAN Y, et al.Pseudoxanthomonas beigongshangi sp.nov, a novel bacteria with predicted nitrite and nitrate reduce ability isolated from pit mud of Baijiu[J].Antonie Van Leeuwenhoek, 2021, 114(8):1 307-1 314.
[25] LU L F, YANG Y, CHAI L J, et al.Blautia liquoris sp.nov., isolated from the mud in a fermentation cellar used for the production of Chinese strong-flavour liquor[J].International Journal of Systematic and Evolutionary Microbiology, 2021, 71(10).DOI:10.1099/ijsem.0.005041.
[26] LIU C L, HUANG D, LIU L Y, et al.Clostridium swellfunianum sp.nov., a novel anaerobic bacterium isolated from the pit mud of Chinese Luzhou-flavor liquor production[J].Antonie Van Leeuwenhoek, 2014, 106(4):817-825.
[27] XU P X, CHAI L J, QIU T, et al.Clostridium fermenticellae sp.nov., isolated from the mud in a fermentation cellar for the production of the Chinese liquor, Baijiu[J].International Journal of Systematic and Evolutionary Microbiology, 2019, 69(3):859-865.
[28] CHAI L J, FANG G Y, XU P X, et al.Novisyntrophococcus fermenticellae Gen.nov., sp.nov., isolated from an anaerobic fermentation cellar of Chinese strong-flavour Baijiu[J].International Journal of Systematic and Evolutionary Microbiology, 2021, 71(9).DOI:10.1099/ijsem.0.004991.
[29] GU Y, ZHU X J, LIN F, et al.Caproicibacterium amylolyticum Gen.nov., sp.nov., a novel member of the family Oscillospiraceae isolated from pit clay used for making Chinese strong aroma-type liquor[J].International Journal of Systematic and Evolutionary Microbiology, 2021, 71(4).DOI:10.1099/ijsem.0.004789.
[30] WANG H L, GU Y, ZHAO D, et al.Caproicibacterium lactatifermentans sp.nov., isolated from pit clay used for the production of Chinese strong aroma-type liquor[J].International Journal of Systematic and Evolutionary Microbiology, 2022, 72(1).DOI:10.1099/ijsem.0.005206.
[31] 张应刚, 许涛, 郑蕾, 等.窖泥群落结构及功能微生物研究进展[J].微生物学通报, 2021, 48(11):4 327-4 343.
ZHANG Y G, XU T, ZHENG L, et al.Research progress on community structure and functional microorganisms of pit mud[J].Microbiology China, 2021, 48(11):4 327-4 343.
[32] 孙乐平. 扩增子测序与培养组学联用分析白酒酒糟和窖泥中微生物的多样性[D].北京:北京工商大学, 2020.
SUN L P.Culturing the Baijiu microorganism from fermentation vinasse and pit mud with culturomics and amplicon-based metagenomic approaches[D].Beijing:Beijing Technology and Business University, 2020.
[33] 邓杰, 卫春会, 边名鸿, 等.浓香型白酒不同窖龄窖池窖泥中古菌群落结构分析[J].食品科学, 2017, 38(8):37-42.
DENG J, WEI C H, BIAN M H, et al.Archaeal community analysis of pit mud from cellars of different ages for Luzhou-flavor liquor[J].Food Science, 2017, 38(8):37-42.
[34] CAI W C, XUE Y A, TANG F X, et al.The depth-depended fungal diversity and non-depth-depended aroma profiles of pit mud for strong-flavor Baijiu[J].Frontiers in Microbiology, 2022, 12:789845.
[35] ZHANG H M, MENG Y J, WANG Y L, et al.Prokaryotic communities in multidimensional bottom-pit-mud from old and young pits used for the production of Chinese strong-flavor Baijiu[J].Food Chemistry, 2020, 312:126084.
[36] YAN Y, XING X, SUN Z B, et al.Brevibacterium renqingii sp.nov, isolated from the Daqu of Baijiu[J].Archives of Microbiology, 2021, 203(5):2 291-2 296.
[37] LI D N, HUANG W, QIU S Y.Thermoflavimicrobium daqui sp.nov., a thermophilic microbe isolated from Moutai-flavour Daqu[J].International Journal of Systematic and Evolutionary Microbiology, 2019, 69(9):2 709-2 716.
[38] GAO Z Q, SU C L, YANG X Y, et al.Franconibacter daqui sp.nov., a facultatively alkaliphilic species isolated from a Daqu sample[J].International Journal of Systematic and Evolutionary Microbiology, 2017, 67(12):4 962-4 966.
[39] YAO S, ZHAI L, XIN C H, et al.Scopulibacillus daqui sp.nov., a thermophilic bacterium isolated from high temperature Daqu[J].International Journal of Systematic and Evolutionary Microbiology, 2016, 66(11):4 723-4 728.
[40] GAN S H, YANG F, SAHU S K, et al.Deciphering the composition and functional profile of the microbial communities in Chinese Moutai liquor starters[J].Frontiers in Microbiology, 2019, 10:1540.
[41] YANG Y, WANG S T, LU Z M, et al.Metagenomics unveils microbial roles involved in metabolic network of flavor development in medium-temperature Daqu starter[J].Food Research International, 2021, 140:110037.
[42] CAI W C, WANG Y R, NI H, et al.Diversity of microbiota, microbial functions, and flavor in different types of low-temperature Daqu[J].Food Research International (Ottawa, Ont.), 2021, 150(Pt A):110734.
[43] 熊盈盈. 第三代测序技术和培养法联合使用对酱香大曲细菌多样性及其功能的研究[D].贵阳:贵州大学, 2021.
XIONG Y Y.Study on bacterial diversity and function of Maotai-flavor Daqu by the combination of the third generation sequencing technology and culture method[D].Guiyang:Guizhou University, 2021.
[44] 王柏文, 吴群, 徐岩, 等.中国白酒酒曲微生物组研究进展及趋势[J].微生物学通报, 2021, 48(5):1 737-1 746.
WANG B W, WU Q, XU Y, et al.Recent advances and perspectives in study of microbiome in Chinese Jiuqu starter[J].Microbiology China, 2021, 48(5):1 737-1 746.
[45] NIU L L, XIONG M J, TANG T Y, et al.Aeromicrobium camelliae sp.nov., isolated from Pu’er tea[J].International Journal of Systematic and Evolutionary Microbiology, 2015, 65(12):4 369-4 373.
[46] NIU L L, XIONG M J, ZHU D C, et al.Pullulanibacillus camelliae sp.nov., isolated from Pu’er tea[J].International Journal of Systematic and Evolutionary Microbiology, 2016, 66(11):4 760-4 765.
[47] NIU L L, TANG T Y, MA Z L, et al.Paenibacillus yunnanensis sp.nov., isolated from Pu’er tea[J].International Journal of Systematic and Evolutionary Microbiology, 2015, 65(11):3 806-3 811.
[48] WANG D J, XIANG Y Q, JIANG C Y, et al.Pueribacillus theae Gen.nov., sp.nov., isolated from Pu’er tea[J].International Journal of Systematic and Evolutionary Microbiology, 2018, 68(9):2 878-2 882.
[49] NIU L L, XIONG M J, ZHANG J, et al.Bacillus camelliae sp.nov., isolated from Pu’er tea[J].International Journal of Systematic and Evolutionary Microbiology, 2018, 68(2):564-569.
[50] LI Q, LAI X, TENG C Q.Characterization of key aroma compounds and core functional microorganisms in different aroma types of Liupao tea[J].Food Research International, 2022, 152:110925.
[51] LI M Y, XIAO Y, ZHONG K, et al.Study on taste characteristics and microbial communities in Pingwu Fuzhuan brick tea and the correlation between microbiota composition and chemical metabolites[J].Journal of Food Science and Technology, 2022, 59(1):34-45.
[52] 王桥美, 彭文书, 杨瑞娟, 等.普洱茶发酵过程中可培养微生物的群落结构分析[J].食品与发酵工业, 2020, 46(20):88-93.
WANG Q M, PENG W S, YANG R J, et al.Community structure of culturable microbes during the fermentation of Pu-erh tea[J].Food and Fermentation Industries, 2020, 46(20):88-93.
[53] 黄紫衡, 陈欢, 黄丽, 等.六堡茶渥堆过程中可培养微生物的分离与鉴定[J/OL].食品科学, 2021:1-16.(2021-11-19).https://kns.cnki.net/kcms/detail/11.2206.TS.20211118.1117.018.html.
HUANG Z H, CHEN H, HUANG L, et al.Isolation and identification of culturable microbes during the fermentation of Liupao tea[J/OL].Food Science, 2021:1-16.(2021-11-19).https://kns.cnki.net/kcms/detail/11.2206.TS.20211118.1117.018.html.
[54] LIU X Y, QIAN M, SHEN Y X, et al.An high-throughput sequencing approach to the preliminary analysis of bacterial communities associated with changes in amino acid nitrogen, organic acid and reducing sugar contents during soy sauce fermentation[J].Food Chemistry, 2021, 349:129131.
[55] 赵才贤. 酱香型白酒大曲和酒醅的微物群落结构特征及其变化规律研究[D].贵阳:贵州大学, 2021.
ZHAO C X.Study on the structure characteristics and variation for microbial community in Daqu and fermented grains of Jiang-flavour Chinese spirits production[D].Guiyang:Guizhou University, 2021.
[56] TANG H, MA H M, HOU Q C, et al.Profiling of koumiss microbiota and organic acids and their effects on koumiss taste[J].BMC Microbiology, 2020, 20(1):85.
[57] LIU Z G, LI J Y, ZHOU X W, et al.The lactic acid bacteria and yeast community of home-made sauerkraut from three provinces in Southwest China[J].Archives of Microbiology, 2021, 203(6):3 171-3 182.
[58] ZHI Y, WU Q, XU Y.Genome and transcriptome analysis of surfactin biosynthesis in Bacillus amyloliquefaciens MT45[J].Scientific Reports, 2017, 7(1):1-13.
[59] 魏军林, 聂尧, 杜海, 等.营养相互作用对传统发酵食品微生物群落构建的推动作用研究进展[J].微生物学通报, 2021, 48(11):4 412-4 424.
WEI J L, NIE Y, DU H, et al.How trophic interactions drive the spontaneous construction of microbial community in traditional fermented foods:A review[J].Microbiology China, 2021, 48(11):4 412-4 424.
[60] LIU C L, HUANG D, ZHANG W X.Combining culture-dependent and culture-independent molecular methods for the isolation and purification of a potentially novel anaerobic species from pit mud in a Chinese liquor distillery[J].Journal of the Institute of Brewing, 2016, 122(4):754-762.
[61] MU D S, LIANG Q Y, WANG X M, et al.Metatranscriptomic and comparative genomic insights into resuscitation mechanisms during enrichment culturing[J].Microbiome, 2018, 6(1):230.
[62] GAO J J, LIU G Y, LI A J, et al.Domination of pit mud microbes in the formation of diverse flavour compounds during Chinese strong aroma-type Baijiu fermentation[J].LWT, 2021, 137:110442.
[63] 卢萌萌. 浓香型白酒窖泥中未培养厌氧菌的可培养化研究[D].无锡:江南大学, 2020.
LU M M.Cultivating uncultured anaerobes from pit mud of Chinese strong-aroma type liquor[D].Wuxi:Jiangnan University, 2020.
[64] DUSSÉAUX S, CROUX C, SOUCAILLE P, et al.Metabolic engineering of Clostridium acetobutylicum ATCC 824 for the high-yield production of a biofuel composed of an isopropanol/butanol/ethanol mixture[J].Metabolic Engineering, 2013, 18:1-8.
[65] 韩颖. 古井窖泥菌群中厌氧菌的纯化培养与分子鉴定[D].哈尔滨:哈尔滨工业大学, 2017.
HAN Y.Purification and molecular identification of anaerobic bacteria in Gujing tribute pit mud[D].Harbin:Harbin Institute of Technology, 2017.
[66] 高凤. 东北酸菜发酵过程亚硝酸盐微生物酶降解机制及应用[D].镇江:江苏大学, 2019.
GAO F.Mechanism and application of nitrite enzymatic degradation by microorganisms in saukraut in northeast China[D].Zhenjiang:Jiangsu University, 2019.
[67] 刘文容. 黄酒陈酿过程中酸败乳酸菌的分离鉴定及其特性研究[D].无锡:江南大学, 2017.
LIU W R.Studies on the isolation, identification and characteristics of spoilage lactic acid bacteria in storage of Chinese rice wine[D].Wuxi:Jiangnan University, 2017.
[68] LAGIER J C, DUBOURG G, MILLION M, et al.Culturing the human microbiota and culturomics[J].Nature Reviews Microbiology, 2018, 16(9):540-550.
[69] XU J L, SUN L P, XING X, et al.Culturing bacteria from fermentation pit muds of Baijiu with culturomics and amplicon-based metagenomic approaches[J].Frontiers in Microbiology, 2020, 11:1223.
[70] DU R B, LIU J, JIANG J, et al.Construction of a synthetic microbial community for the biosynthesis of volatile sulfur compound by multi-module division of labor[J].Food Chemistry, 2021, 347:129036.
[71] CHANG Y X, HOU F Y, PAN Z Y, et al.Optimization of culturomics strategy in human fecal samples[J].Frontiers in Microbiology, 2019, 10:2891.
[72] XU J L, SUN L P, SUN Z B, et al.Pontibacter beigongshangensis sp.nov, isolated from the mash of wine[J].Current Microbiology, 2019, 76(12):1 525-1 530.
[73] 郝淑月, 任清.北工商海洋杆菌诱导戊糖片球菌产生细菌素及降黄酒生物胺功效研究[J].食品科学技术学报, 2021, 39(1):88-95.
HAO S Y, REN Q.Study on bacteriocin production of Pediococcus pentosaceus induced by Pontibacter beigongshangensis and reduction the biogenic amines content in Huangjiu[J].Journal of Food Science and Technology, 2021, 39(1):88-95.
[74] 李意涵, 任清, 徐嘉良, 等.北工商海洋杆菌色素的提取与稳定性的初步研究[J].食品科技, 2021, 46(7):7-13.
LI Y H, REN Q, XU J L, et al.Extraction and stability of the Pontibacter beigongshangensis pigment[J].Food Science and Technology, 2021, 46(7):7-13.
[75] LI K X, SANG X, ZHU Y L, et al.Lentibacillus panjinensis sp.nov, isolated from shrimp paste, a traditional Chinese fermented seafood[J].Current Microbiology, 2020, 77(9):1 997-2 001.
[76] ZHU X Y, ZHOU Y, WANG Y, et al.Production of high-concentration n-caproic acid from lactate through fermentation using a newly isolated Ruminococcaceae bacterium CPB6[J].Biotechnology for Biofuels, 2017, 10:102.
[77] LI P, TIAN W N, JIANG Z, et al.Genomic characterization and probiotic potency of Bacillus sp.DU-106, a highly effective producer of L-lactic acid isolated from fermented yogurt[J].Frontiers in Microbiology, 2018, 9:2216.
[78] YU P, HUANG X X, REN Q, et al.Purification and characterization of a H2O2-tolerant alkaline protease from Bacillus sp.ZJ1502, a newly isolated strain from fermented bean curd[J].Food Chemistry, 2019, 274:510-517.
[79] WU Q, ZHU Y, FANG C, et al.Can we control microbiota in spontaneous food fermentation? -Chinese liquor as a case example[J].Trends in Food Science &Technology, 2021, 110:321-331.
[80] YU Y J, LI X, ZHANG J H, et al.Lactobacillus jinshani sp.nov, isolated from solid-state vinegar culture of Zhenjiang aromatic vinegar[J].Antonie Van Leeuwenhoek, 2020, 113(1):43-54.
[81] 孙佳. 金山醋酸乳杆菌比较基因组分析与酿醋功能评价[D].无锡:江南大学, 2021.
SUN J.Comparative genome analysis of Acetilactobacillus jinshanensis and its function in vinegar fermentation[D].Wuxi:Jiangnan University, 2021.
[82] PARK B J, PARK S J, YOON D N, et al.Cultivation of autotrophic ammonia-oxidizing Archaea from marine sediments in coculture with sulfur-oxidizing bacteria[J].Applied and Environmental Microbiology, 2010, 76(22):7 575-7 587.
[83] CROSS K L, CAMPBELL J H, BALACHANDRAN M, et al.Targeted isolation and cultivation of uncultivated bacteria by reverse genomics[J].Nature Biotechnology, 2019, 37(11):1 314-1 321.